一种烟叶醇化过程中微生物群落演替的分析方法与流程
本发明属于环境微生物
技术领域:
,具体涉及一种烟叶醇化过程中微生物群落演替的分析方法。
背景技术:
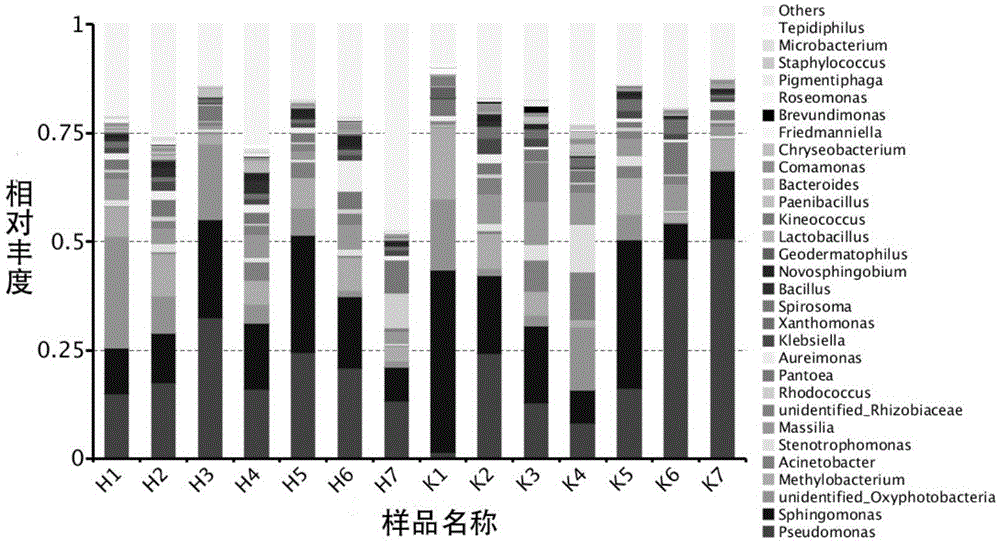
:烟叶原料在刚收获后,内在品质存在着不同程度的缺陷,如生青味重、刺激性大、烟气粗糙、烟香味被杂气掩盖,另外还会存在苦、辣、涩等不良感受,不能直接用于制造卷烟,必须进行适当的醇化处理。烟叶发酵方法主要有人工醇化、自然醇化两种。自然醇化是一种缓慢的自然发酵过程。人工醇化是指人为的控制温度和相对湿度,加速烟叶上微生物的生长变化、酶促反应及其他物质与烟草之间的化学变化,促使烟叶品质在短时间内得到改善。自然醇化工艺简单,操作方便,发酵后烟叶色泽鲜艳,吸食品质好,但耗时长,需要占用大量空间,占压资金多、生产成本高,并且由于存放时间过长,烟叶容易霉变生虫,造成烟叶损失;目前人工醇化技术,大多仅是涉及到人为控制温度和湿度,对醇化相关的菌株的应用极其匮乏,效果也不尽理想。其主要原因是,烟叶醇化过程是一个微生物演替生长的过程,目前的研究多集中在对醇化过程中的微生物分离和生物酶制剂对醇化的作用上,自然醇化的烟叶作为一个微生物宝库,微生物群落多样性及丰富度较高,并在香气物质的产生和有毒有害物质的降解方面具有极大的优势。但目前缺乏对烟叶醇化过程中微生物菌群结构及动态变化方面的研究和认知。技术实现要素:本发明为明确烟叶醇化过程中微生物种群结构和其动态变化以及微生物基因功能预测,提供了一种烟叶醇化过程中微生物群落演替的分析方法,包括如下步骤:1)取样:将自然醇化的烟叶样品,从醇化开始,间隔相同时间进行取样;2)构建基因文库:提取每个时间段采集的样品的基因组dna,分别构建16srrna基因文库和内转录间区基因文库;3)分别对步骤2)中构建的基因文库数据进行分析,得出烟叶醇化过程中微生物群落演替规律。进一步地限定,步骤1)所述自然醇化的烟叶包括自然醇化的烤烟烟叶以及自然醇化的雪茄烟叶。进一步地限定,步骤1)所述取样,是指自然醇化2-3年的烟叶从醇化开始每隔2~3个月取一次样。进一步地限定,步骤2)所述16srrna基因文库构建所用的上游引物为799f,其序列如seqidno.1所示,下游引物为1193r,其序列如seqidno:2所示。进一步地限定,步骤2)所述内转录间区基因文库构建所用的上游引物为its1-1f-f,其序列如seqidno.3所示,下游引物为its1-1f-r,其序列如seqidno:4所示。进一步地限定,步骤3)所述分析包括自然醇化的烟叶中细菌和真菌的群落组成分析以及基因功能分析。进一步地限定,步骤3)所述分析是指先利用qiime软件对基因文库中序列进行提取后,利用uclust将序列分类,再利用usearch版本8.0鉴定和去除嵌合序列,16srrna基因使用uclustsliva数据库进行比对,进行物种注释,内转录间区基因使用unite数据库进行比对,进行物种注释,最后细菌利用picrust预测基因的功能,真菌使用funguild预测基因的功能。本发明提供的烟叶醇化过程中微生物群落演替的分析方法得出的演替规律可用于人工醇化烟草中以及建立微生物促烟叶醇化菌库。有益效果1)通过本发明所述的分析方法获得的烟叶醇化过程中微生物群落演替规律,可用于指导人工醇化,提高烟叶品质,降低发酵成本。2)通过本发明所述的分析方法获得的烟叶醇化过程中微生物群落演替规律,可获得烟叶醇化过程中微生物的群落组成和优势种群,建立微生物菌库,不仅可用于人工醇化,还可用于降解有害物质以及产香微生物的研究和应用。3)通过本发明所述的分析方法获得的烟叶醇化过程中微生物群落演替规律,所得数据可通过生物信息学分析获得微生物的来源,为优质烟叶品种的寻找以及优质烟叶种植条件的优化提供理论研究基础,可靠实用,应用前景广阔。4)本发明可通过基因功能预测获得烟叶醇化过程中微生物的群落功能信息,不仅可以准确的明晰烟叶醇化过程中微生物群落与环境因子间的密切关系,为微生物的应用方向提供理论指导,还为研究解决微生物种群结果以及动态变化研究问题提供了实用便捷的路径。附图说明图1为本发明获得的烟叶醇化过程中细菌的群落组成图,横坐标为样品编号,纵坐标为属水平上,各种属的相对含量,总量为100%。图2为本发明获得的烟叶醇化过程中真菌的群落组成图,横坐标为样品编号,纵坐标为属水平上,各种属的相对含量,总量为100%。图3为本发明获得的烟叶醇化过程中细菌的群落功能差异图,横坐标为样品编号。图4为本发明获得的烟叶醇化过程中真菌的群落功能差异图,横坐标为样品编号,其中rs1代表红大样品组,共计7个样品,rs2代表k326样品组,共计7个样品。具体实施方式本发明采用自然醇化的烟叶,定期取样,并提取其基因组dna,构建基因文库,通过对所构建文库进行分析比较,明确烟叶醇化过程中微生物的群落结构、优势类群、演替规律及基因功能。下面结合实施例对本发明作进一步的详细描述。本领域技术人员将会理解,下列实施例仅用于说明本发明,而不应视为限定本发明的范围。实施例中未注明具体技术或条件者,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用材料或仪器未注明生产厂商者,均为可以通过购买获得的常规产品。其中基因组dna的提取试剂盒购买自美国莫比奥公司。nanodrop-1000分光光度计购买自thermoscientific公司。实施例1.烟叶醇化过程中微生物群落演替的分析方法。1)取样:将自然醇化的烟叶样品,从醇化开始,间隔相同时间进行取样。本实施例中烟叶样品采用云南的红大和k326烟叶品种,从自然醇化开始至醇化结束共计24个月,每隔3个月取一次样品,共计14个样品。样品收集采用随机抽样的原则,按照取样顺序,标记样品编号依次为:h1、h2、h3、h4、h5、h6、h7、k1、k2、k3、k4、k5、k6、k7,其中h1~h7均为云南的红大烟叶样品,k1~k7均为k326烟叶样品。2)构建基因文库:提取每个时间段采集的样品的基因组dna,分别构建16srrna基因文库和内转录间区(its)基因文库。具体方法如下:称取一定量烟叶样品,按照厂家说明书,使用dna提取试剂盒进行基因组dna的提取,样品dna的质量和浓度采用nanodrop-1000分光光度计测定,样品dna的完整性通过1%琼脂糖凝胶电泳的方法检测。之后细菌利用引物799f-1193r扩增16srrna位于原核细胞核糖体小亚基上v5、v6和v7高变区域,真菌利用its1引物进行pcr扩增。扩增子经纯化后,在ions5xl测序平台上进行测序,分别构建获得细菌16srrna基因文库以及its基因文库。结果如下表所示。表1细菌16srrna基因文库以及its基因文库构建所产生的数据样品编号细菌reads(个)细菌otus(个)真菌reads(个)真菌otus(个)h16354518280200257h26447320363025219h39311217987743210h46488219680032271h55528220980094237h65101017380190247h78723515480159257k17193216780177182k25356115880112259k38161922280115220k48020323680297208k58022520080136237k66091214580211202k76735719780077217所述细菌16srrna基因文库建立所用的引物序列分别为:799f:aacmggattagataccckg(seqidno.1)1193r:acgtcatccccaccttc(seqidno.2)所述真菌its基因文库建立所用的引物序列分别为:its1-1f-f:cttggtcatttagaggaagtaa(seqidno.3)。its1-1f-r:gctgcgttcttcatcgatgc(seqidno.4)。pcr扩增体系(30μl):pcr扩增程序:98℃预变性1min;30个循环包括(98℃,10s;50℃,30s;72℃;30s);72℃,5min。3)分别对步骤2)中构建的基因文库数据进行分析,得出烟叶醇化过程中微生物群落演替规律。首先使用qiime软件包进行高质量序列的提取。基于序列长度,质量,引物和标签选择原始序列,并去除低质量序列。使用uclust将独特序列集分类(具有97%同一性阈值的操作分类单位(otu))。使用usearch(版本8.0)鉴定和去除嵌合序列。16srrna基因使用sliva数据库进行比对,进行物种注释。its基因使用unite数据库进行比对,进行物种注释。最后细菌使用picrust预测基因的功能,真菌使用funguild预测基因的功能。数据分析结果:根据生物信息学分析结果,将不同样品中微生物群落组成,以及优势菌种作成柱状图,图1,图2分别为样品中细菌和真菌的群落组成,图中不同的柱形代表不同样品的微生物物种在属水平上的含量,不同颜色代表不同的属,从图中可以看出,不同醇化期中,同一属的微生物的相对含量存在明显差异,处于演替状态,此外不同烟叶表面的优势微生物种群也存在差异,这表明,微生物在特定烟叶香气的形成中也具有一定的作用。在醇化初期,红大烟叶表面真菌优势菌群为枝孢菌属(cladosporium)、茎点霉属(phoma)以及附球菌属(epicoccum),随着时间的变化,symmetrospora菌属成为最主要的微生物菌群。然和,样品k326烟叶表面真菌优势菌群为链格孢属(alternaria)symmetrospora、枝孢菌属(cladosporium),但随着时间的变化,boeremia、symmetrospora、茎点霉属(phoma)、附球菌属(epicoccum)逐渐成为主要的菌群。而在醇化初期,红大烟叶表面细菌优势菌群为假单胞菌属(pseudomonas)、甲基杆菌属(methylobacterium)、寡养单胞菌属(stenotrophomonas),随着时间的变化,假单胞菌属(pseudomonas)、红球菌属(rhodococcus)、泛菌属(pantoea)菌属成为最主要的微生物菌群。然而,样品k326烟叶表面细菌优势菌群为鞘氨醇菌属(sphingomonas)、甲基杆菌属(methylobacterium)、马西利亚菌属(massilia),但随着时间的变化假单胞菌属(pseudomonas)、鞘氨醇菌属(sphingomonas)、甲基杆菌属(methylobacterium)逐渐成为主要的菌群。根据picrust基因功能预测细菌基因功能结果,做热图(图3),根据funguild基因功能预测真菌基因功能结果,做热图(图4),不同颜色深度代表功能基因的含量,并从颜色可直观分析基因功能间含量的差异。最终得出微生物的演替规律。发现烟叶醇化过程中微生物处于此消彼长的变化状态,促成优质烟叶的形成。综上所述,本发明通过对烟叶醇化过程中微生物群落演替的分析,了解烟叶发酵小生境中微生物的演替规律,明确各类微生物在不同发酵阶段的比例,获得不同阶段优势微生物的种类,并对相关基因功能进行了预测。清楚的认识烟叶醇化过程中微生物种群结构和其动态变化以及烟草醇化过程中优势微生物对烟草醇化的贡献度。从而为烟叶醇化过程中的关键微生物的研究应用,建立了微生物菌库,为进一步实现高效人工醇化奠定一定的基础。sequencelisting<110>中国科学院青岛生物能源与过程研究所<120>一种烟叶醇化过程中微生物群落演替的分析方法<130><160>4<170>patentinversion3.5<210>1<211>19<212>dna<213>799f<400>1aacmggattagataccckg19<210>2<211>17<212>dna<213>1193r<400>2acgtcatccccaccttc17<210>3<211>22<212>dna<213>its1-1f-f<400>3cttggtcatttagaggaagtaa22<210>4<211>20<212>dna<213>its1-1f-r<400>4gctgcgttcttcatcgatgc20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1