基于KCNQ1的心源性猝死相关SNP检测试剂盒及检测方法与流程
本发明涉及基因检测及其应用领域,特别是涉及一种心源性猝死相关kcnq1基因的检测试剂盒。
背景技术:
心源性猝死(suddencardiacdeath,scd)可见于以下情况:1)、在原有器质性心脏病(如冠心病或心肌缺血)的基础上导致的猝死;2)、隐匿性离子通道类疾病或遗传因素等等。随着dna测序技术和snp突变检测技术的发展,突变位点的检测能力有了很大的提高,但是还没有专门针对scd相关基因kcmq1的snp筛查试剂盒。
检测基因突变常用技术可大致分为两种策略:1)、间接检测。即蛋白层次的检测,主要借助蛋白分析技术,分析突变体产生的变异蛋白,以及由变异蛋白引起的性状或功能的改变。2)、直接检测。主要指遗传物质的检测,如单链构象异构多态分析技术(sscp)、变性高效液相色谱分析(dhplc)、测序分析(主要是一代和二代测序)、高分辨率熔解曲线(hrm)等。
上述检测方法各有其不足之处。
1、单链构象异构多态分析技术(sscp):sscp技术是在检测突变领域中应用较为广泛的一种方法,其基本原理是基于dna在某种非变性环境中二级构象的差异,通过电泳迁移率区分正常链和异常链。缺点是这种技术的突变检出率只有50~80%,且不能准确找到突变的碱基类型和位置。
2、变性高效液相色谱分析(dhplc):dhplc技术代表了突变检测手段的新进展。该技术通过自动化程序式操作即可完成对外显子大小片段的全部检测,缺点是只可检测出突变与否,对突变的类型无法准确预测。另外,该方法对实验室要求较高,价格贵,不能普遍应用于广泛的法医实验室。
3、一代测序(sanger测序)分析:一代测序读长较长,准确性高,是法医学分析小样本dna的主要方法。其主要缺点一是测序成本较高,不能满足一部分法医实验室的需求;二是通量较低,虽然可以准确分析出基因组的序列,但是不能检测出大片段缺失或拷贝数变异等基因突变的类型,因此对于一些与此相关的遗传性疾病,还不能做出基因学诊断。
4、二代dna测序技术:该技术可以通过软件等链接获得每一个检测的片段,从而合成分析的序列。但该技术有以下缺点:1)、仪器和试剂等造成方法昂贵,且需要专业人员分析,难以普及;2)、较snp分析而言,该方法可以得到一条dna链上的所有信息,多态性大大提高,但有一定的错误率,对于法医学分析而言,尚需仔细评估。
5、高分辨率熔解曲线检测:是国际上发展的一种新型检测单核苷酸多态性及扫描突变新技术。该方法通过单核苷酸熔解温度不同而形成不同形态熔解曲线,从而进行突变基因分析。该方法的缺点是设备成本昂贵、软件分析复杂、重复性差、分析范围狭窄等。
技术实现要素:
本发明的目的是克服现有技术缺陷,提供一种基于kcnq1的心源性猝死相关snp检测试剂盒。
提供所述基于kcnq1的心源性猝死相关snp的检测方法,是本发明的另一发明目的。
本发明所述基于kcnq1的心源性猝死相关snp检测试剂盒包括了seqidno.1~32所示的16对特异性多重pcr引物对,以及seqidno.33~83所示的51个snp的snapshot单碱基延伸引物,用于检测以下51个snp分子标记中的任意一个或一个以上:
rs120074195、rs199472700、rs12720459、rs1800170、rs199472688、rs1085307580、rs199472694、rs199473662、rs75813654、rs755789171、rs533669726、rs760047145、rs120074187、rs120074182、rs199472751、rs199472765、rs199473405、rs775362401、rs1805118、rs199472768、rs12720457、rs199472779、rs12720449、rs199473476、rs199472811、rs199472815、rs199472814、rs120074189、rs606231414、rs199472783、rs17215465、rs1057128、rs199472728、rs199472806、rs199473482、rs199472817、rs17215500、rs199472788、rs397508097、rs139042529、rs199472704、rs139893266、rs397508102、rs1800172、rs34150427、rs201698592、rs11601907、rs397508108、rs370435862、rs199472677、rs199472678。
其中,所述seqidno.1~32所示的16对特异性多重pcr引物对通过扩增能够同时得到包含不同snp的扩增子,每段扩增子至少含有1个snp,最多含有6个snp。
更具体地,本发明所述的16对特异性多重pcr引物对分别用于对应扩增各自所述的snp分子标记。
rs397508108、rs370435862、rs199472677、rs199472678的pcr引物对为序列seqidno.1所示的上游引物和seqidno.2所示的下游引物。
rs1800170、rs199472688的pcr引物对为序列seqidno.3所示的上游引物和seqidno.4所示的下游引物。
rs1085307580、rs139042529、rs199472694、rs199473662、rs606231414、rs199472700的pcr引物对为序列seqidno.5所示的上游引物和seqidno.6所示的下游引物。
rs75813654、rs199472704、rs755789171的pcr引物对为序列seqidno.7所示的上游引物和seqidno.8所示的下游引物。
rs533669726的pcr引物对为序列seqidno.9所示的上游引物和seqidno.10所示的下游引物。
rs199472728、rs760047145、rs120074187、rs120074195的pcr引物对为序列seqidno.11所示的上游引物和seqidno.12所示的下游引物。
rs120074182、rs199472751、12720459的pcr引物对为序列seqidno.13所示的上游引物和seqidno.14所示的下游引物。
rs199472765、rs199473405、rs775362401、rs1805118的pcr引物对为序列seqidno.15所示的上游引物和seqidno.16所示的下游引物。
rs199472768、rs12720457的pcr引物对为序列seqidno.17所示的上游引物和seqidno.18所示的下游引物。
rs199472779、rs12720449、rs199473476、rs199472783的pcr引物对为序列seqidno.19所示的上游引物和seqidno.20所示的下游引物。
rs17215465的pcr引物对为序列seqidno.21所示的上游引物和seqidno.22所示的下游引物。
rs17215500、rs199472788、rs397508097的pcr引物对为序列seqidno.23所示的上游引物和seqidno.24所示的下游引物。
rs1057128的pcr引物对为序列seqidno.25所示的上游引物和seqidno.26所示的下游引物。
rs199472806的pcr引物对为序列seqidno.27所示的上游引物和seqidno.28所示的下游引物。
rs199473482、rs199472811、rs120074189、rs199472814、rs199472815、rs199472817的pcr引物对为序列seqidno.29所示的上游引物和seqidno.30所示的下游引物。
rs139893266、rs397508102、rs1800172、rs34150427、rs201698592、rs11601907的pcr引物对为序列seqidno.31所示的上游引物和seqidno.32所示的下游引物。
进而,所述seqidno.33~83所示的snp的snapshot单碱基延伸引物分别对应以下各自的snp分子标记。
rs120074195的延伸引物序列为seqidno.33;rs199472700的延伸引物序列为seqidno.34;rs12720459的延伸引物序列为seqidno.35;rs1800170的延伸引物序列为seqidno.36;rs199472688的延伸引物序列为seqidno.37;rs1085307580的延伸引物序列为seqidno.38;rs199472694的延伸引物序列为seqidno.39;rs199473662的延伸引物序列为seqidno.40;rs75813654的延伸引物序列为seqidno.41;rs755789171的延伸引物序列为seqidno.42;rs533669726的延伸引物序列为seqidno.43;rs760047145的延伸引物序列为seqidno.44;rs120074187的延伸引物序列为seqidno.45;rs120074182的延伸引物序列为seqidno.46;rs199472751的延伸引物序列为seqidno.47;rs199472765的延伸引物序列为seqidno.48;rs199473405的延伸引物序列为seqidno.49;rs775362401的延伸引物序列为seqidno.50;rs1805118的延伸引物序列为seqidno.51;rs199472768的延伸引物序列为seqidno.52;rs12720457的延伸引物序列为seqidno.53;rs199472779的延伸引物序列为seqidno.54;rs12720449的延伸引物序列为seqidno.55;rs199473476的延伸引物序列为seqidno.56;rs199472811的延伸引物序列为seqidno.57;rs199472815的延伸引物序列为seqidno.58;rs199472814的延伸引物序列为seqidno.59;rs120074189的延伸引物序列为seqidno.60;rs606231414的延伸引物序列为seqidno.61;rs199472783的延伸引物序列为seqidno.62;rs17215465的延伸引物序列为seqidno.63;rs1057128的延伸引物序列为seqidno.64;rs199472728的延伸引物序列为seqidno.65;rs199472806的延伸引物序列为seqidno.66;rs199473482的延伸引物序列为seqidno.67;rs199472817的延伸引物序列为seqidno.68;rs17215500的延伸引物序列为seqidno.69;rs199472788的延伸引物序列为seqidno.70;rs397508097的延伸引物序列为seqidno.71;rs139042529的延伸引物序列为seqidno.72;rs199472704的延伸引物序列为seqidno.73;rs139893266的延伸引物序列为seqidno.74;rs397508102的延伸引物序列为seqidno.75;rs1800172的延伸引物序列为seqidno.76;rs34150427的延伸引物序列为seqidno.77;rs201698592的延伸引物序列为seqidno.78;rs11601907的延伸引物序列为seqidno.79;rs397508108的延伸引物序列为seqidno.80;rs370435862的延伸引物序列为seqidno.81;rs199472677的延伸引物序列为seqidno.82;rs199472678的延伸引物序列为seqidno.83。
本发明所述检测试剂盒中还包括了pcr反应常用的酶和试剂。
进一步地,本发明还提供了一种基于kcnq1的心源性猝死相关snp的检测方法,包括以下步骤。
1、以样本提取的基因组dna为模板,通过选自所述seqidno.1~32所示的16对特异性多重pcr引物对,制备multiplexpcr产物,并进行纯化处理。
2、针对所述multiplexpcr产物,通过选自所述seqidno.33~83所示的snp的snapshot单碱基延伸引物,进行单碱基延伸(sbe)反应制备snapshot单碱基延伸产物,并进行纯化处理。
3、对所述snapshot单碱基延伸产物进行毛细管电泳,并进行结果分析。
更进一步地,所述检测方法还包括对检测体系进行灵敏度检测。
本发明检测方法所述的样本包括健康个体和尸检结果为非心源性猝死的甲醛固定组织构成的阴性对照样本,以及尸检结果为心源性猝死的甲醛固定组织构成的心源性猝死样本。
从所述样本中提取基因组dna的步骤为:采用omega全血基因组dna试剂盒提取人体静脉血基因组dna,采用qiagen血液组织dna提取试剂盒提取甲醛固定组织基因组dna,将提取的基因组dna特异性结合到硅胶膜上,洗涤去除pcr抑制剂,使用水或试剂盒中的缓冲液将结合在离心柱上的纯核酸洗脱收集,以光密度值法定量分析提取的基因组dna,确定基因组dna的含量和浓度。
本发明提供的检测试剂盒可以一次性检测kcnq1基因上的51个心源性猝死(scd)相关snp,并得到清晰结论。本发明检测方法不再利用二代测序技术和全外显子测序进行比对,不需要特殊仪器和手段筛查心源性猝死相关的基因遗传背景。
附图说明
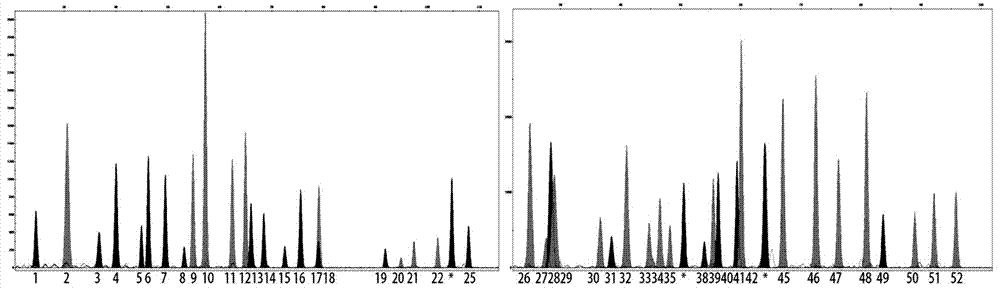
图1是kcnq1基因51个snp分子标记的分型图。
图2是p448r突变位点的检测与确证。
图3是g643s突变位点的检测与确证。
具体实施方式
以下结合具体实施例对本发明技术方案进行进一步的说明。
snp分析技术有很多种。由于单个snp的突变类型较多,因此,很少用直接测序或者dhplc检测突变。对于调查kcnq1相关snp的突变,目前国外已有相应试剂盒,用于分析临床上与其相关的遗传性疾病和离子通道型疾病,如longqtsyndrome(lqts),致命性心律失常,心房纤维颤动(atrialfibrillation)等,但未见法医学中对scd遗传背景和遗传基础的分析。
本方法采用多重pcr技术和snapshot微测序技术相结合的方法,以解决位于kcnq1基因上scd相关的51个snp的检测问题。
首先选取出国内外已报道过的,确定在kcnq1基因上与scd关联的51个snp。至此,本实施例的检测试剂盒一共涵盖了位于kcnq1基因上51个scd相关的snp,以检测kcnq1基因与山西scd人群的联系。
1)、对含有snp位点的每一对pcr引物进行单独pcr扩增(共16对引物),以验证每个扩增产物的特异性。其中,每对上下游引物之间最少含有1个待测snp,最多含有6个待测snp(见表2)。
2)、对复合扩增产物所包含的snp,分两个体系进行检测(体系1st含有24个snp,体系2nd含有27个snp),其原理结合了snapshot微测序技术和毛细管电泳(ce)技术检测突变碱基的类型和位置,从而确定样本在kcnq1上51个snp的分型。
本实施例提供了一种基于kcnq1的心源性猝死相关snp检测试剂盒,其中用于检测的snp选自以下表1所示snp分子标记中一个或一个以上。表1中同时列出了所述snp分子标记的突变类型。
本实施例的试剂盒中包含了针对上述表1所列snp位点的16对特异性多重pcr引物对,该16对多重pcr引物对具体列于表2中,通过扩增能够同时得到包含不同snp的扩增子,每段扩增子至少含有1个snp,最多含有6个snp。
进而,在本实施例的试剂盒中还包含了表3所述的snp分子标记的snapshot单碱基延伸引物。
本实施例利用上述基因检测试剂盒检测基于kcnq1的心源性猝死相关snp的变异情况。
1、以样本提取的基因组dna为模板,通过上述16对特异性多重pcr引物对制备multiplexpcr产物,并进行纯化处理。
2、通过上述单碱基延伸引物,对multiplexpcr产物进行snapshot单碱基延伸(sbe)反应,并进行纯化处理。
3、对snapshot单碱基延伸产物进行毛细管电泳,并进行结果分析。
同时,还包括对检测体系进行灵敏度检测。
所述样本包括由健康个体以及尸检结果为非心源性猝死的甲醛固定组织构成的对照组、尸检结果为心源性猝死的甲醛固定组织构成的心源性猝死组。
本实施例共采样120例,分为两组。1)、采用50例山西健康个体(男女不限)及10例尸检结果为交通事故、颅脑损伤、中毒、溺死等的甲醛固定组织样本,共同作为本实验的对照组;2)、采用60例尸检结果为心源性猝死的甲醛固定组织样本,作为心源性猝死(scd)组。
从样本中提取基因组dna的步骤为:采用omega全血基因组dna试剂盒提取人体静脉血基因组dna,用qiagen血液组织dna提取试剂盒提取甲醛固定组织基因组dna,将样品中的基因组dna特异性结合到硅胶膜上,通过两次洗涤完全去除pcr抑制剂,最后结合在离心柱上的纯核酸用水或试剂盒中的缓冲液进行洗脱收集,将提取的基因组dna按照光密度值法定量分析,确定基因组dna的含量和浓度。
以样本中提取的基因组dna为模板(10ng),进行如下反应。
1、multiplexpcr及其纯化处理。
1)、multiplexpcr的组成成分、体积和扩增条件。
多重pcr体系含16对引物,体系组成成分包括:amplitaqgold®360mastermix(appliedbiosystems,ca,usa)12μl;360gcenhancer(appliedbiosystems,ca,usa)2μl;dna10ng;pcrprimersmix2μl;补水至20μl。
多重pcr体系扩增条件:95℃10min;95℃30s,54℃30s,72℃30s,共35个循环;72℃7min。
2)、多重pcr产物纯化处理的组成成分、体积和纯化条件。
多重扩增产物3.8μl、sap(虾碱性磷酸酶)1.3u、exoι(核酸外切酶ι)6u、补水至10μl。纯化条件37℃,1h;95℃,15min。
2、snapshot单碱基延伸反应(sbe)及其纯化处理。
为了更清晰更准确的检测51个snp的分型,通过在有效延伸序列的5'端加不同长度的-ct尾来区分同一延伸反应体系的所有snp。由于snp位点较多,将51个snps随机分布在两个snapshot单碱基延伸(sbe)反应体系中。
1)、snapshot单碱基延伸反应的组成成分、体积和扩增条件。
snapshot延伸体系1st含24个snp,体系体积5μl,组成成分包括pcr纯化产物2μl,snapshotmix2μl,延伸引物1μl。
snapshot延伸体系2nd含27个snp,体系体积5μl,组成成分包括pcr纯化产物2μl,snapshotmix2μl,延伸引物1μl。
snapshot延伸体系1st和2nd的延伸条件相同,具体为96℃1min;96℃10s,50℃5s,60℃30s,共38个循环;60℃1min。
2)、snapshot延伸产物纯化处理的组成成分、体积和纯化条件。
单碱基延伸产物1μl、sap(虾碱性磷酸酶)1u、补水至5μl。纯化条件37℃,1h;95℃,15min。
3、snapshot单碱基延伸产物的毛细管电泳及结果分析。
毛细管电泳前变性处理组成成分和条件如下:snapshot纯化后产物1.5μl、0.1μl内标(genescan-liz120)、10μl甲酰胺(hi-diformamide);条件:95℃5min,0℃3min后,通过abi®3130dna测序仪完成对延伸引物的检测。
对延伸产物的分析通过abi®genemapperidv3.2完成。
kcnq1上51个scd相关snp的分型结果如图1所示。
4、灵敏度检测。
灵敏度实验采用multiplexpcr(d)方法进行,将dna按照浓度10ng、1ng、0.1ng分成各组。多重pcr和snapshot延伸反应的组成成分、体积和扩增条件如上。
发现上述16对基础引物的灵敏度均在0.5ng以上,即需要对于dna含量在0.5ng以上的dna扩增,本实施例方法才有效。低于0.5ng的dna不建议采用本实施例方法。
对120例dna样本进行51个snp的检测,以检测本实施例试剂盒的应用效能。参与检测的120例样本分成两组:阴性对照(control)组60例;心源性猝死(scd)组60例。各组检测的具体数据如下。
1)、阴性对照(control)组:延伸体系1st和2nd包含的51个snp在60例dna样本中均成功检测。
其中,对于位点rs12720449(p448r),54(90%)例样本检测到纯合c/c(胞嘧啶/胞嘧啶)分型,5(8.3%)例样本检测到杂合c/g(胞嘧啶/鸟嘌呤)分型,1(1.7%)例样本检测到纯合g/g(鸟嘌呤/鸟嘌呤)分型(图2)。
图2中,上层为延伸体系1中的snapshot分型结果,其中a、d、g分别表示位点p448r(rs12720449)正常分型、杂合突变、纯合突变的检测结果;中层为单独pcr并单独延伸的snapshot分型结果,其中b、e、h分别表示单独延伸时位点p448r(rs12720449)对应的检测结果;下层是对包含位点p448r(rs12720449)的pcr产物进行一代测序的结果,其中c、f、i分别表示位点12720449(p448r)相对应的测序结果。
对于位点rs1800172(g643s),54(90%)例样本检测到纯合g/g(鸟嘌呤/鸟嘌呤)分型,6(10.0%)例样本检测到杂合g/a(鸟嘌呤/腺嘌呤)分型(图3)。
图3中,上层为延伸体系1中的snapshot分型结果,其中a、d分别表示位点rs1800172(g643s)正常分型、杂合突变的检测结果;中层为单独pcr并单独延伸的snapshot分型结果,其中b、e分别表示单独延伸时位点rs1800172(g643s)对应的检测结果;下层是对包含位点rs1800172(g643s)的pcr产物进行一代测序的结果,其中c、f分别表示位点rs1800172(g643s)相对应的测序结果。
2)、心源性猝死(scd)组:反应体系1st和2nd包含的51个snp在60例dna样本中均成功检测到与ncbi数据库一致的snp分型。其中,位点rs12720449(p448r),39(65%)例样本检测到纯合c/c(胞嘧啶/胞嘧啶)分型,21(35%)例样本检测到杂合c/g(胞嘧啶/鸟嘌呤)分型(图2)。位点rs1800172(g643s),36(60%)例样本检测到纯合g/g(鸟嘌呤/鸟嘌呤)分型,24(40%)例样本检测到杂合g/a(鸟嘌呤/腺嘌呤)分型(图3)。
分别对心源性猝死(scd)组和对照(control)组进行统计学检测。
数据经统计学计算得出,位点rs12720449(p448r)与rs1800172(g643s)在两组中存在差异(p=0.05水平)。位点rs12720449与rs1800172为突变型snp,突变类型为错义突变,且有可能是潜在的scd相关突变。
为了更具体的分析51个snp在两组中的分型情况,对突变位点rs12720449(p448r)、rs1800172(g643s)进行了ⅹ2检验。
rs12720449(p448r):ⅹ2检验计算p=0.001(p=0.05水平),即两组间有明显差异。为了进一步确定该位点的突变碱基对scd的影响,针对该突变进行了or值和95%ci(可信区间)计算。对照(control)组与心源性猝死(scd)组or=4.200,95%ci=[1.638~10.772]。该位点为突变型snp,突变类型为错义突变,且有可能是潜在的scd相关突变。
rs1800172(g643s):ⅹ2检验计算p<0.0001(p=0.05水平),即两组间有明显差异。为了进一步确定该位点的突变碱基对scd的影响,针对该突变进行了or值和95%ci(可信区间)计算。对照(control)组与心源性猝死(scd)组or=4.000,95%ci=[1.696~9.434]。该位点为突变型snp,突变类型为错义突变,且有可能是潜在的scd相关突变。
通过上述实施例可已看出本发明具有以下优点。
1)、分型准确。由于snapshot微测序技术是以纯化后的多重pcr产物为模板,使用测序酶、四种荧光标记ddntp和5'-端紧靠snp位点的延伸引物进行反应,引物延伸一个碱基即终止,经测序仪检测后,根据峰的移动位置确定该延伸产物对应的snp位点,根据峰的颜色可得知掺入的碱基种类,从而确定该样本的基因型。目标snp的检测相对全基因组测序较为精准,降低了错配率。
2)、一次性可检测多重pcr反应体系包含的目标snp51个,且无需专业的技术人员进行分析,也无需昂贵的仪器设备和试剂,节省了检测成本,并在大部分法医实验室都可进行,适合法医学检测。
3)、不受snp位点多态性特性的限制。本实施例中51个snp位点的突变类型包括错义突变、框移突变(碱基的缺失或插入)、终止突变等,均能准确检测到snp的分型。另外,利用本实施例试剂盒可以灵敏的检测到杂合的snp位点,其应用性优于一代测序,准确性优于二代测序,适用于日常的法医学检测或者临床类诊断。
4)、可用于系统性分析kcnq1相关遗传性心肌离子通道疾病和心房纤维颤动的构成,或对scd患者的亲属进行基因筛查和诊断,从而达到预防和遗传咨询的意义。
尽管上述实施例已经对本发明的技术方案做了较为详细的阐述和列举,应当理解,对于本领域技术人员来说,对上述实施例做出修改或者采用等同替代方案,对本领域技术人员而言是显而易见的,在不偏离本发明精神的基础上,所做的这些修改或改进,均属于本发明要求保护的范围。
sequencelisting
<110>山西医科大学
<120>基于kcnq1的心源性猝死相关snp检测试剂盒及检测方法
<160>83
<170>siposequencelisting1.0
<210>1
<211>18
<212>dna
<213>人工序列
<220>
<400>1
agtgccccttctcgctgg18
<210>2
<211>20
<212>dna
<213>人工序列
<220>
<400>2
cgatactcacacggcgaagt20
<210>3
<211>20
<212>dna
<213>人工序列
<220>
<400>3
tcgaagcactgtctgttcct20
<210>4
<211>20
<212>dna
<213>人工序列
<220>
<400>4
tccagaagagagtccccgtg20
<210>5
<211>18
<212>dna
<213>人工序列
<220>
<400>5
gcatggctgggttcaaac18
<210>6
<211>20
<212>dna
<213>人工序列
<220>
<400>6
caggcatgactcaccgatga20
<210>7
<211>20
<212>dna
<213>人工序列
<220>
<400>7
acgagagcagggtgtatgct20
<210>8
<211>18
<212>dna
<213>人工序列
<220>
<400>8
gcaaggtggatggggcgt18
<210>9
<211>19
<212>dna
<213>人工序列
<220>
<400>9
gccccacaccatctccttc19
<210>10
<211>20
<212>dna
<213>人工序列
<220>
<400>10
tcaagctgtcctagtgtggg20
<210>11
<211>22
<212>dna
<213>人工序列
<220>
<400>11
agctgataaccaccctgtacat22
<210>12
<211>20
<212>dna
<213>人工序列
<220>
<400>12
caaggcccgaagtctcaaga20
<210>13
<211>20
<212>dna
<213>人工序列
<220>
<400>13
gggtttgggttaggcagttg20
<210>14
<211>21
<212>dna
<213>人工序列
<220>
<400>14
ccctcttggttctgaacgtaa21
<210>15
<211>19
<212>dna
<213>人工序列
<220>
<400>15
gtaaccacgtcctgaggct19
<210>16
<211>18
<212>dna
<213>人工序列
<220>
<400>16
atggttctgacaggtggc18
<210>17
<211>18
<212>dna
<213>人工序列
<220>
<400>17
tgggagctgggaccatgt18
<210>18
<211>20
<212>dna
<213>人工序列
<220>
<400>18
tgggctactcaccaccacag20
<210>19
<211>23
<212>dna
<213>人工序列
<220>
<400>19
tgtccaggaaccgctaatctgtt23
<210>20
<211>20
<212>dna
<213>人工序列
<220>
<400>20
gaaactcactggcttgctcg20
<210>21
<211>20
<212>dna
<213>人工序列
<220>
<400>21
gtccccacactttctcctca20
<210>22
<211>20
<212>dna
<213>人工序列
<220>
<400>22
atgggtgtcagcagagtctc20
<210>23
<211>20
<212>dna
<213>人工序列
<220>
<400>23
ccactcacaatctcctctcc20
<210>24
<211>20
<212>dna
<213>人工序列
<220>
<400>24
gccttgacaccctccactat20
<210>25
<211>19
<212>dna
<213>人工序列
<220>
<400>25
cagggaggagaagtgatgc19
<210>26
<211>18
<212>dna
<213>人工序列
<220>
<400>26
ctgcagctccttgatgcg18
<210>27
<211>20
<212>dna
<213>人工序列
<220>
<400>27
aggtgtgaactggtgtctgt20
<210>28
<211>20
<212>dna
<213>人工序列
<220>
<400>28
tctgacatcatggggagagc20
<210>29
<211>20
<212>dna
<213>人工序列
<220>
<400>29
ctaccaccccacttcccaag20
<210>30
<211>20
<212>dna
<213>人工序列
<220>
<400>30
caggagcttcacgttcacac20
<210>31
<211>22
<212>dna
<213>人工序列
<220>
<400>31
ctcatcaccgacatgcttcacc22
<210>32
<211>19
<212>dna
<213>人工序列
<220>
<400>32
ccctctcactcaggcccat19
<210>33
<211>22
<212>dna
<213>人工序列
<220>
<400>33
cctggaagtttccgacttacca22
<210>34
<211>15
<212>dna
<213>人工序列
<220>
<400>34
tttgcccggaagccc15
<210>35
<211>32
<212>dna
<213>人工序列
<220>
<400>35
ctgcttctctgtctttgccatctccttctttg32
<210>36
<211>17
<212>dna
<213>人工序列
<220>
<400>36
agcgtgctgtccaccat17
<210>37
<211>16
<212>dna
<213>人工序列
<220>
<400>37
agggcggcatactgct16
<210>38
<211>19
<212>dna
<213>人工序列
<220>
<400>38
tactccgtcccgaagaaca19
<210>39
<211>16
<212>dna
<213>人工序列
<220>
<400>39
gaccagaggcggacca16
<210>40
<211>15
<212>dna
<213>人工序列
<220>
<400>40
gtgggcctctggggg15
<210>41
<211>20
<212>dna
<213>人工序列
<220>
<400>41
ccacagacctcatcgtggtc20
<210>42
<211>17
<212>dna
<213>人工序列
<220>
<400>42
cgacgtggcaaacacct17
<210>43
<211>21
<212>dna
<213>人工序列
<220>
<400>43
tgcagatcctgaggatgctac21
<210>44
<211>20
<212>dna
<213>人工序列
<220>
<400>44
gtgtacctggctgagaagga20
<210>45
<211>18
<212>dna
<213>人工序列
<220>
<400>45
gtggagttcggcagctac18
<210>46
<211>18
<212>dna
<213>人工序列
<220>
<400>46
tgcaggtcacagtcacca18
<210>47
<211>17
<212>dna
<213>人工序列
<220>
<400>47
cgtctggggcaccttgt17
<210>48
<211>18
<212>dna
<213>人工序列
<220>
<400>48
cccaggggattcttggct18
<210>49
<211>16
<212>dna
<213>人工序列
<220>
<400>49
gccctgaaggtgcagc16
<210>50
<211>16
<212>dna
<213>人工序列
<220>
<400>50
aaccggcagatcccgg16
<210>51
<211>19
<212>dna
<213>人工序列
<220>
<400>51
cctgaatgagtgaggctgc19
<210>52
<211>16
<212>dna
<213>人工序列
<220>
<400>52
ccgacctcagaccgca16
<210>53
<211>18
<212>dna
<213>人工序列
<220>
<400>53
cgactcctccacctggaa18
<210>54
<211>19
<212>dna
<213>人工序列
<220>
<400>54
tccaggagtcaccccattg19
<210>55
<211>15
<212>dna
<213>人工序列
<220>
<400>55
tcacgtgcgaccccc15
<210>56
<211>14
<212>dna
<213>人工序列
<220>
<400>56
agcggcggctggac14
<210>57
<211>16
<212>dna
<213>人工序列
<220>
<222>(12)
<223>r=a或g
<400>57
gcgccgatcgtrttgc16
<210>58
<211>14
<212>dna
<213>人工序列
<220>
<222>(6)
<223>d=g或a或t
<400>58
cgcccdcctgaacc14
<210>59
<211>14
<212>dna
<213>人工序列
<220>
<222>(3)
<223>y=c或t
<400>59
caygatcggcgccc14
<210>60
<211>16
<212>dna
<213>人工序列
<220>
<222>(5,7)
<223>r=a或g
<400>60
gatcrcrgcagcaaca16
<210>61
<211>14
<212>dna
<213>人工序列
<220>
<222>(10)
<223>h=a或t或c
<400>61
ctctggggghggct14
<210>62
<211>25
<212>dna
<213>人工序列
<220>
<400>62
gggttctcaccagaactgtcatagc25
<210>63
<211>16
<212>dna
<213>人工序列
<220>
<400>63
ggtccaggtcctcggc16
<210>64
<211>22
<212>dna
<213>人工序列
<220>
<400>64
gggacgtcattgagcagtactc22
<210>65
<211>29
<212>dna
<213>人工序列
<220>
<400>65
agccaggtacacaaagtacgaggagaaga29
<210>66
<211>18
<212>dna
<213>人工序列
<220>
<400>66
ggctggaccagtccattg18
<210>67
<211>24
<212>dna
<213>人工序列
<220>
<400>67
cccccatagaaaagagcaaggatc24
<210>68
<211>15
<212>dna
<213>人工序列
<220>
<400>68
ggtaggctcacgcgc15
<210>69
<211>20
<212>dna
<213>人工序列
<220>
<400>69
gggccaccattaaggtcatt20
<210>70
<211>21
<212>dna
<213>人工序列
<220>
<400>70
ttggccacaaagtactgcatg21
<210>71
<211>19
<212>dna
<213>人工序列
<220>
<400>71
ctcagcacagggcttacct19
<210>72
<211>19
<212>dna
<213>人工序列
<220>
<400>72
tgttcttcgggacggagta19
<210>73
<211>18
<212>dna
<213>人工序列
<220>
<400>73
acgcagaggaccaccatg18
<210>74
<211>17
<212>dna
<213>人工序列
<220>
<400>74
cagctgctctccttgca17
<210>75
<211>14
<212>dna
<213>人工序列
<220>
<400>75
gtggcagcaccccc14
<210>76
<211>16
<212>dna
<213>人工序列
<220>
<400>76
catcacccagccctgc16
<210>77
<211>14
<212>dna
<213>人工序列
<220>
<400>77
gcagtggcggctcc14
<210>78
<211>19
<212>dna
<213>人工序列
<220>
<400>78
gcaggaagagctcagggtc19
<210>79
<211>16
<212>dna
<213>人工序列
<220>
<400>79
gcacggtcagctgctc16
<210>80
<211>19
<212>dna
<213>人工序列
<220>
<400>80
cgcgtctccatctacagca19
<210>81
<211>15
<212>dna
<213>人工序列
<220>
<400>81
gtgcgcgccaacacc15
<210>82
<211>14
<212>dna
<213>人工序列
<220>
<400>82
acgtccagggccgc14
<210>83
<211>18
<212>dna
<213>人工序列
<220>
<400>83
ggacgctcgaggaagttg18
- 还没有人留言评论。精彩留言会获得点赞!