用于TERT基因全外显子二代测序的探针组合物、试剂及控制系统的制作方法
本发明涉及基因测序领域,具体涉及一种用于tert基因全外显子二代测序的探针组合物、试剂及控制系统。
背景技术:
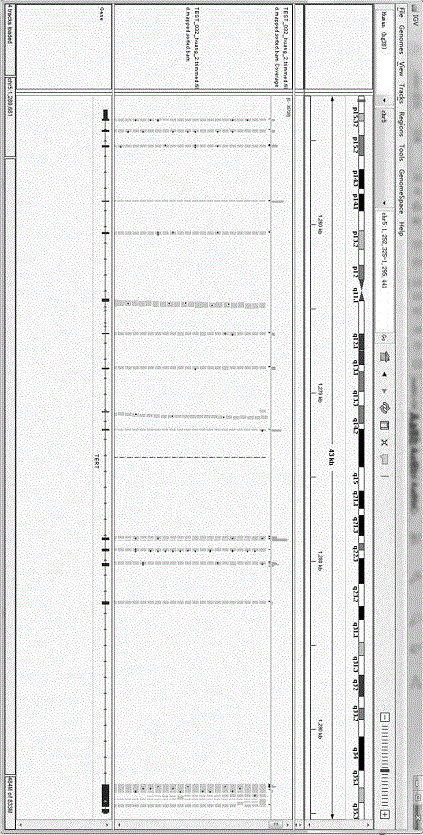
:tert(telomerasereversetranscriptase,端粒酶逆转录酶)是指的端粒酶里面具有逆转录酶活性的催化亚基,是端粒酶的重要催化部分,影响着端粒的调控机理,从而维持细胞的永生化和致癌。tert基因位于第5号染色体,其转录本nm_001193376.1具有15个外显子,外显子全长3275bp,tert基因突变在黑色素瘤、脂肪瘤、肝细胞瘤、神经胶质瘤等恶性肿瘤中都有一定的检出率,且阳性患者的预后大多不良,是相关肿瘤的生物标记。关于tert基因的突变,目前已知研究较多的是能够增强tert启动子活性的c228t和c250t两个点突变。现有技术的tert基因突变检测多为针对tert基因启动子活性的c228t和c250t位点进行检测,存在以下局限性:1.实时荧光定量pcr检测和基因芯片检测,不能检测未知位点;2.sanger测序的检测位点局限在800bp以内;3.涉及多个位点检测时,原有技术操作繁琐、费用高;4.不能检测低频突变。全外显子测序的难点在于探针的设计和优化,要求探针序列组合成型后,性能稳定、操作简便,且其片段长度更加适合illumina测序平台。技术实现要素:为解决现有技术的tert基因突变检测多为针对tert基因启动子活性的c228t和c250t位点进行检测,存在多种局限性的技术问题,本发明提供一种解决上述问题的用于tert基因全外显子二代测序的探针组合物、试剂及控制系统。用于tert基因全外显子二代测序的探针组合物,包括seqidno:01至seqidno:36所示的序列。用于tert基因全外显子二代测序的试剂,包括上述的探针组合物及二代测序所需试剂。用于tert基因全外显子二代测序的控制系统包括:检测模块,采用上文所述的用于tert基因全外显子二代测序的试剂进行二代测序;显示模块,将检测结果转换为可视化数据,并将可视化的数据于显示设备进行展示;分析模块:基于预设的质控程序对检测结果进行验证,并将验证结果于同一显示设备进行展示。其中,运行所述检测模块包括以下步骤:步骤一:文库构建,包括gdna浓度检测及稀释、gdna片段化、末端修复和a碱基的添加、接头的连接及连接产物的纯化、文库片段筛选、pcr扩增及扩增产物的纯化、终文库qc;步骤二:杂交捕获,包括文库pooling、变性与杂交、捕获洗脱、pcr扩增与扩增产物的纯化;步骤三:上机测序,经过稀释、变性等步骤,调整至符合测序仪的标准,并按测序仪的标准操作进行测序、输出检测结果。所述显示模块将步骤三中的检测结果转化为包括“5号染色体概图”、“测序结果”、“参考序列”三项数据的图表。所述分析模块直接以所述显示模块转化得到的图表为原始数据,与预设的数据进行对比,并向所述显示模块输出检测结果。相较于现有技术,本发明提供的所述用于tert基因全外显子二代测序的探针组合物,经设计优化后得到的探针一共36条,具有以下优点:1.能够100%覆盖tert基因的全外显子区域,能够全面、准确的检测待测样本tert基因的突变情况;2.能够检测出sanger测序、实时荧光定量pcr、基因芯片所不能检测出的低频突变,具有极高的科研、临床价值,且操作简单、成本较低。附图说明图1是探针法tert二代测序结果概图;图2是探针法tert二代测序结果细节图。具体实施方式下面将对本发明实施例中的技术方案进行清楚、完整地描述,所描述的实施例仅是本发明的一部分实施例,而不是全部的实施例。实施例1:用于tert基因全外显子二代测序的探针组合物包括seqidno:01~seqidno:36所示的序列。将36条探针按照每管100p的标准稀释,取一个新的1.5ml离心管,将每种探针分别加入2ul,补水至总体积为200ul。维持终浓度是1pmol,震荡混匀,-20℃保存备用。用于tert基因全外显子二代测序的试剂包括上述探针组合物及二代测序所需的试剂,具体的内容在以下用于tert基因全外显子二代测序的方法中说明。用于tert基因全外显子二代测序的方法为采用上述的试剂对tert基因全外显子进行二代测序,包括以下步骤:1、gdna浓度检测及稀释:qubit3.0测定gdna样品浓度,将样品稀释至10ng/ul,样品体积大于35ul,总dna质量大于350ng。2、gdna片段化、末端修复和a碱基的添加,包括:2.1、将fxbuffer和fxenzymemix从-20℃取出冰上解冻,混匀,微离心;2.2、配制反应体系:2.3、吹打混匀,微离心。2.4、将pcr仪的热盖温度调至70℃并使加热板预先冷却至4℃,按下表设置反应程序进行反应:3、接头的连接及连接产物的纯化,包括:3.1、将ligationbuffer、dnaligase和aapter从-20℃取出冰上解冻,混匀,微离心;并预先取出agencourtampurexpbeads放置室温;3.2、配制接头连接反应体系:3.3、吹打混匀,微离心;3.4、20℃孵育30分钟(pcr仪的热盖调为:lifoff);3.5、取一新的ep管,将连接产物与80ul(0.8x)agencourtampurexpbeads混匀,室温孵育5分钟;3.6、磁力架上放置2分钟,弃上清;3.7、加入200ul新鲜配制的80%乙醇,转动ep管几次,静置30s后吸去上清;3.8、重复(3.7)步骤一次;3.9、室温放置5-10分钟干燥磁珠;3.10、将ep管从磁力架上取下,加入52.5ulresuspensionbuffer重悬磁珠,室温静置2分钟,磁力架上放置2分钟,转移50ul上清至新的ep管中。4、文库片段筛选,包括:4.1、在50ul文库中加入35ulagencourtampurexpbeads,吹打混匀,室温放置5分钟;4.2、微离心后放置磁力架上2分钟,移取上清至新的96孔板中,并加入10ulagencourtampurexpbeads,吹打混匀,室温静置5分钟;4.3、微离心,放置磁力架上2分钟,弃上清;4.4、加入100ul新鲜配制的80%乙醇,静置30s,吸去上清;4.5、重复上一步骤一次;4.6、室温放置5-10分钟干燥磁珠;4.7、将96孔板从磁力架上取下,加入26ulresuspensionbuffer重悬磁珠,室温静置2分钟,微离心后放置磁力架上2分钟,转移23.5ul上清至新的96孔板中进行下一步pcr反应。5、pcr扩增及扩增产物的纯化,包括:5.1、将hifipcrmastermix和primermix从-20℃取出冰上解冻,混匀,微离心;5.2、配制pcr反应体系:试剂体积(ul)hifipcrmastermix,2x25primermix(10μmeach)1.5librarydna23.5总量505.3、吹打混匀,微离心;5.4、pcr扩增反应程序:温度时间循环数98℃1min198℃20s860℃30s72℃30s72℃1min110℃∞15.5、取一新的ep管,将pcr产物与50ul(1.0x)agencourtampurexpbeads混匀,室温孵育5分钟;5.6、磁力架上放置2分钟,弃上清;5.7、加入200ul新鲜配制的80%乙醇,转动ep管几次,静置30s后吸去上清;5.8、重复(5.7)步骤一次;5.9、室温放置5-10分钟干燥磁珠;5.10、将ep管从磁力架上取下,加入20ulresuspensionbuffer重悬磁珠,室温静置2分钟,磁力架上放置2分钟,转移17ul上清至新的ep管中,-20℃保存。6、终文库qc,包括:6.1、用qubit3.0对终文库进行浓度测定(文库浓度处于30—50ng/ul);6.2、采用琼脂糖凝胶电泳检测文库片段大小,片段大小处于200-500bp左右。7、文库pooling:取出构号的文库,按照200ng/sample;每个pool不超过6000ng的量混合文库于0.2mlpcr管中,建好的文库于-20℃保存。8、变性与杂交,包括:8.1、-20℃冰箱取出cothumandna和xgen®universalblockers,溶解后震荡离心;8.2、向pooling文库中加入以下试剂:试剂体积(ul)耗材类型混合文库<6000ng0.2mlpcr管humancot-1dna®5xgen®universalblockers-tsmix2总量78.3、将pcr管放至真空抽滤系统(60℃)干燥成干粉,约20min;(预热15min,选择v-aq模式,brake-off);8.4、根据下表向干粉中依次加入相应试剂,吹打混匀后室温放置5min,置于pcr仪上进行变性反应:试剂体积(ul)xgen2xhybridizationbuffer8.5xgenhybridizationbufferenhancer2.7nuclease-freewater1.8总量13反应程序为:温度时间循环数95℃10min4℃∞8.5、反应结束后将样本放置冰板上,加入4ul探针混合液;8.6、混匀,置于pcr仪上反应16-18h;反应程序为:温度时间循环数65℃∞(热盖温度:75℃)。9、捕获洗脱,包括:9.1、取出dynabeads®m-270streptavidinbeads捕获磁珠平衡至室温。按下表配制xgen2xbeadwashbuffer;试剂原体积(ul)ddh2o体积(ul)1xbuffer(ul)xgen2xbeadwashbuffer2502505009.2、吸取100ul/poolm270捕获磁珠,放置磁力架上,弃上清,加入200ul1xbeadwashbuffer重悬磁珠,离心后放置磁力架上,弃上清;重复清洗磁珠一次;9.3、捕获磁珠重悬:加入100ul1xbeadwashbuffer重悬磁珠,并转移至0.2mlpcr管中,离心后放置磁力架上,弃上清;9.4、磁珠与杂交产物结合:将杂交产物17ul转移至捕获磁珠中,吹打重悬磁珠后放置pcr仪(65℃,热盖75℃)孵育45min,中间间隔15min振荡混匀一次;9.5、buffer配制:根据下表配制1xwashbufferi、1xwashbufferii、1xwashbufferiii和1xstringentwashbuffer,将100ul1xwashbufferi/pool和400ul1xstringentwashbuffer放置于65℃中温育;按下表配制1xbeadwashbuffer;试剂原体积(ul)ddh2o体积(ul)1xbuffer(ul)备注xgen10xwashbufferi30270300100ul65℃预热xgen10xwashbufferii20180200xgen10xwashbufferiii20180200xgen10xstringentwashbuffer4036040065℃预热xgen2xbeadwashbuffer25025050010、pcr扩增与扩增产物的纯化,包括:10.1、试剂解冻后震荡离心,按下表分装:试剂体积(ul)kapahifihotstartreadymix25post-lm-pcroligos5总体积30带磁珠产物20反应体系总体积5010.2、按上表加入mix,混匀,微离心后置于pcr仪上反应;反应程序为:10.3、扩增完成后,将pcr管置于磁力架上,吸取50ul上清到新的1.5ml的ep管中,并加入75ulagencourtampurexpbeads,混匀,室温放置5min;10.4、微离心后,置于磁力架上2min,弃上清;10.5、加入200.0ul80%乙醇,静置30s,弃上清;10.6、加入200.0ul80%乙醇,静置30s,弃上清;10.7、室温放置10min,待磁珠干燥;10.8、加入22.0ulresuspensionbuffer(rsb),重悬磁珠;10.9、室温静置2min,离心后磁力架上放置2min;10.10、转移20.0ul上清至新0.2mlpcr管中,放置4℃或-20℃中保存;10.11、用qubit®3.0fluorometer测定浓度并记录。11、上机测序,包括:11.1、将文库用resuspensionbuffer(rsb)稀释至1.0nm;11.2、制备20.0ul0.2n的naoh;11.3、将20.0ul1nm的混合文库与20.0ul0.2n的naoh混匀,室温变性5min;11.4、与960.0ulhybridizationbuffer混匀;11.5、移取100.0ul与1200.0ulhybridizationbuffer混匀,加入至reagentcartridge10号孔。12、按illuminanextseq550二代测序仪的标准操作流程测序。13、分析结果:请同时参阅图1、图2,分别是探针法tert二代测序结果概图和细节图。测序结果经过去除接头、重复序列、低质量序列等生物信息学处理之后,利用igv软件打开,结果如下图1和图2所示。图中由上至下表示的信息均依次为:“5号染色体概图”、“测序结果”、“参考序列”,由图示可以直观的看出本款探针有效。实施例2:通过用于tert基因全外显子二代测序控制系统,控制专用设备来实施实施例1中所述的用于tert基因全外显子二代测序方法。以上所述仅为本发明的实施例,并非因此限制本发明的专利范围,凡是利用本发明说明书内容所作的等效结构或等效流程变换,或直接或间接运用在其它相关的
技术领域:
,均同理包括在本发明的专利保护范围之内。序列表<110>长沙金域医学检验所有限公司<120>用于tert基因全外显子二代测序的探针组合物、试剂及控制系统<130>0006<160>36<170>siposequencelisting1.0<210>1<211>120<212>dna<213>artificialsequence<400>1gccccggccacccccgcgatgccgcgcgctccccgctgccgagccgtgcgctccctgctg60cgcagccactaccgcgaggtgctgccgctggccacgttcgtgcggcgcctggggccccag120<210>2<211>120<212>dna<213>artificialsequence<400>2ggctggcggctggtgcagcgcggggacccggcggctttccgcgcgctggtggcccagtgc60ctggtgtgcgtgccctgggacgcacggccgccccccgccgccccctccttccgccaggtg120<210>3<211>120<212>dna<213>artificialsequence<400>3atgcggagagcagcgcaggcgactcagggcgcttcccccgcaggtgtcctgcctgaagga60gctggtggcccgagtgctgcagaggctgtgcgagcgcggcgcgaagaacgtgctggcctt120<210>4<211>120<212>dna<213>artificialsequence<400>4caccccagcagccggtgtctgtgcccgggagaagccccagggctctgtggcggcccccga60ggaggaggacacagacccccgtcgcctggtgcagctgctccgccagcacagcagcccctg120<210>5<211>120<212>dna<213>artificialsequence<400>5gcaggtgtacggcttcgtgcgggcctgcctgcgccggctggtgcccccaggcctctgggg60ctccaggcacaacgaacgccgcttcctcaggaacaccaagaagttcatctccctggggaa120<210>6<211>120<212>dna<213>artificialsequence<400>6gcatgccaagctctcgctgcaggagctgacgtggaagatgagcgtgcgggactgcgcttg60gctgcgcaggagcccaggtgaggaggtggtggccgtcgagggcccaggccccagagctga120<210>7<211>120<212>dna<213>artificialsequence<400>7cggcttcgcgctgctggacggggcccgcgggggcccccccgaggccttcaccaccagcgt60gcgcagctacctgcccaacacggtgaccgacgcactgcgggggagcggggcgtgggggct120<210>8<211>120<212>dna<213>artificialsequence<400>8gctgctgcgccgcgtgggcgacgacgtgctggttcacctgctggcacgctgcgcgctctt60tgtgctggtggctcccagctgcgcctaccaggtgtgcgggccgccgctgtaccagctcgg120<210>9<211>120<212>dna<213>artificialsequence<400>9cgctgccactcaggcccggcccccgccacacgctagtggaccccgaaggcgtctgggatg60cgaacgggcctggaaccatagcgtcagggaggccggggtccccctgggcctgccagcccc120<210>10<211>120<212>dna<213>artificialsequence<400>10gggtgcgaggaggcgcgggggcagtgccagccgaagtctgccgttgcccaagaggcccag60gcgtggcgctgcccctgagccggagcggacgcccgttgggcaggggtcctgggcccaccc120<210>11<211>120<212>dna<213>artificialsequence<400>11gggcaggacgcgtggaccgagtgaccgtggtttctgtgtggtgtcacctgccagacccgc60cgaagaagccacctctttggagggtgcgctctctggcacgcgccactcccacccatccgt120<210>12<211>120<212>dna<213>artificialsequence<400>12gggccgccagcaccacgcgggccccccatccacatcgcggccaccacgtccctgggacac60gccttgtcccccggtgtacgccgagaccaagcacttcctctactcctcaggcgacaagga120<210>13<211>120<212>dna<213>artificialsequence<400>13gcagctgcggccctccttcctactcagctctctgaggcccagcctgactggcgctcggag60gctcgtggagaccatctttctgggttccaggccctggatgccagggactccccgcaggtt120<210>14<211>120<212>dna<213>artificialsequence<400>14gccccgcctgccccagcgctactggcaaatgcggcccctgtttctggagctgcttgggaa60ccacgcgcagtgcccctacggggtgctcctcaagacgcactgcccgctgcgagctgcggt120<210>15<211>120<212>dna<213>artificialsequence<400>15tggcatgtccttctcgtttaaggggttggctgtgttccggccgcagagcaccgtctgcgt60gaggagatcctggccaagttcctgcactggctgatgagtgtgtacgtcgtcgagctgctc120<210>16<211>120<212>dna<213>artificialsequence<400>16aggtctttcttttatgtcacggagaccacgtttcaaaagaacaggctctttttctaccgg60aagagtgtctggagcaagttgcaaagcattggaatcaggtactgtatccccacgccaggc120<210>17<211>120<212>dna<213>artificialsequence<400>17gcaagcctcctgaggggctctctattgcagacagcacttgaagagggtgcagctgcggga60gctgtcggaagcagaggtcaggcagcatcgggaagccaggcccgccctgctgacgtccag120<210>18<211>120<212>dna<213>artificialsequence<400>18actccgcttcatccccaagcctgacgggctgcggccgattgtgaacatggactacgtcgt60gggagccagaacgttccgcagagaaaagagggtggctgtgctttggtttaacttcctttt120<210>19<211>120<212>dna<213>artificialsequence<400>19cctgactgtctcccatgctgtccccgccaggccgagcgtctcacctcgagggtgaaggca60ctgttcagcgtgctcaactacgagcgggcgcggcgccccggcctcctgggcgcctctgtg120<210>20<211>120<212>dna<213>artificialsequence<400>20ctgggcctggacgatatccacagggcctggcgcaccttcgtgctgcgtgtgcgggcccag60gacccgccgcctgagctgtactttgtcaaggtgggtgccggggacccccgtgagcagccc120<210>21<211>120<212>dna<213>artificialsequence<400>21tctgcgtggccactgtcagtctcctcgcctccactcacacaggtggatgtgacgggcgcg60tacgacaccatcccccaggacaggctcacggaggtcatcgccagcatcatcaaaccccag120<210>22<211>120<212>dna<213>artificialsequence<400>22aacacgtactgcgtgcgtcggtatgccgtggtccagaaggccgcccatgggcacgtccgc60aaggccttcaagagccacgtaaggttcacgtgtgatagtcgtgtccaggatgtgtgtctc120<210>23<211>120<212>dna<213>artificialsequence<400>23ctctgccggcaggtctctaccttgacagacctccagccgtacatgcgacagttcgtggct60cacctgcaggagaccagcccgctgagggatgccgtcgtcatcgagcaggtctgggcactg120<210>24<211>120<212>dna<213>artificialsequence<400>24cccgtctgctttcgcagagctcctccctgaatgaggccagcagtggcctcttcgacgtct60tcctacgcttcatgtgccaccacgccgtgcgcatcaggggcaagtgagtcaggtggccag120<210>25<211>120<212>dna<213>artificialsequence<400>25caggtcctacgtccagtgccaggggatcccgcagggctccatcctctccacgctgctctg60cagcctgtgctacggcgacatggagaacaagctgtttgcggggattcggcgggacgggtg120<210>26<211>120<212>dna<213>artificialsequence<400>26gctgtgtattttcccttattttaggctgctcctgcgtttggtggatgatttcttgttggt60gacacctcacctcacccacgcgaaaaccttcctcaggtgaggcccgtgccgtgtgtctgt120<210>27<211>120<212>dna<213>artificialsequence<400>27gggctgacattgcccctctgccttaggaccctggtccgaggtgtccctgagtatggctgc60gtggtgaacttgcggaagacagtggtgaacttccctgtagaagacgaggccctgggtggc120<210>28<211>120<212>dna<213>artificialsequence<400>28acggcttttgttcagatgccggcccacggcctattcccctggtgcggcctgctgctggat60acccggaccctggaggtgcagagcgactactccaggtgagcgcacctggccggaagtgga120<210>29<211>120<212>dna<213>artificialsequence<400>29gagggggctgggtgtggggcaggcacctgtgtctgacattcccccctgtgtctcagctat60gcccggacctccatcagagccagtctcaccttcaaccgcggcttcaaggctgggaggaac120<210>30<211>120<212>dna<213>artificialsequence<400>30atgcgtcgcaaactctttggggtcttgcggctgaagtgtcacagcctgtttctggatttg60caggtgagcaggctgatggtcagcacagagttcagagttcaggaggtgtgtgcgcaagta120<210>31<211>120<212>dna<213>artificialsequence<400>31cgtttctcactctttcttggcgactctaggtgaacagcctccagacggtgtgcaccaaca60tctacaagatcctcctgctgcaggcgtacaggtgagccgccaccaaggggtgcaggccca120<210>32<211>120<212>dna<213>artificialsequence<400>32ccacgagcaccgtctgattaggaggcctttcctctgacgctgtccgccatcctctcaggt60ttcacgcatgtgtgctgcagctcccatttcatcagcaagtttggaagaaccccacatttt120<210>33<211>120<212>dna<213>artificialsequence<400>33tcctgcgcgtcatctctgacacggcctccctctgctactccatcctgaaagccaagaacg60caggtatgtgcaggtgcctggcctcagtggcagcagtgcctgcctgctggtgttagtgtg120<210>34<211>120<212>dna<213>artificialsequence<400>34ggaggggagctgggctgggcctgtgactcctcagcctctgttttcccccagggatgtcgc60tgggggccaagggcgccgccggccctctgccctccgaggccgtgcagtggctgtgccacc120<210>35<211>120<212>dna<213>artificialsequence<400>35aagcattcctgctcaagctgactcgacaccgtgtcacctacgtgccactcctggggtcac60tcaggacaggcaagtgtgggtggaggccagtgcgggccccacctgcccaggggtcatcct120<210>36<211>120<212>dna<213>artificialsequence<400>36gattttggccccgcagcccagacgcagctgagtcggaagctcccggggacgacgctgact60gccctggaggccgcagccaacccggcactgccctcagacttcaagaccatcctggactga120当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1