基于mariner转座子构建自杀型质粒及耐药突变菌株的方法与流程
本发明属于基因工程
技术领域:
,特别涉及一种基于mariner转座子构建自杀型质粒及耐药突变菌株的方法。
背景技术:
:细菌耐药性的产生已成为一个令人担忧的问题,耐药细菌越来越多地出现在动物身上,也越来越多地出现在世界各地的人类身上。某些细菌几乎对所有临床药物都耐药,主要是细菌通过水平传播或者垂直传播或自身基因突变获得某些耐药基因。例如基因编码的抗广谱头孢菌素使得细菌可以耐碳青霉烯,基因编码的16srrna甲基化酶赋予细菌抵抗氨基糖甙类药物的能力,质粒介导的耐喹诺酮(pmqr)基因赋予细菌耐氟喹诺酮类药物的能力。mcr基因赋予细菌抵抗多粘菌素类药物的能力。基因赋予细菌抵抗某种药物的能力,世界范围类已经发现许多耐药基因。然而,随着细菌进化,许多新型耐药基因尚待挖掘。因此,发现新基因,研究细菌进化特点迫在眉睫。研究新基因,现有的方法只要有基因敲除,这种方法主要是失活目标基因,从而研究目标基因失活以后细菌表型的变化,从而探究目标基因的功能。该方法是可以用来敲除靶基因,但对于在偌大的细菌染色体组寻找靶基因无异于大海捞针。此外,测序技术的兴起,也为研究新基因提供了新的武器。但是测序技术成本昂贵,测序技术可以用来比较基因序列的同源性,但是对于研究某些差异比较大的亚型,效果不好,且对技术人员要求很高。原核生物和真核生物基因组中存在着一种可以在基因组中移动的dna序列,被称为转座子。转座子是基因组内相对独立的、可移动序列,他们不必借助质粒或噬菌体形式就可以从一个位置“跳跃”到另一个位置,从而产生基因突变(slotkinetal.,2007)。早在1951年美国遗传学家barbaramclintock在研究玉米的过程中发现了可移动的dna转座子,之后又陆陆续续在细菌、真菌以及一切生物中发现了转座子,转座子的发现说明了基因在染色体上的位置并非是固定的(medhoraetal.,1988)。转座子有以下几个特点:1、转座子是基因组的正常组成成分,不独立存在;2、转座的发生不依赖于转座子和靶位点之间任何的序列同源性,在基因组内由一个位置直接转移到另一个部位;3、转座随机发生;4、转座子插入引起基因重排使得物种进化。由于转座引起基因的重排使得转座子成为各种生物基因分析的有效工具。技术实现要素:本发明的首要目的在于克服现有技术的缺点与不足,提供一种基于mariner转座子构建自杀型质粒的方法。本发明的另一目的在于提供所述基于mariner转座子构建得到的自杀型质粒。本发明的再一目的在于提供所述自杀型质粒在构建耐药突变菌株中的应用。本发明的目的通过下述技术方案实现:一种基于mariner转座子构建自杀型质粒的方法,包括如下步骤:(1)以pcat-marinerr质粒为模板,以序列at-f和catr为引物,进行pcr扩增,得到扩增产物(作为质粒骨架);其中,at-f和catr的核苷酸序列如下所示:at-f:5’-ccagaactcatgttgcatggtat-3’;catr:5’-ccaagttctgagcctttttgcg-3’;(2)以puc57-tpm质粒为模板,以序列tpm-f和tpm-r为引物,进行pcr扩增,得到tpm抗性基因;其中,tpm-f和tpm-r的核苷酸序列如下所示:tpm-f:5’-tcagaacttggcagtgagtatcca-3’;tpm-r:5’-atgcaacatgagttctggcat-3’;(3)将步骤(1)中得到的扩增产物和步骤(2)中得到的tpm抗性基因进行同源重组,得到ptpm-xy质粒,即所述基于mariner转座子构建的自杀型质粒。步骤(1)中所述的扩增产物的大小为3443bp。步骤(2)中所述的puc57-tpm质粒优选为由擎科生物科技有限公司合成的含有tpm基因的puc57-tpm质粒。步骤(3)中所述的同源重组为采用同源重组试剂盒进行同源重组;优选为采用南京诺唯赞生物科技有限公司的同源重组试剂盒onestepcloningkit进行同源重组。一种自杀型质粒,通过上述任一项所述的方法构建得到。所述的自杀型荧光质粒在构建荧光菌株中的应用。一种基于mariner转座突变技术构建耐药突变菌株的方法,包括如下步骤:(a)将上述自杀型质粒(即ptpm-xy质粒)转化大肠杆菌wm3064(e.coliwm3064)感受态细胞,得到大肠杆菌wm3064/ptpm-xy;(b)将大肠杆菌wm3064/ptpm-xy和目标菌株分别培养至对数生长期;(c)将对数生长期的大肠杆菌wm3064/ptpm-xy和目标菌株按体积比1:1~10混合均匀后加入到培养基中进行培养,得到含有细菌的培养液;(d)将含有细菌的培养液涂布到含有亚碲酸盐的平板上继续培养,然后挑取单菌落同时接种到单药板和双药板上,再挑选双药板上不生长而单药板的菌生长的菌株,得到突变菌株,即所述基于mariner转座突变技术构建的耐药突变菌株;其中,单药板为含有亚碲酸盐的平板,双药板为含有亚碲酸盐和原抗性筛选标记药物的平板。步骤(a)中所述的大肠杆菌wm3064(e.coliwm3064)感受态细胞通过cacl2法制得;优选为通过如下方法制备得到:(i)将大肠杆菌wm3064接种到含有二氨荃庚二酸(dap)的lb肉汤培养基进行活化培养,然后转接到含有二氨荃庚二酸(dap)的lb肉汤培养基进行扩大培养,再在冰上预冷后于4℃条件下进行离心,弃上清,得到大肠杆菌菌液;(ii)将预冷的cacl2溶液加入到步骤(i)中得到的大肠杆菌菌液中,混匀后于4℃条件下进行离心,弃上清,再加入预冷的含15%(v/v)甘油的cacl2溶液,重悬细菌,冰上放置,得到大肠杆菌wm3064感受态细胞。步骤(i)中所述的lb肉汤培养基中的二氨荃庚二酸的浓度为50μg/ml。步骤(i)中所述的活化培养的条件优选为:37℃过夜培养。步骤(i)中所述的扩大培养的条件优选为:37℃培养2~3h。步骤(i)中所述的冰上预冷的时间优选为10min。步骤(i)中所述的离心的条件优选为:4000g离心15min。步骤(ii)中所述的cacl2溶液与大肠杆菌菌液的体积比为0.2:1。步骤(ii)中所述的离心的条件优选为:4000g离心10min。步骤(ii)中所述的冰上放置的时间优选为10min。步骤(ii)中所述的大肠杆菌wm3064感受态细胞的保存温度优选为-80℃。步骤(a)中所述的转化优选为通过如下步骤实现:(i)将自杀型质粒(ptpm-xy质粒)加入到大肠杆菌wm3064感受态细胞中,混匀后冰浴30min、42℃水浴45s、冰浴3min,再加入含有二氨荃庚二酸的培养基进行培养,得到菌液;(ii)将步骤(i)中得到的菌液涂布在含有二氨荃庚二酸和亚碲酸钠的培养基中进行培养,得到转化成功的wm3064/ptpm-xy,即荧光标记的大肠杆菌wm3064/ptpm-xy。步骤(i)中所述的自杀型质粒(ptpm-xy质粒)与大肠杆菌wm3064感受态细胞的体积比优选为1:10。步骤(i)中所述的培养的条件优选为:37℃培养1h。步骤(i)中所述的培养基中的二氨荃庚二酸的浓度为50μg/ml。步骤(i)中所述的培养基优选为sob培养基或lb肉汤培养基。步骤(ii)中所述的培养的条件优选为:37℃培养16~24h。步骤(ii)中所述的培养基中的二氨荃庚二酸的浓度为50μg/ml,亚碲酸钠的浓度为25μg/ml。步骤(ii)中所述的培养基优选为lb琼脂培养基。步骤(b)中所述的目标菌株为嗜麦芽寡氧单胞菌,大肠杆菌,肺炎克雷伯菌,沙门氏菌和阴沟肠杆菌中的至少一种;优选为嗜麦芽寡氧单胞菌qb230,阴沟肠杆菌atcc13047,沙门氏菌atcc14028,肺炎克雷伯菌atcc700603或及大肠杆菌c600(e.colic600)。步骤(b)中所述的培养大肠杆菌wm3064/p26-rp4-lux所用的培养基为含有二氨荃庚二酸(dap)的lb肉汤培养基;优选为含50μg/ml二氨荃庚二酸(dap)的lb肉汤培养基。步骤(b)中所述的培养目标菌株所用的培养基为lb肉汤培养基。步骤(c)中所述的培养基为含有二氨荃庚二酸(dap)的lb琼脂培养基;优选为含50μg/ml二氨荃庚二酸(dap)的lb琼脂培养基。步骤(c)中所述的大肠杆菌wm3064/ptpm-xy和目标菌株的体积比优选为1:1。步骤(c)中所述的培养的时间为2~8h;优选为4小时。步骤(d)中所述的培养的条件为:37℃培养20小时。步骤(d)中所述的亚碲酸盐优选为亚碲酸钠。步骤(d)中所述的单药板(含有亚碲酸盐的平板)中亚碲酸盐的浓度为25μg/ml。步骤(d)中所述的原抗性筛选标记药物优选为美罗培南。步骤(d)中所述的双药板(含有亚碲酸盐和原抗性筛选标记药物的平板)中亚碲酸盐的浓度为25μg/ml,原抗性筛选标记药物的浓度为2μg/ml。所述的基于mariner转座突变技术构建耐药突变菌株的方法,还包括将步骤(d)中得到的耐药突变菌株进一步鉴定的步骤;具体为:提取耐药突变菌株的基因组,然后采用二轮半pcr方法进行扩增,然后送测序公司测序,并将测序结果同ncbi数据库比对,研究突变位点,探究突变的基因。一株耐药突变菌株,通过上述任一项所述的方法构建得到。所述的耐药突变菌株在基因工程
技术领域:
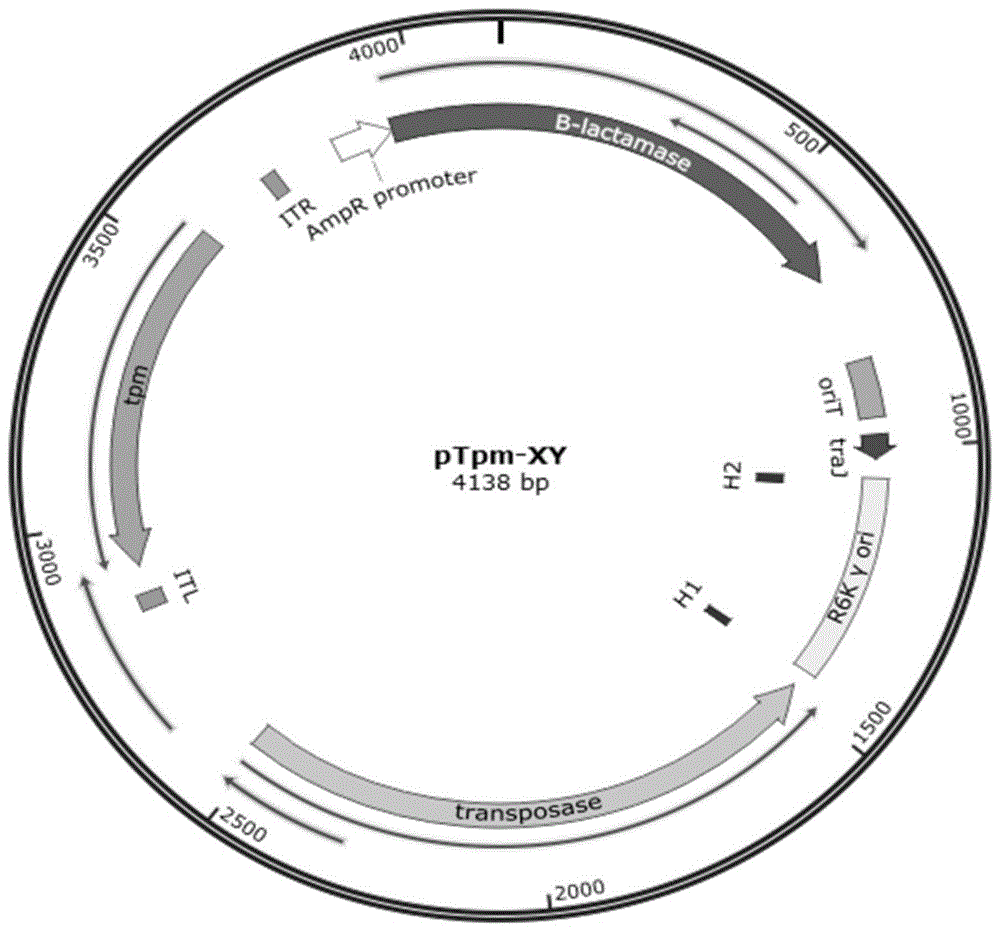
中的应用。所述的基于mariner转座子构建自杀型质粒的方法或基于mariner转座突变技术构建耐药突变菌株的方法在构建耐药菌株、药动药效学评价或筛选药物中的应用。本发明方法包含一个自杀型质粒(即ptpm-xy质粒:由复制起始位点r6k,mariner转座子,tpmr和ampr抗性基因以及oritrp4接合转移区),宿主为含有pir基因的e.coliwm3064,能够编码自杀质粒所需的π蛋白,以及可作为接合型自杀质粒的供体。该质粒可以在不同革兰氏阴性菌之间进行接合转移,该质粒含有一个转座单元,该转座单元由一个分别为29bp的反向末端重复序列itr和个itl单元夹着一个亚碲酸钠耐药基因(tpm)组成。由于临床细菌不能编码π蛋白,故该质粒通过自然接合转移的方式进入临床细菌细胞内后不能复制会随之凋亡,而转座就会发生。两个itl单元形成的转座子从质粒上脱落并高效率的随机插入宿主的质粒或染色体中,而自杀质粒的转座酶基因mariner不会转移,故转座子不会再发生跳跃,因此通过我们的方法可以获得含有itl和itr转座单元的突变株,突变株对亚碲酸钠耐药。本发明相对于现有技术具有如下的优点及效果:1、与现有研究新基因的的技术相比,原技术工作量大,成本高,通量低,精准性差,无法高效筛选到目标基因。本发明操作简单,将营养缺陷型菌株e.coliwm3064,mariner转座子,rp4接合转移基因结合了起来,构建了高通量研究新基因的系统。基于该体系,可以在一天的时间内构建上千株突变株。2、本发明中的e.coliwm3064,分别转入携带tpmr耐药基因的ptpm-xy质粒,该细菌为营养缺性型细菌,只有在培养环境中加入一种氨基酸,即二氨荃庚二酸(dap),该菌便可以增殖。而通常培养基中不含有这一氨基酸,故在不含这一氨基酸的环境中,该细菌不可增殖。3、本发明中,一般工程菌和临床菌不含有tpmr耐药基因也不含有pir基因。故只要ptpm-xy质粒转入目标菌株后,ptpm-xy无法存在,只能以itl和itr单元形成的转座子从质粒上脱落并高效率的随机插入宿主的质粒或染色体使得目标菌株由亚碲酸钠敏感(tpms)突变为亚碲酸钠耐药(tpmr)。4、本发明将e.coliwm3064/ptpm-xy和目标菌株混合培养后涂布选择性培养基,即可筛选到新构建的突变菌株,在一天内可以高通量的获得上千株转座突变株,降低劳动强度。6、本发明的方法廉价,快速,高效,其普适性强,可以广泛适用于所有未知基因的研究。本发明研究新基因的发明能够提供一个高通量的获取新基因的新的技术。附图说明图1是本发明ptpm-xy质粒的结构图。图2是实施例1中在含有亚碲酸钠的lb琼脂单药板上,以及在含有亚碲酸盐和美罗培南的lb琼脂双药板上的菌株生长情况图(图中:a为lb琼脂单药板;b为lb琼脂双药板)。图3是本发明所用两轮半pcr原理图。图4是pcr产物的电泳结果图;其中,m为dnamarker;泳道1~3均为pcr产物。图5是基于本发明的测序结果比对图。具体实施方式下面结合实施例对本发明作进一步详细的描述,但本发明的实施方式不限于此。除非特别说明,本发明采用的试剂、方法和设备为本
技术领域:
常规试剂、方法和设备。除非特别说明,本发明所用试剂、培养基、生物材料等均可通过市售获得。实施例1:基于mariner转座突变技术筛选耐碳青霉烯酶类抗生素新基因1、突变菌株的构建(1)ptpm-xy质粒的构建:ptpm-xy质粒的结构图如图1所示,ptpm-xy质粒由复制子r6k,mariner转座子,tpmr和ampr抗性基因以及traj负责接合转移的基因组成,其构建过程如下:以pcat-marinerr质粒(购自上海吉凯基因科技有限公司)为母体(模板),用引物at-f和catr(at-f:ccagaactcatgttgcatggtat;catr:ccaagttctgagcctttttgcg)进行pcr扩增,得到3443bp的扩增产物(作为质粒骨架);然后以擎科生物科技有限公司合成的含有tpm基因的puc57-tpm质粒为模板,用引物tpm-f和tpm-r(tpm-f:tcagaacttggcagtgagtatcca;tpm-r:atgcaacatgagttctggcat)扩增tpm抗性基因,dpni酶消化模板质粒(puc57-tpm质粒);再用南京诺唯赞生物科技有限公司的同源重组试剂盒onestepcloningkit将两个片段同源重组,从而构建出ptpm-xy质粒。将ptpm-xy质粒转化e.coliwm3064(购自广州成康生物科技有限公司)感受态细胞,再用是lb琼脂培养基(含亚碲酸钠25μg/ml)筛选出单菌落,pcr验证正确的转化子,从而构建出e.coliwm3064/ptpm-xy。其中,e.coliwm3064感受态细胞的制备方法如下:①、挑e.coliwm3064单菌落于3~5mllb肉汤培养基中(培养基购自上海源叶生物科技有限公司,并添加终浓度为50μg/ml的二氨荃庚二酸(dap)),37℃过夜培养;②、按体积比1:50~100转接至100mllb肉汤培养基中(添加终浓度为50μg/ml的二氨荃庚二酸(dap),37℃培养2~3h,至od600约0.5;③、菌液分装于50ml离心管,冰上预冷10min,4℃,4000g,15min;同时提前将cacl2溶液置于冰上预冷;④、弃上清,加入10ml(约0.2倍菌液体积)预冷的cacl2溶液,轻轻地充分悬浮细胞;⑤、4℃,4000g,离心10min,弃上清;⑥、加入4ml预冷的含15%(v/v)甘油的cacl2溶液,轻悬细胞,冰上放置10min;⑦、分装100μl于1.5ml离心管,-80℃保存。ptpm-xy质粒转化e.coliwm3064感受态细胞的过程如下:①、在100μle.coliwm3064感受态细胞中加入100ngptpm-xy质粒(体积小于10μl);②、混匀后,冰浴30min;③、42℃水浴45s;④、冰浴3min,加入890μlsob培养基或lb肉汤培养基(培养基中添加终浓度为50μg/ml的二氨荃庚二酸(dap)),37℃培养1h;⑤、取100μl菌液涂布于含有终浓度为50μg/ml的二氨荃庚二酸(dap)以及25μg/ml亚碲酸钠的lb琼脂培养基中(同时以没有转化的感受态细胞为阴性对照),37℃培养16~24小时。2、突变菌株的筛选嗜麦芽寡氧单胞菌qb230购自广州成康生物科技有限公司,通过pcr鉴定其不含有imp等碳青酶烯酶耐药相关基因,其具体步骤如下:(1)划线培养嗜麦芽寡氧单胞菌qb230在lb琼脂培养基(含2μg/ml美罗培南),培养及上随机挑选1株肉眼可见的菌落,将单菌落挑入到50μlddh2o中,吹打均匀,将ep管在100℃煮10min后,室温12000rpm离心5min,将上清40μl转移到新管中,作为模板备用。设计引物如下,进行菌落pcr检测:针对imp(肌醇单磷酸酶)基因设计上下游引物imp-f和imp-r;同时,针对spm(亚磷酸脱氢酶)基因、aim基因、vim基因、oxa基因、gim基因、bic基因、sim基因、ndm基因、dim基因和kpc基因分别设计上下游引物,引物序列如表2所示。(2)pcr(东盛生物)反应体系如下表1所示。表1.pcr反应体系体系组分体积(μl)灭菌ddh2o82×pcrmix10引物10.5引物20.5模板dna1totalvolume20(3)pcr反应运行参数pcr反应运行参数:(94℃,5min)+{(94℃,30s)+(55℃,30s)+(72℃,2min)}×30+(72℃,10min)(4)pcr产物经1%的琼脂糖电泳实验分析,并拍照。表2.引物基因序列3、突变菌株的构建(1)将e.coliwm3064/ptpm-xy和目标菌株(嗜麦芽寡氧单胞菌qb230,购自广州成康生物科技有限公司)分配用lb肉汤培养基增菌培养(37℃、200rpm)至生长对数期;其中e.coliwm3064/ptpm-xy的培养条件中添加终浓度为50μg/ml二氨荃庚二酸(dap)。(2)将生长对数期的e.coliwm3064/ptpm-xy和目标菌(嗜麦芽寡氧单胞菌qb230)按体积比1:1混合后滴在lb琼脂培养基(含50μg/ml的二氨荃庚二酸(dap))上,静置培养4小时。(3)将步骤(2)中静置培养后的孵育物用生理盐水稀释合适梯度(103cfu/ml)后,涂布于含有25μg/ml亚碲酸钠的lb琼脂培养基中,37℃培养20小时。(4)将步骤(3)中在含有25μg/ml亚碲酸盐的lb琼脂培养基上长出的所有单菌落分别增菌培养(lb肉汤培养基,37℃培养8小时)后取1μl滴于含有25μg/ml亚碲酸钠的lb琼脂的单药板和含有25μg/ml亚碲酸盐/2μg/ml美罗培南(购自上海源叶生物科技有限公司)的双药板上,37℃培养20小时。(5)挑选双药板上不长而在单药板长出的嗜麦芽寡氧单胞菌qb230突变株,如图2所示。按照天根生化科技有限公司细菌基因组dna提取试剂盒中提细菌基因组的步骤,提取qb230突变株细菌基因组。3、两轮半pcr方法扩增如图3所示,采用两轮半pcr方法扩增基因组,具体步骤如下:(1)引物设计及合成构建序列明确突变体文库最重要的是突变株定位。为了获得每一株突变株在基因组上的插入位点,需要用pcr的方法扩增出插入位点的侧翼序列。本实验所采用的方法为两轮半任意pcr,该方法最关键的部分在于设计引物,其引物序列见表1。表1.本发明筛选新基因所用到的随机引物序列名称引物序列(5’-3’)ab1ggccacgcgtcgactagtacnnnnnnnnnnacgccab2ggccacgcgtcgactagtacnnnnnnnnnncctggab3ggccacgcgtcgactagtacnnnnnnnnnncctcgabsggccacgcgtcgactagtacitpmf3ccaagttctgagcctttttgcgitpmr3tcaacggctgatatcactgacg表中,“n”表示“a或c或g或t”。引物ab1、ab2、ab3为随机引物,引物结构分为三段:第一段部分引物都相同,为20个碱基的特异性序列;第二段为10个随机碱基;第三段为5个特异性碱基,这一段序列不相同,并且是特异性序列,该段序列的选取是整个pcr中最关键的部分,需要考虑模板菌株的基因组中gc含量,以及五个特异性碱基在基因组上随机出现的频率,使用embosscompseq软件来找寻合适的特异性碱基。引物abs序列与ab1、ab2、ab3中第一段20个特异性碱基相同。引物itpmf3和itpmr3为低gc含量的高退火温度的特异性引物。第一轮pcr退火温度较低,使用的ab1、ab2、ab3中的10个随机碱基,能够最大程度的扩增出插入位点的侧翼序列。第二轮pcr以第一轮产物为模板,采用高退火温度和高特异性的巢氏引物,能够特异性的扩增出插入位点侧翼的序列。(2)两轮半任意pcr以提取qb230突变株细菌基因组为模板进行第一轮反应,反应体系如表2所示:表2.第一轮pcr反应体系体系组分体积2×pcrmix10μlab1引物0.2μm(终浓度)ab2引物0.2μm(终浓度)ab3引物0.2μm(终浓度)itpmf3引物0.6μm(终浓度)模板dna1灭菌ddh2o加至20pcr反应运行参数:预变性,95℃,5min;(变性,95℃,30s;退火,42~36℃,30s,每个循环减1℃;延伸,72℃,3min),6个循环;(变性,95℃,30s;退火,58℃,30s;延伸,72℃,3min),26个循环;补延伸,72℃,10min;最终保温,4℃;将第一轮pcr产物,使用qiangendna纯化试剂盒纯化,去掉第一轮的引物以及缓冲液成分,作为第二轮pcr的模板dna,反应体系如表3所示:表3第二轮pcr反应体系体系组分体积(μl)2×pcrmix25abs引物0.5μm(终浓度)itpmf3引物0.5μm(终浓度)模板dna1灭菌ddh2o加至50pcr反应运行参数:预变性,98℃,3min;(变性,98℃,10s;退火,58℃,30s;延伸,72℃,1min),30个循环;补延伸,72℃,10min;最终保温,4℃;第二轮pcr产物取5μl跑电泳,如图4所示,将有亮带的pcr反应液送生工测序生物公司测序,测序引物用itpmf3。使用blast比对分析测序结果,确定插入基因位置,blast比对分析结果如图5所示,可知通过mariner转座突变技术,将qb230的细菌原yh67_01495基因失活了,该基因原功能为产β-内酰胺酶(b-lactamase),使得细菌耐亚碲酸钠药物。实施例2基于mariner突变技术构建突变文库为了研究该突变技术能否用于构建其他菌株突变文库,本实施例选择4株标准菌:阴沟肠杆菌atcc13047、沙门氏菌atcc14028、肺炎克雷伯菌atcc700603、以及大肠杆菌c600(e.colic600),以上均购自广州成康生物科技有限公司。1.实验菌株对亚碲酸盐敏感性测定将实验菌株划线接种于麦康凯琼脂培养基(购自广州环凯生物科技有限公司)中进行培养,第二天取单菌落传代划线培养于含25μg/ml亚碲酸钠的lb琼脂培养基(购自广州环凯生物科技有限公司)中,发现该琼脂板上无菌落长出,证明以上菌株均对亚碲酸盐敏感。2、突变菌株的构建(1)将e.coliwm3064/ptpm-xy和目标菌株(即上述4株实验菌株)分配用lb肉汤培养基增菌培养至生长对数期;其中e.coliwm3064/ptpm-xy的培养条件中添加终浓度为50μg/ml二氨荃庚二酸(dap)。(2)将生长对数期的e.coliwm3064/ptpm-xy和目标菌按体积比1:1混合后滴在lb琼脂培养基(含50μg/ml的二氨荃庚二酸(dap))上,静置培养4小时。(3)将步骤(2)中静置培养后的孵育物用生理盐水稀释合适梯度后(103cfu/ml),涂布于含有25μg/ml亚碲酸盐的lb琼脂培养基中,37℃培养20小时。(4)将步骤(3)中在含有25μg/ml亚碲酸盐的lb琼脂培养基上长出的所有单菌落分别增菌培养(lb肉汤培养基,37℃培养8小时)后取1μl滴于含有25μg/ml亚碲酸盐的lb琼脂的单药板和含有25μg/ml亚碲酸盐/2μg/ml美罗培南(购自上海源叶生物科技有限公司)的双药板上,37℃培养20小时)。(5)挑选双药板上不长而单药板长出的突变株进行pcr验证。实验结果表明,本发明成功获得以上四株细菌的突变株。说明本发明的方法可用于以上四种细菌的相关基因的筛查。上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。序列表<110>华南农业大学<120>基于mariner转座子构建自杀型质粒及耐药突变菌株的方法<160>32<170>siposequencelisting1.0<210>1<211>23<212>dna<213>人工序列(artificialsequence)<220><223>引物at-f<400>1ccagaactcatgttgcatggtat23<210>2<211>22<212>dna<213>人工序列(artificialsequence)<220><223>引物catr<400>2ccaagttctgagcctttttgcg22<210>3<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物tpm-f<400>3tcagaacttggcagtgagtatcca24<210>4<211>21<212>dna<213>人工序列(artificialsequence)<220><223>引物tpm-r<400>4atgcaacatgagttctggcat21<210>5<211>22<212>dna<213>人工序列(artificialsequence)<220><223>imp-f<400>5ggaatagagtggcttaaytctc22<210>6<211>20<212>dna<213>人工序列(artificialsequence)<220><223>imp-r<400>6ggtttaayaaaacaaccacc20<210>7<211>20<212>dna<213>人工序列(artificialsequence)<220><223>spm-f<400>7aaaatctgggtacgcaaacg20<210>8<211>20<212>dna<213>人工序列(artificialsequence)<220><223>spm-r<400>8acattatccgctggaacagg20<210>9<211>20<212>dna<213>人工序列(artificialsequence)<220><223>aim-f<400>9ctgaaggtgtacggaaacac20<210>10<211>19<212>dna<213>人工序列(artificialsequence)<220><223>aim-r<400>10gttcggccacctcgaattg19<210>11<211>19<212>dna<213>人工序列(artificialsequence)<220><223>vim-f<400>11gatggtgtttggtcgcata19<210>12<211>17<212>dna<213>人工序列(artificialsequence)<220><223>vim-r<400>12cgaatgcgcagcaccag17<210>13<211>20<212>dna<213>人工序列(artificialsequence)<220><223>oxa-f<400>13gcgtggttaaggatgaacac20<210>14<211>20<212>dna<213>人工序列(artificialsequence)<220><223>oxa-r<400>14catcaagttcaacccaaccg20<210>15<211>20<212>dna<213>人工序列(artificialsequence)<220><223>gim-f<400>15tcgacacaccttggtctgaa20<210>16<211>20<212>dna<213>人工序列(artificialsequence)<220><223>gim-r<400>16aacttccaactttgccatgc20<210>17<211>20<212>dna<213>人工序列(artificialsequence)<220><223>bic-f<400>17tatgcagctcctttaagggc20<210>18<211>20<212>dna<213>人工序列(artificialsequence)<220><223>bic-r<400>18tcattggcggtgccgtacac20<210>19<211>19<212>dna<213>人工序列(artificialsequence)<220><223>sim-f<400>19tacaagggattcggcatcg19<210>20<211>20<212>dna<213>人工序列(artificialsequence)<220><223>sim-r<400>20taatggcctgttcccatgtg20<210>21<211>20<212>dna<213>人工序列(artificialsequence)<220><223>ndm-f<400>21ggtttggcgatctggttttc20<210>22<211>20<212>dna<213>人工序列(artificialsequence)<220><223>ndm-r<400>22cggaatggctcatcacgatc20<210>23<211>21<212>dna<213>人工序列(artificialsequence)<220><223>dim-f<400>23gcttgtcttcgcttgctaacg21<210>24<211>20<212>dna<213>人工序列(artificialsequence)<220><223>dim-r<400>24cgttcggctggattgatttg20<210>25<211>20<212>dna<213>人工序列(artificialsequence)<220><223>kpc-fm<400>25cgtctagttctgctgtcttg20<210>26<211>20<212>dna<213>人工序列(artificialsequence)<220><223>kpc-rm<400>26cttgtcatccttgttaggcg20<210>27<211>35<212>dna<213>人工序列(artificialsequence)<220><223>ab1<220><221>misc_feature<222>(21)..(21)<223>nisa,c,g,toru<220><221>misc_feature<222>(22)..(22)<223>nisa,c,g,toru<220><221>misc_feature<222>(23)..(23)<223>nisa,c,g,toru<220><221>misc_feature<222>(24)..(24)<223>nisa,c,g,toru<220><221>misc_feature<222>(25)..(25)<223>nisa,c,g,toru<220><221>misc_feature<222>(26)..(26)<223>nisa,c,g,toru<220><221>misc_feature<222>(27)..(27)<223>nisa,c,g,toru<220><221>misc_feature<222>(28)..(28)<223>nisa,c,g,toru<220><221>misc_feature<222>(29)..(29)<223>nisa,c,g,toru<220><221>misc_feature<222>(30)..(30)<223>nisa,c,g,toru<400>27ggccacgcgtcgactagtacnnnnnnnnnnacgcc35<210>28<211>35<212>dna<213>人工序列(artificialsequence)<220><223>ab2<220><221>misc_feature<222>(21)..(21)<223>nisa,c,g,toru<220><221>misc_feature<222>(22)..(22)<223>nisa,c,g,toru<220><221>misc_feature<222>(23)..(23)<223>nisa,c,g,toru<220><221>misc_feature<222>(24)..(24)<223>nisa,c,g,toru<220><221>misc_feature<222>(25)..(25)<223>nisa,c,g,toru<220><221>misc_feature<222>(26)..(26)<223>nisa,c,g,toru<220><221>misc_feature<222>(27)..(27)<223>nisa,c,g,toru<220><221>misc_feature<222>(28)..(28)<223>nisa,c,g,toru<220><221>misc_feature<222>(29)..(29)<223>nisa,c,g,toru<220><221>misc_feature<222>(30)..(30)<223>nisa,c,g,toru<400>28ggccacgcgtcgactagtacnnnnnnnnnncctgg35<210>29<211>35<212>dna<213>人工序列(artificialsequence)<220><223>ab3<220><221>misc_feature<222>(21)..(21)<223>nisa,c,g,toru<220><221>misc_feature<222>(22)..(22)<223>nisa,c,g,toru<220><221>misc_feature<222>(23)..(23)<223>nisa,c,g,toru<220><221>misc_feature<222>(24)..(24)<223>nisa,c,g,toru<220><221>misc_feature<222>(25)..(25)<223>nisa,c,g,toru<220><221>misc_feature<222>(26)..(26)<223>nisa,c,g,toru<220><221>misc_feature<222>(27)..(27)<223>nisa,c,g,toru<220><221>misc_feature<222>(28)..(28)<223>nisa,c,g,toru<220><221>misc_feature<222>(29)..(29)<223>nisa,c,g,toru<220><221>misc_feature<222>(30)..(30)<223>nisa,c,g,toru<400>29ggccacgcgtcgactagtacnnnnnnnnnncctcg35<210>30<211>20<212>dna<213>人工序列(artificialsequence)<220><223>abs<400>30ggccacgcgtcgactagtac20<210>31<211>22<212>dna<213>人工序列(artificialsequence)<220><223>itpmf3<400>31ccaagttctgagcctttttgcg22<210>32<211>22<212>dna<213>人工序列(artificialsequence)<220><223>itpmr3<400>32tcaacggctgatatcactgacg22当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1