小麦远缘杂交后代小片段易位系中发掘外源功能候选基因的方法与流程
本发明涉及基因组学、转录组学及生物信息学领域,具体地说,涉及小麦远缘杂交后代小片段易位系中发掘外源功能候选基因的方法。
背景技术:
从小麦进化历史和育种进程可以看出,远缘杂交是拓宽小麦遗传基础,创制新种质,实现小麦高产、稳产育种的有效途径之一。国内外学者已经实现了小麦族的所有属与小麦间的杂交,获得了大量的且具有可利用价值的小麦新种质,从这些新种质中发掘新的功能基因并加以利用,将在小麦抗性育种中发挥着举足轻重的作用。由于多数小麦近缘植物没有参考基因组序列,且在小麦背景下,对外源功能基因进行图位克隆的研究相对困难。有研究人员通过突变体筛选、抗病基因富集测序和易位染色体分拣测序组装等技术,成功从簇毛麦的6v染色体上克隆了抗白粉病基因pm21。类似研究工作耗时长、工作量大、成本高,且相关的研究主要集中在抗病相关的基因,但与小麦复杂的数量性状相关的基因克隆还没有相关的研究报道。因此,如何有效地在小麦背景下快速筛选外源功能基因,特别是与产量等复杂性状相关的基因,是亟需解决的问题。
技术实现要素:
本发明的目的是提供小麦远缘杂交后代小片段易位系中发掘外源功能候选基因的方法。
为了实现本发明目的,本发明提供小麦远缘杂交后代小片段易位系中发掘外源功能候选基因的方法,包括以下步骤:
a、小麦近缘植物的全长转录组测序:获取小麦近缘植物多个组织且包含所研究的目的性状发生前和发生时的组织,分别提取各个组织mrna且等量混合;为了获得小麦近缘植物的全长转录组参考序列,利用第三代测序技术和平台对混合样品进行全长转录组的建库和测序。
b、小麦远缘杂交后代小片段易位系和小麦受体的转录组测序:分别获取小麦远缘杂交后代小片段易位系和小麦受体植株多个组织且包含所研究的目的性状发生前和发生时的组织,分别提取各个组织mrna且等量混合;为了对转录本的表达进行定量,利用第二代测序技术和平台对各自的混合样品进行高通量转录组的建库和测序,分别得到小麦受体的转录本序列和易位系的转录本测序数据。
c、易位系中外源染色体片段中特异表达转录本分析:对小麦近缘植物的全长转录组测序数据进行生物信息学分析(生物信息学分析包括:使用isoseq3软件处理原始测序数据。具体步骤如下:首先,使用ccs算法进行分类,产生每个零模式波导孔中的共有序列,保留具有至少一个完整通道的序列用于后续分析;其次,使用lima算法获得全长序列;最后,进行聚类和矫正),获得非嵌合且全长的转录本序列,去除冗余后,与小麦参考基因组序列和注释文件进行整合作为总的参考序列;将小麦受体的转录本序列以及易位系的转录本序列分别与所述总的参考序列进行比对,并过滤,去重复,仅保留唯一比对的序列比对结果,然后统计比对到每个转录本上的序列数目,最后进行小麦受体和易位系之间转录本的序列差异表达显著性检验,根据易位系和小麦受体之间转录本的序列比对数目,使用参数log2(差异倍数)≤-4和假阳性率<0.05进行过滤(为了获得可信的以及统计上显著差异表达的转录本,必须控制表达的差异倍数以及多重检验的p-value),获得易位系中外源染色体片段中特异表达的转录本,作为候选转录本序列。
d、分子标记开发:将获得的候选转录本序列与小麦参考基因序列进行比对,根据比对结果找出候选转录本与小麦基因组上同源基因之间的差异,根据差异序列设计并开发分子标记及其检测引物;利用设计的引物在小麦近缘植物、小麦受体和易位系中进行pcr验证,确定所获得的外源染色体片段中特异表达的转录本的真实性。
e、候选基因预测:对于步骤d中获得的真实候选转录本序列,与小麦参考基因序列进行比对,进行比较基因组学分析,构建易位系中外源染色体片段与小麦基因组间的比较基因组学图谱;对共线性候选区间内的基因进行功能注释,获得外源功能候选基因。
可选地,步骤c中,对小麦近缘植物的全长转录组测序数据进行生物信息学分析,获得非嵌合且全长的转录本序列,利用cd-hit-est软件去冗余,然后与小麦参考基因组序列和注释文件进行整合作为总的参考序列。
利用star软件将小麦受体的转录本序列以及易位系的转录本序列分别与所述总的参考进行序列比对,并过滤,去重复,仅保留唯一比对的序列比对结果。然后,用featurecounts软件统计比对到每个转录本上的序列数目。最后,用deseq2软件进行小麦受体和易位系之间转录本的差异表达显著性检验。
步骤d中,将获得的候选转录本序列通过blast与小麦基因序列进行比对。
步骤d中,所开发的分子标记包括但不限于est、kasp标记。
在本发明的一个具体实施方式中,提供小麦-冰草易位系中发掘外源功能候选基因的方法,包括以下步骤:
s1、冰草的全长转录组测序:获取冰草z559根、茎、叶、幼穗、籽粒多个组织,分别提取各个组织mrna且等量混合,构建pacbio测序文库,利用pacbiosequel平台的2个cell对混合样品进行全长转录组测序。共获得了约15g大小的测序数据量。
s2、小麦受体fukuhokumugi和易位系的转录组测序:分别获取fukuhokumugi和易位系植株的根、茎、叶、籽粒(包括灌浆前、中、后期)多个组织,然后提取各个组织mrna且等量混合,对fukuhokumugi和易位系各自的混合样品构建高通量测序文库,并使用illuminahiseq2500高通量测序平台进行转录组测序;
s3、易位系中冰草外源染色体易位片段中特异表达的转录本分析:整合冰草全长转录组测序数据和小麦参考基因组序列作为总的参考序列,用star软件将fukuhokumugi和易位系的转录本序列与所述总的参考序列进行比对,仅保留唯一比对的序列比对结果,然后用featurecounts软件分别计算fukuhokumugi和易位系中比对到每个转录本上的序列数目,最后使用deseq2软件进行fukuhokumugi和易位系之间转录本的序列差异表达显著性检验,用log2(差异倍数)≤-4且假阳性率<0.05进行过滤,获得易位系中外源染色体片段中特异表达的转录本,作为候选转录本序列。共获得普冰3035中冰草外源染色体易位片段中特异表达的12个转录本(表1)。
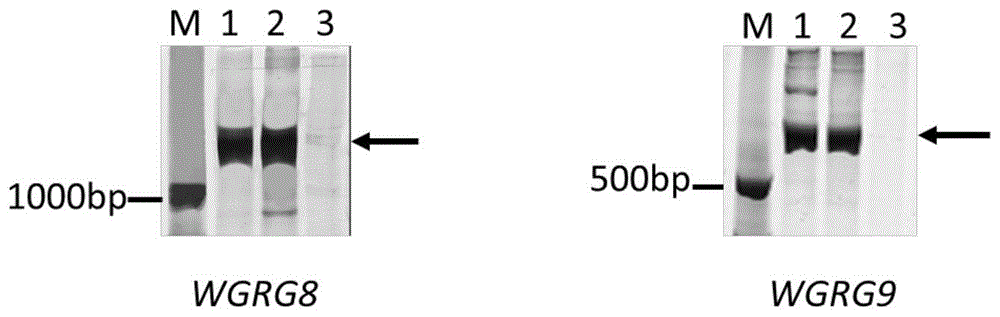
s4、分子标记开发:将获得的候选转录本序列与小麦参考基因序列进行比对,并根据比对结果找出候选转录本与小麦a/b/d基因组上同源基因之间的差异,根据差异序列设计est标记及其检测引物;利用设计的est标记在冰草z559、fukuhokumugi和易位系中进行pcr验证,分别对每个候选转录本开发了相对应的多态性分子标记(图1),从而验证外源染色体片段中特异表达的转录本的真实性。
s5、候选基因预测:对于步骤s4中获得的真实候选转录本序列,与小麦参考基因序列进行比对,进行比较基因组学分析,构建易位系中外源染色体片段与小麦a/b/d基因组间的比较基因组学图谱(图2);对共线性候选区间内的基因进行功能注释,获得外源功能候选基因。
本发明中,所述小麦-冰草易位系为普冰3035。
步骤s1还包括对冰草全长转录组测序数据进行过滤、质控;具体方法为主要使用isoseq3软件处理原始测序数据,首先,使用ccs算法进行分类,产生每个零模式波导孔中的共有序列,保留具有至少一个完整通道的序列用于后续分析;其次,使用lima算法获得全长序列;最后,进行聚类和矫正获得高质量的冰草全长转录本。
经过对测序数据进行过滤、质量控制等分析,共得到了44372个全长非冗余的冰草转录本序列。
步骤s2还包括对原始测序数据进行质控、去污染和接头,得到高质量的测序数据;具体方法为:去除两端测序质量值小于20的碱基,小于25bp的测序读长将被去除,过滤使用trimmomatic软件执行。
步骤s4中所述est标记包括wgrg8和wgrg9,它们的检测引物分别如下(seqidno:3-6):
wgrg8_f:5′-tgccagtggtgaccaatgca-3′;
wgrg8_r:5′-acttggggaagagtctcact-3′;
wgrg9_f:5′-tccaaatcctccagcaaatc-3′;
wgrg9_r:5′-cccgagaccgagcactatac-3′。
步骤s5获得的外源功能候选基因包括与小麦千粒重相关的候选基因transcript/2、transcript/4,它们的核苷酸序列分别如seqidno:1和2所示。
其中,转录本transcript/2在水稻中的同源基因为asl2,其编码质体核糖体蛋白l21,突变后会导致叶绿体发育缺陷和幼苗死亡,主要影响植株的光合作用;转录本transcript/4在水稻中的同源基因为osubp15/lg1,其与gw2基因相互作用,是水稻粒宽和籽粒大小的正调节因子。因此基于此方法从小麦-冰草易位系普冰3035中发掘的冰草特异转录本transcript/2和transcript/4可以作为普冰3035中控制千粒重和粒长的候选基因。
借由上述技术方案,本发明至少具有下列优点及有益效果:
本发明提供一种仅需对小麦远缘杂交衍生的小片段易位系及其小麦受体和野生近缘种供体三个样品进行转录组测序,结合生物信息学分析、比较基因组学和功能标记开发验证,可实现从小麦远缘杂交衍生系中快速挖掘外源功能候选基因。首先对小麦野生近缘种多个组织的混合样品进行全长转录组测序,并与小麦参考基因组序列进行整合作为总的参考序列;其次对小麦远缘杂交衍生的小片段易位系和小麦受体进行高通量转录组测序,鉴定在衍生系中特异表达的外源转录本;再次进行与小麦基因组的比较基因组学分析,开发功能标记;最后结合功能注释确定与目的性状相关的外源功能候选基因。本发明不依赖小麦近缘植物的参考基因组序列,实现了低成本且高效地筛选功能候选基因,为功能基因的克隆奠定基础。
附图说明
图1为本发明实施例1中根据转录本transcript/2开发的多态性标记wgrg8和transcript/4开发的多态性标记wgrg9的聚丙烯酰胺凝胶电泳图。其中,m:dnamarker;1:冰草z559;2:普冰3035;3:fukuhokumugi。
图2为本发明实施例1中普冰3035中冰草6p染色体易位片段与小麦6a染色体间的比较基因组学图谱。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例均按照常规实验条件,如sambrook等分子克隆实验手册(sambrookj&russelldw,molecularcloning:alaboratorymanual,2001),或按照制造厂商说明书建议的条件。实施例1小麦-冰草易位系中发掘外源功能候选基因的方法
前期研究表明,普冰3035(ti1as-6pl-1as·1al,遗传背景为小麦受体品系fukuhokumugi)为小麦-冰草易位系,其来自上麦近缘植物冰草6p染色体上的易位片段能够显著提高小麦的千粒重。本实施例提供了该小麦背景下外源染色体片段中功能候选基因快速发掘的方法,在筛选小麦-冰草小片段插入易位系普冰3035中与小麦千粒重相关的候选功能基因的实例。
具体方法为:
a、小麦近缘植物冰草的全长转录组测序:
为了能够获得尽可能多的冰草全长转录组序列,获取冰草z559根、茎、叶、幼穗、籽粒多个组织,分别提取各个组织mrna且等量混合,最后构建pacbio的测序文库,使用pacbiosequel平台的2个cell对混合样品进行全长转录组测序,共获得约15g大小的测序数据量。使用isoseq3软件处理原始测序数据。具体步骤如下:首先,使用ccs算法进行分类,产生每个零模式波导孔中的共有序列,保留具有至少一个完整通道的序列用于后续分析;其次,使用lima算法获得全长序列;最后,进行聚类和矫正,获得高44372个全长非冗余的高质量冰草全长转录本。
b、小麦受体fukuhokumugi和衍生系普冰3035的转录组测序:
分别获取小麦受体fukuhokumugi和普冰3035植株根、茎、叶、籽粒(包括灌浆前、中、后期)多个组织,然后提取各个组织mrna且等量混合,最后对小麦受体fukuhokumugi和普冰3035两个混合的样品构建高通量测序文库,使用illuminahiseq2500高通量测序平台进行转录组测序。对原始测序数据进行质控、去污染和接头,去除两端测序质量值小于20的碱基,小于25bp的测序读长将被去除,过滤使用trimmomatic软件执行,得到高质量的测序数据。
c、普冰3035中冰草外源染色体易位片段中特异表达的转录本分析:
整合冰草非冗余全长转录组序列和小麦参考基因组(iwgscrefseqv1.0)作为总的参考序列,使用star软件将小麦fukuhokumugi和普冰3035的转录组测序序列与参考序列进行比对,只保留唯一比对的序列比对结果,然后使用featurecounts软件分别计算fukuhokumugi和普冰3035中比对到每个转录本上的序列数目,最后使用deseq2进行fukuhokumugi和普冰3035间转录本的差异表达显著性检验,用log2(差异倍数)或log2(变化倍数)≤-4且假阳性率<0.05进行过滤,共获得普冰3035中冰草外源染色体易位片段中特异表达的12个转录本(表1)。
d、分子标记开发
根据获得的12个候选转录本序列,与小麦基因序列进行比对,并依据比对的结果找出候选转录本与小麦a/b/d基因组上同源基因之间的差异,设计est标记及其检测引物。对设计的est标记在冰草z559、fukuhokumugi和衍生系普冰3035中进行pcr验证,分别对每个候选转录本开发了相对应的多态性分子标记(图1),从而验证外源染色体片段中特异表达的转录本的真实性。
所述est标记包括wgrg8和wgrg9,它们的检测引物分别如下:
wgrg8_f:5′-tgccagtggtgaccaatgca-3′;
wgrg8_r:5′-acttggggaagagtctcact-3′;
wgrg9_f:5′-tccaaatcctccagcaaatc-3′;
wgrg9_r:5′-cccgagaccgagcactatac-3′。
e、候选基因预测
根据真实的候选转录本序列,与小麦基因序列进行比对,进行比较基因组学分析,构建衍生系中外源染色体片段与小麦染色体a/b/d间的比较基因组学图谱(图2);对共线性候选区间内的基因进行功能注释,其中转录本transcript/2在水稻中的同源基因为asl2,其编码质体核糖体蛋白l21,突变后会导致叶绿体发育缺陷和幼苗死亡,主要影响植株的光合作用;转录本transcript/4在水稻中的同源基因为osubp15/lg1,其与gw2基因相互作用,是水稻粒宽和籽粒大小的正调节因子。因此基于此方法从小麦-冰草易位系普冰3035中发掘的冰草特异转录本transcript/2和transcript/4可以作为普冰3035中控制千粒重和粒长的候选基因。
表1普冰3035中冰草外源染色体中特异表达转录本结果统计
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之做一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>中国农业科学院作物科学研究所
<120>小麦远缘杂交后代小片段易位系中发掘外源功能候选基因的方法
<130>khp191111756.2
<160>6
<170>siposequencelisting1.0
<210>1
<211>839
<212>dna
<213>冰草(agropyroncristatum)
<400>1
gacgcgatttcgtcctatccacaatattatcccactcccctcttcgtctgaaaaccctcc60
agtccagtatccgtgttccccctcgccatcgttccgagccccgtccatggccaccgccac120
gctccctctccgcctcctcccctccagaaccccactcctccacgccgccgccttcctccc180
cgcgagcacctctctctccgtcaccgcgtccgcgccccgcaactggcgcatcttcgccgc240
cgccgaggaggcgcccgctctggtggaagccgaggcggaggaggtggtagaggatgccgc300
ggttccggagcctgttgagctgcagctggctgctgctggcgcggggaaggacgctgacat360
cttcgccgttgtcatgattgggtccagacaatacattgtgatgccaggtcggtacatata420
cacgcagaggctgaaagacgccaatgtcaatgatcagatcattttgaacaaggtactact480
ggtgtcaacaagagacaaagcttatattggcatgccagtggtgaccaatgcagctgttca540
tgcaattgttgaagaacagggactggacgataaagtgattgttttcaagttcaagaagaa600
gaagaagtaccagaggaaagctggtcacagacagccaaatacgaggttaagaattaccgg660
cataagtggatatgaggaataccctgctgatccaatacttcaagttccagcttaagtgta720
actgtaagttgtttagttacaaatgattggtagtacattagaatgcaagagcattttccc780
cactgttccccaagtgagactcttccccaagttaatgtacttgaaatgctgccattcgt839
<210>2
<211>3631
<212>dna
<213>冰草(agropyroncristatum)
<400>2
gactataccaaagcaaaaacgcgcacacatgagcagagcacagtagccaagcaacagcga60
gcgagccacagtcggtcactcccaccgccgctcccgattccttcagccgccggcaccgct120
gccatcctcggctagagcttctccatcccctacccctatataccgcgatctcctccagaa180
ccaccggccgggactgctgctccgcttgctcgcccccgttggagatccgcccggagcgac240
atttgtctttcttggtagtctagtggttgatttgctggaggatttggagatagtaaagag300
gtgatgtgagccacaaagtgaacttatttctctggccctaacattctaaggtaaatcagt360
gatttggaatgtttttccgagctgaagcagagaattcttggagctggataataataatct420
tttagggatatgctccaaccaagggaagctgatgtgcctgcactctttgttgtatttatc480
atacttccggtgatagcatattttcttcttgggagatggcatgattctgtaagtaagaaa540
acaagagtaggtgtgcttggccagaaagctgcagaagaagccttcaaagtggaaacaatg600
gcatgcccagatgttatattgccaggaccgtctctgagacccatgccttatttgagatct660
gttccttctttaagatccgaataccacgagtgtgctacttgtcgtggccctgcaaacact720
aggtgctcgaggtgcaaatctgttagatactgctctggaaagtgtcaaattatacactgg780
aggcaagggcataaacaagcatgccagcagtggcatgttaacggtggtagcaactctggt840
ggactatctctgatggagagttctgaacaaatgccgttcttgactaacctgaattcacct900
cttccagggggtgacagtcacctgcatgacatgaattttgacacagtatcagagccatcc960
tttgcgacaactgatagctatattcttgatactgatctattcctgacagacagaagcaac1020
atgaatgaatcaaatcaaagtcttctttcaagagtaaatagcgcttctgttgcatcttct1080
gaaaagagcaattacagtgttgatgaagaaaccaactcatctgagattttatcagcaaat1140
aaggtttcaaacaacagttatggttgtttggatgaaaagaatggcaaccatgattttact1200
tatcctctcaataatacggtacaacaacccaataattgtgctcctgaaataccaaaatgt1260
ccaaaagcaagcatcacagtttatgaacccgacatgggtgtctatttaacttctgatatg1320
atgagttcttgcgaggggccatattcttccgcaacagagtcactacagaggagcaaatca1380
tctggaaaatatagtgggagaggaaatgtgatctatatgaagcctccttatccaccaggt1440
aaggtggcttcatcacaaaaagcacaggaggtgttggcatcatatcaatacaatgtccat1500
gaaaagaacacttcctgcaaaaatgaacaaagatctgcaaaatcaagtgtttcaacaaac1560
aacaatttacaaggacgcactcgaatctcaaaattaggagcatccaaggttgaagtcttg1620
aagaagccctcaaaatttctcaaaaccagcctggtgggtttaatcaatgataacaagagg1680
agtaaggtattgtttcgctatgaagatctcgttaagttcttccagtatgaagtacggggc1740
gtttctcccagaggtcttttcaactgtgggaacagctgctatgcaaatgctgttctacaa1800
tgcctcatgtgcacaaaacccctgatgatccacctgcttctgagattgcattctaaagac1860
tgttgctcaaagaactggtgtcttatgtgtgaacttgagcaatatgcttcaactttacgt1920
gaaagtggtggacctgtgtccccaagcagaatcctttcgaatctaaggaacattggatgt1980
cgcttgggtggtggaagtcaggaagatgctcatgaatttttaaggcatcttgtgatgtct2040
atgcaagcagcgtgcctggactggactgggtggtgagaagcaagtagaacaaagcttgca2100
ggaaactacactgatacaacagatgtttggtggacgccttaaatcgaaggttaagtgcct2160
cagatgccatcatgaatctgaaagatacgagaatataatggatcttactttggagattca2220
tggttgggtggagtccttgcaagatgctttgacacagttcactgctcctgaagatttaga2280
tggggataatatgtataaatgtggaaggtgttgtgcttatgttaaagctagaaaacaact2340
aagcgtgcatgaagtgccaaacatattaacagtagttttaaaaagattccagacaggaaa2400
gtatggcaagattaacaaatgtgtcacttttcctgatatgttggacatggttccttttgt2460
gactggggctggtgatcacccgcctctttacttcttgtatgctgtggttgtacatgtgga2520
tacagaaatgcatcattctctggtcactacatatcgtatgtcaaagatatgcagggaaca2580
tggttaagaattgatgactcagaggtcaaggctgtatcattgaatcaagttatgtccgaa2640
ggtgcatatatgctattctacttgagatcttttcctcgccctccgaggatatacattgag2700
aaaggcctattgcctgacccatcatcttcatatcatcactcatcaaaatcctccaagggc2760
tcttctaaacaagagcagaagcagacagaatcactctttacttctgatgatcaaatccat2820
ggtatttatgattttagaccagaggaggaaggttacaggcaagatcagcatgtcaagttg2880
aggtcccaaaatttatatcacaccgatgatgcttttgccgattcggttagcacggacttc2940
tcggacgctacatcaagtgaatggtccctgtttaccagctctgatgaatcttcgtttacc3000
actgaaagcactagagattcattcagtgttgtggattatggtgacaatgctggccttgat3060
ccaatcacctcaatttttgggccatattatgctcctgaccatcctcttgacaactttgcc3120
tcatgtacaaggctctcgccttccaatccgcaaacaagatactttccggaaagcatgggt3180
tttgtctcagattcttccttgccaactcacccctacggcaatgtacatagaggaagatat3240
ccagacagggcttgcgcgtcttcagccgaacctcttgcttcagcaaaccagcgaagtttg3300
tacggtaggtataaccatagtagagatggttttgttcaaacatctgggttttgtcatatg3360
taatggtctgcatctgatagttttagctggagctggtctgatatggttagcagtgccatt3420
aatatcgcctcatgttcttttatttcagatgaggcatatttaattgtagctgcatattct3480
agatgtagttcactcagtgtagtgaagtaactagatctgtctagatgcctagcttttggg3540
ttgacatgttagaattcaaggactagctttgtgaaagcaaagatggccaagcatttattt3600
actgtgtgcgaaaagcttccatttgcttcag3631
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
tgccagtggtgaccaatgca20
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
acttggggaagagtctcact20
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
tccaaatcctccagcaaatc20
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
cccgagaccgagcactatac20
- 还没有人留言评论。精彩留言会获得点赞!