一种高通量测序文库构建的DNA转化效率的检测方法与流程
本发明涉及高通量测序
技术领域:
,具体涉及一种高通量测序文库构建的dna转化效率的检测方法。
背景技术:
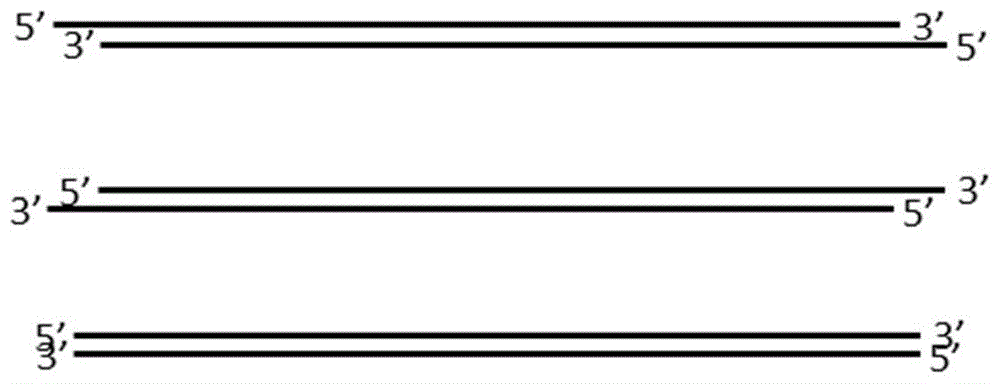
:文库构建是二代测序过程中的核心步骤之一。文库构建过程包括末端补平、磷酸化、末端加a、接头连接和pcr扩增等过程。其中,末端补平、磷酸化、末端加a和接头连接效率的高低决定了原始模板转化为两端都连接上接头的dna片段的效率(即dna转化效率)的高低。文库构建过程的dna转化效率是反映建库试剂和建库方法优劣的重要指标,是筛选建库试剂和建库方法的关键考虑因素。目前,在检测文库构建过程中的dna转化效率时,需要对dna模板中的dna分子和构建文库中两端都连接上接头的dna分子分别进行定量。对于dna模板和两端连接上接头的dna片段进行定量,通常使用定量pcr的方法,利用该方法检测文库构建过程中的dna转化效率存在以下缺点:(1)需要针对原始dna模板和两端都连接接头的dna片段分别建立两个标准曲线,过程繁琐;(2)通过定量pcr进行dna定量时,耗时较长;(3)由于不同测序平台所使用的接头序列不同,需要准备多种定量pcr引物配合不同测序平台使用;(4)受接头序列的影响,连接接头后的dna分子进行定量pcr时很容易出现非特异扩增,导致定量不准确;(5)接头二聚体的残留会明显降低dna转化效率检测的准确性。因此,开发快速、便捷、准确性较高的dna转化效率的检测方法具有重要意义。技术实现要素:为了解决现有技术中存在的技术问题,本发明的目的是提供一种高通量测序文库构建的dna转化效率的检测方法。为实现上述目的,本发明采用毛细管电泳技术区分两端连接接头和未连接接头的dna片段,定量检测文库中dna片段长度分布情况;在测序实践中,dna样品需要先进行片段化处理,经片段化处理的dna样品会产生不同类型的dna片段,本发明在研发过程中发现在检测dna转化效率时,由于不同类型的dna片段在dna修复过程以及与接头的连接效率不同,导致用于评价dna转化效率的dna模板类型极大地影响dna转化效率评价的准确性,因此,本发明进一步针对毛细管电泳技术特点以及真实测序过程中片段化dna样品的特点,设计了能够高效模拟真实的片段化dna模板样品的各种末端情况的模拟dna模板,将该模拟dna模板进行文库构建后,再利用毛细管电泳检测文库中不同dna片段的含量,进而开发了可用于建库试剂和建库方法筛选等建库优化过程的dna转化效率检测方法。具体地,本发明的技术方案如下:本发明提供一种高通量测序文库构建的dna转化效率的检测方法,利用用于模拟真实样品末端情况的模拟dna模板进行文库构建,利用毛细管电泳检测所述文库中dna片段的分布情况,根据所述dna片段的分布情况分析两端连接接头的dna片段在所述文库中所占比例。具体地,所述模拟dna模板包括目的序列以及位于目的序列两侧的侧翼序列。通过在目的序列的两侧引入侧翼序列,可更加便于确定合成的模拟dna模板的准确性。优选地,所述侧翼序列的长度为50~150bp。为能够更真实地反映文库构建过程中dna转化效率的高低,所述目的序列优选为末端碱基平衡的序列。上述目的序列的选择可更有利于排除其他因素影响dna转化效率。所述末端碱基平衡是指末端为a、t、g和c的dna片段在所述模拟dna模板中的含量相等。合适的模拟dna模板长度能够更好地保证毛细管电泳的检测效率,优选地,所述模拟dna模板的长度为100~350bp。以dna为模板进行文库构建时首先需要将dna片段的粘性末端修复为平末端。dna片段通常存在两种粘性末端形式(如图1所示),一种为5’端突出粘性末端,一种为3’端突出粘性末端。为更好地模拟真实样品模板的情况,提高dna转化效率检测的准确性,本发明所述目的序列的5’端和3’端分别含有能够产生5’突出粘性末端和3’突出粘性末端的酶切位点,所述目的序列经酶切和末端修复后,其5’端和3’端的碱基相同。优选地,所述目的序列的5’端和3’端分别含有能够产生5’突出末端和3’突出末端的酶切位点。为更好地提高dna转化效率检测的准确性,本发明所述目的序列优选包括经酶切后末端分别为a、t、g和c碱基突出的4种目的序列。作为本发明的一种优选实施方式,所述模拟dna模板采用酶切的方法构建得到,其构建方法包括如下步骤:(1)分别扩增所述目的序列和所述侧翼序列;(2)将所述目的序列的扩增产物与所述侧翼序列的扩增产物拼接;(3)利用所述目的序列含有的酶切位点对应的酶对步骤(2)的拼接产物进行酶切,切除侧翼序列,分别得到末端分别为a、t、g和c碱基突出的4种dna片段,将4种dna片段等比例混合,得到所述所述模拟dna模板。作为本发明的一种实施方式,上述4种目的序列的5’端和3’端包含的酶切位点分别如下:bamhi/psti、nhei/kpni、sali/nsii和ecori/ecori。作为本发明的具体示例,本发明以puc57质粒序列为模板,合成模拟dna模板,其中,如seqidno.1(未融合侧翼序列)和seqidno.5(两端融合侧翼序列)所示的序列经bamhi/psti酶切后产生5’端和3’端突出为碱基g的模拟dna模板;如seqidno.2(未融合侧翼序列)和seqidno.6(两端融合侧翼序列)所示的序列经nhei/kpni酶切后产生5’端和3’端突出为碱基c的模拟dna模板;如seqidno.3(未融合侧翼序列)和seqidno.7(两端融合侧翼序列)所示的序列经sali/nsii酶切后产生5’端和3’端为碱基t的模拟dna模板;如seqidno.4(未融合侧翼序列)和seqidno.8(两端融合侧翼序列)所示的序列经ecori/ecori酶切后产生5’端和3’端突出为碱基a的模拟dna模板。将上述4种碱基末端突出的酶切产物混合后即为模拟dna模板,将该模拟dna模板建库后,利用毛细管电泳检测其dna转化效率,在保证快速、便捷的同时,具有较高的dna转化效率检测准确性,可更为准确地评估建库试剂或建库方法的dna转化效率。具体地,所述模拟dna模板序列的构建方法包括如下步骤:(1)采用如seqidno.12-19所示的引物扩增4种末端的目的序列;采用如seqidno.20-23所示的引物扩增位于目的序列两侧的侧翼序列(5’和3’端侧翼序列分别如seqidno.9和seqidno.10所示);(2)将4种末端的目的序列的扩增产物分别与所述侧翼序列的扩增产物进行拼接;(3)利用所述目的序列含有的酶切位点对应的酶对步骤(2)的拼接产物进行酶切,切除侧翼序列,分别得到末端分别为a、t、g和c碱基突出的4种dna片段,将4种dna片段等比例混合,得到所述所述模拟dna模板。在本发明所述模拟dna模板的基础上,利用待评估dna转化效率的文库构建试剂或文库构建方法对模拟dna模板进行文库构建。文库构建基本流程可为:(1)末端修复和加a;(2)接头连接;(3)文库中y型接头的双链化。在上述文库构建后,分别将所述文库以及所述文库与所述模拟dna模板的混合物进行毛细管电泳检测,分析其中未连接接头的dna片段的浓度以及两端连接接头的dna片段的浓度,计算两端连接接头的dna片段在所述文库中所占比例。上述毛细管电泳检测具体可分为两组:(1)文库产物(待检片段溶液);(2)文库产物和模拟dna模板的混合物(对照片段溶液)。优选地,以所述文库为待检片段溶液,以所述文库与所述模拟dna模板的混合物为对照片段溶液,当所述待检片段溶液中所述文库的含量与所述对照片段溶液中所述文库的含量相等,且所述对照片段溶液中所述文库与所述模拟dna模板含量相等时,所述dna转化效率的计算公式如下:其中,t为dna转化效率,t1为所述文库中未连接接头的dna片段的浓度,s1为所述文库中两端连接接头的dna片段的浓度,t2为所述文库与所述模拟dna模板的混合物中未连接接头的dna片段的浓度,s2为所述文库与所述模拟dna模板的混合物中两端连接接头的dna片段的浓度。另一方面,本发明还提供一种用于检测高通量测序文库构建的dna转化效率的试剂,其包含dna片段和/或所述dna片段的扩增引物;所述dna片段作为模拟dna模板用于高通量测序的文库构建;所述dna片段包括目的序列以及位于目的序列两侧的侧翼序列;所述目的序列的碱基平衡;所述dna片段的长度为100~350bp;所述目的序列的5’端和3’端分别含有能够产生5’突出粘性末端和3’突出粘性末端的酶切位点,经酶切和末端修复后,其5’端和3’端的碱基相同;所述目的序列包括经酶切后末端分别为a、t、g和c碱基突出的4种目的序列。优选地,所述侧翼序列的长度为50~150bp;更优选地,所述4种目的序列的5’端和3’端包含的酶切位点分别如下:bamhi/psti、nhei/kpni、sali/nsii和ecori/ecori。作为本发明的一种优选方案,所述试剂包括序列如seqidno.1~4所示的dna片段、序列如seqidno.5~8所示的dna片段和/或序列如seqidno.12~23所示的扩增引物。进一步地,本发明还提供所述高通量测序文库构建的dna转化效率的检测方法或所述用于检测高通量测序文库构建的dna转化效率的试剂的如下任一种应用:(1)在高通量测序文库构建的dna转化效率检测中的应用;(2)在筛选高通量测序文库构建试剂中的应用;(3)在优化高通量测序文库构建方法中的应用。本发明的有益效果在于:本发明基于毛细管电泳技术,提供了一种高通量测序文库构建过程中dna转化效率的评价方法,与传统的定量pcr方法相比,该方法具有操作简单、快速、可同时适用于多种测序平台的优势;同时,除可不受接头序列非特异性扩增和接头二聚体污染的影响外,该方法通过利用特异设计的模拟dna模板有效提高了其评价的准确性,可更真实、全面地反映待评价的建库试剂或建库方法的dna转化效率性能,对于文库构建试剂和方法的筛选,提高文库构建和高通量测序的效率和质量具有重要意义,具有优异的应用价值。附图说明图1为本发明
发明内容部分不同dna粘性末端的示意图,由上至下依次为5’端突出,3’端突出和平末端。图2为本发明实施例2中毛细管电泳检测dna片段分布的结果图;其中,a为待检片段溶液的检测结果,b为对照片段溶液的检测结果。图3为本发明实施例3中不同二代测序建库试剂盒构建所得文库中dna片段的分布检测结果;其中,a1为二代测序建库试剂盒a所得文库(待检片段溶液)的毛细管电泳中dna分布情况;a2为二代测序建库试剂盒a所得文库与模板混合物(对照片段溶液)的毛细管电泳中dna分布情况;b1为二代测序建库试剂盒b所得文库(待检片段溶液)的毛细管电泳中dna分布情况;b2为二代测序建库试剂盒b所得文库与模板混合物(对照片段溶液)的毛细管电泳中dna分布情况;c1为二代测序建库试剂盒c所得文库(待检片段溶液)的毛细管电泳中dna分布情况;c2为二代测序建库试剂盒c所得文库与模板混合物(对照片段溶液)的毛细管电泳中dna分布情况。图4为本发明实施例4中采用不同接头用量构建所得文库中dna片段分布检测结果;其中,a1为接头用量为0.1μl(10μm)时所得文库中的dna片段分布情况;a2为接头用量为0.1μl(10μm)时所得文库与模板混合物的dna分布情况;b1为接头用量为0.5μl(10μm)时所得文库中的dna片段分布情况;b2为接头用量为0.5μl(10μm)时所得文库与模板混合物的dna分布情况;c1为接头用量为2.0μl(10μm)时所得文库中的dna片段分布情况;c2为接头用量为2.0μl(10μm)时所得文库与模板混合物的dna分布情况。具体实施方式下面将结合实施例对本发明的优选实施方式进行详细说明。需要理解的是以下实施例的给出仅是为了起到说明的目的,并不是用于对本发明的范围进行限制。本领域的技术人员在不背离本发明的宗旨和精神的情况下,可以对本发明进行各种修改和替换。下述实施例中所使用的实验方法如无特殊说明,均为常规方法。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。实施例1模拟dna模板的构建本实施例以质粒puc57的序列(seqidno.11)为模板构建模拟dna模板。1、模拟dna模板的设计如下:(1)在质粒puc57的序列为中选取一段碱基平衡的、长度为209bp的序列作为目的序列;分别向目的序列的两侧各延伸63bp和85bp作为侧翼序列,以区分经酶切和未经酶切的dna片段;(2)设计扩增引物,以puc57为模板进行目的片段和两端侧翼序列的扩增;在用于扩增目的片段的正向引物和反向引物上分别添加能够产生5’突出和3’突出末端的酶切位点,并保证经末端修复后5’端和3’端的碱基相同;共设计经酶切后能够分别产生a、t、g和c碱基末端的4对目的片段扩增引物(4对引物分别携带不同的酶切位点,使得经酶切后能够分别产生a、t、g和c碱基末端);(3)将目的片段与侧翼序列进行拼接,在目的片段的5’端和3’端分别添加63bp和85bp的侧翼序列片段。用于扩增目的片段的引物序列(seqidno.12-19)和侧翼序列片段的引物序列(seqidno.20-23)如表1所示。表1用于扩增目的片段和侧翼序列片段的引物序列2、模拟dna模板的构建(1)侧翼序列片段以及4种不同碱基末端的目的片段的扩增利用如表1所示的扩增引物,以puc57质粒为模板,pcr扩增侧翼序列片段(fll和flr)以及4种不同碱基末端的目的片段(gcbg/c/t/a)。pcr反应体系如下:pcr扩增程序如下:12℃,保存。pcr扩增产物的纯化:利用北京康为世纪生物科技有限公司的快速琼脂糖凝胶dna回收试剂盒对目的dna片段进行纯化回收。(2)侧翼序列片段和目的片段的拼接将侧翼序列片段(fll和flr)以及4种不同碱基末端的目的片段(gcbg/c/t/a)进行拼接,在每种碱基末端的目的片段的两侧均连接上侧翼序列片段。pcr反应体系如下:片段之间退火程序如下:95℃,2min;60℃,2min;72℃,5min;95℃,2min;90℃,1min;85℃,1min;80℃,1min;75℃,1min;70℃,1min;65℃,1min;60℃,1min;72℃,5min;12℃,保存。将pcr管从pcr仪中取出,向pcr管中加入1μl的正向引物fll-f(10μm)和1μl的反向引物flr-r(10μm),按照下面程序进行pcr扩增:拼接产物的纯化:采用北京康为世纪生物科技有限公司的快速琼脂糖凝胶dna回收试剂盒对拼接产物目的片段进行纯化回收。(3)目的片段的酶切将步骤(2)得到的拼接片段纯化产物进行限制性内切酶酶切,切去侧翼序列,得到具有不同末端碱基突出的模拟dna模板。酶切反应体系如下:其中,对于gcbg的拼接片段(seqidno.5),内切酶1和2分别为bamhi和psti;对于gcbc的拼接片段(seqidno.6),内切酶1和2分别为nhei和kpni;对于gcbt的拼接片段(seqidno.7),内切酶1和2分别为sali和nsii;对于gcba的拼接片段(seqidno.8),内切酶1和2均为ecori。将装有酶切反应体系的pcr管放于预热至37℃的pcr仪中孵育15min。酶切产物的纯化:采用用北京康为世纪生物科技有限公司的快速琼脂糖凝胶dna回收试剂盒对目的dna片段(209bp)进行纯化回收。酶切后纯化回收得到4种不同碱基末端(a、t、g、c)的dna片段(209bp),将这4种dna片段等比例混合即为模拟dna模板。实施例2高通量测序文库构建的dna转化效率的检测对实施例1构建的模拟dna模板进行文库构建,利用毛细管电泳检测构建得到的文库中两端连接接头的dna片段的含量比例,评价文库构建过程中的dna转化效率,具体方法如下:1、末端修复与加a反应反应体系如下:将上述反应体系涡旋震荡混匀后短暂离心。反应程序如表2所示:表2末端修复与加a反应的反应程序2、接头连接(1)接头连接的反应体系如下:将上述反应体系涡旋震荡混匀后短暂离心。反应程序如下:20℃连接15min,4℃保存。(2)接头连接产物的纯化:①向1.5ml离心管中加入110μl接头连接产物和110μlampurexp,涡旋震荡或吹吸混匀后室温放置5min;②将离心管固定于磁力架上静置5min,之后用移液器弃去溶液;③向离心管中加入250μl80%乙醇,之后弃去溶液;④重复步骤③一次;⑤保持离心管固定于磁力架上,室温放置3-5min使乙醇充分挥发;⑥向离心管中加入25μl去离子水,涡旋震荡或吹吸使磁珠充分混匀,室温放置5min;⑦将离心管固定于磁力架上静置5min,之后用移液器转移20μl洗脱液至新的pcr管中备用。3、文库中y型接头的双链化:(1)反应体系如下:kapahifihotstart预混液(2×)25μl;kapa文库扩增引物预混液(10×)5μl;接头连接产物20μl。反应程序如表3所示:表3y型接头的双链化的反应程序步骤温度时间循环数变性98℃1min1退火60℃1min1延伸72℃2min1保存72℃forever1(2)pcr产物的纯化:①向1.5ml离心管中加入50μlpcr扩增产物和90μlampurexp,涡旋震荡或吹吸混匀后室温放置5min;②将离心管固定于磁力架上静置5min,之后用移液器弃去溶液;③向离心管中加入250μl80%乙醇,之后弃去溶液;④重复步骤③一次;⑤保持离心管固定于磁力架上,室温放置3-5min使乙醇充分挥发;⑥向离心管中加入23μl去离子水,涡旋震荡或吹吸使磁珠充分混匀,室温放置5min;⑦将离心管固定于磁力架上静置5min,之后用移液器转移20μl洗脱液至新的pcr管中备用,得到文库产物。4、毛细管电泳检测将上述步骤3中的文库产物进行毛细管电泳检测,分别设置如下2组:(1)待检片段溶液:取10μl文库产物与10μl去离子水充分混匀;(2)对照片段溶液:取实施例1制备的模拟dna模板25ng,用去离子水稀释至10μl,之后加入10μl文库产物,充分混匀。取上述待检片段和对照片段的dna溶液放入毛细管电泳仪(qseq100或agilent2100)中进行dna片段分布检测。dna转化效率的计算公式如下:其中,t为dna转化效率,t1为文库产物(待检片段溶液)中未连接接头的dna片段的浓度,s1为文库产物(待检片段溶液)中两端连接接头的dna片段的浓度,t2为文库与模拟dna模板的混合物(对照片段溶液)中未连接接头的dna片段的浓度,s2为文库与模拟dna模板的混合物(对照片段溶液)中两端连接接头的dna片段的浓度。为更好地说明dna转化效率的计算方法,以下进行举例说明:若连接接头的长度为60bp,未连接接头的dna片段长度为209bp(实施例1制备的模拟dna模板,毛细管电泳检测结果为210bp左右),两端均连接接头的dna片段长度为329bp(毛细管电泳检测结果为330bp左右),一端连接接头的dna片段长度为269bp(毛细管电泳检测结果为270bp左右),接头二聚体的dna片段长度120bp,利用毛细管电泳仪(qseq100或agilent2100)中分析上述dna片段的浓度,分析结果的示意图如图2所示,其中,20bp和1000bp的dna片段为毛细管电泳中的marker。实施例3利用dna转化效率检测方法进行建库试剂的筛选本实施例利用不同的建库试剂对实施例1构建的模拟dna模板进行文库构建,并利用实施例2的毛细管电泳检测方法检测文库中dna片段的分布情况,并利用实施例2的dna转化效率计算公式计算文库构建过程中的dna转化效率,进而筛选性能优异的建库试剂。文库构建方法与实施例2的区别仅在于,在文库构建过程中使用了3种不同的商品化二代测序文库构建试剂盒,三种试剂盒分别命名为a、b和c,试剂盒信息如表4所示。表4商品化试剂盒信息利用不同建库试剂进行文库构建得到的文库产物的毛细管电泳检测结果如图3所示,根据毛细管电泳检测结果计算待检片段溶液中未连接接头的dna片段(210bp左右)浓度(t1)和两端连接接头的dna片段(330bp左右)浓度(s1)以及对照片段溶液中未连接接头的dna片段(210bp左右)浓度(t2)和两端连接接头的dna片段(330bp左右)浓度(s2),利用dna转化效率的计算公式得出,二代测序建库试剂盒c中dna片段的转化效率最高,dna的转化效率为44.50%;试剂盒a效果次之,dna的转化效率为35.36%;试剂盒b的效果最差,dna的转化效率为20.19%。实施例4利用dna转化效率检测方法进行建库方法的筛选本实施例利用实施例2的建库方法对实施例1构建的模拟dna模板进行文库构建,采用bioo接头(nextflex-96tmdnabarcodes-96(forillumina),货号:nova-514106,批号:b3068214),设置不同的接头用量,并利用实施例2的毛细管电泳检测方法检测文库构建过程中的dna转化效率,进而筛选最适的接头用量。文库构建方法与实施例2的区别仅在于,文库构建过程中(步骤2的接头连接反应体系中)采用不同的接头用量(接头母液为10μm,接头用量分别为0.1μl、0.5μl和2μl)。采用不同接头用量进行文库构建得到的文库产物的毛细管电泳检测结果如图4所示,根据毛细管电泳检测结果计算待检片段溶液中未连接接头的dna片段(210bp左右)浓度(t1)和两端连接接头的dna片段(330bp左右)浓度(s1)以及对照片段溶液中未连接接头的dna片段(210bp左右)浓度(t2)和两端连接接头的dna片段(330bp左右)浓度(s2),利用dna转化效率的计算公式得出各种接头用量的dna转化效率。结果表明,随着接头用量的增加,dna的转化效率也随之增加:接头用量为0.1μl、0.5μl和2.0μl时的转化效率分别为33.20%、37.64%和40.24%;这与文库构建的一般性规律相符(一般地,在一定范围内增加接头用量,接头连接效率相应提高)。以上所述仅是本发明的优选实施方式,应当指出,对于本
技术领域:
的普通技术人员来说,在不脱离本发明技术原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。序列表<110>北京优迅医学检验实验室有限公司<120>一种高通量测序文库构建的dna转化效率的检测方法<130>khp191117165.2<160>23<170>siposequencelisting1.0<210>1<211>263<212>dna<213>人工序列(artificialsequence)<400>1cacaattccacacaacatacgaggatccatgagtaaacttggtctgacagttaccaatgc60ttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctga120ctccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgca180atgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccactg240cagttacatgatcccccatgttg263<210>2<211>264<212>dna<213>人工序列(artificialsequence)<400>2cacaattccacacaacatacgagctagcatgagtaaacttggtctgacagttaccaatgc60ttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctga120ctccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgca180atgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccaggt240accgttacatgatcccccatgttg264<210>3<211>264<212>dna<213>人工序列(artificialsequence)<400>3cacaattccacacaacatacgagtcgacatgagtaaacttggtctgacagttaccaatgc60ttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctga120ctccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgca180atgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccaatg240catgttacatgatcccccatgttg264<210>4<211>264<212>dna<213>人工序列(artificialsequence)<400>4cacaattccacacaacatacgagaattcatgagtaaacttggtctgacagttaccaatgc60ttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctga120ctccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgca180atgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccagaa240ttcgttacatgatcccccatgttg264<210>5<211>368<212>dna<213>人工序列(artificialsequence)<400>5catggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatac60gagaattcatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctat120ctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataac180tacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacg240ctcaccggctccagatttatcagcaataaaccagccagaattcgttacatgatcccccat300gttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggc360cgcagtgt368<210>6<211>368<212>dna<213>人工序列(artificialsequence)<400>6catggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatac60gagctagcatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctat120ctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataac180tacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacg240ctcaccggctccagatttatcagcaataaaccagccaggtaccgttacatgatcccccat300gttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggc360cgcagtgt368<210>7<211>368<212>dna<213>人工序列(artificialsequence)<400>7catggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatac60gagtcgacatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctat120ctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataac180tacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacg240ctcaccggctccagatttatcagcaataaaccagccaatgcatgttacatgatcccccat300gttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggc360cgcagtgt368<210>8<211>368<212>dna<213>人工序列(artificialsequence)<400>8catggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatac60gagaattcatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctat120ctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataac180tacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacg240ctcaccggctccagatttatcagcaataaaccagccagaattcgttacatgatcccccat300gttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggc360cgcagtgt368<210>9<211>63<212>dna<213>人工序列(artificialsequence)<400>9catggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatac60gag63<210>10<211>85<212>dna<213>人工序列(artificialsequence)<400>10gttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgtt60gtcagaagtaagttggccgcagtgt85<210>11<211>2709<212>dna<213>人工序列(artificialsequence)<400>11tcgcgcgtttcggtgatgacggtgaaaacctctgacacatgcagctcccggagacggtca60cagcttgtctgtaagcggatgccgggagcagacaagcccgtcagggcgcgtcagcgggtg120ttggcgggtgtcggggctggcttaactatgcggcatcagagcagattgtactgagagtgc180accatatgcggtgtgaaataccgcacagatgcgtaaggagaaaataccgcatcaggcgcc240attcgccattcaggctgcgcaactgttgggaagggcgatcggtgcgggcctcttcgctat300tacgccagctggcgaaagggggatgtgctgcaaggcgattaagttgggtaacgccagggt360tttcccagtcacgacgttgtaaaacgacggccagtgaattcgagctcggtacctcgcgaa420tgcatctagatatcggatcccgggcccgtcgactgcagaggcctgcatgcaagcttggcg480taatcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaac540atacgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcaca600ttaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcat660taatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcc720tcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactca780aaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgtgagca840aaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccatagg900ctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccg960acaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgtt1020ccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctt1080tctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggc1140tgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtctt1200gagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaacaggatt1260agcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggc1320tacactagaagaacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaa1380agagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtt1440tgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttct1500acggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagattt1560caaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaa1620gtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatct1680cagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataacta1740cgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgct1800caccggctccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtg1860gtcctgcaactttatccgcctccatccagtctattaattgttgccgggaagctagagtaa1920gtagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgt1980cacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggcgagtta2040catgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtca2100gaagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctctta2160ctgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattct2220gagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccg2280cgccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaac2340tctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaact2400gatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaa2460atgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatactcttccttt2520ttcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaat2580gtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctg2640acgtctaagaaaccattattatcatgacattaacctataaaaataggcgtatcacgaggc2700cctttcgtc2709<210>12<211>55<212>dna<213>人工序列(artificialsequence)<400>12cacaattccacacaacatacgaggatccatgagtaaacttggtctgacagttacc55<210>13<211>50<212>dna<213>人工序列(artificialsequence)<400>13caacatgggggatcatgtaactgcagtggctggtttattgctgataaatc50<210>14<211>55<212>dna<213>人工序列(artificialsequence)<400>14cacaattccacacaacatacgagctagcatgagtaaacttggtctgacagttacc55<210>15<211>51<212>dna<213>人工序列(artificialsequence)<400>15caacatgggggatcatgtaacggtacctggctggtttattgctgataaatc51<210>16<211>55<212>dna<213>人工序列(artificialsequence)<400>16cacaattccacacaacatacgagtcgacatgagtaaacttggtctgacagttacc55<210>17<211>50<212>dna<213>人工序列(artificialsequence)<400>17caacatgggggatcatgtaacatgcatggctggtttattgctgataaatc50<210>18<211>55<212>dna<213>人工序列(artificialsequence)<400>18cacaattccacacaacatacgagaattcatgagtaaacttggtctgacagttacc55<210>19<211>51<212>dna<213>人工序列(artificialsequence)<400>19caacatgggggatcatgtaacgaattctggctggtttattgctgataaatc51<210>20<211>23<212>dna<213>人工序列(artificialsequence)<400>20catggtcatagctgtttcctgtg23<210>21<211>23<212>dna<213>人工序列(artificialsequence)<400>21ctcgtatgttgtgtggaattgtg23<210>22<211>21<212>dna<213>人工序列(artificialsequence)<400>22gttacatgatcccccatgttg21<210>23<211>21<212>dna<213>人工序列(artificialsequence)<400>23acactgcggccaacttacttc21当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1