用于对细胞内ATP进行活细胞分析的方法和组合物与流程
交叉参考
本申请主张2018年3月9日提交的美国临时专利申请第62/640983号的优先权,所述申请以全文引用的方式并入本文中。
背景技术:
监测药物诱导的代谢扰动的标准途径受限于提供基于群体的测量和有限的动力学信息的端点分析。根据本公开的方面,提供能够对细胞atp含量进行直接、自动化活细胞分析的经遗传编码的atp传感器以及使用方法。
技术实现要素:
在一个方面中,公开了融合蛋白,其包含通式x1-x2-x3-x4-x5的多肽,其中:
x1和x5中的一个包含具有受体激发波长和fret发射波长的荧光共振能量传递(fret)受体多肽,并且x1和x5中的另一个包含具有供体激发波长和供体发射波长的fret供体多肽。
x2和x4独立地是任选的氨基酸接头;并且
x3包含atp结合蛋白;
其中atp结合蛋白对atp的结合引起所述fret受体多肽与所述fret供体多肽的相互作用。
在一个实施例中,x1包含fret受体多肽并且x5包含fret供体多肽;在另一个实施例中,x1包含fret供体多肽并且x5包含fret受体多肽。在各种实施例中,x2为长度在1到2个氨基酸之间的氨基酸接头,或不存在。在其它实施例中,x2为选自由以下组成的群组的氨基酸接头:a、s、p、v、t、ts、id,或其中x2不存在。在其它实施例中,x4为长度在1到5个氨基酸之间的氨基酸接头,或其中x4不存在。在其它实施例中,x4为选自由以下组成的群组的氨基酸接头:at、a、sa、ga、ff、pppp(seqidno:20)、fl、gtsg(seqidno:21)、p、s、anefm(seqidno:22)或其中x4不存在。在各种实施例中,x2和x4都是a;x2是s并且x4是a;
x2不存在并且x4是ff;x2是v并且x4是fl;x2是t并且x4是gtsg(seqidno:21);或x2是s并且x4是sa。在另一实施例中,x2和x4不包括任何脯氨酸残基。在其他实施例中,所述fret受体多肽具有500到560nm范围内的最大受体激发波长和530到580nm范围内的受体最大发射波长。在其它实施例中,所述fret受体多肽包含沿一或多种seqidno:1-3的所述氨基酸序列的长度至少90%、91%、92%、93%、94%95%、96%、97%、98%、99%或100%一致并且在发色团处一致的氨基酸序列。在另一实施例中,存在所述fret受体多肽中的所有任选的氨基酸残基。在一个实施例中,所述fret供体多肽具有450到500nm范围内的最大供体激发波长和480到515nm范围内的最大供体发射波长。在其他实施例中,所述fret供体多肽包含沿seqidno:4的所述氨基酸序列的长度至少85%、87%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%一致并且在所述发色团处一致的氨基酸序列。在其它实施例中,x3包含atp结合蛋白,其包含沿选自由seqidno:5-6的群组的所述氨基酸序列的长度至少65%、67%、68%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%一致的所述氨基酸序列。在另一个实施例中,所述融合蛋白包含沿选自由seqidno:7-11组成的群组的所述氨基酸序列的长度至少90%、91%、92%、93%、94%95%、96%、97%、98%、99%或100%一致的氨基酸序列。
在另一方面中,公开对照融合蛋白,其包括本文所公开的所述融合蛋白和其组分的所有实施例和实施例的组合,不同之处在于x3包含不结合到atp的对照蛋白。在一个实施例中,x3包含沿seqidno:12的所述氨基酸序列的长度至少65%、67%、68%、70%、75%、80%、85%、90%、91%、92%、93%、94%95%、96%、97%、98%、99%或100%一致的氨基酸序列,其中seqidno:12的所述带下划线和粗体的残基必须是k。在其他实施例中,所述对照融合蛋白包含沿seqidno:13的所述氨基酸序列的长度至少65%、67%、68%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%一致的氨基酸序列,其中seqidno:13所述带下划线和粗体的残基必须是k。
在另一方面中,公开编码本文所公开的任何实施例或实施例的组合的所述融合蛋白或对照蛋白的多核苷酸。在其它方面,本公开提供编码本公开的所述多核苷酸的表达载体,其中所述多核苷酸可操作地连接到能够引导所述多核苷酸表达的启动子序列,和包含本文所公开的任何实施例或实施例的组合的所述多核苷酸或表达载体的重组宿主细胞。在一个实施例中,所述宿主细胞为稳定转化体细胞系的细胞。
在其它方面,本公开提供包含本文所公开的任何实施例或实施例的组合的一或多种融合蛋白和一或多种对照融合蛋白的试剂盒。在另一方面,本公开提供用于测定所关注的细胞中atp的含量的所述融合蛋白的用途的方法。在一个实施例中,所述分析atp的方法包含:
(a)在一或多个第一细胞中表达本文中公开的任何实施例或实施例的组合的所述融合蛋白,并且产生选自由以下组成的群组的一或多个图像:
(i)在使所述一或多个第一细胞暴露于具有所述供体多肽激发波长的光时,通过侦测由从所述一或多个第一细胞发射的具有所述fret受体多肽发射波长的光产生的荧光信号而产生的第一荧光图像;和/或
(ii)在使所述一或多个第一细胞暴露于具有所述受体多肽激发波长的光时,通过侦测由从所述一或多个第一细胞发射的具有所述fret受体多肽发射波长的光产生的荧光信号而产生的第二荧光图像;和/或
(iii)在使所述一或多个第一细胞暴露于具有所述供体多肽激发波长的光时,通过侦测由从所述一或多个第一细胞发射的具有所述fret供体多肽发射波长的光产生的荧光信号而产生的第三荧光图像;和/或
(b)通过比较所述第一荧光图像、所述第二荧光图像和/或所述第三荧光图像中的荧光信号的输出来确定所述一或多个第一细胞中的fret比;
其中所述一或多个第一细胞中atp的水平与所确定的fret比成比例。在一个实施例中,所述方法进一步包含在一或多个第一细胞中表达本文所公开的任何实施例或实施例的组合的所述对照融合蛋白,并且侦测由从所述一或多个第一细胞发射的具有所述受体发射波长的光产生的对照信号。在一个实施例中,侦测所述对照信号包含
(c)在一或多个对照细胞(如所述第一细胞或第二细胞)中表达本文所公开的任何实施例或实施例的组合的所述对照融合蛋白,并且产生选自由以下组成的群组的一或多个图像:
(i)在使所述一或多个对照细胞暴露于具有所述供体多肽激发波长的光时,通过侦测由从所述一或多个对照细胞发射的具有所述fret受体多肽发射波长的光产生的荧光信号而产生的第四荧光图像;和/或
(ii)在使所述一或多个对照细胞暴露于具有所述受体多肽激发波长的光时,通过侦测由从所述一或多个对照细胞发射的具有所述fret受体多肽发射波长的光产生的荧光信号而产生的第五荧光图像;和/或
(iii)在使所述一或多个对照细胞暴露于具有所述供体多肽激发波长的光时,通过侦测由从所述一或多个对照细胞发射的具有所述fret供体多肽发射波长的光产生的荧光信号而产生的第六荧光图像;和/或
(d)通过比较所述第四荧光图像、所述第五荧光图像和/或所述第六荧光图像中的荧光信号的输出,确定所述一或多个对照细胞中的对照融合fret比;
其中将所述对照融合fret比的变化确定为与atp结合无关的实验条件的结果,并且其中基于对照融合fret比的变化校正所测定的fret比。
在其它实施例中,所述一或多个第一细胞在培养箱中培养,其中在不从所述培养箱去除所述一或多个第一细胞的情况下进行所有成像步骤。在另一实施例中,所述方法进一步包含使所述一或多个第一细胞与一或多种测试物质接触以及测定所述测试物质对所述一或多个第一细胞中atp的存在的影响。在另一实施例中,在1分钟到三个月范围内的时间段内连续地或间歇地确定所述一或多种测试物质对所述一或多个第一细胞中atp的存在的影响。
在另一方面,本公开提供编码融合蛋白的多核苷酸,以及所述融合蛋白本身,所述融合蛋白包含:为具有受体激发波长和fret发射波长的fret受体的第一荧光团、具有n端和c端的所述第一荧光团;具有n端和c端的atp结合蛋白;和为具有供体激发波长和供体发射波长的fret供体的第二荧光团、具有n端和c端的所述第二荧光团,其中第一接头安置于所述第一荧光团的所述c端与所述atp结合蛋白的所述n端之间,并且第二接头安置于所述atp结合蛋白的所述c端与所述第二荧光团的所述n端之间,并且其中所述第一荧光团、atp结合蛋白和第二荧光团可操作地连接,使得所述atp结合蛋白对atp的结合引起所述第一荧光团和第二荧光团的相互作用以在暴露于具有所述供体激发波长的光时产生fret发射信号。在其它实施例中,所述多核苷酸进一步包含可操作地连接的启动子;所述多核苷酸包含于表达载体中;并且公开包含所述多核苷酸或所述表达载体的宿主细胞。
附图说明
图1为针对本发明的示例性融合蛋白计算的信号窗口的图形表示,其通过从在治疗后2小时的载体处理的细胞的fret比减去药物处理的细胞的fret比来计算。
图2a-b为使用具有每一传感器的稳定表达的细胞系中的本发明(((a)go-ateam1;和(b)a1)的示例性融合蛋白观察到的fret比的图解比较。
图3a-b为使用与和不与10mmatp一起培养的本发明的示例性融合蛋白观察到的激发光谱的图解比较。传感器a1(go-ateam-a1)的表现(图3a)说明,与go-ateam1传感器的表现(图3b)相比,信号窗口(将atp结合状态(实线)与atp未结合状态(虚线)进行比较)提高了2倍。
图4.在标准培养基中生长的细胞上分别通过2dg和kcn同时抑制糖酵解和oxphos的atp消耗(图4a)。数据表明atp耗尽的时程和同时2dg和kcn处理的浓度依赖性。在葡萄糖或半乳糖培养基中生长的细胞上的细胞毒性化合物(氯丙嗪,图4b)和有丝分裂毒性化合物(鱼藤酮,图4c)的比较。前两个图描绘了如所指示的在生长于葡萄糖或半乳糖中的细胞中在24小时时间段内各化合物的作用。第三幅图描绘在24小时时间点的每种化合物的浓度-反应曲线。细胞毒性化合物在两种培养基条件下展示类似作用,而有丝分裂毒性化合物在半乳糖中生长的细胞中诱导较大atp消耗。
图5a-b.cb-839在具有成纤维细胞的单或共培养物中的乳癌细胞系中的作用。代表性数据展现三重阴性乳癌(tnbc)细胞中的延长的atp消耗,其通过与ccd-1068sk成纤维细胞共培养而减弱(图5a)。相比之下,受体阳性细胞在48小时内展现从cb839处理的回收,并且所述反应不受成纤维细胞的存在影响(图5b)。
具体实施方式
本文所用的科学和技术术语意图具有本领域普通技术人员通常理解的含义。发现这些术语在各种标准参考文献中被定义和使用,包括j.sambrook和dwrussell,《分子克隆:实验室手册》,冷泉港实验室出版社;第三版,2001年;fmausubel编辑,分子生物学简明实验方案,当前方案;第五版,2002年;b.alberts等,细胞分子生物学,第四版,garland,2002;dlnelson和mmcox,lehninger生物化学原理,第4版,whfreeman&company,2004;和herdewijn,p.,寡核苷酸合成(主编):方法与应用,分子生物学方法,humana公司出版社,2004。
除非另外明确陈述或上下文另外明确指示,否则单数术语“一”、“一”和“所述”并不意图为限制性的且包含多个指示物。
除非上下文另外明确指示,否则本文中所公开的所有实施例可以组合。
在第一方面中,本发明提供融合蛋白,其包含通式x1-x2-x3-x4-x5的多肽,其中:
x1和x5中的一个包含具有受体激发波长和fret发射波长的荧光共振能量传递(fret)受体多肽,并且x1和x5中的另一个包含具有供体激发波长和供体发射波长的fret供体多肽。
x2和x4独立地是任选的氨基酸接头;并且
x3包含atp结合蛋白;
其中atp通过atp结合蛋白的结合引起所述fret受体多肽与所述fret供体多肽的相互作用。
此第一方面的融合蛋白可以例如用于侦测和测量活细胞中的atp,如以下实例中详述。
荧光共振能量转移(fret)为能量从激发供体荧光团非辐射转移到接近供体的适合受体荧光团。对于选择供用于本发明融合蛋白的fret荧光团供体/受体多肽对,考虑每一者的吸收和发射波长。基于本文中的教示,本领域的技术人员可以容易地确定在特定应用中各种荧光团中的哪些荧光团待用作fret供体/受体多肽对。
在一个实施例中,x1包含fret受体多肽并且x5包含fret供体多肽;在另一个实施例中,x1包含fret供体多肽并且x5包含fret受体多肽。
可以使用任何适合的多肽荧光团,包括但不限于绿色荧光蛋白和衍生物中的任一种,如本文所述的bfp、ebfp、ebfp2、ecfp、rfp和yfp以及其它多肽荧光团。
在其他实施例中,所述fret受体多肽具有500到560nm范围内的最大受体激发波长和530到580nm范围内的受体最大发射波长。在如以下实例中所描述的另一个实施例中,fret受体多肽包含沿seqidno:1-3中的一或多个的氨基酸序列的长度(由突出显示的残基标注)至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%一致的氨基酸序列。圆括号中的残基在通篇中是任选的。
mkok(seqidno:1)
(mvsvi)kpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvf
mko(seqidno:2)
(msvik)pemkmryymdgsvngheftiegegtgrpyeghqemtlrvtma
mko2(seqidno:3)
(mvsvi)kpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvf
在一个实施例中,存在所述fret受体多肽中的所有任选的氨基酸残基。
在另一个实施例中,所述fret供体多肽具有450到500nm范围内的最大供体激发波长和480到515nm范围内的最大供体发射波长。在如以下实例中所描述的另一个实施例中,fret供体多肽包含沿seqidno:4的氨基酸序列的长度至少85%、87%、90%、91%、92%、93%、94%95%、96%、97%、98%、99%或100%一致并且在发色团处一致的氨基酸序列(突出显示的残基)。
循环排列(cpm)egfp:seqid.no:4
(dg)svqladhyqqntpigdgpvllpdnhylstqs(a/k)lskdpnekrdhmvllefvtaagitlgmdelykggsggmvskgeelftgvvpilveldgdvnghkfsvsgegegdatygkltlkficttgklpvpwptlvttl
与seqidno:4具有必需氨基酸序列一致性并且可以用于本发明的融合蛋白的例示性fret供体多肽在以下表1中列出。
表1.
认为适合于预期用途(即:能够在待分析的细胞类型中表达等)的融合蛋白中可以使用任何适合的atp结合蛋白。在如以下实例中所描述的一个非限制性实施例中,x3包含沿着选自由seqidno:5-6(圆括号中的残基为任选的)组成的群组的氨基酸残基的长度至少65%、67%、68%、70%、75%、80%、85%、90%、91%、92%、93%、94%95%、96%、97%、98%、99%或100%一致的包含氨基酸序列的atp结合蛋白。
seqidno:5:枯草杆菌fof1-atp合成酶的经修饰ε亚单元(m)ktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaararqraqerlnsqsddtdirraelalqralnrldvagk
seqidno:6具有来自芽孢杆菌属ps3fof1-atp合成酶的ε亚单元的两个元素、m到y突变和gk到emk取代(为粗体)的枯草杆菌fof1-atp合成酶的经修饰的ε亚单元。
(m)ktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqte
在下表2中列出了示例性的atp结合蛋白,其与seqidno5-6具有必需的氨基酸序列同一性,并且可以用于本发明的融合蛋白中。
表2.
·多种:atp合成酶ε链[芽孢杆菌],97%,gi|489320353|wp_003227688.1
·atp合成酶ε链[枯草芽孢杆菌],96%,gi|505296717|wp_015483819.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],96%,gi|504291152|wp_014478254.1
·atp合成酶ε链[tequilensis芽孢杆菌],95%,gi|639655008|wp_024713798.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],95%,gi|545118192|wp_021480810.1
·f0f1atp合成酶亚单元[芽孢杆菌属fmq74],95%,gi|1102032633|wp_071577657.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],95%,gi|1280952059|wp_100275394.1
·多种:f0f1atp合成酶亚单元ε[枯草芽孢杆菌组],95%,gi|1039477827|wp_064816018.1
·f0f1atp合成酶亚单元ε[枯草芽孢杆菌],94%,gi|1207627978|wp_087992693.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],94%,gi|745781482|wp_039075208.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],92%,gi|498018072|wp_010332228.1
·f0f1atp合成酶亚单元ε[卤化芽孢杆菌],92%,gi|570758358|wp_024123161.1
·多种:f0f1atp合成酶亚单元ε[枯草芽孢杆菌组],89%,gi|489420817|wp_003326558.1
·多种:atp合成酶ε链[芽孢杆菌],87%,gi|489242972|wp_003151176.1
·f0f1atp合成酶亚单元ε[中村芽孢杆菌],86%,gi|1004344994|wp_061522539.1
·多种:atp合成酶f0f1亚单元ε[芽孢杆菌],86%,gi|921995573|wp_053285344.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],86%,gi|740674878|wp_038460167.1
·多种:atp合成酶ε链[芽孢杆菌],86%,gi|723597160|wp_033574395.1
·多种:atp合成酶ε链[芽孢杆菌],85%,gi|503119226|wp_013353907.1
·f0f1atp合成酶亚单元[velacensis芽孢杆菌],86%,gi|1207676870|wp_088037238.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],95%,gi|489314501|wp_003221882.1
·f0f1atp合成酶亚单元[swezeyi芽孢杆菌],82%,gi|1140910370|wp_076759287.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],82%,gi|651594109|wp_026589081.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],82%,gi|489278336|wp_003186004.1
·f0f1atp合成酶亚单元[索诺拉沙漠芽孢杆菌(bacillussonorensis)],82%,gi|493689352|wp_006639406.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],82%,gi|521288986|wp_020453254.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],82%,gi|806497211|wp_046130316.1
·f0f1atp合成酶亚单元[甘氨酸芽孢杆菌],81%,gi|860247715|wp_048355651.1
·atp合成酶亚单元ε[枯草芽孢杆菌xf-1],95%,gi|449030049|age65288.1
·f0f1atp合成酶亚单元[戈壁芽孢杆菌],75%,gi|926266349|wp_053604535.1
·f0f1atp合成酶亚单元ε[维生素性嗜氧杆菌],73%,gi|1408392372|wp_111644740.1
·f0f1atp合成酶亚单元[芽孢杆菌属j37],73%,gi|651531384|wp_026563116.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],73%,gi|736227472|wp_034318329.1
·f0f1atp合成酶亚单元ε[厦门芽孢杆菌],73%,gi|495630347|wp_008354926.1
·f0f1atp合成酶亚单元ε[苛求芽孢杆菌(bacillusfastidiosus)],72%,gi|1054351039|wp_066230885.1
·f0f1atp合成酶亚单元ε[欧文芽孢杆菌],71%,gi|1272187322|wp_099360911.1
·多种:atp合成酶f0f1亚单元ε[芽孢杆菌],73%,gi|915975869|wp_050944437.1
·f0f1atp合成酶亚单元[芽孢杆菌属mbgli79],81%,gi|1339810892|auz40790.1
·f0f1atp合成酶亚单元ε[短小芽孢杆菌],73%,gi|1002946034|wp_061409853.1
·f0f1atp合成酶亚单元[芽孢杆菌属yn-1],71%,gi|1493450619|wp_121448499.1
·atp合成酶ε链[短小芽孢杆菌],73%,gi|763278706|wp_044140142.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],72%,gi|736652987|wp_034660282.1
·f0f1atp合成酶亚单元ε[onubensis芽孢杆菌],70%,gi|1272175367|wp_099354575.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],72%,gi|495618814|wp_008343393.1
·f0f1atp合成酶亚单元[安全芽孢杆菌],72%,gi|1207611961|wp_087977756.1
·多种:atp合成酶ε链[芽孢杆菌],72%,gi|489307156|wp_003214592.1
·f0f1atp合成酶亚单元ε[短小芽孢杆菌],72%,gi|1274554308|wp_099682181.1
·f0f1atp合成酶亚单元ε[南洋芽孢杆菌(bacillusaustralimaris)],72%,gi|983547050|wp_060698605.1
·f0f1atp合成酶亚单元ε[印度芽孢杆菌],71%,gi|657860862|wp_029566603.1
·f0f1atp合成酶亚单元ε[alveayuensis芽孢杆菌],71%,gi|765542636|wp_044749357.1
·f0f1atp合成酶亚单元[芽孢杆菌属nmtd17],72%,gi|1360795247|wp_106071342.1
·多种:f0f1atp合成酶亚单元ε[厌氧芽孢杆菌(anoxybacillus)],67%,gi|765534407|wp_044741555.1
·f0f1atp合成酶亚单元[平流芽孢杆菌],71%,gi|494762215|wp_007497623.1
·f0f1atp合成酶亚单元ε[sinesaloumensis芽孢杆菌],70%,gi|1148930458|wp_077619903.1
·f0f1atp合成酶亚单元[威海芽孢杆菌],71%,gi|1119658527|wp_072578188.1
·f0f1atp合成酶亚单元ε[印度芽孢杆菌],71%,gi|657040285|wp_029283365.1
·f0f1atp合成酶亚单元[芽孢杆菌属hng],69%,gi|1452393681|wp_116352015.1
·f0f1atp合成酶亚单元[芽孢杆菌属hmsc76g11],69%,gi|1093537853|wp_070877088.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],68%,gi|736761603|wp_034765168.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],68%,gi|692164220|wp_032086917.1
·f0f1atp合成酶亚单元[芽孢杆菌属[umb0893],69%,gi|1325663800|wp_101567345.1
·f0f1atp合成酶亚单元[芽孢杆菌属mku004],68%,gi|1035730730|wp_064567821.1
·多种:f0f1atp合成酶亚单元ε[芽孢杆菌],68%,gi|764371645|wp_044339984.1
·f0f1atp合成酶亚单元ε[枯草芽孢杆菌],71%,gi|1054559304|wp_066328627.1
·atp合成酶ε链[短小芽孢杆菌],71%,gi|1129140162|olp66784.1
·f0f1atp合成酶亚单元[提蒙类芽孢杆菌(bacillustimonensis)],69%,gi|498363035|wp_010677191.1
·f0f1atp合成酶亚单元[热帕拉热芽孢杆菌],69%,gi|1035712219|wp_064552783.1
·多种:f0f1atp合成酶亚单元ε[土芽孢杆菌],69%,gi|496156185|wp_008880692.1
·f0f1atp合成酶亚单元[水生芽孢杆菌],68%,gi|1430927033|wp_113969229.1
·f0f1atp合成酶亚单元ε[喜温地厌氧芽孢杆菌(anoxybacillustepidamans)],67%,gi|653159045|wp_027408017.1
·atp合成酶ε链[就地堆肥地芽孢杆菌(parageobacillustoebii)],69%,gi|1004920835|kyd29843.1
·多种:f0f1atp合成酶亚单元[杆菌科],69%,gi|1011859921|wp_062678125.1
·f0f1atp合成酶亚单元ε[碱性芽孢杆菌],71%,gi|1154169450|wp_078543435.1
·f0f1atp合成酶亚单元ε[山茶芽孢杆菌],70%,gi|1316769867|wp_101356051.1
·f0f1atp合成酶亚单元[巨球形土芽孢杆菌3],70%,gi|530784737|wp_020961503.1
·f0f1atp合成酶亚单元[芽孢杆菌属sjs],68%,gi|737427371|wp_035408062.1
·f0f1atp合成酶亚单元ε[甲醇芽孢杆菌],68%,gi|489441322|wp_003346791.1
·f0f1atp合成酶亚单元[超嗜热古芽孢杆菌(bacillushorikoshii)],68%,gi|1025825400|wp_063559286.1
·f0f1atp合成酶亚单元[咸芽孢杆菌(bacillussalsus)],69%,gi|1222808566|wp_090853330.1
·f0f1atp合成酶亚单元[水生芽孢杆菌],67%,gi|1031446442|wp_064091927.1
·f0f1atp合成酶亚单元ε[枯草芽孢杆菌],67%,gi|953347474|wp_057998216.1
·atp合成酶ε链[嗜热脂肪芽孢杆菌],69%,gi|1017231308|kze96595.1
·多种:f0f1atp合成酶亚单元ε[土芽孢杆菌],70%,gi|1017204529|wp_063167198.1
·f0f1atp合成酶亚单元ε[噬热地芽胞杆菌(geobacilluslituanicus)],70%,gi|1229394455|wp_094239354.1
·f0f1atp合成酶亚单元ε[嗜热脱氮芽孢杆菌(geobacillusthermodenitrificans)],69%,gi|1293369325|wp_100660569.1
·f0f1atp合成酶亚单元ε[巴达维亚芽胞杆菌(bacillusbataviensis)],67%,gi|494146406|wp_007086152.1
·f0f1atp合成酶亚单元[芽孢杆菌属chd6a],68%,gi|983512443|wp_060664609.1
·f0f1atp合成酶亚单元[超嗜热古芽孢杆菌],68%,gi|1207658166|wp_088019729.1
·f0f1atp合成酶亚单元[芽孢杆菌属ll01],68%,gi|847255533|wp_047970236.1
·f0f1atp合成酶亚单元ε[土壤芽孢杆菌(bacillussoli)],68%,gi|1053820122|wp_066072952.1
·f0f1atp合成酶亚单元[土芽孢杆菌属46c-iia],69%,gi|1173351582|wp_081207861.1
·f0f1atp合成酶亚单元ε[甲醇芽孢杆菌],69%,gi|489446343|wp_003351763.1
·多种:f0f1atp合成酶亚单元ε[土芽孢杆菌],68%,gi|765520579|wp_044732826.1
·f0f1atp合成酶亚单元[芽孢杆菌属fjat-14578],68%,gi|654945804|wp_028395968.1
·f0f1atp合成酶亚单元ε[热帕拉热芽孢杆菌],68%,gi|1222906556|wp_090949501.1
·f0f1atp合成酶亚单元[marisflavi芽孢杆菌],68%,gi|850295930|wp_048004873.1
·f0f1atp合成酶亚单元ε[marisflavi芽孢杆菌],68%,gi|850330602|wp_048014185.1
在一个实施例中,氨基酸接头(x2)存在于x1与x3之间,并且氨基酸接头(x4)存在于x3与x5之间。可以使用任何适合的氨基酸接头;在以下实例中提供广泛引导。在各种实施例中,x2
是长度在0-2、1-2、0、1或2个氨基酸之间的氨基酸接头。在各种其他实施例中:
·x2为选自由以下组成的群组的氨基酸接头:a、s、p、v、t、ts、id,或其中x2不存在;
·x2为选自由以下组成的群组的氨基酸接头:a、s、v、t、ts和id;或
·x2为选自由以下组成的群组的氨基酸接头:a、s、v和t。
在其他实施例中,x4是长度为1-5、1-4、1-3、1-2、1、2、3、4或5个氨基酸之间的氨基酸连接基,或其中x4不存在。在各种其他实施例中:
·x4是选自由以下组成的群组的氨基酸接头:at、a、sa、ga、ff、pppp(seqidno:20)、fl、gtsg(seqidno:21)、p、s、anefm(seqidno:22),或其中x4不存在;
·x4是选自由以下组成的群组的氨基酸接头:at、a、sa、ga、ff、fl、p和s;或
·x4是选自由以下组成的群组的氨基酸接头:a、ff、fl和gtsg(seqidno:21)。
在其他特定实施例中:
·x2和x4都是a;
·x2为s并且x4为a;
·x2不存在并且x4为ff;
·x2为v并且x4为fl;
·x2是t并且x4是gtsg(seqidno:21);或
·x2为s并且x4为sa。
在另一个实施例中,x2和x4不包括任何脯氨酸残基,以将接头维持为柔性的。
在其它实施例中,融合蛋白包含沿氨基酸序列的长度至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%一致的氨基酸序列,所述氨基酸序列选自由seqidno:7-11(名称参考以下实例中所使用的指定)组成的群组,或与以下序列相同但下文展示的接头(x2和x4)(突出显示)在以下实例中被表3中展示的其它构建体的接头取代:
seqidno:7go-ateam-a1
融合蛋白,包括mkok荧光蛋白(氨基酸1-218),第一丙氨酸接头(氨基酸219),atp结合蛋白(氨基酸220-351),第二丙氨酸接头(氨基酸352)和cpmegfp荧光蛋白(氨基酸353-598)
(m)vsvikpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvfcyghrvftkypeeipdyfkqafpeglswerslefedggsasvsahislrgntfyhkskftgvnfpadgpimqnqsvdwepstekitasdgvlkgdvtmylklegggnhkcqfkttykaakeilempgdhyighrlvrktegniteqvedavahs
(m)ktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalqralnrldvagk
seqidno:8d5
(m)vsvikpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvfcyghrvftkypeeipdyfkqafpeglswerslefedggsasvsahislrgntfyhkskftgvnfpadgpimqnqsvdwepstekitasdgvlkgdvtmylklegggnhkcqfkttykaakeilempgdhyighrlvrktegniteqvedavahs
seqidno:9f1
(m)vsvikpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvfcyghrvftkypeeipdyfkqafpeglswerslefedggsasvsahislrgntfyhkskftgvnfpadgpimqnqsvdwepstekitasdgvlkgdvtmylklegggnhkcqfkttykaakeilempgdhyighrlvrktegniteqvedavahs(无接头)
(m)ktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalqralnrldvagk
seqidno:10g1
(m)vsvikpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvfcyghrvftkypeeipdyfkqafpeglswerslefedggsasvsahislrgntfyhkskftgvnfpadgpimqnqsvdwepstekitasdgvlkgdvtmylklegggnhkcqfkttykaakeilempgdhyighrlvrktegniteqvedavahs
seqidno:11g9
(m)vsvikpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvfcyghrvftkypeeipdyfkqafpeglswerslefedggsasvsahislrgntfyhkskftgvnfpadgpimqnqsvdwepstekitasdgvlkgdvtmylklegggnhkcqfkttykaakeilempgdhyighrlvrktegniteqvedavahs
(m)ktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalqralnrldvagk
在第二方面中,本发明提供可以与本发明的第一方面的活性融合蛋白一起使用的对照融合蛋白,例如作为侦测和测量活细胞中的atp的分析中的对照。对照融合蛋白具有与本发明的第一方面的融合蛋白相同的通式(x1-x2-x3-x4-x5)。针对第一方面所公开的x1、x2、x4和x5结构域的所有实施例同样可适用于对照融合蛋白中。x3域在对照融合蛋白中的不同之处在于包含不结合到atp的对照蛋白。可使用任何适合的对照蛋白。在一个实施例中,对照蛋白包含沿seqidno:12的氨基酸序列的长度至少65%、67%、68%、70%、75%、80%、85%、90%、91%、92%、93%、94%95%、96%、97%、98%、99%或100%一致的氨基酸序列,其中加下划线的和粗体字体残基必须为k:seqidno:12
(m)ktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalq
这一实施例的对照蛋白与以上所公开的atp结合蛋白密切相关,不同之处在于具有上文所提到的所需赖氨酸残基,其消除atp结合活性。
在一个特定实施例中,对照融合蛋白包含沿seqidno:13的氨基酸序列的长度至少65%、67%、68%、70%、75%、80%、85%、90%、91%、92%、93%、94%95%、96%、97%、98%、99%或100%一致的氨基酸序列,其中带下划线和粗字体残基必须为k:
seqidno:13
mvsvikpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvfcyghrvftkypeeipdyfkqafpeglswerslefedggsasvsahislrgntfyhkskftgvnfpadgpimqnqsvdwepstekitasdgvlkgdvtmylklegggnhkcqfkttykaakeilempgdhyighrlvrktegniteqvedavahsamktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalq
在此实施例中,斜体化的和带下划线的接头残基可被改性为任何适合接头残基,包括但不限于实例的表3中列出的那些接头残基。
如在本申请通篇中所使用,术语“蛋白”或以其最广泛意义使用是指单位氨基酸序列。本文所述的多肽可以化学合成或以重组方式表达。
如所属领域的技术人员将理解,本发明的融合蛋白可以包括不存在于所描述融合蛋白中的n端、c端或这两者处的额外残基;这些额外残基不包括于测定本发明的多肽相对于参考多肽的一致性百分比中。此类残基可以是适用于预定用途的任何残基,包括但不限于适用于纯化目的的配体(his标签等)和向多肽添加功能性的额外肽结构域。
在一个实施例中,相对于参考融合蛋白的改变包含保守氨基酸取代。如本文所使用,“保守性氨基酸取代”意指不改变或实质上改变融合蛋白或结构域功能或其它特征的氨基酸或核酸取代。给定氨基酸可以由具有类似生理化学特征的残基代替,例如用一个脂肪族残基取代另一个(如ile、val、leu或ala彼此),或用一个极性残基取代另一个(如lys与arg之间;glu与asp之间;或gln和gln)。其它此类保守性取代,例如具有类似疏水性特征的整个区域的取代是众所周知的。可以在本文所描述的分析中测试包含保守性氨基酸取代的多肽以确认保留所要活性。
在另一方面,本发明提供编码根据本发明的任何实施例或实施例的组合的融合蛋白或对照融合蛋白的多核苷酸。多核苷酸可以包含rna或dna。此类多核苷酸可包含适用于促进经编码蛋白质的表达和/或纯化的额外序列,包括但不限于polya序列、经修饰的kozak序列和编码表位标签、输出信号和分泌信号的序列、核定位信号和质膜定位信号。基于本文中的教示,对所属领域的技术人员将显而易见的是,何种多核苷酸将编码本发明的融合蛋白。
在另一个方面,本发明提供重组表达载体,其包含可操作地连接到能够引导多核苷酸表达的启动子序列的本发明的任何实施例或实施例的组合的多核苷酸。“重组表达载体”包括可操作地将多核苷酸连接到能够实现融合蛋白表达的任何启动子序列的载体。可操作地连接到本发明的核酸序列的“启动子序列”是能够实现多核苷酸的表达的核酸序列。启动子不需要与多核苷酸邻接,只要其发挥引导多核苷酸表达的作用即可。因此,举例来说,插入的未翻译但转录的序列可存在于启动子序列与多核苷酸之间,且启动子序列可仍视为“可操作地连接”到编码序列。此类表达载体可以是所属领域中已知的任何类型,包括但不限于质体和基于病毒的表达载体。启动子可以是构成性的(由多种启动子中的任一种驱动,包括但不限于cmv、sv40、rsv、肌动蛋白、ef)或诱导型的(由多种诱导型启动子中的任一种驱动,包括但不限于四环素、流感、非对映异构性启动子)。在各种实施例中,表达载体可包含质粒、基于病毒的载体或任何其它适合的表达载体。
在另一方面,本公开提供包含本文所公开的重组表达载体的宿主细胞,其中所述宿主细胞可以是原核或真核细胞。细胞可以短暂地或稳定地工程化以并入本公开的表达载体。根据本公开的产生融合蛋白的方法为本公开的额外部分。所述方法包含以下步骤:(a)在有益于融合蛋白表达的条件下,培养根据本公开的这个方面的宿主,和(b)任选地,回收所表达的融合蛋白。所表达的融合蛋白可以从无细胞提取物或细胞培养基中回收。
在另一方面,本公开提供了试剂盒,其包含:
(a)本公开的任何实施例或实施例的组合的融合蛋白、根据权利要求59所述的多核苷酸、根据权利要求60所述的表达载体和/或根据权利要求61或62所述的重组宿主细胞;和
(b)本发明的任何实施例或实施例的组合的对照融合蛋白,所述多核苷酸编码所述对照融合蛋白,所述表达载体包含编码所述对照融合蛋白的所述多核苷酸和/或包含所述表达载体的所述重组宿主细胞,所述表达载体包含编码所述对照融合蛋白的所述多核苷酸。
此方面的试剂盒可以例如用于侦测和测量活细胞中的atp,如以下实例中详述。
atp结合融合蛋白
根据本公开内容,提供了一种编码融合蛋白的多核苷酸,所述融合蛋白包含:第一荧光团,其为具有受体激发波长和fret发射波长的fret受体的第一荧光团、,所述第一荧光团具有n端和c端的所述第一荧光团;具有n端和c端的atp结合蛋白;和第二荧光团,其为具有供体激发波长和供体发射波长的fret供体的第二荧光团,所述第二荧光具有n端和c端的所述第二荧光团,其中第一接头安置于所述第一荧光团的所述c端与所述atp结合蛋白的所述n端之间,并且第二接头安置于所述atp结合蛋白的所述c端与所述第二荧光团的所述n端之间,并且其中所述第一荧光团、atp结合蛋白和第二荧光团可操作地连接,使得atp通过所述atp结合蛋白对atp的结合引起所述第一荧光团和第二荧光团的相互作用以在暴露于具有所述供体激发波长的光时产生fret发射信号。
根据本公开的方面,所述atp结合融合蛋白的第一接头为丙氨酸残基并且所述第二接头为丙氨酸残基。
根据本公开的方面,所述atp结合融合蛋白的第一接头为丝氨酸残基并且所述第二接头为丙氨酸残基。
根据本发明的方面,所述atp结合融合蛋白的第一接头为无效物并且所述第二接头为苯丙胺酸-苯丙胺酸二肽残基。
根据本发明的方面,所述atp结合融合蛋白的第一接头为缬氨酸残基并且所述第二接头为苯丙胺酸-亮胺酸二肽残基。
根据本发明的方面,所述atp结合融合蛋白的第一接头为苏氨酸残基并且所述第二接头为甘氨酸-苏氨酸-丝氨酸-甘氨酸肽残基。
根据本发明的方面,所述atp结合融合蛋白的第一接头为丝氨酸残基并且所述第二接头为丝氨酸-丙氨酸二肽残基。
根据本发明的方面,所述atp结合融合蛋白的第二接头不包含天冬酰胺残基。
根据本公开的方面提供融合蛋白,所述融合蛋白包含:第一荧光团,其为具有受体激发波长和fret发射波长的fret受体,所述第一荧光团具有n端和c端;具有n端和c端的atp结合蛋白;和第二荧光团,其为具有供体激发波长和供体发射波长的fret供体,所述第二荧光团具有n端和c端,其中第一接头安置于所述第一荧光团的所述c端与所述atp结合蛋白的所述n端之间,并且第二接头安置于所述atp结合蛋白的所述c端与所述第二荧光团的所述n端之间,并且其中所述第一荧光团、atp结合蛋白和第二荧光团可操作地连接,使得所述atp结合蛋白对atp的结合引起所述第一荧光团和第二荧光团的相互作用以在暴露于具有所述供体激发波长的光时产生fret发射信号。
根据本公开的方面,atp结合蛋白包括融合蛋白是seqidno:5的atp结合蛋白:5,由seqidno:14编码。
根据本发明的各方面,包括融合蛋白的atp结合蛋白为结合atp的seqidno:5的变异体,并且与seqidno:5相比具有1、2、3、4、5、6、7、8、9或10个取代突变、添加或缺失。
根据本公开的各方面,包括融合蛋白的atp结合蛋白为结合atp的seqidno:5的变异体,并且由在高严格性杂交条件下结合于seqidno:14的seqidno:14的变异体编码。
对照融合蛋白
根据本公开的方面,提供了编码对照融合蛋白的多核苷酸,其中所述对照融合蛋白:第一荧光团,其为具有受体激发波长和受体发射波长的fret受体,所述第一荧光团具有n端和c端;具有n端和c端的对照突变体atp结合蛋白,所述对照突变atp结合蛋白的突变使得其不结合atp;以及第二荧光团,其为具有供体激发波长和供体发射波长的fret供体,第二荧光团具有n端和c端,其中第一接头安置于第一荧光团的c端和对照突变atp结合蛋白的n端之间,并且第二接头安置于对照突变atp结合蛋白的c端和第二荧光团的n端之间,并且其中第一荧光团、对照突变atp结合蛋白和第二荧光团可操作地连接以充当对照,原因为对照突变atp结合蛋白不结合atp,使得当暴露于具有供体激发波长的光时,第一荧光团和第二荧光团相互作用而产生fret发射信号,所述fret发射信号代表与atp的存在无关的非特异性背景信号。
根据本公开的方面,对照融合蛋白的第一接头是丙氨酸残基且第二接头是丙氨酸残基。
根据本公开的方面,对照融合蛋白的第一接头是丝氨酸残基且第二接头是丙氨酸残基。
根据本公开的方面,对照融合蛋白的第一接头是无效物且第二接头是苯丙氨酸-苯丙氨酸二肽残基。
根据本公开的方面,对照融合蛋白的第一接头是缬氨酸残基且第二接头是苯丙氨酸-亮氨酸二肽残基。
根据本公开的方面,对照融合蛋白的第一接头是苏氨酸残基且第二接头是甘氨酸-苏氨酸-丝氨酸-甘氨酸肽残基。
根据本公开的方面,对照融合蛋白的第一接头是丝氨酸残基且第二接头是丝氨酸-丙氨酸二肽残基。
根据本公开的方面,对照融合蛋白的第二接头不是且/或不包括天冬酰胺残基。
对照融合蛋白,所述对照融合蛋白包括:第一荧光团,其为具有受体激发波长和fret发射波长的fret受体,所述第一荧光团具有n端和c端;和具有n端和c端的对照突变atp结合蛋白;以及第二荧光团,其为具有供体激发波长和供体发射波长的fret供体,第二荧光团具有n端和c端,其中第一接头位于第一荧光团的c端和对照突变atp结合蛋白的n端之间,并且第二接头位于对照突变atp结合蛋白的c端和第二荧光团的n端之间,其中第一荧光团、对照突变atp结合蛋白和第二荧光团可操作地连接以充当对照,原因为对照突变atp结合蛋白不结合atp,使得当暴露于具有供体激发波长的光时,第一荧光团和第二荧光团相互作用而产生fret发射信号,所述fret发射信号代表与atp的存在无关的非特异性背景信号。
根据本公开的方面,对照融合蛋白所包括的atp结合蛋白是由seqidno:16编码的seqidno:15的atp结合蛋白。
根据本公开的方面,对照融合蛋白所包括的不结合atp的对照突变atp结合蛋白是seqidno:15的变异体,其不结合atp并且与seqidno:15相比具有1、2、3、4、5、6、7、8、9或10个取代突变、添加或缺失。
根据本公开的方面,融合蛋白所包括的对照突变atp结合蛋白是seqidno:15的变异体,其不结合atp且由在高严格杂交条件下结合于seqidno:16的seqidno:16变异体编码。
根据本公开的方面,本文所述的多核苷酸包括可操作地连接的启动子。根据本公开的方面,本文所述的多核苷酸包含在表达载体中。根据本公开的各方面,本文所描述的多核苷酸和/或表达载体存在于宿主细胞中,例如但不限于表达多核苷酸和其编码的融合蛋白的稳定转化体细胞系。
根据本公开的方面,供体荧光团是cpmegfp。根据本公开的方面,受体荧光团是mkok。
应当理解的是,虽然在本文中描述了本公开的实施例,其中fret供体荧光团位于atp结合蛋白或对照突变体结合蛋白的c端(接头设置在atp结合蛋白或对照突变体结合蛋白的c端与fret供体荧光团之间),并且fret受体荧光团位于atp结合蛋白或对照突变体结合蛋白的n端(接头设置在atp结合蛋白或对照突变体结合蛋白的n端与fret受体荧光团之间),但是可以任选地变换荧光团的相对位置,使得本文所述的融合蛋白的fret供体荧光团位于atp结合蛋白或对照突变体结合蛋白的n端(接头设置在atp结合蛋白或对照突变体结合蛋白的n端与fret供体荧光团之间,并且fret受体荧光团位于atp结合蛋白或对照突变体结合蛋白的c端(接头设置在atp结合蛋白或对照突变体结合蛋白的c端与fret受体荧光团之间)。
荧光团
fret供体/受体对的实例一般包括但不限于蓝绿色荧光蛋白质/黄色荧光蛋白质(cfp-yfp)fret对、绿色荧光蛋白质/红色荧光蛋白质(gfp-rfp)fret对和其它组合。
在另一个方面,本公开提供使用本文所公开的融合蛋白确定所关注的细胞中atp的水平的方法。融合蛋白侦测atp,因为atp通过融合蛋白中的atp结合蛋白的结合引起fret受体多肽与fret供体多肽的相互作用,产生可以侦测到的光发射。所发射的光的侦测可经由任何适合的分析或成像格式执行。
在一实施例中,这种方法包含
(a)在一或多个第一细胞中表达本文中公开的任何实施例或实施例的组合的融合蛋白,并且产生选自由以下组成的群组的一或多个图像:
(i)在使所述一或多个第一细胞暴露于具有fret供体多肽激发波长的光时,通过侦测由从所述一或多个第一细胞发射的具有fret受体多肽发射波长的光产生的荧光信号而产生的第一荧光图像;和/或
(ii)在使所述一或多个第一细胞暴露于具有fret受体多肽激发波长的光时,通过侦测由从所述一或多个第一细胞发射的具有所述fret受体多肽发射波长的光产生的荧光信号而产生的第二荧光图像;和/或
(iii)在使所述一或多个第一细胞暴露于具有所述fret供体多肽激发波长的光时,通过侦测由从所述一或多个第一细胞发射的具有所述fret供体多肽发射波长的光产生的荧光信号而产生的第三荧光图像;和/或
(b)通过比较所述第一荧光图像、所述第二荧光图像和/或所述第三荧光图像中的荧光信号的输出来测定所述一或多个第一细胞中的fret比;
其中所述一或多个第一细胞中的atp含量与所测定的fret比成比例。
在本实施例中,“比较”荧光信号的输出意味着将一个图像中的荧光信号的输出除以不同图像中的荧光信号的输出。例如:
·所述第一荧光图像中的荧光信号的输出可除以所述第二荧光图像中的荧光信号的输出;
·所述第一荧光图像中的荧光信号的输出可除以所述第三荧光图像中的荧光信号的输出;
·所述第二荧光图像中的荧光信号的输出可除以所述第一荧光图像中的荧光信号的所述输出;
·所述第二荧光图像中的荧光信号的所述输出可除以所述第三荧光图像中的荧光信号的所述输出;
·所述第三荧光图像中的荧光信号的输出可除以第一荧光图像中的荧光信号的输出;或
·所述第三荧光图像中的荧光信号的输出可除以第二荧光图像中的荧光信号的输出。
可基于任何合适的基础(包括但不限于以整个图像为基础、以每小区为基础、以每像素为基础或使用任何替代强度测量值)确定荧光信号的“输出”。
在另一实施例中,所述方法进一步包含在一或多个第一细胞中表达本发明的任何实施例或实施例的组合的对照融合蛋白,且侦测由具有从一或多个第一细胞发射的受体发射波长的光产生的对照信号。可使用用于使用对照信号校正所确定的fret比的任何适合方法。在一实施例中,侦测对照信号包含
(c)在一或多个对照细胞(如所述第一细胞或第二细胞)中表达本文所公开的任何实施例或实施例组合的所述对照融合蛋白,以及产生选自由以下组成的群组的一或多个图像:
(i)在使所述一或多个对照细胞暴露于具有所述fret供体多肽激发波长的光时,通过侦测由从所述一或多个对照细胞发射的具有所述fret受体多肽发射波长的光产生的荧光信号而产生的第四荧光图像;和/或
(ii)在使所述一或多个对照细胞暴露于具有所述fret受体多肽激发波长的光时,通过侦测由从所述一或多个对照细胞发射的具有所述fret受体多肽发射波长的光产生的荧光信号而产生的第五荧光图像;和/或
(iii)在使所述一或多个对照细胞暴露于具有所述fret供体多肽激发波长的光时,通过侦测由从所述一或多个对照细胞发射的具有所述fret供体多肽发射波长的光产生的荧光信号而产生的第六荧光图像;和/或
(d)通过比较所述第四荧光图像、所述第五荧光图像和/或所述第六荧光图像中的荧光信号的输出,测定所述一或多个对照细胞中的对照融合fret比;
其中所述对照融合fret比的变化经确定为与atp结合无关的实验条件的结果,并且其中基于所述对照融合fret比的变化校正所测定的fret比。
一或多个细胞可以是任何需要确定atp水平的细胞或细胞群。在一个实施例中,一或多个第一细胞在培养箱中培养。在另一实施例中,执行所有成像步骤而不从培养箱中移出一或多个第一细胞。在此实施例中,将细胞在适合的细胞培养基中在恒温箱中培养,并且配置恒温箱使得待分析的细胞不必在侦测atp的分析的观察和/或记录期间从恒温箱去除,例如由于使细胞与测试物质接触而改变atp。
所述分析可用于例如测试一或多种测试化合物对所关注的细胞中的atp含量的影响。因此,在一个实施例中,所述方法进一步使所述一或多个第一细胞与一或多种测试物质接触并且确定所述测试物质对所述一或多个第一细胞中atp的存在的影响。一种或多种测试物质对一种或多种第一细胞中atp的存在的影响可以在任何所关注时间段内确定,包括但不限于在1分钟到三个月范围内的时间段内连续地或间歇地确定。
用于细胞内atp的活细胞分析的代谢系统的
台架安装在恒温箱中,且容纳显微镜和相机。控制器控制显微镜系统且充当服务器。控制器插入到通信端口中,例如但不限于以太网端口。图形用户界面(gui)装载到计算机上并且与控制器(即服务器)交互以控制显微镜系统并且与数据交互。在根据本公开的各方面的控制器上完成所有自动化图像处理。
自动图像捕获
以下数据来源于对象(例如细胞、atp存在和/或水平)的成像,和/或针对每一对象、每一孔或每一组孔计算单独的或组合形式的一或多个额外度量,存储在数据库中,且在客户端计算机中通过图形用户界面获取数据之后不久显示给用户。
通常每2小时扫描孔,但更频繁或更不频繁的扫描也是选项。在每一次扫描之后,在那些时间点计算并存储度量,例如存储在数据库中。举例来说,在3天实验的过程中,在36个时间点收集每一度量,串接到时间系列中,且可在整个实验时间范围(即分钟、小时、天、周、月)的过程中制图。
如本文中所描述,本公开的系统和方法允许用户以自动化的中等处理量方式监测长时间内的atp存在和/或水平的变化。相比之下,先前方法为:1)终点方法(仅可获得各种所测量参数的一个或有限的读数),2)极度破坏性方法,3)需要用户将细胞取出恒温箱以用于分析/观测,4)取决于细胞数,5)atp产生的间接测量,和/或6)低处理量(一次一个孔,手动)。
在以下实例中说明本公开的组合物和方法的实施例。这些实例是出于说明性目的提供,且不被视为对所要求保护的组合物和方法的范围的限制。
实例1
接头筛选
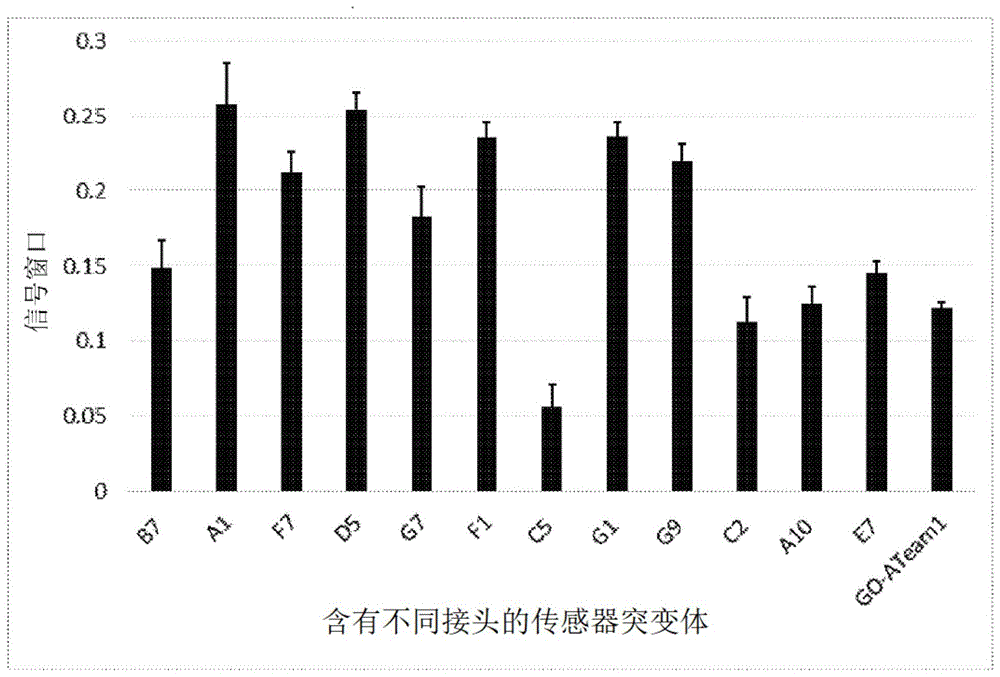
筛选了具有大约14,000个独特突变体的多样性的文库,以优化接头区域。简而言之,筛选是在表达特定突变体的细菌细胞中完成的。使用光学方法评估了超过15,000个单个菌落的亮度和fret比。在这超过15,000个菌落中,选择了400个用于使用蛋白裂解物进行进一步评估,以便可以在atp结合状态和未结合状态下进行测量。选择12个潜在的菌落进行基因测序,并将其克隆到哺乳动物表达载体中,并用于瞬时转染实验。用已知减少细胞内atp的量的药物处理表达这些构建体的细胞。通过从在治疗后2小时的载体处理的细胞的fret比减去药物处理的细胞的fret比计算信号窗口。结果如图1所示。误差线为sem,n=3。a1和go-ateam1的两尾p值等于0.0084(未配对t检验)。
以下表3显示了测试的接头序列:
产生稳定表达构建体a1和go-ateam1的细胞系,即通过慢病毒转导感染细胞,并使用抗生素选择标记进行选择以产生稳定的细胞系。用2.5μm星形孢菌素处理细胞以增加atp,并合并40mm2-脱氧-d-葡萄糖(2-dg)和4mm氰化钾(kcn)以消耗atp。表达传感器a1的细胞中的信号窗口大约是表达go-ateam1的细胞系中信号窗口的两倍(图2a-b)。
将纯化的蛋白在有和没有10mmatp的条件下孵育,并收集其激发光谱,图3a和3b。atp处理的蛋白和未处理的蛋白的光谱之间的差异表示信号窗口。传感器a1(go-ateam-a1)的表现(图3a)说明,与go-ateam1传感器的表现(图3b)相比,信号窗口(将atp结合状态(实线)与atp未结合状态(虚线)进行比较)提高了2倍。
实例2
使用根据本公开的各方面的方法和组合物评估使用葡萄糖/半乳糖交换模型将测试物质分类为无毒、细胞毒性或有丝分裂毒性的活细胞成像方法,所述方法和组合物分析活细胞中的atp。在生长培养基中用半乳糖代替葡萄糖会阻碍细胞通过糖酵解产生atp的能力,从而赋予对用于产生atp的线粒体氧化磷酸化的依赖并增强对线粒体所驱动的毒性的敏感性(ldmarroquin等人,toxicolsci97(2),539-547(2007))。在测试物质处理后最多24小时测量细胞atp水平。无毒的测试物质几乎不会改变atp,在葡萄糖和半乳糖条件下,细胞毒性的测试物质都会降低atp,而在半乳糖的条件下,有丝分裂毒性测试物质的效力向左移动。可以分钟为单位观察atp的降低,且瞬时降低和随后的恢复突出显示了使用活细胞成像方法并使用如下文进一步详细描述的根据本公开的各方面的组合物和方法的动态数据的灵敏度和值。
表达atp传感器的细胞系的产生
通过慢病毒转导产生稳定表达细胞atp(atp结合)和对照(突变atp结合结构域)传感器的细胞系。包含双顺反子表达盒的第三代慢病毒系统用于表达传感器。具体而言,ef-1α启动子被用于使用间插ires序列驱动传感器和嘌呤霉素选择标记的表达。慢病毒感染前一天,将细胞以50,000个细胞/孔接种到6孔板中。细胞在8μg/ml聚凝胺(polybrene)存在下暴露于细胞atp或对照慢病毒。24小时后,将培养基替换为无聚凝胺的培养基。使细胞传代一次,然后进行嘌呤霉素选择,以生成表达细胞atp或对照传感器的稳定且均质的细胞群。
细胞培养
细胞购自
atp分析
atp测量是通过配备有专门的基于fret的过滤器组和数据采集模块的
为了生成浓度反应曲线,使用以下公式对每种处理的数据进行了校正,并在给定的时间点根据载体对照将其归一化为:
图4a:通过2dg和kcn分别同时抑制在标准培养基中生长的细胞上的糖酵解和oxphos引起的atp消耗。数据证明了同时2dg和kcn处理的atp消耗时间和浓度依赖性。图4b和4c显示了在葡萄糖或半乳糖培养基中生长的细胞上细胞毒性化合物(氯丙嗪,图4b)和有丝分裂毒性化合物(鱼藤酮,图4c)的比较。前两幅图描述了在所示葡萄糖或半乳糖中生长的细胞中的每种化合物在24小时的过程中的影响。第三幅图描绘了每种化合物在24小时时间点的浓度-反应曲线。细胞毒性化合物在两种培养基条件下均显示相似的影响,而细胞毒性化合物在半乳糖中生长的细胞中诱导更大的atp消耗。
实例3.
材料与方法
产生了稳定表达遗传编码的荧光atp传感器或对照(非atp结合)传感器的癌细胞系。除评估谷氨酰胺酶-1(gls1)抑制剂cb-839的作用的研究外,在推荐的培养基条件下培养细胞系,在实验之前,使细胞适应补充有10%fbs的rpmi。使用配备有专用过滤器套件和数据采集模块的
数据总结
接着,利用活细胞成像方法评估癌症治疗剂对肿瘤细胞系中atp水平的影响,主要集中于针对代谢脆弱性的化合物。例如,三阴性乳腺癌(tnbc)细胞系已显示出比其受体阳性对应物更依赖于gls1的活性,gls1催化利用谷氨酰胺促进线粒体代谢的第一步。使用活细胞分析方法,在tnbc细胞系中剥夺谷氨酰胺或用cb-839抑制gls1后,atp迅速下降。在三天的时间过程中,atp的水平一直低于载体处理的细胞的atp水平。相反,在48小时内,受体阳性细胞系的反应范围包括atp的无变化到更适度的下降,以及完全恢复。相融合的定量证实与观察到atp水平恢复的条件相比,atp的持续降低与增强的抗增殖功效有关。获得了进一步的数据,该数据证明了atp传感器测量与基质细胞共培养的肿瘤细胞随时间变化的代谢扰动的能力。在具有ccd-1068sk成纤维细胞的单层上培养的tnbc显示出对cb-839处理的抗性,这是通过对atp水平的影响减弱来测量的(图5a)。相反,来自受体阳性乳腺癌细胞系的结果显示,经过cb-839治疗后,atp水平会暂时降低,该结果不受ccd-1068sk成纤维细胞的存在的影响(图5b)。
实例4.
慢病毒转导用于产生表达go-ateama1的原代和ipsc衍生神经元。当慢病毒被递送到神经元和星形胶质细胞共培养物中时,表达由突触蛋白启动子驱动,并且局限于神经元。fret比率下降表明,用2dg和kcn联合处理的神经元对atp的浓度依赖性消耗有反应。反应与在其他哺乳动物细胞(例如癌细胞系,图4a)中联合2dg和kcn处理后观察到的反应是相当的。
序列
seqidno:5-atp结合蛋白
mktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalqralnrldvagk
seqidno:14-编码seqidno:1的atp结合蛋白的dna序列
atgaaaactgtgaaagtgaatataacaacccctgatgggccagtctacgacgctgatatcgagatggtgtccgtgcgggccgagagtggtgatctcggcatcctccccggtcacattcccacaaaggccccactgaagatcggagctgtgcggctgaagaaggacggccaaaccgagatggtcgcagtctcaggcggcactgttgaagtgcggcctgaccacgttaccattaatgctcaagccgctgaaacagccgaaggaatcgacaaagagagagcagaagccgcaagacagagggcccaggagcggctgaactctcaatccgatgacaccgatattcgccgggccgagctggcactgcagagggccctgaacagactggacgtggctgggaag
seqidno:15-不结合atp的对照突变atp结合蛋白
mktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalqkalnkldvagk
seqidno:16-编码seqidno:3的对照突变atp结合蛋白的dna序列atgaaaactgtgaaagtgaatataacaacccctgatgggccagtctacgacgctgatatcgagatggtgtccgtgcgggccgagagtggtgatctcggcatcctccccggtcacattcccacaaaggccccactgaagatcggagctgtgcggctgaagaaggacggccaaaccgagatggtcgcagtctcaggcggcactgttgaagtgcggcctgaccacgttaccattaatgctcaagccgctgaaacagccgaaggaatcgacaaagagagagcagaagccgcaagacagagggcccaggagcggctgaactctcaatccgatgacaccgatattcgccgggccgagctggcactgcagaaggccctgaacaagctggacgtggctgggaaggo-ateam-a1
seqidno:17-编码mkok荧光蛋白(核苷酸1-654)、第一丙氨酸接头(核苷酸655-657)、atp结合蛋白(核苷酸658-1053)、第二丙氨酸接头(核苷酸1054-1056)和cpmegfp荧光蛋白(核苷酸1057-1794)的dna序列
atggtgagtgtgattaaaccagagatgaagatgaggtactacatggacggctccgtcaatgggcatgagttcacaattgaaggtgaaggcacaggcagaccttacgagggacatcaagagatgacactacgcgtcacaatggccgagggcgggccaatgcctttcgcgtttgacttagtgtcacacgtgttctgttacggccacagagtatttactaaatatccagaagagataccagactatttcaaacaagcatttcctgaaggcctgtcatgggaaaggtcgttggagttcgaagatggtgggtccgcttcagtcagtgcgcatataagccttagaggaaacaccttctaccacaaatccaaatttactggggttaactttcctgccgatggtcctatcatgcaaaaccaaagtgttgattgggagccatcaaccgagaaaattactgccagcgacggagttctgaagggtgatgttacgatgtacctaaaacttgaaggaggcggcaatcacaaatgccaattcaagactacttacaaggcggcaaaagagattcttgaaatgccaggagaccattacatcggccatcgcctcgtcaggaaaaccgaaggcaacattactgagcaggtagaagatgcagtagctcattccgctatgaaaactgtgaaagtgaatataacaacccctgatgggccagtctacgacgctgatatcgagatggtgtccgtgcgggccgagagtggtgatctcggcatcctccccggtcacattcccacaaaggccccactgaagatcggagctgtgcggctgaagaaggacggccaaaccgagatggtcgcagtctcaggcggcactgttgaagtgcggcctgaccacgttaccattaatgctcaagccgctgaaacagccgaaggaatcgacaaagagagagcagaagccgcaagacagagggcccaggagcggctgaactctcaatccgatgacaccgatattcgccgggccgagctggcactgcagagggccctgaacagactggacgtggctgggaaggctgacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcacccagtccaagctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagggtggcagcggtggcatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggaagct
seqidno:7-由seqidno:17编码的融合蛋白-包括mkok荧光蛋白(氨基酸1-218)、第一丙氨酸接头(氨基酸219)、atp结合蛋白(氨基酸220-351)、第二丙氨酸接头(氨基酸352)和cpmegfp荧光蛋白(氨基酸352-598)mvsvikpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvfcyghrvftkypeeipdyfkqafpeglswerslefedggsasvsahislrgntfyhkskftgvnfpadgpimqnqsvdwepstekitasdgvlkgdvtmylklegggnhkcqfkttykaakeilempgdhyighrlvrktegniteqvedavahsamktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalqralnrldvagkadgsvqladhyqqntpigdgpvllpdnhylstqsklskdpnekrdhmvllefvtaagitlgmdelykggsggmvskgeelftgvvpilveldgdvnghkfsvsgegegdatygkltlkficttgklpvpwptlvttltygvqcfsrypdhmkqhdffksampegyvqertiffkddgnyktraevkfegdtlvnrielkgidfkedgnilghkleynynshnvyimadkqkngikvnfkirhnieea
go-ateam-a1对照
seqidno:18-编码mkok荧光蛋白(核苷酸1-654)、第一丙氨酸接头(核苷酸655-657)、经突变以便不结合atp的对照atp结合蛋白(核苷酸658-1053)、第二丙氨酸接头(核苷酸1054-1056)和cpmegfp荧光蛋白(核苷酸1057-1794)的dna
atggtgagtgtgattaaaccagagatgaagatgaggtactacatggacggctccgtcaatgggcatgagttcacaattgaaggtgaaggcacaggcagaccttacgagggacatcaagagatgacactacgcgtcacaatggccgagggcgggccaatgcctttcgcgtttgacttagtgtcacacgtgttctgttacggccacagagtatttactaaatatccagaagagataccagactatttcaaacaagcatttcctgaaggcctgtcatgggaaaggtcgttggagttcgaagatggtgggtccgcttcagtcagtgcgcatataagccttagaggaaacaccttctaccacaaatccaaatttactggggttaactttcctgccgatggtcctatcatgcaaaaccaaagtgttgattgggagccatcaaccgagaaaattactgccagcgacggagttctgaagggtgatgttacgatgtacctaaaacttgaaggaggcggcaatcacaaatgccaattcaagactacttacaaggcggcaaaagagattcttgaaatgccaggagaccattacatcggccatcgcctcgtcaggaaaaccgaaggcaacattactgagcaggtagaagatgcagtagctcattccgctatgaaaactgtgaaagtgaatataacaacccctgatgggccagtctacgacgctgatatcgagatggtgtccgtgcgggccgagagtggtgatctcggcatcctccccggtcacattcccacaaaggccccactgaagatcggagctgtgcggctgaagaaggacggccaaaccgagatggtcgcagtctcaggcggcactgttgaagtgcggcctgaccacgttaccattaatgctcaagccgctgaaacagccgaaggaatcgacaaagagagagcagaagccgcaagacagagggcccaggagcggctgaactctcaatccgatgacaccgatattcgccgggccgagctggcactgcagaaggccctgaacaagctggacgtggctgggaaggctgacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcacccagtccaagctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagggtggcagcggtggcatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggaagct
seqidno:19-由seqidno:18编码的融合蛋白-包括mkok荧光蛋白(氨基酸1-218)、第一丙氨酸接头(氨基酸219)、不结合atp的对照突变atp结合蛋白(氨基酸220-351)、第二丙氨酸接头(氨基酸352)和cpmegfp荧光蛋白(氨基酸352-598)
mvsvikpemkmryymdgsvngheftiegegtgrpyeghqemtlrvtmaeggpmpfafdlvshvfcyghrvftkypeeipdyfkqafpeglswerslefedggsasvsahislrgntfyhkskftgvnfpadgpimqnqsvdwepstekitasdgvlkgdvtmylklegggnhkcqfkttykaakeilempgdhyighrlvrktegniteqvedavahsamktvkvnittpdgpvydadiemvsvraesgdlgilpghiptkaplkigavrlkkdgqtemvavsggtvevrpdhvtinaqaaetaegidkeraeaarqraqerlnsqsddtdirraelalqkalnkldvagkadgsvqladhyqqntpigdgpvllpdnhylstqsklskdpnekrdhmvllefvtaagitlgmdelykggsggmvskgeelftgvvpilveldgdvnghkfsvsgegegdatygkltlkficttgklpvpwptlvttltygvqcfsrypdhmkqhdffksampegyvqertiffkddgnyktraevkfegdtlvnrielkgidfkedgnilghkleynynshnvyimadkqkngikvnfkirhnieea
本说明书中提及的任何专利或出版物都以相同的程度通过引用并入本文,就如同每个单独的出版物均被明确地和单独地指出通过引用并入一样。
在此描述的组合物和方法目前是优选实施例的代表,是示例性的,并且不意在限制本公开的范围。本领域技术人员将想到其中的改变和其他用途。可以进行这样的改变和其他使用,而不脱离如权利要求中阐述的本公开的范围。
序列表
<110>埃森仪器公司dba埃森生物科学公司
d·阿普多恩
c·施拉姆
g·菲罗诺夫
k·施罗德
<120>用于对细胞内atp进行活细胞分析的方法和组合物
<130>18-1221-pct
<150>62/640983
<151>2018-03-09
<160>22
<170>patentin3.5版
<210>1
<211>218
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(5)
<223>任选地缺失
<220>
<221>misc_feature
<222>(217)..(218)
<223>任选地缺失
<400>1
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnphe
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahisser
210215
<210>2
<211>217
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(5)
<223>任选地缺失
<220>
<221>misc_feature
<222>(216)..(217)
<223>任选地缺失
<400>2
metservalilelysproglumetlysmetargtyrtyrmetaspgly
151015
servalasnglyhisgluphethrilegluglygluglythrglyarg
202530
protyrgluglyhisglnglumetthrleuargvalthrmetalalys
354045
glyglyprometprophealapheaspleuvalserhisvalphecys
505560
tyrglyhisargprophethrlystyrproglugluileproasptyr
65707580
phelysglnalapheprogluglyleusertrpgluargserleuglu
859095
phegluaspglyglyseralaservalseralahisileserleuarg
100105110
glyasnthrphetyrhislysserlysphethrglyvalasnphepro
115120125
alaaspglyproilemetglnasnglnservalasptrpgluproser
130135140
thrglulysilethralaseraspglyvalleulysglyaspvalthr
145150155160
mettyrleulysleugluglyglyglyasnhislyscysglnphelys
165170175
thrthrtyrlysalaalalyslysileleulysmetproglyserhis
180185190
tyrileserhisargleuvalarglysthrgluglyasnilethrglu
195200205
leuvalgluaspalavalalahisser
210215
<210>3
<211>218
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(5)
<223>任选地缺失
<220>
<221>misc_feature
<222>(217)..(218)
<223>任选地缺失
<400>3
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnmet
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahisser
210215
<210>4
<211>246
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(2)
<223>任选地缺失
<220>
<221>misc_feature
<222>(34)..(34)
<223>xaa是a或k
<220>
<221>misc_feature
<222>(245)..(246)
<223>任选地缺失
<400>4
aspglyservalglnleualaasphistyrglnglnasnthrproile
151015
glyaspglyprovalleuleuproaspasnhistyrleuserthrgln
202530
serxaaleuserlysaspproasnglulysargasphismetvalleu
354045
leugluphevalthralaalaglyilethrleuglymetaspgluleu
505560
tyrlysglyglyserglyglymetvalserlysglyglugluleuphe
65707580
thrglyvalvalproileleuvalgluleuaspglyaspvalasngly
859095
hislyspheservalserglygluglygluglyaspalathrtyrgly
100105110
lysleuthrleulyspheilecysthrthrglylysleuprovalpro
115120125
trpprothrleuvalthrthrleuthrtyrglyvalglncyspheser
130135140
argtyrproasphismetlysglnhisaspphephelysseralamet
145150155160
progluglytyrvalglngluargthrilephephelysaspaspgly
165170175
asntyrlysthrargalagluvallysphegluglyaspthrleuval
180185190
asnargilegluleulysglyileaspphelysgluaspglyasnile
195200205
leuglyhislysleuglutyrasntyrasnserhisasnvaltyrile
210215220
metalaasplysglnlysasnglyilelysvalasnphelysilearg
225230235240
hisasnileglugluala
245
<210>5
<211>132
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(1)
<223>任选地缺失
<400>5
metlysthrvallysvalasnilethrthrproaspglyprovaltyr
151015
aspalaaspileglumetvalservalargalagluserglyaspleu
202530
glyileleuproglyhisileprothrlysalaproleulysilegly
354045
alavalargleulyslysaspglyglnthrglumetvalalavalser
505560
glyglythrvalgluvalargproasphisvalthrileasnalagln
65707580
alaalagluthralagluglyileasplysgluargalaglualaala
859095
argglnargalaglngluargleuasnserglnseraspaspthrasp
100105110
ileargargalagluleualaleuglnargalaleuasnargleuasp
115120125
valalaglylys
130
<210>6
<211>133
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(1)
<223>任选地缺失
<400>6
metlysthrvallysvalasnilethrthrproaspglyprovaltyr
151015
aspalaaspileglumetvalservalargalagluserglyaspleu
202530
glyileleuproglyhisileprothrlysalaproleulysilegly
354045
alavalargleulyslysaspglyglnthrglutyrvalalavalser
505560
glyglythrvalgluvalargproasphisvalthrileasnalagln
65707580
alaalagluthralagluglyileasplysgluargalaglualaala
859095
argglnargalaglngluargleuasnserglnseraspaspthrasp
100105110
ileargargalagluleualaleuglnargalaleuasnargleuasp
115120125
valalaglumetlys
130
<210>7
<211>598
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(1)
<223>任选地缺失
<220>
<221>misc_feature
<222>(220)..(220)
<223>任选地缺失
<400>7
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnphe
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahisseralametlysthrvallys
210215220
valasnilethrthrproaspglyprovaltyraspalaaspileglu
225230235240
metvalservalargalagluserglyaspleuglyileleuprogly
245250255
hisileprothrlysalaproleulysileglyalavalargleulys
260265270
lysaspglyglnthrglumetvalalavalserglyglythrvalglu
275280285
valargproasphisvalthrileasnalaglnalaalagluthrala
290295300
gluglyileasplysgluargalaglualaalaargglnargalagln
305310315320
gluargleuasnserglnseraspaspthraspileargargalaglu
325330335
leualaleuglnargalaleuasnargleuaspvalalaglylysala
340345350
aspglyservalglnleualaasphistyrglnglnasnthrproile
355360365
glyaspglyprovalleuleuproaspasnhistyrleuserthrgln
370375380
serlysleuserlysaspproasnglulysargasphismetvalleu
385390395400
leugluphevalthralaalaglyilethrleuglymetaspgluleu
405410415
tyrlysglyglyserglyglymetvalserlysglyglugluleuphe
420425430
thrglyvalvalproileleuvalgluleuaspglyaspvalasngly
435440445
hislyspheservalserglygluglygluglyaspalathrtyrgly
450455460
lysleuthrleulyspheilecysthrthrglylysleuprovalpro
465470475480
trpprothrleuvalthrthrleuthrtyrglyvalglncyspheser
485490495
argtyrproasphismetlysglnhisaspphephelysseralamet
500505510
progluglytyrvalglngluargthrilephephelysaspaspgly
515520525
asntyrlysthrargalagluvallysphegluglyaspthrleuval
530535540
asnargilegluleulysglyileaspphelysgluaspglyasnile
545550555560
leuglyhislysleuglutyrasntyrasnserhisasnvaltyrile
565570575
metalaasplysglnlysasnglyilelysvalasnphelysilearg
580585590
hisasnileglugluala
595
<210>8
<211>598
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(1)
<223>任选地缺失
<220>
<221>misc_feature
<222>(220)..(220)
<223>任选地缺失
<400>8
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnphe
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahissersermetlysthrvallys
210215220
valasnilethrthrproaspglyprovaltyraspalaaspileglu
225230235240
metvalservalargalagluserglyaspleuglyileleuprogly
245250255
hisileprothrlysalaproleulysileglyalavalargleulys
260265270
lysaspglyglnthrglumetvalalavalserglyglythrvalglu
275280285
valargproasphisvalthrileasnalaglnalaalagluthrala
290295300
gluglyileasplysgluargalaglualaalaargglnargalagln
305310315320
gluargleuasnserglnseraspaspthraspileargargalaglu
325330335
leualaleuglnargalaleuasnargleuaspvalalaglylysala
340345350
aspglyservalglnleualaasphistyrglnglnasnthrproile
355360365
glyaspglyprovalleuleuproaspasnhistyrleuserthrgln
370375380
serlysleuserlysaspproasnglulysargasphismetvalleu
385390395400
leugluphevalthralaalaglyilethrleuglymetaspgluleu
405410415
tyrlysglyglyserglyglymetvalserlysglyglugluleuphe
420425430
thrglyvalvalproileleuvalgluleuaspglyaspvalasngly
435440445
hislyspheservalserglygluglygluglyaspalathrtyrgly
450455460
lysleuthrleulyspheilecysthrthrglylysleuprovalpro
465470475480
trpprothrleuvalthrthrleuthrtyrglyvalglncyspheser
485490495
argtyrproasphismetlysglnhisaspphephelysseralamet
500505510
progluglytyrvalglngluargthrilephephelysaspaspgly
515520525
asntyrlysthrargalagluvallysphegluglyaspthrleuval
530535540
asnargilegluleulysglyileaspphelysgluaspglyasnile
545550555560
leuglyhislysleuglutyrasntyrasnserhisasnvaltyrile
565570575
metalaasplysglnlysasnglyilelysvalasnphelysilearg
580585590
hisasnileglugluala
595
<210>9
<211>598
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(1)
<223>任选地缺失
<220>
<221>misc_feature
<222>(219)..(219)
<223>任选地缺失
<400>9
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnphe
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahissermetlysthrvallysval
210215220
asnilethrthrproaspglyprovaltyraspalaaspileglumet
225230235240
valservalargalagluserglyaspleuglyileleuproglyhis
245250255
ileprothrlysalaproleulysileglyalavalargleulyslys
260265270
aspglyglnthrglumetvalalavalserglyglythrvalgluval
275280285
argproasphisvalthrileasnalaglnalaalagluthralaglu
290295300
glyileasplysgluargalaglualaalaargglnargalaglnglu
305310315320
argleuasnserglnseraspaspthraspileargargalagluleu
325330335
alaleuglnargalaleuasnargleuaspvalalaglylysphephe
340345350
aspglyservalglnleualaasphistyrglnglnasnthrproile
355360365
glyaspglyprovalleuleuproaspasnhistyrleuserthrgln
370375380
serlysleuserlysaspproasnglulysargasphismetvalleu
385390395400
leugluphevalthralaalaglyilethrleuglymetaspgluleu
405410415
tyrlysglyglyserglyglymetvalserlysglyglugluleuphe
420425430
thrglyvalvalproileleuvalgluleuaspglyaspvalasngly
435440445
hislyspheservalserglygluglygluglyaspalathrtyrgly
450455460
lysleuthrleulyspheilecysthrthrglylysleuprovalpro
465470475480
trpprothrleuvalthrthrleuthrtyrglyvalglncyspheser
485490495
argtyrproasphismetlysglnhisaspphephelysseralamet
500505510
progluglytyrvalglngluargthrilephephelysaspaspgly
515520525
asntyrlysthrargalagluvallysphegluglyaspthrleuval
530535540
asnargilegluleulysglyileaspphelysgluaspglyasnile
545550555560
leuglyhislysleuglutyrasntyrasnserhisasnvaltyrile
565570575
metalaasplysglnlysasnglyilelysvalasnphelysilearg
580585590
hisasnileglugluala
595
<210>10
<211>599
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(1)
<223>任选地缺失
<220>
<221>misc_feature
<222>(220)..(220)
<223>任选地缺失
<400>10
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnphe
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahisservalmetlysthrvallys
210215220
valasnilethrthrproaspglyprovaltyraspalaaspileglu
225230235240
metvalservalargalagluserglyaspleuglyileleuprogly
245250255
hisileprothrlysalaproleulysileglyalavalargleulys
260265270
lysaspglyglnthrglumetvalalavalserglyglythrvalglu
275280285
valargproasphisvalthrileasnalaglnalaalagluthrala
290295300
gluglyileasplysgluargalaglualaalaargglnargalagln
305310315320
gluargleuasnserglnseraspaspthraspileargargalaglu
325330335
leualaleuglnargalaleuasnargleuaspvalalaglylysphe
340345350
leuaspglyservalglnleualaasphistyrglnglnasnthrpro
355360365
ileglyaspglyprovalleuleuproaspasnhistyrleuserthr
370375380
glnserlysleuserlysaspproasnglulysargasphismetval
385390395400
leuleugluphevalthralaalaglyilethrleuglymetaspglu
405410415
leutyrlysglyglyserglyglymetvalserlysglyglugluleu
420425430
phethrglyvalvalproileleuvalgluleuaspglyaspvalasn
435440445
glyhislyspheservalserglygluglygluglyaspalathrtyr
450455460
glylysleuthrleulyspheilecysthrthrglylysleuproval
465470475480
protrpprothrleuvalthrthrleuthrtyrglyvalglncysphe
485490495
serargtyrproasphismetlysglnhisaspphephelysserala
500505510
metprogluglytyrvalglngluargthrilephephelysaspasp
515520525
glyasntyrlysthrargalagluvallysphegluglyaspthrleu
530535540
valasnargilegluleulysglyileaspphelysgluaspglyasn
545550555560
ileleuglyhislysleuglutyrasntyrasnserhisasnvaltyr
565570575
ilemetalaasplysglnlysasnglyilelysvalasnphelysile
580585590
arghisasnileglugluala
595
<210>11
<211>601
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(1)
<223>任选地缺失
<220>
<221>misc_feature
<222>(220)..(220)
<223>任选地缺失
<400>11
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnphe
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahisserthrmetlysthrvallys
210215220
valasnilethrthrproaspglyprovaltyraspalaaspileglu
225230235240
metvalservalargalagluserglyaspleuglyileleuprogly
245250255
hisileprothrlysalaproleulysileglyalavalargleulys
260265270
lysaspglyglnthrglumetvalalavalserglyglythrvalglu
275280285
valargproasphisvalthrileasnalaglnalaalagluthrala
290295300
gluglyileasplysgluargalaglualaalaargglnargalagln
305310315320
gluargleuasnserglnseraspaspthraspileargargalaglu
325330335
leualaleuglnargalaleuasnargleuaspvalalaglylysgly
340345350
thrserglyaspglyservalglnleualaasphistyrglnglnasn
355360365
thrproileglyaspglyprovalleuleuproaspasnhistyrleu
370375380
serthrglnserlysleuserlysaspproasnglulysargasphis
385390395400
metvalleuleugluphevalthralaalaglyilethrleuglymet
405410415
aspgluleutyrlysglyglyserglyglymetvalserlysglyglu
420425430
gluleuphethrglyvalvalproileleuvalgluleuaspglyasp
435440445
valasnglyhislyspheservalserglygluglygluglyaspala
450455460
thrtyrglylysleuthrleulyspheilecysthrthrglylysleu
465470475480
provalprotrpprothrleuvalthrthrleuthrtyrglyvalgln
485490495
cyspheserargtyrproasphismetlysglnhisaspphephelys
500505510
seralametprogluglytyrvalglngluargthrilephephelys
515520525
aspaspglyasntyrlysthrargalagluvallysphegluglyasp
530535540
thrleuvalasnargilegluleulysglyileaspphelysgluasp
545550555560
glyasnileleuglyhislysleuglutyrasntyrasnserhisasn
565570575
valtyrilemetalaasplysglnlysasnglyilelysvalasnphe
580585590
lysilearghisasnileglugluala
595600
<210>12
<211>132
<212>prt
<213>人工序列
<220>
<223>合成
<220>
<221>misc_feature
<222>(1)..(1)
<223>任选地缺失
<400>12
metlysthrvallysvalasnilethrthrproaspglyprovaltyr
151015
aspalaaspileglumetvalservalargalagluserglyaspleu
202530
glyileleuproglyhisileprothrlysalaproleulysilegly
354045
alavalargleulyslysaspglyglnthrglumetvalalavalser
505560
glyglythrvalgluvalargproasphisvalthrileasnalagln
65707580
alaalagluthralagluglyileasplysgluargalaglualaala
859095
argglnargalaglngluargleuasnserglnseraspaspthrasp
100105110
ileargargalagluleualaleuglnlysalaleuasnlysleuasp
115120125
valalaglylys
130
<210>13
<211>598
<212>prt
<213>人工序列
<220>
<223>合成
<400>13
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnphe
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahisseralametlysthrvallys
210215220
valasnilethrthrproaspglyprovaltyraspalaaspileglu
225230235240
metvalservalargalagluserglyaspleuglyileleuprogly
245250255
hisileprothrlysalaproleulysileglyalavalargleulys
260265270
lysaspglyglnthrglumetvalalavalserglyglythrvalglu
275280285
valargproasphisvalthrileasnalaglnalaalagluthrala
290295300
gluglyileasplysgluargalaglualaalaargglnargalagln
305310315320
gluargleuasnserglnseraspaspthraspileargargalaglu
325330335
leualaleuglnlysalaleuasnlysleuaspvalalaglylysala
340345350
aspglyservalglnleualaasphistyrglnglnasnthrproile
355360365
glyaspglyprovalleuleuproaspasnhistyrleuserthrgln
370375380
serlysleuserlysaspproasnglulysargasphismetvalleu
385390395400
leugluphevalthralaalaglyilethrleuglymetaspgluleu
405410415
tyrlysglyglyserglyglymetvalserlysglyglugluleuphe
420425430
thrglyvalvalproileleuvalgluleuaspglyaspvalasngly
435440445
hislyspheservalserglygluglygluglyaspalathrtyrgly
450455460
lysleuthrleulyspheilecysthrthrglylysleuprovalpro
465470475480
trpprothrleuvalthrthrleuthrtyrglyvalglncyspheser
485490495
argtyrproasphismetlysglnhisaspphephelysseralamet
500505510
progluglytyrvalglngluargthrilephephelysaspaspgly
515520525
asntyrlysthrargalagluvallysphegluglyaspthrleuval
530535540
asnargilegluleulysglyileaspphelysgluaspglyasnile
545550555560
leuglyhislysleuglutyrasntyrasnserhisasnvaltyrile
565570575
metalaasplysglnlysasnglyilelysvalasnphelysilearg
580585590
hisasnileglugluala
595
<210>14
<211>396
<212>dna
<213>人工序列
<220>
<223>合成
<400>14
atgaaaactgtgaaagtgaatataacaacccctgatgggccagtctacgacgctgatatc60
gagatggtgtccgtgcgggccgagagtggtgatctcggcatcctccccggtcacattccc120
acaaaggccccactgaagatcggagctgtgcggctgaagaaggacggccaaaccgagatg180
gtcgcagtctcaggcggcactgttgaagtgcggcctgaccacgttaccattaatgctcaa240
gccgctgaaacagccgaaggaatcgacaaagagagagcagaagccgcaagacagagggcc300
caggagcggctgaactctcaatccgatgacaccgatattcgccgggccgagctggcactg360
cagagggccctgaacagactggacgtggctgggaag396
<210>15
<211>132
<212>prt
<213>人工序列
<220>
<223>合成
<400>15
metlysthrvallysvalasnilethrthrproaspglyprovaltyr
151015
aspalaaspileglumetvalservalargalagluserglyaspleu
202530
glyileleuproglyhisileprothrlysalaproleulysilegly
354045
alavalargleulyslysaspglyglnthrglumetvalalavalser
505560
glyglythrvalgluvalargproasphisvalthrileasnalagln
65707580
alaalagluthralagluglyileasplysgluargalaglualaala
859095
argglnargalaglngluargleuasnserglnseraspaspthrasp
100105110
ileargargalagluleualaleuglnlysalaleuasnlysleuasp
115120125
valalaglylys
130
<210>16
<211>396
<212>dna
<213>人工序列
<220>
<223>合成
<400>16
atgaaaactgtgaaagtgaatataacaacccctgatgggccagtctacgacgctgatatc60
gagatggtgtccgtgcgggccgagagtggtgatctcggcatcctccccggtcacattccc120
acaaaggccccactgaagatcggagctgtgcggctgaagaaggacggccaaaccgagatg180
gtcgcagtctcaggcggcactgttgaagtgcggcctgaccacgttaccattaatgctcaa240
gccgctgaaacagccgaaggaatcgacaaagagagagcagaagccgcaagacagagggcc300
caggagcggctgaactctcaatccgatgacaccgatattcgccgggccgagctggcactg360
cagaaggccctgaacaagctggacgtggctgggaag396
<210>17
<211>1794
<212>dna
<213>人工序列
<220>
<223>合成
<400>17
atggtgagtgtgattaaaccagagatgaagatgaggtactacatggacggctccgtcaat60
gggcatgagttcacaattgaaggtgaaggcacaggcagaccttacgagggacatcaagag120
atgacactacgcgtcacaatggccgagggcgggccaatgcctttcgcgtttgacttagtg180
tcacacgtgttctgttacggccacagagtatttactaaatatccagaagagataccagac240
tatttcaaacaagcatttcctgaaggcctgtcatgggaaaggtcgttggagttcgaagat300
ggtgggtccgcttcagtcagtgcgcatataagccttagaggaaacaccttctaccacaaa360
tccaaatttactggggttaactttcctgccgatggtcctatcatgcaaaaccaaagtgtt420
gattgggagccatcaaccgagaaaattactgccagcgacggagttctgaagggtgatgtt480
acgatgtacctaaaacttgaaggaggcggcaatcacaaatgccaattcaagactacttac540
aaggcggcaaaagagattcttgaaatgccaggagaccattacatcggccatcgcctcgtc600
aggaaaaccgaaggcaacattactgagcaggtagaagatgcagtagctcattccgctatg660
aaaactgtgaaagtgaatataacaacccctgatgggccagtctacgacgctgatatcgag720
atggtgtccgtgcgggccgagagtggtgatctcggcatcctccccggtcacattcccaca780
aaggccccactgaagatcggagctgtgcggctgaagaaggacggccaaaccgagatggtc840
gcagtctcaggcggcactgttgaagtgcggcctgaccacgttaccattaatgctcaagcc900
gctgaaacagccgaaggaatcgacaaagagagagcagaagccgcaagacagagggcccag960
gagcggctgaactctcaatccgatgacaccgatattcgccgggccgagctggcactgcag1020
agggccctgaacagactggacgtggctgggaaggctgacggcagcgtgcagctcgccgac1080
cactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactac1140
ctgagcacccagtccaagctgagcaaagaccccaacgagaagcgcgatcacatggtcctg1200
ctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagggtggc1260
agcggtggcatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtc1320
gagctggacggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgat1380
gccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccc1440
tggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgac1500
cacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgc1560
accatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggc1620
gacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatc1680
ctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaag1740
cagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggaagct1794
<210>18
<211>1794
<212>dna
<213>人工序列
<220>
<223>合成
<400>18
atggtgagtgtgattaaaccagagatgaagatgaggtactacatggacggctccgtcaat60
gggcatgagttcacaattgaaggtgaaggcacaggcagaccttacgagggacatcaagag120
atgacactacgcgtcacaatggccgagggcgggccaatgcctttcgcgtttgacttagtg180
tcacacgtgttctgttacggccacagagtatttactaaatatccagaagagataccagac240
tatttcaaacaagcatttcctgaaggcctgtcatgggaaaggtcgttggagttcgaagat300
ggtgggtccgcttcagtcagtgcgcatataagccttagaggaaacaccttctaccacaaa360
tccaaatttactggggttaactttcctgccgatggtcctatcatgcaaaaccaaagtgtt420
gattgggagccatcaaccgagaaaattactgccagcgacggagttctgaagggtgatgtt480
acgatgtacctaaaacttgaaggaggcggcaatcacaaatgccaattcaagactacttac540
aaggcggcaaaagagattcttgaaatgccaggagaccattacatcggccatcgcctcgtc600
aggaaaaccgaaggcaacattactgagcaggtagaagatgcagtagctcattccgctatg660
aaaactgtgaaagtgaatataacaacccctgatgggccagtctacgacgctgatatcgag720
atggtgtccgtgcgggccgagagtggtgatctcggcatcctccccggtcacattcccaca780
aaggccccactgaagatcggagctgtgcggctgaagaaggacggccaaaccgagatggtc840
gcagtctcaggcggcactgttgaagtgcggcctgaccacgttaccattaatgctcaagcc900
gctgaaacagccgaaggaatcgacaaagagagagcagaagccgcaagacagagggcccag960
gagcggctgaactctcaatccgatgacaccgatattcgccgggccgagctggcactgcag1020
aaggccctgaacaagctggacgtggctgggaaggctgacggcagcgtgcagctcgccgac1080
cactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactac1140
ctgagcacccagtccaagctgagcaaagaccccaacgagaagcgcgatcacatggtcctg1200
ctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagggtggc1260
agcggtggcatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtc1320
gagctggacggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgat1380
gccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccc1440
tggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgac1500
cacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgc1560
accatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggc1620
gacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatc1680
ctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaag1740
cagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggaagct1794
<210>19
<211>598
<212>prt
<213>人工序列
<220>
<223>合成
<400>19
metvalservalilelysproglumetlysmetargtyrtyrmetasp
151015
glyservalasnglyhisgluphethrilegluglygluglythrgly
202530
argprotyrgluglyhisglnglumetthrleuargvalthrmetala
354045
gluglyglyprometprophealapheaspleuvalserhisvalphe
505560
cystyrglyhisargvalphethrlystyrproglugluileproasp
65707580
tyrphelysglnalapheprogluglyleusertrpgluargserleu
859095
gluphegluaspglyglyseralaservalseralahisileserleu
100105110
argglyasnthrphetyrhislysserlysphethrglyvalasnphe
115120125
proalaaspglyproilemetglnasnglnservalasptrpglupro
130135140
serthrglulysilethralaseraspglyvalleulysglyaspval
145150155160
thrmettyrleulysleugluglyglyglyasnhislyscysglnphe
165170175
lysthrthrtyrlysalaalalysgluileleuglumetproglyasp
180185190
histyrileglyhisargleuvalarglysthrgluglyasnilethr
195200205
gluglnvalgluaspalavalalahisseralametlysthrvallys
210215220
valasnilethrthrproaspglyprovaltyraspalaaspileglu
225230235240
metvalservalargalagluserglyaspleuglyileleuprogly
245250255
hisileprothrlysalaproleulysileglyalavalargleulys
260265270
lysaspglyglnthrglumetvalalavalserglyglythrvalglu
275280285
valargproasphisvalthrileasnalaglnalaalagluthrala
290295300
gluglyileasplysgluargalaglualaalaargglnargalagln
305310315320
gluargleuasnserglnseraspaspthraspileargargalaglu
325330335
leualaleuglnlysalaleuasnlysleuaspvalalaglylysala
340345350
aspglyservalglnleualaasphistyrglnglnasnthrproile
355360365
glyaspglyprovalleuleuproaspasnhistyrleuserthrgln
370375380
serlysleuserlysaspproasnglulysargasphismetvalleu
385390395400
leugluphevalthralaalaglyilethrleuglymetaspgluleu
405410415
tyrlysglyglyserglyglymetvalserlysglyglugluleuphe
420425430
thrglyvalvalproileleuvalgluleuaspglyaspvalasngly
435440445
hislyspheservalserglygluglygluglyaspalathrtyrgly
450455460
lysleuthrleulyspheilecysthrthrglylysleuprovalpro
465470475480
trpprothrleuvalthrthrleuthrtyrglyvalglncyspheser
485490495
argtyrproasphismetlysglnhisaspphephelysseralamet
500505510
progluglytyrvalglngluargthrilephephelysaspaspgly
515520525
asntyrlysthrargalagluvallysphegluglyaspthrleuval
530535540
asnargilegluleulysglyileaspphelysgluaspglyasnile
545550555560
leuglyhislysleuglutyrasntyrasnserhisasnvaltyrile
565570575
metalaasplysglnlysasnglyilelysvalasnphelysilearg
580585590
hisasnileglugluala
595
<210>20
<211>4
<212>prt
<213>人工序列
<220>
<223>合成
<400>20
propropropro
1
<210>21
<211>4
<212>prt
<213>人工序列
<220>
<223>合成
<400>21
glythrsergly
1
<210>22
<211>5
<212>prt
<213>人工序列
<220>
<223>合成
<400>22
alaasngluphemet
15
- 还没有人留言评论。精彩留言会获得点赞!