与OX40L非竞争结合的抗OX40抗体的制作方法
本发明涉及抗体药物领域,具体而言,本发明涉及一种抗ox40抗体及其用途。
背景技术:
ox40是1型跨膜糖蛋白,近30年前作为一个激活的t细胞表达的细胞表面抗原得到报道。自发现以来,它被证实是t细胞和肿瘤坏死因子受体家族成员的共刺激分子,也称为“肿瘤坏死因子受体超家族成员4”或cd134,基因的名称为tnfrsf4。
ox40的分子量为50kd,具有胞质尾、跨膜结构域和胞外区。目前ox40只有一个已知配体,即ox40l(cd252),其表达在活化的apc细胞上,ox40与其配体的结合会共刺激t细胞增殖和细胞因子的产生。ox40对t细胞的刺激是通过胞浆内细胞信号传导实现的,如nf-κb以及t细胞活化核因子(nfat)的激活,后者可以提高如survivin、细胞周期蛋白依赖性激酶、bcl-2抗凋亡分子、细胞因子和细胞因子受体的表达。
ox40是长期t细胞免疫的共刺激分子,同时在cd4+t细胞应答和t细胞依赖b细胞增殖分化中起着积极的促进作用。发现ox40在小鼠t细胞表面的表达通常发生在同源抗原识别后的24小时和96小时之间。使用激活性的ox40抗体,直接促进了不同的效应t细胞亚群增加生存。
此外,ox40在肿瘤浸润淋巴细胞(til)的表达已经有了不同深入的研究,如在乳腺癌、黑色素瘤、淋巴瘤、头颈部肿瘤中。另外发现ox40在结肠癌肿瘤组织中高表达,在肠系膜淋巴结或在浸润性边缘淋巴的ox40的表达与整体生存率相关。
正是因为ox40对t细胞具有激活作用,其涉及多种疾病,并可应用于肿瘤免疫治疗及器官移植的抗排异等多个领域。因此,共刺激分子ox40属于下一代目前在肿瘤学早期临床试验中检测的免疫治疗靶点。
目前国内外已有一些公司或者机构针对ox40开始了一些研究,包括开发针对该靶点的激动剂,例如小分子及生物大分子抗体的药物开发。几种ox40单克隆抗体目前正在进行早期癌症临床试验。但是,目前的数据显示,此类激动剂大都和ox40l有对ox40结合的竞争关系,因此在此类激动剂激活ox40的同时也失去了天然配体对ox40的激活作用。例如,国际上现有的ox40抗体大都与ox40l竞争性结合ox40,即抗体与ox40的结合位点和ox40l与ox40的结合位点有部分重合,所以当抗体作为激动剂激活了ox40及其下游信号通路时,却失去了ox40l作为天然配体也同时激活ox40的功能。
因此,本领域仍然存在对新型抗ox40抗体的需要。
技术实现要素:
本发明要解决的技术问题是,提供一种新型抗ox40抗体,所述抗体对ox40具有高亲和力,能有效激活ox40蛋白及其下游的信号通路,同时所述抗体与ox40配体ox40l为非竞争结合关系,因此与ox40l具有协同激活作用,从而克服现有抗ox40抗体的不足。
针对上述技术问题,本发明的目的是提供一种新型抗ox40抗体或其功能性片段,并基于该新型ox40抗体或片段,提供其用途。
本发明的技术方案如下:
本发明的发明人以ox40蛋白的胞外区与小鼠igg2afc标签构建的融合蛋白作为免疫原,免疫小鼠,利用杂交瘤技术,筛选在分子水平和细胞水平上与ox40亲和力最好的杂交瘤细胞株,利用ht1080-ox40稳转细胞在亲和力高的细胞株里采用elisa方法筛选激活功能最好的杂交瘤细胞株并测定抗体序列。
一方面,本发明提供一种能够特异性结合ox40的抗体,所述抗体与ox40的天然配体结合ox40的不同表位。换言之,本发明提供的抗体与ox40的天然配体ox40l没有与ox40结合的竞争关系,在结合、进而激活ox40时仍不影响天然配体对ox40的结合、激活作用。
检测本发明的抗体与ox40的结合,发现本发明的抗体以2-15nm、优选4-6nm的亲和力与ox40结合。所述亲和力通过本申请实施例9中所述的biacore实验测得。
所述抗体包含重(h)链和/或轻(l)链,其中:
所述重链在其重链可变区(vh)中包含h-cdr1、h-cdr2和h-cdr3,h-cdr1具有如seqidno:1、4、7或10所示的氨基酸序列,h-cdr2具有如seqidno:2、5、8或11所示的氨基酸序列,h-cdr3具有如seqidno:3、6、9或12所示的氨基酸序列;
和/或,
所述轻链在其轻链可变区(vl)中包含l-cdr1、l-cdr2和l-cdr3,其中l-cdr1具有如seqidno:13、16、19或22所示的氨基酸序列,l-cdr2具有如seqidno:14、17、20或23所示的氨基酸序列,l-cdr3具有如seqidno:15、18、21或24所示的氨基酸序列。
优选地,关于本发明的抗体:
所述重链在其重链可变区中包含选自以下的h-cdr1、h-cdr2和h-cdr3的组合:
(1)具有如seqidno:1所示的氨基酸序列的h-cdr1、具有如seqidno:2所示的氨基酸序列的h-cdr2和具有如seqidno:3所示的氨基酸序列的h-cdr3;
(2)具有如seqidno:4所示的氨基酸序列的h-cdr1、具有如seqidno:5所示的氨基酸序列的h-cdr2和具有如seqidno:6所示的氨基酸序列的h-cdr3;
(3)具有如seqidno:7所示的氨基酸序列的h-cdr1、具有如seqidno:8所示的氨基酸序列的h-cdr2和具有如seqidno:9所示的氨基酸序列的h-cdr3;和
(4)具有如seqidno:10所示的氨基酸序列的h-cdr1、具有如seqidno:11所示的氨基酸序列的h-cdr2和具有如seqidno:12所示的氨基酸序列的h-cdr3;
和/或
所述轻链在其轻链可变区中包含选自以下的l-cdr1、l-cdr2和l-cdr3的组合:
(1)具有如seqidno:13所示的氨基酸序列的l-cdr1、具有如seqidno:14所示的氨基酸序列的l-cdr2和具有如seqidno:15所示的氨基酸序列的l-cdr3;
(2)具有如seqidno:16所示的氨基酸序列的l-cdr1、具有如seqidno:17所示的氨基酸序列的l-cdr2和具有如seqidno:18所示的氨基酸序列的l-cdr3;
(3)具有如seqidno:19所示的氨基酸序列的l-cdr1、具有如seqidno:20所示的氨基酸序列的l-cdr2和具有如seqidno:21所示的氨基酸序列的l-cdr3;和
(4)具有如seqidno:22所示的氨基酸序列的l-cdr1、具有如seqidno:23所示的氨基酸序列的l-cdr2和具有如seqidno:24所示的氨基酸序列的l-cdr3。
优选地,所述重链可变区包含如seqidno:25、26、27或28所示的氨基酸序列或者包含与如seqidno:25、26、27或28所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列;
和/或
所述轻链可变区包含如seqidno:29、30、31或32所示的氨基酸序列或者包含与如seqidno:29、30、31或32所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列。
更优选地,所述抗体包含选自以下的重链可变区和轻链可变区的组合:
(1)包含如seqidno:25所示的氨基酸序列或者包含与如seqidno:25所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的重链可变区;和
包含如seqidno:29所示的氨基酸序列或者包含与如seqidno:29所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的轻链可变区;
(2)包含如seqidno:26所示的氨基酸序列或者包含与如seqidno:26所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的重链可变区;和
包含如seqidno:30所示的氨基酸序列或者包含与如seqidno:30所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的轻链可变区;
(3)包含如seqidno:27所示的氨基酸序列或者包含与如seqidno:27所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的重链可变区;和
包含如seqidno:31所示的氨基酸序列或者包含与如seqidno:31所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的轻链可变区;以及
(4)包含如seqidno:28所示的氨基酸序列或者包含与如seqidno:28所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的重链可变区;和
包含如seqidno:32所示的氨基酸序列或者包含与如seqidno:32所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的轻链可变区。
除了可变区之外,本发明的抗体在其重链中还包含小鼠源重链恒定区;和/或,在其轻链中还包含小鼠源轻链恒定区;
更优选地,所述抗体包含选自以下的重链和轻链的组合:
(1)包含如seqidno:33所示的氨基酸序列或者包含与如seqidno:33所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的重链;和
包含如seqidno:37所示的氨基酸序列或者包含与如seqidno:37所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的轻链;
(2)包含如seqidno:34所示的氨基酸序列或者包含与如seqidno:34所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的重链;和
包含如seqidno:38所示的氨基酸序列或者包含与如seqidno:38所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的轻链;
(3)包含如seqidno:35所示的氨基酸序列或者包含与如seqidno:35所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的重链;和
包含如seqidno:39所示的氨基酸序列或者包含与如seqidno:39所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的轻链;以及
(4)包含如seqidno:36所示的氨基酸序列或者包含与如seqidno:35所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的重链;和
包含如seqidno:40所示的氨基酸序列或者包含与如seqidno:40所示的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、进一步优选至少90%、甚至优选至少91%、92%、93%、94%、95%、96%、97%、98%或99%同一性的氨基酸序列的轻链。
本发明的抗体是单克隆抗体、单链抗体、单域抗体、完全或部分人源化的抗体或者嵌合抗体;
更优选地,所述抗体是免疫球蛋白,优选为iga、igd、ige、igg或igm,更优选为igg1;
具体地,所述抗体选自抗体i3e3、h1e6、161b3和h1k1。
另一方面,本发明还提供一种核酸分子,所述核酸分子编码本发明抗体的重链可变区、轻链可变区、重链和/或轻链;
优选地,所述核酸分子包含如seqidno:41、42、43、44、45、46、47或48所示的核苷酸序列。
本发明的核酸分子可以被克隆到载体中,进而转化或转染宿主细胞。因此又一方面,本发明还提供一种载体,所述载体包含本发明的核酸分子。所述载体可以为真核表达载体、原核表达载体、人工染色体及噬菌体载体等。
本发明的载体或核酸分子可以用于转化或转染宿主细胞,用于保存或抗体表达等目的。因此,还一方面,本发明提供一种宿主细胞,所述宿主细胞包含本发明的核酸分子和/或载体,或者所述宿主细胞被本发明的核酸分子和/或载体转化或转染。宿主细胞可以是任何原核或真核细胞,例如细菌或昆虫、真菌、植物或动物细胞。
本发明提供的抗体可以利用本领域已知的任何方法获得。例如,可以先由本发明提供的核酸分子获得所述抗体的重链可变区和/或轻链可变区,或者获得所述抗体的重链和/或轻链,然后与所述抗体的任选其他结构域组装成抗体;或者,在允许本发明提供的宿主细胞表达所述抗体的重链可变区和/或轻链可变区或者所述抗体的重链和/或轻链以组装成所述抗体的情况下,培养所述宿主细胞。任选地,所述方法还包括回收产生的抗体的步骤。
本发明提供的抗体、核酸分子、载体和/或宿主细胞可以被包含在药物组合物中,更特别地被包含在药物制剂中,从而根据实际需要用于各种目的。因此,在又一方面,本发明还提供一种药物组合物,所述药物组合物包含本发明所述的抗体或其片段、核酸分子、载体和/或宿主细胞,以及任选的药学上可接受的辅料。
再一方面,本发明还提供所述抗体、核酸分子、载体、宿主细胞和/或药物组合物在制备用于预防或治疗疾病的药物中的用途,所述疾病包括肿瘤或癌症,例如黑色素瘤、肺癌、霍奇金淋巴瘤、膀胱癌、肾癌、肝癌、食管癌、非霍奇金淋巴瘤、乳腺癌、甲状腺癌、胃癌、肠癌、鼻咽癌、胰腺癌、前列腺癌、白血病、喉癌、口腔癌、耳部眼部肿瘤、胆道癌、胆囊癌、肾上腺癌、生殖系统肿瘤、多发性骨髓瘤、神经系统肿瘤。
另一方面,本发明还提供一种预防或治疗疾病的方法,所述方法包括给有此需要的受试者施用本发明的抗体、核酸分子、载体、宿主细胞和/或药物组合物,所述疾病包括肿瘤或癌症,例如黑色素瘤、肺癌、霍奇金淋巴瘤、膀胱癌、肾癌、肝癌、食管癌、非霍奇金淋巴瘤、乳腺癌、甲状腺癌、胃癌、肠癌、鼻咽癌、胰腺癌、前列腺癌、白血病、喉癌、口腔癌、耳部眼部肿瘤、胆道癌、胆囊癌、肾上腺癌、生殖系统肿瘤、多发性骨髓瘤、神经系统肿瘤;
优选地,所述受试者为哺乳类动物;更优选地,所述受试者为人。
并且,本发明还提供一种增强t细胞免疫应答的方法,所述方法包括给有此需要的受试者施用本发明的抗体、核酸分子、载体、宿主细胞和/或药物组合物;
优选地,所述受试者为哺乳类动物;更优选地,所述受试者为人。
其中,所述增强t细胞免疫应答包括增强t细胞的细胞因子产生;优选地,细胞因子包括il-8和/或ifn-γ。
在本发明的上述方法中,还可以联合施用其他药物,例如所述方法还包括给受试者施用至少一种选自下述的药物:tlr9拮抗剂例如cpg、ido1抑制剂、pd1、pdl1、ctla4等抑制剂类药物,以及肿瘤疫苗类药物。
再一方面,本发明还提供所述抗体、核酸分子、载体、宿主细胞和/或药物组合物在制备用于诊断疾病的试剂中的用途,所述疾病包括肿瘤或癌症,例如黑色素瘤、肺癌、霍奇金淋巴瘤、膀胱癌、肾癌、肝癌、食管癌、非霍奇金淋巴瘤、乳腺癌、甲状腺癌、胃癌、肠癌、鼻咽癌、胰腺癌、前列腺癌、白血病、喉癌、口腔癌、耳部眼部肿瘤、胆道癌、胆囊癌、肾上腺癌、生殖系统肿瘤、多发性骨髓瘤、神经系统肿瘤。
另一方面,本发明还提供一种诊断疾病的方法,所述方法包括使本发明的抗体、核酸分子、载体、宿主细胞和/或药物组合物与来自受试者的样品相接触,所述疾病包括肿瘤或癌症,例如黑色素瘤、肺癌、霍奇金淋巴瘤、膀胱癌、肾癌、肝癌、食管癌、非霍奇金淋巴瘤、乳腺癌、甲状腺癌、胃癌、肠癌、鼻咽癌、胰腺癌、前列腺癌、白血病、喉癌、口腔癌、耳部眼部肿瘤、胆道癌、胆囊癌、肾上腺癌、生殖系统肿瘤、多发性骨髓瘤、神经系统肿瘤;
优选地,所述受试者为哺乳类动物;更优选地,所述受试者为人。
又一方面,本发明提供一种试剂盒,所述试剂盒包括本发明的抗体、核酸分子、载体、宿主细胞和/或药物组合物。所述试剂盒可以用于治疗或诊断用途。
与现有技术相比,本发明提供了一种新型的能够特异性结合ox40的抗体。本发明的抗体如下进行筛选和制备:以ox40蛋白的胞外区与小鼠igg2afc标签构建的融合蛋白作为免疫原,免疫小鼠,利用杂交瘤技术,筛选在分子水平和细胞水平上与ox40亲和力最好的杂交瘤细胞株,利用ht1080-ox40稳转细胞在亲和力高的细胞株里采用elisa方法筛选激活功能最好的杂交瘤细胞株并测定抗体序列。最终,本发明筛选出具有出乎意料的高ox40亲和力和t细胞活化能力的抗体,例如抗体i3e3、h1e6、161b3和h1k1。
与现有抗ox40抗体相比,本发明的抗体具有以下特点:
本发明的抗ox40抗体对人源ox40蛋白具有高亲和力,能有效激活ox40蛋白及其下游的信号通路,同时本发明的抗ox40抗体与ox40l非竞争地结合,因此并不影响ox40与其配体ox40l的结合。因此,本发明的抗ox40抗体能够与ox40l协同激活ox40蛋白及其下游的信号通路,从而与下游蛋白traf2和traf5的相互作用而激活nf-κb,同时促进凋亡抑制剂bcl2和bcl2ll1/bcl2-xl的表达,从而抑制t细胞凋亡,达到长期刺激t细胞免疫的功能。因此该ox40抗体能够运用于治疗肿瘤疾病药物的制备中。另外,本发明的抗ox40抗体还可作为诊断试剂,用于诊断涉及ox40蛋白表达的疾病。
附图说明
以下,结合附图来详细说明本发明的实施方案,其中:
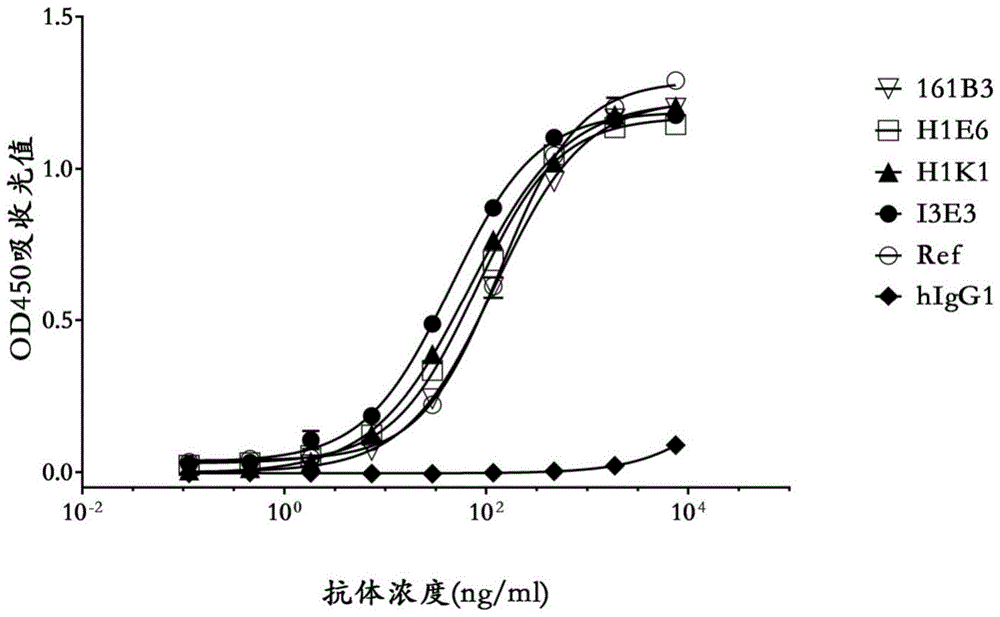
图1显示了本发明的抗体与抗原ox40结合的elisa检测结果。
图2显示了本发明的抗体与抗原ox40结合的facs检测结果。
图3显示了本发明的抗体刺激ox40稳转细胞产生il-8的elisa检测结果。
图4显示了本发明的抗体与ox40l非竞争地结合ox40的facs检测结果。
图5显示了本发明的抗体与抗原ox40结合的biacore检测结果。
图6显示了本发明的抗体增强okt3激活原代t细胞释放ifn-γ的检测结果。
具体实施方式
以下参照具体的实施例来说明本发明。本领域技术人员能够理解,这些实施例仅用于说明本发明,其不以任何方式限制本发明的范围。
下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的药材原料、试剂材料等,如无特殊说明,均为市售购买产品。
实施例1本发明杂交瘤细胞的制备
用包含ox40蛋白(来自genbank登记号nm_003327.3)的胞外区(seqidno:49)与小鼠igg2a-fc(来自genbank登记号aah31470.1)的融合蛋白作为免疫原免疫小鼠,5只小鼠皮下免疫,5只小鼠肌肉免疫,佐剂采用quickantibody5w水溶性佐剂,加强免疫以后2周测效价,选取效价高的两只小鼠进行免疫冲击,3天之后进行下文所述的细胞融合。
取准备融合的两只小鼠,取血清,然后解剖后,取脾脏,分离脾细胞,与培养的骨髓瘤细胞进行融合,铺96孔板,同时加入选择性培养基进行筛选,7天后换液,10天后进行elisa检测,选择od值大于阴性对照10倍以上的再进行流式细胞仪检测。
挑选双阳的细胞,通过细胞有限稀释法进行亚克隆铺板,挑选单克隆细胞。将挑选出的单克隆细胞,取培养上清,进行elisa检测及流式细胞仪检测,挑选双阳的细胞扩大培养。
实施例2本发明杂交瘤细胞的培养上清与ox40结合的elisa检测
用包含ox40蛋白(来自genbank登记号nm_003327.3)的胞外区(seqidno:49)与人的igg1-fc(来自genbank登记号cac20454.1)用包被液稀释至1-2μg/ml,然后以50-100μl/孔加入酶标板孔中,置4℃过夜或37℃吸附2小时。弃去孔内的液体,同时用洗涤液洗3次,每次3-5分钟,拍干。每孔加入200μl封闭液4℃过夜或37℃封闭2小时。用洗涤液洗3次,此时包被板可在-20℃或4℃保存备用。
向每孔中加入50-100μl待检杂交瘤细胞的培养上清,同时设立阳性对照(加入融合小鼠的血清)、阴性对照(加入正常小鼠的血清)和空白对照(加入培养基)。在37℃孵育1-2小时,洗涤,拍干。然后向每孔中加入酶标二抗,为1:10000稀释的辣根过氧化物酶标记的羊抗小鼠igg(sigma,货号a9044-2ml),每孔50-100μl,37℃孵育1-2小时,洗涤,拍干。向每孔中加入新鲜配制的底物显色液tmb50-100μl,37℃孵育10-30分钟。
加入2mol/lh2so4终止反应,在酶联免疫阅读仪上读取od值。
结果判定:以p/n>2:1(p代表阳性数值,n代表正常小鼠血清数值)为阳性。若阴性对照孔无色或接近无色,阳性对照孔明确显色,则可直接用肉眼观察结果。
实施例3本发明杂交瘤细胞的培养上清与ox40结合的facs检测
将ox40蛋白(来自genbank登记号nm_003327.3)的胞外区(seqidno:49)构建到plvx病毒包装载体(clontech,viruspackagemix,货号631275),转染293t细胞包装病毒,用该病毒感染hek293细胞,加药嘌呤霉素(puromycin)筛选得到耐药细胞株即稳定转染ox40的hek293。然后将hek293-ox40细胞在含2%fbs的pbs中制备成细胞浓度为107个细胞/ml的细胞悬液。
向每个流式管(样品管)中放入50μl细胞悬液,然后加入50μl待检杂交瘤细胞的培养上清,4℃孵育60分钟。向每个流式管中加入1ml的流式缓冲液,1200rpm下离心5分钟,弃上清,重复洗涤三次。同时设置对照管1(不加入培养上清和下文的二抗,仅加入细胞悬液)和对照管2(不加入培养上清,仅加入细胞悬液和下文的二抗)。
然后向每个流式管中加入100μl流式缓冲液进行重悬,根据实验要求加入5μlpe标记的抗人fc标签的二抗(biolegend,货号409304),4℃避光孵育30分钟,然后加入1ml流式缓冲液,室温下1200rpm离心5分钟,弃上清,重复洗涤三次。
再次向每个流式管中加入250μl流式缓冲液,重悬混匀,上机检测。
实施例4本发明杂交瘤细胞的培养上清刺激ox40稳转细胞产生il-8的检测
用ox40蛋白(来自genbank登记号nm_003327.3)的胞外区(seqidno:49)构建到plvx病毒包装载体(clontech,viruspackagemix,货号631275),转染293t细胞包装病毒,用该病毒感染ht1080细胞,加药嘌呤霉素(puromycin)筛选得到耐药细胞株即稳定转染ox40的ht1080。然后收集处于对数生长期的ht1080-ox40细胞,在补充10%fbs的1640培养基中制备成细胞浓度为1x106个细胞/ml的细胞悬液。
将100μl细胞悬液加入96孔板中,细胞培养箱中孵育过夜。然后弃去96孔板中的培养基,用pbs洗涤一次。加入50μl~100μl待检杂交瘤细胞的培养上清,培养箱中孵育6小时后收集上清。
用eilsa试剂盒检测上清中il-8含量。基于检测到的il-8含量,选择相应的单克隆细胞,提rna,再反转录cdna,连入测序载体中进行测序分析。示例性抗体示于下表1。
表1
对于测序得到的针对ox40靶点的抗体dna序列,重新设计引物,合成基因,连入真核表达载体中,转化dh5a感受态细胞,37℃恒温培养箱培养过夜,挑单克隆菌株送公司测序鉴定。挑选序列正确的菌株,摇菌,大提质粒,转染哺乳动物表达细胞293f,置于37℃、5%co2的培养箱中,表达培养7天。
收取表达上清,离心,过滤,选择proteing亲和层析柱,进行纯化,纯化的抗体通过sds-page电泳检测纯度,用bca蛋白检测试剂盒检测抗体浓度,分装,保存于-80℃冰箱中备用。
实施例5本发明的抗体与ox40结合的elisa检测
实验过程参照实施例2,不同之处在于,向包被板的每孔中加入待检抗体、阳性参考抗体(ref;genetech,1a7抗体,参见us2015/307617a1),和阴性参考抗体(higg1),浓度参见图1。
实验结果见图1。
实施例6本发明的抗体与ox40结合的facs检测
实验过程参照实施例3,不同之处在于,向每个流式管中放入50μl细胞悬液之后,加入待检抗体、阳性参考抗体(ref;genetech,1a7抗体,参见us2015/307617a1),和阴性参考抗体(higg1),浓度参见图2。同时设置对照管1(不加入抗体和下文的二抗,仅加入细胞悬液)和对照管2(不加入抗体,仅加入细胞悬液和下文的二抗)。
实验结果见图2。
实施例7本发明的抗体刺激ox40稳转细胞产生il-8的检测
实验过程参照实施例4,不同之处在于,向96孔板的每孔中加入待检抗体、阳性参考抗体(ref;genetech,1a7抗体,参见us2015/307617a1),和阴性参考抗体(higg1),浓度参见图3。
实验结果见图3。
实施例8本发明的抗体与ox40l非竞争关系的facs检测
将hek293-ox40细胞在含2%fbs的pbs中制备成细胞浓度为107个细胞/ml的细胞悬液。
向每个流式管(样品管)中放入50μl细胞悬液,然后加入ox40l-hfc融合蛋白(sinobiologicalinc,货号13127-h01h-50)和不同的20μg/ml本发明的抗体或阳性参考抗体(ref;genetech,1a7抗体,参见us2015/307617a1),4℃孵育60分钟。向每个流式管中加入1ml的流式缓冲液,1200rpm下离心5分钟,弃上清,重复洗涤三次。同时设置对照管1(不加入抗体、融合蛋白和下文的二抗,仅加入细胞悬液)和对照管2(不加入抗体和融合蛋白,仅加入细胞悬液和下文的二抗)。
然后向每个流式管中加入100μl流式缓冲液进行重悬,根据实验要求加入5μlpe标记的抗人fc二抗(biolegend,货号:409304),4℃避光孵育30分钟,然后加入1ml流式缓冲液,室温下1200rpm离心5分钟,弃上清,重复洗涤三次。
再次向每个流式管中加入250μl流式缓冲液,重悬混匀,上机检测。
实验结果见图4。结果表明,抗体i3e3、h1e6、161b3和h1k1在5μg/ml的浓度下均与ox40l同时结合ox40,由此证明抗体i3e3、h1e6、161b3和h1k1与ox40l均非竞争地结合ox40。
实施例9本发明表达纯化后的抗体与ox40结合的亲和力检测
将实施例1中的融合蛋白在pbs缓冲溶液中制备成最高浓度为30nm的溶液,并3倍稀释为6个浓度并设0浓度对照。将本发明的抗体在pbs中制备为20nm的溶液。
用biacoret200测定抗体抗原的亲和力。使用cm5芯片捕获本发明的抗体之后,将抗原ox40小鼠igg2afc标签蛋白流过芯片,具体条件为:流速30μl/min;抗原抗体结合时间200秒,解离时间500秒。测得的结果用仪器专用配套软件拟合,以分析抗体与抗原的亲和力。
实验结果见图5。结果表明,i3e3的kd值为4.4nm,h1k1的kd值为11nm,h1e6的kd值为5.1nm。
实施例10本发明的抗体增强okt3激活原代t细胞释放ifn-γ的检测
采用常规方法制备pmbc,然后在rpmi-1640培养基中制备成细胞浓度为5x107个细胞/ml的细胞悬液,取2ml置于5ml流式管中。然后向每ml细胞悬液中加入50μlcd4+t细胞富集混合物(cellenrichmentcocktail),混匀置于室温孵育10分钟。
每ml细胞悬液中加入100μleasyseptmd磁珠,混匀,置于室温孵育5分钟。然后补加rpmi-1640培养基至总体积2.5ml,混匀,然后将流式管置于磁铁中5分钟。颠倒磁铁和流式管,将混合物导入新的5ml流式管中。对于待分离的cd4+t细胞,按照2x106个细胞/ml的浓度加入5μg/mlpha及20u/mlil2,激活48小时。
用1μg/ml抗小鼠fc抗体(sigma,货号m3534)包被96孔板12小时。然后每孔加入105个/40μl上文激活的cd4+t细胞,再加入10μl10μg/mlokt3抗体。
之后,以20μg/ml起始以10x梯度在pbs中稀释待检抗体、阳性参考抗体(ref;genetech,1a7抗体,参见us2015/307617a1)和阴性参考抗体(higg1),加入50μl抗体稀释液,混匀,继续培养72小时。收集上清,用inf-gammaelisa试剂盒检测抗体对t细胞的激活作用。
实验结果见图6。结果表明,抗体i3e3、h1e6、161b3和h1k1在不同浓度下均可刺激产生ifn-γ,在2μg/ml浓度左右达到最大刺激。
以上对本发明具体实施方式的描述并不限制本发明,本领域技术人员可以根据本发明作出各种改变或变形,只要不脱离本发明的精神,均应属于本发明所附权利要求的范围。
序列表
<110>康源博创生物科技(北京)有限公司
<120>与ox40l非竞争结合的抗ox40抗体
<130>lc17110072-d
<160>49
<170>patentinversion3.3
<210>1
<211>10
<212>prt
<213>artificialsequence
<400>1
glyphethrpheseraspphetyrmetgly
1510
<210>2
<211>19
<212>prt
<213>artificialsequence
<400>2
alaserargasnlysalatyrasptyrthrthralatyrseralaser
151015
vallysgly
<210>3
<211>9
<212>prt
<213>artificialsequence
<400>3
aspaspglytyrtyrprometasptyr
15
<210>4
<211>10
<212>prt
<213>artificialsequence
<400>4
glytyrthrphethrsertyraspileasn
1510
<210>5
<211>17
<212>prt
<213>artificialsequence
<400>5
trpilepheproglyaspglyserthrlystyrasnglulysphelys
151015
gly
<210>6
<211>12
<212>prt
<213>artificialsequence
<400>6
trpaspglytyrtyrgluglyasptyrpheaspasn
1510
<210>7
<211>10
<212>prt
<213>artificialsequence
<400>7
glyphethrphesersertyraspmetser
1510
<210>8
<211>17
<212>prt
<213>artificialsequence
<400>8
thrileserserglyglythrtyrthrtyrpheleuaspservallys
151015
gly
<210>9
<211>12
<212>prt
<213>artificialsequence
<400>9
argtyrglyglnglyglyglytyralametasptyr
1510
<210>10
<211>10
<212>prt
<213>artificialsequence
<400>10
glytyrthrphethrsersertrpilehis
1510
<210>11
<211>17
<212>prt
<213>artificialsequence
<400>11
aspilehisproasnserglyasnthrasntyrasnglulysphelys
151015
gly
<210>12
<211>13
<212>prt
<213>artificialsequence
<400>12
sertyrglyasntyrlysasptyrtyrglymetasptyr
1510
<210>13
<211>15
<212>prt
<213>artificialsequence
<400>13
argalaserlysservalserthrserglytyrsertyrmethis
151015
<210>14
<211>7
<212>prt
<213>artificialsequence
<400>14
leualaserasnleugluser
15
<210>15
<211>9
<212>prt
<213>artificialsequence
<400>15
glnhisserarggluleuproleuthr
15
<210>16
<211>11
<212>prt
<213>artificialsequence
<400>16
lysalaserglnaspileasnlystyrileala
1510
<210>17
<211>7
<212>prt
<213>artificialsequence
<400>17
tyrthrserthrleuglnpro
15
<210>18
<211>8
<212>prt
<213>artificialsequence
<400>18
leuglntyrvalasnleutyrthr
15
<210>19
<211>10
<212>prt
<213>artificialsequence
<400>19
seralaserserservalsertyrmettyr
1510
<210>20
<211>7
<212>prt
<213>artificialsequence
<400>20
leuthrserasnleualaser
15
<210>21
<211>9
<212>prt
<213>artificialsequence
<400>21
glnglntrpserargasnproleuthr
15
<210>22
<211>15
<212>prt
<213>artificialsequence
<400>22
lysalaserglnservalasptyraspglyaspsertyrmetasn
151015
<210>23
<211>7
<212>prt
<213>artificialsequence
<400>23
alaalaserasnleugluser
15
<210>24
<211>9
<212>prt
<213>artificialsequence
<400>24
glnglnserasngluaspprotyrthr
15
<210>25
<211>120
<212>prt
<213>artificialsequence
<400>25
gluvallysleuvalgluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalathrserglyphethrpheseraspphe
202530
tyrmetglytrpvalargglnproproglylysargleuglutrpile
354045
alaalaserargasnlysalatyrasptyrthrthralatyrserala
505560
servallysglyargpheilevalserargaspthrserglnserile
65707580
leutyrleuglnmetasnalaleuargalagluaspthralailetyr
859095
tyrcysalaargaspaspglytyrtyrprometasptyrtrpglygln
100105110
glythrservalthrvalserser
115120
<210>26
<211>121
<212>prt
<213>artificialsequence
<400>26
gluvallysleuvalgluserglyalagluleuvallysproglythr
151015
servalargleusercyslysalaserglytyrthrphethrsertyr
202530
aspileasntrpvalargglnargprogluglnglyleuglutrpile
354045
glytrpilepheproglyaspglyserthrlystyrasnglulysphe
505560
lysglylysalathrleuthrthrasplysserserserthrvaltyr
65707580
metglnleuserargleuthrsergluaspseralavaltyrphecys
859095
alaargtrpaspglytyrtyrgluglyasptyrpheaspasntrpgly
100105110
glnglythrthrleuthrvalserser
115120
<210>27
<211>121
<212>prt
<213>artificialsequence
<400>27
glnvalglnleulysgluserglyglyaspleuvallysproglygly
151015
serleulysleusercysalaalapheglyphethrphesersertyr
202530
aspmetsertrpileargglnthrproasplysargleuglutrpval
354045
alathrileserserglyglythrtyrthrtyrpheleuaspserval
505560
lysglyargphethrileserargaspasnalalysasnthrleutyr
65707580
leuglnmetserserleuargsergluaspthralamettyrtyrcys
859095
alaargargtyrglyglnglyglyglytyralametasptyrtrpgly
100105110
glnglythrservalthrvalserser
115120
<210>28
<211>122
<212>prt
<213>artificialsequence
<400>28
gluvalglnleuglnglnproglyservalleuvalargproglythr
151015
servallysleusercyslysalaserglytyrthrphethrserser
202530
trpilehistrpalaargglnargproglyglnglyleuglutrpile
354045
glyaspilehisproasnserglyasnthrasntyrasnglulysphe
505560
lysglylysalathrleuthrvalaspthrserserserthralatyr
65707580
valaspleuserserleuthrsergluaspseralavaltyrtyrcys
859095
valargsertyrglyasntyrlysasptyrtyrglymetasptyrtrp
100105110
glyglnglythrservalthrvalserser
115120
<210>29
<211>111
<212>prt
<213>artificialsequence
<400>29
aspileglnleuthrglnserproalaserleualavalserleugly
151015
glnargalathrilesercysargalaserlysservalserthrser
202530
glytyrsertyrmethistrptyrglnglnlysproglyglnpropro
354045
lysleuleuiletyrleualaserasnleugluserglyvalproala
505560
argpheserglyserglyserglythraspphethrleuasnilehis
65707580
provalgluglugluaspalaalathrtyrtyrcysglnhisserarg
859095
gluleuproleuthrpheglyalaglythrlysleugluleulys
100105110
<210>30
<211>107
<212>prt
<213>artificialsequence
<400>30
aspilevalmetthrglnserproserserleuseralaserleugly
151015
glylysvalthrilethrcyslysalaserglnaspileasnlystyr
202530
ilealatrptyrglnhislysproglylysglyproargleuleuile
354045
histyrthrserthrleuglnproglyileproserargphesergly
505560
serglyserglyargasptyrserpheasnileserasnleuglupro
65707580
gluaspilealathrtyrtyrcysleuglntyrvalasnleutyrthr
859095
pheglyglyglythrlysleugluilelysarg
100105
<210>31
<211>106
<212>prt
<213>artificialsequence
<400>31
glnilevalleuthrglnserproalaleumetseralaserprogly
151015
glulysvalthrmetthrcysseralaserserservalsertyrmet
202530
tyrtrptyrglnglnlysproargserserprolysalatrpiletyr
354045
leuthrserasnleualaserglyvalproalaargpheserglyser
505560
glyserglythrsertyrserleuthrilesersermetglualaglu
65707580
aspalaalathrtyrtyrcysglnglntrpserargasnproleuthr
859095
pheglyalaglythrlysleugluleulys
100105
<210>32
<211>112
<212>prt
<213>artificialsequence
<400>32
aspileleumetthrglnserproalaserleualavalserleugly
151015
glnargalathrilesercyslysalaserglnservalasptyrasp
202530
glyaspsertyrmetasntrptyrglnglnlysproglyglnpropro
354045
lysleuleuiletyralaalaserasnleugluserglyileproala
505560
argpheserglyserglyserglythraspphethrleuasnilehis
65707580
provalgluglugluaspalaalathrtyrtyrcysglnglnserasn
859095
gluaspprotyrthrpheglyglyglythrlysleugluilelysarg
100105110
<210>33
<211>450
<212>prt
<213>artificialsequence
<400>33
gluvallysleuvalgluserglyglyglyleuvalglnproglygly
151015
serleuargleusercysalathrserglyphethrpheseraspphe
202530
tyrmetglytrpvalargglnproproglylysargleuglutrpile
354045
alaalaserargasnlysalatyrasptyrthrthralatyrserala
505560
servallysglyargpheilevalserargaspthrserglnserile
65707580
leutyrleuglnmetasnalaleuargalagluaspthralailetyr
859095
tyrcysalaargaspaspglytyrtyrprometasptyrtrpglygln
100105110
glythrservalthrvalserseralaserthrlysglyproserval
115120125
pheproleualaproserserlysserthrserglyglythralaala
130135140
leuglycysleuvallysasptyrpheprogluprovalthrvalser
145150155160
trpasnserglyalaleuthrserglyvalhisthrpheproalaval
165170175
leuglnserserglyleutyrserleuserservalvalthrvalpro
180185190
serserserleuglythrglnthrtyrilecysasnvalasnhislys
195200205
proserasnthrlysvalasplyslysvalgluprolyssercysasp
210215220
lysthrhisthrcysproprocysproalaprogluleuleuglygly
225230235240
proservalpheleupheproprolysprolysaspthrleumetile
245250255
serargthrprogluvalthrcysvalvalvalaspvalserhisglu
260265270
aspprogluvallyspheasntrptyrvalaspglyvalgluvalhis
275280285
asnalalysthrlysproargglugluglntyrasnserthrtyrarg
290295300
valvalservalleuthrvalleuhisglnasptrpleuasnglylys
305310315320
glutyrlyscyslysvalserasnlysalaleuproalaproileglu
325330335
lysthrileserlysalalysglyglnproarggluproglnvaltyr
340345350
thrleuproproserargaspgluleuthrlysasnglnvalserleu
355360365
thrcysleuvallysglyphetyrproseraspilealavalglutrp
370375380
gluserasnglyglnprogluasnasntyrlysthrthrproproval
385390395400
leuaspseraspglyserphepheleutyrserlysleuthrvalasp
405410415
lysserargtrpglnglnglyasnvalphesercysservalmethis
420425430
glualaleuhisasnhistyrthrglnlysserleuserleuserpro
435440445
glylys
450
<210>34
<211>451
<212>prt
<213>artificialsequence
<400>34
gluvallysleuvalgluserglyalagluleuvallysproglythr
151015
servalargleusercyslysalaserglytyrthrphethrsertyr
202530
aspileasntrpvalargglnargprogluglnglyleuglutrpile
354045
glytrpilepheproglyaspglyserthrlystyrasnglulysphe
505560
lysglylysalathrleuthrthrasplysserserserthrvaltyr
65707580
metglnleuserargleuthrsergluaspseralavaltyrphecys
859095
alaargtrpaspglytyrtyrgluglyasptyrpheaspasntrpgly
100105110
glnglythrthrleuthrvalserseralaserthrlysglyproser
115120125
valpheproleualaproserserlysserthrserglyglythrala
130135140
alaleuglycysleuvallysasptyrpheprogluprovalthrval
145150155160
sertrpasnserglyalaleuthrserglyvalhisthrpheproala
165170175
valleuglnserserglyleutyrserleuserservalvalthrval
180185190
proserserserleuglythrglnthrtyrilecysasnvalasnhis
195200205
lysproserasnthrlysvalasplyslysvalgluprolyssercys
210215220
asplysthrhisthrcysproprocysproalaprogluleuleugly
225230235240
glyproservalpheleupheproprolysprolysaspthrleumet
245250255
ileserargthrprogluvalthrcysvalvalvalaspvalserhis
260265270
gluaspprogluvallyspheasntrptyrvalaspglyvalgluval
275280285
hisasnalalysthrlysproargglugluglntyrasnserthrtyr
290295300
argvalvalservalleuthrvalleuhisglnasptrpleuasngly
305310315320
lysglutyrlyscyslysvalserasnlysalaleuproalaproile
325330335
glulysthrileserlysalalysglyglnproarggluproglnval
340345350
tyrthrleuproproserargaspgluleuthrlysasnglnvalser
355360365
leuthrcysleuvallysglyphetyrproseraspilealavalglu
370375380
trpgluserasnglyglnprogluasnasntyrlysthrthrpropro
385390395400
valleuaspseraspglyserphepheleutyrserlysleuthrval
405410415
asplysserargtrpglnglnglyasnvalphesercysservalmet
420425430
hisglualaleuhisasnhistyrthrglnlysserleuserleuser
435440445
proglylys
450
<210>35
<211>451
<212>prt
<213>artificialsequence
<400>35
glnvalglnleulysgluserglyglyaspleuvallysproglygly
151015
serleulysleusercysalaalapheglyphethrphesersertyr
202530
aspmetsertrpileargglnthrproasplysargleuglutrpval
354045
alathrileserserglyglythrtyrthrtyrpheleuaspserval
505560
lysglyargphethrileserargaspasnalalysasnthrleutyr
65707580
leuglnmetserserleuargsergluaspthralamettyrtyrcys
859095
alaargargtyrglyglnglyglyglytyralametasptyrtrpgly
100105110
glnglythrservalthrvalserseralaserthrlysglyproser
115120125
valpheproleualaproserserlysserthrserglyglythrala
130135140
alaleuglycysleuvallysasptyrpheprogluprovalthrval
145150155160
sertrpasnserglyalaleuthrserglyvalhisthrpheproala
165170175
valleuglnserserglyleutyrserleuserservalvalthrval
180185190
proserserserleuglythrglnthrtyrilecysasnvalasnhis
195200205
lysproserasnthrlysvalasplyslysvalgluprolyssercys
210215220
asplysthrhisthrcysproprocysproalaprogluleuleugly
225230235240
glyproservalpheleupheproprolysprolysaspthrleumet
245250255
ileserargthrprogluvalthrcysvalvalvalaspvalserhis
260265270
gluaspprogluvallyspheasntrptyrvalaspglyvalgluval
275280285
hisasnalalysthrlysproargglugluglntyrasnserthrtyr
290295300
argvalvalservalleuthrvalleuhisglnasptrpleuasngly
305310315320
lysglutyrlyscyslysvalserasnlysalaleuproalaproile
325330335
glulysthrileserlysalalysglyglnproarggluproglnval
340345350
tyrthrleuproproserargaspgluleuthrlysasnglnvalser
355360365
leuthrcysleuvallysglyphetyrproseraspilealavalglu
370375380
trpgluserasnglyglnprogluasnasntyrlysthrthrpropro
385390395400
valleuaspseraspglyserphepheleutyrserlysleuthrval
405410415
asplysserargtrpglnglnglyasnvalphesercysservalmet
420425430
hisglualaleuhisasnhistyrthrglnlysserleuserleuser
435440445
proglylys
450
<210>36
<211>452
<212>prt
<213>artificialsequence
<400>36
gluvalglnleuglnglnproglyservalleuvalargproglythr
151015
servallysleusercyslysalaserglytyrthrphethrserser
202530
trpilehistrpalaargglnargproglyglnglyleuglutrpile
354045
glyaspilehisproasnserglyasnthrasntyrasnglulysphe
505560
lysglylysalathrleuthrvalaspthrserserserthralatyr
65707580
valaspleuserserleuthrsergluaspseralavaltyrtyrcys
859095
valargsertyrglyasntyrlysasptyrtyrglymetasptyrtrp
100105110
glyglnglythrservalthrvalserseralaserthrlysglypro
115120125
servalpheproleualaproserserlysserthrserglyglythr
130135140
alaalaleuglycysleuvallysasptyrpheprogluprovalthr
145150155160
valsertrpasnserglyalaleuthrserglyvalhisthrphepro
165170175
alavalleuglnserserglyleutyrserleuserservalvalthr
180185190
valproserserserleuglythrglnthrtyrilecysasnvalasn
195200205
hislysproserasnthrlysvalasplyslysvalgluprolysser
210215220
cysasplysthrhisthrcysproprocysproalaprogluleuleu
225230235240
glyglyproservalpheleupheproprolysprolysaspthrleu
245250255
metileserargthrprogluvalthrcysvalvalvalaspvalser
260265270
hisgluaspprogluvallyspheasntrptyrvalaspglyvalglu
275280285
valhisasnalalysthrlysproargglugluglntyrasnserthr
290295300
tyrargvalvalservalleuthrvalleuhisglnasptrpleuasn
305310315320
glylysglutyrlyscyslysvalserasnlysalaleuproalapro
325330335
ileglulysthrileserlysalalysglyglnproarggluprogln
340345350
valtyrthrleuproproserargaspgluleuthrlysasnglnval
355360365
serleuthrcysleuvallysglyphetyrproseraspilealaval
370375380
glutrpgluserasnglyglnprogluasnasntyrlysthrthrpro
385390395400
provalleuaspseraspglyserphepheleutyrserlysleuthr
405410415
valasplysserargtrpglnglnglyasnvalphesercysserval
420425430
methisglualaleuhisasnhistyrthrglnlysserleuserleu
435440445
serproglylys
450
<210>37
<211>218
<212>prt
<213>artificialsequence
<400>37
aspileglnleuthrglnserproalaserleualavalserleugly
151015
glnargalathrilesercysargalaserlysservalserthrser
202530
glytyrsertyrmethistrptyrglnglnlysproglyglnpropro
354045
lysleuleuiletyrleualaserasnleugluserglyvalproala
505560
argpheserglyserglyserglythraspphethrleuasnilehis
65707580
provalgluglugluaspalaalathrtyrtyrcysglnhisserarg
859095
gluleuproleuthrpheglyalaglythrlysleugluleulysarg
100105110
thrvalalaalaproservalpheilepheproproseraspglugln
115120125
leulysserglythralaservalvalcysleuleuasnasnphetyr
130135140
proargglualalysvalglntrplysvalaspasnalaleuglnser
145150155160
glyasnserglngluservalthrgluglnaspserlysaspserthr
165170175
tyrserleuserserthrleuthrleuserlysalaasptyrglulys
180185190
hislysvaltyralacysgluvalthrhisglnglyleuserserpro
195200205
valthrlysserpheasnargglyglucys
210215
<210>38
<211>213
<212>prt
<213>artificialsequence
<400>38
aspilevalmetthrglnserproserserleuseralaserleugly
151015
glylysvalthrilethrcyslysalaserglnaspileasnlystyr
202530
ilealatrptyrglnhislysproglylysglyproargleuleuile
354045
histyrthrserthrleuglnproglyileproserargphesergly
505560
serglyserglyargasptyrserpheasnileserasnleuglupro
65707580
gluaspilealathrtyrtyrcysleuglntyrvalasnleutyrthr
859095
pheglyglyglythrlysleugluilelysargthrvalalaalapro
100105110
servalpheilepheproproseraspgluglnleulysserglythr
115120125
alaservalvalcysleuleuasnasnphetyrproargglualalys
130135140
valglntrplysvalaspasnalaleuglnserglyasnserglnglu
145150155160
servalthrgluglnaspserlysaspserthrtyrserleuserser
165170175
thrleuthrleuserlysalaasptyrglulyshislysvaltyrala
180185190
cysgluvalthrhisglnglyleuserserprovalthrlysserphe
195200205
asnargglyglucys
210
<210>39
<211>213
<212>prt
<213>artificialsequence
<400>39
glnilevalleuthrglnserproalaleumetseralaserprogly
151015
glulysvalthrmetthrcysseralaserserservalsertyrmet
202530
tyrtrptyrglnglnlysproargserserprolysalatrpiletyr
354045
leuthrserasnleualaserglyvalproalaargpheserglyser
505560
glyserglythrsertyrserleuthrilesersermetglualaglu
65707580
aspalaalathrtyrtyrcysglnglntrpserargasnproleuthr
859095
pheglyalaglythrlysleugluleulysargthrvalalaalapro
100105110
servalpheilepheproproseraspgluglnleulysserglythr
115120125
alaservalvalcysleuleuasnasnphetyrproargglualalys
130135140
valglntrplysvalaspasnalaleuglnserglyasnserglnglu
145150155160
servalthrgluglnaspserlysaspserthrtyrserleuserser
165170175
thrleuthrleuserlysalaasptyrglulyshislysvaltyrala
180185190
cysgluvalthrhisglnglyleuserserprovalthrlysserphe
195200205
asnargglyglucys
210
<210>40
<211>218
<212>prt
<213>artificialsequence
<400>40
aspileleumetthrglnserproalaserleualavalserleugly
151015
glnargalathrilesercyslysalaserglnservalasptyrasp
202530
glyaspsertyrmetasntrptyrglnglnlysproglyglnpropro
354045
lysleuleuiletyralaalaserasnleugluserglyileproala
505560
argpheserglyserglyserglythraspphethrleuasnilehis
65707580
provalgluglugluaspalaalathrtyrtyrcysglnglnserasn
859095
gluaspprotyrthrpheglyglyglythrlysleugluilelysarg
100105110
thrvalalaalaproservalpheilepheproproseraspglugln
115120125
leulysserglythralaservalvalcysleuleuasnasnphetyr
130135140
proargglualalysvalglntrplysvalaspasnalaleuglnser
145150155160
glyasnserglngluservalthrgluglnaspserlysaspserthr
165170175
tyrserleuserserthrleuthrleuserlysalaasptyrglulys
180185190
hislysvaltyralacysgluvalthrhisglnglyleuserserpro
195200205
valthrlysserpheasnargglyglucys
210215
<210>41
<211>360
<212>dna
<213>artificialsequence
<400>41
gaggtgaagctggtggaatctggaggaggcttggtacagcctgggggttctctgagactc60
tcctgtgcaacttctgggttcaccttcagtgatttctacatggggtgggtccgccagcct120
ccagggaagagactggagtggattgctgcaagtagaaacaaagcttatgattatacaaca180
gcatacagtgcatctgtgaagggtcggttcatcgtctccagagacacttcccaaagcatc240
ctctaccttcagatgaatgccctgagagctgaggacactgccatttattactgtgcaaga300
gatgatggttactaccctatggactactggggtcaaggaacctcagtcaccgtctcctca360
<210>42
<211>363
<212>dna
<213>artificialsequence
<400>42
gaggtgaagctggtggagtctggagctgagctggtaaagcctgggacttcagtgaggttg60
tcctgcaaggcttctggctacaccttcacaagctatgatataaattgggtgaggcagagg120
cctgaacagggacttgagtggattggatggatttttcctggagatggtagtactaagtac180
aatgagaagttcaagggcaaggccacactgactacagacaaatcctccagcacagtctac240
atgcagctcagcaggctgacatctgaggactctgctgtctatttctgtgcaagatgggat300
ggttactacgagggggactactttgacaactggggccaaggcaccactctcacagtctcc360
tca363
<210>43
<211>363
<212>dna
<213>artificialsequence
<400>43
caggtgcagctgaaggagagcggcggcgacctggtgaagcccggcggcagcctgaagctg60
agctgcgccgccttcggcttcaccttcagcagctacgacatgagctggatcagacagacc120
cccgacaagagactggagtgggtggccaccatcagcagcggcggcacctacacctacttc180
ctggacagcgtgaagggcagattcaccatcagcagagacaacgccaagaacaccctgtac240
ctgcagatgagcagcctgagaagcgaggacaccgccatgtactactgcgccagaagatac300
ggccagggcggcggctacgccatggactactggggccagggcaccagcgtgaccgtgagc360
agc363
<210>44
<211>366
<212>dna
<213>artificialsequence
<400>44
gaggtccagctgcagcagcctgggtctgtgctggtgaggcctggaacttcagtgaagctg60
tcctgcaaggcttctggctacaccttcaccagctcctggattcactgggcgaggcagagg120
cctggacaaggccttgagtggattggagacattcatcctaatagtggtaatactaactat180
aatgagaagttcaagggcaaggccacactgactgtagacacatcctccagtacagcctac240
gtggatctcagcagcctgacatctgaggactctgcggtctattattgtgtaagaagctat300
ggtaactacaaggattactatggtatggactattggggtcaaggaacctcagtcaccgtc360
tcctca366
<210>45
<211>334
<212>dna
<213>artificialsequence
<400>45
gacatccagctgactcagtctcctgcttccttagctgtatctctggggcagagggccacc60
atctcatgcagggccagcaaaagtgtcagtacatctggctatagttatatgcactggtac120
caacagaaaccaggacagccacccaaactcctcatctatcttgcatccaacctagaatct180
ggggtccctgccaggttcagtggcagtgggtctgggacagacttcaccctcaacatccat240
cctgtggaggaggaggatgctgcaacctattactgtcagcacagtagggagcttcccctc300
acgttcggtgctgggaccaagctggagctgaaac334
<210>46
<211>321
<212>dna
<213>artificialsequence
<400>46
gacattgtgatgacccagtctccatcctcactgtctgcatctctgggaggcaaagtcacc60
atcacttgcaaggcaagccaagacattaacaagtatatagcttggtaccaacacaagcct120
ggaaaaggtcctaggctgctcattcattacacatctacattacagccaggcatcccatca180
agattcagtggaagtgggtctgggagagattattccttcaacatcagcaacctggagcct240
gaagatattgcaacttattattgtctacagtatgttaatctgtacacgttcggagggggg300
accaagctggaaataaaacgt321
<210>47
<211>318
<212>dna
<213>artificialsequence
<400>47
cagatcgtgctgacccagagccccgccctgatgagcgccagccccggcgagaaggtgacc60
atgacctgcagcgccagcagcagcgtgagctacatgtactggtaccagcagaagcccaga120
agcagccccaaggcctggatctacctgaccagcaacctggccagcggcgtgcccgccaga180
ttcagcggcagcggcagcggcaccagctacagcctgaccatcagcagcatggaggccgag240
gacgccgccacctactactgccagcagtggagcagaaaccccctgaccttcggcgccggc300
accaagctggagctgaag318
<210>48
<211>336
<212>dna
<213>artificialsequence
<400>48
gacattctgatgacccagtctccagcttctttggctgtgtctctagggcagagggccacc60
atctcctgcaaggccagccaaagtgttgattatgatggtgatagttacatgaactggtat120
caacagaaaccaggacagccacccaaactcctcatctatgctgcatccaatctagaatct180
gggatcccagccaggtttagtggcagtgggtctgggacagacttcaccctcaacatccat240
cctgtggaggaggaggatgctgcaacctattactgtcagcaaagtaatgaggatccgtac300
acgttcggaggggggaccaagctggaaataaaacgt336
<210>49
<211>214
<212>prt
<213>homosapiens
<400>49
metcysvalglyalaargargleuglyargglyprocysalaalaleu
151015
leuleuleuglyleuglyleuserthrvalthrglyleuhiscysval
202530
glyaspthrtyrproserasnaspargcyscyshisglucysargpro
354045
glyasnglymetvalserargcysserargserglnasnthrvalcys
505560
argprocysglyproglyphetyrasnaspvalvalserserlyspro
65707580
cyslysprocysthrtrpcysasnleuargserglysergluarglys
859095
glnleucysthralathrglnaspthrvalcysargcysargalagly
100105110
thrglnproleuaspsertyrlysproglyvalaspcysalaprocys
115120125
proproglyhispheserproglyaspasnglnalacyslysprotrp
130135140
thrasncysthrleualaglylyshisthrleuglnproalaserasn
145150155160
serseraspalailecysgluaspargaspproproalathrglnpro
165170175
glngluthrglnglyproproalaargproilethrvalglnprothr
180185190
glualatrpproargthrserglnglyproserthrargprovalglu
195200205
valproglyglyargala
210
- 还没有人留言评论。精彩留言会获得点赞!