一种对绒毛组织中Siglecs家族的检测方法及其试剂盒和应用与流程
本发明是一种对绒毛组织中siglecs家族的检测方法及其试剂盒和应用,具体涉及对绒毛组织中siglecs家族核酸水平的检测方法及其使用到的引物和试剂盒,以及该检测方法在预测反复性不明原因流产中的应用,属于医学和生物技术领域。
背景技术:
反复性自然流产(recurrentspontaneousabortion,rsa)是指临床上连续2次或2次以上的自然流产,是一种常见的妊娠并发症,其发病率为0.4~0.8%,其中,40~80%的rsa原因不明,称为不明原因反复自然流产(unexplainedrecurrentspontaneousabortion,ursa)。
当前研究发现,ursa的病因复杂多样,包括母体因素(如子宫内膜容受性、免疫性抗体异常)和胚胎因素(如染色体异常)。然而,目前临床上仅能对其中部分因素进行排查,通过抽取血液检测免疫性不孕七项以判断母体免疫学因素,通过高通量测序或基因芯片都清宫术后胚胎组织进行染色体异常的鉴定。例如:
徐静等在“hif-1α、vegf在不明原因反复自然流产患者绒毛组织中的表达及与mvd的关系”,《陕西医学杂志》,2007年12月第36卷第12期,1620-1622,1627的研究中发现,hif-1α(缺氧诱导因子-1α)、vegf(血管内皮生长因子)的表达均与mvd(微血管密度)呈正相关,其结果表明:hif-1α在不明原因反复自然流产患者绒毛组织表达水平下降,通过调节vegf(血管内皮生长因子)基因的转录,进而影响滋养细胞的浸润和胎盘血管重铸,可能参与了不明原因反复自然流产的发生。
白慧玲等在“绒毛中trail及其受体的表达与不明原因反复自然流产的相关性的探讨”,《免疫学杂质》,2010年1月第26卷第1期,53-54中提到:在取不明原因反复流产患者绒毛组织进行免疫组化法检测trail及其受体的表达时发现,绒毛组织中trail及其受体dr4、dr5蛋白表达较强,而诱骗受体dcr1及dcr2蛋白与正常对照相比表达较少,p值均小于0.05,该结果表明,trail及其受体可能是造成自然流产的机制之一。
现有研究发现,由于胚胎因素(如胚胎植入能力不足)导致的不明原因流产更为复杂,因此,在不明原因反复流产病因检测过程中,目前暂未出现判断胚胎植入能力的检测方式。
技术实现要素:
本发明的目的在于提供一种对绒毛组织中siglecs家族的检测方法及其对应的试剂盒,提供了对绒毛组织中siglecs家族siglec1-3、siglec5-12、siglec13-15基因序列进行的pcr检测,用于测定绒毛组织中siglecs家族的核酸水平。
本发明的另一目的在于提供一种对绒毛组织中siglecs家族的检测和试剂盒不明原因反复自然流产中的应用,特别是因胚胎植入能力受到影响,而导致的胚胎黏附能力失败等造成的不明原因反复自然流产的检测。
本发明通过下述技术方案实现:一种对绒毛组织中siglecs家族的检测方法,提供绒毛组织siglecs家族中的siglec1-3、siglec5-12、siglec14-15制备特异性引物,将该特异性引物包被于细胞培养板上,将待测样品与反应液混合后,加入包被后的细胞培养板中进行pcr定量检测。
所述特异性引物包括:
siglec1:f-primer:ggtacgcctccaaaccaaga,r-primer:gacgttctgcacctcacaga;
siglec2:f-primer:atacctgccctgacatgcac,r-primer:gggcccctttctacttcgtt;
siglec3:f-primer:cctgtcaggtgaagttcgct,r-primer:gaaccactcctgctctggtc;
siglec5:f-primer:ccaacagctccctgatcctc,
r-primer:ctgcgggctttcactattaaaaaga;
siglec6:f-primer:tatggcagaccacaggactt,r-primer:tcagggcctttgttagctgtt;
siglec7:f-primer:agatgataacccccgacacc,r-primer:agcctcagactcctttgctg;
siglec8:f-primer:atggcaaccccctgaagaag,r-primer:tgctggaggggttgtgattc;
siglec9:f-primer:gggaaggggccaatacagac,r-primer:cacattcacagagagccggt;
siglec10:f-primer:gcggaatcagaaagccacac,r-primer:agccgaagtcccagtcctaa;
siglec11:f-primer:ccagggtcaccagcatgaat,r-primer:ttggggatggggctgaaatc;
siglec12:f-primer:aaccttgaggaatggctcgg,r-primer:gggagcctagagggttctga;
siglec14:f-primer:agggaaaagccctcaatcct,
r-primer:gttctgtcttgagcgggagg;
siglec15:f-primer:cgtctacctgttccgcttcc,r-primer:atggatgttggtggctgagg。
所述待测样品是取受试者的绒毛组织,采用trizol法或吸附柱法提取其中的总rna的含量。
所示吸附柱法中,采用吸附柱提取总rna的含量并以取至少0.5μg进行逆转录,所得cdna待检存放。
检测时,rna的用量至少为0.5μg/板。
所述反应液为rt2 real-timetm sybr green pcr master mix。
所述细胞培养板为96孔板。
一种对绒毛组织中siglecs家族检测的试剂盒,包括包被了特异性引物的细胞培养板以及反应液,所述特异性引物由绒毛组织siglecs家族中的siglec1-3、siglec5-12、siglec13-15制备得到。
一种根据所述对绒毛组织中siglecs家族的检测方法在检测不明原因反复自然流产中的应用。
一种根据所述对绒毛组织中siglecs家族检测的试剂盒在检测不明原因反复自然流产中的应用。
本发明与现有技术相比,具有以下优点及有益效果:
(1)本发明提供了一种通过对绒毛组织siglecs家族表达量的检测来鉴定不明原因反复自然流产的病因,该检测方法及其使用的试剂盒可用于反映胚胎的黏附及侵袭能力,弥补了临床上对胚胎植入能力检测的空白,为解释不明原因反复流产提供了新的思考和方向。
(2)本发明采用pcr芯片检测绒毛组织中siglecs家族核酸水平以反映胚胎黏附能力,为解释反复性不明原因流产提供新的思路,以此作为开发的检测试剂盒以流产后绒毛组织为样本,通过提取rna逆转录为cdna,采用pcr芯片检测绒毛中siglecs家族水平。最低所需rna量为0.5μg/板,为微量样本的检测提供了可能性,同时减少了相应试剂如taq酶、逆转录酶的用量,节省了实验样本及经费。
(3)本发明采用96孔板操作系统,可实现多基因同时扩增,提高了实验效率,减少误差,提高了实验可重复性。
附图说明
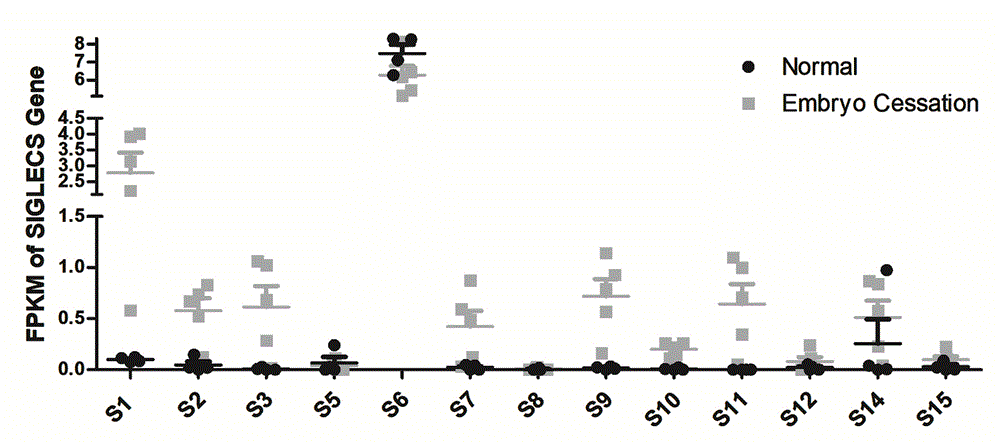
图1为实施例2中对ursa患者绒毛组织中siglecs家族的mrna水平检测的转录组数据。
图2为实施例2中对ursa患者绒毛组织中siglecs家族在ursa疾病状态下所参与的信号通路。
图3为96孔板引物排列设计图。
具体实施方式
下面结合实施例对本发明作进一步地详细说明,但本发明的实施方式不限于此。
实施例1:
本实施例涉及对绒毛组织中siglecs家族的检测方法。
(1)制备特异性引物的过程:
选取siglecs家族中的siglec1-3、siglec5-12、siglec14-15的基因序列,制备其特异性探针序列,如下表1所示:
具体制备过程如下:
采用mermade192e合成仪,通过固相亚磷酰胺三酯法合成dna引物,首先,将预先连接在固相载体cpg上的活性基团被保护的核苷酸与三氯乙酸反应,脱去其5'-羟基的保护基团dmt,获得游离的5'-羟基;然后,通过亚磷酰胺保护核苷酸单体,与活化剂四氮唑混合,得到核苷亚磷酸活化中间体,它的3'端被活化,5'-羟基仍然被dmt保护,与溶液中游离的5'-羟基发生缩合反应,合成dna的原料;缩合反应中可能有极少数5'-羟基没有参加反应(少于2%),用乙酸酐和1-甲基咪唑终止其后继续发生反应;最后,在氧化剂碘的作用下,亚磷酰形式转变为更稳定的磷酸三酯,完成dna引物的合成。
(2)包被96孔板的制备过程:将制备的特异性引物干粉用1xtebuffer溶解后放在96孔板中,密封后置于4℃过夜。包被完成后,用纯水洗涤3次,真空密封包装后存放于4℃即可。
(3)待测样品的制备过程:取受试者的绒毛组织,利用trizol法或吸附柱法提取受试者绒毛组织中总rna的含量,其中,trizol法是:采用异硫氰酸胍提取组织内总rna并逆转录为cdna后,待检存放;吸附柱法是:采用吸附柱提取总rna的含量并逆转录为cdna后,待检存放。两者制备好的rna通过nanodrop微量分光光度计测量其质量及浓度,质量要求:260/280在1.8-2.2间;浓度要求:推荐浓度为1000ng/μl。
(4)pcr定量检测及相关检测参数的设定:根据实验需要,准备至少0.5μgrna逆转录后的cdna,取一只5ml无菌eppendorf管,依次加入待检cdna,2.4ml反应液,2.4ml纯水,涡旋振荡。按每孔100μl依次加入96孔板包被孔中。操作需定量移液,建议每次更换枪头,全程置于冰上操作。上机进行pcr反应,采用三步法进行pcr,温度及时间分别为94℃3min—(94℃15s—58℃15s—72℃15s)循环35次—72℃3min。
实施例2:
本实施例涉及采用实施例1所述方法对ursa患者绒毛组织中siglecs家族的mrna水平的检测。
样本纳入条件:选取30名成都市华西第二医院患者纳入研究,其中15名为人工流产,15名为ursa患者。具体地,人工流产为正常妊娠因无生育要求,行人工流产术患者,主要表现为:既往无不良妊娠结局,本次妊娠无阴道流血、腹痛等,b超显示胚胎发育正常。ursa患者为复发性流产患者,既往无解剖、内分泌、感染等方面的疾病,本次妊娠以无任何征兆发生胚胎停育及自然流产为主要表现。
将上述两组患者的绒毛组织中siglecs家族的mrna水平通过实施例1所述方法进行检测,参见图1和图2,通过图1可证实从mrna水平上,siglecs家族的水平能够反映ursa疾病状态,通过图2可证实siglecs家族的水平能够反映胚胎的黏附能力,为试剂盒提供有力的数据基础。
实施例3:
本实施例涉及采用所述试剂盒对ursa患者绒毛组织中siglecs家族的mrna水平的检测。
该试剂盒包括包被了特异性引物的96孔板以及反应液rt2 real-timetm sybrgreen pcr master mix,特异性引物由绒毛组织siglecs家族中的siglec1-3、siglec5-12、siglec13-15制备得到,具体参见实施例1。
样本纳入条件:选取10名成都市华西第二医院患者纳入研究,其中5名为人工流产,5名为ursa患者,具体要求参见实施例2。
样本处理:待人流/清宫手术后取患者的绒毛组织少量,去除血水并冰浴研磨。用trizol法或吸附柱法提取总rna并测定浓度及质量,尽量将浓度维持于1000ng/μl,-80℃存放待检。取至少0.5μgrna进行去基因组及逆转录,将制备好的cdna置于-20℃存放待检。
试剂盒操作流程:
步骤1,根据实验需要,准备至少0.5μgrna逆转录后的cdna;
步骤2,取一只5ml无菌eppendorf管,依次加入待检cdna,2.4ml反应液,2.4ml纯水,涡旋振荡。
步骤3,按每孔100μl依次加入96孔板包被孔中,图3为96孔板引物排列设计图。操作需定量移液,建议每次更换枪头,全程置于冰上操作。
步骤4,上机进行pcr反应,采用三步法进行pcr,温度及时间分别为94℃3min—(94℃15s—58℃15s—72℃15s)循环35次—72℃3min。
步骤5,数据处理。
经上述数据处理可以发现,本实施例中,通过患者绒毛组织中siglecs家族的mrna水平反映ursa的疾病状态,并预测其胚胎的黏附能力,为后续辅助生殖提供理论基础及方向。
以上所述,仅是本发明的较佳实施例,并非对本发明做任何形式上的限制,凡是依据本发明的技术实质对以上实施例所作的任何简单修改、等同变化,均落入本发明的保护范围之内。
sequencelisting
<110>四川大学华西第二医院
<120>一种对绒毛组织中siglecs家族的检测方法及其试剂盒和应用
<130>2020
<160>39
<170>patentinversion3.3
<210>1
<211>345
<212>dna
<213>siglec1
<400>1
ggtacgcctccaaaccaagactggtgtgctgcacctgccccaggcagcctggagcgatgc60
tggcgtctacacctgccaagctgagaacggcgtgggctctttggtctcacccccatcaga120
atccacatcttcatggctgaggtccaggtgagcccagcaggtcccatcctggagaaccag180
acagtgacactagtctgcaacacacccaatgaggcacccagtgatctccgctacagatgg240
tacaagaaccatgtcctgctggaggatgcccactcccataccctccggctgcacttggcc300
actagggctgatactggcttctacttctgtgaggtgcagaacgtc345
<210>2
<211>20
<212>dna
<213>siglec1f-primer
<400>2
ggtacgcctccaaaccaaga20
<210>3
<211>20
<212>dna
<213>siglec1r-primer
<400>3
gacgttctgcacctcacaga20
<210>4
<211>121
<212>dna
<213>siglec2
<400>4
tggggagaaaaatggcaggcttctggggaaagaaagccagctgaattttgactccatctc60
cccagaagatgctgggagttacagctgctgggtgaacaactccataggacagacagcgtc120
c121
<210>5
<211>20
<212>dna
<213>siglec2f-primer
<400>5
atacctgccctgacatgcac20
<210>6
<211>20
<212>dna
<213>siglec2r-primer
<400>6
gggcccctttctacttcgtt20
<210>7
<211>153
<212>dna
<213>siglec3
<400>7
acctgtcaggtgaagttcgctgggagctggtgtgactacggagagaaccatccagctcaa60
cgtcaacgtcacctatgttccacagaacccaacaactggtatctttccaggagatggctc120
agggaaacaagagaccagagcaggagtggttca153
<210>8
<211>20
<212>dna
<213>siglec3f-primer
<400>8
cctgtcaggtgaagttcgct20
<210>9
<211>20
<212>dna
<213>siglec3r-primer
<400>9
gaaccactcctgctctggtc20
<210>10
<211>224
<212>dna
<213>siglec5
<400>10
ccaacagctccctgatcctccacggggggctcagctccgacctcaaagtcagctgcaagg60
cctggaacatctatgggtcccagagcggctctgtcctgctgctgcaagggagatcgaacc120
tccgggacaggagtggttcctgcagcccttggtggtgctggtgtcatggccctgctctgt180
atctgtctgtgcctcatcttctttttaatagtgaaagcccgcag224
<210>11
<211>20
<212>dna
<213>siglec5f-primer
<400>11
ccaacagctccctgatcctc20
<210>12
<211>25
<212>dna
<213>siglec5r-primer
<400>12
ctgcgggctttcactattaaaaaga25
<210>13
<211>200
<212>dna
<213>siglec6
<400>13
tatggcagaccacaggacttctgtccaagagaactgagttcaactccatctatgccagct60
attgagcaagtcgctttatgtccctgctctgtaaggcagggaaataatttccatctaacc120
agattattgtgaaaggtcaaagaaagcatacagctaacatacagctttgttagctgtaaa180
acagctaacaaaggccctga200
<210>14
<211>20
<212>dna
<213>siglec6f-primer
<400>14
tatggcagaccacaggactt20
<210>15
<211>21
<212>dna
<213>siglec6r-primer
<400>15
tcagggcctttgttagctgtt21
<210>16
<211>225
<212>dna
<213>siglec7
<400>16
cagatgataacccccgacaccatggcctggctgcccactcctcaggggaggaaagagaga60
tccagtatgcacccctcagctttcataagggggagcctcaggacctatcaggacaagaag120
ccaccaacaatgagtactcagagatcaagatccccaagtaagaaaatgcagaggctcggg180
cttgtttgagggttcacgacccctccagcaaaggagtctgaggct225
<210>17
<211>20
<212>dna
<213>siglec7f-primer
<400>17
agatgataacccccgacacc20
<210>18
<211>20
<212>dna
<213>siglec7r-primer
<400>18
agcctcagactcctttgctg20
<210>19
<211>214
<212>dna
<213>siglec8
<400>19
gatggcaaccccctgaagaagcctcccccagctgttgccccctcgtcaggggaggaagga60
gagctccattatgcaaccctcagcttccataaagtgaatcctcaggacccgcagggacag120
gaggccactgacagtgaatactcggagatcaagatccacaagcgagaaactgcagagact180
caggcctgtttgaggaatcacaacccctccagca214
<210>20
<211>20
<212>dna
<213>siglec8f-primer
<400>20
atggcaaccccctgaagaag20
<210>21
<211>20
<212>dna
<213>siglec8r-primer
<400>21
tgctggaggggttgtgattc20
<210>22
<211>230
<212>dna
<213>siglec9
<400>22
gggaaggggccaatacagaccaggatgctccagtggccacaaacaacccagctcgggcag60
tgtgggaggagactcgggaccgattccacctccttggggacccacataccaagaattgca120
ccctgagcatcagagatgccagaagaagtgatgcggggagatacttctttcgtatggaga180
aaggaagtataaaatggaattataaacatcaccggctctctgtgaatgtg230
<210>23
<211>20
<212>dna
<213>siglec9f-primer
<400>23
gggaaggggccaatacagac20
<210>24
<211>20
<212>dna
<213>siglec9r-primer
<400>24
cacattcacagagagccggt20
<210>25
<211>300
<212>dna
<213>siglec10
<400>25
gcggaatcagaaagccacaccaaacagtcctcggacccctcttccaccaggtgctccctc60
cccagaatccaaagaagaaccagaaaaagcagtatcagttgcccagtttcccagaaccca120
aatcatccactcaagccccagaatcccaggagagccaagaggagctccattatgccacgc180
tcaacttcccaggcgtcagacccaggcctgaggcccggatgcccaagggcacccaggcgg240
attatgcagaagtcaagttccaatgagggtctcttaggctttaggactgggacttcggct300
<210>26
<211>20
<212>dna
<213>siglec10f-primer
<400>26
gcggaatcagaaagccacac20
<210>27
<211>20
<212>dna
<213>siglec10r-primer
<400>27
agccgaagtcccagtcctaa20
<210>28
<211>367
<212>dna
<213>siglec11
<400>28
ccagggtcaccagcatgaatgctcggcaggcagctcccaagaccacccgcccccaggtgc60
agccacctacaccccggggaagggggaagagcaggagctccactatgcctccctcagctt120
ccagggcctgaggctctgggagcctgcggaccaggaggcccccagcaccaccgagtactc180
ggagatcaagatccacacaggacagcccctcaggggcccaggctttgggcttcaattgga240
gagggagatgtcagggatggttccaaagtgaagaggtctccatggcaacaggacaaccag300
caagtgtgtgggagtcgcactggtgtgacggccagaactggactcagatttcagccccat360
ccccaat367
<210>29
<211>20
<212>dna
<213>siglec11f-primer
<400>29
ccagggtcaccagcatgaat20
<210>30
<211>20
<212>dna
<213>siglec11r-primer
<400>30
ttggggatggggctgaaatc20
<210>31
<211>221
<212>dna
<213>siglec12
<400>31
caaccttgaggaatggctcggccctttcagtcctggagggccagtccctgcaccttgtct60
gtgctgtcgatagcaatccccctgccaggctgagctggacctgggggagcctgaccctga120
gcccctcacagtcctcgaaccttggggtgctggagctgcctcgagtgcatgtgaaggatg180
aaggggaattcacctgccgagctcagaaccctctaggctcc221
<210>32
<211>20
<212>dna
<213>siglec12f-primer
<400>32
aaccttgaggaatggctcgg20
<210>33
<211>20
<212>dna
<213>siglec12r-primer
<400>33
gggagcctagagggttctga20
<210>34
<211>349
<212>dna
<213>siglec14
<400>34
agggaaaagccctcaatccttcccagacctcaatgtctgggaccctggagctgcctaaca60
taggagctagagagggaggggaattcacctgccgggttcagcatccgctgggctcccagc120
acctgtccttcatcctttctgtgcagagaagctcctcttcctgcatatgtgtaactgaga180
aacagcagggctcctggcccctcgtcctcaccctgatcaggggggctctcatgggggctg240
gcttcctcctcacctatggcctcacctggatctactataccaggtgtggaggcccccagc300
agagcagggctgagaggcctggctgagcccctcccgctcaagacagaac349
<210>35
<211>20
<212>dna
<213>siglec14f-primer
<400>35
agggaaaagccctcaatcct20
<210>36
<211>20
<212>dna
<213>siglec14r-primer
<400>36
gttctgtcttgagcgggagg20
<210>37
<211>209
<212>dna
<213>siglec15
<400>37
gcgtctacctgttccgcttccatggcgccagcggggcctcgacggtcgccctcctgctcg60
gcgctctcggcttcaaggcgctgctgctgctcggggtcctggccgcccgcgctgcccgcc120
gccgcccagagcatctggacaccccggacaccccaccacggtcccaggcccaggagtcca180
attatgaaaatttgagccagatgaacccc209
<210>38
<211>20
<212>dna
<213>siglec15f-primer
<400>38
cgtctacctgttccgcttcc20
<210>39
<211>20
<212>dna
<213>siglec15r-primer
<400>39
atggatgttggtggctgagg20
- 还没有人留言评论。精彩留言会获得点赞!