OsDUF6基因在提高水稻耐旱性中的应用的制作方法
本发明涉及基因工程技术领域,更具体的说是涉及osduf6基因在提高水稻耐旱性中的应用。
背景技术:
水稻是世界上重要的粮食作物之一,养活世界上大约60%以上的人口,在我国乃至世界的粮食作物中占有非常重要的地位。但水稻产量损失日趋增多,ray等发现大约三分之一的水稻产量损失是由气候变化引起的。由于全球气候多变,水稻的产量受干旱、高温以及重金属等非生物胁迫的影响很大,其中干旱对水稻的生产的影响最为严重。近年来,随着城乡用水的加剧以及淡水资源的大面积污染,同时由于降水时空分布不均,造成水稻用水日益紧张,使水稻在生长阶段缺水从而造成减产。因此,加强对水稻抗旱和耐旱机理的研究就显得极为重要。
水稻在干旱胁迫下,为提高存活率,其自身进化出一系列的抗旱机制提高自身耐旱性。在这其中水稻调节一系列的基因参与干旱响应,这些干旱胁迫响应基因一类是功能蛋白基因(如渗透调节基因、抗氧化相关基因等),另一类是功能调控蛋白基因(如蛋白磷酸化基因和抗旱转录因子)。
当前功能调控蛋白分为两大类:蛋白激酶类和转录因子,蛋白激酶普遍存在于生物体内,并参与各种生命活动。mapk类激酶中osmpk5在根和茎中被干旱诱导表达,过表达该基因会提高水稻的耐旱性;osmpk12可以磷酸化激活oserebp1和oswrky33使其结合相应的元件而执行功能,此外该基因和osmpk8、osmpk44被干旱诱导,而osmpk4是干旱抑制基因。杨丹丹等发现osmapk15对干旱胁迫响应灵敏,说明该基因可能参与水稻耐旱机制调节。dsm1基因属于mapkkk家族,ning等发现dsm1突变体中过氧化物酶基因表达下降和相关酶的活性减弱,导致ros的积累引起突变体对干旱敏感。但该基因过表达后水稻抗旱能力增强,这可能是dsm1通过调控ros清除能力来应对水稻对干旱胁迫的应答。过表达oscdpk4后,水稻的耐旱性得到提高,保水能力增强,同时膜脂过氧化减弱以及电解质等物质渗透水平降低。受体蛋白激酶基因ossik1受干旱诱导表达,并通过多种途径提高水稻的耐旱性。
抗旱转录因子的研究是研究抗旱的一个重要领域,当前,水稻中发现许多转录因子,但是与抗旱相关的转录因子有wrky、bzip、nac、myb和ap2/erf家族等。例如,osbzip23过表达可以提高水稻的耐旱性,调节机制是脱水蛋白、膜转运蛋白的表达被该基因调控;wrky类转录因子wrky30的过表达可以增强水稻的耐旱性;nac家族转录因子在水稻耐旱性中发挥着重要的作用,如osnac6、osnac9的过表达提高水稻根数和直径,进而提高水稻的抗旱性。
此外,还有其它基因在水稻干旱胁迫过程中发挥着作用,如osdt11基因是crp(富含半胱氨酸短肽)短肽,其在水稻过表达后显著提高水稻的耐旱性,这是首次发现crp类短肽参与抗旱,对抗旱机制的研究和生产具有重大的意义。罗广宇等发现丝氨酸羧肽酶(scp)家族基因osscpd1突变体的耐旱性比对照低,过表达该基因后水稻的耐旱性增强,根据这一表型深入研究后揭示了该基因调节水稻耐旱的机理。
水稻受到干旱胁迫时,水稻体内对干旱响应是一个复杂的调控反应,各种调节机制不是单独作用,而是多种途径相互作用形成复杂的网络调控体系,进而提高水稻抗旱能力。
未知功能结构域蛋白(domainsofunknownfunctionproteinfamilies,dufs)一直是科研工作者研究的热点。研究发现,dufs家族基因具有的生物学功能是各不相同的。植物中dufs能够调控植物生长发育、参与非生物胁迫反应以及参与病虫害的防御反应等生理生化过程。水稻dufs家族仅有少数基因的功能被揭示,大部分家族基因尚未被研究。
因此,提供osduf6基因在提高水稻耐旱性中的应用是本领域技术人员亟需解决的问题。
技术实现要素:
有鉴于此,本发明提供了osduf6基因在提高水稻耐旱性中的应用。
为了实现上述目的,本发明采用如下技术方案:
一种水稻osduf6基因,其核苷酸序列如seqidno.3所示。
所述水稻osduf6基因编码的多肽,其氨基酸序列如seqidno.4所示。
所述的水稻osduf6基因在提高水稻耐旱性中的应用。
所述的水稻osduf6基因在干旱胁迫条件下,在提高抗氧化酶活性和降低mda含量中的应用。
所述抗氧化酶为sod、cat、pod。
经由上述的技术方案可知,与现有技术相比,本发明公开提供了osduf6基因在提高水稻耐旱性中的应用,本发明克隆获得了一个水稻未知功能结构域基因osduf6,通过遗传转化获得该基因的过表达植株;对osduf6过表达植株进行干旱处理,发现与野生型植株相比,其存活率上升、抗氧化酶的活性升高、膜脂过氧化程度减弱、aba含量变化升高、活性氧(ros)的含量下降,表明过表达osduf6可显著提高水稻对干旱的耐受性。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据提供的附图获得其他的附图。
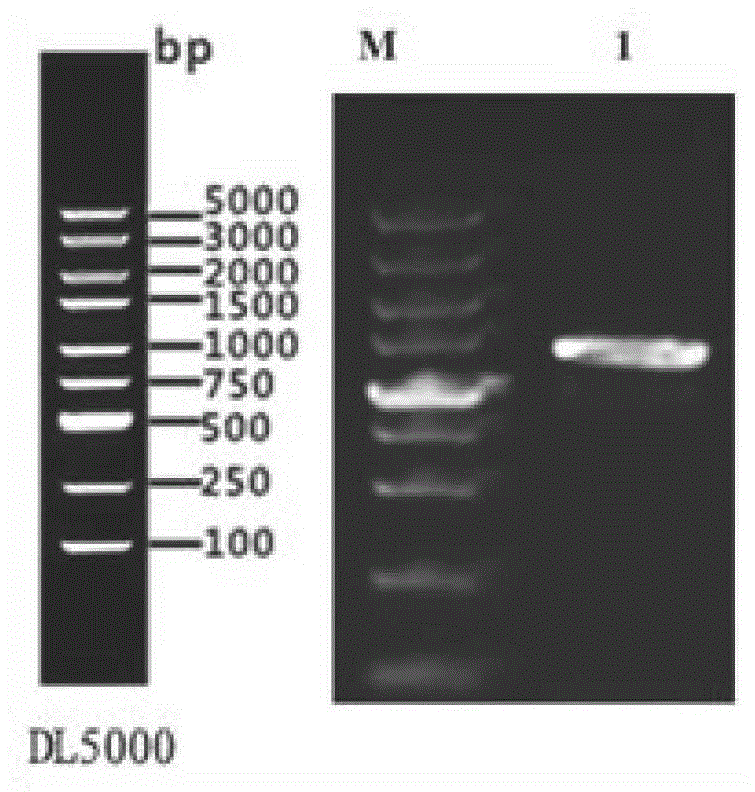
图1附图为本发明osduf6cds扩增产物的琼脂糖凝胶电泳图;
其中,m为dl5000marker;1为osduf6cds扩增产物;
图2附图为本发明osduf6cds序列与日本晴同源基因序列比对结果;
图3附图为本发明osduf6氨基酸序列比对结果;
图4附图为本发明pcubi1390载体图谱;
图5附图为本发明osduf6基因过表达载体构建策略示意图;
图6附图为本发明农杆菌介导水稻遗传转化的流程图;
其中,a:水稻愈伤诱导;b:水稻愈伤继代;c:侵染后愈伤筛选;d:筛选后愈伤预分化;e:筛选后愈伤分化成苗;f:转化幼苗生根;
图7附图为本发明部分转基因苗pcr检测结果;
其中,a:阴性对照;b:阳性对照;1-10为10株转基因苗;
图8附图为本发明干旱处理前盆中土壤含水量的情况;
图9附图为本发明转基因水稻植株在干旱胁迫下的表型;
图10附图为本发明复水后转基因植株的存活率;
其中,**表示t检验中与野生型相比p<0.01的显著差异;
图11附图为本发明干旱胁迫下osduf6过表达植株抗氧化酶pod活性的测定;
图12附图为本发明干旱胁迫下osduf6过表达植株抗氧化酶cat活性的测定;其中,**表示t检验中与野生型相比p<0.01的显著差异;
图13附图为本发明干旱胁迫下osduf6过表达植株抗氧化酶sod活性的测定;
图14附图为本发明干旱胁迫下osduf6过表达植株膜脂过氧化程度mda的测定;其中,**表示t检验中与野生型相比p<0.01的显著差异。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1osduf6基因的克隆分析
以籼稻突变体材料75-1-127和籼稻品种93-11为亲本,进行正反交,获得f1植株继续进行自交得到f2群体。利用f2群体为实验材料,以日本晴数据库为参考序列对插入缺失标记indel-l06和indel-115的目标定位区间进行候选基因的分析,结合预测网站gramene、rgap和softberry的fgenesh等数据库预测获得候选基因loc_os06g44810(http://www.ricedata.cn/gene/)。
根据日本晴参考基因组,对候选基因进行序列上的比对分析,设计相应的特异性引物,具体引物序列如下:
810f:5’-atggagtcacccgccgcgggcg-3’;seqidno.1;
810r:5’-tcattttgccttccttggcctc-3’;seqidno.2。
利用高保真酶
扩增后利用琼脂糖凝胶电泳将pcr产物进行检测,待确定目的条带正确后,将扩增产物通过胶回收试剂盒进行产物回收,回收步骤参照说明书进行。最后利用回收后的产物与平末端克隆试剂盒(南京诺唯赞生物科技有限公司)的克隆载体进行重组,重组反应体系如下:ptopo-bluntsimplevector0.5μl,10×enhancer0.5μl,纯化后的pcr产物xμl(100ng),ddh2o补至5μl。
混匀,低速离心,室温(20-30℃)连接5min。重组后将连接产物转化入trans1-t1(dh5α)感受态细胞中进行转化,转化程序如下:
1)将上述转化产物进行30min冰浴处理,随后42℃热激60s;
2)冰浴2min后加入500μllb液体培养基,37℃,180rpm,振荡培养60min;
3)2000rpm,离心1min,弃掉上清,留下约100μllb液体重悬浮菌体;
4)在含有氨苄抗性的固体培养基上涂布转化菌液,置于37℃恒温箱培养12h;
5)挑选5~10个单克隆菌落置于700μl含有氨苄抗性的lb液体培养基;
6)37℃,220rpm振荡培养4~5h,随后进行菌液pcr鉴定,菌液样品可4℃冰箱中短暂保存,亦可直接将菌液送样进行测序分析。
将osduf6cds序列测序结果(seqidno.3)与日本晴同源基因序列比对,结果见图2,图2结果显示,75-1-127中该基因cds序列存在7个碱基的突变,突变位点分别为:第132位碱基由t突变为c,第254位碱基由c突变为t,第345位碱基由g突变为a,第703位碱基由a突变为g,第935位碱基由g突变为a,第997位碱基由t突变为a,第1097位碱基由a突变为c。osduf6氨基酸序列(seqidno.4)与日本晴氨基酸序列比对结果见图3,图3结果显示,氨基酸序列存在5个氨基酸残基发生改变,分别是第85位氨基酸由p替换为l,第235位氨基酸由t替换为a,第312位氨基酸由g替换为d,第333位氨基酸由f替换为i,第366位氨基酸由y替换为s。(图2的cds序列比对结果以及图3的氨基酸序列比对结果,均只截取了存在突变的位点,并没有截取全部结果。)
atggagtcacccgccgcgggcggcgcgtctccggcgccggaggctcctccgcccccgccgagggggtggatctccggcctcgtctccggagccggccggatcctcgcctccgtgctaggtcccgactcgcccgccgccgcctccggctccgccacgacgacctcagccacgtcgccgtccgcgtcctcctccccggcgtcgtccaggcatcccggttatattcgagatcatggtaattccccactattttttctgaaagctaataagttaaacaagagtgaaaatgaagcaattatgaaagattattctgaggcatcactagcaattatctcagagattgagccaaaggatgccataatgcagttacttaagcaggagacttattcaaggtctgaatgcaatgcactagtaaagataattcaagagcgggttgtggactcaaatttaaatggagttgatgctggtgggcttgctcttccaatcaattggaagactggcagacaagcaaatattggatattcttcattgagtccaaaaggattgttgcctgcaacttcaattcctccagtccaggaccatgtttttgataatagtgctggtgccggtgcatctacaacaatagctcatgataggggtcctttcgcccacgctactgataagattcaatccgtactcaaaagaagttgctctgttgcaacggatgcccctgaccctgaggactcccgaagagtcaggccaaaaattaatggaaattcattagaaatttcaaattttaagcaagtcgatgttattcgaactcattcaggagatgacaataaattatcagatgttcccctttttgggaccaacaatttaatatattctaatattgtttcaattgttgggagtgccgatgaaaaaataggcatacctaacaaaccttcagctggtgatgacaataaaaactatgattctgaatttttaaatccatgtaccaataaggatctgaagaacagcattccattgaaggtggaacctttggatgtttgtattcctttcgagcagcagatgatggatctctcacaccagaagcatgagcatgctgcatgtgatgattcttgttctgtctcaaaattaatgtttaaggaggatattgagactgcactcagcatgccagtgggcgtgccgctagaaaatggttctaagaaccggagacgaagggcacccaacacgcagaggattaccccggcaaggtctcctgccaagggttcacgtcgcaagagtaatgatgtcacagtaaaatcagagaccgacttgctagagcaaagcaagggttcacatgatgtcacagtaaagtcagagatcgacttgctagagcaaagtaaattggcactgatggagcagtcaccagatttaggcgatattccggtgaagagaccagttgggaggccaaggaaggcaaaatga;seqidno.3。
mespaaggaspapeappppprgwisglvsgagrilasvlgpdspaaasgsatttsatspsassspassrhpgyirdhgnsplfflkanklnkseneaimkdyseaslaiiseiepkdaimqllkqetysrsecnalvkiiqervvdsnlngvdagglalpinwktgrqanigysslspkgllpatsippvqdhvfdnsagagasttiahdrgpfahatdkiqsvlkrscsvatdapdpedsrrvrpkingnsleisnfkqvdvirthsgddnklsdvplfgtnnliysnivsivgsadekigipnkpsagddnknydseflnpctnkdlknsiplkvepldvcipfeqqmmdlshqkhehaacddscsvsklmfkedietalsmpvgvplengsknrrrrapntqritparspakgsrrksndvtvksetdlleqskgshdvtvkseidlleqsklalmeqspdlgdipvkrpvgrprkak*;seqidno.4。
实施例2osduf6基因过表达载体的构建
将ubiquitin驱动的pcubi1390(载体图谱见图4)作为过表达载体,用psti进行酶切,回收,获得线性化克隆载体片段;选择酶切位点(psti),设计相应引物(ubi:810过f、ubi:810过r)对osduf6基因进行扩增、酶切、回收,参照clon
具体引物序列如下:
ubi:810过f:5’-ttctgcactaggtacctgcagatggagtcacccgccgcg-3’;psti;seqidno.5;
ubi:810过r:5’-cggggatccgtcgacctgcagtcattttgccttccttggcc-3’;psti;seqidno.6。
实施例3农杆菌转化(冻融法)
将构建好的过表达载体质粒转化到农杆菌感受态细胞eha105中,以便后续遗传转化组织侵染实验,具体步骤如下:
(1)农杆菌感受态eha105的制备
1)将-80℃保存的甘油菌eha105置于冰上解冻,随后在超净台上在含利福平(rif)抗性(50μg/ml)的固体培养基中划线,置于28℃暗培养2-3d;
2)挑取单克隆菌落加入到700μl含rif抗性(50μg/ml)的lb液体培养基中振荡培养,150rpm,28℃暗培养,待菌体培养一定浓度后,置于50ml三角瓶中继续扩大培养;
3)待培养菌体od值约0.4-0.6范围内时,将菌液冰浴处理30min,4℃4,000rpm离心10min,去除上清液;
4)加入10ml预冷的nacl溶液(0.15mol/l),轻轻混匀重悬菌体,4℃,4,000rpm离心10min,去上清;
5)加入1ml预冷的cacl2溶液(0.02mol/l),轻轻吹打悬浮细胞;
6)加入1ml预冷的30%浓度甘油,轻轻混均,并进行分装处理(每管50μl),随后进行液氮速冻并置于-80℃超低温冰箱保存备用。
(2)重组载体质粒的转化
1)冰上冻融制备好的eha105感受态细胞,加入2μg重组载体质粒,轻柔混匀后再冰浴静置30min;
2)液氮速冻3min,立即放入37℃水浴锅水浴3min,再冰浴2min;
3)加入500μl的lb液体培养基,150rpm,28℃复苏培养3-5h;
4)将上述样品涂布于含50μg/ml的rif和50μg/mlkan抗性的lb固体培养基上,28℃进行暗培养处理2-3d;
5)挑取单克隆菌落加入到含kan+rif抗性(均为50μg/ml)的700μllb液体培养基中,220rpm,37℃振荡培养约4h左右,再经菌液pcr鉴定将阳性克隆扩大培养,加入一定量甘油(15%-25%甘油浓度)与菌株混合并冻存于-80℃冰箱备用。
实施例4农杆菌介导水稻遗传转化及转基因苗的检测
(一)水稻愈伤组织的诱导与继代培养
(1)材料的挑选
水稻愈伤组织的诱导可以挑选幼胚或者当季新鲜种子进行诱导,幼胚挑选谷壳嫩绿谷粒偏软带汁为佳,成熟胚则挑选饱满,完整性好的种子。
(2)消毒
先无菌水清洗3-5次,去除表面杂质,用75%乙醇溶液进行表面消毒处理2min,随后直接用含有2.5%的次氯酸钠溶液浸泡10min(为了更好的消毒效果,可添加一滴吐温20),期间置于摇床不停摇晃以达更好的效果,再经过无菌水清洗数次后将其置于暗处30min,随后将消毒清洗后的谷粒铺于灭菌的滤纸上超净工作台吹干(约10min)。
(3)愈伤组织的诱导与继代培养
消毒处理后的幼胚用解剖针取下胚接到诱导培养基上,成熟胚可直接接种到诱导培养基上,并放于28℃暗培养。7-10d后将诱导长出的新愈伤组织去芽后转接到继代培养基上,根据愈伤组织生长情况,约2w继代一次。
(二)农杆菌转化水稻愈伤组织
(1)菌体活化
吸取适量经鉴定为阳性的菌液直接在含kan(50mg/l)和rif(50mg/l)抗性的lb固体培养基上划线,倒置于28℃生化培养箱中进行1-2d的暗培养。
(2)愈伤组织的侵染
1)挑选直径约2-3mm,色泽鲜黄且结构致密的胚性愈伤组织于预培养培养基中28℃培养2-3d;
2)取适量含乙酰丁香酮(as)(100μmol/l)的aam液体培养基吹洗已活化的菌体,调整od浓度,使od600在0.2-0.3的范围内,激烈振荡1min,静置1h,使菌体形成悬浮液同时增强其侵染活性;
3)挑取预培养的胚性愈伤组织,倒入悬浮液,略微摇动后静置10min,于无菌滤纸上晾干愈伤组织后置于含有100μmol/l乙酰丁香酮的共培养培养基上,28℃暗培养2d。
(3)抗性愈伤组织的继代筛选
共培养2d后的愈伤组织转接到不同羧苄青霉素浓度的筛选培养基上进行筛选培养,新长出的愈伤组织继代2-3次,每次继代培养10-12d。
(4)分化与生根
挑选经筛选继代处理后长出的新愈伤组织转接至预分化培养基中进行再生,成团,一周后愈伤组织出现绿点,再将其转接到分化培养基中,约2-3w后长出幼芽。当长出的植株到一定高度后转到生根培养基中生根处理至成苗。
(5)炼苗
生根10d后开盖,加入无菌水浸泡培养基即可,2d后洗苗转移至营养液中进行生长,在此期间可进行转基因植株的阳性鉴定。其中,农杆菌介导水稻遗传转化的流程见图6。
水稻遗传转化组培试剂配制培养基配方如下:
1)诱导(继代、预培养)培养基(j0):msbasalmedium+2,4-d(3mg/l)+谷氨酰胺(500mg/l)+脯氨酸(500mg/l)+3%蔗糖+0.25%gelrite(植物凝胶),ph=5.8;
2)共培养培养基:dl3+2,4-d(2mg/l)+乙酰丁香酮(as,100μm),ph=5.8;
3)筛选培养基ⅰ:dl3+2,4-d(2mg/l)+羧苄青霉素400mg/l+潮霉素50mg/l,ph=5.8;
4)筛选培养基ⅱ:dl3+2,4-d(2mg/l)+羧苄青霉素200mg/l+潮霉素50mg/l,ph=5.8;
5)筛选培养基ⅲ:dl3+2,4-d(2mg/l)+羧苄青霉素100mg/l+潮霉素50mg/l,ph=5.8;
6)预分化培养基:dl3+羧卞(250mg/l)+hyg(50mg/l),ph=5.9;
7)分化培养基:dl+ba(2mg/l)+naa(0.2mg/l)+kt(2mg/l)+iaa(0.2mg/l)+谷氨酰胺(500mg/l)+脯氨酸(500mg/l)+水解酪蛋白(800mg/l),ph=5.9;
8)生根培养基:1/2ms无机盐+ms有机成分+30g蔗糖,ph=5.8。
(三)转基因植株阳性检测
提取所有待检测转基因植株叶片的dna,使用潮霉素引物进行pcr反应进行检测,检测引物如下:
hyg458-f:5’-cgagagcctgacctattg-3’;seqidno.7;
hyg458-r:5’-gcctccagaagaagatgt-3’;seqidno.8。
pcr反应体系如下:2×mixture5μl,hyg458-f0.5μl,hyg458-r0.5μl,基因组dna1μl(≈100ng),ddh2o补至10μl。
pcr扩增程序如下:95℃预变性5min;95℃变性15s,62℃退火15s,72℃延伸1min,共35个循环;72℃延伸7min;4℃保温。
将扩增后的pcr产物进行琼脂糖凝胶电泳,部分转基因苗pcr检测结果见图7。
实施例5osduf6转基因植株在干旱胁迫下的表型分析
(1)土壤含水量的测定
在osduf6过表达植株(生长一致、三叶一心期)中选取2个osduf6基因表达量较高的水稻株系(分别命名为oe-1,oe-2)进行干旱胁迫表型分析。
在干旱处理前,分别从种植osduf6过表达植株和wt植株的盆子中称取一定量的新鲜土壤,称重后记录数据。待土壤烘干后,称重并记录数据。之后按以下公式计算:土壤含水量(%)=(烘干前的重量-烘干后的重量)/烘干前的重量。以上的测定设置三组重复防止误差。结果见图8,在干旱处理前wt、oe-1、oe-2的土壤含水量分别为31.3%、32.1%、31.7%,两个osduf6过表达株系的土壤含水量与wt的土壤含水量无显著性差异。
(2)osduf6转基因植株干旱胁迫处理
选取适量的osduf6过表达种子以及野生型(wt)种子于浸种袋,用水清洗3~5次以去除种子表皮的一些杂质及霉菌等,接着置于37℃催芽1~2d,然后将种子均匀的铺于含滤纸的培养皿中,放于28℃光照培养室中培养至一叶一心期后放置到营养液中继续培养。待长到三叶一心期时,挑选长势一致的转基因过表达植株和野生型植株移植于室外进行土培,每一盆种20株水稻。在此期间,保证生长环境一致,培养6周后不再浇水,进行干旱处理,随时观察植株生长情况并拍照。待出现明显的表型后正常进行浇水,随时观察植株在复水之后的生长情况并拍照,结果见图9。接着统计植株的存活率,结果见图10。以上的处理设置三组重复防止误差,另外再设置一组重复用于处理阶段的取样。
图9结果显示,在干旱处理前osduf6过表达株系和wt的长势是一致的,干旱处理16d后,所有wt植株的叶片均出现卷曲、植株发生萎焉的症状,而两个osduf6过表达株系的叶片仅出现轻微的卷曲,只有少量植株出现萎焉的症状;复水5天后,转基因过表达株系从干旱胁迫损伤中恢复的效果更好,而wt植株的恢复效果比较差,仍然有萎焉的植株。此外复水5d后,oe-1和oe-2的存活率(图10)分别为75.8%、72.4%,而wt仅为34.06%,说明osduf6过表达株系的存活率要显著高于wt株系,osduf6过表达株系更耐旱。
实施例6干旱胁迫下osduf6过表达植株抗氧化能力及膜受损程度分析
为检测osduf6过表达植株与wt植株在干旱胁迫下的抗氧化能力以及膜系统受损的情况,分别取处理前(0d)以及处理后(16d)的osduf6过表达植株和wt植株的样品进行sod、cat、pod活性以及mda含量检测。
(1)过氧化物酶(pod)活性检测
分别取osduf6过表达植株(oe-1)和野生型植株干旱处理前后的叶片,测定二者干旱处理前后pod的活性变化。具体操作步骤参考pod活性检测试剂盒(索莱宝)内的说明书。检测结果见图11。
图11结果显示,干旱处理前osduf6过表达植株(oe-1)pod的活性与wt相比差异不显著;而干旱处理16d后,osduf6过表达植株pod的活性相对于wt有明显的升高,两者达到显著性差异。
(2)过氧化氢酶(cat)活性检测
分别取osduf6过表达植株(oe-1)和野生型植株干旱处理前后的叶片,测定二者干旱处理前后cat的活性变化。具体操作步骤参考cat活性检测试剂盒(索莱宝)内的说明书。检测结果见图12。
图12结果显示,干旱处理前osduf6过表达植株(oe-1)cat的活性与wt相比差异不显著;而干旱处理16d后,osduf6过表达植株cat的活性相对于wt有明显的升高,两者达到显著性差异。
(3)超氧化物歧化酶(sod)活性检测
分别取osduf6过表达植株(oe-1)和野生型植株干旱处理前后的叶片,测定二者干旱处理前后sod的活性变化。具体操作步骤参考sod活性检测试剂盒(索莱宝)内的说明书。检测结果见图13。
图13结果显示,两者sod的活性在干旱处理前后都无显著性的差异。
上述指标都是与活性氧清除能力相关的,这些指标的活性越强,清除活性氧的能力就越强,植物遭受的损害就越低。
(4)丙二醛(mda)含量检测
分别取osduf6过表达植株(oe-1)和野生型植株干旱处理前后的叶片,测定二者干旱处理前后md的含量变化。具体操作步骤参考mda含量检测试剂盒(索莱宝)内的说明书。检测结果见图14。
图14结果显示,在干旱处理前,osduf6过表达植株(oe-1)的mda含量与wt之间没有明显的差异,干旱处理后,二者的mda含量都升高,但osduf6过表达植株的mda含量相对于wt有明显的降低,该指标是与膜损伤有关,mda的值越大说明细胞膜所遭受的损伤就越大。
上述结果表明,osduf6过表达植株在干旱条件下,受到的损伤要低于wt,说明osduf6过表达植株通过提高抗氧化酶的活性和降低mda的含量来提高水稻耐旱性。
对所公开的实施例的上述说明,使本领域专业技术人员能够实现或使用本发明。对这些实施例的多种修改对本领域的专业技术人员来说将是显而易见的,本文中所定义的一般原理可以在不脱离本发明的精神或范围的情况下,在其它实施例中实现。因此,本发明将不会被限制于本文所示的这些实施例,而是要符合与本文所公开的原理和新颖特点相一致的最宽的范围。
序列表
<110>福建省农业科学院水稻研究所
<120>osduf6基因在提高水稻耐旱性中的应用
<160>8
<170>siposequencelisting1.0
<210>1
<211>22
<212>dna
<213>artificialsequence
<400>1
atggagtcacccgccgcgggcg22
<210>2
<211>22
<212>dna
<213>artificialsequence
<400>2
tcattttgccttccttggcctc22
<210>3
<211>1446
<212>dna
<213>artificialsequence
<400>3
atggagtcacccgccgcgggcggcgcgtctccggcgccggaggctcctccgcccccgccg60
agggggtggatctccggcctcgtctccggagccggccggatcctcgcctccgtgctaggt120
cccgactcgcccgccgccgcctccggctccgccacgacgacctcagccacgtcgccgtcc180
gcgtcctcctccccggcgtcgtccaggcatcccggttatattcgagatcatggtaattcc240
ccactattttttctgaaagctaataagttaaacaagagtgaaaatgaagcaattatgaaa300
gattattctgaggcatcactagcaattatctcagagattgagccaaaggatgccataatg360
cagttacttaagcaggagacttattcaaggtctgaatgcaatgcactagtaaagataatt420
caagagcgggttgtggactcaaatttaaatggagttgatgctggtgggcttgctcttcca480
atcaattggaagactggcagacaagcaaatattggatattcttcattgagtccaaaagga540
ttgttgcctgcaacttcaattcctccagtccaggaccatgtttttgataatagtgctggt600
gccggtgcatctacaacaatagctcatgataggggtcctttcgcccacgctactgataag660
attcaatccgtactcaaaagaagttgctctgttgcaacggatgcccctgaccctgaggac720
tcccgaagagtcaggccaaaaattaatggaaattcattagaaatttcaaattttaagcaa780
gtcgatgttattcgaactcattcaggagatgacaataaattatcagatgttccccttttt840
gggaccaacaatttaatatattctaatattgtttcaattgttgggagtgccgatgaaaaa900
ataggcatacctaacaaaccttcagctggtgatgacaataaaaactatgattctgaattt960
ttaaatccatgtaccaataaggatctgaagaacagcattccattgaaggtggaacctttg1020
gatgtttgtattcctttcgagcagcagatgatggatctctcacaccagaagcatgagcat1080
gctgcatgtgatgattcttgttctgtctcaaaattaatgtttaaggaggatattgagact1140
gcactcagcatgccagtgggcgtgccgctagaaaatggttctaagaaccggagacgaagg1200
gcacccaacacgcagaggattaccccggcaaggtctcctgccaagggttcacgtcgcaag1260
agtaatgatgtcacagtaaaatcagagaccgacttgctagagcaaagcaagggttcacat1320
gatgtcacagtaaagtcagagatcgacttgctagagcaaagtaaattggcactgatggag1380
cagtcaccagatttaggcgatattccggtgaagagaccagttgggaggccaaggaaggca1440
aaatga1446
<210>4
<211>481
<212>prt
<213>artificialsequence
<400>4
metgluserproalaalaglyglyalaserproalaproglualapro
151015
proproproproargglytrpileserglyleuvalserglyalagly
202530
argileleualaservalleuglyproaspserproalaalaalaser
354045
glyseralathrthrthrseralathrserproseralaserserser
505560
proalaserserarghisproglytyrileargasphisglyasnser
65707580
proleuphepheleulysalaasnlysleuasnlyssergluasnglu
859095
alailemetlysasptyrserglualaserleualaileileserglu
100105110
ilegluprolysaspalailemetglnleuleulysglngluthrtyr
115120125
serargserglucysasnalaleuvallysileileglngluargval
130135140
valaspserasnleuasnglyvalaspalaglyglyleualaleupro
145150155160
ileasntrplysthrglyargglnalaasnileglytyrserserleu
165170175
serprolysglyleuleuproalathrserileproprovalglnasp
180185190
hisvalpheaspasnseralaglyalaglyalaserthrthrileala
195200205
hisaspargglyprophealahisalathrasplysileglnserval
210215220
leulysargsercysservalalathraspalaproaspprogluasp
225230235240
serargargvalargprolysileasnglyasnserleugluileser
245250255
asnphelysglnvalaspvalileargthrhisserglyaspaspasn
260265270
lysleuseraspvalproleupheglythrasnasnleuiletyrser
275280285
asnilevalserilevalglyseralaaspglulysileglyilepro
290295300
asnlysproseralaglyaspaspasnlysasntyraspsergluphe
305310315320
leuasnprocysthrasnlysaspleulysasnserileproleulys
325330335
valgluproleuaspvalcysileprophegluglnglnmetmetasp
340345350
leuserhisglnlyshisgluhisalaalacysaspaspsercysser
355360365
valserlysleumetphelysgluaspilegluthralaleusermet
370375380
provalglyvalproleugluasnglyserlysasnargargargarg
385390395400
alaproasnthrglnargilethrproalaargserproalalysgly
405410415
serargarglysserasnaspvalthrvallyssergluthraspleu
420425430
leugluglnserlysglyserhisaspvalthrvallyssergluile
435440445
aspleuleugluglnserlysleualaleumetgluglnserproasp
450455460
leuglyaspileprovallysargprovalglyargproarglysala
465470475480
lys
<210>5
<211>39
<212>dna
<213>artificialsequence
<400>5
ttctgcactaggtacctgcagatggagtcacccgccgcg39
<210>6
<211>41
<212>dna
<213>artificialsequence
<400>6
cggggatccgtcgacctgcagtcattttgccttccttggcc41
<210>7
<211>18
<212>dna
<213>artificialsequence
<400>7
cgagagcctgacctattg18
<210>8
<211>18
<212>dna
<213>artificialsequence
<400>8
gcctccagaagaagatgt18
- 还没有人留言评论。精彩留言会获得点赞!