适用于地衣芽胞杆菌的启动子及其在高效表达目的产物中的应用的制作方法
本发明涉及基因工程和微生物代谢工程领域,具体涉及适用于地衣芽胞杆菌的启动子及其在高效表达目的产物中的应用。
背景技术:
启动子是位于基因转录单元上游的一段特殊dna序列,具有启动基因转录的功能,并在调控基因转录水平中发挥着重要的作用。随着合成生物学和代谢途径工程的发展,人们逐渐发现,为了实现目标产物的高产,强化目的蛋白的表达水平和目的代谢物的代谢通量是至关重要的。由于启动子是基因转录的开关,并被认定为是建立基因高效表达工具的主要表达元件,从而启动子改造是开发能优化蛋白表达水平和代谢物代谢途径技术的首选策略。目前,启动子改造策略主要围绕着σ因子识别和结合的启动子核心区(-35区、核心间隔区、-10区)做改良工作。如周等人在枯草芽胞杆菌中,通过随机突变策略改造启动子psrfa的核心间隔区,获得的最佳突变启动子pbh4较原始启动子psrfa,使gfp的表达水平提高了2倍(doi:10.1186/s12934-019-1148-3),又如严等人在枯草芽胞杆菌中,通过对启动子pylb的-35区和-10区进行突变,获得的最佳突变启动子pnbp3510较原始启动子pylb,使β-半乳糖苷酶(bgab)活性提高近25倍(doi:10.1186/s12934-019-1159-0),以及王等人对t7启动子核心区(-35区和-10区)进行组合,得到的最佳启动子p21285提高了l-天冬酰胺酶和酸性脲酶在大肠杆菌中的产量(doi:10.1002/biot.201800298)。
地衣芽胞杆菌是一个重要的工业生产模式菌株,具有分泌蛋白能力强特征,且被美国fda认定为生物安全性菌株,从而其可作为蛋白和目标代谢物生产的优良宿主。目前,其已经实现多种蛋白和代谢物高效生产,如α-淀粉酶、碱性蛋白酶,杆菌肽等。然而,地衣芽胞杆菌在表达多数外源蛋白时,存在表达难或表达效率低的问题,而启动子被认定为是建立基因高效表达工具的主要表达元件,且启动子改造是开发能优化蛋白表达水平和代谢物代谢途径技术的首选策略。在地衣芽胞杆菌中,启动子主要包括σa型和σb型启动子,其中σa型启动子主要介导与细胞生长和代谢相关基因的表达,而σb型启动子主要介导细胞响应内外各种压力(温度、渗透压、ph等)相关基因的表达。
技术实现要素:
本发明的目的是提供了适用于地衣芽胞杆菌的启动子,所述的启动子序列为seqidno.2-seqidno.5中的任意一个。
适用于地衣芽胞杆菌的启动子在高效表达目的蛋白中的应用,将本发明的启动子导入地衣芽胞杆菌中,可提高地衣芽胞杆菌生产目的蛋白的能力。
为了达到上述目的,本发明采取以下技术措施:
适用于地衣芽胞杆菌的启动子,所述的启动子的核苷酸序列为seqidno.2所示;或seqidno.3所示;或seqidno.4所示;或seqidno.5所示。
适用于地衣芽胞杆菌的启动子在提高地衣芽胞杆菌目的蛋白表达量中的应用,将本发明提供的启动子,导入地衣芽胞杆菌中,可提高蛋白表达和代谢产物的产量;将所述的启动子启动外源蛋白的表达可提高蛋白表达量,将所述的启动子启动增强代谢产物产量的基因表达,可提高代谢产物的产量。
以上所述的应用中,优选的,所述外源蛋白为绿色荧光蛋白和角蛋白酶;
以上所述的应用中,优选的,所述代谢产物为γ-pga和杆菌肽;
以上所述的应用中,优选的,所述的地衣芽胞杆菌为地衣芽胞杆菌dw2和地衣芽胞杆菌wx-02。
与现有技术相比,本发明具有以下优点:
1.本发明人首次尝试通过核心区四组合镶嵌模式改造启动子的常规核心区(-35区(6bp)、-10区(6bp)、核心间隔区(16-18bp)),使其核心区完全由σ因子识别的四个位点组成(a-35-b-35-c-35-d-35-a-10-b-10-c-10-d-10,a、b、c和d序列长度为5-6bp),进而,在不同σ因子识别作用下,改善该启动子在发酵对数期和稳定期的活性,从而使其介导表达的基因在整个发酵周期都能有效表达。
2.本发明人首次表明启动子核心区四组合镶嵌模式“σb-σb-σa-σb”可作为启动子优化的有效策略,且该核心区四组合镶嵌模式的序列长度为43-46bp,导致其构建非常方便,以非常低廉的费用直接通过引物设计导入表达载体中。
3.本发明人先获得一组基于启动子核心区四组合的启动子(r1-r4)。接着,与启动子p43相比,研究发现启动子r2效果最佳,显著提高了gfp、ker的产量。最后,将启动子r2应用于代谢工程中,与对照菌相比,通过将其应用于代谢途径强化使聚γ-谷氨酸,杆菌肽的产率分别提高了52.09%,20.39%,从而说明改造得到的启动子r2不仅性能良好,而且具有一定的普适性。
附图说明
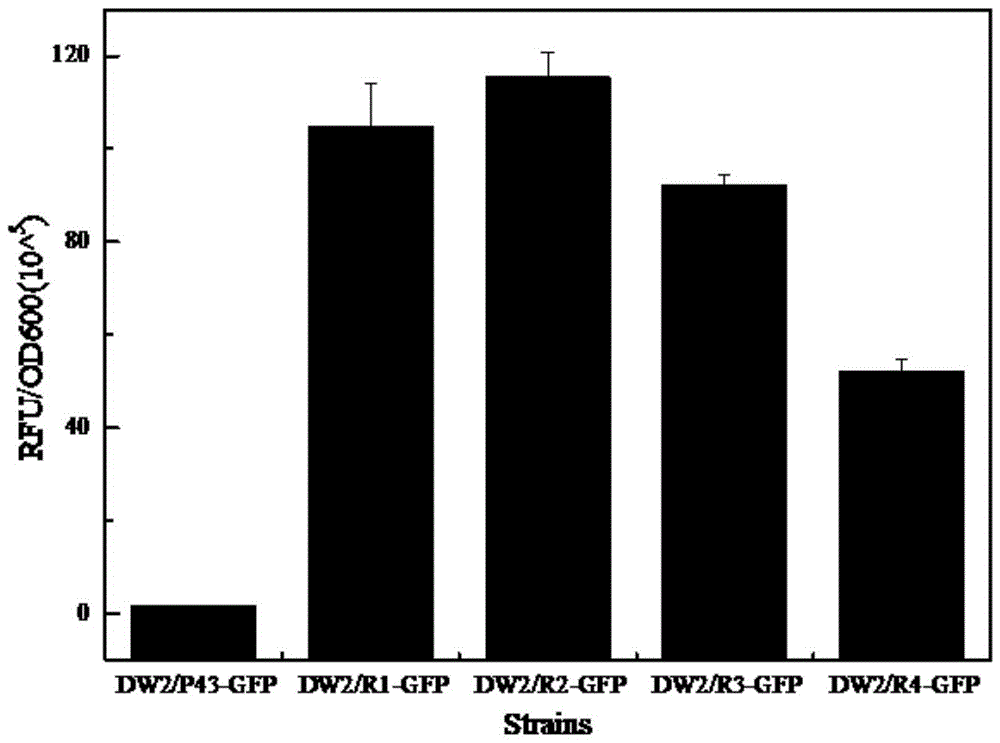
图1为r1-r4启动子对gfp表达的影响示意图。
图2为r2启动子对角蛋白酶表达的影响示意图。
图3为r2启动子对wx-02产γ-pga的影响示意图。
图4为r2启动子对dw2产杆菌肽的影响示意图。
具体实施方式
本发明所述技术方案,如未特别说明,均为本领域的常规方案;所述试剂或材料,如未特别说明,均来源于商业渠道。
本发明以2种外源蛋白(绿色荧光蛋白gfp、角蛋白酶)和两种代谢产物(γ-pga、杆菌肽)为例,说明本发明技术方案的优越性。
实施例1:
适用于地衣芽胞杆菌的启动子的获得:
本发明的启动子,是通过在σa和σb型因子识别的五个结合位点中,在保证启动子核心区完全由σa和σb型因子识别位点组成的前提下,有选择的选取四个位点嵌合而成,所述的五个结合位点的核苷酸序列分别为:
ttgaca…tataat;
gccttga…ccccat;
ttctca…taaaat;
aagcca…tatatt;
gtaaaa…tattat;
根据上述5个结合位点,选取了四种嵌合方式后,人工设计并合成了四个优化启动子,命名为r1-r4,其对应的核苷酸序列分别为seqidno.2-seqidno.5所示。
实施例2:
优化启动子r1-r4在提高地衣芽胞杆菌外源蛋白表达量中的应用:
在本实施例中,通过引物直接引入了r1-r4启动子,替换了表达载体中的p43启动子核心区,具体步骤如下:
1、以本实验早期构建的质粒phy-p43-gfp(doi:10.1016/j.jbiotec.2020.02.015)作为模板,通过表1的引物,pcr扩增出包含优化启动子的载体骨架,并进行dna回收;
表1:含有不同优化启动子的表达载体引物设计
注:划线为同源臂区,18-19bp。
2、利用dpni酶对残留的phy-p43-gfp模板进行消化;
3、采用vazyme的同源重组酶(exnaseii)对载体骨架进行处理,并在37℃条件下反应30min,使载体骨架自连;随后进行钙转,将其转入大肠杆菌中;最后通过测序验证后,提取质粒,即得到含有优化启动子的重组质粒,分别为phy-r1-gfp、phy-r2-gfp、phy-r3-gfp和phy-r4-gfp;
4、将步骤3得到的重组质粒分别转入地衣芽胞杆菌dw2中,以四环抗生素作为筛选标记,随后,通过菌落pcr筛选得到阳性转化子,从而获得四种的工程菌dw2/r1-gfp、dw2/r2-gfp、dw2/r3-gfp、dw2/r4-gfp。
对照菌株dw2/p43-gfp的制备:直接将phy-p43-gfp转入地衣芽胞杆菌dw2中获得。
5、工程菌种子活化:从甘油管以体积百分比1%将步骤4得到的工程菌分别接种于装有5mllb培养基中,230r/min、温度37℃,培养12小时,然后将菌种活化后的菌液以体积百分比按1%接种量接种于种子发酵培养基于230r/min、37℃中培养10小时,得到种子培养的菌液;
所述的发酵培养基即为lb培养基。
6、工程菌发酵:向500ml三角瓶中装入50mllb液体发酵培养基,然后将种子培养的菌液以接种量为2%(体积百分比),转速230r/min,温度37℃,发酵培养48小时,得到生产发酵的菌液。
7、gfp表达水平检测:取0.2ml发酵液于2mlep管,加入1.8mlpbs缓冲液,用可见光分光光度计测其生物量(od600)。然后用pbs缓冲液将发酵液相应稀释到终浓度(od600)为1,终体积2ml,随后7000rpm,2min,接着用2mlpbs缓冲液重悬,重复一次,最后用酶标仪检测其荧光强度,该结果见图1。由图1可知:对照菌株dw2/p43-gfp表达的gfp荧光强度为1.79×105,而含有r1、r2、r3和r4介导表达的gfp荧光强度依次为104.74×105,115.55×105,92.27×105,52.13×105,其中,r2效果最佳,使gfp产量提高63.5倍。由于r2的提升效果最好,因此本发明以下实施例以r2为例,对其在提升地衣芽胞杆菌外源蛋白或代谢产物表达量的效果进行说明,其余优化启动子也有相同提升功能。
实施例3:
r2启动子提高地衣芽胞杆菌对角蛋白酶的表达亮:
1、以phy-r2-gfp和地衣芽胞杆菌wx-02基因组dna为模板,通过设计引物t5-f、t5-r,ker-f、ker-r来分别扩增载体骨架phy-r2和角蛋白酶基因ker片段(seqidno.6所示),并进行dna回收;
2、利用dpni酶对载体骨架phy-r2中残留的phy-r2-gfp模板进行消化;
3、采用vazyme的同源重组酶(exnaseii)对纯化的载体骨架phy-r2和ker基因片段进行处理,并在37℃条件下反应30min,使载体骨架和片段相连;随后进行钙转,将其转入大肠杆菌中;最后通过测序验证后,即得到重组质粒(phy-r2-ker);
4、将重组质粒(phy-r2-ker)转入地衣芽胞杆菌dw2中,以四环抗生素作为筛选标记,随后,通过菌落pcr筛选得到阳性转化子,从而获得r2介导的ker工程菌dw2/r2-ker;同理,先构建质粒phy-p43-ker(即将phy-p43-gfp中的gfp基因替换为ker基因),再将其电转入地衣芽胞杆菌dw2中,以获得对照菌株dw2/p43-ker。
5、工程菌dw2/p43-ker和dw2/r2-ker的种子活化和发酵阶段的相关操作除了发酵培养基外,与实施例2中的步骤5和6相同;
角蛋白酶发酵培养基:20g/l葡萄糖、10g/l大豆蛋白胨、10g/l玉米浆,5g/l酵母粉,10g/l氯化钠,1g/lk2hpo4和1g/l(nh4)2so4。
6、角蛋白酶酶活测定参照中华人民共和国国家标准中所描述的福林法进行,该角蛋白酶发酵检测结果见图2。由图2可知:对照菌株dw2/p43-ker表达的ker酶活为293.12u/ml,而r2介导表达的ker酶活为2576.89u/ml,使ker酶活提高7.8倍。同时表明本发明涉及的人工启动子r2在地衣芽胞杆菌的蛋白表达中具有一定的普适性。
一个单位酶活(u)定义为:一定温度、一定ph条件下,蛋白酶液在1min内水解酪蛋白产生1μg酪氨酸所需要的酶量。
注:划线为同源臂区,18-19bp。
实施例4:
启动子r2在提高地衣芽胞杆菌的代谢产物产量中应用:
谷氨酸脱氢酶rocg启动子强化工程菌株wx-02::r2(rocg)的制备:
1、申请早期研究发现:强化谷氨酸脱氢酶rocg的表达水平,可以提高wx-02产γ-pga的能力(doi:10.1016/j.jbiotec.2020.02.015)。根据地衣芽胞杆菌wx-02基因组dna序列中的rocg基因启动子procg的序列,设计procg的上游同源臂引物(rocg-f1、rocg-r1)、下游同源臂引物(rocg-f3、rocg-r3)和启动子r2引物(rocg-f2、rocg-r2);然后以地衣芽胞杆菌wx-02的基因组dna和质粒phy-r2-gfp为模板,分别pcr扩增扩增以得到procgdna序列的上游同源臂片段、下游同源臂片段和启动子r2片段(procg的上游同源臂片段为523bp;procg的下游同源臂片段为531bp;启动子r2片段为307bp);
其中,rocg-f1、rocg-r1、rocg-f2、rocg-r2、rocg-f3、rocg-r3的序列为:
rocg-f1:gctctagagccaggcgcttcagcagg、
rocg-r1:cgaaaacataccacctatcaatgagcatcttatcccgt、
rocg-f2:tgataggtggtatgttttcg、
rocg-r2:ctcgccgtctggaaatttgtgatccttcctcctttag、
rocg-f3:caaatttccagacggcgag、
rocg-r3:ccgagctccccttgctccgacaccaat;
2、以procg序列的上游同源臂、下游同源臂和r2启动子片段为模板,上游同源臂引物rocg-f1、下游同源臂引物rocg-r3为引物,通过重叠延伸pcr将procg序列的上游同源臂、r2启动子片段和下游同源臂连接到一起,得到目的片段,seqidno.7所示;
3、采用xbai和saci限制性内切酶对步骤2中的目的基因片段进行双酶切得到双酶切基因片段;
4、并采用xbai和saci限制性内切酶对质粒t2(2)-ori进行双酶切得到线性质粒片段;
5、将步骤3的酶切基因片段和步骤4的线性质粒片段经t4dna连接酶进行连接,得到连接产物;通过氯化钙转化法将该连接产物转入大肠杆菌dh5α,在37℃的条件下经含有卡那青霉素抗性的培养基进行筛选,筛选得到转化子,对转化子挑质粒进行菌落pcr验证(所用引物为:t2-f和t2-r)。若转化子的pcr验证结果为:在1661bp处出现电泳条带,说明启动子替换载体构建成功,上述转化子为阳性转化子(命名为:启动子替换载体t2(2)-r2(rocg));
6、将启动子替换载体t2(2)-r2(rocg)转入地衣芽胞杆菌wx-02中,在37℃的条件下经含有卡那青霉素抗性的培养基进行筛选,筛选得到转化子,对转化子挑质粒进行菌落pcr验证(所用引物为:t2-f和t2-r)。若转化子的pcr验证结果为:在1661bp处出现电泳条带,证明:启动子替换载体t2(2)-r2(rocg)成功转入地衣芽胞杆菌wx-02中,此时,该转化子为阳性转化子(即转入了启动子替换载体t2(2)-r2(rocg)的地衣芽胞杆菌wx-02);
其中,t2-f和t2-r的序列为:
t2-f:atgtgataactcggcgta、
t2-r:gcaagcagcagattacgc;
7、将步骤6得到的阳性转化子在45℃条件下、含有卡那青霉素抗性的培养基上转接培养3次,每次培养12h,并以t2-f和rocg-kr为引物进行菌落pcr检测单交换菌株,扩增出1811长度的条带,即证明为单交换菌株;
其中,rocg-kf和rocg-kr的序列为:
rocg-kf:ggatccagacaaaccagg、
rocg-kr:aatctttttatactgtcc;
8、将步骤7得到的单交换成功的菌株接种培养,开始双交换传代。在37℃、不含有卡那青霉素的培养基中经过数次转接培养,挑转化子进行菌落pcr验证(引物为rocg-kf和rocg-kr)。若转化子的pcr验证结果为:在1685bp处出现电泳条带时,说明wx-02的基因组上的rocg基因的启动子procg成功被替换成r2,该转化子为阳性转化子。随后针对阳性转化子进行dna测序进一步验证,得到双交换成功的rocg基因启动子强化菌株(即地衣芽胞杆菌wx-02::r2(rocg))。
9、构建对照菌株,即地衣芽胞杆菌wx-02::p43(rocg),同理,采取相似的操作构建,用p43替代了该菌株中的procg,即获得地衣芽胞杆菌wx-02::p43(rocg),作为对照组。
谷氨酸脱氢酶rocg启动子强化对wx-02产γ-pga的影响:
1、γ-pga生产菌株wx-02、dw2::p43(rocg)和wx-02::r2(rocg)的种子活化和发酵阶段除发酵培养基外,与实施例2中的步骤5和6相同,γ-pga发酵检测的方法参考申请人已发表的文献(doi:10.1016/j.jbiotec.2020.02.015)。
γ-pga液体发酵培养基:葡萄糖80g/l,柠檬酸钠10g/l,硝酸钠10g/l,氯化铵8g/l,k2hpo4·3h2o1g/l,znso4·7h2o1g/l,mnso4·h2o0.15g/l,cacl21g/l,ph7.2。
其γ-pga发酵检测结果见图3。由图3可知:菌株wx-02、wx-02::p43(rocg)和wx-02::r2(rocg)生产的γ-pga产量依次为10.08g/l、12.79g/l、15.29g/l。与野生型菌株相比wx-02,启动子r2效果优化p43,其使γ-pga产量提高52.09%。从而,启动子r2可用于代谢工程以强化γ-pga的代谢通量。
实施例5:
杆菌肽合成基因簇bacabc启动子强化工程菌dw2-r2(bacabc)制备:
1、根据地衣芽胞杆菌dw2基因组dna序列中的bacabc基因簇启动子pbaca的序列,设计pbaca的上游同源臂引物(baca-f1、baca-r1)、下游同源臂引物(baca-f3、baca-r3)和启动子r2引物(baca-f2、baca-r2);然后以地衣芽胞杆菌dw2的基因组dna和质粒phy-r2-gfp为模板,分别pcr扩增扩增以得到pbacadna序列的上游同源臂片段、下游同源臂片段和启动子r2片段(pbaca的上游同源臂片段为520bp;pbaca的下游同源臂片段为528bp;启动子r2片段为307bp);
其中,baca-f1、baca-r1、baca-f2、baca-r2、baca-f3、baca-r3的序列为:
baca-f1:gctctagacgaaaaggtgacgactacgc,
baca-r1:cgaaaacataccacctatcattaaaacgtccggctggctg,
baca-f2:tgataggtggtatgttttcg,
baca-r2:ctaatgaatgtttagcaaccattgatccttcctcctttag,
baca-f3:atggttgctaaacattcattag,
baca-r3:ccgagctcgtctgaagcctctctgtc;
2、以pbaca序列的上游同源臂、下游同源臂和r2启动子片段为模板,上游同源臂引物baca-f1、下游同源臂引物baca-r3为引物,通过重叠延伸pcr将pbaca序列的上游同源臂、r2启动子片段和下游同源臂连接到一起,得到目的片段,seqidno.8所示;
3、采用xbai和saci限制性内切酶对步骤2中的目的基因片段进行双酶切得到双酶切基因片段;
4、并采用xbai和saci限制性内切酶对质粒t2(2)-ori进行双酶切得到线性质粒片段;
5、将步骤3的酶切基因片段和步骤4的线性质粒片段经t4dna连接酶进行连接,得到连接产物;通过氯化钙转化法将该连接产物转入大肠杆菌dh5α,在37℃的条件下经含有卡那青霉素抗性的培养基进行筛选,筛选得到转化子,对转化子挑质粒进行菌落pcr验证(所用引物为:t2-f和t2-r)。若转化子的pcr验证结果为:在1655bp处出现电泳条带,说明启动子替换载体构建成功,上述转化子为阳性转化子(命名为:启动子替换载体t2(2)-r2(bacabc));
6、将启动子替换载体t2(2)-r2(bacabc)转入地衣芽胞杆菌dw2中,在37℃的条件下经含有卡那青霉素抗性的培养基进行筛选,筛选得到转化子,对转化子挑质粒进行菌落pcr验证(所用引物为:t2-f和t2-r)。若转化子的pcr验证结果为:在1655bp处出现电泳条带,证明:启动子替换载体t2(2)-r2(bacabc)成功转入地衣芽胞杆菌dw2中,此时,该转化子为阳性转化子(即转入了启动子替换载体t2(2)-r2(bacabc)的地衣芽胞杆菌dw2);
其中,t2-f和t2-r的序列为:
t2-f:atgtgataactcggcgta、
t2-r:gcaagcagcagattacgc;
7、将步骤6得到的阳性转化子在45℃条件下、含有卡那青霉素抗性的培养基上转接培养3次,每次培养12h,并以t2-f和baca-kr为引物进行菌落pcr检测单交换菌株,扩增出1625bp长度的条带,即证明为单交换菌株;
其中,baca-kf和baca-kr的序列为:
baca-kf:catatgcagcccgacattgag,
baca-kr:gacagaactgagcggttc;
8、将步骤7得到的单交换成功的菌株接种培养,开始双交换传代。在37℃、不含有卡那青霉素的培养基中经过数次转接培养,挑转化子进行菌落pcr验证(引物为baca-kf和baca-kr)。若转化子的pcr验证结果为:在1575bp处出现电泳条带时,说明dw2的基因组上的bacabc基因簇的启动子pbaca成功被替换成r2,该转化子为阳性转化子。随后针对阳性转化子进行dna测序进一步验证,得到双交换成功的bacabc基因簇启动子强化菌株(即地衣芽胞杆菌dw2::r2(bacabc))。
9、同理,采取相似的操作构建,用p43替代了该菌株中的启动子pbaca,即获得地衣芽胞杆菌dw2::p43(bacabc)。
杆菌肽合成基因簇bacabc启动子强化对dw2产杆菌肽的影响:
1、杆菌肽生产菌株dw2、dw2::p43(bacabc)和dw2::r2(bacabc)的种子活化和发酵阶段的相关操作除发酵培养基外,与实施例2中的步骤5和6相同,杆菌肽发酵检测结果见图4。
杆菌肽发酵培养基:100g/l豆粕,45g/l玉米淀粉,6g/l轻钙,1%硫酸铵,ph7.0。
由图4可知:菌株dw2、dw2::p43(bacabc)和dw2::r2(bacabc)生产的杆菌肽效价依次为817.54u/ml、536.72u/ml、989.22u/ml。与野生型菌株相比dw2,核心区四组合启动子r2效果明显优化p43,其使杆菌肽效价提高20.39%。从而,表明核心区四组合启动子r2可用于代谢工程以强化代谢产物的代谢通量。
序列表
<110>湖北大学
<120>适用于地衣芽胞杆菌的启动子及其在高效表达目的产物中的应用
<160>45
<170>siposequencelisting1.0
<210>1
<211>290
<212>dna
<213>人工序列(artificialsequence)
<400>1
tgataggtggtatgttttcgcttgaacttttaaatacagccattgaacatacggttgatt60
taataactgacaaacatcaccctcttgctaaagcggccaaggacgctgccgccggggctg120
tttgcgtttttgccgtgatttcgtgtatcattggtttacttatttttttgccaaagctgt180
aatggctgaaaattcttacatttattttacatttttagaaatgggcgtgaaaaaaagcgc240
gcgattatgtaaaatataaagtgatagcagatctaaaggaggaaggatca290
<210>2
<211>304
<212>dna
<213>人工序列(artificialsequence)
<400>2
tgataggtggtatgttttcgcttgaacttttaaatacagccattgaacatacggttgatt60
taataactgacaaacatcaccctcttgctaaagcggccaaggacgctgccgccggggctg120
tttgcgtttttgccgtgatttcgtgtatcattggtttacttatttttttgccaaagctgt180
aatggctgaaaattcttacatttattttacatttttagaaatgggcgtaaaagccattga240
cattctcatattatatattataataaaatataaagtgatagcagatctaaaggaggaagg300
atca304
<210>3
<211>307
<212>dna
<213>人工序列(artificialsequence)
<400>3
tgataggtggtatgttttcgcttgaacttttaaatacagccattgaacatacggttgatt60
taataactgacaaacatcaccctcttgctaaagcggccaaggacgctgccgccggggctg120
tttgcgtttttgccgtgatttcgtgtatcattggtttacttatttttttgccaaagctgt180
aatggctgaaaattcttacatttattttacatttttagaaatgggcgtaaaaagccattg240
acattctcatattatatatttataattaaaatataaagtgatagcagatctaaaggagga300
aggatca307
<210>4
<211>308
<212>dna
<213>人工序列(artificialsequence)
<400>4
tgataggtggtatgttttcgcttgaacttttaaatacagccattgaacatacggttgatt60
taataactgacaaacatcaccctcttgctaaagcggccaaggacgctgccgccggggctg120
tttgcgtttttgccgtgatttcgtgtatcattggtttacttatttttttgccaaagctgt180
aatggctgaaaattcttacatttattttacatttttagaaatgggcgccttgaagccatt240
gacattctcaccccatatatttataattaaaatataaagtgatagcagatctaaaggagg300
aaggatca308
<210>5
<211>306
<212>dna
<213>人工序列(artificialsequence)
<400>5
tgataggtggtatgttttcgcttgaacttttaaatacagccattgaacatacggttgatt60
taataactgacaaacatcaccctcttgctaaagcggccaaggacgctgccgccggggctg120
tttgcgtttttgccgtgatttcgtgtatcattggtttacttatttttttgccaaagctgt180
aatggctgaaaattcttacatttattttacatttttagaaatgggcgccttgaagccatt240
gacattctcaccccatatattataataaaatataaagtgatagcagatctaaaggaggaa300
ggatca306
<210>6
<211>1149
<212>dna
<213>人工序列(artificialsequence)
<400>6
atgagaggcaaaaaggtatggatcagtttgctgtttgctttagcgttaatctttacgatg60
gcgttcggcagcacgacttctgcccaggctgcagggaaatcaaacggggaaaagaaatat120
attgtcggatttaagcagacaatgagcacgatgagcgccgccaagaaaaaagatgtcatt180
tctgaaaaaggcgggaaagtggaaaagcaattcaaatatgtagacgcagcttcagctaca240
ttaaatgaaaaagctgtaaaagagctgaaaaaagaccctagcgtcgcttacgttgaagaa300
gatcacattgcacaggcgtacgcgcagtccgtgccttacggcgtatcacagattaaagcc360
cctgctctgcactctcaaggcttcaccggatcaaatgttaaagtagcggttatcgacagc420
ggtatcgattcttctcatcctgatttaaaggtagcaggcggagccagcatggttccttct480
gaaacaaatcctttccaagataacaactctcacggaactcacgttgccggtacagttgcg540
gctcttaataactcagtcggtgtattaggcgttgcgccaagcgcatctctttacgctgta600
aaagttctcggcgctgacggttccggccagtacagctggatcattaacggaattgagtgg660
gcgatcgcaaacaatatggacgttattaacatgagcctcggcggaccttctggttctgca720
gcgttaaaagcggcagttgacaaagccgttgcttccggcgtcgtagtggttgcggcagcc780
ggtaacgaaggcacttccggcggctcaagcacagtgggctaccctggtaaatacccttct840
gtcattgcggtaggcgctgttaacagcagcaaccaaagagcatctttctcaagcgtaggt900
tctgagcttgatgtcatggcaccaggcgtctctatccaaagcacgcttcctggaaacaaa960
tacggcgcgtacaatggtacgtcaatggcatctccgcacgttgccggagcggctgctttg1020
attctttctaagcacccgaactggacaaacactcaagtccgcagcagtttagaaaacacc1080
actacaaaacttggtgatgctttctactatggaaaagggctgatcaacgtacaggcggca1140
gctcagtaa1149
<210>7
<211>2245
<212>dna
<213>人工序列(artificialsequence)
<400>7
ggatccagacaaaccaggccatgaaccaaatgagccaggcgcttcagcaggcgcagcagt60
catttaaccagcaagggcaacaaggacaacagggtcagcaaaatcaacaaaaccagcagc120
aaaatcaaaattttcaacagcctctacaataaaaagctgtaaagagccgtttagcacggc180
tcttttttatatcggtcattcctcgttttgatcctcttcgtcatgggttggatgcgctcc240
gggatattgacgcggcgttccttgatgaagattgcggaattcgtatgcgaaattgctgta300
gtattgctggccttccttccatggcgtgcttttgttctcaacgggctcgtctttgccctg360
tgcagctccatacgcaccttcaggaaattcttcagctgttaaaaagtttctttgcgtttc420
aacgttcgacatttcttttttgtcgtccatcagatcacccctgttttagttttattgttg480
ccatgaaaaaggggagctatgtgttttcgttttgtatgcagtaggtttcattttaaaaac540
gggataagatgctcattctctgccgggagtacaaggggggaaatgcgccgatcacaaaga600
aaatagacattttgtacagaaagaacctgtcagataaagtggtattttatggtttattgg660
tccttttctcctcagcttttaagagctgtccttctatcttatgacccgtctattctcgct720
ttttgttatacatctcaattcagcaatggttttatctgttttccagattgatgtaaatgg780
ggcattggttgtaaaatttaccatgagagaaaatcttacaatgttcgttctgcttttatt840
ttagaaagggagctgaaaaaaggtgcatacgctagaaaaaatggagcaaacaaatttcca900
gacggcgagagattatgtgacacaagcatacgagacagtacagaagcgaaatttttacga960
aagcgaatttcttcaagctgtaaaggaaatatttgattcccttgtccctgtattggcaag1020
gcatccaaagtatatcgaacaccgcattcttgagaggatcgcagagccggaacggatgat1080
caccttcagggtgccgtgggtcgatgatgaaggcaatatccgggttaaccgagggttccg1140
ggttcaatttaacagtgcaatcggtccgtataaaggcggcatccgctttcacccttctgt1200
gaacgcgagcattattaaatttttgggttttgagcagatttttaaaaattctttgaccgg1260
actgccgatcggaggcggaaaaggcggggctgattttgatccgaagggcaaatcggacag1320
ggagattatgagttttacgcagagcttcatgaatgaactgtacagacatatcggaccgga1380
cacggatatccctgccggcgatattggtgtcggagcaagggaagtcgggtttatgttcgg1440
acagtataaaaagattcggggccgctatgatgcaggcgtgttaacaggcaaaggccttga1500
atacgggggcagtttaacgaggaaagaagcgacagggtacggtctggtttatttcgtgga1560
agaaatgctgaaggatcaggggatgcgctttgaaaacagcaccgttgtcgtctccggttc1620
agggaatgtggcgctgtacgcgatggaaaaagccgctcaattcggtgcgaaggtggtggc1680
ctgcagcgattctgacggctatgtctatgacgaaaaaggcatctgtcttgagacggtgaa1740
gcggctcaaagaagacgggaacggaaggattcgcgagtatgtcagcgagcatccggaagc1800
acactatttcgagggatgtaccggcatttggtctattccatgcgatatcgcgcttccgtg1860
cgcgacccagaacgaaattgacgaagaggcggccgaagtgctcatttcaaatggggtcaa1920
agctgtcggagaaggagcaaatatgccgtctgaagagggcgccgtcaaacgctttttgga1980
tgcgggagttctattcggaccggctaaggctgcaaatgccggcggtgtagccgtttcagc2040
gctcgaaatggcgcagaacagcgcacggcttcactggacggcggaagaaacggatgcgaa2100
gctcagggcgatcatggctgatattcacaagagaagcgttgaagcggcttcagaatacgg2160
acggcccggaaatctgctcgacggctgcaatatagccggatttatcaaagtggcggatgc2220
gatgatcgctcagggagtcgtttaa2245
<210>8
<211>1580
<212>dna
<213>人工序列(artificialsequence)
<400>8
catatgcagcccgacattgagatttgtccgattcagctgaagggaaggggccggcgtttc60
aacgagccttgttacgaaagccttgaagaagcagttcaagacatttttgagcaggttcaa120
gctgaacgaaaaggtgacgactacgctcttttcgggcacagcatgggaagccttttggca180
tatgaactttactatcaaatgagcggggcgggagctgaaaaaccggttcacatttttttc240
tcgggctataaagcgccaaacaggatcagaaagacagaaaaactgcataccttgcccaat300
cctatttttaagaaaaaaattgtcgagctcgggggaacgcctgaggagctcatcaatcat360
gaagagctatttgaattgtttatccccattctcaaaagcgactttaaaatggtagaaaac420
tatatctatcaagaaagaaacagcaaaatagattgcgacattaccgttctcaacggaaaa480
gaagacgccatgagcaaggaagatgtatccgattggaaacatcatacttcaggacacttt540
acagcctattactttgaggggaatcatttctttttgcaccatcacgttgaaaagatcacc600
gaaatcatcaatcattcactgacagccagccggacgttttaacctgcgatttcggcgaga660
ttcaagcccgggtctaatctatttttccttcttcggacgcttcaaaaattacttttatta720
taatcggaacagtgttttttagatcttttgatctatttggtgtttatcttgtctcataaa780
tacatgtttaaacaatgtaaaatataaaatatccaattcataaaaaattaaccattatta840
aacaatattcctatggaaaataatgattatttttgataatctgttttcacaagacggagg900
ttcaataaaaaatcggtaaaagagcaactacagaccaatattatggtgaatattttatca960
aaaaggagaatttttatatggttgctaaacattcattagaaaatggggtatttcacaaaa1020
tgacagagaatgaaaaagaactgatcctacattttaacaacacaaaaaccgattatccaa1080
aaaacaagacgcttcatgagctttttgaagaacaggccatgaagacgcctgaccatacgg1140
ctcttgtgttcggtgctcagcgcatgacctacagagagctgaatgagaaggcaaaccaaa1200
ccgcgagactcctcagagaaaaagggatcggcagaggctcgattgccgcgatcatcgcgg1260
accgctcctttgaaatgatcatcggcatcatcggtattttaaaagcgggaggtgcgtatc1320
tgccgatcgaccctgaaacgccgaaagacagaattgccttcatgctcagcgacacgaaag1380
ccgcggtgctgcttacgcaaggaaaagcggcggacggaatcgattgtgaggctgacatcg1440
ttcagttggacagagaggcttcagacggattcagtaaagaaccgctcagttctgtcaatg1500
attccggcgataccgcctatattatctacacatcaggttccacgggaacgccaaaaggcg1560
tcatcaccccgcactacagt1580
<210>9
<211>12
<212>dna
<213>人工序列(artificialsequence)
<400>9
ttgacatataat12
<210>10
<211>13
<212>dna
<213>人工序列(artificialsequence)
<400>10
gccttgaccccat13
<210>11
<211>12
<212>dna
<213>人工序列(artificialsequence)
<400>11
ttctcataaaat12
<210>12
<211>12
<212>dna
<213>人工序列(artificialsequence)
<400>12
aagccatatatt12
<210>13
<211>12
<212>dna
<213>人工序列(artificialsequence)
<400>13
aagccatatatt12
<210>14
<211>51
<212>dna
<213>人工序列(artificialsequence)
<400>14
tatataatatgagaatgtcaatggcttttacgcccatttctaaaaatgtaa51
<210>15
<211>54
<212>dna
<213>人工序列(artificialsequence)
<400>15
gacattctcatattatatattataataaaatataaagtgatagcagatctaaag54
<210>16
<211>52
<212>dna
<213>人工序列(artificialsequence)
<400>16
tatataatatgagaatgtcaatggctttttacgcccatttctaaaaatgtaa52
<210>17
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>17
acattctcatattatatatttataattaaaatataaagtgatagcagatctaaag55
<210>18
<211>53
<212>dna
<213>人工序列(artificialsequence)
<400>18
tatatggggtgagaatgtcaatggcttcaaggcgcccatttctaaaaatgtaa53
<210>19
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>19
gacattctcaccccatatatttataattaaaatataaagtgatagcagatctaaag56
<210>20
<211>52
<212>dna
<213>人工序列(artificialsequence)
<400>20
atatggggtgagaatgtcaatggcttcaaggcgcccatttctaaaaatgtaa52
<210>21
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>21
tgacattctcaccccatatattataataaaatataaagtgatagcagatctaaag55
<210>22
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>22
gtttattatccatacccttac21
<210>23
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>23
cagatttcgtgatgcttgtc20
<210>24
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>24
tgatccttcctcctttagatctg23
<210>25
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>25
aagagcagagaggacggatttcc23
<210>26
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>26
ctaaaggaggaaggatcaatgagaggcaaaaaggtatg38
<210>27
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>27
tccgtcctctctgctcttttactgagctgccgcctgtac39
<210>28
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>28
gctctagagccaggcgcttcagcagg26
<210>29
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>29
cgaaaacataccacctatcaatgagcatcttatcccgt38
<210>30
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>30
tgataggtggtatgttttcg20
<210>31
<211>37
<212>dna
<213>人工序列(artificialsequence)
<400>31
ctcgccgtctggaaatttgtgatccttcctcctttag37
<210>32
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>32
caaatttccagacggcgag19
<210>33
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>33
ccgagctccccttgctccgacaccaat27
<210>34
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>34
atgtgataactcggcgta18
<210>35
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>35
gcaagcagcagattacgc18
<210>36
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>36
ggatccagacaaaccagg18
<210>37
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>37
aatctttttatactgtcc18
<210>38
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>38
gctctagacgaaaaggtgacgactacgc28
<210>39
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>39
cgaaaacataccacctatcattaaaacgtccggctggctg40
<210>40
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>40
tgataggtggtatgttttcg20
<210>41
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>41
ctaatgaatgtttagcaaccattgatccttcctcctttag40
<210>42
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>42
atggttgctaaacattcattag22
<210>43
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>43
ccgagctcgtctgaagcctctctgtc26
<210>44
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>44
catatgcagcccgacattgag21
<210>45
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>45
gacagaactgagcggttc18
- 还没有人留言评论。精彩留言会获得点赞!