一种催化活性和热稳定性提高的谷氨酰胺转氨酶变体
1.本发明涉及一种催化活性和热稳定性提高的谷氨酰胺转氨酶变体,属于生物领域和食品领域。
背景技术:
2.茂源链霉菌(streptomyces mobaraenesis aat65817)来源的谷氨酰胺转氨酶 (transglutaminase,以下称tgase,ec 2.3.2.13)是一种广泛应用于食品领域的工业酶制剂。它可以催化蛋白及小分子中谷氨酰胺残基的γ羧酰胺基与酰基受体中氨基发生反应促使二者形成共价交联。目前,tgase在食品中以添加剂形式应用,尤其在食品预处理期间应用频繁,如肉类、豆制品及乳制品加工中进行tgase添加以提升食品品相和口感。
3.tgase的稳定性差一直是限制其应用空间的主要问题。稳定性差致使tgase不易保存、在运输以及使用中损耗均比较大。而在食品加工中,加入tgase后往往伴随着升温以辅助食品处理,这期间tgase损耗量大则造成成本增高。此外,酶的催化活性,也一直是酶制剂领域的关注热点。因此,挖掘稳定性提升和/或催化活性得到提高的tgase突变体非常重要。
技术实现要素:
4.为了解决上述问题,本发明提供了热稳定性改善的谷氨酰胺转氨酶变体。
5.本发明的谷氨酰胺转氨酶变体,包含对应于seq id no.1所示的多肽的第287位的取代,其中,
6.i)所述变体是与如seq id no:1所示的多肽具有至少60%、至少70%、至少80%、至少 90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%、但小于100%序列同一性的多肽;和/或
7.ii)所述变体是由以下多核苷酸编码的多肽,所述多核苷酸与如seq id no:4所示的成熟多肽编码序列具有至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%、但小于100%序列同一性。
8.其中,所述第287位的氨基酸为丙氨酸(简写为a)。
9.在一种实施方式中,所述第287位的取代,取代成脯氨酸(pro)。
10.在一种实施方式中,所述谷氨酰胺转氨酶变体,在实际生产中,与seq id no.1的谷氨酰胺转氨酶相比,可以与茂源链霉菌streptomyces mobaraenesis谷氨酰胺转氨酶酶原区(seq id no.2)相连进行表达。
11.在一种实施方式中,所述谷氨酰胺转氨酶变体,在实际生产中,与seq id no.1的谷氨酰胺转氨酶相比,可以与streptomyces caniferus谷氨酰胺转氨酶(seq id no.3)的酶原区相连进行表达。
12.在一种实施方式中,所述的谷氨酰胺转氨酶变体还包含对应于seq id no.1所示的多肽的第2、23、24、199、294位的氨基酸取代。
13.在一种实施方式中,所述第2、23、24、199、294位的氨基酸取代,是发生如下取代
s2p、 s23v、y24n、s199a、k294l。
14.在一种实施方式中,所述谷氨酰胺转氨酶变体为a287p,与seq id no.1所示的多肽相比,第287位取代成为脯氨酸。
15.本发明还涉及编码所述变体的多核苷酸;包含所述多核苷酸的核酸构建体、载体、和宿主细胞;以及产生所述变体的方法。此外,本发明涉及包含本发明的谷氨酰胺转氨酶变体的组合物。
16.本发明还涉及产生本发明的谷氨酰胺转氨酶变体的方法,这些方法包括:
17.a)在适合于表达所述变体的条件下培养本发明的宿主细胞;以及
18.b)任选地回收所述变体。
19.本发明还涉及在鲜肉加工、肠类制品、鱼丸、肉糜加工、豆制品和/或乳制品中改变食品品相、口感和/或稳定性的方法,所述方法包括在上述食品加工过程中添加本发明所述的谷氨酰胺转氨酶变体或本发明所述的组合物进行处理。
20.本发明还涉及所述的谷氨酰胺转氨酶变体或本发明所述的组合物在食品处理、加工和转化方面的应用。
21.在一种实施方式中,用于保持或改善食品的品质、稠度、弹性、水分或粘度。
22.在一种实施方式中,其中所述食品选自奶酪、酸乳、冰淇淋、蛋黄酱和肉类。
23.在一种实施方式中,其中所述食品为鱼类。
24.在一种实施方式中,用于形成不同密度的明胶和制备少脂的预烹调食品。
25.定义或术语:
26.谷氨酰胺转氨酶:术语“谷氨酰胺转氨酶(transglutaminase,tgase)”、“r-谷氨酰胺酰-肽酰胺酶-γ-谷氨酰-转移酶”、“转谷氨酰胺酶”是指如酶命名法所定义的ec 2.3.2.13类中的酶。出于本发明的目的,根据实例中所述的程序确定谷氨酰胺转氨酶活性。在一方面,本发明的变体具有seq id no:1的多肽的谷氨酰胺转氨酶活性的至少20%,例如至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、或100%。
27.编码序列:术语“编码序列”意指多核苷酸,所述多核苷酸直接规定了谷氨酰胺转氨酶变体的氨基酸序列。编码序列的边界通常由可读框确定,所述可读框以起始密码子(例如atg、 gtg或ttg)开始并且以终止密码子(例如taa、tag或tga)结束。编码序列可以为基因组 dna、cdna、合成dna或其组合。
28.控制序列:术语“控制序列”意指对于表达编码本发明的谷氨酰胺转氨酶变体的多核苷酸所必需的核酸序列。每个控制序列对于编码所述谷氨酰胺转氨酶变体的多核苷酸来说可以是原生的(即,来自相同基因)或外源的(即,来自不同基因),或相对于彼此是原生的或外源的。此类控制序列包括但不限于前导序列、多腺苷酸化序列、前肽序列、启动子、信号肽序列、以及转录终止子。最少,控制序列包括启动子、以及转录和翻译终止信号。出于引入促进控制序列与编码本发明的谷氨酰胺转氨酶变体的多核苷酸的编码区域连接的特异性限制位点的目的,控制序列可以提供有接头。
29.表达:术语“表达”包括涉及谷氨酰胺转氨酶变体产生的任何步骤,包括但不限于转录、转录后修饰、翻译、翻译后修饰、以及分泌。
30.表达载体:术语“表达载体”意指直链或环状dna分子,所述分子包含编码本发明的
谷氨酰胺转氨酶变体的多核苷酸并且可操作地连接至提供用于其表达的控制序列。
31.片段:术语“片段”意指在多肽的氨基和/或羧基末端缺失一个或多个(例如,若干个)氨基酸的多肽;其中所述片段具有谷氨酰胺转氨酶活性。在一方面,片段含有seq id no:1的氨基酸1至331(即不包含酶原区序列长度)的数目的至少50%、至少55%、至少60%、至少 65%、至少70%、至少75%、至少80%、至少85%、至少90%、或至少95%、至少96%、至少97%、至少98%、至少99%,但小于100%。
32.宿主细胞:术语“宿主细胞”意指易于用包含本发明的多核苷酸的核酸构建体或表达载体进行转化、转染、转导等的任何细胞类型。术语“宿主细胞”涵盖由于复制期间出现的突变而与亲本细胞不完全相同的任何亲本细胞子代。
33.改善的热稳定性:术语“改善的热稳定性”意指与相对于亲本谷氨酰胺转氨酶有所改善的谷氨酰胺转氨酶变体的特征。
34.分离的:术语“分离的”意指处于自然界中不存在的形式或环境中的物质。分离的物质的非限制性实例包括(1)任何非天然存在的物质;(2)至少部分地从与其在自然界中相关的一种或多种或全部天然存在的组分中去除的任何物质,包括但不限于任何酶、变体、核酸、蛋白质、肽或辅因子;(3)相对于自然界中发现的那种物质通过人工手动修饰的任何物质;或(4)通过相对于与其天然相关的其他组分增加所述物质的量而修饰的任何物质(例如,编码所述物质的基因的多个拷贝;比与编码所述物质的基因天然相关的启动子更强的启动子的使用)。分离的物质可以存在于发酵液样品中。
35.成熟多肽:术语“成熟多肽”意指在翻译和任何翻译后修饰如n-末端加工、c-末端截短、糖基化作用、磷酸化作用等之后处于其最终形式的多肽。在一方面,所述成熟多肽是seq idno:1的氨基酸1至331。本领域中已知,宿主细胞可以产生由相同多核苷酸表达的两种或更多种不同成熟多肽(即,具有不同的c-末端和/或n-末端氨基酸)的混合物。
36.成熟多肽编码序列:术语“成熟多肽编码序列”意指编码具有谷氨酰胺转氨酶活性的成熟多肽的多核苷酸。在一方面,所述成熟多肽编码序列是seq id no:2的核苷酸1至993(即不包含酶原区所对应的密码子序列)。
37.突变体:术语“突变体”意指编码变体的多核苷酸。
38.核酸构建体:术语“核酸构建体”意指单链或双链的核酸分子,所述核酸分子是从天然存在的基因中分离的,或以本来不存在于自然界中的方式被修饰成含有核酸的区段,或是合成的,所述核酸分子包含一个或多个控制序列。
39.母本或母本谷氨酰胺转氨酶:术语“母本”或“母本谷氨酰胺转氨酶”意指进行改变以产生本发明的谷氨酰胺转氨酶变体的谷氨酰胺转氨酶。本专利所述母本1为来源于茂源链霉菌 streptomyces mobaraenesis的谷氨酰胺转氨酶,包含促溶蛋白标签trxa(氨基酸序列如seqid no:7)streptomyces mobaraenesis来源的酶原区(seq id no:2)与成熟区(seq id no:1)。所述母本2包含促溶蛋白标签trxa(氨基酸序列如seq id no:7)、来源于streptomycescaniferus的谷氨酰胺转氨酶酶原区(seq id no:3)及茂源链霉菌streptomyces mobaraenesis 的谷氨酰胺转氨酶成熟区(seq id no:1),并在seq id no:1上含有突变s2p、s23v、y24n、 s199a、k294l的酶。所述母本成熟酶,即只包含成熟区多肽所对应序列的谷氨酰胺转氨酶。
40.序列同一性:两个氨基酸序列之间或两个核苷酸序列之间的关联度通过参数“序
为蛋白质量标记marker,单位为kda;1为母本2大肠杆菌e.coli bl21表达后全细胞电泳分析;2变体2大肠杆菌e.coli bl21表达后全细胞电泳分析。
52.图5:母本1、2与变体1、2成熟酶经纯化所得样品经sds-page分析;m为蛋白质量标记marker,单位为kda;1为母本1;2为母本2;3为变体1;4变体2.
具体实施方式
53.本发明涉及与亲本谷氨酰胺转氨酶相比已得到改善的谷氨酰胺转氨酶变体。更确切地说,本发明涉及与母本谷氨酰胺转氨酶成熟酶(特别是如seq id no:1所示的谷氨酰胺转氨酶,或者在seq id no:1的基础上发生了酶原区替换及s2p、s23v、y24n、s199a、k294l突变的酶)相比,具有改善的热稳定性的谷氨酰胺转氨酶变体。
54.根据本发明如下方法测定谷氨酰胺转氨酶的酶活、热稳定性、动力学参数。
55.酶活测试
56.酶活测试底物溶液a:200mm tris-hcl,100mm羟胺,10mm还原型谷胱甘肽,30mm n-苄氧羰基-l-谷氨酰甘氨酸,调节ph至6.0.
57.酶活测试终止液b:3mol/l hcl、5%fecl3·
6h2o(溶于0.1mol/lhcl)以及12%tca(三氯乙酸)进行等体积混合即完成溶液配置。
58.酶活定义:一个单位的酶活定义为每分钟催化1μmol底物形成产物的酶量。
59.酶活测试的标准曲线制作:称取648mg的标准品l-谷氨酸-γ-单异羟肟酸加100ml的 tris-hcl 200mm,ph 6.0溶液,并以tris-hcl 200mm,ph 6.0溶液通过2倍稀释法依次稀释5个梯度,将该溶液与底物溶液a分别进行37℃保温5min,之后取60μl标准品溶液到 150μl底物溶液a中,37℃水浴10min后然加入60μl终止液b,通过10000rpm离心1min 后取200μl上清液测定525nm吸光值。以吸光值比氧肟酸的量做直线,由直线的斜率得到一个转换系数k,在样品酶活的测定中得到吸光度后就可以通过k计算出氧肟酸的生成量。
60.测定方法:蛋白样品和150μl底物溶液a进行37℃孵育5min,之后取60μl蛋白样品加入150μl底物溶液a,37℃水浴10min后加入60μl测试终止液b。反应液10000rpm离心1min,取200μl上清液测定波长为525nm的吸光值。空白对照为60μl测试终止液b加 60μl蛋白样品,再加入150μl底物溶液a经10000rpm离心1min后取200μl上清液测定525nm吸光值。以实验组获得的吸光值减去对照组获得的吸光值,并带入到酶活标准曲线中,即可获得蛋白加入的质量对应的酶活,以该酶活除以蛋白浓度即获得蛋白比酶活u/mg。
61.热稳定性测试
62.测定50℃水浴条30min后残余酶活百分比,具体方法为,首先将谷氨酰胺转氨酶成熟酶及变体成熟酶溶液稀释至0.5mg/ml,取一定量该样品在50℃水浴进行连续热孵育,取出的样品放于20℃条件下进行冷敷。分别对所取样品进行酶活测定,以随时间变化谷氨酰胺转氨酶成熟酶残余酶活比初始酶活获得残余酶活百分比。
63.测定60℃水浴条件下半衰期,具体方法为,首先将谷氨酰胺转氨酶成熟酶及不同突变体蛋白溶液稀释至0.5mg/ml,取一定量该样品在60℃水浴进行连续热孵育,在0~10min内每分钟进行取样,10~40min则采用每2min取样,取出的样品均立即放于20℃条件下进行冷敷。分别对所取样品进行酶活测定,以获得随时间变化谷氨酰胺转氨酶成熟酶残余酶活比初始酶活获得残余酶活百分比,通过original 2018中exponential-expdec1对其
进行非线性拟合,获得拟合公式后计算出酶活下降至初始的50%所对应的时间即为半衰期。
64.本发明的谷氨酰胺转氨酶变体,包含对应于seq id no.1所示的多肽的第287位(第287 位的氨基酸为丙氨酸(简写为a))的取代,其中,
65.i)所述变体是与如seq id no:1所示的多肽具有至少60%、至少70%、至少80%、至少 90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、或至少99%、但小于100%序列同一性的多肽;和/或
66.ii)所述变体是由以下多核苷酸编码的多肽,所述多核苷酸与如seq id no:4所示的成熟多肽编码序列具有至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%、但小于100%序列同一性。
67.在一种实施例中,所述第287位的氨基酸是取代成了脯氨酸(p)。
68.在一种实施例中,所述谷氨酰胺转氨酶变体,与seq id no.1所示的多肽相比,仅发生了第287的取代,取代成脯氨酸(p)。与seq id no.1所示的谷氨酰胺转氨酶相比,具有提高的热稳定性,即50℃水浴条件下处理30分钟后残余酶活百分比提高了186.17%;具有提高的比酶活,提高了9.04%。
69.在一种实施例中,所述谷氨酰胺转氨酶变体,与母本2相比,发生了对应seq id no.1 的a287p的突变,突变后具有提高的热稳定性和催化活性,其中60℃半衰期提高了317.56%,比酶活提高了22.04%。其中,母本2,在表达过程中,与seq id no.1所示的多肽相比,在 seq id no.1所示的多肽氮端添加了序列为seq id no.3的streptomyces caniferus谷氨酰胺转氨酶酶原区,且在seq id no.1所示的多肽发生了s2p、s23v、y24n、s199a、k294l突变。
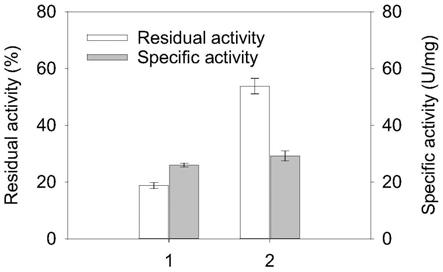
70.表1列出了与母本1即包含seq id no.1所示的多肽相比,第287位的取代的谷氨酰胺转氨酶变体1的相对热稳定性和比酶活。
71.表1
[0072][0073]
表2列出了母本2和变体2的相对热稳定性和比酶活。
[0074]
表2
[0075][0076]
变体的制备
[0077]
可以使用本领域已知的任何诱变程序(例如定点诱变、合成基因构建、半合成基因构建、随机诱变、改组等)来制备本发明的谷氨酰胺转氨酶变体。
[0078]
定点诱变是在编码所述亲本谷氨酰胺转氨酶的多核苷酸中的一个或多个限定位点处引入一个或多个(例如,若干个)突变的技术。
[0079]
通过涉及使用含有所希望的突变的寡核苷酸引物的pcr可以体外实现定点诱变。也可以通过盒式诱变进行体外定点诱变,所述盒式诱变涉及由限制酶在包含编码亲本谷氨酰胺转氨酶的多核苷酸的质粒中的位点处切割并且随后将含有突变的寡核苷酸连接在多核苷酸中。通常,消化质粒和寡核苷酸的限制酶是相同的,从而允许质粒和插入物的粘性末端彼此连接。
[0080]
还可以通过本领域中已知的方法在体内实现定点诱变。
[0081]
可以在本发明中使用任何定点诱变程序。存在可用于制备变体的许多可商购的试剂盒。
[0082]
合成基因构建需要设计的多核苷酸分子的体外合成以编码感兴趣的多肽。基因合成可以利用多种技术来进行。
[0083]
使用已知的诱变、重组和/或改组方法,随后进行相关的筛选程序可以做出单或多氨基酸取代、缺失和/或插入并对其进行测试。
[0084]
诱变/改组方法可以与高通量、自动化的筛选方法组合以检测由宿主细胞表达的克隆的、诱变的多肽的活性。可从宿主细胞回收编码活性多肽的诱变的dna分子,并使用本领域的标准方法快速测序。这些方法允许快速确定多肽中各个氨基酸残基的重要性。
[0085]
通过组合合成基因构建、和/或定点诱变、和/或随机诱变、和/或改组的多方面来实现半合成基因构建。半合成构建典型地是利用合成的多核苷酸片段的过程结合pcr技术。因此,基因的限定区域可以从头合成,而其他区域可以使用位点特异性诱变引物来扩增,而还有其他区域可以进行易错pcr或非易错pcr扩增。然后可以对多核苷酸子序列进行改组。
[0086]
多核苷酸
[0087]
本发明还涉及编码本发明的谷氨酰胺转氨酶变体的分离的多核苷酸。在某些方面,本发明涉及包含本发明的多核苷酸的核酸构建体。在某些方面,本发明涉及包含本发明的多核苷酸的表达载体。在某些方面,本发明涉及包含本发明的多核苷酸的宿主细胞。在某些方面,本发明涉及产生谷氨酰胺转氨酶变体的方法,所述方法包括:(a)在适合于表达所述谷氨酰胺转氨酶变体的条件下培养本发明的宿主细胞;和(b)回收所述谷氨酰胺转氨酶变体。
[0088]
核酸构建体
[0089]
本发明还涉及包含编码本发明的谷氨酰胺转氨酶变体的、可操作地连接至一个或多个控制序列的多核苷酸的核酸构建体,所述一个或多个控制序列在与控制序列相容的条件下指导编码序列在适合的宿主细胞中的表达。
[0090]
可以按多种方式来操纵多核苷酸以提供谷氨酰胺转氨酶变体的表达。取决于表达载体,在多核苷酸插入载体之前对其进行操作可以是理想的或必需的。用于利用重组dna方法修饰多核苷酸的技术是本领域熟知的。
[0091]
控制序列可以是启动子,即由宿主细胞识别用于表达所述多核苷酸的多核苷酸。启动子含有介导谷氨酰胺转氨酶变体的表达的转录控制序列。启动子可以是在宿主细胞中显示转录活性的任何多核苷酸,包括突变型、截短型和杂合型启动子,并且可以获得自编码与宿主细胞同源或异源的细胞外或细胞内多肽的基因。
[0092]
表达载体
[0093]
本发明还涉及包含编码本发明的谷氨酰胺转氨酶变体的多核苷酸、启动子、以及转录和翻译终止信号的重组表达载体。各种核苷酸和控制序列可以连接在一起以产生重组表达载体,所述重组表达载体可以包括一个或多个合宜的限制位点以允许在此类位点处插入或取代编码谷氨酰胺转氨酶变体的多核苷酸。可替代地,可以通过将多核苷酸或包含所述多核苷酸的核酸构建体插入用于表达的适当载体中而表达所述多核苷酸。在产生表达载体时,编码序列如此位于载体中,使得编码序列与用于表达的适当控制序列可操作地连接。
[0094]
重组表达载体可以是可以方便地经受重组dna程序并且可以引起多核苷酸表达的任何载体(例如,质粒或病毒)。载体的选择将典型地取决于载体与待引入载体的宿主细胞的相容性。载体可以是直链或闭合环状质粒。
[0095]
载体可以是自主复制载体,即作为染色体外实体存在的载体,其复制独立于染色体复制,例如质粒、染色体外元件、微染色体或人工染色体。载体可以含有用于确保自我复制的任何手段。可替代地,载体可以是这样一种载体,当被引入宿主细胞中时,它被整合到基因组中并与其整合的一个或多个染色体一起复制。此外,可以使用单独的载体或质粒或两个或更多个载体或质粒,其共同含有待引入宿主细胞基因组的总dna,或可以使用转座子。
[0096]
载体优选地含有允许方便地选择转化细胞、转染细胞、转导细胞等细胞的一个或多个选择性标记。选择性标记是一种基因,其产物提供了杀生物剂抗性或病毒抗性、对重金属抗性、对营养缺陷型的原养型等。
[0097]
细菌选择性标记的实例是地衣芽孢杆菌或枯草芽孢杆菌dal基因、或赋予抗生素抗性(如氨苄青霉素、氯霉素、卡那霉素、新霉素、大观霉素、或四环素抗性)的标记。酵母宿主细胞的适合的标记包括但不限于:ade2、his3、leu2、lys2、met3、trp1和ura3。用于在丝状真菌宿主细胞中使用的选择性标记包括但不限于amds(乙酰胺酶)、argb(鸟氨酸氨甲酰基转移酶)、bar(草胺膦乙酰转移酶)、hph(潮霉素磷酸转移酶)、niad(硝酸还原酶)、pyrg(乳清苷
ꢀ‑5’‑
磷酸脱羧酶)、sc(硫酸腺苷基转移酶)、以及trpc(邻氨基苯甲酸合酶),连同其等同物。优选的用于曲霉细胞中的是构巢曲霉或米曲霉amds和pyrg基因以及吸水链霉菌(streptomyceshygroscopicus)bar基因。
[0098]
载体优选地含有允许载体整合到宿主细胞的基因组中或载体在细胞中独立于基
因组自主复制的一个或多个元件。
[0099]
为了整合至宿主细胞基因组中,载体可以依赖于编码谷氨酰胺转氨酶变体的多核苷酸序列或用于借助同源或非同源重组整合至基因组中的任何其他载体元件。可替代地,所述载体可以含有用于指导通过同源重组而整合入宿主细胞基因组中的染色体中的精确位置处的另外的多核苷酸。为了增加在精确位置处整合的可能性,整合元件应当含有足够数目的核酸,例如100至10,000个碱基对、400至10,000个碱基对和800至10,000个碱基对,所述核酸与相应的靶序列具有高度序列同一性以增强同源重组的概率。整合元件可以是与宿主细胞基因组内的靶序列同源的任何序列。此外,整合元件可以是非编码或编码的多核苷酸。另一方面,载体可以通过非同源重组整合入宿主细胞的基因组中。
[0100]
为了自主复制,载体还可以另外包含复制起点,所述复制起点使得载体在讨论中的宿主细胞中自主复制成为可能。复制起点可以是在细胞中发挥作用的介导自主复制的任何质粒复制子。术语“复制起点”或“质粒复制子”意指使质粒或载体能够在体内复制的多核苷酸。
[0101]
细菌复制起点的实例是允许在大肠杆菌中复制的质粒pbr322、puc19、pacyc177、和 pacyc184的复制起点,以及允许在芽孢杆菌属中复制的质粒pub110、pe194、pta1060、和pamβ1的复制起点。
[0102]
用于酵母宿主细胞中的复制起点的实例是2微米复制起点、ars1、ars4、ars1与cen3 的组合、及ars4与cen6的组合。
[0103]
在丝状真菌细胞中有用的复制起点的实例是ama1和ans1(gems等人,1991,gene[基因]98:61-67;cullen等人,1987,nucleic acids res.[核酸研究]15:9163-9175;wo00/24883)。可以根据wo 00/24883中披露的方法完成ama1基因的分离和包含所述基因的质粒或载体的构建。
[0104]
可以将多于一个拷贝的本发明的多核苷酸插入宿主细胞中以增加谷氨酰胺转氨酶变体的产生。通过将序列的至少一个另外的拷贝整合到宿主细胞基因组中或通过包括一个与所述多核苷酸一起的可扩增的选择性标记基因可以获得所述多核苷酸的增加的拷贝数目,其中通过在适当的选择性试剂的存在下培养细胞可以选择含有选择性标记基因的经扩增的拷贝的细胞、以及由此所述多核苷酸的另外的拷贝。
[0105]
用于连接以上所述的元件以构建本发明的重组表达载体的程序是本领域的普通技术人员熟知的。
[0106]
宿主细胞
[0107]
本发明还涉及重组宿主细胞,所述重组宿主细胞包含编码本发明的谷氨酰胺转氨酶变体的、可操作地连接至一个或多个控制序列的多核苷酸,所述一个或多个控制序列指导本发明的谷氨酰胺转氨酶变体的产生。将包含多核苷酸的构建体或载体引入到宿主细胞中,这样使得所述构建体或载体被维持作为染色体整合体或作为自主复制的染色体外载体,如早前所描述。术语“宿主细胞”涵盖由于复制期间出现的突变而与亲本细胞不完全相同的任何亲本细胞子代。宿主细胞的选择在很大程度上取决于编码谷氨酰胺转氨酶变体的基因及其来源。
[0108]
宿主细胞可以是在谷氨酰胺转氨酶变体的重组生产中有用的任何细胞,例如原核细胞或真核细胞。
[0109]
原核宿主细胞可以是任何革兰氏阳性或革兰氏阴性细菌。革兰氏阳性细菌包括但不限于:芽孢杆菌属、梭菌属、肠球菌属、土芽孢杆菌属(geobacillus)、乳杆菌属、乳球菌属、大洋芽孢杆菌属、葡萄球菌属、链球菌属和链霉菌属。革兰氏阴性细菌包括但不限于弯曲杆菌属、大肠杆菌、黄杆菌属、梭杆菌属、螺杆菌属、泥杆菌属、奈瑟氏菌属、假单胞菌属、沙门氏菌属、以及脲原体属。
[0110]
宿主细胞还可以是真核生物,如哺乳动物、昆虫、植物或真菌细胞。
[0111]
产生方法
[0112]
本发明还涉及产生本发明的谷氨酰胺转氨酶变体的方法,所述方法包括:(a)在适合于表达所述谷氨酰胺转氨酶变体的条件下培养本发明的宿主细胞;和(b)回收所述谷氨酰胺转氨酶变体。
[0113]
使用本领域已知的方法在适合于产生谷氨酰胺转氨酶变体的营养介质中培养宿主细胞。例如,可以通过摇瓶培养,或者在适合的培养基中并在允许谷氨酰胺转氨酶或变体表达和/或分离的条件下在实验室或工业发酵罐中进行小规模或大规模发酵(包括连续发酵、分批发酵、分批给料发酵或固态发酵)来培养细胞。使用本领域中已知的程序,培养发生在包含碳和氮来源及无机盐的适合的营养培养基中。适合的培养基可从商业供应商获得或可以根据公开的组成制备。如果谷氨酰胺转氨酶变体被分泌到营养介质中,则所述谷氨酰胺转氨酶变体可以直接从培养基中回收。如果谷氨酰胺转氨酶变体没有分泌,则它可以从细胞裂解液中回收。
[0114]
可以使用本领域已知的对谷氨酰胺转氨酶变体特异的方法检测谷氨酰胺转氨酶变体。这些检测方法包括但不限于:特异性抗体的使用、酶产物的形成或酶底物的消失。例如,可以使用酶测定来确定谷氨酰胺转氨酶变体的活性(如实例中所述的那些)。
[0115]
可以使用本领域已知的方法回收谷氨酰胺转氨酶变体。例如,可以通过常规程序从营养介质中回收谷氨酰胺转氨酶变体,所述常规程序包括但不限于收集、离心、过滤、提取、喷雾干燥、蒸发或沉淀。
[0116]
可以通过本领域中已知的多种程序来纯化谷氨酰胺转氨酶变体以获得基本上纯的谷氨酰胺转氨酶变体,所述程序包括但不限于色谱法(例如,离子交换色谱、亲和色谱、疏水作用色谱、色谱聚焦、以及尺寸排阻色谱)、电泳程序(例如,制备型等电点聚焦)、差别溶解度(例如,硫酸铵沉淀)、sds-page或萃取。
[0117]
在可替代的方面,没有回收谷氨酰胺转氨酶变体,而是将表达谷氨酰胺转氨酶变体的本发明的宿主细胞用作所述谷氨酰胺转氨酶变体的来源。
[0118]
发酵液配制品或细胞组合物
[0119]
本发明还涉及包含本发明的多肽的发酵液配制品或细胞组合物。所述发酵液产物进一步包含在发酵过程中使用的另外的成分,例如像细胞(包括含有编码本发明的多肽的基因的宿主细胞,这些宿主细胞用于产生目的多肽)、细胞碎片、生物质、发酵培养基和/或发酵产物。在一些实施例中,组合物是含有一种或多种有机酸、杀灭的细胞和/或细胞碎片以及培养基的细胞杀灭的全培养液。
[0120]
如本文使用的术语“发酵液”是指由细胞发酵产生的、不经历或经历最少的回收和/或纯化的制剂。例如,当微生物培养物在允许蛋白质合成(例如,由宿主细胞表达酶)并且将蛋白质分泌到细胞培养基中的碳限制条件下孵育生长到饱和时,产生发酵液。所述发
酵液可以含有在发酵结束时得到的发酵材料的未分级的或分级的内容物。典型地,所述发酵液是未分级的并且包含用过的培养基以及例如通过离心去除微生物细胞(例如,丝状真菌细胞)之后存在的细胞碎片。在一些实施例中,发酵液含有用过的细胞培养基、胞外酶以及有活力的和/或无活力的微生物细胞。
[0121]
在一个实施例中,发酵液配制品和细胞组合物包含第一有机酸组分(包含至少一种1-5个碳的有机酸和/或其盐)和第二有机酸组分(包含至少一种6个或更多个碳的有机酸和/或其盐)。在一个具体实施例中,第一有机酸组分是乙酸、甲酸、丙酸、其盐或前述两种或更多种的混合物;并且所述第二有机酸组分是苯甲酸、环己烷羧酸、4-甲基戊酸、苯乙酸、其盐或前述两种或更多种的混合物。
[0122]
在一方面,组合物含有一种或多种有机酸,并且任选地进一步含有杀灭的细胞和/或细胞碎片。在一个实施例中,从细胞杀灭的全培养液中去除这些杀灭的细胞和/或细胞碎片,以提供不含这些组分的组合物。
[0123]
这些发酵液配制品或细胞组合物可以进一步包含防腐剂和/或抗微生物(例如,抑菌)剂,包括但不限于山梨醇、氯化钠、山梨酸钾、以及本领域已知的其他试剂。
[0124]
所述细胞杀灭的全培养液或组合物可以含有在发酵结束时得到的发酵材料的未分级的内容物。典型地,所述细胞杀灭的全培养液或组合物含有用过的培养基以及在微生物细胞(例如,丝状真菌细胞)生长至饱和、在碳限制条件下孵育以允许蛋白合成之后存在的细胞碎片。在一些实施例中,所述细胞杀灭的全培养液或组合物含有用过的细胞培养基、胞外酶和杀灭的丝状真菌细胞。在一些实施例中,可以使用本领域已知的方法来使细胞杀灭的全培养液或组合物中存在的微生物细胞透性化和/或裂解。
[0125]
如本文所述的全培养液或细胞组合物典型地是液体,但是可以含有不溶性组分,例如杀灭的细胞、细胞碎片、培养基组分和/或一种或多种不溶性酶。在一些实施例中,可以去除不溶性组分以提供澄清的液体组合物。
[0126]
组合物
[0127]
本发明还涉及包括本发明的变体谷氨酰胺转氨酶的组合物。
[0128]
这些组合物可以包含本发明的变体谷氨酰胺转氨酶作为主要酶组分,例如,单组分组合物。可替代地,组合物可以包含多种酶活性,例如选自由以下组成的组的一种或多种(例如,若干种)酶:蛋白酶、葡糖淀粉酶、β-淀粉酶、支链淀粉酶。
[0129]
实施例1
[0130]
下面结合其中一种具体实施方式对部分谷氨酰胺转氨酶变体的制备和热稳定性能进行阐述。
[0131]
涉及的大肠杆菌jm109以及大肠杆菌e.coli bl21(de3)购自takara-宝日医生物技术(北京)有限公司,pet-22b(+)质粒购自novagen公司(上述菌株大肠杆菌e.coli bl21(de3)可以购买得到,不需要进行用于专利程序的保藏),中性蛋白酶购于北京索莱宝科技有限公司(货号z8032),blunting kination ligation(bkl)kit和hs dna polymerase购于宝日医生物技术(北京)有限公司,bradford蛋白浓度测定试剂盒(去垢剂兼容型)购于上海碧云天生物技术有限公司。
[0132]
涉及的培养基如下:
[0133]
lb液体培养基:酵母粉5.0g/l、胰蛋白胨10.0g/l、nacl 10.0g/l、氨苄青霉素100μ
g/l。
[0134]
lb固体培养基:酵母粉5.0g/l、胰蛋白胨10.0g/l、nacl 10.0g/l、琼脂粉15g/l、氨苄青霉素100μg/l。
[0135]
tb培养基:酵母提取物24g/l、胰蛋白胨12g/l、三水合磷酸氢二钾12.84g/l、磷酸二氢钾2.31g/l,甘油4ml/l、氨苄青霉素100μg/l。
[0136]
1、突变体的构建
[0137]
母本1的成熟区如seq id no:1,即来源于茂源链霉菌streptomyces mobaraenesis的谷氨酰胺转氨酶的成熟区,表达框包含促溶蛋白标签trxa(氨基酸序列如seq id no:7)、茂源链霉菌streptomyces mobaraenesis自身的酶原区(seq id no:2)与成熟区(seq id no:1)。所述母本2的成熟区,包含对应于seq id no:1的s2p、s23v、y24n、s199a、k294l突变,表达框包含促溶蛋白标签trxa(氨基酸序列如seq id no:7)、来源于streptomyces caniferus 的谷氨酰胺转氨酶酶原区(seq id no:3)后连接突变后的茂源链霉菌streptomycesmobaraenesis的谷氨酰胺转氨酶成熟区(seq id no:1)。
[0138]
具体构建如下:
[0139]
含有母本1和母本2的基因委托苏州金唯智公司进行合成,并通过限制性内切酶位点ndei 和blpi插入到质粒pet-22b(+)中得到pet-22b-tgase(包含载体整体序列如seq id no.5)和 pet-22b-proc/tgm(包含载体整体序列如seq id no.6)。变体1(与母本1相比,发生对应 seq id no:1的a287p突变)和变体2(与母本2相比,发生对应seq id no:1的a287p突变)在分别以pet-22b-tgase和pet-22b-proc/tgm作为模板,分别对应287f/287r和287mf/287mr 作为引物进行pcr和线性dna环化获得,以上所有应用到的引物见表2。pcr过程参考宝日医生物技术(北京)有限公司hs dnapolymerase说明书,线性dna环化方法参考宝日医生物技术(北京)有限公司blunting kination ligation(bkl)kit说明书。
[0140]
表3引物基因序列
[0141][0142]
2、突变体酶的制备方法
[0143]
将构建好能表达母本、变体的质粒转化大肠杆菌jm109,转化产物涂布于lb固体培养基,于37℃培养10h并挑取转化子进行序列测定,获得测序正确的重组质粒转化大肠杆菌 e.coli bl21(de3)得到能表达对应谷氨酰胺转氨酶变体的重组大肠杆菌。
[0144]
获得的重组大肠杆菌涂布于lb固体培养基,在37℃培养10h挑取转化子接入lb液体培养基(含100μg/ml氨苄青霉素),于37℃培养10h以1%转接量转接至tb液体培养基(含100μg/ml氨苄青霉素)。通过37℃培养大肠杆菌e.coli bl21(de3)至od
600
到1.0~1.5之间,加入iptg至终浓度0.01mm对重组蛋白表达进行诱导。接入iptg后培养温度改为20℃并连续培养36h。所有液体培养均采用摇床培养,转速为220rpm。
[0145]
发酵后对样品进行7500rpm离心10min并收集菌体,加入发酵液总体积五分之一的 tris-hcl 50mm,ph 8.0进行菌体重悬,并冰敷10min。将样品置于冰上进行超声破壁,并于12000rpm离心15min后回收上清液,向上清液中加入200mg/ml的中性蛋白酶溶液(中性蛋白酶购自北京索莱宝科技有限公司,货号z8032)对谷氨酰胺转氨酶进行活化,孵育条件为37℃水浴30min。对孵育后样品进行12000rpm离心20min后取上清,并通过镍离子亲和纯化方式进行蛋白提纯,具体方法为:取镍离子亲和纯化柱先后分别过水和tris-hcl 50 mm,20mm imidazole,ph 7.8溶液至电导平衡,之后通过样品,并用tris-hcl 50mm,20mmimidazole,ph 7.8溶液进行冲洗至电导再次平衡,然后通过溶液tris-hcl 50mm,180mmimidazole,ph 7.8溶液对蛋白进行洗脱,并回收蛋白。整个纯化过程在aktapure机器上完成,通过观察a280波长吸收峰来确定蛋白收集时间及收集量。收集后蛋白样品通过凝胶色谱法进行蛋白溶液脱盐,具体方法为:取凝胶柱先后分别过水和tris-hcl 50mm,ph 8.0溶液至电导平衡,然后使凝胶柱通过蛋白样品,之后继续通过tris-hcl 50mm,ph 8.0溶液至 a280波长出现吸收峰并收集蛋白,在电导出现变化时及时停止收集。蛋白浓度采用bca测试法,具体方法参考上海碧云天生物技术有限公司bradford蛋白浓度测定试剂盒(去垢剂兼容型)说明书。
[0146]
对母本和变体的比酶活、残余酶活百分比、60℃半衰期等进行测试,结果如表1、表2 图1、图2、图3和图4所示。
[0147]
从表1和图1中,可以看出:变体1相较于母本1的催化活性和热稳定性均所有提高,其中50℃处理30分钟后残余酶活百分比有186.17%的显著提高,同时比酶活也有9.04%的提高。
[0148]
从表2和图2中,可以看出:变体2相较于母本2的催化活性和热稳定性均所有提高,其中60℃(t
1/260℃
)下半衰期提高了371.56%、比酶活提高了22.04%。
[0149]
从图3中,可以看出:母本1和变体1分别实现了在大肠杆菌e.coli bl21中的表达。在两个泳道中,在蛋白分子量略微低于49kda的地方可以看到明显的条带,与理论大小43.3 kda相近。
[0150]
从图4中,可以看出:母本2和变体2分别实现了在大肠杆菌e.coli bl21中的表达。在两个泳道中,在蛋白分子量49-62kda之间可以看到明显的条带,与理论大小56.5kda相近。
[0151]
从图5中,可以看出:蛋白经纯化后条带单一,表明蛋白纯度符合预期。
[0152]
实施例2:谷氨酰胺转氨酶变体在肉制品加工中的应用
[0153]
利用实施例1制备的谷氨酰胺转氨酶变体2成熟酶进行兔肉脯加工,具体是:
[0154]
s1、将兔肉绞碎、鸡肉切丁,得到混合肉;
[0155]
s2、将食盐、复合磷酸盐和水混合均匀后,加入s1中得到的混合肉,混合均匀、用保鲜膜封口后,腌制10h,得到腌制肉;
[0156]
s3、腌制肉匀浆,得到混合肉糜;
[0157]
s4、向s3中得到的混合肉糜中加入谷氨酰胺转氨酶、卵清蛋白、姜粉等,在温度为2℃下搅拌均匀,得到混合肉糜;
[0158]
s5、将s4中得到的混合肉糜用保鲜膜封口后,在温度为60℃的水浴条件下放置0.2h后,挤压成型,烘干,自然冷却,得到半成品;
[0159]
s6、将s5中得到的半成品烤制,得到复合兔肉脯。
[0160]
相关序列
[0161]
seq id no:1的序列如下:
[0162]
dsddrvtppaepldrmpdpyrpsygraetvvnnyirkwqqvyshrdgrkqqmteeqrew lsygcvgvtwvnsgqyptnrlafasfdedrfknelkngrprsgetraefegrvakesfdee kgfqrarevasvmnralenahdesayldnlkkelangndalrnedarspfysalrntpsf kernggnhdpsrmkaviyskhfwsgqdrsssadkrkygdpdafrpapgtglvdmsrdrni prsptspgegfvnfdygwfgaqteadadktvwthgnhyhapngslgamhvyeskfrnw segysdfdrgayvitfipkswntapdkvkqgwp
[0163]
seq id no:2的序列如下:
[0164]
dngageetksyaetyrltaddvaninalnesapaassagpsfrap
[0165]
seq id no:3的序列如下:
[0166]
masggdeewegsyaathgltaedvkninalnkraltagqpgnfpaelppsatalfrap d
[0167]
seq id no:4的序列如下:
[0168]
gactccgacgacagggtcacccctcccgccgagccgctcgacaggatgcccgacc cgtaccgtccctcgtacggcagggccgagacggtcgtcaacaactacatacgcaagtgg cagcaggtctacagccaccgcgacggcaggaagcagcagatgaccgaggagcagcgg gagtggctgtcctacggctgcgtcggtgtcacctgggtcaattcgggtcagtacccgac gaacagactggccttcgcgtccttcgacgaggacaggttcaagaacgagctgaagaac ggcaggccccggtccggcgagacgcgggcggagttcgagggccgcgtcgcgaaggag agcttcgacgaggagaagggcttccagcgggcgcgtgaggtggcgtccgtcatgaaca gggccctggagaacgcccacgacgagagcgcttacctcgacaacctcaagaaggaact ggcgaacggcaacgacgccctgcgcaacgaggacgcccgttccccgttctactcggcg ctgcggaacacgccgtccttcaaggagcggaacggaggcaatcacgacccgtccagga tgaaggccgtcatctactcgaagcacttctggagcggccaggaccggtcgagttcggc cgacaagaggaagtacggcgacccggacgccttccgccccgccccgggcaccggcctg gtcgacatgtcgagggacaggaacattccgcgcagccccaccagccccggtgagggat tcgtcaatttcgactacggctggttcggcgcccagacggaagcggacgccgacaagac cgtctggacccacggaaatcactatcacgcgcccaatggcagcctgggtgccatgcatg tctacgagagcaagttccgcaactggtccgagggttactcggacttcgaccgcggagc ctatgtgatcaccttcatccccaagagctggaacaccgcccccgacaaggtaaagcagg gctggccg
[0169]
seq id no:5的序列如下:(其中单下划线部分为trxa所对应的基因、双下划线为茂源链霉菌streptomyces mobaranesis谷氨酰胺转氨酶酶原区所对应的基因、波浪线为茂源链霉菌 streptomyces mobaranesis谷氨酰胺转氨酶成熟区所对应的基因)
[0170]
tggcgaatgggacgcgccctgtagcggcgcattaagcgcggcgggtgtggtggttacgcgcagcgtgaccgctacacttgccagcgc cctagcgcccgctcctttcgctttcttcccttcctttctcgccacgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctgatagacggtttttcgccctttga cgttggagtccacgttctttaatagtggactcttgttccaaactggaacaacactcaaccctatctcggtctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaatattaacgtttacaatttcaggtggcacttttcggg gaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgcttcaataatattga aaaaggaagagtatgagtattcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaa agtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctc
aacagcggtaagatccttgagagttttcgccccgaag aacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtattgacgccgggcaagagcaactcggtcgccgcataca ctattctcagaatgacttggttgagtactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataac catgagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcacaacatgggggatcatgtaa ctcgccttgatcgttgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgcagcaatggcaacaacgttg cgcaaactattaactggcgaactacttactctagcttcccggcaacaattaatagactggatggaggcggataaagttgcaggaccacttctgcg ctcggcccttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggtatcattgcagcactggggccagatggta agccctcccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactgatt aagcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcctttttgata atctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgc gcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcg ctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcag cggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgag aaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctat ggaaaaacgccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctgattctgtggataa ccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgc ctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcatatatggtgcactctcagtacaatctgctctgatgccgcatagttaagcc agtatacactccgctatcgctacgtgactgggtcatggctgcgccccgacacccgccaacacccgctgacgcgccctgacgggcttgtctgctc ccggcatccgcttacagacaagctgtgaccgtctccgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcgaggcagctg cggtaaagctcatcagcgtggtcgtgaagcgattcacagatgtctgcctgttcatccgcgtccagctcgttgagtttctccagaagcgttaatgtct ggcttctgataaagcgggccatgttaagggcggttttttcctgtttggtcactgatgcctccgtgtaagggggatttctgttcatgggggtaatgata ccgatgaaacgagagaggatgctcacgatacgggttactgatgatgaacatgcccggttactggaacgttgtgagggtaaacaactggcggtat ggatgcggcgggaccagagaaaaatcactcagggtcaatgccagcgcttcgttaatacagatgtaggtgttccacagggtagccagcagcatc ctgcgatgcagatccggaacataatggtgcagggcgctgacttccgcgtttccagactttacgaaacacggaaaccgaagaccattcatgttgtt gctcaggtcgcagacgttttgcagcagcagtcgcttcacgttcgctcgcgtatcggtgattcattctgctaaccagtaaggcaaccccgccagcct agccgggtcctcaacgacaggagcacgatcatgcgcacccgtggggccgccatgccggcgataatggcctgcttctcgccgaaacgtttggt ggcgggaccagtgacgaaggcttgagcgagggcgtgcaagattccgaataccgcaagcgacaggccgatcatcgtcgcgctccagcgaaa gcggtcctcgccgaaaatgacccagagcgctgccggcacctgtcctacgagttgcatgataaagaagacagtcataagtgcggcgacgatagt catgccccgcgcccaccggaaggagctgactgggttgaaggctctcaagggcatcggtcgagatcccggtgcctaatgagtgagctaacttac attaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgc
cagctgcattaatgaatcggccaacgcgcggggagaggcggtt tgcgtattgggcgccagggtggtttttcttttcaccagtgagacgggcaacagctgattgcccttcaccgcctggccctgagagagttgcagcaa gcggtccacgctggtttgccccagcaggcgaaaatcctgtttgatggtggttaacggcgggatataacatgagctgtcttcggtatcgtcgtatcc cactaccgagatatccgcaccaacgcgcagcccggactcggtaatggcgcgcattgcgcccagcgccatctgatcgttggcaaccagcatcg cagtgggaacgatgccctcattcagcatttgcatggtttgttgaaaaccggacatggcactccagtcgccttcccgttccgctatcggctgaatttg attgcgagtgagatatttatgccagccagccagacgcagacgcgccgagacagaacttaatgggcccgctaacagcgcgatttgctggtgacc caatgcgaccagatgctccacgcccagtcgcgtaccgtcttcatgggagaaaataatactgttgatgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccacagcaatggcatcctggtcatccagcggatagttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttcgacgccgcttcgttctaccatcgacaccaccacgctggcacccagttgatcggcgcgagattta atcgccgcgacaatttgcgacggcgcgtgcagggccagactggaggtggcaacgccaatcagcaacgactgtttgcccgccagttgttgtgcc acgcggttgggaatgtaattcagctccgccatcgccgcttccactttttcccgcgttttcgcagaaacgtggctggcctggttcaccacgcgggaa acggtctgataagagacaccggcatactctgcgacatcgtataacgttactggtttcacattcaccaccctgaattgactctcttccgggcgctatc atgccataccgcgaaaggttttgcgccattcgatggtgtccgggatctcgacgctctcccttatgcgactcctgcattaggaagcagcccagtagt aggttgaggccgttgagcaccgccgccgcaaggaatggtgcatgcaaggagatggcgcccaacagtcccccggccacggggcctgccacc atacccacgccgaaacaagcgctcatgagcccgaagtggcgagcccgatcttccccatcggtgatgtcggcgatataggcgccagcaaccgc acctgtggcgccggtgatgccggccacgatgcgtccggcgtagaggatcgagatctcgatcccgcgaaattaatacgactcactataggggaa ttgtgagcggataacaattcccctctagaaataattttgtttaactttaagaaggagatataca
caccaccaccaccaccactgagatccg gctgctaacaaagcccgaaaggaagctgagttggctgctgccaccgctgagcaataactagcataaccccttggggcctctaaacgggtcttga ggggttttttgctgaaaggaggaactatatccggat
[0171]
seq id no:6的序列如下:(其中波浪线部分表示促溶蛋白trxa所对应的基因序列、单画线部分表示来源于streptomyces caniferus谷氨酰胺转氨酶酶原区的基因序列,双画线部分表示在序列seq id no:1所示氨基酸序列上做s2p、s23v、y24n、s199a、k294l突变后所对应的基因序列)
[0172]
tggcgaatgggacgcgccctgtagcggcgcattaagcgcggcgggtgtggtggttacgcgcagcgtgaccgctacacttgccagcgc cctagcgcccgctcctttcgctttcttcccttcctttctcgccacgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctgatagacggtttttcgccctttga cgttggagtccacgttctttaatagtggactcttgttccaaactggaacaacactcaaccctatctcggtctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaatattaacgtttacaatttcaggtggcacttttcggg gaaatgtgcgcggaacccctatttgtttattttt
ctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgcttcaataatattga aaaaggaagagtatgagtattcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaa agtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcggtaagatccttgagagttttcgccccgaag aacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtattgacgccgggcaagagcaactcggtcgccgcataca ctattctcagaatgacttggttgagtactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataac catgagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcacaacatgggggatcatgtaa ctcgccttgatcgttgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgcagcaatggcaacaacgttg cgcaaactattaactggcgaactacttactctagcttcccggcaacaattaatagactggatggaggcggataaagttgcaggaccacttctgcg ctcggcccttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggtatcattgcagcactggggccagatggta agccctcccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactgatt aagcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcctttttgata atctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgc gcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcg ctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcag cggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgag aaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctat ggaaaaacgccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctgattctgtggataa ccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgc ctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcatatatggtgcactctcagtacaatctgctctgatgccgcatagttaagcc agtatacactccgctatcgctacgtgactgggtcatggctgcgccccgacacccgccaacacccgctgacgcgccctgacgggcttgtctgctc ccggcatccgcttacagacaagctgtgaccgtctccgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcgaggcagctg cggtaaagctcatcagcgtggtcgtgaagcgattcacagatgtctgcctgttcatccgcgtccagctcgttgagtttctccagaagcgttaatgtct ggcttctgataaagcgggccatgttaagggcggttttttcctgtttggtcactgatgcctccgtgtaagggggatttctgttcatgggggtaatgata ccgatgaaacgagagaggatgctcacgatacgggttactgatgatgaacatgcccggttactggaacgttgtgagggtaaacaactggcggtat ggatgcggcgggaccagagaaaaatcactcagggtcaatgccagcgcttcgttaatacagatgtaggtgttccacagggtagccagcagcatc ctgcgatgcagatccggaacataatggtgcagggcgctgacttccgcgtttccagactttacgaaacacggaaaccgaagaccattcatgttgtt gctcaggtcgcagacgttttgcagcagcagtcgcttcacgttcgctcgcgtatcggtgattcattctgctaaccagtaaggcaaccccgccagcct agccgggtcctcaacgacaggagcacgatcatgcgcacccgtggggccgccatgccggcgataatggcctgcttctcgccgaaacgtttggt ggcgggaccagtgacgaaggcttgagcgagggcgtgcaagattccgaataccgcaagcgacaggccgatcatcgtcgcgctccagcgaaa gcggtcctcgcc
gaaaatgacccagagcgctgccggcacctgtcctacgagttgcatgataaagaagacagtcataagtgcggcgacgatagt catgccccgcgcccaccggaaggagctgactgggttgaaggctctcaagggcatcggtcgagatcccggtgcctaatgagtgagctaacttac attaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtt tgcgtattgggcgccagggtggtttttcttttcaccagtgagacgggcaacagctgattgcccttcaccgcctggccctgagagagttgcagcaa gcggtccacgctggtttgccccagcaggcgaaaatcctgtttgatggtggttaacggcgggatataacatgagctgtcttcggtatcgtcgtatcc cactaccgagatatccgcaccaacgcgcagcccggactcggtaatggcgcgcattgcgcccagcgccatctgatcgttggcaaccagcatcg cagtgggaacgatgccctcattcagcatttgcatggtttgttgaaaaccggacatggcactccagtcgccttcccgttccgctatcggctgaatttg attgcgagtgagatatttatgccagccagccagacgcagacgcgccgagacagaacttaatgggcccgctaacagcgcgatttgctggtgacc caatgcgaccagatgctccacgcccagtcgcgtaccgtcttcatgggagaaaataatactgttgatgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccacagcaatggcatcctggtcatccagcggatagttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttcgacgccgcttcgttctaccatcgacaccaccacgctggcacccagttgatcggcgcgagattta atcgccgcgacaatttgcgacggcgcgtgcagggccagactggaggtggcaacgccaatcagcaacgactgtttgcccgccagttgttgtgcc acgcggttgggaatgtaattcagctccgccatcgccgcttccactttttcccgcgttttcgcagaaacgtggctggcctggttcaccacgcgggaa acggtctgataagagacaccggcatactctgcgacatcgtataacgttactggtttcacattcaccaccctgaattgactctcttccgggcgctatc atgccataccgcgaaaggttttgcgccattcgatggtgtccgggatctcgacgctctcccttatgcgactcctgcattaggaagcagcccagtagt aggttgaggccgttgagcaccgccgccgcaaggaatggtgcatgcaaggagatggcgcccaacagtcccccggccacggggcctgccacc atacccacgccgaaacaagcgctcatgagcccgaagtggcgagcccgatcttccccatcggtgatgtcggcgatataggcgccagcaaccgc acctgtggcgccggtgatgccggccacgatgcgtccggcgtagaggatcgagatctcgatcccgcgaaattaatacgactcactataggggaa ttgtgagcggataacaattcccctctagaaataattttgtttaactttaagaaggagatatacatagaaggagatatacat
cacacc accaccaccaccactgagatccggctgctaacaaagcccgaaaggaagctgagttggctgctgccaccgctgagcaataacta gcataaccccttggggcctctaaacgggtcttgaggggttttttgctgaaaggaggaactatatccggat
[0173]
seq id no:7的序列如下:
[0174]
msdkiihltddsfdtdvlkadgailvdfwaewcgpckmiapildeiadeyqgkltvakl nidqnpgtapkygirgiptlllfkngevaatkvgalskgqlkefldanla。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1