用于控制肠道沙门氏菌的细菌素的制作方法
1.本发明提供了能够对沙门氏菌(salmonella)产生细胞毒性作用的蛋白,称为沙门菌素(salmocin)。本发明还提供了包含一种或更多种所述蛋白的组合物,包括药物组合物。还提供了预防或减少沙门氏菌对目标物的感染或污染的方法,治疗有此需要的受试者或患者的沙门氏菌感染的方法,以及生产包含所述蛋白的组合物的方法。
背景技术:
2.沙门氏菌是肠杆菌(enterobacteriaceae)科的杆状革兰氏阳性菌。肠道沙门氏菌(salmonella enterica)是模式种并进一步分成6个亚种,其中肠道沙门氏菌肠道亚种(s.enterica ssp.enterica)为包括超过2500种血清型的亚种。沙门氏菌感染是常见的,并且可以导致多变的临床表现,从无症状状态到非常严重的疾病。据估计,肠道沙门氏菌在美国每年导致100万例病例,估计导致19,000例住院病例和380人死亡。在过去的5年中,美国已经记录了46次沙门氏菌爆发,大多数食物中毒是由于被污染的家禽或蔬菜,以及红肉和鱼(cdc网站)。
3.预防沙门氏菌感染或减少食品受沙门氏菌污染需要在食物链的所有阶段采取控制措施,从农场的农业生产到商业机构和家庭厨房的食品加工、制造和制备。良好的卫生习惯减少了沙门氏菌对食物的污染,但不能保证产品中不含沙门氏菌。在家中对沙门氏菌采取的预防措施类似于对其他食源性细菌采取的措施。基本的食品卫生习惯,例如"煮熟",被推荐作为针对沙门氏菌病的预防措施,参见
4.www.who.int/mediacentre/factsheets/fs139/en/。
5.抗微生物疗法可用于治疗感染沙门氏菌的人或动物。然而,抗微生物剂的耐药性是全球公共健康关注的问题,并且沙门氏菌是已经出现了一些耐药血清型并影响食物链的微生物的一种。
6.上述预防或治疗沙门氏菌感染或减少沙门氏菌污染的大多数方法是基本上不依赖于特定的病原菌或沙门氏菌的特定血清型的方法。这样做的好处是,在采取应对措施之前,对所涉及的特定沙门氏菌菌株或肠道沙门氏菌血清型几乎没有必要事先了解。然而,上述预防沙门氏菌感染或减少沙门氏菌污染的方法,例如加热,并不总是适用的,或者以不希望的方式改变了处理的商品或食品。其他方法可能对特定患者无效。因此,需要预防或治疗沙门氏菌感染或污染,或减少或预防沙门氏菌,特别是肠道沙门氏菌肠道亚种对目标物的污染的另外方法。
7.之前描述了一些对沙门氏菌有活性的蛋白(wo 2018/172065),它们被称为“沙门菌素”。虽然wo 2018/172065中描述的沙门菌素对沙门氏菌物种和菌株显示出高活性,但局限性在于它们的毒性活性相似,即使将一些不同的已知沙门菌素组合在一起,其活性谱也受到限制。此外,中期或长期使用相同或相似沙门菌素类型可能选择出对所用沙门菌素具有抗性的沙门氏菌菌株。因此,非常需要通过其他机制起作用的其他沙门菌素。值得注意的是,期望包含两种或更多种不同类型沙门菌素的组合物来拓宽针对许多不同沙门氏菌物种
的活性范围。
8.对于用于技术应用的蛋白,易于生产、纯化和储存是重要的方面,其可以决定技术应用是否可行。
9.本发明的目的是提供预防或治疗沙门氏菌感染(如食源性沙门氏菌感染)的方法。另一个目的是提供预防或减少沙门氏菌对目标物(特别是食物)的污染的方法。本发明的另一个目的是提供预防或治疗沙门氏菌感染的方法和/或减少沙门氏菌对目标物污染的方法,所述方法对大范围的沙门氏菌血清群有效。另一个目的是提供更多的沙门菌素,它们通过不同的或另外的抗沙门氏菌的机制起作用。本发明的另一个目的是提供抗沙门氏菌的活性剂,其可以方便地以高稳定性生产和/或纯化和/或储存。此外,用于这些方法的化合物、试剂和组合物是期望的。
技术实现要素:
10.因此,本发明提供了权利要求中定义的保护主题。本发明还提供了:
11.(1)一种优选能够对沙门氏菌产生细胞毒性作用的蛋白,所述蛋白包含至少任何一个下列氨基酸序列区段(a-i)至(a-x)或(b-i)至(b-x)、(c-i)至(c-x)或(d-i)至(d-x)中所定义的其衍生物:
12.(a-i)来自scole2(seq id no:1)的氨基酸残基316至449的区段,
13.(a-ii)来自scole3(seq id no:2)的氨基酸残基315至483的区段,
14.(a-iii)来自scole7(seq id no:3)的氨基酸残基318至451的区段,
15.(a-iv)来自scole1a(seq id no:4)的氨基酸残基174至297的区段,
16.(a-v)来自scole1b(seq id no:5)的氨基酸残基198至322的区段,
17.(a-vi)包含seq id no:6的spst的至少200个连续氨基酸残基的区段;
18.(a-vii)来自scole1c(seq id no:25)的氨基酸残基195至319的区段,
19.(a-viii)来自scole1d(seq id no:26)的氨基酸残基195至319的区段,
20.(a-ix)来自scole1e(seq id no:27)的氨基酸残基193至317的区段,
21.(a-x)来自scolma(seq id no:28)的氨基酸残基38至138的区段,
22.(a-xi)来自scolmb(seq id no:33)的氨基酸残基38至138的区段,或
23.(a-xii)来自scolmc(seq id no:34)的氨基酸残基38至138的区段;或
24.(b-i)与来自scole2(seq id no:1)的氨基酸残基316至449的区段具有至少75%序列同一性的区段,
25.(b-ii)与来自scole3(seq id no:2)的氨基酸残基315至483的区段具有至少70%序列同一性的区段,
26.(b-iii)与来自scole7(seq id no:3)的氨基酸残基318至451的区段具有至少77%序列同一性的区段,
27.(b-iv)与来自scole1a(seq id no:4)的氨基酸残基174至297的区段具有至少70%序列同一性的区段,
28.(b-v)与来自scole1b(seq id no:5)的氨基酸残基198至322的区段具有至少70%序列同一性的区段,
29.(b-vi)与包含seq id no:6的spst的至少200个连续氨基酸残基的区段具有至少
70%序列同一性的区段,
30.(b-vii)与来自scole1c(seq id no:25)的氨基酸残基195至319的区段具有至少70%序列同一性的区段,
31.(b-viii)与来自scole1d(seq id no:26)的氨基酸残基195至319的区段具有至少70%序列同一性的区段,
32.(b-ix)与来自scole1e(seq id no:27)的氨基酸残基193至317的区段具有至少70%序列同一性的区段,
33.(b-x)与来自scolma(seq id no:28)的氨基酸残基38至138的区段具有至少70%序列同一性的区段,
34.(b-xi)与来自scolmb(seq id no:33)的氨基酸残基38至138的区段具有至少70%序列同一性的区段,或
35.(b-xii)与来自scolmc(seq id no:34)的氨基酸残基38至138的区段具有至少70%序列同一性的区段;
36.或
37.(c-i)与来自scole2(seq id no:1)的氨基酸残基316至449的区段具有至少85%序列相似性的区段,
38.(c-ii)与来自scole3(seq id no:2)的氨基酸残基315至483的区段具有至少80%序列相似性的区段,
39.(c-iii)与来自scole7(seq id no:3)的氨基酸残基318至451的区段具有至少85%序列相似性的区段,
40.(c-iv)与来自scole1a(seq id no:4)的氨基酸残基174至297的区段具有至少80%序列相似性的区段,
41.(c-v)与来自scole1b(seq id no:5)的氨基酸残基198至322的区段具有至少80%序列相似性的区段,
42.(c-vi)与包含seq id no:6的spst的至少200个连续氨基酸残基的区段具有至少80%序列相似性的区段,
43.(c-vii)与来自scole1c(seq id no:25)的氨基酸残基195至319的区段具有至少80%序列同一性的区段,
44.(c-viii)与来自scole1d(seq id no:26)的氨基酸残基195至319的区段具有至少80%序列同一性的区段,
45.(c-ix)与来自scole1e(seq id no:27)的氨基酸残基193至317的区段具有至少80%序列同一性的区段,
46.(c-x)与来自scolma(seq id no:28)的氨基酸残基38至138的区段具有至少80%序列同一性的区段,
47.(c-xi)与来自scolmb(seq id no:33)的氨基酸残基38至138的区段具有至少80%序列同一性的区段,或
48.(c-xii)与来自scolmc(seq id no:34)的氨基酸残基38至138的区段具有至少80%序列同一性的区段;
49.或
50.(d-i)与来自scole2(seq id no:1)的氨基酸残基316至449的区段相比具有1至25个氨基酸取代、添加、插入或缺失的区段,
51.(d-ii)与来自scole3(seq id no:2)的氨基酸残基315至483的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
52.(d-iii)与来自scole7(seq id no:3)的氨基酸残基318至451的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段,
53.(d-iv)与来自scole1a(seq id no:4)的氨基酸残基174至297的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
54.(d-v)与来自scole1b(seq id no:5)的氨基酸残基198至322的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
55.(d-vi)与包含seq id no:6的spst的至少200个连续氨基酸残基的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
56.(d-vii)与来自scole1c(seq id no:25)的氨基酸残基195至319的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
57.(d-viii)与来自scole1d(seq id no:26)的氨基酸残基195至319的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
58.(d-ix)与来自scole1e(seq id no:27)的氨基酸残基193至317的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
59.(d-x)与来自scolma(seq id no:28)的氨基酸残基38至138的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段,
60.(d-xi)与来自scolmb(seq id no:33)的氨基酸残基38至138的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段,或
61.(d-xii)与来自scolmc(seq id no:34)的氨基酸残基38至138的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段。
62.(2)根据(1)所述的蛋白,其包含具有任何一种或更多种以下活性的细胞毒性或催化结构域:膜成孔活性、dna酶活性、rna酶活性或细胞壁降解活性如溶菌酶活性。
63.(3)根据(1)或(2)所述的蛋白,其包含细胞毒性或催化结构域,所述细胞毒性或催化结构域包含下列氨基酸序列区段(a-i)’至(a-x)’或其衍生物,或由(b-i)’至(b-x)’、(c-i)’至(c-x)’或(d-i)’至(d-x)’所定义的氨基酸序列区段中的任何一个,或由其组成:
64.(a-i)’来自scole2(seq id no:1)的氨基酸残基453至582的区段,
65.(a-ii)’来自scole3(seq id no:2)的氨基酸残基501至584的区段,
66.(a-iii)’来自scole7(seq id no:3)的氨基酸残基455至584的区段,
67.(a-iv)’来自scole1a(seq id no:4)的氨基酸残基306至478的区段,
68.(a-v)’来自scole1b(seq id no:5)的氨基酸残基350至522的区段,
69.(a-vi)’来自spst(seq id no:6)的氨基酸残基112至288的区段,
70.(a-vii)’来自scole1c(seq id no:25)的氨基酸残基347至519的区段,
71.(a-viii)’来自scole1d(seq id no:26)的氨基酸残基347至519的区段,
72.(a-ix)’来自scole1e(seq id no:27)的氨基酸残基345至517的区段,
73.(a-x)’来自scolma(seq id no:28)的氨基酸残基139至269的区段,
74.(a-xi)’来自scolmb(seq id no:33)的氨基酸残基139至269的区段,或
75.(a-xii)’来自scolmc(seq id no:34)的氨基酸残基139至269的区段;
76.或
77.(b-i)’与来自scole2(seq id no:1)的氨基酸残基453至582的区段具有至少70%序列同一性的区段,
78.(b-ii)’与来自scole3(seq id no:2)的氨基酸残基501至584的区段具有至少70%序列同一性的区段,
79.(b-iii)’与来自scole7(seq id no:3)的氨基酸残基455至584的区段具有至少70%序列同一性的区段,
80.(b-iv)’与来自scole1a(seq id no:4)的氨基酸残基306至478的区段具有至少70%序列同一性的区段,
81.(b-v)’与来自scole1b(seq id no:5)的氨基酸残基350至522的区段具有至少70%序列同一性的区段,
82.(b-vi)’与来自spst(seq id no:6)的氨基酸残基112至288的区段具有至少70%序列同一性的区段,
83.(b-vii)’与来自scole1c(seq id no:25)的氨基酸残基347至519的区段具有至少70%序列同一性的区段,
84.(b-viii)’与来自scole1d(seq id no:26)的氨基酸残基347至519的区段具有至少70%序列同一性的区段,
85.(b-ix)’与来自scole1e(seq id no:27)的氨基酸残基345至517的区段具有至少70%序列同一性的区段,
86.(b-x)’与来自scolma(seq id no:28)的氨基酸残基139至269的区段具有至少70%序列同一性的区段,
87.(b-xi)’与来自scolmb(seq id no:33)的氨基酸残基139至269的区段具有至少70%序列同一性的区段,或
88.(b-xii)’与来自scolmc(seq id no:34)的氨基酸残基139至269的区段具有至少70%序列同一性的区段;
89.或
90.(c-i)’与来自scole2(seq id no:1)的氨基酸残基453至582的区段具有至少80%序列相似性的区段,
91.(c-ii)’与来自scole3(seq id no:2)的氨基酸残基501至584的区段具有至少80%序列相似性的区段,
92.(c-iii)’与来自scole7(seq id no:3)的氨基酸残基455至584的区段具有至少80%序列相似性的区段,
93.(c-iv)’与来自scole1a(seq id no:4)的氨基酸残基306至478的区段具有至少80%序列相似性的区段,
94.(c-v)’与来自scole1b(seq id no:5)的氨基酸残基350至522的区段具有至少80%序列相似性的区段,
95.(c-vi)’与来自spst(seq id no:6)的氨基酸残基112至288的区段具有至少80%
序列相似性的区段;
96.(c-vii)’与来自scole1c(seq id no:25)的氨基酸残基347至519的区段具有至少80%序列相似性的区段,
97.(c-viii)’与来自scole1d(seq id no:26)的氨基酸残基347至519的区段具有至少80%序列相似性的区段,
98.(c-ix)’与来自scole1e(seq id no:27)的氨基酸残基345至517的区段具有至少80%序列相似性的区段,
99.(c-x)’与来自scolma(seq id no:28)的氨基酸残基139至269的区段具有至少80%序列相似性的区段,
100.(c-xi)’与来自scolmb(seq id no:33)的氨基酸残基139至269的区段具有至少80%序列相似性的区段,或
101.(c-xii)’与来自scolmc(seq id no:34)的氨基酸残基139至269的区段具有至少80%序列相似性的区段;
102.或
103.(d-i)’与来自scole2(seq id no:1)的氨基酸残基453至582的区段相比具有1至20个氨基酸取代、添加、插入或缺失的区段,
104.(d-ii)’与来自scole3(seq id no:2)的氨基酸残基501至584的区段相比具有1至20个氨基酸取代、添加、插入或缺失的区段,
105.(d-iii)’与来自scole7(seq id no:3)的氨基酸残基455至584的区段相比具有1至20个氨基酸取代、添加、插入或缺失的区段,
106.(d-iv)’与来自scole1a(seq id no:4)的氨基酸残基306至478的区段相比具有1至20个氨基酸取代、添加、插入或缺失的区段,
107.(d-v)’与来自scole1b(seq id no:5)的氨基酸残基350至522的区段相比具有1至20个氨基酸取代、添加、插入或缺失的区段,或
108.(d-vi)’与来自spst(seq id no:6)的氨基酸残基112至288的区段相比具有1至20个氨基酸取代、添加、插入或缺失的区段,
109.(d-vii)’与来自scole1c(seq id no:25)的氨基酸残基347至519的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
110.(d-viii)’与来自scole1d(seq id no:26)的氨基酸残基347至519的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
111.(d-ix)’与来自scole1e(seq id no:27)的氨基酸残基345至517的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
112.(d-x)’与来自scolma(seq id no:28)的氨基酸残基139至269的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段,
113.(d-xi)’与来自scolmb(seq id no:33)的氨基酸残基139至269的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段,或
114.(d-xii)’与来自scolmc(seq id no:34)的氨基酸残基139至269的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段。
115.(4)根据(1)、(2)或(3)所述的蛋白,其包含易位结构域,所述易位结构域包含下列
氨基酸区段(a-i)”至(a-v)”、(a-vii)”至(a-x)”、(a-xi)”和(a-xii)”,或由(b-i)”至(b-v)”、(b-vii)”至(b-x)”、(b-xi)”和(b-xii)”、(c-i)”至(c-v)”、(c-vii)”至(c-x)”、(c-xi)”和(c-xii)”或(d-i)”至(d-v)”、(d-vii)”至(d-x)”、(d-xi)”和(d-xii)”所定义的其衍生物(或氨基酸序列区段)中的任何一个(或由其组成):
116.(a-i)”来自scole2(seq id no:1)的氨基酸残基43至313的区段,
117.(a-ii)”来自scole3(seq id no:2)的氨基酸残基35至315的区段,
118.(a-iii)”来自scole7(seq id no:3)的氨基酸残基的43至316的区段,
119.(a-iv)”来自scole1a(seq id no:4)的氨基酸残基1至170的区段,
120.(a-v)”来自scole1b(seq id no:5)的氨基酸残基1至195的区段,
121.(a-vii)’来自scole1c(seq id no:25)的氨基酸残基6至194的区段,
122.(a-viii)’来自scole1d(seq id no:26)的氨基酸残基6至194的区段,
123.(a-ix)’来自scole1e(seq id no:27)的氨基酸残基5至192的区段,
124.(a-x)’来自scolma(seq id no:28)的氨基酸残基1至37的区段,
125.(a-xi)’来自scolmb(seq id no:33)的氨基酸残基1至37的区段,或
126.(a-xii)’来自scolmc(seq id no:34)的氨基酸残基1至37的区段;
127.或
128.(b-i)”与来自scole2(seq id no:1)的氨基酸残基43至313的区段具有至少75%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
129.(b-ii)”与来自scole3(seq id no:2)的氨基酸残基35至315的区段具有至少75%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
130.(b-iii)”与来自scole7(seq id no:3)的氨基酸残基43至316的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段;
131.(b-iv)”与来自scole1a(seq id no:4)的氨基酸残基1至170的区段具有至少70%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
132.(b-v)”与来自scole1b(seq id no:5)的氨基酸残基1至195的区段具有至少70%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
133.(b-vii)”与来自scole1c(seq id no:25)的氨基酸残基6至194的区段具有至少70%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
134.(b-viii)”与来自scole1d(seq id no:26)的氨基酸残基6至194的区段具有至少70%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
135.(b-ix)”与来自scole1e(seq id no:27)的氨基酸残基5至192的区段具有至少70%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
136.(b-x)”与来自scolma(seq id no:28)的氨基酸残基1至37的区段具有至少70%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
137.(b-xi)”与来自scolmb(seq id no:33)的氨基酸残基1至37的区段具有至少70%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,或
138.(b-xii)”与来自scolmc(seq id no:34)的氨基酸残基1至37的区段具有至少70%、优选至少80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段;
139.或
140.(c-i)”与来自scole2(seq id no:1)的氨基酸残基43至313的区段具有至少85%、优选至少90%、更优选至少95%序列相似性的区段,
141.(c-ii)”与来自scole3(seq id no:2)的氨基酸残基35至315的区段具有至少85%、优选至少90%、更优选至少95%序列相似性的区段,
142.(c-iii)”与来自scole7(seq id no:3)的氨基酸残基43至316的区段具有至少90%、优选至少95%序列相似性的区段,
143.(c-iv)”与来自scole1a(seq id no:4)的氨基酸残基1至170的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段,
144.(c-v)”与来自scole1b(seq id no:5)的氨基酸残基1至195的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段,
145.(c-vii)”与来自scole1c(seq id no:25)的氨基酸残基6至194的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段,
146.(c-viii)”与来自scole1d(seq id no:26)的氨基酸残基6至194的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段,
147.(c-ix)”与来自scole1e(seq id no:27)的氨基酸残基5至192的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段,
148.(c-x)”与来自scolma(seq id no:28)的氨基酸残基1至37的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段,
149.(c-xi)”与来自scolmb(seq id no:33)的氨基酸残基1至37的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段,或
150.(c-xii)”与来自scolmc(seq id no:34)的氨基酸残基1至37的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段;
151.或
152.(d-i)”与来自scole2(seq id no:1)的氨基酸残基43至313的区段相比具有1至50个、优选1至40个、更优选1至30个、甚至更优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段,
153.(d-ii)”与来自scole3(seq id no:2)的氨基酸残基35至315的区段相比具有1至50个、优选1至40个、更优选1至30个、甚至更优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段,
154.(d-iii)”与来自scole7(seq id no:3)的氨基酸残基43至316的区段相比具有1至30个、优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段,
155.(d-iv)”与来自scole1a(seq id no:4)的氨基酸残基1至170的区段相比具有1至40个、优选1至30个、更优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段,
156.(d-v)”与来自scole1b(seq id no:5)的氨基酸残基1至195的区段相比具有1至40个、优选1至30个、更优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段,
157.(d-vii)”与来自scole1c(seq id no:25)的氨基酸残基6至194的区段相比具有1至40个、优选1至30个、更优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段,
158.(d-viii)”与来自scole1d(seq id no:26)的氨基酸残基6至194的区段相比具有1至40个、优选1至30个、更优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段,
159.(d-ix)”与来自scole1e(seq id no:27)的氨基酸残基5至192的区段相比具有1至40个、优选1至30个、更优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段,
160.(d-x)”与来自scolma(seq id no:28)的氨基酸残基1至37的区段相比具有1至7个、优选1至5个、更优选1至3个、最优选1至3个氨基酸取代、添加、插入或缺失的区段,
161.(d-xi)”与来自scolmb(seq id no:33)的氨基酸残基1至37的区段相比具有1至7个、优选1至5个、更优选1至3个、最优选1至3个氨基酸取代、添加、插入或缺失的区段,或
162.(d-xii)”与来自scolmc(seq id no:34)的氨基酸残基1至37的区段相比具有1至7个、优选1至5个、更优选1至3个、最优选1至3个氨基酸取代、添加、插入或缺失的区段。
163.(5)根据(1)至(4)中任一项所述的蛋白,其中所述蛋白如以下项中任一项所定义:
164.(b-x)、(c-x)、(d-x)、(b-xi)、(c-xi)、(d-xi)、(b-xii)、(c-xii)、(d-xii),
165.(b-x)’、(c-x)’、(d-x)’、(b-xi)’、(c-xi)’、(d-xi)’、(b-xii)’、(c-xii)’、(d-xii)’,
166.(b-x)”、(c-x)”、(d-x)”、(b-xi)”、(c-xi)”、(d-xi)”、(b-xii)”、(c-xii)’、或(d-xii)”;
167.并且
168.所述蛋白对应于seq id no:33的残基155的氨基酸残基是pro和/或对应于seq id no:33的残基246的氨基酸残基是arg或lys,优选arg。
169.(6)根据(5)所述的蛋白,其中对应于seq id no:33的残基76和84的氨基酸残基是gln。
170.(7)根据(1)至(6)中任一项所述的蛋白,其用于治疗沙门氏菌如肠道沙门氏菌,优选肠道沙门氏菌肠道亚种感染或污染的方法。
171.(8)根据(1)至(7)中任一项所述的蛋白,其中权利要求1的蛋白,特别是权利要求1中的(b)至(d)类的蛋白的针对肠道沙门氏菌的毒性是这样的:在点样5微升所述(b)至(d)类蛋白和seq id no:1的蛋白的溶液于软琼脂覆盖的平板上并随后在37℃下孵育琼脂平板12小时后,其与seq id no:1的蛋白产生相同直径的不含肠道沙门氏菌肠道亚种血清型纽波特(newport)菌株6962
tm
*的活菌的菌斑,所述软琼脂覆盖的平板用0.14ml每cm
2 1
×
107cfu/ml的敏感的肠道沙门氏菌菌株的细菌溶液接种,其中(b)至(d)类蛋白的浓度是seq id no:1的蛋白的相当溶液的至多5倍。
172.(9)一种蛋白,其包含以下氨基酸序列中的任何一种或由其组成:
173.(a-x)seq id no:28,
174.(a-xi)seq id no:33,或
175.(a-xii)seq id no:34;
176.或
177.(b-x)与seq id no:28的氨基酸序列具有至少70%序列同一性的氨基酸序列,
178.(b-xi)与seq id no:33的氨基酸序列具有至少70%序列同一性的氨基酸序列,或
179.(b-xii)与seq id no:34的氨基酸序列具有至少70%序列同一性的氨基酸序列;
180.或
181.(c-x)与seq id no:28的氨基酸序列具有至少80%序列相似性的氨基酸序列,
182.(c-xi)与seq id no:33的氨基酸序列具有至少80%序列相似性的氨基酸序列,或
183.(c-xii)与seq id no:34的氨基酸序列具有至少80%序列相似性的氨基酸序列;
184.或
185.(d-x)与seq id no:28的氨基酸序列相比具有1至40个氨基酸取代、添加、插入或缺失的氨基酸序列,
186.(d-xi)与seq id no:33的氨基酸序列相比具有1至40个氨基酸取代、添加、插入或缺失的氨基酸序列,或
187.(d-xii)与seq id no:34的氨基酸序列相比具有1至40个氨基酸取代、添加、插入或缺失的氨基酸序列;
188.或
189.(e-x)包含seq id no:28的至少215个连续氨基酸残基或由其组成的氨基酸序列,
190.(e-xi)包含seq id no:33的至少215个连续氨基酸残基或由其组成的氨基酸序列,或
191.(e-xii)包含seq id no:34的至少215个连续氨基酸残基或由其组成的氨基酸序列。
192.(10)根据(9)所述的蛋白,其中所述蛋白如(b-x)、(c-x)、(d-x)或(e-x)中所定义的,优选如(b-xi)、(c-xi)、(d-xi)、(e-xi)、(b-xii)、(c-xii)、(d-xii)或(e-xii)中所定义的,并且,(所述蛋白的)对应于seq id no:33的残基155的氨基酸残基是pro,和/或对应于seq id no:33的残基246的氨基酸残基是arg或lys,优选arg。
193.(11)根据(9)或(10)所述的蛋白,其中对应于seq id no:33的残基76和84的氨基酸残基是gln。
194.(12)一种组合物,其包含(1)至(11)中任一项所定义的一种或多种蛋白。
195.(13)根据(12)所述的组合物,其中所述一种或多种蛋白是或包含scole1c、scole1d、scole1e、scolma、scolmb或scolmc,或scole1c、scole1d、scole1e、scolma、scolmb或scolmc的衍生物,所述衍生物如第(1)项的(b)、(c)和(d)项中所定义,或优选地,如第(9)项的(b)、(c)和(d)项中所定义。
196.(14)根据(12)或(13)所述的组合物,其包含选自如(1)、(2)或(3)所定义的至少两个不同的类别(i)至(x)、(xi)和(xii)的两种或更多种蛋白,优选选自如(1)、(2)或(3)所定义的至少两个不同的类别(i)至(xii)的两种或更多种蛋白,甚至更优选选自如(1)至(4)中任一项所定义的至少两个不同的类别(iv)、(v)和(x)至(xii)的两种或更多种蛋白。
197.(15)根据(14)所述的组合物,其至少包含((a)至(d)类中任一种的)(x)亚类的蛋白和((a)至(d)类中任一种的)(v)亚类的蛋白;或至少包含((a)至(d)类中任一种的)(xi)
亚类的蛋白和((a)至(d)类中任一种的)(v)亚类的蛋白;或至少包含((a)至(d)类中任一种的)(xii)亚类的蛋白和((a)至(d)类中任一种的)(v)亚类的蛋白。
198.(16)根据(12)至(15)中任一项所述的组合物,其用于治疗沙门氏菌,优选肠道沙门氏菌,更优选肠道沙门氏菌肠道亚种感染的方法。
199.(17)根据(12)至(16)中任一项所述的组合物,其中所述组合物是植物材料或其提取物,其中所述植物材料是来自具有表达的所述蛋白的植物、优选具有表达的所述蛋白的可食用植物的材料。
200.(18)根据(17)所述的组合物,其中所述植物材料是选自菠菜、叶甜菜(chard)、甜菜根(beetroot)、胡萝卜、糖用甜菜(sugar beet)、多叶甜菜(leafy beet)、苋菜、烟草的植物的材料,和/或所述植物材料是一种或多种叶、根、块茎或种子,或所述叶、根、块茎或种子的压碎、磨碎或粉碎的产物。
201.(19)根据(12)至(18)中任一项所述的组合物,其中所述组合物是含有所述蛋白的水溶液。
202.(20)根据(19)所述的组合物,其中所述水溶液中的所述蛋白的浓度,或如果组合物含有两种或更多种不同的蛋白时,所述两种或更多种蛋白的浓度为0.0001至1mg/ml,优选0.001至0.1mg/ml,更优选0.005至0.05mg/ml;或0.1至15mg/kg食物,优选0.5至10mg/kg食物,更优选0.1至5mg/kg食物。
203.(21)根据(12)至(20)中任一项所述的组合物,其包含根据(a-iv)、(b-iv)、(c-iv)、(d-iv)或(e-iv)项的蛋白,和/或根据(a-x)、(b-x)、(c-x)、(d-x)或(e-x)项的蛋白;或包含根据(a-v)、(b-v)、(d-v)或(e-v)项的蛋白,和/或根据(a-x)、(b-x)、(d-x)或(e-x)项的蛋白;其中本文定义的优选实施方案可以与该第(21)项中定义的实施方案组合。
204.(22)一种预防或减少沙门氏菌对目标物感染或污染的方法,其包括将所述目标物与(1)至(11)中任一项所定义的蛋白或(12)至(21)中任一项所定义的组合物接触。
205.(23)根据(22)所述的方法,其中用所述水溶液喷洒所述目标物或将所述目标物浸入到所述水溶液中。
206.(24)根据(22)至(23)所述的方法,其中将所述目标物浸入所述蛋白的水溶液中至少10秒钟,优选至少1分钟,优选至少5分钟。
207.(25)根据(22)至(24)中任一项所述的方法,其中所述目标物为食品或动物饲料。
208.(26)根据(25)所述的方法,其中所述食品是完整的动物躯体、肉、蛋、生的水果或蔬菜,优选所述食品是肉、生的水果或蔬菜,更优选所述食品是肉。
209.(27)一种治疗有此需要的受试者的沙门氏菌感染的方法,其包括将(1)至(11)中任一项所定义的蛋白或(12)至(21)中任一项所定义的组合物施用于所述受试者。
210.(28)根据(22)至(27)任一项所述的方法,其中所述沙门氏菌是肠道沙门氏菌,优选肠道沙门氏菌肠道亚种。
211.(29)一种生产包含(1)至(11)中任一项所定义的蛋白的组合物的方法,所述方法包括以下步骤:
212.(i)在植物、优选可食用植物或烟草(nicotiana)中表达所述蛋白,
213.(ii)从所述植物中收获含有表达的蛋白的植物材料,
214.(iii)用含水缓冲液从所述植物材料中提取所述蛋白,以获得含有所述蛋白的组
合物,
215.(iv)任选地从所述组合物中除去不需要的污染物。
216.(30)根据(1)或(9)所述的蛋白,其中所述蛋白是以下项的蛋白:
[0217]-(a-vii)、(b-vii)、(c-vii)或(d-vii),每一项任选地与第(3)项组合,或
[0218]-(a-vii)、(b-vii)、(c-vii)、(d-vii)或(e-vii);
[0219]
或
[0220]
其中所述蛋白是以下项的蛋白:
[0221]-(a-viii)、(b-viii)、(c-viii)或(d-viii),每一项任选地与第(3)项组合,或
[0222]-(a-viii)、(b-viii)、(c-viii)、(d-viii)或(e-viii);
[0223]
或
[0224]
其中所述蛋白是以下项的蛋白:
[0225]-(a-x)、(b-x)、(c-x)或(d-x),每一项任选地与第(3)项组合,或
[0226]-(a-x)、(b-x)、(c-x)、(d-x)或(e-x);
[0227]
或
[0228]
其中所述蛋白是以下项的蛋白:
[0229]-(a-xi)、(b-xi)、(c-xi)或(d-xi),每一项任选地与第(3)项组合,或
[0230]-(a-xi)、(b-xi)、(c-xi)、(d-xi)或(e-xi);
[0231]
或
[0232]
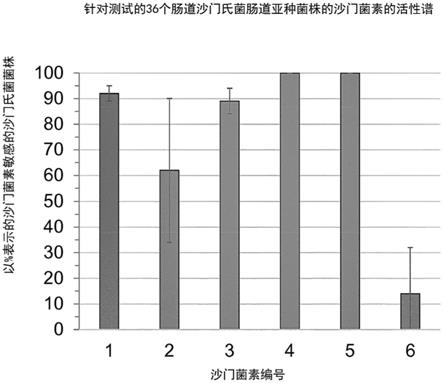
其中所述蛋白是以下项的蛋白:
[0233]-(a-xii)、(b-xii)、(c-xii)或(d-xii),每一项任选地与第(3)项组合,或
[0234]-(a-xii)、(b-xii)、(c-xii)、(d-xii)或(e-xii)。
[0235]
(31)根据(12)所述的组合物,其包含:
[0236]-根据(a-x)、(b-x)、(c-x)、(d-x)或(e-x)项;和/或根据(a-xi)、(b-xi)、(c-xi)、(d-xi)或(e-xi)项;和/或(a-xii)、(b-xii)、(c-xii)、(d-xii)或(e-xii)项所述的蛋白,任选地如权利要求7或8所进一步定义的;和
[0237]-根据(a-viii)、(b-viii)、(c-viii)、(d-viii)或(e-iii)项所述的蛋白。
[0238]
沙门氏菌细菌素,在此与其衍生物一起称为"沙门菌素"(本文中缩写为“scol”或“sal”),是由某些沙门氏菌菌株产生的天然非抗生素抗微生物蛋白,其杀死其它沙门氏菌菌株或抑制其生长。与相对充分研究的称为大肠菌素的大肠杆菌(escherichia coli)蛋白类似物不同,沙门菌素几乎没有引起注意。在公众可获得的基因组数据库中有许多与大肠菌素序列相似的沙门氏菌序列,它们中的大多数显示出与大肠菌素m、ia、ib、5和10的高度同一性。本发明人已经鉴定了与大肠菌素相似但不同的沙门菌素,其可用于预防或减少沙门氏菌,特别是肠道沙门氏菌肠道亚种的感染或污染。
[0239]
本发明人发现沙门菌素都可以在植物中有效表达。本研究中所用的表达方法已经达到gmp合规水平,并且目前在不同的临床试验中用作制造方法。大多数沙门菌素以高产率表达(高达1.7g活性蛋白/千克新鲜绿色生物质),这意味着低的商业可行的制造成本。尤其可以使用烟草和可食用植物如叶甜菜或菠菜来进行生产。在不同的沙门菌素中,沙门菌素ma(scolma)、mb(scolmb)和mc(scolmc)是优选的,并且这些m型沙门菌素与e1a(scole1a)和(更优选的)e1b(scole1b)或其衍生物的组合物是更优选的,因为它们被发现具有针对主要
致病性沙门氏菌菌株的非常广泛的抗微生物活性。这两种沙门菌素scole1a和scole1b中的每一种还显示出针对所有36种测试的主要致病菌株的非常高的活性。而且,scolma显示出针对主要致病性沙门氏菌菌株具有广泛的抗微生物活性(参见图32)。有趣的是,scolma针对scole1沙门菌素对其活性较低的几种致病性沙门氏菌菌株具有高抗微生物活性,使scolma以及scolmb和scolmc成为在本发明的组合物中与scole1沙门菌素组合使用的理想沙门菌素,并因此可用于本发明的方法中。scolmb和scolmc针对沙门氏菌菌株的抗微生物活性谱与scolma非常相似(参见图37)。因此,对于本文所述的用途和方法,scolmb和scolmc具有与scolma相同或相似的效用。就易于纯化而言,在scolm沙门菌素中,scolmb和scolmc(及其如本文定义的衍生物)是优选的,并且scolmc(及其如本文定义的衍生物)是最优选的。scolmc(及其衍生物)组合了针对沙门氏菌菌株的广泛抗微生物活性谱与理想的处理性能以及高抗微生物活性。
[0240]
在所使用的测定中,用少量大肠菌素(例如,每kg处理的食品产品少于10mg大肠菌素)的处理降低不同致病菌株的细菌负荷3至>6个log。在使用加有2至4种病原体血清型的禽肉的加标(spike)实验中,大肠菌素(大肠菌素m、ia和5)有效地降低了致病菌的滴度。因此,预期到上文提到的对肠道沙门氏菌肠道亚种血清型具有较提到的大肠菌素高的抗微生物活性的沙门菌素能更有效地降低沙门氏菌对家禽污染的滴度。
[0241]
本发明的实验数据证明,非抗生素抗菌沙门菌素可以在植物如本氏烟草(nicotiana benthamiana)(目前正在进行临床试验的多种生物药物的标准生产宿主)中以非常高的水平表达,并且植物表达的蛋白显然具有完全活性。在大多数情况下,表达水平在没有工艺优化的情况下达到总可溶性蛋白的37%或1.74g/kg新鲜叶生物量,这意味着沙门菌素对植物无毒性,并且可以开发廉价的转基因宿主中的优化的转染或诱导工业程序。相反,在细菌宿主中高水平表达细菌素蛋白的尝试通常遇到这种细菌素类的一般毒性,甚至在同源细菌以外的物种中也是如此(例如,medina等人,plos one,2011;6(8):e23055;diaz等人,1994)。因此,植物是产生沙门菌素的极佳宿主。
[0242]
本发明的数据证明在实际暴露(exposure)建模下,沙门菌素可有效控制肠道沙门氏菌肠道亚种的大多数或所有的主要致病血清型。沙门氏菌产生的沙门菌素种类有限。所研究的沙门菌素在一般的三结构域(易位、受体和细胞毒性结构域)结构内具有相当多样的结构,类似于更多研究的大肠杆菌大肠菌素。令人惊奇的是,实际上所有测试的沙门菌素和大肠菌素,单独或与抗毒素(免疫蛋白)一起,在植物中都非常好地表达。这可以解释为沙门菌素和大肠菌素对植物细胞的低毒性,以及这些细菌素蛋白(是"固有无序蛋白"(在细菌细胞壁和膜易位过程中解折叠/重折叠的能力所必需的特征)的经典代表)可能不对植物细胞的翻译和翻译后机制施加不寻常的要求的事实。
[0243]
与fda根据对由大肠杆菌引起的食物中毒的历史分析所定义的主要大肠杆菌菌株的列表不同,管理机构尚未定义主要食物源沙门氏菌菌株的列表,这主要是由于引起爆发的致病变型的较高多样性。面对现有技术中缺乏指导,本发明人决定将三种现有的主要研究集中起来,这些研究基于致病变型的流行和中毒严重性对致病变型进行分级。在我们的研究中,选择在2003-2012年向疾病控制中心报道的(national entitical disease surveillance:salmonella annual report,2013(cdc,2016年6月):laboratory-confirmed human salmonella infections(us)reported至cdc 2003-2012)36种血清型进
行分析,其中29种引起至少100次发病,其中有17种最著名的致病变型在cdc的前20个名单上;该数目比fda定义的大肠杆菌致病株数高5倍(7)。
[0244]
本文提供的数据表明,基于它们控制主要致病性沙门氏菌菌株的能力,可以将这5种不同的沙门氏菌的沙门菌素scole1a、scole1b、scole2、scole3和scole7分成3组。沙门氏菌沙门菌素e1a和e1b被证明具有普遍活性,均能够杀死所有测试的致病变型并显示出最高的平均活性。两种沙门菌素在所有测试菌株上的平均活性超过107au/μg。例如,沙门菌素e1a的个体活性是:36个菌株中有35个菌株>103au/μg,36个菌株中有24个菌株>104au/μg,36个菌株中有13个菌株>106au/μg。剩余的沙门菌素分成两组,其中沙门菌素e2和e7抑制超过80%的菌株,但具有低100倍的平均活性(低于105au/μg),而沙门菌素e3以较低的平均活性(约102au/μg)抑制大约60%的菌株。本发明人还发现,沙门菌素scole1c、scole1d、scole1e和scolma表现出显著的抗微生物活性。发现scole1b和scole1d在处理性能方面出奇地更好,尤其是在纯化方面。令人惊讶地发现,scole1d作为溶液(特别是当在低于室温下例如在0
°
至10℃,优选在3
°
至7℃之间,或在约4℃下时冷却的水溶液)具有优异的储存稳定性。在scolm沙门菌素中,发现scolmb和scolmc,特别是scolmc,在处理性能方面出奇地更好,特别是在纯化方面,并且针对沙门氏菌物种的抗微生物活性高。
[0245]
这些结果是出乎意料的,因为大肠菌素(由大肠杆菌细胞产生的沙门菌素类似物)针对七种大肠杆菌致病变型显示出非常窄的抗微生物活性谱,并且二至五种大肠菌素的混合物必须优选用于有效地抑制由fda定义的所有七种stec血清型。大肠菌素还表现出针对"大7"(big seven)stec菌株的低得多的平均活性(平均值<103au/μg),尽管在菌株h104:h4(>105au/μg)(2011年其在欧洲引起重大爆发)和普通的实验室菌株上观察到高得多的活性。
[0246]
本发明人分别对沙门菌素和大肠菌素对大肠杆菌和沙门氏菌的交叉特异活性的分析表明,针对不同属/种的细菌的低的活性。特别地,沙门菌素针对"大七"stec菌株的活性是低(小于102au/μg)的,尽管一些沙门菌素(例如e2、e7和e1b,但令人惊讶地不是e1a)对h104:h4(103au/μg)和实验室菌株dh10b(105au/μg)具有相当的活性。类似地,发现大肠菌素对沙门氏菌致病变型的活性低,其中大肠菌素ia和ib对超过80%的菌株具有活性,但仅大肠菌素ia的平均活性高于3
×
103au/μg(或比沙门菌素e1a/b低3-4个数量级)。本发明人从这些研究中得出结论,为了对抗两种致病物种,必须使用大肠菌素和沙门菌素的混合物。这些结果也与最近对大肠菌素样蛋白在不同属细菌之间竞争的生态学功效的研究似乎部分不一致(nedialkova等人,plos pathog.2014jan;10(1):e1003844)。
[0247]
本发明提供了用于控制沙门氏菌的新的试剂和组合物。本发明的沙门菌素具有可以以简单的方式获得销售授权的优点。例如,fda最近批准了植物产生的大肠菌素gras(通常认为是安全的)状态(grn573,fda网站)。由于对用于沙门氏菌控制的天然非抗生素抗菌剂的需求未得到满足,本发明人设想开发沙门氏菌细菌素("沙门菌素")。因此,完成了本发明。
附图说明
[0248]
图1示意性显示了在实施例中使用的用于表达沙门菌素及对应的免疫蛋白的病毒载体。表达沙门菌素的构建体基于烟草花叶病毒(tmv),而表达免疫蛋白的构建体则基于马
铃薯病毒x(pvx)。
[0249]
沙门菌素表达载体包括pnmd28161、pnmd28151和pnmd28172,分别用于表达沙门菌素scole2、scole3和scole7(图1a),包括pnmd28191、pnmd28204和pnmd28182,分别用于表达沙门菌素scole1a、scole1b和spst(图1b)。
[0250]
rb和lb表示双元载体的t-dna的右和左边界。pact2:拟南芥(arabidopsis)肌动蛋白2基因的启动子;o:来自tvcv(芜菁脉明病毒)(turnip vein clearing virus)的5'末端;rdrp:来自cr-tmv(感染十字花科植物的烟草花叶病毒)的rna依赖性rna聚合酶开放阅读框(orf);mp:来自cr-tmv的移动蛋白orf;scole2:沙门菌素scole2编码序列;scole3:沙门菌素scole3编码序列;scole7:沙门菌素scole7编码序列;scole1a:沙门菌素scole1a编码序列;scole1b:沙门菌素scole1b编码序列;spst:沙门菌素spst编码序列;n:来自cr-tmv的3'-非翻译区;t:农杆菌(agrobacterium)胭脂碱合酶终止子;在rdrp和mp orf中中断灰色区段的白色区段表示插入这些orf中的内含子,用于增加在植物细胞的细胞质中形成rna复制子的可能性,其详细描述于wo2005049839中。为了防止这些蛋白对用于质粒克隆的大肠杆菌细胞的细胞毒性作用,还将内含子插入到scole2、scole3和scole7 orf中。
[0251]
用于表达免疫蛋白的基于pvx的载体包括pnmd28222和pnmd28232,分别用于表达沙门菌素scole2和scole7免疫蛋白(图1a)。p35s:花椰菜花叶病毒35s启动子;pvx-pol:来自pvx的rna依赖性rna聚合酶;cp:外壳蛋白orf;25k、12k和8k一起表示来自pvx的25kda、12kda和8kda的三基因块模块(block module);n:来自pvx的3'-非翻译区。simme2和simme7分别代表沙门菌素scole2和scole7免疫蛋白的编码序列。
[0252]
图2显示了在用携带病毒载体的农杆菌浸润本氏烟草(nicotiana benthamiana)植物后,沙门菌素表达的比较sds-page分析。用5倍体积的缓冲液提取植物叶材料,所述缓冲液含有50mm hepes(ph7.0)、10mm乙酸钾、5mm乙酸镁、10%(v/v)甘油、0.05%(v/v)tween-20和300mm nacl。将蛋白提取物溶解在12%聚丙烯酰胺凝胶中。对于凝胶上样,使用含有相当于0.4mg植物组织鲜重的提取物体积的等分试样。在凝胶上样之前,将等分试样的蛋白提取物与2
×
laemmli缓冲液以1:1的比例混合,并在95℃下孵育10分钟。凝胶泳道上的数字代表来自表达下列重组蛋白的植物组织的蛋白提取物:1-沙门菌素scole2;2-沙门菌素scole3;3-沙门菌素scole7;4-沙门菌素scole1a;5-沙门菌素scole1b;6-沙门菌素spst。数字7对应于用作负对照的未感染叶组织的提取物。l-pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,美国),#sm0671)。箭头表示对应于表达的重组大肠菌素的特定的蛋白条带。
[0253]
图3显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素的植物提取物的特定的抗微生物活性的半定量评价。使用径向扩散测定通过斑点法(spot-on-lawn-method)检测抗微生物活性。沙门菌素敏感的沙门氏菌株的百分比(3个独立实验的平均值)是针对沙门菌素给出的:1-沙门菌素scole2;2-沙门菌素scole3;3-沙门菌素scole7;4-沙门菌素scole1a;5-沙门菌素scole1b;6-沙门菌素spst。
[0254]
图4显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素的植物提取物的平均抗微生物活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每mg表达重组沙门菌素的植物生物量鲜重(fw)的任意单位(au)计算(3个独立实验的平均值)。因此它反映了每单位生物量的特定活性剂的产率,即宿主的特定生产能
力。任意单位被计算为在径向扩散测定中引起可检测的清除效应的蛋白提取物的最高稀释度的稀释因子。所测试的重组沙门菌素被给出为:1
–
沙门菌素scole2;2-沙门菌素scole3;3-沙门菌素scole7;4-沙门菌素scole1a;5-沙门菌素scole1b;6-沙门菌素spst。
[0255]
图5显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每μg重组沙门菌素的任意单位(au)计算(3个独立实验的平均值),其反映沙门菌素针对特定菌株的比活性;即评价了沙门菌素的特定的抗微生物效力。所测试的重组沙门菌素被给出为:1-沙门菌素scole2;2-沙门菌素scole3;3-沙门菌素scole7;4-沙门菌素scole1a;5-沙门菌素scole1b;6-沙门菌素spst。
[0256]
图6显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole2的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每mg fw植物生物量的任意单位(au)计算(3个独立实验的平均值)。
[0257]
图7显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole2的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每μg重组沙门菌素的任意单位(au)计算(3个独立实验的平均值)。
[0258]
图8显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole3的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每mg fw植物生物量的任意单位(au)计算(3个独立实验的平均值)。
[0259]
图9显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole3的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每μg重组沙门菌素的任意单位(au)计算(3个独立实验的平均值)。
[0260]
图10显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole7的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每mg fw植物生物量的任意单位(au)计算(3个独立实验的平均值)。
[0261]
图11显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole7的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每μg重组沙门菌素的任意单位(au)计算(3个独立实验的平均值)。
[0262]
图12显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole1a的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每mg fw植物生物量的任意单位(au)计算(3个独立实验的平均值)。
[0263]
图13显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole7的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每μg重组沙门菌素的任意单位(au)计算(3个独立实验的平均值)。
[0264]
图14显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole1b的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑
点法检测抗微生物活性并以每mg fw植物生物量的任意单位(au)计算(3个独立实验的平均值)。
[0265]
图15显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含沙门菌素scole1b的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每μg重组沙门菌素的任意单位(au)计算(3个独立实验的平均值)。
[0266]
图16示意性地显示了用于表达实施例中使用的大肠菌素的基于烟草花叶病毒(tmv)的病毒载体。大肠菌素表达载体包括pnmd25856、pnmd15311、pnmd25848、pnmd19141、pnmd25861和pnmd102221,分别用于表达大肠菌素cols4、col5、col10、colla、collb和colm。
[0267]
rb和lb表示双元载体的t-dna的右和左边界。pact2:拟南芥肌动蛋白2基因的启动子;o:来自tvcv(芜菁脉明病毒)的5'末端;rdrp:来自cr-tmv(感染十字花科植物的烟草花叶病毒)的rna依赖性rna聚合酶开放阅读框(orf);mp:来自cr-tmv的移动蛋白orf;cols4:大肠菌素s4编码序列;cols5:大肠菌素5编码序列;cols10:大肠菌素10编码序列;colia:大肠菌素ia编码序列;colib:大肠菌素ib编码序列;colm:大肠菌素m编码序列;n:来自cr-tmv的3'-非翻译区;t:农杆菌胭脂碱合酶终止子;在rdrp和mp orf中中断灰色区段的白色区段表示插入这些orf中的内含子,用于增加在植物细胞的细胞质中形成rna复制子的可能性,其详细描述于wo2005049839中。
[0268]
图17显示了针对列于表5a和5b中的35个肠道沙门氏菌肠道亚种菌株(no.1至35)的含沙门菌素的植物提取物的平均抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性。给出了大肠菌素的大肠菌素敏感性沙门氏菌菌株的百分比(3个独立实验的平均值):1-大肠菌素s4;2-大肠菌素5;3-大肠菌素10;4-大肠菌素ia;5-大肠菌素ib;6-大肠菌素m。
[0269]
图18显示了在用包含大肠菌素m、大肠菌素ia和大肠菌素5的三组分大肠菌素混合物处理的污染的鸡胸肉片中的肠道沙门氏菌肠道亚种细胞群的减少。分别用血清型鼠伤寒和肠炎的肠道沙门氏菌肠道亚种菌株14028
tm
*和13076
tm
*的双菌株混合物污染肉。星号表明细菌数量统计学上的显著差异。
[0270]
图19显示了用包含大肠菌素m、大肠菌素ia和大肠菌素5的三组分大肠菌素混合物处理的污染的鸡胸肉片中肠道沙门氏菌肠道亚种细胞群的减少。分别用血清型鼠伤寒、肠炎、鸭沙门氏菌(anatum)和纽波特的肠道沙门氏菌肠道亚种菌株14028
tm
*、13076
tm
*、9270
tm
*和6962
tm
*的四菌株混合物污染肉。星号表明细菌数量统计学上的显著差异。
[0271]
图20a-c显示了使用clustal omega工具(fast,scalable generation of high-quality protein multiple sequence alignments using clustal omega(2011年10月11日)molecular systems biology 7:539)产生的沙门菌素氨基酸序列的多序列比对。scole2、scole3、scole7、scole1a、scole1b和spst分别指seq id no:1-6。颜色标记表示氨基酸残基的性质(红色残基为avfpmilw(小的、疏水的和芳香族-y);蓝色残基为de(酸性);品红色残基为rk(碱性)和绿色残基为styhcngq(羟基巯基胺和g))。共有符号*(星号)表示比对中的位置,其具有单个完全保守的残基,:(冒号)表示在具有强相似性质的组之间的保守性和.(句号)表示弱相似性质的组之间的保守性。
[0272]
图21是用于稳定植物转化的编码scole1b的质粒构建体(pnmd35541)的t-dna区的示意图。t-dna区由4个表达盒组成,所述的4个表达盒用于:1)卡那霉素抗性转基因植物选择标记的组成型表达,2)alcr转录激活因子的组成型表达,3)沙门菌素scole1b的乙醇诱导型表达,和4)tmv mp的乙醇诱导型表达。箭头指示表达盒的方向。为了严格控制非诱导状态的病毒复制子活化,将病毒载体在2个组分(复制子和mp(表达盒3和4))中解构(werner等.proc.natl.acad.sci.usa 108,14061-14066(2011)。lb和rb,分别为双元载体的左和右边界;tnos和pnos,农杆菌胭脂碱合酶基因的终止子和启动子;nptii,新霉素磷酸转移酶ii;pstls,马铃薯st-ls1基因的启动子;alcr,构巢曲霉(aspergillus nidulans)alcr orf;tact2,拟南芥肌动蛋白2终止子;t35s,camv35s终止子;3
′
tmv,tmv的3
′
非翻译区;rdrp,rna依赖性rna聚合酶;paica,scole1b,沙门菌素e1b的编码序列(scole1b);构巢曲霉醇脱氢酶(aica)启动子;mp,运动蛋白;农杆菌章鱼碱合酶基因的tocs终止子;[]表达盒3中mp的缺失。
[0273]
图22显示了在稳定的转基因本氏烟草植物中沙门菌素scole1b的诱导型表达的结果。粗提物上样相当于用2
×
laemmli缓冲液从(泳道1、3、5、7)未诱导的植物材料或(泳道2、4、6、8)用乙醇诱导的植物材料4dp中提取的3mg fw。(泳道1、2)本氏烟草野生型(wt)植物,(泳道3、4),(泳道5、6),(泳道7、8)用于t0代的单拷贝t-dna插入的候选的不同转基因植物(scole1b#4、12、37)。箭头标记重组蛋白。
[0274]
图23显示了在菠菜(spinacia oleracea cv)中瞬时表达沙门菌素的结果。用携带tmv或tmv和pvx载体的农杆菌注射器浸润牛肝菌。tsp提取物上样相当于用5倍体积150mm nacl提取的3mg fw植物材料。(a)5dpi(浸润后天数)收获scole1b的植物材料,6dpi收获scole3、scole7和scole1a的植物材料,或7dpi收获scole2的植物材料或(b)4dpi收获scole1b的植物材料,5dpi收获scole3、scole7和scole1a的植物材料,6dpi收获scole2的植物材料或(d)8dpi收获scole2、scole3、scole7、scole1a和scole1b的植物材料。从表达scole2(泳道1)、scole3(泳道2)、scole7(泳道3)、scole1a(泳道4)和scole1b(泳道5)的植物材料或从(wt)未转染的叶组织制备(a,b,d)分析的提取物。scole2和scole7与它们各自的免疫蛋白共表达。箭头标记重组蛋白。
[0275]
图24显示了针对肠道沙门氏菌肠道亚种和大肠杆菌大七stec血清型的沙门氏菌和大肠杆菌的细菌素活性谱。通过径向扩散测定经斑点法对针对表9中列出的36个(a、e)或35个(b、f)肠道沙门氏菌肠道亚种菌株或表10中列出的7个大肠杆菌大七stec菌株(c、d、g、h)的含有(a、c、e、g)沙门菌素和(b、d、f、h)大肠菌素的植物提取物的抗微生物比活性的半定量评价。对于细菌素敏感菌株(e、f、g、h)的百分比和以每μg重组蛋白任意单位(au)(a,b,c,d)计算的所有测试菌株的细菌素比活性,分别在(a、b、e、f)和(c、d、g、h)中给出了n=3和n=2独立实验的平均值和stdv。1-scole2(a、c、e、g)或cols4(b、d、f、h);2-scole3(a、c、e、g)或col5(b、d、f、h);3-scole7(a、c、e、g)或col10(b、d、f、h);4-scole1a(a、c、e、g)或colia(b、d、f、h);5-scole1b(a、c、e、g)或colib(b、d、f、h);6-colm(b、d、f、h)。
[0276]
图25显示了沙门菌素减少肠道沙门氏菌肠道亚种对新鲜鸡胸肉片的污染。(a)通过喷洒施用沙门菌素处理污染的肉后,从在10℃下不同时间储存后从肉中回收的细菌群(0h黑色条,初始污染水平;白色条,载体处理;在1小时、24小时、48小时和72小时:浅灰色条,在浓度为3mg/kg肉的细菌素处理scole1a;灰色条,在浓度为3+1+1+1mg/kg肉的细菌素
处理scole1a+scole1b+scole2+scole7;深灰色条,在浓度为0.3+0.1+0.1+0.1mg/kg肉中的细菌素处理scole1a+scole1b+scole2+scole7)。误差条表示生物学重复试验的标准偏差,n=4。(b)(a)中使用的鸡胸肉修整物。
[0277]
图26显示了使用clustal omega工具产生的沙门菌素氨基酸序列的多序列比对。scole1a、scole1c、scole1d、scole1e分别指seq id no:4、25、26和27。共有符号*(星号)表示比对中的位置,其具有单个完全保守的残基,:(冒号)表示在具有强相似性质的组之间的保守性,.(句号)表示弱相似性质的组之间的保守性。
[0278]
图27显示了使用clustal omega工具产生的沙门菌素scolma和大肠菌素colm氨基酸序列的多序列比对。scolma和colm分别指seq id no:28和14。
[0279]
图28示意性显示了分别用于表达沙门菌素scole1c、scole1d、scole1e和scolma的病毒载体pnmd47710、pnmd47720、pnmd48260和pnmd47730。这些构建体基于烟草花叶病毒(tmv)。
[0280]
scole1c:沙门菌素scole1c编码序列;scole1d:沙门菌素scole1d编码序列;scole1e:沙门菌素scole1e编码序列;scolma:沙门菌素scolma编码序列。
[0281]
图29显示了在用携带病毒载体的农杆菌浸润本氏烟草植物后,沙门菌素表达的比较sds-page分析。用5倍体积的缓冲液提取植物叶材料,所述缓冲液含有50mm hepes(ph 7.0)、10mm乙酸钾、5mm乙酸镁、10%(v/v)甘油、0.05%(v/v)tween-20和300mm nacl。将蛋白提取物溶解在12%聚丙烯酰胺凝胶中。对于凝胶上样,使用含有相当于0.4mg植物组织鲜重的提取物体积的等分试样。在凝胶上样之前,将等分试样的蛋白提取物与2
×
laemmli缓冲液以1:1的比例混合,并在95℃下孵育10分钟。凝胶泳道上的数字代表来自表达下列重组蛋白的植物组织的蛋白提取物:1-沙门菌素scole1c;2-沙门菌素scole1d;3-沙门菌素scole1e;4-沙门菌素scolma;5-沙门菌素scole1b;6-沙门菌素scole1a,7-大肠菌素m。数字8对应于用作阴性对照的未感染叶组织的提取物。l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671)。箭头表示对应于表达的重组大肠菌素的特定的蛋白条带。
[0282]
图30显示了用于稳定植物转化的编码scole1d和scolma的质粒构建体(分别为pnmd49621和pnmd49632)的t-dna区的示意图。t-dna区由4个表达盒组成,所述的4个表达盒用于:1)卡那霉素抗性转基因植物选择标记的组成型表达,2)alcr转录激活因子的组成型表达,3)沙门菌素scoie1b的乙醇诱导型表达,和4)tmv mp的乙醇诱导型表达。箭头指示表达盒的方向。scole1d代表沙门菌素e1d(scole1d)的编码序列;scolma代表沙门菌素ma(scolma)的编码序列。详情请参见图21的说明。
[0283]
图31显示了在t0代的稳定转基因本氏烟草植物中沙门菌素scole1d的诱导型表达结果。粗提物上样相当于用2x laemmli缓冲液从本氏烟草野生型(wt)植物和用乙醇诱导4天后的用pnmd49621构建体转化的单独t0植物(泳道29、88、101、151、152、153、154、155、156、157、158和159)中提取的3mg fw。箭头标记重组蛋白。
[0284]
图32显示了针对列于表5a和5b中的36个肠道沙门氏菌肠道亚种菌株的含scole1b、scole1d和scolma的植物提取物的抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性并以每μg重组沙门菌素的任意单位(au)计算(3个独立实验的平均值)。
[0285]
图33显示了对沙门菌素scole1a、scole1b和scole1d在其以冻干的纯化蛋白和溶液形式储存时的抗微生物活性的评价。通过径向扩散测定来评价沙门菌素针对敏感的鼠伤寒沙门氏菌菌株atcc14028的活性。抗微生物活性表示为每mg重组蛋白的比活性单位(au)。使用bradford测定法测定沙门菌素浓度。(a)沙门菌素scole1a在以冻干干粉和溶液形式在4℃和室温(rt,20-20℃)下储存时的活性。(b)沙门菌素scole1b(批次3)在以冻干干粉和溶液形式在4℃和室温(rt,20-20℃)下储存时的活性。(c)沙门菌素(批次2)在以冻干干粉和溶液形式在4℃和室温(rt,20-20℃)下储存时的活性。
[0286]
图34显示了使用clustal omega(1.2.4)在线工具(https://www.ebi.ac.uk/tools/msa/clustalo/)产生的沙门菌素氨基酸序列的多序列比对。scolma、scolmb和scolmc分别指seq id no:28、33和34。共有符号*(星号)表示比对中的位置,其具有单个完全保守的残基,:(冒号)表示在具有强相似性质的组之间的保守性,.(句号)表示弱相似性质的组之间的保守性。
[0287]
图35示意性显示了分别用于表达沙门菌素scolmb和scolmc的病毒载体pnmd51280和pnmd51290。这些构建体基于烟草花叶病毒(tmv)。scolmb:沙门菌素scolmb编码序列;scolmc:沙门菌素scolmc编码序列。
[0288]
图36显示了在用携带病毒载体的农杆菌浸润本氏烟草叶后,沙门菌素scolmb和scolmc表达的sds-page分析。在浸润后(dpi)4、5、6和7天收获植物叶材料。用5倍体积的缓冲液提取植物叶材料,所述缓冲液含有50mm hepes(ph 7.0)、10mm乙酸钾、5mm乙酸镁、10%(v/v)甘油、0.05%(v/v)tween-20和300mm nacl。将蛋白提取物溶解在12%聚丙烯酰胺凝胶中。在凝胶上样之前,将等分试样的蛋白提取物与2
×
laemmli缓冲液以1:1的比例混合,并在95℃下孵育10分钟。凝胶泳道上的数字代表收获叶样品时浸润后天数。scolmb和scolmc分别表示来自表达scolmb和scolmc的植物组织的蛋白提取物。nc-来自用作阴性对照的未感染叶组织的提取物。l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671)。箭头表示对应于表达的重组大肠菌素的特定的蛋白条带。
[0289]
图37显示了针对列于表5a和5b中的10个选择的肠道沙门氏菌肠道亚种菌株的含scolma、scolmb和scolmc沙门菌素的植物提取物的抗微生物比活性的半定量评价。使用径向扩散测定通过斑点法检测抗微生物活性,并以每μg重组沙门菌素的任意单位(au)对其进行计算。
[0290]
图38显示了当使用hic hitraptm苯基ff(ls)树脂(a)和hitrap
tm
丁基ff树脂(b)时沙门菌素scolma蛋白纯化级分的sds-page分析。l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671);cl
–
柱上样;ft
–
流通;w
–
柱洗涤;f4、f8-f16、f18
–
洗脱级分。箭头表示对应于表达的重组沙门菌素scolma和rubisco大亚基(rbcl)的特定的蛋白条带。
[0291]
图39显示了对于hitrap
tm capto
tm mmc树脂的沙门菌素scolma蛋白纯化级分的sds-page分析。l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671);cl
–
柱上样;ft
–
流通;w
–
柱洗涤;f11-f16、f18、f19
–
洗脱级分。
[0292]
图40显示了使用不同ph值的提取缓冲液从本氏烟草叶生物量提取的scolma、scolmb和scolmc重组蛋白的sds-page分析。提取在4℃(a)和室温(约22℃)(b)下进行。接下来,使用缓冲液进行蛋白提取:1)50mm hepes(ph 7.0)、10mm乙酸钾、5mm乙酸镁、10%甘油、
0.05%tween-20、300mm nacl;2)15mm乙酸钠(ph 5.0)、20mm nah2po4、30mm nacl、0.05%tween-80和3)15mm乙酸钠(ph 4.0)、20mm nah2po4、30mm nacl、0.05%tween 80。
[0293]
l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671);1
–
用ph 7.0的提取缓冲液获得的提取物;2-用ph 5.0的提取缓冲液获得的提取物;3-用ph 4.0的提取缓冲液获得的提取物。
[0294]
图41显示了对于hitrap
tm capto
tm mmc树脂的沙门菌素scolmb蛋白纯化级分的sds-page分析。l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671);cl
–
柱上样;ft
–
流通;w
–
柱洗涤;f10-f20
–
洗脱级分。箭头表示特定的scolmb蛋白条带。
[0295]
图42显示了对于hitrap
tm
苯基ff(ls)树脂的沙门菌素scolmb蛋白纯化级分的sds-page分析。l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671);cl
–
柱上样;ft
–
流通;w
–
柱洗涤;f9-f18
–
洗脱级分。箭头表示特定的scolmb蛋白条带。
[0296]
图43显示了对于hitrap
tm capto
tm mmc树脂的沙门菌素scolmc蛋白纯化级分的sds-page分析。l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671);cl
–
柱上样;ft
–
流通;w
–
柱洗涤;f10-f20
–
洗脱级分。箭头表示特定的scolmc蛋白条带。
[0297]
图44显示了对于hitrap
tm
苯基ff(ls)树脂的沙门菌素scolmc蛋白纯化级分的sds-page分析。l
–
pageruler
tm
预染色的蛋白梯(thermo fisher scientific inc.(waltham,usa),#sm0671);cl
–
柱上样;ft
–
流通;w
–
柱洗涤;f9-f18
–
洗脱级分。箭头表示特定的scolmc蛋白条带。
[0298]
发明详述
[0299]
本发明的蛋白是对沙门氏菌具有细胞毒性作用的蛋白,在本文称为"沙门菌素"。沙门菌素通常具有至少一个结合结构域(也称为"受体结合结构域"),其允许沙门菌素结合到靶沙门氏菌细胞的表面受体结构上。沙门菌素还具有细胞毒性结构域,其可以是催化结构域或成孔结构域。催化结构域可具有rna酶或dna酶催化活性、针对细胞壁肽聚糖(胞壁质)生物合成的抑制活性,或可降解沙门氏菌的细胞壁结构。此外,沙门菌素可具有易位结构域,其可与靶沙门氏菌的细胞膜蛋白相互作用,从而使沙门菌素易位至沙门菌素发挥其细胞毒性功能的区室。
[0300]
发明人假设本发明的m型沙门菌素(scolm或salm)是肽聚糖酶,其特异性地裂解位于肽聚糖脂质i和脂质ii中间体的脂质部分与焦磷酰基之间的键,其位于内膜的周质侧(通过与以下文献类似的方法:gross和braun,mol.gen.genet.251(1996)388-396;barreteau等人,microbial drug resistance 18(2012),222-229)。释放的c55-聚异戊烯醇不再将murnac-五肽-glcnac易位穿过细胞质膜。scolm沙门菌素在其被摄取穿过外膜进入周质后杀死敏感的沙门氏菌菌株。scolm的作用方式被认为包括吸附到fhua外膜受体、通过tonb输入机制(tonb、exbb和exbd)通过细胞外膜能量依赖性地易位到周质中以及其底物的催化作用的步骤。这些步骤中的每一个都由特定的蛋白结构域执行。因此,scolm沙门菌素也具有三结构域结构组织和窄抗微生物谱,因为针对沙门氏菌以外的细菌的抗菌活性是有限的。
[0301]
对于沙门氏菌的特异性来说,结合结构域是重要的,并且(尤其)将沙门菌素与其
它类似的大肠菌素区别开来。因此,本发明的蛋白可以定义为具有至少一个结合结构域,所述结合结构域包含如上文第(1)项或权利要求1中定义的任一氨基酸序列区段,或由其组成,或包含于其中。第(1)项或权利要求1的(a-i)至(a-v)项分别定义沙门菌素scole2、scole3、scole7、scole1a和scole1b的结合结构域。上文第(1)项的(a-vii)至(a-x)以及(a-xi)和(a-xii)项分别定义沙门菌素scole1c、scole1d、scole1e、scolma、scolmb和scolmc的结合结构域。spst的结合结构域包含在上文第(1)项的(a-vi)项中定义的氨基酸序列区段中。沙门菌素scole2、scole3、scole7、scole1a、scole1b和spst的氨基酸序列分别以seq id no:1至6给出。沙门菌素scole1c、scole1d、scole1e、scolma、scolmb和scolmc的氨基酸序列分别以seq id no:25至28、33和34给出。上文第(1)项的(b)-(d)项定义了scole2、scole3、scole7、scole1a、scole1b、spst、scole1c、scole1d、scole1e、scolma、scolmb和scolmc的衍生物,它们具有或含有衍生物结合结构域(或氨基酸序列区段)。类似地,(b)至(e)项和(α)至(δ)项(在下文定义)定义了scole2、scole3、scole7、scole1a、scole1b、spst、scole1c、scole1d、scole1e、scolma、scolmb和scolmc的衍生物。scole2、scole3、scole7、scole1a、scole1b、spst、scole1c、scole1d、scole1e、scolma、scolmb和scolmc优选能够对沙门氏菌产生细胞毒性作用。
[0302]
在本发明中,优选沙门菌素scole1c、scole1d、scole1e、scolma、scolmb和scolmc(及如所定义的其衍生物);更优选scolma、scolmb和scolmc(及如所定义的其衍生物)。scolmb,并且特别是scolmc(及如所定义的其衍生物)是最优选的,因为它们在表达后纯化异常容易并且具有抗微生物活性。由于其异常高的抗微生物活性(参见例如实施例23),scolmc(及如所定义的其衍生物)也是优选的。如本文所定义的scole1a和scole1b以及scole1d及其衍生物优选与如本文所定义的scolm沙门菌素(例如scolma、scolmb和scolmc)及其衍生物组合使用。
[0303]
在本文中,氨基酸序列区段(或简称为区段)是指蛋白或具有比该区段更多的氨基酸残基数目的多肽的多个连续氨基酸残基。本文也将结构域称为"氨基酸序列区段"或简称为"区段"。术语“蛋白”和“多肽”在本文中可互换使用。
[0304]
本发明的蛋白至少包含结合结构域。下列(b)至(d)项中每项的(i)至(v)、(vii)至(x)、(xi)和(xii)项均定义了优选的结合结构域。最优选的结合结构域是(a-i)至(a-v)、(a-vii)至(a-x)、(a-xi)和(a-xii)项的结合结构域。下列(b)至(d)项中每项的(vi)项(即(b-vi)、(c-vi)和(d-vi)子项)均定义了优选的氨基酸序列区段,其含有结合结构域并且是沙门菌素spst的衍生物。本发明的蛋白优选包含任何一个下列氨基酸序列区段:
[0305]
(b-i)与scole2(seq id no:1)的氨基酸残基316至449的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0306]
(b-ii)与scole3(seq id no:2)的氨基酸残基315至483的区段具有至少75%、优选80%、更优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0307]
(b-iii)与scole7(seq id no:3)的氨基酸残基318至451的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0308]
(b-iv)与scole1a(seq id no:4)的氨基酸残基174至297的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0309]
(b-v)与scole1b(seq id no:5)的氨基酸残基198至322的区段具有至少80%、优
选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0310]
(b-vi)与包含seq id no:6的spst的至少200个连续氨基酸残基的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段:在一个实施方案中,与包含seq id no:6的spst的至少250个连续氨基酸残基的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段;在另一个实施方案中;在另一个实施方案中,蛋白包含与seq id no:6的(全部)氨基酸序列具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的氨基酸序列,或由其组成,
[0311]
(b-vii)与scole1c(seq id no:25)的氨基酸残基195至319的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0312]
(b-viii)与scole1d(seq id no:26)的氨基酸残基195至319的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0313]
(b-ix)与scole1e(seq id no:27)的氨基酸残基193至317的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0314]
(b-x)与scolma(seq id no:28)的氨基酸残基38至138的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段;
[0315]
(b-xi)与scolmb(seq id no:33)的氨基酸残基38至138的区段具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%、最优选至少97%序列同一性的区段;或
[0316]
(b-xii)与scolmc(seq id no:34)的氨基酸残基38至138的区段具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%、最优选至少97%序列同一性的区段;
[0317]
或
[0318]
(c-i)与scole2(seq id no:1)的氨基酸残基316至449的区段具有至少90%、优选至少95%序列相似性的区段,
[0319]
(c-ii)与scole3(seq id no:2)的氨基酸残基315至483的区段具有至少85%、优选至少90%、更优选至少95%序列相似性的区段,
[0320]
(c-iii)与scole7(seq id no:3)的氨基酸残基318至451的区段具有至少90%、优选至少95%序列相似性的区段,
[0321]
(c-iv)与scole1a(seq id no:4)的氨基酸残基174至297的区段具有至少90%、优选至少95%序列相似性的区段,
[0322]
(c-v)与scole1b(seq id no:5)的氨基酸残基198至322的区段具有至少90%、优选至少95%序列相似性的区段,
[0323]
(c-vi)与包含seq id no:6的spst的至少200个连续氨基酸残基的区段具有至少90%、优选至少95%序列相似性的区段:在一个实施方案中,与包含seq id no:6的spst的至少250个连续氨基酸残基的区段具有至少90%、优选至少95%序列相似性的区段;在另一个实施方案中,蛋白包含与seq id no:6的(全部)氨基酸序列具有至少80%、优选至少90%、最优选至少95%序列相似性的氨基酸序列,或由其组成,
[0324]
(c-vii)与scole1c(seq id no:25)的氨基酸残基195至319的区段具有至少90%、优选至少95%序列相似性的区段,
[0325]
(c-viii)与scole1d(seq id no:26)的氨基酸残基195至319的区段具有至少
90%、优选至少95%序列相似性的区段,
[0326]
(c-ix)与scole1e(seq id no:27)的氨基酸残基193至317的区段具有至少90%、优选至少95%序列相似性的区段,
[0327]
(c-x)与scolma(seq id no:28)的氨基酸残基38至138的区段具有至少90%、优选至少95%序列相似性的区段,
[0328]
(c-xi)与scolmb(seq id no:33)的氨基酸残基38至138的区段具有至少90%、优选至少95%、更优选至少97%序列相似性的区段,或
[0329]
(c-xii)与scolmc(seq id no:34)的氨基酸残基38至138的区段具有至少90%、优选至少95%、更优选至少97%序列相似性的区段;
[0330]
或
[0331]
(d-i)与scole2(seq id no:1)的氨基酸残基316至449的区段相比具有1至20个、优选1至15个、更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0332]
(d-ii)与scole3(seq id no:2)的氨基酸残基315至483的区段相比具有1至30个、优选1至29个、更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0333]
(d-iii)与scole7(seq id no:3)的氨基酸残基318至451的区段相比具有1至20个、优选1至15个、更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0334]
(d-iv)与scole1a(seq id no:4)的氨基酸残基174至297的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0335]
(d-v)与scole1b(seq id no:5)的氨基酸残基198至322的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0336]
(d-vi)与包含seq id no:6的spst的至少200个连续氨基酸残基的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个氨基酸取代、添加、插入或缺失的区段:在一个实施方案中,与包含seq id no:6的spst的至少250个连续氨基酸残基的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个氨基酸取代、添加、插入或缺失的区段;在另一个实施方案中,蛋白包含与seq id no:6的(全部)氨基酸序列相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,或由其组成。
[0337]
(d-vii)与scole1c(seq id no:25)的氨基酸残基195至319的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0338]
(d-viii)与scole1d(seq id no:26)的氨基酸残基195至319的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0339]
(d-ix)与scole1e(seq id no:27)的氨基酸残基193至317的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0340]
(d-x)与scolma(seq id no:28)的氨基酸残基38至138的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个、最优选1至5个氨基酸取代、添加、插入
或缺失的区段,
[0341]
(d-xi)与scolmb(seq id no:33)的氨基酸残基38至138的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个、最优选1至5个氨基酸取代、添加、插入或缺失的区段,或
[0342]
(d-xii)与scolmc(seq id no:34)的氨基酸残基38至138的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个、最优选1至5个氨基酸取代、添加、插入或缺失的区段。在上述蛋白(或多肽)中,由数字(vii)至(xii)标识的亚类中的那些是优选的(在所有(a)至(d)类中),(x)、(xi)和(xii)亚类中的那些是更优选的。
[0343]
在另一个实施方案中,本发明提供了优选能够对沙门氏菌产生细胞毒性作用的蛋白,其中所述蛋白的氨基酸序列如上文(a-i)至(a-vi)、(a-vii)至(a-x)、(a-xi)、(a-xii)、(b-i)至(b-vi)、(b-vii)至(b-x)、(b-xi)、(b-xii)、(c-i)至(c-vi)、(c-vii)至(c-x)、(c-xi)、(c-xii)、(d-i)至(d-vi)、或(d-vii)至(d-x)、(d-xi)、(d-xii)项中任一项所定义或通过包含所述项的区段来定义。在一个优选的实施方案中,所述蛋白(或多肽)的氨基酸序列由上文定义的(a-vii)至(a-x)、(a-xi)、(a-xii)、(b-vii)至(b-x)、(b-xi)、(b-xii)、(c-vii)至(c-x)、(c-xi)、(c-xii)、或(d-vii)至(d-x)、(d-xi)、(d-xii)项中任一项所定义或通过包含所述项的区段来定义。在更优选的实施方案中,所述蛋白的氨基酸序列由上文定义的(a-x)、(a-xi)、(a-xii)、(b-x)、(b-xi)、(b-xii)、(c-x)、(c-xi)、(c-xii)、或(d-x)、(d-xi)、(d-xii)项中任一项所定义或通过包含所述项的区段来定义。
[0344]
优选地、替代地或另外地,所述蛋白如(b-x)、(c-x)、(d-x)、(b-xi)、(c-xi)、(d-xi)、(b-xii)、(c-xii)、(d-xii)中任一项所定义,并且对应于seq id no:33的残基155的氨基酸残基是pro和/或对应于seq id no:33的残基246的氨基酸残基是arg或lys,优选arg。替代地或另外地,对应于seq id no:33的残基76和84的氨基酸残基可以是gln。
[0345]
在本文中,措辞“对应于seq id no:yy的残基xx
…
的氨基酸残基”是指所述蛋白的氨基酸序列在对应于seq id no:yy的残基xx的位置具有指定氨基酸残基。此处,xx代表蛋白的氨基酸序列中氨基酸残基的编号(从n末端开始),yy代表指定的seq id no:。
[0346]
可以通过将蛋白与seq id no:33的氨基酸序列进行比对以给出最佳比对来确定相应的氨基酸残基(如例如图34完成并显示的)。措辞“对应于残基
…
的氨基酸残基”是指如图34所示的比对,并且表示在seq id no:yy(此处:seq id no:33)中具有在所述比对中与seq id no:yy中指定的氨基酸残基相同位置(即,在彼此的顶部上书写)的氨基酸残基。
[0347]
其中蛋白在本文中由氨基酸取代、添加、插入或缺失的数目或数目范围定义,氨基酸取代、添加、插入或缺失可以组合,但给定的数目或数目范围是指所有氨基酸取代、添加、插入和缺失的总和。在氨基酸取代、添加、插入或缺失中,优选氨基酸取代、添加和缺失。术语"插入"是指在参考序列的氨基酸序列内部的插入,即排除在c-或n-末端的添加。术语添加是指在参考序列的氨基酸序列的c-或n-末端添加。缺失可以是参考序列的末端或内部氨基酸残基的缺失。在此,蛋白或其任何结构域由相对于所示的氨基酸序列区段的氨基酸取代、添加、插入或缺失的数目或数目范围定义,在进一步的实施方案中,蛋白或结构域可以具有相对于所示的氨基酸序列区段的1至数个氨基酸取代、添加、插入或缺失。
[0348]
本发明蛋白的细胞毒性或催化结构域可以如上文第(3)项所定义。在优选的实施方案中,本发明的蛋白包含细胞毒性或催化结构域,其包含下列氨基酸序列区段中的任何
一个,或由其组成:
[0349]
(b-i)’与scole2(seq id no:1)的氨基酸残基453至582的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0350]
(b-ii)’与scole3(seq id no:2)的氨基酸残基501至584的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0351]
(b-iii)’与scole7(seq id no:3)的氨基酸残基455至584的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0352]
(b-iv)’与scole1a(seq id no:4)的氨基酸残基306至478的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0353]
(b-v)’与scole1b(seq id no:5)的氨基酸残基350至522的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0354]
(b-vi)’与spst(seq id no:6)的氨基酸残基112至288的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0355]
(b-vii)’与来自scole1c(seq id no:25)的氨基酸残基347至519的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0356]
(b-viii)’与来自scole1d(seq id no:26)的氨基酸残基347至519的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0357]
(b-ix)’与来自scole1e(seq id no:27)的氨基酸残基345至517的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0358]
(b-x)’与来自scolma(seq id no:28)的氨基酸残基139至269的区段具有至少80%、优选至少85%、更优选至少90%、最优选至少95%序列同一性的区段,
[0359]
(b-xi)’与来自scolmb(seq id no:33)的氨基酸残基139至269的区段具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%、最优选至少97%序列同一性的区段,或
[0360]
(b-xii)’与来自scolmc(seq id no:34)的氨基酸残基139至269的区段具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%、最优选至少97%序列同一性的区段;
[0361]
或
[0362]
(c-i)’与scole2(seq id no:1)的氨基酸残基453至582的区段具有至少90%、优选至少95%序列相似性的区段,
[0363]
(c-ii)’与scole3(seq id no:2)的氨基酸残基501至584的区段具有至少90%、优选至少95%序列相似性的区段,
[0364]
(c-iii)’与scole7(seq id no:3)的氨基酸残基455至584的区段具有至少90%、优选至少95%序列相似性的区段,
[0365]
(c-iv)’与scole1a(seq id no:4)的氨基酸残基306至478的区段具有至少90%、优选至少95%序列相似性的区段,
[0366]
(c-v)’与scole1b(seq id no:5)的氨基酸残基350至522的区段具有至少90%、优选至少95%序列相似性的区段,
[0367]
(c-vi)’与spst(seq id no:6)的氨基酸残基112至288的区段具有至少90%、优选
至少95%序列相似性的区段,
[0368]
(c-vii)’与scole1c(seq id no:25)的氨基酸残基347至519的区段具有至少90%、优选至少95%序列相似性的区段,
[0369]
(c-viii)’与scole1d(seq id no:26)的氨基酸残基347至519的区段具有至少90%、优选至少95%序列相似性的区段,
[0370]
(c-ix)’与scole1e(seq id no:27)的氨基酸残基345至517的区段具有至少90%、优选至少95%序列相似性的区段,
[0371]
(c-x)’与scolma(seq id no:28)的氨基酸残基139至269的区段具有至少90%、优选至少95%序列相似性的区段,
[0372]
(c-xi)’与scolmb(seq id no:33)的氨基酸残基139至269的区段具有至少90%、优选至少95%、更优选至少97%序列相似性的区段,或
[0373]
(c-xii)’与scolmc(seq id no:34)的氨基酸残基139至269的区段具有至少90%、优选至少95%、更优选至少97%序列相似性的区段;
[0374]
或
[0375]
(d-i)’与scole2(seq id no:1)的氨基酸残基453至582的区段相比具有1至20个、优选1至15个、更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0376]
(d-ii)’与scole3(seq id no:2)的氨基酸残基501至584的区段相比具有1至20个、优选1至15个、优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0377]
(d-iii)’与scole7(seq id no:3)的氨基酸残基455至584的区段相比具有1至20个、优选1至15个、优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0378]
(d-iv)’与scole1a(seq id no:4)的氨基酸残基306至478的区段相比具有1至30个、优选1至20个、更优选1至15个、优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0379]
(d-v)’与scole1b(seq id no:5)的氨基酸残基350至522的区段相比具有1至30个、优选1至20个、更优选1至15个、优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0380]
(d-vi)’与spst(seq id no:6)的氨基酸残基112至288的区段相比具有1至20个、优选1至15个、更优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0381]
(d-vii)’与scole1c(seq id no:25)的氨基酸残基347至519的区段相比具有1至30个、优选1至20个、更优选1至15个、优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0382]
(d-viii)’与scole1d(seq id no:26)的氨基酸残基347至519的区段相比具有1至30个、优选1至20个、更优选1至15个、优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0383]
(d-ix)’与scole1e(seq id no:27)的氨基酸残基345至517的区段相比具有1至30个、优选1至20个、更优选1至15个、优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0384]
(d-x)’与scolma(seq id no:28)的氨基酸残基139至269的区段相比具有1至30个、优选1至20个、更优选1至15个、优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0385]
(d-xi)’与scolmb(seq id no:33)的氨基酸残基139至269的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个、最优选至少1至5个氨基酸取代、添加、插入或缺失的区段,或
[0386]
(d-xii)’与scolmc(seq id no:34)的氨基酸残基139至269的区段相比具有1至30个、优选1至20个、更优选1至15个、甚至更优选1至10个、最优选1至5个氨基酸取代、添加、插
入或缺失的区段。
[0387]
在更优选的实施方案中,本发明的蛋白包含细胞毒性或催化结构域,该结构域包含序列区段(a-vii)’至(a-x)’、(a-xi)’或(a-xii)’中的任何一个或由其组成。
[0388]
蛋白可以如(b-x)’、(c-x)’、(d-x)’、(b-xi)’、(c-xi)’、(d-xi)’、(b-xii)’、(c-xii)’或(d-xii)’中的任一项所定义,并且所述蛋白对应于seq id no:33的残基155的氨基酸残基是pro和/或对应于seq id no:33的残基246的氨基酸残基是arg或lys,优选arg。替代地或另外地,对应于seq id no:33的残基76和84的氨基酸残基可以是gln。
[0389]
在本文中,在任何(x-y)’项(其中x代表a、b、c或d中的任何一个,y代表任何罗马数字i至xii)中,引号’表示催化结构域或区段。缺少引号的(x-y)项表示结合结构域或区段。携带双引号”的(x-y)”表示易位结构域或区段。在(a)至(d)项中,优选(a)、(b)和(d)项的那些,并且更优选(a)和(d)项。类似地,在(a)’至(d)’项中,优选(a)’、(b)’和(d)’项的那些,并且更优选(a)’和(d)’项。类似地,在(a)”至(d)”项中,优选(a)”、(b)”和(d)”项的那些,并且更优选(a)”和(d)”项:
[0390]
当本发明的蛋白包含如本文定义的结合结构域和如本文定义的催化结构域时,如上定义的任何结合结构域(或区段)可以与任何催化结构域(或区段)组合。在优选的实施方案中,将(i)至(x)中任一子项的结合结构域在本发明的蛋白中分别与子项(i)’至(x)’的催化结构域组合(例如,第(iii)项的结合结构域与第(iii)’项的催化结构域组合),由此催化结构域可以在蛋白的c末端侧。在一个实施方案中,将(a)至(d)中任意项的结合结构域分别与(a)'至(d)'项的催化结构域结合,由此催化结构域可以在所述蛋白的c末端侧。
[0391]
在某些实施方案中,本发明的蛋白能够对沙门氏菌产生细胞毒性作用,并且该蛋白包含至少任何一个下列氨基酸序列区段的组合,优选从蛋白的n-末端至c-末端给定的顺序:
[0392]
(α-i)来自seq id no:1的氨基酸残基316至449的区段和来自seq id no:1的氨基酸残基453至582的区段,
[0393]
(α-ii)来自seq id no:2的scole3的氨基酸残基315至483的区段和来自seq id no:2的氨基酸残基501至584的区段,
[0394]
(α-iii)来自seq id no:3的氨基酸残基318至451的区段和来自seq id no:3的氨基酸残基455至584的区段,
[0395]
(α-iv)来自seq id no:4的氨基酸残基174至297的区段和来自seq id no:4的氨基酸残基306至478的区段,
[0396]
(α-v)来自seq id no:5的氨基酸残基198至322的区段和来自seq id no:5的氨基酸残基350至522的区段,
[0397]
(α-vi)包含包括来自seq id no:6的氨基酸残基112至288的区段的seq id no:6的至少200个连续氨基酸残基的区段,
[0398]
(α-vii)来自seq id no:25的氨基酸残基195至319的区段和来自seq id no:25的氨基酸残基347至519的区段,
[0399]
(α-viii)来自seq id no:26的氨基酸残基195至319的区段和来自seq id no:26的氨基酸残基347至519的区段,
[0400]
(α-ix)来自seq id no:27的氨基酸残基193至317的区段和来自seq id no:27的
氨基酸残基345至517的区段,
[0401]
(α-x)来自seq id no:28的氨基酸残基38至138的区段和来自seq id no:28的氨基酸残基139至269的区段,
[0402]
(α-xi)来自seq id no:33的氨基酸残基38至138的区段和来自seq id no:33的氨基酸残基139至269的区段,或
[0403]
(α-xii)来自seq id no:34的氨基酸残基38至138的区段和来自seq id no:34的氨基酸残基139至269的区段;
[0404]
或
[0405]
(β-i)与来自seq id no:1的氨基酸残基316至449的区段具有至少75%序列同一性的区段和与来自seq id no:1的氨基酸残基453至582的区段具有至少70%序列同一性的区段,
[0406]
(β-ii)与来自seq id no:2的氨基酸残基315至483的区段具有至少70%序列同一性的区段和与来自seq id no:2的氨基酸残基501至584的区段具有至少70%序列同一性的区段,
[0407]
(β-iii)与来自seq id no:3的氨基酸残基318至451的区段具有至少77%序列同一性的区段和与来自seq id no:3的氨基酸残基455至584的区段具有至少70%序列同一性的区段,
[0408]
(β-iv)与来自seq id no:4的氨基酸残基174至297的区段具有至少70%序列同一性的区段和与来自seq id no:4的氨基酸残基306至478的区段具有至少70%序列同一性的区段,
[0409]
(β-v)与来自seq id no:5的氨基酸残基198至322的区段具有至少70%序列同一性的区段和与来自seq id no:5的氨基酸残基350至522的区段具有至少70%序列同一性的区段,
[0410]
(β-vi)与包含包括与来自seq id no:6的氨基酸残基112至288的区段具有至少70%序列同一性的区段的seq id no:6的至少200个连续氨基酸残基的区段具有至少70%序列同一性的区段,
[0411]
(β-vii)与来自seq id no:25的氨基酸残基195至319的区段具有至少70%序列同一性的区段和与来自seq id no:25的氨基酸残基347至519的区段具有至少70%序列同一性的区段,
[0412]
(β-viii)与来自seq id no:26的氨基酸残基195至319的区段具有至少70%序列同一性的区段和与来自seq id no:26的氨基酸残基347至519的区段具有至少70%序列同一性的区段,
[0413]
(β-ix)与来自seq id no:27的氨基酸残基193至317的区段具有至少70%序列同一性的区段和与来自seq id no:27的氨基酸残基345至517的区段具有至少70%序列同一性的区段,
[0414]
(β-x)与来自seq id no:28的氨基酸残基38至138的区段具有至少70%序列同一性的区段和与来自seq id no:28的氨基酸残基139至269的区段具有至少70%序列同一性的区段,
[0415]
(β-xi)与来自seq id no:33的氨基酸残基38至138的区段具有至少70%、优选至
少80%、更优选至少90%、最优选至少95%序列同一性的区段和与来自seq id no:33的氨基酸残基139至269的区段具有至少70%、优选至少80%、更优选至少90%、最优选至少95%序列同一性的区段,
[0416]
(β-xii)与来自seq id no:34的氨基酸残基38至138的区段具有至少70%、优选至少80%、更优选至少90%、最优选至少95%序列同一性的区段和与来自seq id no:34的氨基酸残基139至269的区段具有至少70%、优选至少80%、更优选至少90%、最优选至少95%序列同一性的区段;
[0417]
或
[0418]
(χ-i)与来自seq id no:1的氨基酸残基316至449的区段具有至少85%序列相似性的区段和与来自seq id no:1的氨基酸残基453至582的区段具有至少80%序列相似性的区段,
[0419]
(χ-ii)与来自seq id no:2的氨基酸残基315至483的区段具有至少80%序列相似性的区段和与来自seq id no:2的氨基酸残基501至584的区段具有至少80%序列相似性的区段,
[0420]
(χ-iii)与来自seq id no:3的氨基酸残基318至451的区段具有至少85%序列相似性的区段和与来自seq id no:3的氨基酸残基455至584的区段具有至少80%序列相似性的区段,
[0421]
(χ-iv)与来自seq id no:4的氨基酸残基174至297的区段具有至少80%序列相似性的区段和与来自seq id no:4的氨基酸残基306至478的区段具有至少80%序列相似性的区段,
[0422]
(χ-v)与来自seq id no:5的氨基酸残基198至322的区段具有至少80%序列相似性的区段和与来自seq id no:5的氨基酸残基350至522的区段具有至少80%序列相似性的区段,
[0423]
(χ-vi)与包含包括与来自seq id no:6的氨基酸残基112至288的区段具有至少80%序列相似性的区段的seq id no:6的至少200个连续氨基酸残基的区段具有至少80%序列相似性的区段,
[0424]
(χ-vii)与来自seq id no:25的氨基酸残基195至319的区段具有至少80%序列相似性的区段和与来自seq id no:25的氨基酸残基347至519的区段具有至少80%序列相似性的区段,
[0425]
(χ-viii)与来自seq id no:26的氨基酸残基195至319的区段具有至少80%序列相似性的区段和与来自seq id no:26的氨基酸残基347至519的区段具有至少80%序列相似性的区段,
[0426]
(χ-ix)与来自seq id no:27的氨基酸残基193至317的区段具有至少80%序列相似性的区段和与来自seq id no:27的氨基酸残基345至517的区段具有至少80%序列相似性的区段,或
[0427]
(χ-x)与来自seq id no:28的氨基酸残基38至138的区段具有至少80%序列相似性的区段和与来自seq id no:28的氨基酸残基139至269的区段具有至少80%序列相似性的区段,
[0428]
(χ-xi)与来自seq id no:33的氨基酸残基38至138的区段具有至少80%、优选至
少90%、最优选至少95%序列相似性的区段和与来自seq id no:33的氨基酸残基139至269的区段具有至少80%、优选至少90%、最优选至少95%序列相似性的区段,
[0429]
(χ-xii)与来自seq id no:34的氨基酸残基38至138的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段和与来自seq id no:34的氨基酸残基139至269的区段具有至少80%、优选至少90%、更优选至少95%序列相似性的区段;
[0430]
或
[0431]
(δ-i)与来自seq id no:1的氨基酸残基316至449的区段相比具有1至25个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:1的氨基酸残基453至582的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段,
[0432]
(δ-ii)与来自seq id no:2的氨基酸残基315至483的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:2的氨基酸残基501至584的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段,
[0433]
(δ-iii)与来自seq id no:3的氨基酸残基318至451的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:3的氨基酸残基455至584的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段,
[0434]
(δ-iv)与来自seq id no:4的氨基酸残基174至297的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:4的氨基酸残基306至478的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
[0435]
(δ-v)与来自seq id no:5的氨基酸残基198至332的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:5的氨基酸残基350至522的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
[0436]
(δ-vi)与包含包括与来自seq id no:6的氨基酸残基112至288的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段的seq id no:6的至少200个连续氨基酸残基的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
[0437]
(δ-vii)与来自seq id no:25的氨基酸残基195至319的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:25的氨基酸残基347至519的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
[0438]
(δ-viii)与来自seq id no:26的氨基酸残基195至319的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:26的氨基酸残基347至519的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
[0439]
(δ-ix)与来自seq id no:27的氨基酸残基193至317的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:27的氨基酸残基345至517的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,或
[0440]
(δ-x)与来自seq id no:28的氨基酸残基38至138的区段相比具有1至30个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:28的氨基酸残基139至269的区段相比具有1至40个氨基酸取代、添加、插入或缺失的区段,
[0441]
(δ-xi)与来自seq id no:33的氨基酸残基38至138的区段相比具有1至30个、优选1至20个、更优选1至10个、最优选1至5个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:33的氨基酸残基139至269的区段相比具有1至40个、优选1至30个、更优选1至20个、
最优选1至10个氨基酸取代、添加、插入或缺失的区段,
[0442]
(δ-xii)与来自seq id no:34的氨基酸残基38至138的区段相比具有1至30个、优选1至20个、更优选1至10个、最优选1至5个氨基酸取代、添加、插入或缺失的区段和与来自seq id no:34的氨基酸残基139至269的区段相比具有1至40个、优选1至30个、更优选1至20个、最优选1至10个氨基酸取代、添加、插入或缺失的区段。
[0443]
蛋白优选包含(vii)至(xii)、甚至更优选(x)、(xi)或(xii)亚类中定义的氨基酸序列区段的至少任何一种组合。
[0444]
所有这些实施方案可以与本文定义的各个区段的最小序列同一性或相似性的优选值或氨基酸取代、添加、插入或缺失的优选数组合。
[0445]
上文(发明内容的)第(4)项定义了scole2、scole3、scole7、scole1a、scole1b、scole1c、scole1d、scole1e、scolma、scolmb和scolmc的易位结构域及其衍生物。易位结构域及其衍生物的定义可以与细胞毒性和结合结构域或其衍生物的定义组合。易位结构域及其衍生物的定义可以与以下特别定义的蛋白的定义组合。
[0446]
本发明的蛋白可以具有根据(a-i)至(a-x)、(a-xi)和(a-xii)项中任一项或根据(b-i)至(b-x)、(b-xi)和(b-xii)、(c-i)至(c-x)、(c-xi)和(c-xii)、或(d-i)至(d-x)、(d-xi)和(d-xii)项中任何衍生物的结合结构域(或结合区段)。优选地,本发明的蛋白可具有根据(a-vii)至(ax)、(a-xi)和(a-xii)项中任一项的结合结构域(或结合区段)或根据(b-vii)至(b-x)、(b-xi)和(b-xii)、(c-vii)至(c-x)、(c-xi)和(c-xii)、或(d-vii)至(d-x)、(d-xi)和(d-xii)项的任何衍生物。任何这样的结合结构域可以与根据(a-i)’至(a-x)’、(a-xi)’、(a-xii)’、(b-i)’至(b-x)’、(b-xi)’、(b-xii)’、(c-i)’至(c-x)’、(c-xi)’、(c-xii)’、或(d-i)’至(d-x)、(d-xi)’、或(d-xii)’任一项的催化/细胞毒性结构域组合。优选地,任何这样的结合结构域可以与根据(a-vii)’至(a-x)’、(a-xi)’、(a-xii)’、(b-vii)’至(b-x)’、(b-xi)’、(b-xii)’、(c-vii)’至(c-x)’、(c-xi)’、(c-xii)’、或(d-i)’至(d-x)、(d-xi)’、(d-xii)’任一项的催化/细胞毒性结构域组合,由此该优选实施方案可以优选地与本段上面给出的优选结合域组合。
[0447]
沙门菌素的结构域结构允许建立人工合成沙门菌素,其中将来自本发明的不同沙门菌素的结构域或如本文定义的其衍生物进行组合以形成新的沙门菌素(嵌合沙门菌素)。在这类嵌合沙门菌素中,易位结构域(如果存在的话)、结合结构域和催化或活性结构域的n-末端到c-末端的天然沙门菌素的结构域序列可以保持或不保持;优选地,它是保持的。因此,本发明的蛋白从n末端至c末端可包含(a-i)至(a-x)、(a-xi)和(a-xii)项中任一项的结合结构域,或根据(b-i)至(b-x)、(b-xi)、(b-xii)、(c-i)至(c-x)、(c-xi)、(c-xii)、或(d-i)至(d-x)、(d-xi)、(d-xii)项的任何衍生物的结合结构域,以及(a-i)’至(a-x)’、(a-xi)’、(a-xii)’、(b-i)’至(b-x)’、(b-xi)’、(b-xii)’、(c-i)’至(c-x)’、(c-xi)’、(c-xii)’或(d-i)’至(d-x)’、(d-xi)’、(d-xii)’项中任一项的催化结构域(区段)。在优选的实施方案中,本发明的蛋白从n-末端到c-末端可包含(a-i)”至(a-ix)”、(b-i)”至(b-ix)”、(c-i)”至(c-ix)”或(d-i)”至(d-ix)”项中任一项的易位结构域,(a-i)至(a-x)项中任一项的结合结构域或根据(b-i)至(b-x)、(c-i)至(c-x)或(d-i)至(d-x)项的任何衍生物的结合结构域,以及(a-i)’至(a-x)’、(b-i)’至(b-x)’、(c-i)’至(c-x)’或(d-i)’至(d-x)’项中任一项的催化结构域(区段)。
[0448]
在沙门菌素核酸酶、成孔酶和溶菌酶的三种细胞毒性活性中(表1),结构域可在具有相同类型细胞毒性活性的沙门菌素之间交换。例如,具有rna酶类型细胞毒性的新沙门菌素可以从scole2或scole7的易位和结合结构域(或这些结构域的衍生物)和scole3的细胞毒性结构域中形成。然而,优选地,(i)至(x)子项中任一项的结合结构域分别与(i)’至(x)’子项中任一项的催化结构域组合,以增加与天然沙门菌素的相似性,优选(a)至(d)项中的任意项的每项的相似性。然而,更优选地,(i)至(v)或(vii)至(x)子项中任一项的结合结构域可分别与(i)’至(v)’或(vii)’至(x)’子项中任一项的催化结构域组合,并分别与(i)”至(v)”或(vii)”至(x)”子项中任一项,优选(a)至(d)项中的任何项的易位结构域组合,以增加与天然沙门菌素的相似性。
[0449]
在另一个实施方案中,(i)至(v)子项的结合结构域分别与(i)’至(vi)’(优选(i)’至(v)’)子项的催化结构域组合,以增加与天然沙门菌素的相似性,优选(a)至(d)项中的每一项。在进一步的实施方案中,(i)至(v)子项中任一项的结合结构域分别与(i)’至(v)’子项中任一项的催化结构域以及分别与(i)”至(v)”子项中任一项、优选(a)至(d)项中任一项的易位结构域组合,以增加与天然沙门菌素的相似性。
[0450]
本发明还提供了一种优选能够对沙门氏菌产生细胞毒性作用的蛋白,所述蛋白包含下列氨基酸序列,或由其组成:
[0451]
(a-i)seq id no:1,
[0452]
(a-ii)seq id no:2,
[0453]
(a-iii)seq id no:3,
[0454]
(a-iv)seq id no:4,
[0455]
(a-v)seq id no:5,
[0456]
(a-vi)seq id no:6,
[0457]
(a-vii)seq id no:25,
[0458]
(a-viii)seq id no:26,
[0459]
(a-ix)seq id no:27,
[0460]
(a-x)seq id no:28,
[0461]
(a-x)seq id no:33,或
[0462]
(a-x)seq id no:34;
[0463]
或
[0464]
(b-i)与seq id no:1的氨基酸序列具有至少75%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%序列同一性的氨基酸序列,
[0465]
(b-ii)与seq id no:2的氨基酸序列具有至少80%、优选至少85%、更优选至少93%、甚至更优选至少96%序列同一性的氨基酸序列,
[0466]
(b-iii)与seq id no:3的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%序列同一性的氨基酸序列,
[0467]
(b-iv)与seq id no:4的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%序列同一性的氨基酸序列,
[0468]
(b-v)与seq id no:5的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%序列同一性的氨基酸序列,
[0469]
(b-vi)与seq id no:6的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%序列同一性的氨基酸序列,
[0470]
(b-vii)与seq id no:25的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%序列同一性的氨基酸序列,
[0471]
(b-viii)与seq id no:26的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%序列同一性的氨基酸序列,
[0472]
(b-ix)与seq id no:27的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%序列同一性的氨基酸序列,
[0473]
(b-x)与seq id no:28的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%、最优选至少97%序列同一性的氨基酸序列,
[0474]
(b-xi)与seq id no:33的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%、最优选至少97%序列同一性的氨基酸序列,或
[0475]
(b-xii)与seq id no:34的氨基酸序列具有至少70%、优选至少80%、更优选至少85%、甚至更优选至少90%、甚至更优选至少95%、最优选至少97%序列同一性的氨基酸序列;
[0476]
或
[0477]
(c-i)与seq id no:1的氨基酸序列具有至少85%、优选至少90%、更优选至少95%序列相似性的氨基酸序列,
[0478]
(c-ii)与seq id no:2的氨基酸序列具有至少85%、优选至少90%、更优选至少95%序列相似性的氨基酸序列,
[0479]
(c-iii)与seq id no:3的氨基酸序列具有至少85%、优选至少90%、更优选至少95%序列相似性的氨基酸序列,
[0480]
(c-iv)与seq id no:4的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%序列相似性的氨基酸序列,
[0481]
(c-v)与seq id no:5的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%序列相似性的氨基酸序列,
[0482]
(c-vi)与seq id no:6的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%序列相似性的氨基酸序列,
[0483]
(c-vii)与seq id no:25的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%序列相似性的氨基酸序列,
[0484]
(c-viii)与seq id no:26的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%序列相似性的氨基酸序列,
[0485]
(c-ix)与seq id no:27的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%序列相似性的氨基酸序列,
[0486]
(c-x)与seq id no:28的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%、最优选至少97%序列相似性的氨基酸序列,
[0487]
(c-xi)与seq id no:33的氨基酸序列具有至少80%、优选至少85%、更优选至少
90%、甚至更优选至少95%、最优选至少97%序列相似性的氨基酸序列,或
[0488]
(c-xii)与seq id no:34的氨基酸序列具有至少80%、优选至少85%、更优选至少90%、甚至更优选至少95%、最优选至少97%序列相似性的氨基酸序列;
[0489]
或
[0490]
(d-i)与seq id no:1的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0491]
(d-ii)与seq id no:2的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0492]
(d-iii)与seq id no:3的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0493]
(d-iv)与seq id no:4的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0494]
(d-v)与seq id no:5的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0495]
(d-vi)与seq id no:6的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0496]
(d-vii)与seq id no:25的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0497]
(d-viii)与seq id no:26的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0498]
(d-ix)与seq id no:27的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、并最优选1至10个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0499]
(d-x)与seq id no:28的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、甚至更优选1至10个、并最优选1至5个氨基酸取代、添加、插入或缺失的氨基酸序列,
[0500]
(d-xi)与seq id no:33的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、甚至更优选1至10个、并最优选1至5个氨基酸取代、添加、插入或缺失的氨基酸序列,或
[0501]
(d-xii)与seq id no:34的氨基酸序列相比具有1至40个、优选1至30个、更优选1至20个、甚至更优选1至10个、并最优选1至5个氨基酸取代、添加、插入或缺失的氨基酸序列;
[0502]
或
[0503]
(e-i)包含seq id no:1的至少470个、优选至少525个、更优选至少555个连续氨基酸残基或由其组成的氨基酸序列,
[0504]
(e-ii)包含seq id no:2的至少470个、优选至少525个、更优选至少555个连续氨基酸残基或由其组成的氨基酸序列,
[0505]
(e-iii)包含seq id no:3的至少470个、优选至少525个、更优选至少555个连续氨基酸残基或由其组成的氨基酸序列,
[0506]
(e-iv)包含seq id no:4的至少390个、优选至少435个、更优选至少460个连续氨基酸残基或由其组成的氨基酸序列,
[0507]
(e-v)包含seq id no:5的至少425个、优选至少475个、更优选至少500个连续氨基酸残基或由其组成的氨基酸序列,
[0508]
(e-vi)包含seq id no:6的至少250个、优选至少270个、更优选至少282个连续氨基酸残基或由其组成的氨基酸序列,
[0509]
(e-vii)包含seq id no:25的至少425个、优选至少475个、更优选至少500个连续氨基酸残基或由其组成的氨基酸序列,
[0510]
(e-viii)包含seq id no:26的至少425个、优选至少475个、更优选至少500个连续氨基酸残基或由其组成的氨基酸序列,
[0511]
(e-ix)包含seq id no:27的至少425个、优选至少475个、更优选至少500个连续氨基酸残基或由其组成的氨基酸序列,
[0512]
(e-x)包含seq id no:28的至少215个、优选至少240个、更优选至少260个连续氨基酸残基或由其组成的氨基酸序列,
[0513]
(e-xi)包含seq id no:33的至少215个、优选至少240个、更优选至少260个连续氨基酸残基或由其组成的氨基酸序列,或
[0514]
(e-xii)包含seq id no:34的至少215个、优选至少240个、更优选至少260个连续氨基酸残基或由其组成的氨基酸序列。
[0515]
如通常理解的并且为避免任何疑问,措辞“包含以下任一氨基酸序列的蛋白”是指所述蛋白的氨基酸序列与定义的那些相比可包含额外的氨基酸残基或序列延伸(例如纯化标签或其他标签)。措辞“由以下任一氨基酸序列组成的蛋白”是指所述蛋白的氨基酸序列与定义的那些相比不具有额外的氨基酸残基。如上所述,在上述蛋白(或多肽)中,由数字(vii)至(xii)标识的亚类中的那些(在所有(a)至(d)类中)是优选的,(x)、(xi)和(xii)亚类中的那些是更优选的。这也适用于下文描述的优选实施方案。
[0516]
在另一个实施方案中,本发明提供了优选能够对沙门氏菌产生细胞毒性作用的蛋白,其中所述蛋白的氨基酸序列如(a-i)至(a-x)、(a-xi)、(a-xii)、(b-i)至(b-x)、(b-xi)、(b-xii)、(c-i)至(c-x)、(c-xi)、(c-xii)、(d-i)至(d-x)、(d-xi)、(d-xii)、或(e-i)至(e-x)、(e-xi)或(e-xii)项中任一项所定义。在一个优选实施方案中,本发明提供了优选能够对沙门氏菌产生细胞毒性作用的蛋白,其中所述蛋白的氨基酸序列如(a-vii)至(a-x)、(a-xi)、(a-xii)、(b-vii)至(b-x)、(b-xi)、(b-xii)、(c-vii)至(c-x)、(c-xi)、(c-xii)、(d-vii)至(d-x)、(d-xi)、(d-xii)、或(e-vii)至(e-x)、(e-xi)、或(e-xii)项中任一项所定义。
[0517]
上述关于seq id no 1至6、25至28、33或34的完整序列的蛋白的定义可以与上述基于一个或更多个特定结构域的蛋白定义组合,所述特定结构域如结合和/或催化或细胞毒性结构域和/或适用时易位结构域。
[0518]
在本文中,使用align sequences protein blast(blastp 2.6.1+)(stephen f.altschul,thomas l.madden,alejandro a.jinghui zhang,zheng zhang,webb miller和david j.lipman(1997),"gapped blast and psi-blast:a new generation of protein database search programs",nucleic acids res.25:3389-3402.)测定序列同一性和相似性。
[0519]
如上文(b)至(d)、(b)’至(d)’、(b)”至(d)”项或(b)至(d)或(e)项中定义的本发明的结构域和/或蛋白的衍生物,尽管上文定义的实施方案允许序列变化,但仍保留了如下定
义的氨基酸残基。在优选的实施方案中,氨基酸残基对应于:
[0520]
seq id no:4的残基125是asn或ser;
[0521]
seq id no:4的残基145是lys或arg;
[0522]
seq id no:4的残基151是ala或gly;
[0523]
seq id no:4的残基154是ala、ser或gly;
[0524]
seq id no:4的残基155是phe、leu或iie;
[0525]
seq id no:4的残基158是ala或gly;
[0526]
seq id no:4的残基163是glu、asp、ser、leu或ile,优选glu、asp或ser;
[0527]
seq id no:4的残基165是ala、thr、val或ser,优选ala、thr或val;
[0528]
seq id no:4的残基167是arg;
[0529]
seq id no:4的残基172是thr、ala或ser;
[0530]
seq id no:4的残基175是gln;
[0531]
seq id no:4的残基176是val或leu;
[0532]
seq id no:4的残基178是gln或leu,优选gln;
[0533]
seq id no:4的残基181是glu或asp,优选glu;
[0534]
seq id no:4的残基184是arg或gln,优选arg;
[0535]
seq id no:4的残基192是ala或thr;
[0536]
seq id no:4的残基195是ala或val;
[0537]
seq id no:4的残基196是glu或gln,优选glu;
[0538]
seq id no:4的残基198是ala或thr;
[0539]
seq id no:4的残基209是leu或iie,优选leu;
[0540]
seq id no:4的残基273是leu或iie;
[0541]
seq id no:4的残基280是arg;
[0542]
seq id no:4的残基283是lys;
[0543]
seq id no:4的残基286是gln或lys;
[0544]
seq id no:4的残基290是ala或thr;
[0545]
seq id no:1的残基299是asp、asn或glu;
[0546]
seq id no:4的残基301是leu;
[0547]
seq id no:4的残基302是asn或asp;
[0548]
seq id no:4的残基346是asn、asp或glu;
[0549]
seq id no:4的残基363是lys、asn或arg;
[0550]
seq id no:4的残基364是lys或gln。
[0551]
措辞"对应于氨基酸残基
…
的氨基酸残基"是指图20a-c和图26所示的比对,并且表示seq id no:4(scole1a)的氨基酸残基或在所述比对中具有与seq id no:4(scole1a)所示的氨基酸残基相同的位置(即写在彼此上方)的seq id no:1至3、5、6、25、26或27的氨基酸残基。
[0552]
在scole1a和scole1b的衍生物中,和/或scole1a和scole1b的衍生结构域中,与图20的scole1a和scole1b比对中相同的相应氨基酸残基可以与scole1a和scole1b中的氨基酸残基相同;和/或对应于scole1a和scole1b之间不同的氨基酸残基,一些或所有这些不同
的氨基酸残基可以是scole1a或scole1b中的氨基酸残基(但不是其它氨基酸残基)。
[0553]
在scole2和scole7的衍生物中,和/或scole2和scole7的衍生结构域中,与图20的scole2和scole7比对中相同的相应氨基酸残基可以是scole2或scole7相同的氨基酸残基;和/或对应于scole2和scole7之间不同的氨基酸残基,一些或所有这些不同的氨基酸残基可以是scole2或scole7中的氨基酸残基(但不是其它氨基酸残基)。
[0554]
根据本发明的沙门菌素可以包含额外的n-或c-末端氨基酸序列段,例如纯化标签,例如6个或更多个连续组氨酸残基的his标签;优选地,衍生物没有n-末端氨基酸残基的添加。
[0555]
本发明的蛋白(沙门菌素)优选能够对沙门氏菌,特别是对肠道沙门氏菌,更优选肠道沙门氏菌肠道亚种产生细胞毒性作用。可使用径向扩散测定通过斑点法检测是否满足该条件。待测蛋白对肠道沙门氏菌的细胞毒性是这样的:在点样5微升所述待测蛋白和seq id no:1的蛋白的溶液于软琼脂覆盖的平板上并随后在37℃下孵育琼脂平板12小时后,其和seq id no:1的蛋白产生相同直径的不含肠道沙门氏菌肠道亚种血清型纽波特菌株6962
tm
*活菌的斑点,所述软琼脂覆盖的平板用0.14ml 1
×
107cfu/ml/cm2的敏感的肠道沙门氏菌菌株的细菌溶液接种,其中待测蛋白的浓度是seq id no:1的蛋白的相当溶液的至多5倍。在优选的实施方案中,在其它方面相同的条件下,参考点不是seq id no:1的蛋白,而是seq id no:4或5的蛋白。
[0556]
本发明的组合物包含如上所述的蛋白(沙门菌素)和任选的其它组分,视情况需要,例如载体。组合物优选包含如上所述的scole1a和/或scole1b或其衍生物,以及任选的其它组分,视情况需要,例如载体。组合物可以包含一种或更多种如本文定义的不同蛋白(沙门菌素),例如两种、三种或四种如本文定义的不同蛋白(沙门菌素)。"不同"是指蛋白在至少一个氨基酸残基上不同。该组合物可以包含两种、三种或更多种来自由上文(i)至(x)、(xi)或(xii)项中任一项所代表的相同亚类的沙门菌素,或者优选地,来自由上文(i)至(x)项中任一项所代表的不同类别的沙门菌素。该组合物还可以包含一种或更多种大肠杆菌大肠菌素或其衍生物,例如如ep 3 097 783a1中所述的,例如用于相伴地控制致病性大肠杆菌如ehec。
[0557]
由于本发明的蛋白优选通过在植物或其细胞中表达而产生,因此组合物可以是植物材料或其提取物,其中植物材料是来自具有表达的蛋白的植物,优选烟草或具有表达的所述蛋白的可食用植物。植物材料提取物是含有水溶性蛋白的水溶液,其包括在所述植物材料中存在或表达的本发明的沙门菌素,或这种水溶液的干燥产物。提取物优选具有例如通过过滤或离心除去的植物材料的水不溶性组分。植物材料可以是选自由以下组成的组的植物的材料:菠菜、叶甜菜、甜菜根、胡萝卜、糖用甜菜、多叶甜菜、苋菜、烟草,和/或所述植物材料是一种或多种叶、根、块茎或种子,或所述叶、根、块茎或种子的压碎、磨碎或粉碎的产物。
[0558]
该组合物或来自植物材料的所述提取物可以是含有所述沙门菌素的固体或液体组合物,如溶液或分散液。液体组合物可以是含水的,例如水溶液。所述蛋白在所述水分散液或溶液中的浓度可以是0.0001-1mg/ml,优选0.001-0.1mg/ml,更优选0.005-0.05mg/ml。如果使用多于一种能够对沙门氏菌产生细胞毒性作用的沙门菌素,这些浓度与所有这些沙门菌素的总浓度有关。
[0559]
除了一种或多种沙门菌素外,水溶液还可以含有缓冲剂。缓冲剂可以是无机或有机酸或其盐。无机酸的实例是磷酸或其盐。有机酸的实例是hepes、乙酸、琥珀酸、酒石酸、苹果酸、苯甲酸、肉桂酸、乙醇酸、乳酸、柠檬酸和抗坏血酸。优选的有机酸是苹果酸、乳酸、柠檬酸和抗坏血酸。溶液的ph通常可为4至8,优选5至8,更优选6.0至7.5。如果组合物所施用的目标物是肉,则溶液的ph通常可为4至8,优选4.5至7,更优选5.0至6.5,甚至更优选5.0至6.0。此外,溶液可以含有诸如甘油或盐的等渗剂。优选使用的盐是氯化钠。含有一种或多种沙门菌素的水溶液可以是缓冲的水溶液,其可以含有另外的溶质,例如盐,如50-400mm nacl,优选100-200mm nacl。水溶液中还可以含有巯基化合物,如二硫苏糖醇(dtt)、二硫赤藓糖醇、硫代乙醇或谷胱甘肽,优选dtt。总巯基化合物在水溶液中的浓度可以是1-50mm,优选2-20mm,更优选4-10mm。
[0560]
如果本发明的组合物是固体组合物,它可以是粉末,例如通过冻干上述提取物或溶液获得的冻干的固体组合物。粉末可以含有另外的固体组分,例如上面对于水溶液所提到的那些。在使用前,可以用合适的液体例如水或缓冲液将其重构。固体组合物可以含有缓冲剂、盐或如上所述的其它成分,以便在固体组合物重组或溶解时可以达到上面给出的浓度。
[0561]
组合物的载体的实例是溶剂如水或含水缓冲液(如上所述)、盐、糖如单糖和二糖、糖醇和其它载体如药物组合物中已知的那些。后者的例子是淀粉、纤维素和其它蛋白如白蛋白。糖的实例是葡萄糖、果糖、乳糖、蔗糖和麦芽糖。
[0562]
基于组合物中蛋白的总重量,本发明的组合物可以含有至少10重量%,优选至少20重量%,更优选至少30重量%,甚至更优选至少50重量%,甚至更优选至少75重量%的一种或更多种本发明的沙门菌素。组合物中沙门菌素的含量可以通过将组合物进行sds-page并分析染色后获得的凝胶,通过测定凝胶上的条带强度来测定。因此,可以相对于组合物中归于所有蛋白的条带强度之和确定归于沙门菌素的条带强度。组合物中的总蛋白含量可以使用公知的bradford蛋白测定法来测定。
[0563]
在一个实施方案中,本发明的组合物是药物组合物。除了本发明的一种或多种沙门菌素外,药物组合物可以任选地含有大肠杆菌大肠菌素,和/或一种或更多种合适的药学上可接受的赋形剂。
[0564]
本发明提供了预防或减少沙门氏菌对目标物的感染或污染的方法,包含使所述目标物与一种或更多种如上所述的蛋白(沙门菌素)或如上所述的组合物接触。目标物可以是任何非有机目标物或有机目标物如食物的表面。用沙门氏菌污染目标物意指活沙门氏菌细胞粘附到目标物上。减少沙门氏菌污染意指减少粘附于目标物的活沙门氏菌细胞的数量。确定沙门氏菌对目标物的污染是公知常识的一部分。例如,可以使用如实施例中所做的匀浆食物的溶液或分散液的稀释铺板或其它目标物的漂洗液的稀释铺板,随后计数细菌菌落。优选地,所述目标物是食物或动物饲料。食品可以是肉,如家禽的整个躯体、生肉、熟肉、肉末,蛋如生蛋、全蛋、去皮熟蛋、炒鸡蛋,生水果,或生或熟蔬菜。
[0565]
为了用蛋白或组合物处理或接触目标物,通常使如上所述的蛋白溶液或液体组合物与目标物接触。例如,所述目标物用水溶液喷洒或浸入作为本发明的组合物的水溶液中。将目标物浸入水溶液中至少10秒钟,优选至少1分钟,优选至少5分钟。使目标物与液体组合物接触有助于将组合物分布在目标物的表面上。当可以实现足够均匀的分布时,可以将目
标物与依据本发明的固体组合物接触,例如在绞肉时。
[0566]
本发明还提供了治疗有此需要的受试者的沙门氏菌感染的方法,包括对所述受试者施用一种或更多种如上所述的蛋白(沙门菌素)或如上所述的组合物。所述受试者可以是人类或哺乳动物,例如农场动物。农场动物的实例是禽和牛。通常,制备含有沙门菌素和任选的如上所述的其它成分的液体或固体药物组合物,以用于施用于动物或人。液体组合物可以是如上所述的水溶液。固体组合物可以是含有至少一种沙门菌素的粉末,例如冻干形式的粉末,或从这种粉末获得的片剂或用这种粉末填充的胶囊。施用可以是口服。在这种情况下,药物制剂是一种允许穿过胃而不受胃中酸性介质侵袭的药物制剂。然后,沙门菌素应在肠内从药物制剂中释放。这样的药物制剂是本领域已知的。实例是对胃中的酸性介质具有抗性的片剂和胶囊。还可以将含有表达的沙门菌素的生物物质如大肠杆菌或植物材料口服施用于患者。可以将沙门菌素以每天1mg至1000mg,优选每天10mg至250mg的量施用于成年人。这类量也可以施用于动物。在益生菌方法中,可以通过向患者施用表达至少一种沙门菌素的遗传修饰的微生物来治疗患者。遗传修饰的微生物可以是遗传修饰的非致病性大肠杆菌或通常用于乳制品发酵的产乳酸的微生物。产乳酸的微生物的实例是来自乳杆菌属(lactobacillus)的细菌,例如乳酸乳杆菌(lactobacillus lactis),和双歧杆菌属(bifidobacterium)的细菌,例如两歧双歧杆菌(bifidobacterium bifidum)或短双歧杆菌(bifidobacterium breve)。另一种施用途径是通过注射到患者的血流中以预防沙门氏菌感染。为此,可以将沙门菌素溶解在生理盐水中,并将溶液灭菌。
[0567]
在上述方法中,沙门氏菌是肠道沙门氏菌,优选肠道沙门氏菌肠道亚种。
[0568]
沙门菌素scole1a和scole1b针对沙门氏菌,特别是肠道沙门氏菌,优选肠道沙门氏菌肠道亚种的许多不同血清型具有特别广泛的活性,如以下实例中所证明的。因此,scole1a和scole1b或其衍生物优选用于治疗任意肠道沙门氏菌,优选任意肠道沙门氏菌肠道亚种的感染或用于预防或减少任意肠道沙门氏菌,优选任意肠道沙门氏菌肠道亚种的污染。沙门菌素e2、e3、e7和spst也具有针对靶沙门氏菌的广泛活性。然而,scole2及其衍生物可优选针对表5a和5b中定义的菌株1、3、4、15、20、22至30使用。scole3及其衍生物可优选针对表5a和5b中定义的菌株1、3、4、17和20至25使用。scole7及其衍生物可优选针对表5a和5b中定义的菌株1、3、4、5、15、20、22至30和32使用。
[0569]
依据本发明的沙门菌素可以在标准表达系统中通过已知的蛋白表达方法产生。为了产生沙门菌素,编码其的核苷酸序列可以在合适的宿主生物体中表达。现有技术中已经描述了可用于产生和纯化目的蛋白的方法,并且可以使用任意这样的方法。例如,可以使用本领域公知的大肠杆菌表达系统。如果使用真核表达系统,可以在沙门菌素的编码序列中插入一个或更多个内含子以防止对用于克隆的细菌生物体的毒性。
[0570]
特别有效的表达方法是现有技术中也已知的植物表达系统。根据本发明的可用于表达沙门菌素的植物表达系统描述于实施例中。实现目标核苷酸序列在植物中表达的一种可能方式是使用含有编码沙门菌素的核苷酸序列的自我复制(病毒)复制子。沙门菌素的编码序列可以是密码子优化的,用于在植物中或在用作表达宿主的特定植物中表达。植物病毒表达系统已在许多出版物中描述,例如在wo2012019660、wo2008028661、wo2006003018、wo2005071090、wo2005049839、wo2006012906、wo02101006、wo2007137788或wo02068664中,并且在这些文献中引用了更多的出版物。已知有各种方法可将核酸分子如dna分子导入植
物或植物部分中以瞬时表达。农杆菌可用于用核酸分子(载体)或核酸构建体转染植物,例如通过农杆菌浸润或用农杆菌悬液喷雾。作为参考,参见wo 2012019660、wo 2014187571或wo 2013149726。
[0571]
在需要作为目的蛋白的沙门菌素的强表达的实施方案中,含有编码沙门菌素的核苷酸序列的核酸构建体可编码可在植物细胞中复制以形成病毒载体复制子的病毒载体。为了进行复制,病毒载体和复制子可含有复制起点,其可被存在于植物细胞中的核酸聚合酶识别,例如被复制子表达的病毒聚合酶识别。在rna病毒载体(称为"rna复制子")的情况下,在dna构建体导入植物细胞核之后,复制子可通过在植物细胞中有活性的启动子的控制下,从dna构建体转录形成。在dna复制子的情况下,复制子可通过在dna构建体中编码病毒复制子的序列侧翼的两个重组位点之间重组而形成,例如如在wo00/17365和wo 99/22003中所述。如果复制子由dna构建体编码,则优选rna复制子。多年来,文献中广泛描述了dna和rna病毒载体(dna或rna复制子)的使用。一些实例是下列专利出版物:wo2008028661、wo2007137788、wo 2006003018、wo2005071090、wo2005049839、wo02097080、wo02088369、wo02068664。dna病毒载体的实例是那些基于双生病毒的载体。对于本发明,可以优选使用基于植物rna病毒的病毒载体或复制子,特别是那些基于正义单链rna病毒。因此,病毒复制子可以是正义单链rna复制子。这类病毒载体的实例是那些基于烟草花叶病毒(tmv)和马铃薯x病毒组x(pvx)。"基于"是指病毒载体使用复制系统,例如参与这些病毒复制的复制酶和/或其它蛋白。基于马铃薯x病毒组的病毒载体和表达系统描述于ep2061890或wo2008/028661中。
[0572]
沙门菌素可以在多细胞植物或其部分,特别是高等植物或其部分中表达。单子叶和双子叶(作物)植物都可以使用。可用于表达目的蛋白的常见植物包括本氏烟草、烟草(nicotiana tabacum)、菠菜、油菜(brassica campestris)、芥菜(b.juncea)、甜菜(甜菜(beta vulgaris))、水芹、田芥、芥菜、草莓、头状藜(chenopodium capitatum)、莴苣、向日葵、黄瓜、大白菜、卷心菜、胡萝卜、葱、洋葱、萝卜、莴苣、豌豆、花椰菜、牛蒡、芜菁、番茄、茄子、南瓜、西瓜、王子瓜和甜瓜。优选的植物是菠菜、叶甜菜、甜菜根、胡萝卜、糖用甜菜、烟草和本氏烟草。在可食用植物中的表达可用于防止沙门氏菌污染植物或由其制备的食物。在一个实施方案中,使用通常不进入人或动物食物链的植物,例如烟草属物种,例如烟草和本氏烟草。
[0573]
通常,作为目的蛋白的沙门菌素在植物或植物部分的细胞的细胞质中表达。在这种情况下,没有将指导目的蛋白进入特定区室的信号肽加入到蛋白中。或者,目的蛋白可在植物叶绿体中表达或靶向植物叶绿体;在后一种情况下,将通常称为质体转运肽或叶绿体靶向肽的n-末端前序列作为目的蛋白加入到沙门菌素的n-末端或c-末端,优选n-末端。
[0574]
如实验部分所述,沙门菌素可以与免疫蛋白一起共表达,特别是如果沙门菌素具有核酸酶活性,为了防止对植物组织的毒性。可共表达的合适的免疫蛋白是下表2中给出的那些。
[0575]
在生产含有至少一种沙门菌素的组合物的方法中,在第一步中,沙门菌素在植物或植物细胞如可食用植物中表达。在下一步中,收获来自具有表达的沙门菌素的植物的含有表达的沙门菌素的植物材料。植物材料可以例如是叶、根、块茎或种子,或叶、根、块茎或种子的压碎、磨碎或粉碎的产物。在步骤(iii)中,使用含水缓冲液从植物材料中提取沙门
菌素。这可以包括将植物材料均质化,并且可以通过离心或过滤除去不溶性物质。包括沙门菌素的可溶性组分将被提取到含水缓冲液中以在含水缓冲液中产生沙门菌素溶液。含水缓冲液可以含有无机酸或有机酸或其盐,并且可以具有如上文对作为本发明组合物的水溶液所定义的ph。另外,含水缓冲液可以含有盐和/或巯基化合物,也如上文对作为本发明组合物的水溶液所述。如果需要相对纯的沙门菌素组合物,可以根据已知的蛋白纯化方法通过在步骤(iv)中除去不需要的成分来进一步纯化在含水缓冲液中的沙门菌素溶液。
[0576]
因此,本发明提供了一种生产包含依据本发明蛋白的组合物的方法,所述方法包含以下步骤:
[0577]
(i)在如上所述的植物中表达所述蛋白,优选可食用植物或烟草,
[0578]
(ii)从所述植物收获含有表达的蛋白的植物材料,
[0579]
(iii)用含水缓冲液从所述植物材料中提取所述蛋白,以获得含有所述蛋白的组合物,
[0580]
任选地从所述组合物中除去不需要的污染物。
[0581]
如果在植物中表达沙门菌素,则收获具有表达的蛋白的植物或其组织,可以将组织匀浆,并且可以通过离心或过滤除去不溶性物质。如果需要相对纯的沙门菌素,可以通过通常已知的蛋白纯化方法进一步纯化沙门菌素,例如通过层析方法可除去其它宿主细胞蛋白和诸如生物碱和多酚的植物代谢物。纯化的沙门菌素溶液可以被浓缩和/或冻干。
[0582]
如果在可食用植物中表达沙门菌素,来自可食用植物的粗蛋白提取物或半纯化浓缩物可用于防止或减少沙门氏菌对诸如食物的目标物的污染。
[0583]
优选实施方案
[0584]
一种蛋白,其氨基酸序列包含氨基酸序列(a-x)、(b-x)、(c-x)、(d-x)或(e-x)中的任一个或由其组成。
[0585]
一种蛋白,其氨基酸序列包含氨基酸序列(a-xi)、(b-xi)、(c-xi)、(d-xi)或(e-xi)中的任一个或由其组成。
[0586]
一种蛋白,其氨基酸序列包含氨基酸序列(a-xii)、(b-xii)、(c-xii)、(d-xii)或(e-xii)或由其组成。
[0587]
根据前述三个优选实施方案(句子)中任一项所述的蛋白,其中所述蛋白如(b-x)、(c-x)、(d-x)、(e-x)、(b-xi)、(c-xi)、(d-xi)、(e-xi)、(b-xii)、(c-xii)、(d-xii)或(e-xii)中任一项所定义,优选如(b-xi)、(c-xi)、(d-xi)、(e-xi)、(b-xii)、(c-xii)、(d-xii)或(e-xii)中任一项所定义,并且
[0588]
其中所述蛋白的对应于seq id no:33的残基155的氨基酸残基是pro和/或对应于seq id no:33的残基246的氨基酸残基是arg或lys,优选arg,并且
[0589]
其中对应于seq id no:33的残基76和84的氨基酸残基均为gln。
[0590]
一种蛋白(seq id no:26的scole1d或其衍生物),其氨基酸序列包含氨基酸序列(a-viii)、(b-viii)、(c-viii)、(d-viii)或(e-viii)中的任一个,任选地如上文关于对应于seq id no:4的氨基酸残基进一步定义的,或由其组成。
[0591]
一种组合物,所述组合物包含其氨基酸序列包含氨基酸序列(a-x)、(b-x)、(c-x)、(d-x)或(e-x)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白,以及其氨基酸序列包含氨基酸序列(a-v)、(b-v)、(c-v)、(d-v)或(e-v)中的任一个
或由其组成的蛋白;
[0592]
一种组合物,所述组合物包含其氨基酸序列包含氨基酸序列(a-xi)、(b-xi)、(c-xi)、(d-xi)或(e-xi)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白,以及其氨基酸序列包含氨基酸序列(a-v)、(b-v)、(c-v)、(d-v)或(e-v)中的任一个或由其组成的蛋白;
[0593]
一种组合物,所述组合物包含其氨基酸序列包含氨基酸序列(a-xii)、(b-xii)、(c-xii)、(d-xii)或(e-xii)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白,以及其氨基酸序列包含氨基酸序列(a-v)、(b-v)、(c-v)、(d-v)或(e-v)中的任一个或由其组成的蛋白;
[0594]
一种组合物,所述组合物包含其氨基酸序列包含氨基酸序列(a-x)、(b-x)、(c-x)、(d-x)或(e-x)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白,以及其氨基酸序列包含氨基酸序列(a-iv)、(b-iv)、(c-iv)、(d-iv)或(e-iv)中的任一个或由其组成的蛋白;
[0595]
一种组合物,所述组合物包含其氨基酸序列包含氨基酸序列(a-xi)、(b-xi)、(c-xi)、(d-xi)或(e-xi)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白,以及其氨基酸序列包含氨基酸序列(a-iv)、(b-iv)、(c-iv)、(d-iv)或(e-iv)中的任一个或由其组成的蛋白;
[0596]
一种组合物,所述组合物包含其氨基酸序列包含氨基酸序列(a-xii)、(b-xii)、(c-xii)、(d-xii)或(e-xii)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白,以及其氨基酸序列包含氨基酸序列(a-iv)、(b-iv)、(c-iv)、(d-iv)或(e-iv)中的任一个或由其组成的蛋白。
[0597]
前述句子中任一项所述的蛋白或组合物,其用于治疗受试者被沙门氏菌感染的方法中。
[0598]
一种预防或减少目标物被沙门氏菌感染或污染的方法,包括使所述目标物与前述句子中任一项所定义的蛋白或前述句子中任一项所定义的组合物接触。
[0599]
一种预防或减少目标物被沙门氏菌感染或污染的方法,包括使所述目标物与蛋白接触,所述蛋白包含氨基酸序列(a-x)、(b-x)、(c-x)、(d-x)或(e-x)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成,或使所述目标物与一种组合物接触,所述组合物包含其氨基酸序列包含氨基酸序列(a-x)、(b-x)、(c-x)、(d-x)或(e-x)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白以及其氨基酸序列包含氨基酸序列(a-v)、(b-v)、(c-v)、(d-v)或(e-v)中的任一个或由其组成的蛋白。
[0600]
一种预防或减少目标物被沙门氏菌感染或污染的方法,包括使所述目标物与蛋白接触,所述蛋白的氨基酸序列包含氨基酸序列(a-xi)、(b-xi)、(c-xi)、(d-xi)或(e-xi)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成,或使所述目标物与组合物接触,所述组合物包含其氨基酸序列包含根据(a-xi)、(b-xi)、(c-xi)、(d-xi)或(e-xi)的氨基酸序列中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白和其氨基酸序列包含氨基酸序列(a-v)、(b-v)、(c-v)、(d-v)或(e-v)中的任一个或由其组成的蛋白。
[0601]
一种预防或减少目标物被沙门氏菌感染或污染的方法,包括使所述目标物与蛋白
接触,所述蛋白的氨基酸序列包含氨基酸序列(a-xii)、(b-xii)、(c-xii)、(d-xii)或(e-xii)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成,或使所述目标物与组合物接触,所述组合物包含其氨基酸序列包含氨基酸序列(a-xii)、(b-xii)、(c-xii)、(d-xii)或(e-xii)中的任一个(任选地,如上文关于特定氨基酸残基进一步定义的)或由其组成的蛋白和其氨基酸序列包含氨基酸序列(a-v)、(b-v)、(c-v)、(d-v)或(e-v)中的任一个或由其组成的蛋白。
[0602]
一种治疗有需要的受试者的沙门氏菌感染的方法,包括向所述受试者施用如前述句子中任一项所定义的蛋白或前述句子中任一项所定义的组合物。
[0603]
这些优选实施方案可以与本文描述的其他实施方案或优选实施方案组合。
实施例
[0604]
实施例1:质粒构建体(沙门菌素)
[0605]
选择代表四个活性组的六种沙门菌素(表1)
[0606]
表1.实施例中使用的沙门氏菌细菌素(沙门菌素)列表
[0607][0608][0609]
该列表包含沙门菌素scole2、scole3、scole7、scole1a、scole1b和spst。从genbank中检索出各自的氨基酸序列;由thermo fisher scientific inc.合成具有针对本氏烟草优化的密码子使用的相应核苷酸序列。在沙门菌素scole2、scole3和scole7的情况下,通过插入cat 1内含子(针对过氧化氢酶cat1的来自蓖麻(ricinus communis)cat1基因的第一个内含子(genbank:d21161.1,679和867之间的核苷酸位置))中断编码序列以防止在用于克隆的大肠杆菌细胞中的细胞毒性。将沙门菌素编码序列插入基于tmv的组装病毒载体pnmd035中(详细描述于wo2012/019660中),得到图1a-b中所示的质粒构建体。
[0610]
在初步表达研究中,发现具有核酸酶(rna酶和dna酶)活性的细菌素通常对表达它们的植物组织具有高毒性。它们的表达导致组织坏死和重组蛋白的积累不良。然而,与适当的免疫蛋白共表达降低了毒性作用并显著增加了这些细菌素的积累。我们研究中使用的沙门菌素免疫蛋白列于表2中。
[0611]
表2.实施例中使用的免疫蛋白列表
[0612]
no.免疫蛋白特异性登录号seq id no:1simme2scole2(dna酶)ktm78571.17
2simme7scole7(dna酶)ksu39546.18
[0613]
免疫蛋白simme2和simme7分别用于沙门菌素scole2和scole7。从genbank中检索到免疫蛋白的氨基酸序列;通过thermo fisher scientific inc.合成具有针对本氏烟草优化的密码子使用的相应核苷酸序列,并如wo2012/019660中所述亚克隆到基于pvx的组装病毒载体pnmd670中。得到的质粒构建体如图1a所示。
[0614]
实施例2:沙门菌素表达筛选
[0615]
使用无针注射器将携带基于tmv的组装载体的稀释的根癌农杆菌(agrobacterium tumefaciens)培养物渗入六周龄的本氏烟草植物,用于细胞溶质沙门菌素表达。在沙门菌素scole2和scole7的情况下,将用于沙门菌素表达的携带基于tmv的载体的农杆菌培养物与其他携带用于表达相应的免疫蛋白的基于pvx的载体的培养物以相等的比例混合。将各个过夜培养物调节至od
600
=1.5,并用含有10mm mes,ph 5.5和10mm mgso4的浸润缓冲液以1:100进一步稀释。表3中总结了在该实验中使用的质粒构建体。为了确定最佳收获时间点,在浸润后的几个时间点收获植物材料并用于用5倍体积的含有50mm hepes(ph7.0),10mm乙酸钾,5mm乙酸镁,10%(v/v)甘油,0.05%(v/v)tween-20和300mm nacl的缓冲液进行蛋白提取。使用bradford测定法测定总可溶性蛋白(tsp)浓度,并使用sds-page用考马斯染色分析tsp提取物。在我们的实验中,如通过与牛血清白蛋白(bsa)蛋白的比较测定的,所有检测的沙门菌素在相当高的水平上表达,在1.2和1.8mg重组大肠菌素/g fw之间或在18-47%tsp之间变化(表4)。
[0616]
表3.沙门菌素表达筛选小结
[0617]
no.沙门菌素构建体构建体(特征)1scole2/simme2pnmd28161/pnmd28222tmv/pvx2scole3pnmd28151tmv3scole7/simme7pnmd28172/pnmd28232tmv4scole1apnmd28191tmv/pvx5scole1bpnmd28204tmv/pvx6spstpnmd28182tmv
[0618]
表4.本氏烟草植物中表达的重组沙门菌素的产量。fw代表鲜重,tsp表示总可溶性蛋白,dpi表示浸入后天数,av表示平均值,sd表示标准偏差,n表示独立实验数。
[0619][0620]
[0621]
实施例3:沙门菌素活性筛选
[0622]
我们分析了植物产生的重组沙门菌素针对肠道沙门氏菌肠道亚种的33个不同的血清型的36个菌株的抗微生物活性。在表5a和5b中给出了实验中使用的菌株的详细信息。
[0623]
表5a.用于抗微生物活性筛选的肠道沙门氏菌肠道亚种菌株。
[0624][0625][0626]
表5b.用于抗微生物活性筛选的肠道沙门氏菌肠道亚种菌株。
20ml lb琼脂培养基(1.5%w/v琼脂)倒入10
×
10cm四方(quadratic)培养皿中。将lb软琼脂培养基(0.8%(w/v)琼脂)融化,将20ml等分试样转移到50ml塑料管中,并将它们的温度调节至50至55℃。将用lb培养基调节至od
600
=1.0的沙门氏菌过夜培养物以1:100的比例加入到软琼脂培养基中,得到最终的od
600
=0.01或约1
×
107个细胞/ml,并将20ml含有沙门氏菌测试菌株的lb软琼脂倒入预浇lb平板上,得到0.14ml每平方厘米1
×
107cfu/ml的细菌溶液。
[0630]
如实施例2中所述提取植物叶材料。我们通过使用相同的提取缓冲液从未稀释的样品开始制备1:1稀释组的植物提取物。将5μl等分的tsp稀释系列应用于琼脂平板;将板在37℃下过夜孵育。基于清除区(clearing zone)评价抗微生物活性。
[0631]
在6个测试的沙门菌素中,一个显示出窄的抗微生物活性(spst
–
12%的菌株被抑制),一个沙门菌素具有中等活性谱(scole3-60%的菌株被抑制),另外4个具有广泛的活性谱:scole2和scole7-抑制大约90%的菌株和scolela和scolelb-抑制了100%的菌株(图3)。
[0632]
沙门菌素scole1和scolelb表现出针对测试的沙门氏菌菌株的不仅广泛而且非常高的活性(图4、5)。
[0633]
对于半定量比较,我们展示了重组大肠菌素在任意单位(au)下的相对抗微生物活性,计算为蛋白提取物在径向扩散测定中引起可检测清除效应的最高稀释度的稀释因子。在图6、8、10、12和14中分别显示了scole2、scole3、scole7、scole1a和scole1b的以au/mg fw植物组织来计算的针对沙门氏菌菌株的沙门菌素抗微生物活性。因此,每单位生物量的特定活性剂的产量;即反映了宿主的特定的生产能力。
[0634]
图7、9、11、13和15分别显示了以au每μg重组沙门菌素蛋白scole2、scole3、scole7、scole1a和scole1b计算的相同活性,反映了沙门菌素的抗微生物比效力。
[0635]
实施例4:质粒构建体(大肠菌素)
[0636]
选择代表两个活性组的六种大肠菌素(表6)。该列表包括大肠菌素cois4、coi5、coi10、coiia、coiib和coim。从genbank中检索出相应的氨基酸序列;由thermo fisher scientific inc.合成具有针对本氏烟草优化的密码子使用的相应核苷酸序列。将大肠菌素编码序列插入基于tmv的组装病毒载体pnmd035(在wo2012/019660中详细描述)中,得到图16中所示的质粒构建体。通过插入cat 1内含子(来自过氧化氢酶cat1的蓖麻cat1基因的第一个内含子(genbank:d21161.1,核苷酸位置在679和867之间))中断大肠菌素m的编码序列。
[0637]
表6.实施例中使用的大肠杆菌细菌素(大肠菌素)列表。
[0638][0639]
实施例5:大肠菌素表达筛选
[0640]
使用无针注射器将携带基于tmv的组装载体的稀释的农杆菌培养物渗入六周龄的本氏烟草植物,用于细胞溶质大肠菌素表达。将农杆菌过夜培养物调节至od
600
=1.5,并用含有10mm mes,ph 5.5和10mm mgso4的浸润缓冲液以1:100进一步稀释。在表7中总结了在该实验中使用的质粒构建体。为了确定最佳收获时间点,在浸润后的几个时间点收获植物材料并用于用5倍体积的含有50mm hepes(ph7.0),10mm乙酸钾,5mm乙酸镁,10%(v/v)甘油,0.05%(v/v)tween-20和300mm nacl的缓冲液进行蛋白提取。使用bradford测定法测定总可溶性蛋白(tsp)浓度,并使用sds-page用考马斯染色分析tsp提取物。在我们的实验中,如通过与牛血清白蛋白(bsa)蛋白的比较测定的,所有测试的大肠菌素在相当高的水平上表达,在1.5-4.7mg重组大肠菌素/g fw或在16-41%tsp之间变化(表8)。
[0641]
表7.大肠菌素表达筛选小结
[0642]
no.大肠菌素构建体构建体(特征)1cols4pnmd25856tmv2col5pnmd15311tmv3col10pnmd25848tmv4coliapnmd19141tmv5colibpnmd25861tmv6colmpnmd10221tmv
[0643]
表8.在本氏烟草植物中表达的重组大肠菌素的产量。fw代表鲜重,tsp表示总可溶性蛋白,dpi表示浸润后数天,av表示平均值,sd表示标准偏差,n表示独立实验数。
[0644]
[0645][0646]
实施例6:大肠菌素活性筛选
[0647]
我们分析了植物产生的重组大肠菌素针对肠道沙门氏菌肠道亚种的32个不同的血清型的35个菌株的抗微生物活性。在表5a和5b(菌株编号1至35)中给出了在我们的实验中使用的菌株的详细信息。
[0648]
如实施例3中所述,在径向扩散测定中通过斑点法测试含重组大肠菌素的植物提取物的抗微生物活性。
[0649]
在6种测试的大肠菌素中,一种显示出窄的抗微生物活性(cois4
–
抑制25%的菌株),三种大肠菌素具有中等活性谱(coi5、coi10和coim-分别抑制48%、46%和42%的菌株),两种大肠杆菌具有广泛的活性谱:coiia和coiib-分别抑制96%和89%的菌株(图17)。
[0650]
实施例7:细菌素(大肠菌素混合物)对应用于肉基质的肠道沙门氏菌肠道亚种的致病菌株的杀菌作用的效果测定
[0651]
测试植物产生的大肠菌素对受致病性沙门氏菌污染的鸡胸肉样品的抗菌活性。
[0652]
效果评价包含在受污染的肉样品上分析致病性肠道沙门氏菌肠道亚种群,随后用植物产生的重组大肠菌素的混合物或由来自相同生产宿主但没有大肠菌素的植物提取物组成的对照载体溶液处理,并且在4℃下将处理过的肉样品储存不同时间段。
[0653]
使用没有特殊来源的肉类样品以确保在代表性消费品中评价细菌素活性。在实验前一天,在零售店(用于这些研究,aldi超市,halle,德国)购买生鸡胸肉片。将肉储存在4℃,在实验暴露之前不对肉进行清洗或预处理。
[0654]
用分别代表血清型鼠伤寒沙门氏菌和肠炎沙门氏菌(9270
tm
*,13076
tm
*)的2种肠道沙门氏菌肠道亚种菌株1:1的混合物或用分别代表鼠伤寒沙门氏菌、肠炎沙门氏菌、纽波特和anatum的4种肠道沙门氏菌肠道亚种菌株1:1:1:1的混合物(9270
tm
*、13076
tm
*、6962
tm
*和9270
tm
*)实验性污染肉测试基质(分别为图18和19)。在肉污染之前,单独培养菌株至od
600
=0.3并以1:1或1:1:1:1混合。用lb液体培养基将菌株混合物进一步稀释至所需的细胞数(od
600
=0.005至0.001,2
×
106至1.8
×
105cfu/ml),用作肉污染悬浮液,以获得~2
×
104cfu/g肉的初始接种物。将鸡胸肉切片(~25克重量的3片)浸入12ml细菌悬浮液中并翻转并再次浸入以接种两面。允许受污染的肉和细菌在室温下分别干燥和定殖基质样品30分钟,在此期间每15分钟翻转鸡胸肉切片。
[0655]
通过使用雾化瓶低压喷洒(2-4bar)载体或大肠菌素混合物溶液(从未处理的植物材料或用用于大肠菌素表达的农杆菌注射接种的植物材料的本氏烟草制备tsp提取物,50mm hepes ph7.0,10mm乙酸钾,5mm乙酸镁,10%(v/v)甘油,0.05%(v/v)tween-200,300mm nacl)来处理污染的肉。对于大肠菌素m,建议施用率为3mg/kg,对于混合物中使用的任何其他大肠菌素(大肠菌素ia和大肠菌素5),施用率为1mg/kg。将肉在室温下进一步孵育30分钟,同时每15分钟翻转一次。
[0656]
在应用大肠菌素30分钟后,将等分的~25g鸡胸肉切片置于无菌样品袋(400p),一式两份操作,记录每个样品的精确重量,并使用闭合夹闭合样品袋(
400p)。总之,肉样品在大肠菌素处理后在室温下孵育1小时,然后将密封的肉样品储存在4℃。
[0657]
对在4℃下储存1小时、48小时和72小时的肉样品进行取样,以测定基质上微生物污染水平。为了从肉样品中回收致病性沙门氏菌,向每等分~25g肉样品中加入~100ml缓冲蛋白胨水。将样品在实验室混合器(400cc;设置:间隙0,时间30秒,速度4)中均质处理。收集来自储存袋的过滤部分的微生物悬浮液,并制备1:10稀释组(dilution series)。将100μl等分未稀释或稀释的微生物悬浮液涂布在xld琼脂上。将板在37℃下孵育18至24小时,并计数cfu(菌落形成单位)。每g样品的cfu数目计算如下:
[0658][0659]
通过单向方差分析(one-way anova)(tukey's多重比较测试)和使用graphpad prism v.6.01的非配对参数t-检验比较用载体处理的对照样品和大肠菌素处理的样品获得的数据来评价大肠菌素处理减少实验性污染的肉样品中的活致病性沙门氏菌的数目的效力。
[0660]
图18和19分别显示二菌株或四菌株沙门氏菌污染的细菌计数结果。在大肠菌素处理后储存1小时后,已经发生细菌群的最显著减少(1.8个对数)。
[0661]
总之,通过用大肠菌素混合物处理肉,可以在受污染的肉上实现沙门氏菌群的统计学上显著的减少。
[0662]
测试沙门菌素混合物或沙门菌素/大肠菌素混合物对食物产品的去沙门氏菌污染,并计划用于食品工业以减少沙门氏菌污染。沙门菌素scole1a和scole1b显示出针对测试的沙门氏菌菌株最广泛的抗微生物活性。因此,它们可以用作沙门菌素混合物的主要成分,用于控制沙门氏菌。
[0663]
实施例8:在稳定的转基因宿主中产生沙门菌素
[0664]
使用用于沙门菌素表达的t0代转基因植物的分离叶的用于etoh可诱导的转基因表达和诱导的载体,通过农杆菌介导的叶盘转化来转化本氏烟草。这是按照schulz等人.proc.natl.acad.sci.usa.112,e5454-e5460(2015)的描述来完成。
[0665]
稳定的转基因本氏烟草植物(含有基于tmv的病毒载体的基因组插入,其用乙醇双重可诱导scolelb表达(图21))表现出正常的生长和发育,并且选择的转基因品系在用乙醇诱导后累积的沙门菌素达到预期水平(图22)。
[0666]
实施例9:菠菜中沙门菌素的产生
[0667]
菠菜栽培种fr
ü
hes riesenblatt(spinacia oleracea cv.fr
ü
hes riesenblatt)植物在温室中生长(白天和夜晚温度分别为19至23℃和17至20℃,12小时光照和35至70%湿度)。如实施例2中所述,将6周龄的植物用于注射器浸润。使用sds-page用考马斯染色确认重组蛋白的表达(图23)。
[0668]
实施例10:扩展的沙门菌素活性筛选
[0669]
我们进一步分析了如实施例6中所述的植物产生的重组沙门菌素针对其他沙门氏菌菌株的抗微生物活性。为了确定沙门菌素抗微生物活性谱,选择并筛选代表105个肠道沙门氏菌肠道亚种血清型的109个菌株(表9)。筛选包括在美国疾病控制和预防中心(cdc)记
录(www.cdc.gov/nationalsurveillance/pdfs/salmonella-annual-report-2013-508c.pdf)的从2003至2012年引起至少100例人沙门氏菌感染的所有血清型(除了血清型typhi和14,5:12:r:-)每一种的一个菌株,血清型鼠伤寒沙门氏菌、肠炎沙门氏菌和javiana沙门氏菌的两个菌株和6种导致发病率低于100的血清型或未向cdc报告的血清型。
[0670]
表9.用于抗微生物药物敏感性分析的肠道沙门氏菌肠道亚种菌株列表。血清型抗原配方如供应商所提供的,以(亚种[空间]o抗原[结肠]1期h抗原[结肠]2期h抗原)中给出。供应来源中的数字对应于1-microbiologics,inc.(st.cloud,美国),2-lgc标准(teddington,英国),3-robert koch institute,沙门氏菌病和其他肠道病原体国立参考中心(national reference centre for salmonellosis and other enteric pathogens)(wernigerode,德国),4-leibnitz institute dsmz
–
德国微生物和细胞培养物保藏中心(german collection of microorganisms and cell cultures)(braunschweig,德国),5
–
国立典型培养物保藏中心(national collection of type cultures)(salisbury,英国)。菌株标有“用于一式三份重复的抗微生物药敏试验实验。发病次数是指在2003-2012年报告至cdc并发表在national enteric disease surveillance:salmonella annual report,2013(cdc,2016年6月;www.cdc.gov/nationalsurveillance/pdfs/salmonella-annual-report-2013-508c.pdf.中的实验室确认的人沙门氏菌感染(us)。
[0671]
[0672]
[0673]
[0674]
[0675]
[0676][0677]
表10.本研究使用的大肠杆菌stec菌株列表
[0678][0679]
为了评估活性谱的宽度,对所有菌株进行至少一次测试,随后分别用沙门菌素和大肠菌素在一式三份重复实验中重复筛选36或35个菌株(图24)。对于沙门菌素scole1a和scole1b,再次鉴定了最广泛的抗微生物活性谱,其分别针对100%和99%所评价的所有菌株显示出阳性抗微生物活性。对于沙门菌素scole2(94%)、scole3(70%)和scole7(95%)也观察到显著的活性宽度,如它们对图24e中所示的36个菌株子集的活性所反映的。
[0680]
基于它们控制主要致病性沙门氏菌菌株的能力,将分析的五种沙门菌素分为四组。沙门菌素scole1a和scole1b具有普遍活性,每一种均能够杀死所有测试的致病变型并且在所有测试的菌株上显示出高于105au/μg重组蛋白的最高平均活性(图24a)并且在大多数情况下对各个菌株为高于103au/μg蛋白。剩余的沙门菌素分为两组,其中一组中的沙门菌素scole2和scole7具有低100倍的平均活性(《105au/μg蛋白,图24a),而另一组中的scole3显示了显著更低的平均活性(102au/μg,图24a)。
[0681]
与沙门菌素抑制致肠病肠道沙门氏菌菌株的高效力相比,大肠菌素ia、ib、m、5、10和s4(表6)的比活性低2至4个数量级(2-3log au/μg,图24b),尽管109个菌株大部分受到大
肠菌素ia(92%)和ib(90%)的抑制,大约三分之一的菌株受到大肠菌素s4(45%)、5(25%)、10(29%)和m(34%)的抑制,也反映在35个菌株子集的易感性模式中(图24f)。一般来说,针对沙门氏菌,沙门菌素表现出比大肠杆菌大肠菌素更高和更广泛的活性。相反,沙门菌素针对大肠杆菌stec(表10)菌株显示出低(低于102au/μg)的种间特异性和窄的活性(图24c,g)。
[0682]
实施例11:单独的沙门菌素scole1a和沙门菌素混合物控制受污染的鸡肉基质上的沙门氏菌
[0683]
在模拟研究中分析了植物产生的单独的沙门菌素scole1a以及沙门菌素混合物控制沙门氏菌污染的肉表面的杀菌效力。
[0684]
鸡胸肉片从当地超市购买。肠道沙门氏菌肠道变种血清型肠炎沙门氏菌(菌株13076
tm*
)、鼠伤寒沙门氏菌菌株(菌株14028
tm*
)、纽波特沙门氏菌菌株(菌株6962
tm*
)、javiana沙门氏菌菌株(菌株10721
tm*
)、海德堡沙门氏菌菌株(菌株8326
tm*
)、婴儿沙门氏菌菌株(菌株baa-1675
tm*
)和慕尼黑沙门氏菌菌株(菌株8388
tm*
)的萘啶酸抗性突变体在补充有25μg/ml萘啶酸的lb培养基中单独生长至静止期,用新鲜lb稀释并生长至指数期。对于禽肉的污染,用lb培养基稀释细菌培养物至od
600
=0.001(~2
×
105cfu/ml)并以1:1:1:1:1:1:1混合。将切成约20g重块的一定量的鸡胸肉片在室温下以每100g肉~2
×
105cfu/ml密度接种1ml的7个肠道沙门氏菌菌株的混合物,得到肉基质的初始污染水平,为约3log cfu/g致病性肠道沙门氏菌的7个血清型的混合物;在室温下允许细菌附着到肉表面30分钟。随后,通过喷洒(10ml/kg)植物提取物对照(不含沙门菌素的wt本氏烟草植物材料的tsp提取物,用50mm hepes ph7.0,10mm乙酸钾,5mm乙酸镁,10%(v/v)甘油,0.05%(v/v)tween-20,300mm nacl制备)或浓度为3mg/kg的scole1a、或3mg/kg的scole1a、1mg/kg的scolelb、1mg/kg的scole2、1mg/kg的scole7或0.3mg/kg的scole1a、0.1mg/kg的scole1b、0.1mg/kg的scole2和0.1mg/kg的scole7的沙门菌素溶液(用与植物提取物对照相同的缓冲液制备的表达scole1a、scole1b、scole2和scole7的本氏烟草植物材料的tsp提取物的个体或混合物)处理鸡胸肉片。将处理的肉片在室温下进一步孵育30分钟。将相当于~40g的肉片等分装入400p无菌袋(interscience)中并在10℃下储存1h、1d和3d,这代表了对于细菌生长是许可的但不是最理想的实际工业肉类加工条件。
[0685]
总之,在沙门菌素处理期间将肉样品在室温下孵育1.5小时,然后将它们密封并在10℃下储存。为了分析细菌群,使用bag均质器(设置:间隙0,时间30秒,速度4;interscience)用4倍体积蛋白胨水将禽肉等分试样进行均质化并且在用连续稀释的微生物悬浮液涂布后,在补充有25μg/ml萘啶酸的xld培养基(sifin diagnostics)计数肠道沙门氏菌的菌落形成单位(cfu)。一式四份分析样品。
[0686]
通过比较用载体处理的对照样品和沙门菌素处理的样品获得的数据,通过具有6个自由度的双尾不成对参数t检验使用graphpad prism v.6.01来评价沙门菌素处理在减少实验性污染的肉样品中的活的致病性沙门氏菌数量方面的效力。
[0687]
评估了沙门菌素处理对沙门菌素处理的(分别为施用率为3mg/kg肉的个体scole1a和施用率为3+1+1+1mg/kg肉的由scole1a+scole1b+scole2+scole7组成的沙门菌
素混合物)致病性细菌群水平降低程度的效力,发现在所有分析的时间点上肉类样品和2-3log cfu/g肉中的活菌计数的统计学上显著的净减少两者都与植物提取物对照处理相关(图25)。在4种沙门菌素混合物(浓度为3+1+1+1mg/kg肉)中观察到细菌群的最高水平减少,在储存48小时后与载体处理比较达到3.39平均log减少,这对应于细菌平均值减少99.6%。单一的沙门菌素scolela(以3mg/kg肉施用)能够控制肉类上的沙门氏菌污染,其效果与以双倍浓度(6mg/kg肉总沙门菌素)施用的四种沙门菌素的混合物相似。甚至用非常低剂量的沙门菌素处理(总沙门菌素0.6mg/kg肉;scole1a+scole1b+scole2+scole7的混合物,0.3+0.1+0.1+0.1mg/kg肉)也产生统计学上显著的细菌群减少,最长储存48小时时大约1log cfu。在最初减少细菌污染后,在72小时后观察到活细菌的再生长,表明沙门菌素迅速起作用但对食物没有长期的技术影响。
[0688]
实施例12:植物正确表达重组沙门菌素
[0689]
通过基质辅助激光解吸/电离(maldi)飞行时间(tof)质谱(ms)分析包含在植物tsp提取物中的植物表达的重组沙门菌素的包括翻译后修饰在内的一级结构。
[0690]
为了蛋白水解消化,将使用5倍体积的20mm柠檬酸钠,20mm nah2po4,30mm nacl,ph 5.5从表达沙门菌素的植物材料制备的tsp提取物进行sds-page并切下含有5μg蛋白的考马斯染色sds凝胶条带,并通过用100mm nh4hco3和乙腈(acn)/h2o(50;50,v/v)中的100mm nh4hco3连续洗涤而脱色。在50℃下用10mm dtt还原二硫键45分钟,然后用10mg/ml碘乙酰胺烷基化60分钟。然后将脱色和烷基化的凝胶条带用不同的测序级内切蛋白酶(promega,madison,美国)进行蛋白水解消化。在消化溶液中将蛋白酶:蛋白比率调整至1:20(w/w),在25℃(胰凝乳蛋白酶)或37℃(asp-n,glu-c,lys-c,胰蛋白酶)进行消化12小时。通过分别用h2o、acn/h2o/三氟乙酸(50;45;5,v/v/v)和acn连续洗涤来提取蛋白水解肽。合并提取溶液,在真空离心机中浓缩,并在h2o/乙酸(90;10,v/v)中重新溶解。
[0691]
通过固相萃取使用c4或c18键合的二氧化硅材料(millipore,darmstadt,德国)纯化如上所述获得的蛋白水解的沙门菌素肽或纯化的完整的植物产生的沙门菌素scole1a,scolelb和scole7蛋白用于质谱分析,并将洗脱溶液与2,5-二羟基苯乙酮以及2,5-二羟基苯甲酸基质(bruker daltonics,bremen,德国)在maldi地面钢靶上共结晶。
[0692]
在maldi-tof/tof质谱仪(autoflex speedtm,bruker daltonics,bremen,德国)上获得质谱,其具有用于分子量测定的线性模式的正极性和用于通过in源衰变(isd)分析的蛋白测序的反射器模式。用nd:yag激光器(smart beam-iitm,bruker daltonics,bremen,德国)以355nm的发射波长并设定脉冲速率为1khz照射基质晶体。
[0693]
用flexcontrol(3.4版,bruker daltonics,bremen,德国)分别通过累积至少5000或10000激光照射(每个样品点)记录ms和ms/ms光谱。激光能量设定为略高于ms实验的阈值,并为ms/ms分析设定为最大值。使用flexanalysis(版本3.4,bruker daltonics,bremen,德国)通过应用具有tophat算法的基线减法,使用savitzky-golay算法进行平滑处理并使用snap算法进行峰值检测来进行光谱处理。
[0694]
使用一组标准肽和具有已知质量的蛋白(peptide calibration standard ii,protein calibration standard i和ii,bruker daltonics,bremen,德国)校准质谱仪。
[0695]
完整分子量的确定基于单个和多个带电荷分子离子的质荷比(m/z)。
[0696]
通过isd分析进行蛋白末端的测序。使用biotools(版本3.2,bruker daltonics,bremen,德国)通过计算机生成碎片离子的m/z值,并将其与获得的isd谱中观察到的碎片信号的m/z值进行比较进行isd碎片光谱的注释。该方法使得能够鉴定末端氨基酸序列以及本发明的修饰。
[0697]
为了蛋白测序分析,仅使用碎片(ms/ms)光谱来鉴定蛋白水解肽,并且使用peaks studio(版本7.5,bioinformatics solutions inc.,waterloo,加拿大)进行注释。分别通过针对ncbi nr数据库和附加有沙门菌素序列的uniprot/swissprot数据库搜索ms/ms数据来进行蛋白的鉴定和它们的氨基酸序列的验证。进行数据库搜索,亲本质量误差容限为50ppm,碎片质量误差容限为0.5da。错过的切割以及一个蛋白水解片段的翻译后修饰的最大数目设定为3。两个蛋白末端都允许非特异性切割。
[0698]
来自针对uniprot/swissprot数据库的沙门菌素的蛋白水解肽的每个ms/ms数据集的搜索结果证实了每种分析的沙门菌素的特征(表11)。通过使用isd和分子量测定方法的基于ms的蛋白末端测序进一步分析纯化的沙门菌素scole1a、scole1b和scole7的完整性,其证实所有沙门菌素蛋白在植物表达后是完整的。观察到的翻译后修饰限于在scole2、scole7、scole1a和scole1b的情况下n-末端甲硫氨酸的裂解,和scole7和scole1a的n-末端乙酰化(表11)。
[0699]
表11.植物产生的沙门菌素的特征和完整性研究。分别通过肽质量指纹分析或通过源内衰变和分子量测定对蛋白末端测序对本氏烟草的含有沙门菌素的tsp提取物或纯化的沙门氏菌进行maldi-tof/tof质谱分析。显示了用于产生肽片段的蛋白酶。通过搜索针对ncbi非冗余数据库获得的ms/ms数据集来确认沙门菌素的特征。获得的分子量表明蛋白是完整的。nd,未检测到;ptm翻译后修饰;aa,氨基酸序列。
[0700]
[0701][0702]
实施例13:沙门菌素scole1c、scole1d scole1e和scolma的鉴定
[0703]
如实施例10所示,两种成孔沙门菌素scole1a和scole1b对所有测试的沙门氏菌菌株表都现出最高和最广泛的抗微生物活性。为了鉴定控制沙门氏菌的其他沙门菌素,我们在ncbi数据库中对与scole1a和scole1b相似但在n末端部分与大肠菌素不同的沙门氏菌蛋白进行了同源性检索。该检索揭示了三个新序列,我们将其称为scole1c(seq id no:25)、scole1d(seq id no:26)和scole1e(seq id no:27)(表12)。这些序列的clustal omega比对如图26所示。
[0704]
我们还检索了与大肠菌素m相似的沙门氏菌蛋白,以便具有另一个表达抗微生物活性的功能结构域,其与核酸酶无关(因为这通常需要免疫蛋白的共表达,而这些蛋白中的大多数不容易纯化)。该检索得到了scolma序列(seq id no:28)(表12)。
[0705]
表12.实施例中使用的沙门氏菌细菌素(沙门菌素)列表。
[0706][0707][0708]
实施例14:沙门菌素scole1c、scole1d、scole1e和scolma的质粒构建体
[0709]
从genbank中检索出沙门菌素scole1c、scole1d、scole1e和scolma的氨基酸序列;由thermo fisher scientific inc.合成具有针对本氏烟草优化的密码子使用的相应核苷酸序列。seq id no:29编码scole1c,seq id no:30编码scole1d,seq id no:31编码scole1e,seq id no:32编码scolma。
[0710]
将沙门菌素编码序列插入基于tmv的组装病毒载体pnmd035中(详细描述于wo2012/019660中),得到图28中所示的质粒构建体。
[0711]
实施例15:沙门菌素scole1c、scole1d、scole1e和scolma的表达筛选
[0712]
沙门菌素的表达筛选如实施例2中所述进行。沙门菌素scole1c、scole1d和scolma在本氏烟草叶中的积累很高。相比之下,scole1e的表达较差(图29)。
[0713]
实施例16:对scole1c、scole1d、scole1e和scolma的沙门菌素活性筛选
[0714]
我们比较了植物产生的重组沙门菌素scole1c、scole1d、scole1e和scolma相对于scole1a和scole1b的抗微生物活性。对于这一比较,我们使用了10种肠道沙门氏菌肠道亚种菌株(表13)。使用如实施例6中所述的径向扩散斑点测定法对含有沙门菌素的植物提取物中的抗微生物活性进行评价。来自未转染植物组织的提取物(wt)用作阴性对照。所有测试的新沙门菌素都表现出显著的抗微生物活性,尽管它们对于scole1a和scoleb并不是更优的(表13)。
[0715]
基于表达和抗微生物活性水平,我们选择scole1d和scolma来产生乙醇可诱导的稳定转基因本氏烟草宿主。
[0716]
表13.沙门菌素scole1c、scole1d、scole1e和scolma针对选择的肠道沙门氏菌肠道亚种菌株的抗微生物活性。
[0717][0718]
实施例17:在稳定的转基因宿主中产生沙门菌素scole1d和scolma。
[0719]
使用用于etoh可诱导的转基因表达的载体(对于scole1d为pnmd49621,对于scolma为pnmd49632,图30)通过农杆菌介导的叶盘转化来转化本氏烟草。进行乙醇诱导t0代转基因植物的分离叶以表达沙门菌素。这些实验如实施例8中所述进行。对于用于分析的用pnmd49621构建体转化的169个t0转基因品系,126个品系对于etoh可诱导的sale1d表达呈阳性(图31)。
[0720]
实施例18:沙门菌素scole1b、scole1d和scolma的抗微生物活性:比较。
[0721]
如实施例3中所述,分析了植物产生的重组沙门菌素scole1b、scole1d和scolma的抗微生物活性。在这些实验中,我们使用径向扩散测定通过斑点法针对33种不同血清型肠道沙门氏菌肠道亚种的36个菌株测试了含有重组沙门菌素的植物提取物。细菌菌株列于表
5a和5b中。
[0722]
对于半定量比较,我们以任意单位(au)表示重组沙门菌素的相对抗微生物活性,计算为蛋白提取物在径向扩散测定中引起可检测清除效应的最高稀释度的稀释因子。蛋白电泳后,在考马斯染色的聚丙烯酰胺凝胶上,通过与bsa标准品的目视比较来评价tsp提取物中沙门菌素蛋白的浓度。作为3个独立实验的平均值,以任意单位(au)/μg重组沙门菌素计算特定的抗微生物活性。
[0723]
我们已发现scole1d的抗微生物活性谱与scole1b非常相似,尽管在大多数情况下scole1d的整体活性较低,并且仅在少数情况下其与scole1b相当(图32a和32b)。
[0724]
对于20个菌株,scolma的抗微生物活性低于scole1b,对于10个菌株则相当,而对于6个菌株则更高(图32a和32b)。基于不同的(互补)抗微生物活性模式和不同的抗微生物活性机制,scolma可以被认为是与scole1沙门菌素混合的沙门菌素合剂(cocktail)的良好候选物。
[0725]
实施例19:沙门菌素scole1a、scole1b和scole1d在储存时的稳定性
[0726]
为了进行稳定性评价,将纯化的沙门菌素蛋白样品以干燥的冻干粉末和溶液形式储存在4℃和室温(20-25℃)下。基于93天(scole1a)、387天(scole1b)和200天(scole1d)的抗微生物活性评估蛋白稳定性。通过径向扩散测定评价针对敏感细菌的抗微生物活性。使用相同的细菌菌株:鼠伤寒沙门氏菌14028对所有沙门菌素进行测试。
[0727]
将冻干的蛋白样品重新悬浮在蒸馏水(0.2-0.4mg/ml)中。使用bradford测定法测量每个样品的可溶性蛋白浓度。对于径向扩散测定,制备溶解在pbs缓冲液中的蛋白的系列1:2稀释液。将5μl蛋白稀释液(稀释前为1-1.8μg蛋白),点在具有易感细菌菌株的软琼脂板上。过夜孵育板后评价沙门菌素的残留活性。抗微生物活性被评价为比活性单位(au)-对未受影响的细菌生长区域产生差异的最高稀释度,该区域通过将板保持在光源前方对细菌生长抑制进行目视检查来确定。将抑制生长的最高稀释度记录为活性,以au/mg沙门菌素表示。
[0728]
当以干粉形式在室温或4℃下储存时,所有三种沙门菌素在整个研究期间都保持稳定(图33a-c)。在溶液的情况下,所有三种沙门菌素在室温下在一周内都失去其活性(图33a-c)。当沙门菌素溶液在4℃下储存时,scole1d是最稳定的:它在储存183天后仍保持明显的抗微生物活性(图33c)。
[0729]
实施例20:沙门菌素scolmb和scolmc的鉴定
[0730]
为了鉴定具有磷酸酶m活性的其他沙门菌素,我们在ncbi数据库中对类似于scolma(seq id no:28)的沙门氏菌蛋白进行了同源性检索。作为该检索的结果,我们鉴定了两种蛋白:scolmb(suf52254.1;seq id no:33)和scolmc(oin38022.1;seq id no:34)。scolma、scolmb和scolmc序列的clustal omega(1.2.4)多序列比对在图34中示出。所有三个序列都具有高度(97-98%)的同一性,只有很少的氨基酸错配。
[0731]
实施例21:沙门菌素scolmb和scolmc的质粒构建体和表达筛选
[0732]
由thermo fisher scientific inc.合成具有针对本氏烟草优化的密码子使用的scolmb和scolmc编码核苷酸序列。seq id no:35编码scolmb,seq id no:36编码scolmc。
[0733]
将沙门菌素编码序列插入基于tmv的组装病毒载体pnmd035(详细描述于wo2012/019660中)中,得到图35中所示的质粒构建体。
[0734]
如实施例2中所述进行scolmb和scolmc表达筛选。在7dpi时,发生植物组织坏死,表明更早的收获时间点是优选的。scolmb和scolmc重组蛋白在本氏烟草叶中积累的水平相当(图36)。基于蛋白电泳后在考马斯染色的聚丙烯酰胺凝胶上与bsa标准品进行比较的目视评价,发现scolmb表达产量为约2.6mg/g fw植物材料或30%tsp。对于scolmc,表达产量为2.3mg/g fw或37.5%tsp。对于这两种蛋白,都高于scolma(1.6mg/g fw或18.8%tsp)。
[0735]
实施例22:沙门菌素scolma、scolmb和scolmc的抗微生物活性:比较
[0736]
为了进行比较,如实施例3中所述,分析了植物产生的重组沙门菌素scolma、scolmb和scolmc的抗微生物活性。我们使用径向扩散测定通过斑点法针对8种不同血清型肠道沙门氏菌肠道亚种的10个菌株测试了含有重组沙门菌素的植物提取物。细菌菌株列于表5a和5b中。
[0737]
对于半定量比较,我们以任意单位(au)表示重组沙门菌素的相对抗微生物活性,计算为蛋白提取物在径向扩散测定中引起可检测清除效应的最高稀释度的稀释因子。蛋白电泳后,在考马斯染色的聚丙烯酰胺凝胶上,通过与bsa标准品的目视比较来评价tsp提取物中沙门菌素蛋白的浓度。作为3个独立实验的平均值,以任意单位(au)/μg重组沙门菌素计算特定的抗微生物活性。
[0738]
所有三种测试的沙门菌素都具有非常相似的抗微生物模式:它们针对鼠伤寒、爪哇安纳、慕尼黑、海德堡、都柏林和奥博尼菌株都有活性,对肠炎和纽波特菌株没有活性或活性非常低(图37)。
[0739]
实施例23:测定沙门菌素scole1b、scole7、scole1d、scolmb和scolmc的最小抑制浓度(mic)
[0740]
最小抑制浓度(mic)被定义为防止相应细菌菌株的可见生长的细菌素最低浓度。我们使用mhb培养基(m
ü
ller-hinton bouillon;carl roth gmbh&co.kg,karlsruhe,德国)通过琼脂稀释法测定了scole1b、scole7、scole1d、scolmb和scolmc沙门菌素的mic。将测试的沙门氏菌菌株(肠炎atcc13076、鼠伤寒atcc14028、鼠伤寒atcc13311和都柏林atcc14028)单菌落划线到mhb琼脂板(补充有0.75%(w/v)琼脂的mhb培养基,细菌级(applichem gmbh,darmstadt,德国)),并将板在37℃下孵育至少16小时。通过挑取8个相同大小的菌落接种4ml mhb培养基来制备主培养物,将其在37℃和180rpm下进一步孵育2-3小时直至达到od600为约0.2。将细菌培养物预稀释至od600=0.02,然后进一步稀释至潜在的2x10
6 cfu/ml。
[0741]
为了评价mic,将测试的冻干纯化沙门菌素蛋白重新悬浮于补充有0.1mg/ml bsa(applichem gmbh,darmstadt,德国)的pbs缓冲液(磷酸盐缓冲盐水(137mm nacl,2.7mm kcl,10mm na2hpo4,2mm kh2po4))。将蛋白浓度调整到适当的量(比将要测试的浓度高100倍),并在pbs+0.1mg/ml bsa中制备11种进一步的1:1蛋白稀释液。在融化mhb琼脂、分成5ml等分样品并将琼脂冷却至50℃后,使用6孔无菌细胞培养板(tpp techno plastics products ag,trasadingen,瑞士)制备不同的含有沙门菌素的mhb琼脂板。添加50μl沙门菌素/5ml mhb琼脂后,将溶液填充到6孔板的一个孔中。将板冷却至室温后,在每个孔中滴入4个5μl稀释的沙门氏菌培养物斑点。将液滴干燥并将板在37℃下孵育~16小时。孵育后,检查板在不同浓度下的细菌生长情况。mic被定义为抑制测试微生物可见生长的抗微生物物质的最低浓度。
[0742]
表14.scole1b、scole7、scole1d、scolmb和scolmc针对选择的沙门氏菌菌株的最低抑制浓度(mic)。
[0743][0744]
n.d.-未测定到
[0745]
对个体沙门菌素测定的mic显示出一定的批次间差异,这可能与批次质量(即蛋白质杂质或蛋白水解降解产物的存在)有关。在我们之前的实验中,scole1b和scole1d显示出最广泛的抗微生物活性(实施例3);它们对测试菌株的mic也非常低。sale1b具有更广泛的高活性,因为人们还可以检测到对第二株测试的鼠伤寒菌株的低mic。与scolmb相比,scolmc对测试的atcc14028菌株具有更高的活性。对于scolmc,出人意料地检测到最低mic(《0.98ng/ml),参见例如采用鼠伤寒菌株atcc13311的数据。
[0746]
实施例24:scolma蛋白的纯化
[0747]
使用农杆菌介导的基于tmv的病毒载体pnmd47730的递送产生含有本氏烟草叶生物量的沙门菌素scolma(图28)。将携带pnmd477390质粒的根癌农杆菌菌株icf320的液体培养物从甘油原液中接种到lbs培养基中,并在28℃下摇动孵育过夜。将农杆菌过夜培养物调节至od
600
=1.3,并用lbs培养基按1:100进一步稀释。使用无针注射器浸润4-6周龄的本氏烟草植物的叶子。在5dpi时,收获植物材料,用液氮冷冻,使用研钵和研杵研磨,并进一步储存在-80℃。
[0748]
对于scolma蛋白分离,我们测试了各种纯化策略。优选在ph 4.0-5.5下从本氏烟草叶生物量中提取目的重组蛋白,因为在该ph范围内,最丰富的植物宿主蛋白rubisco沉淀,并且可以与其他固体一起进一步容易地去除。不幸的是,在ph 4.0-5.5时,scolma的提取收率非常差。在ph 6.0下,scolma的产量高得多,尽管得到的提取物也含有大量的rubisco。使用各种方法对此类提取物进一步进行柱色谱纯化。使用pure色谱系
统(cytiva europe gmbh,freiburg im breisgau,德国)进行所有蛋白纯化实验。
[0749]
例如,我们使用hitrap
tm
苯基ff(ls)和hitrap
tm
丁基ff树脂(cytiva europe gmbh,freiburg im breisgau,德国)测试了疏水相互作用色谱法(hic)。在这两种情况下,都使用由20mm柠檬酸盐(ph6.0)、20mm nah2po4和2m nacl组成的缓冲液进行提取。该缓冲液还用于柱平衡和样品加载后的柱洗涤。使用具有0-100%nacl梯度的相同缓冲液进行洗脱。对于这两种树脂,没有实现scolma和宿主蛋白之间的有效分离(图38a和38b)。
[0750]
使用hitrap
tm capto
tm mmc、hitrap
tm capto
tm adhere、hitrap
tm capto
tm q、hitrap
tm capto
tm adhere、hitrap
tm capto
tm mmc、hitrap
tm capto
tm mmc、hitrap
tm capto
tm s、hitrap
tm capto
tm mmc和hitrap
tm capto
tm deae树脂(全都来自cytiva europe gmbh,freiburg im breisgau,德国)的其他众多努力未能获得令人满意的scolma收率和纯度。这些实验的结果总结在表15中。作为一个实例,图39显示了使用hitrap
tm capto
tm deae树脂进行scolma纯化的不成功的结果。使用20mm tris-hcl(ph 8.0)、0.05%tween-80进行提取。用20mm tris-hcl(ph 8.0)洗涤柱子。用含有20mm tris-hcl(ph 8.0)、1m nacl的0-100%线性梯度洗脱缓冲液进行洗脱。尽管scolma提取是有效的,但蛋白未正确结合至树脂;也没有实现rubisco的分离。
[0751]
表15.用于scolma纯化的方法总结。
[0752]
[0753][0754]
cv:柱体积
[0755]
实施例25:scolmb和scolmc蛋白的纯化
[0756]
我们使用ph 4.0、ph 5.0和ph 7.0的缓冲液测试了从本氏烟草叶生物量提取scolma、scolmb和scolmc蛋白。如实施例26中所述生产含有目的重组蛋白的叶生物量。使用pnmd47730(图28)、pnmd51280和pnmd51290(图35)构建体分别来表达scolma、scolmb和scolmc蛋白。对于提取,我们测试了以下缓冲液:1)50mm hepes(ph 7.0),10mm乙酸钾,5mm乙酸镁,10%甘油,0.05%tween-20,300mm nacl;2)15mm乙酸钠(ph 5.0),20mm nah2po4,
30mm nacl,0.05%tween-80,和3)15mm乙酸钠(ph 4.0),20mm nah2po4,30mm nacl,0.05%tween-80。
[0757]
在4℃或室温(约22℃)下进行提取。对于在4℃下进行的提取,将冷冻磨碎的植物材料与提取缓冲液充分混合,并在冰上孵育30分钟。在4℃下以13000rpm离心10分钟后,将上清液转移到新管中(重复两次)。使用sds-page(15%凝胶)分析所得上清液。对于在室温下进行的提取,将冷冻磨碎的植物材料与提取缓冲液充分混合,并在室温下孵育15分钟。在22℃下以7100g离心5分钟后,将上清液转移到新管中并在22℃下以7100g再次离心20分钟。使用sds-page(15%凝胶)分析所得上清液。
[0758]
图40a和40b分别显示了4℃和室温下提取效率的sds-page分析。在两种温度下对于ph 7.0的缓冲液,scolma的回收都是有效的。当使用ph 5.0和ph 4.0的缓冲液时,该蛋白的回收显著不太有效。使用这些缓冲液,scolma收率在室温下特别低,表明蛋白在这样的条件下的不稳定性(图40a)。相比之下,scolmb和scolmc在测试的所有三种缓冲液和两种温度下都具有更高的产量(图40a和40b)。
[0759]
使用hitrap
tm capto
tm mmc树脂有效纯化了scolmb。在室温下用5体积的提取缓冲液(15mm乙酸钠(ph 4.0),20mm nah2po4,30mm nacl,0.05%tween-80)提取本氏烟草叶材料。使用相同的缓冲液进行柱平衡和柱洗涤。使用由10mm乙酸钠(ph 7.5)、20mm nah2po4、50mm nacl组成的洗脱缓冲液的线性0-100%梯度进行蛋白洗脱。使用sds-page分析蛋白样品(图41)。洗脱级分f18高度富集有scolmb蛋白。
[0760]
使用hitrap
tm
苯基ff(ls)树脂也有效纯化了scolmb。在室温下用5体积的提取缓冲液(15mm乙酸钠(ph 4.0),20mm nah2po4,30mm nacl,0.05%tween-80)提取植物生物量。将过滤的植物提取物与50mm乙酸钠(ph 4.0)、30mm nacl、3m(nh4)2so4混合以获得最终的1m(nh4)2so4浓度。用含有15mm乙酸钠(ph 4.0)、20mm nah2po4、30mm nacl、0.05%tween 80、1m(nh4)2so4的缓冲液平衡和洗涤柱子。使用含有10mm乙酸钠(ph 7.5)、20mm nah2po4、50mm nacl的洗脱缓冲液的线性0-100%梯度进行蛋白洗脱。使用sds-page分析蛋白样品(图42)。洗脱级分f11-f14最有效地富集有纯化的scolmb蛋白。
[0761]
根据与scolmb相同的方案,使用hitrap
tm capto
tm mmc树脂纯化scolmc。蛋白纯化级分的sds-page分析如图43所示。洗脱级分f18高度富集有scolmb蛋白。
[0762]
同样,使用与scolmc相同的方案,用hitrap
tm
苯基ff(ls)树脂纯化scolmc。图43显示蛋白纯化级分的sds-page分析。洗脱级分f10-f15高度富集有scolmc。
[0763]
氨基酸和核苷酸序列
[0764]
seq id no:1沙门菌素scole2的氨基酸序列
[0765][0766]
seq id no:2沙门菌素scole3的氨基酸序列
[0767][0768][0769]
seq id no:3沙门菌素scole7的氨基酸序列
[0770]
[0771]
seq id no:4沙门菌素scole1a的氨基酸序列
[0772][0773][0774]
seq id no:5沙门菌素scole1b的氨基酸序列
[0775][0776]
seq id no:6沙门菌素spst的氨基酸序列
[0777][0778]
seq id no:7沙门菌素scole2免疫蛋白simme2的氨基酸序列
[0779][0780]
seq id no:8沙门菌素scole7免疫蛋白simme7的氨基酸序列
[0781]
[0782]
seq id no:9大肠菌素s4的氨基酸序列
[0783][0784]
seq id no:10大肠菌素5的氨基酸序列
[0785][0786]
seq id no:11大肠菌素10的氨基酸序列
[0787][0788][0789]
seq id no:12大肠菌素ia的氨基酸序列
[0790][0791]
seq id no:13大肠菌素ib的氨基酸序列
[0792][0793][0794]
seq id no:14大肠菌素m的氨基酸序列
[0795][0796]
seq id no:15实施例中用于沙门菌素scole2表达的核苷酸序列
[0797]
atgtctggtggtgatggtatcggtcacaatagcggtgctcattctactggtggtgtgaacggttcttc
atctggtaggggtggtagttcttcaggtggtggtaacaaccctaactctggtcctggttggggtactactcatactcctgatggtcacgatatccacaactacaaccctggtgagtttggtggtggtggacataagcctggtggaaacggtggtaatcactctggtggtactggtgatggacaacctcctggtgctgctatggcttttggtttccctgctcttgttcctgctggtgctggtggtcttgctgttactgtttctggtgatgctctggctgctgcaattgctgatgtgcttgctgttctgaagggacctttcaagtttggtgcttggggtatcgctctgtacggtattcttcctaccgagatcgctaaggatgatccaaggatgatgagcaagatcgtgacctctttgcctgctgatgctgtgactgagtctcctgtgtcatctctgcctcttgatcaggctactgtgagcgttaccaagagggttaccgatgtggttaaggatgagaggcagcacattgctgttgttgctggtgtgcctgcttctatccctgttgttgatgctaagcctactacccaccctggtgtgttctctgtttctgttcctggtctgcctgatctgcaggtttcaactgtgaagaacgctcctgctatgactgctttgcctaggggtgttactgatgagaaggataggactgttcaccctgctggtttcaccttcggtggttcttctcatgaggctgtgatcaggttccctaaagagtctggtcaggctcctgtttacgtgtcagtgaccgatgttcttacccctgagcaggttaagcagagacaggatgaagagaatagaaggcagcaagagtgggatgctactcaccctgttgaagtggctgagaggaattacaggctggcttctgatgagctgaacagggctaatgtggatgtggctggtaagcaagagaggcagattcaagctgctcaagctgttgctgctagaaagggtgaactggatgctgctaacaagaccttcgctgatgctaaagaagagatcaagaagttcgagaggttcgctcacgatcctatggctggtggacacagaatgtggcaaatggctggtcttaaggctcagagggctcagaatgaggttaaccagaaacaagctgagttcaacgctgctgagaaagaaaaggctgatgcagatgctgctctgaacgtggcacttgagtctaggaagcagaaagaacaaaaggcaaaggatgctagcgataagctggataaggaaaacaagaggaaccaccctggaaaggctactggtaagggtcaacctgttggtgataagtggcttgaggatgctggtaaagaagctggagcacctgttccagataggatcgctgataagctgagagataaggaattcaagaacttcgatgattttaggaagaagttctgggaagaggtaaatttctagtttttctccttcattttcttggttaggacccttttctctttttatttttttgagctttgatctttctttaaactgatctattttttaattgattggttatggtgtaaatattacatagctttaactgataatctgattactttatttcgtgtgtctatgatgatgatgataactgcaggttagcaaggatcctgagctgagcaagcagttcatccctggtaacaagaaaaggatgagccagggtcttgctcctagggctagaaacaaggatactgtgggtggtagaagatccttcgagctgcatcacgataagccaatctctcaggatggtggtgtttacgatatggataacatcagggtgaccaccccaaagctgcacatcgatattcataggggaaagtaa
[0798]
seq id no:16实施例中用于沙门菌素scole3表达的核苷酸序列
[0799]
atgtctggtggtgatggtaggggtcataataccggtgctcatagcaccagcggtaacattaacggtggtcctactggtcttggtgtgtcaggtggtgcttctgatggttctggttggtcctctgagaacaatccttggggtggtggtagcggttctggtattcactggggaggtggaagtggtagaggtaatggtggtggaaacggtaacagtggtggtggttctggaactggtggtaacctttctgctgttgctgctcctgttgctttcggtttccctgctctttctactcctggtgctggtggtttggctgtgtctatttctgcttctgagctgagcgctgctatcgctggtattatcgctaagctgaagaaggtgaacctgaagttcacccctttcggtgtggtgctgtcctctttgattcctagcgagatcgctaaggatgatcctaacatgatgagcaagatcgtgaccagcctgcctgctgatgatattaccgagtctcctgtgtcctctctgcctcttgataaggctactgtgaatgtgaacgtgagggtggtggatgatgtgaaggatgagaggcagaacatcagcgttgtgtctggtgttcctatgtctgtgcctgttgtggatgctaagcctactgaaaggcctggtgtgttcaccgcttctattccaggtgctcctgtgctgaacatctccgtgaacaattctacccctgctgtgcagactctttctcctggtgtgactaacaacaccgataaggatgttaggcctgctggtttcactcagggtggtaataccagggatgctgtgatcaggttccctaaggattctggtcacaacgcagtgtacgtgtccgtgtctgatgtgttgtctccagatcaggttaagcagaggcaggatgaagagaatagaaggcagcaagagtgggatgctactcaccctgttgaagttgctgagagagagtacgagaacgc
tagagctgaacttgaggctgaaaacaagaacgtgcacagccttcaggtggcacttgatggtcttaagaataccgctgagggtctggctctttctgatgctggtagacatcctctgaccagcagcgagtctagatttgttgctgtgcctggttactccggtggtggtgttcattttgatgctaccgctaccgtggatagcagggataggcttaactctcttctgtctcttggtggtgctgcttacgtgaacaacgtgttggagcttggtgaggtgtcagctcctactgaggatggtttgaaggtgggaaacgctatcaagaacgctatgatcgaggtgtacgataagctgaggcagaggcttattaccaggcagaacgagatcaaccacgctcaggtgtcacttaacaccgctatcgagtctaggaacaagaaagaggaaaagaagaggtccgcagagaacaagctgaacgaagagagaaacaagcctagaaagggtactaaggattacggacacgattaccatcctgctccagagactgaagaaatcaagggtctgggtgatatcaagaagggtatccctaagacccctaagcagaacggtggtggtaagagaaagagatggatcggagataagggtagaaagatctacgagtgggatagccagcatggtgagcttgaaggtaaatttctagtttttctccttcattttcttggttaggacccttttctctttttatttttttgagctttgatctttctttaaactgatctattttttaattgattggttatggtgtaaatattacatagctttaactgataatctgattactttatttcgtgtgtctatgatgatgatgataactgcaggttatagggcttcagatggtcagcacctgggaagctttgatcctaagactggtaagcagctgaagggtcctgatccaaagaggaacatcaagaagtacctttaa
[0800]
seq id no:17实施例中用于沙门菌素scole7表达的核苷酸序列
[0801]
atgtctggtggtgatggtatcggtcacaatagcggtgctcattctactggtggtgtgaacggttcctcttctggttctggtggaagctcatctggaagcggtaacaaccctaattctggtcctggttggggtactactcatacccctaacggtgatatccacaactacaaccctggtgagtttggtggtggtggaaacaagcctggtggacatggtggtaactctggtaaccacgatggtagctctggaaacggtcaaccttctgctgctcctatggcttttggtttccctgctcttgctcctgctggtgctggttctcttgctgttactgtttctggtgaggctctgtctgctgctatcgctgatattttcgctgctctgaagggacctttcaagttcggtgcttggggtattgctctgtacggtattatgcctaccgagatcgctaaggatgatcctaacatgatgagcaagatcatgaccagcctgcctgctgatactgtgactgatactcctgtgtcctctctgcctcttgatcaggctactgtgtctgtgactaagagggttgcagatgtggtgaaggatgagaggcagcatattgctgttgttgctggtgtgcctatgtctgtgcctgttgttgatgctaagcctaccactaggcctggtatcttctctgctactgttcctggacttcctgctttggaggtgtcaaccggtaagtctattcctgcttctaccgctctgcctaggggtattactgaggataaggataggactgagcaccctgctggtttcactttcggtggttcttctcacgatgctgtgatcaggttccctaaagagtctggtcaggctccagtttacgtgtcagtgactgatgtgcttacccctgagcaggttaagcagagacaggatgaagagtctagaaggcagcaagagtgggatgctactcatcctgttgaagtggctgagaggaactacaggcttgcttctgatgagctgaacagggtgaacgctgatgtggctggtaagcaagaaagacaagctcaagctggacaggctgttgctgctagaaagggtgaacttgatgctgctaacaagaccttcgctgatgctaaagaagagatcaagaagttcgagcacttcgctagggatccaatggctggtggtcatagaatgtggcagatggctggtcttaaggctcagagggctcagaatgaggttaaccagaaacaagctgagttcgatgctgcagagaaagaaaaggctgatgctgatgcagctctgaacgctgctcttgaatctaggaagcagaaagagcagaaggctaaggataccaaagagaggctggataaggaaaacaagaggaatcagcctggtaaggctaccggtaagggtcagccagtttctgataagtggcttgaggatgctggtaaagagagcggttctcctatccctgatagcattgctgataagcttagagataaggaattcagaaacttcgatgattttaggaagaagttctgggaggaagttagcaaggatcctgagctgagcaagcagttcatcaagggtaacagagataggatgcaggtaaatttctagtttttctccttcattttcttggttaggacccttttctctttttatttttttgagctttgatctttctttaaactgatctattttttaattgattggttatggtgtaaatattacatagctttaactgataatctgattactttatttcgtgtgtctatgatgatgatgataactgcaggttggaaaggctcctaagtccagaaagaaggatgctgctggtaagaggacctctttcgagcttcatcacgataagcctgtgagccaggatggtggtgtttacgatatgg
ataacctgaggatcaccacccctaagaggcacatcgatattcataggggacagtaa
[0802]
seq id no:18实施例中用于沙门菌素scole1a表达的核苷酸序列
[0803]
atggctgataacaccattgcttactacgaggatggtgtgcctcacagcgctgatggtaaggtggtgattgtgatcgatggtaagatgcctgtggataccggtgctggtggtactggtggtggtggaggtggtaaggttggaggaacttctgaaagctctgctgctattcacgctaccgctaagtggtctaccgctcagcttaagaaaaccctggctgagaaggctgctagagagagagaaactgctgctgcaatggctgctgctaaggctaagagagatgctcttacccagcacctgaaggatatcgtgaacgatgtgcttaggcacaacgcttctaggaccccttctgctactgatcttgctcacgctaacaacatggctatgcaggctgaagctcagagacttggtagagctaaggctgaggaaaaggctagaaaagaggctgaggctgctgagcttgctttccaagaagctgaaagacagagggaagaggctgttagacagcttgctgaaactgagaggcagcttaagcaagctgaggaagagaagaggcttgctgctctttctgatgaggctagggctgttgagaacgctaggaagaatctggataccgcaaagtccgagctggctaatgtggattctgatatcgagaggcagaggtcccagctgtcatctcttgatgctgatgtgaagaaggctgaagagaacctgaggctgaccatgaggattaagggtaggatcggtaggaagatgcaggctaagtcacaggctatcgtggatgataagaaaaggatctactccgatgctgagaacgtgctgaataccatgaccgtgaataggaacctgaaggctcagcaggttaccgatgcagagaatgagcttaaggtggcaatcgataacctgaacagcagccagatgaagaacgctgtggatgctaccgtgtctttctaccagactctgaccgagaagtacggtgagaagtacagccttatcgctcaagagctggcagagaagtccaagggtaagaaaatcggaaatgtggatgaggctctggctgcattcgagaagtataaggatgtgctggataagaagttcagcaaggctgatagggatgctattgtgaacgctctgaagtccttcaactacgatgattgggctaagcacctggatcagttcgctaagtacctgaagatcaccggtcacgtgagcttcggttacgatgttgtgtctgatgtgctgaaggctagcgagactggtgattggaagcctctgttcattacccttgagcagaaggtgttggatactggtatgagctacctggtggtgctgatgttctctcttattgctggaaccaccctgggaatcttcggtgtggctattattaccgctatcctgtgcagcttcgtggataagtacatcctgaacgcactgaacgatgctctgggaatctaa
[0804]
seq id no:19实施例中用于沙门菌素scole1b表达的核苷酸序列
[0805]
atgagcgataacaccattgcttactacgaggatggtgtgccttacagcgctgatggtcaagtggtgattgtgatcgatggtaagatgcctgtggataccggtgctggtggtactggtggtggtggaggtggtaaggttggaggaacttctgaaagctctgctgctattcacgctaccgctaagtggtctaaggctcagcttcagaagtccctggaagagaaggctgctagagagagagaaactgctgctgcaatggctgctgctaaggctaagagagatgctcttacccagcacctgaaggatatcgtgaacgatgtgctgaggtacaacgcttctaggactccttctgctaccgatcttgctcacgctaacaacatggctatgcaggctgaagctcagagacttggtagagctaaggctgaggaaaaggctagaaaagaggctgaggctgctgagaagtctcttcaagaagctgagagacagagggaagaagctgctaggcaaagagctgaagcagagaggcaacttaagcaggcagaggctgaagagaagaggttggctgctctttctgaagaggctagggcagttgagatcacccagaagaatcttgctgctgctcagagcgagctgtccaagatggatggtgagatcaagagccttaacgtgaggctgtctacctctatccatgctagggatgctgagatgaacagcctgtctggtaagaggaacgagctggctcaagagagcgctaagtacaaagaactggatgagctggtgaagaagcttgagcctagggctaatgatcctctgcagaacaggcctttcttcgatgctacatctagaagggcaagggctggtgatactttggctgagaagcagaaagaggtgaccgcttctgagactaggatcaacgagcttaacaccgagatcaaccaggtgaggggtgctatttcacaggcaaacaacaataggaacctgaaggtgcagcaggttaccgagactgagaacgctcttaaggtggcaatcgataacctgaacagcagccagatgaagaacgctgtggatgctaccgtgtctttctaccagaccctgactgctaagtacggtgagaagtacagcctgatcgctcaagaacttgctgagcagtccaagggtaagaaaatcagcaatgtggatgaggctctggctgcattcgagaagtataagga
tgtgctggataagaagttcagcaaggctgatagggatgcaattgtgaacgctctgaagtccgtggattacgctgattgggctaagcacctggatcagttcagcagatacctgaagatcagcggtagggtgtcaaccggttacgatatctacagcgatatcagaaagggtatggataccaacgattggaggcctctgttcctgacccttgagaagcttgctgttgatgctggtgtgggttacatcgtggctcttggtttctctgtgatcgcttctaccgctcttggtatttggggtgtggctattatcaccggtgtgatctgcagcttcgttgataagaaggatttggagaagctgaacgaggcactgggaatctaa
[0806]
seq id no:20实施例中用于沙门菌素spst表达的核苷酸序列
[0807]
atgttcatcaagagcggtggtaacctgaccatcaggacttttggtggtcttggtgtgggtggtgatttcgatagcgatacttggagaagaaggtccaccgattcttgggtgccatacagcgagtacattgctatcgagtgcatcgtggctcctaaccagctttaccagcttcttactgatgtggctcaggtggaaactgtggctgctcaacttgctcaggttggataccagtatcttcagggtaggcttaggctggtgagagaggatggttcttgcaccgatttcagcggtaaggctatgctggataacctgctgaacaagagcaaggatattctggatctggatttcctgcacgtgagcgagggttataggtctgaagcttattggcctggtcagtcctctggtatcaccattggttacggtgtggatatcggtcaccagtctgaagagggacttcataagtggggtgtgcctcagagcatcatcgataagatcaaggattacttcggtattaccggtgaggctgctaacacccttcttaagggtctgaaggataagaccctgggactgagcgatagagagatcaagcagttctccgatatcgtgaagaagcaggctaccgctgatatcatcaacaagtacaacgctgctaccaagggtatcacctttgataagatcccttacaacaccaggaccgctatcatcgatctgttctaccagtacagcgctcctaagggtgctcctaagtcttggggtttcattatcaacaacgattggaacggtttctacaacgagctgatgaacttcggtgataagcacaccaccagaagagagagggaagctgctctggttctgtctgatattgtgaacaaccagtacatctacaagtaa
[0808]
seq id no:21实施例中用于沙门菌素scole2免疫蛋白simme2表达的核苷酸序列
[0809]
atggaactgaagaagtccatcagcgattacaccgaggctgagttcaagaagatcatcgaggctatcatcaactgcgagggtgatgagaaaacccaggatgataaccttgagttcttcatcagggtgaccgagtacccttctggtagcgatcttatctactaccctgagggtgataacgatggtagcaccgaggcaattatcaaagaaatcaaggaatggagggctgctaacggtaagcctggttttaagcaagcttaa
[0810]
seq id no:22实施例中用于沙门菌素scole7免疫蛋白simme7表达的核苷酸序列
[0811]
atggaactgaagaacagcatcagcgattacaccgaggctgagttcatcgagttcatgaaagaaatcgataaggaaaacgtggcagagactgatgataagctggatctgctgctgaaccacttcgagcaggttacagaacaccctgatggaaccgatctgatctactacgctgcttccgatgctgagtctacccctgaggctatcaccaagaaaatcaaagaatggagggctgctaacggtaagcctggttttaagcaaggttaa
[0812]
seq id no:23实施例中用于沙门菌素scolea表达的双元的基于tmv的载体的核苷酸序列
[0813]
catggctgataacaccattgcttactacgaggatggtgtgcctcacagcgctgatggtaaggtggtgattgtgatcgatggtaagatgcctgtggataccggtgctggtggtactggtggtggtggaggtggtaaggttggaggaacttctgaaagctctgctgctattcacgctaccgctaagtggtctaccgctcagcttaagaaaaccctggctgagaaggctgctagagagagagaaactgctgctgcaatggctgctgctaaggctaagagagatgctcttacccagcacctgaaggatatcgtgaacgatgtgcttaggcacaacgcttctaggaccccttctgctactgatcttgctcacgctaacaacatggctatgcaggctgaagctcagagacttggtagagctaaggctgaggaaaaggctagaaaagaggctgaggctgctgagcttgctttccaagaagctgaaagacagagggaagaggctgttagacagcttgctgaaactgagaggcagcttaagcaagctgaggaagagaagaggcttgctgctctttctgatgaggctagggctgttgagaacgctaggaagaatctggataccgcaaagtccgagctggctaatgtggattctgatatcgagaggcagaggtcccagctgtcatct
cttgatgctgatgtgaagaaggctgaagagaacctgaggctgaccatgaggattaagggtaggatcggtaggaagatgcaggctaagtcacaggctatcgtggatgataagaaaaggatctactccgatgctgagaacgtgctgaataccatgaccgtgaataggaacctgaaggctcagcaggttaccgatgcagagaatgagcttaaggtggcaatcgataacctgaacagcagccagatgaagaacgctgtggatgctaccgtgtctttctaccagactctgaccgagaagtacggtgagaagtacagccttatcgctcaagagctggcagagaagtccaagggtaagaaaatcggaaatgtggatgaggctctggctgcattcgagaagtataaggatgtgctggataagaagttcagcaaggctgatagggatgctattgtgaacgctctgaagtccttcaactacgatgattgggctaagcacctggatcagttcgctaagtacctgaagatcaccggtcacgtgagcttcggttacgatgttgtgtctgatgtgctgaaggctagcgagactggtgattggaagcctctgttcattacccttgagcagaaggtgttggatactggtatgagctacctggtggtgctgatgttctctcttattgctggaaccaccctgggaatcttcggtgtggctattattaccgctatcctgtgcagcttcgtggataagtacatcctgaacgcactgaacgatgctctgggaatctaagcttactagagcgtggtgcgcacgatagcgcatagtgtttttctctccacttgaatcgaagagatagacttacggtgtaaatccgtaggggtggcgtaaaccaaattacgcaatgttttgggttccatttaaatcgaaaccccttatttcctggatcacctgttaacgcacgtttgacgtgtattacagtgggaataagtaaaagtgagaggttcgaatcctccctaaccccgggtaggggcccagcggccgctctagctagagtcaagcagatcgttcaaacatttggcaataaagtttcttaagattgaatcctgttgccggtcttgcgatgattatcatataatttctgttgaattacgttaagcatgtaataattaacatgtaatgcatgacgttatttatgagatgggtttttatgattagagtcccgcaattatacatttaatacgcgatagaaaacaaaatatagcgcgcaaactaggataaattatcgcgcgcggtgtcatctatgttactagatcgacctgcatccaccccagtacattaaaaacgtccgcaatgtgttattaagttgtctaagcgtcaatttgtttacaccacaatatatcctgccaccagccagccaacagctccccgaccggcagctcggcacaaaatcaccactcgatacaggcagcccatcagtcagatcaggatctcctttgcgacgctcaccgggctggttgccctcgccgctgggctggcggccgtctatggccctgcaaacgcgccagaaacgccgtcgaagccgtgtgcgagacaccgcggccgccggcgttgtggatacctcgcggaaaacttggccctcactgacagatgaggggcggacgttgacacttgaggggccgactcacccggcgcggcgttgacagatgaggggcaggctcgatttcggccggcgacgtggagctggccagcctcgcaaatcggcgaaaacgcctgattttacgcgagtttcccacagatgatgtggacaagcctggggataagtgccctgcggtattgacacttgaggggcgcgactactgacagatgaggggcgcgatccttgacacttgaggggcagagtgctgacagatgaggggcgcacctattgacatttgaggggctgtccacaggcagaaaatccagcatttgcaagggtttccgcccgtttttcggccaccgctaacctgtcttttaacctgcttttaaaccaatatttataaaccttgtttttaaccagggctgcgccctgtgcgcgtgaccgcgcacgccgaaggggggtgcccccccttctcgaaccctcccggcccgctaacgcgggcctcccatccccccaggggctgcgcccctcggccgcgaacggcctcaccccaaaaatggcagcgctggccaattcgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatggctaaaatgagaatatcaccggaattgaaaaaactgatcgaaaaataccgctgcgtaaaagatacggaaggaatgtctcctgctaaggtatataagctggtgggagaaaatgaaaacctatatttaaaaatgacggacagccggtataaagggaccacctatgatgtggaacgggaaaaggacatgatgctatggctggaaggaaagctgcctgttccaaaggtcctgcactttgaacggcatgatggctggagcaatctgctcatgagtgaggccgatggcgtcctttgctcggaagagtatgaagatgaacaaagccctgaaaagattatcgagctgtatgcggagtgcatcaggctctttcactccatcgacatatcggattgtccctatacgaatagcttagacagccgcttagccgaattggattacttactgaataacgatctggccgatgtggattgcgaaaactgggaagaagacactccatttaaagatccgcgcgagctgtatgattttttaaagacggaaaagcccgaagaggaacttgtcttttcccacggcgacctgggagacagcaacatctttgtgaaagatggcaaagtaagtggctttattgatcttgggagaagcggcagggcggacaagtggta
tgacattgccttctgcgtccggtcgatcagggaggatatcggggaagaacagtatgtcgagctattttttgacttactggggatcaagcctgattgggagaaaataaaatattatattttactggatgaattgttttagctgtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttacggttcctggcagatcctagatgtggcgcaacgatgccggcgacaagcaggagcgcaccgacttcttccgcatcaagtgttttggctctcaggccgaggcccacggcaagtatttgggcaaggggtcgctggtattcgtgcagggcaagattcggaataccaagtacgagaaggacggccagacggtctacgggaccgacttcattgccgataaggtggattatctggacaccaaggcaccaggcgggtcaaatcaggaataagggcacattgccccggcgtgagtcggggcaatcccgcaaggagggtgaatgaatcggacgtttgaccggaaggcatacaggcaagaactgatcgacgcggggttttccgccgaggatgccgaaaccatcgcaagccgcaccgtcatgcgtgcgccccgcgaaaccttccagtccgtcggctcgatggtccagcaagctacggccaagatcgagcgcgacagcgtgcaactggctccccctgccctgcccgcgccatcggccgccgtggagcgttcgcgtcgtctcgaacaggaggcggcaggtttggcgaagtcgatgaccatcgacacgcgaggaactatgacgaccaagaagcgaaaaaccgccggcgaggacctggcaaaacaggtcagcgaggccaagcaggccgcgttgctgaaacacacgaagcagcagatcaaggaaatgcagctttccttgttcgatattgcgccgtggccggacacgatgcgagcgatgccaaacgacacggcccgctctgccctgttcaccacgcgcaacaagaaaatcccgcgcgaggcgctgcaaaacaaggtcattttccacgtcaacaaggacgtgaagatcacctacaccggcgtcgagctgcgggccgacgatgacgaactggtgtggcagcaggtgttggagtacgcgaagcgcacccctatcggcgagccgatcaccttcacgttctacgagctttgccaggacctgggctggtcgatcaatggccggtattacacgaaggccgaggaatgcctgtcgcgcctacaggcgacggcgatgggcttcacgtccgaccgcgttgggcacctggaatcggtgtcgctgctgcaccgcttccgcgtcctggaccgtggcaagaaaacgtcccgttgccaggtcctgatcgacgaggaaatcgtcgtgctgtttgctggcgaccactacacgaaattcatatgggagaagtaccgcaagctgtcgccgacggcccgacggatgttcgactatttcagctcgcaccgggagccgtacccgctcaagctggaaaccttccgcctcatgtgcggatcggattccacccgcgtgaagaagtggcgcgagcaggtcggcgaagcctgcgaagagttgcgaggcagcggcctggtggaacacgcctgggtcaatgatgacctggtgcattgcaaacgctagggccttgtggggtcagttccggctgggggttcagcagccagcgcctgatctggggaaccctgtggttggcacatacaaatggacgaacggataaaccttttcacgcccttttaaatatccgattattctaataaacgctcttttctcttaggtttacccgccaatatatcctgtcaaacactgatagtttaaactgaaggcgggaaacgacaatctgatctaagctagcttggaattggtaccacgcgtttcgacaaaatttagaacgaacttaattatgatctcaaatacattgatacatatctcatctagatctaggttatcattatgtaagaaagttttgacgaatatggcacgacaaaatggctagactcgatgtaattggtatctcaactcaacattatacttataccaaacattagttagacaaaatttaaacaactattttttatgtatgcaagagtcagcatatgtataattgattcagaatcgttttgacgagttcggatgtagtagtagccattatttaatgtacatactaatcgtgaatagtgaatatgatgaaacattgtatcttattgt
ataaatatccataaacacatcatgaaagacactttctttcacggtctgaattaattatgatacaattctaatagaaaacgaattaaattacgttgaattgtatgaaatctaattgaacaagccaaccacgacgacgactaacgttgcctggattgactcggtttaagttaaccactaaaaaaacggagctgtcatgtaacacgcggatcgagcaggtcacagtcatgaagccatcaaagcaaaagaactaatccaagggctgagatgattaattagtttaaaaattagttaacacgagggaaaaggctgtctgacagccaggtcacgttatctttacctgtggtcgaaatgattcgtgtctgtcgattttaattatttttttgaaaggccgaaaataaagttgtaagagataaacccgcctatataaattcatatattttcctctccgctttgaagttttagttttattgcaacaacaacaacaaattacaataacaacaaacaaaatacaaacaacaacaacatggcacaatttcaacaaacaattgacatgcaaactctccaagccgctgcgggacgcaacagcttggtgaatgatttggcatctcgtcgcgtttacgataatgcagtcgaggagctgaatgctcgttccagacgtcccaaggtaataggaactttctggatctactttatttgctggatctcgatcttgttttctcaatttccttgagatctggaattcgtttaatttggatctgtgaacctccactaaatcttttggttttactagaatcgatctaagttgaccgatcagttagctcgattatagctaccagaatttggcttgaccttgatggagagatccatgttcatgttacctgggaaatgatttgtatatgtgaattgaaatctgaactgttgaagttagattgaatctgaacactgtcaatgttagattgaatctgaacactgtttaaggttagatgaagtttgtgtatagattcttcgaaactttaggatttgtagtgtcgtacgttgaacagaaagctatttctgattcaatcagggtttatttgactgtattgaactctttttgtgtgtttgcaggtccacttctccaaggcagtgtctacggaacagaccctgattgcaacaaacgcatatccggagttcgagatttcctttactcatacgcaatccgctgtgcactccttggccggaggccttcggtcacttgagttggagtatctcatgatgcaagttccgttcggttctctgacgtacgacatcggcggtaacttttccgcgcaccttttcaaagggcgcgattacgttcactgctgcatgcctaatctggatgtacgtgacattgctcgccatgaaggacacaaggaagctatttacagttatgtgaatcgtttgaaaaggcagcagcgtcctgtgcctgaataccagagggcagctttcaacaactacgctgagaacccgcacttcgtccattgcgacaaacctttccaacagtgtgaattgacgacagcgtatggcactgacacctacgctgtagctctccatagcatttatgatatccctgttgaggagttcggttctgcgctactcaggaagaatgtgaaaacttgtttcgcggcctttcatttccatgagaatatgcttctagattgtgatacagtcacactcgatgagattggagctacgttccagaaatcaggtaacattccttagttacctttcttttctttttccatcataagtttatagattgtacatgctttgagatttttctttgcaaacaatctcaggtgataacctgagcttcttcttccataatgagagcactctcaattacacccacagcttcagcaacatcatcaagtacgtgtgcaagacgttcttccctgctagtcaacgcttcgtgtaccacaaggagttcctggtcactagagtcaacacttggtactgcaagttcacgagagtggatacgttcactctgttccgtggtgtgtaccacaacaatgtggattgcgaagagttttacaaggctatggacgatgcgtggcactacaaaaagacgttagcaatgcttaatgccgagaggaccatcttcaaggataacgctgcgttaaacttctggttcccgaaggtgctcttgaaattggaagtcttcttttgttgtctaaacctatcaatttctttgcggaaatttatttgaagctgtagagttaaaattgagtcttttaaacttttgtaggtgagagacatggttatcgtccctctctttgacgcttctatcacaactggtaggatgtctaggagagaggttatggtgaacaaggacttcgtctacacggtcctaaatcacatcaagacctatcaagctaaggcactgacgtacgcaaacgtgctgagcttcgtggagtctattaggtctagagtgataattaacggtgtcactgccaggtaagttgttacttatgattgttttcctctctgctacatgtattttgttgttcatttctgtaagatataagaattgagttttcctctgatgatattattaggtctgaatgggacacagacaaggcaattctaggtccattagcaatgacattcttcctgatcacgaagctgggtcatgtgcaagatgaaataatcctgaaaaagttccagaagttcgacagaaccaccaatgagctgatttggacaagtctctgcgatgccctgatgggggttattccctcggtcaaggagacgcttgtgcgcggtggttttgtgaaagtagcagaacaagccttagagatcaaggttagtatcatatgaagaaatacctagtttcagttgatgaatgctattttctgacctcagttgttctcttttgagaattatttcttttctaatttgcctgatttttctattaattcattaggttcccgagctatactgtacctt
cgccgaccgattggtactacagtacaagaaggcggaggagttccaatcgtgtgatctttccaaacctctagaagagtcagagaagtactacaacgcattatccgagctatcagtgcttgagaatctcgactcttttgacttagaggcgtttaagactttatgtcagcagaagaatgtggacccggatatggcagcaaaggtaaatcctggtccacacttttacgataaaaacacaagattttaaactatgaactgatcaataatcattcctaaaagaccacacttttgttttgtttctaaagtaatttttactgttataacaggtggtcgtagcaatcatgaagtcagaattgacgttgcctttcaagaaacctacagaagaggaaatctcggagtcgctaaaaccaggagaggggtcgtgtgcagagcataaggaagtgttgagcttacaaaatgatgctccgttcccgtgtgtgaaaaatctagttgaaggttccgtgccggcgtatggaatgtgtcctaagggtggtggtttcgacaaattggatgtggacattgctgatttccatctcaagagtgtagatgcagttaaaaagggaactatgatgtctgcggtgtacacagggtctatcaaagttcaacaaatgaagaactacatagattacttaagtgcgtcgctggcagctacagtctcaaacctctgcaaggtaagaggtcaaaaggtttccgcaatgatccctctttttttgtttctctagtttcaagaatttgggtatatgactaacttctgagtgttccttgatgcatatttgtgatgagacaaatgtttgttctatgttttaggtgcttagagatgttcacggcgttgacccagagtcacaggagaaatctggagtgtgggatgttaggagaggacgttggttacttaaacctaatgcgaaaagtcacgcgtggggtgtggcagaagacgccaaccacaagttggttattgtgttactcaactgggatgacggaaagccggtttgtgatgagacatggttcagggtggcggtgtcaagcgattccttgatatattcggatatgggaaaacttaagacgctcacgtcttgcagtccaaatggtgagccaccggagcctaacgccaaagtaattttggtcgatggtgttcccggttgtggaaaaacgaaggagattatcgaaaaggtaagttctgcatttggttatgctccttgcattttaggtgttcgtcgctcttccatttccatgaatagctaagattttttttctctgcattcattcttcttgcctcagttctaactgtttgtggtatttttgttttaattattgctacaggtaaacttctctgaagacttgattttagtccctgggaaggaagcttctaagatgatcatccggagggccaaccaagctggtgtgataagagcggataaggacaatgttagaacggtggattccttcttgatgcatccttctagaagggtgtttaagaggttgtttatcgatgaaggactaatgctgcatacaggttgtgtaaatttcctactgctgctatctcaatgtgacgtcgcatatgtgtatggggacacaaagcaaattccgttcatttgcagagtcgcgaactttccgtatccagcgcattttgcaaaactcgtcgctgatgagaaggaagtcagaagagttacgctcaggtaaagcaactgtgttttaatcaatttcttgtcaggatatatggattataacttaatttttgagaaatctgtagtatttggcgtgaaatgagtttgctttttggtttctcccgtgttataggtgcccggctgatgttacgtatttccttaacaagaagtatgacggggcggtgatgtgtaccagcgcggtagagagatccgtgaaggcagaagtggtgagaggaaagggtgcattgaacccaataaccttaccgttggagggtaaaattttgaccttcacacaagctgacaagttcgagttactggagaagggttacaaggtaaagtttccaactttcctttaccatatcaaactaaagttcgaaactttttatttgatcaacttcaaggccacccgatctttctattcctgattaatttgtgatgaatccatattgacttttgatggttacgcaggatgtgaacactgtgcacgaggtgcaaggggagacgtacgagaagactgctattgtgcgcttgacatcaactccgttagagatcatatcgagtgcgtcacctcatgttttggtggcgctgacaagacacacaacgtgttgtaaatattacaccgttgtgttggacccgatggtgaatgtgatttcagaaatggagaagttgtccaatttccttcttgacatgtatagagttgaagcaggtctgtctttcctatttcatatgtttaatcctaggaatttgatcaattgattgtatgtatgtcgatcccaagactttcttgttcacttatatcttaactctctctttgctgtttcttgcaggtgtccaatagcaattacaaatcgatgcagtattcaggggacagaacttgtttgttcagacgcccaagtcaggagattggcgagatatgcaattttactatgacgctcttcttcccggaaacagtactattctcaatgaatttgatgctgttacgatgaatttgagggatatttccttaaacgtcaaagattgcagaatcgacttctccaaatccgtgcaacttcctaaagaacaacctattttcctcaagcctaaaataagaactgcggcagaaatgccgagaactgcaggtaaaatattggatgccagacgatattctttcttttgatttgtaactttttcctgtcaaggtcgataaattttattttttttggtaaaaggtcgataatttttttttggagccattatgtaattttcctaattaactgaaccaaaatta
tacaaaccaggtttgctggaaaatttggttgcaatgatcaaaagaaacatgaatgcgccggatttgacagggacaattgacattgaggatactgcatctctggtggttgaaaagttttgggattcgtatgttgacaaggaatttagtggaacgaacgaaatgaccatgacaagggagagcttctccaggtaaggacttctcatgaatattagtggcagattagtgttgttaaagtctttggttagataatcgatgcctcctaattgtccatgttttactggttttctacaattaaaggtggctttcgaaacaagagtcatctacagttggtcagttagcggactttaactttgtggatttgccggcagtagatgagtacaagcatatgatcaagagtcaaccaaagcaaaagttagacttgagtattcaagacgaatatcctgcattgcagacgatagtctaccattcgaaaaagatcaatgcgattttcggtccaatgttttcagaacttacgaggatgttactcgaaaggattgactcttcgaagtttctgttctacaccagaaagacacctgcacaaatagaggacttcttttctgacctagactcaacccaggcgatggaaattctggaactcgacatttcgaagtacgataagtcacaaaacgagttccattgtgctgtagagtacaagatctgggaaaagttaggaattgatgagtggctagctgaggtctggaaacaaggtgagttcctaagttccatttttttgtaatccttcaatgttattttaacttttcagatcaacatcaaaattaggttcaattttcatcaaccaaataatatttttcatgtatatataggtcacagaaaaacgaccttgaaagattatacggccggaatcaaaacatgtctttggtatcaaaggaaaagtggtgatgtgacaacctttattggtaataccatcatcattgccgcatgtttgagctcaatgatccccatggacaaagtgataaaggcagctttttgtggagacgatagcctgatttacattcctaaaggtttagacttgcctgatattcaggcgggcgcgaacctcatgtggaacttcgaggccaaactcttcaggaagaagtatggttacttctgtggtcgttatgttattcaccatgatagaggagccattgtgtattacgatccgcttaaactaatatctaagttaggttgtaaacatattagagatgttgttcacttagaagagttacgcgagtctttgtgtgatgtagctagtaacttaaataattgtgcgtatttttcacagttagatgaggccgttgccgaggttcataagaccgcggtaggcggttcgtttgctttttgtagtataattaagtatttgtcagataagagattgtttagagatttgttctttgtttgataatgtcgatagtctcgtacgaacctaaggtgagtgatttcctcaatctttcgaagaaggaagagatcttgccgaaggctctaacgaggttaaaaaccgtgtctattagtactaaagatattatatctgtcaaggagtcggagactttgtgtgatatagatttgttaatcaatgtgccattagataagtatagatatgtgggtatcctaggagccgtttttaccggagagtggctagtgccagacttcgttaaaggtggagtgacgataagtgtgatagataagcgtctggtgaactcaaaggagtgcgtgattggtacgtacagagccgcagccaagagtaagaggttccagttcaaattggttccaaattactttgtgtccaccgtggacgcaaagaggaagccgtggcaggtaaggatttttatgatatagtatgcttatgtattttgtactgaaagcatatcctgcttcattgggatattactgaaagcatttaactacatgtaaactcacttgatgatcaataaacttgattttgcaggttcatgttcgtatacaagacttgaagattgaggcgggttggcagccgttagctctggaagtagtttcagttgctatggtcaccaataacgttgtcatgaagggtttgagggaaaaggtcgtcgcaataaatgatccggacgtcgaaggtttcgaaggtaagccatcttcctgcttatttttataatgaacatagaaataggaagttgtgcagagaaactaattaacctgactcaaaatctaccctcataattgttgtttgatattggtcttgtattttgcaggtgtggttgacgaattcgtcgattcggttgcagcatttaaagcggttgacaactttaaaagaaggaaaaagaaggttgaagaaaagggtgtagtaagtaagtataagtacagaccggagaagtacgccggtcctgattcgtttaatttgaaagaagaaaacgtcttacaacattacaaacccgaatcagtaccagtatttcgataagaaacaagaaac
[0814]
seq id no:24实施例中用于沙门菌素scole2免疫蛋白immscole2表达的双元的基于pvx的载体的核苷酸序列
[0815]
gatcggtcgtatcactggaacaacaaccgctgaggctgttgtcactctaccaccaccataactacgtctacataaccgacgcctaccccagtttcatagtattttctggtttgattgtatgaataatataaataaaaaaaaaaaaaaaaaaaaaaaactagtgagctcttctgtcagcgggcccactgcatccaccccagtacattaaaaacgtccgcaatgtgttattaagttgtctaagcgtcaatttgtttacaccacaatatatcctgccaccagccagccaacagctcccc
gaccggcagctcggcacaaaatcaccactcgatacaggcagcccatcagtcagatcaggatctcctttgcgacgctcaccgggctggttgccctcgccgctgggctggcggccgtctatggccctgcaaacgcgccagaaacgccgtcgaagccgtgtgcgagacaccgcggccgccggcgttgtggatacctcgcggaaaacttggccctcactgacagatgaggggcggacgttgacacttgaggggccgactcacccggcgcggcgttgacagatgaggggcaggctcgatttcggccggcgacgtggagctggccagcctcgcaaatcggcgaaaacgcctgattttacgcgagtttcccacagatgatgtggacaagcctggggataagtgccctgcggtattgacacttgaggggcgcgactactgacagatgaggggcgcgatccttgacacttgaggggcagagtgctgacagatgaggggcgcacctattgacatttgaggggctgtccacaggcagaaaatccagcatttgcaagggtttccgcccgtttttcggccaccgctaacctgtcttttaacctgcttttaaaccaatatttataaaccttgtttttaaccagggctgcgccctgtgcgcgtgaccgcgcacgccgaaggggggtgcccccccttctcgaaccctcccggcccgctaacgcgggcctcccatccccccaggggctgcgcccctcggccgcgaacggcctcaccccaaaaatggcagcgctggccaattcgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatggctaaaatgagaatatcaccggaattgaaaaaactgatcgaaaaataccgctgcgtaaaagatacggaaggaatgtctcctgctaaggtatataagctggtgggagaaaatgaaaacctatatttaaaaatgacggacagccggtataaagggaccacctatgatgtggaacgggaaaaggacatgatgctatggctggaaggaaagctgcctgttccaaaggtcctgcactttgaacggcatgatggctggagcaatctgctcatgagtgaggccgatggcgtcctttgctcggaagagtatgaagatgaacaaagccctgaaaagattatcgagctgtatgcggagtgcatcaggctctttcactccatcgacatatcggattgtccctatacgaatagcttagacagccgcttagccgaattggattacttactgaataacgatctggccgatgtggattgcgaaaactgggaagaagacactccatttaaagatccgcgcgagctgtatgattttttaaagacggaaaagcccgaagaggaacttgtcttttcccacggcgacctgggagacagcaacatctttgtgaaagatggcaaagtaagtggctttattgatcttgggagaagcggcagggcggacaagtggtatgacattgccttctgcgtccggtcgatcagggaggatatcggggaagaacagtatgtcgagctattttttgacttactggggatcaagcctgattgggagaaaataaaatattatattttactggatgaattgttttagctgtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttacggttcctggcagatcctagatgtggcgcaacgatgccggcgacaagcaggagcgcaccgacttcttccgcatcaagtgttttggctctcaggccgaggcccacggcaagtatttgggcaaggggtcgctggtattcgtgcagggcaagattcggaataccaagtacgagaaggacggccagacggtctacgggaccgacttcattgccgataaggtggattatctggacaccaaggcaccaggcgggtcaaatcaggaataagggcacattgccccggcgtgagtcggggcaatcccgcaaggagggtgaatgaatcggacgtttgaccggaaggcatacaggcaagaactgatcgacgcggggttttccgccgaggatgccgaaaccatcgcaagccgcaccgtcatgcgtgcgccccgcgaaaccttccagtccgtcggctcgatggtccagcaagctacggccaagatcgagcgcgacagcgtgcaact
ggctccccctgccctgcccgcgccatcggccgccgtggagcgttcgcgtcgtctcgaacaggaggcggcaggtttggcgaagtcgatgaccatcgacacgcgaggaactatgacgaccaagaagcgaaaaaccgccggcgaggacctggcaaaacaggtcagcgaggccaagcaggccgcgttgctgaaacacacgaagcagcagatcaaggaaatgcagctttccttgttcgatattgcgccgtggccggacacgatgcgagcgatgccaaacgacacggcccgctctgccctgttcaccacgcgcaacaagaaaatcccgcgcgaggcgctgcaaaacaaggtcattttccacgtcaacaaggacgtgaagatcacctacaccggcgtcgagctgcgggccgacgatgacgaactggtgtggcagcaggtgttggagtacgcgaagcgcacccctatcggcgagccgatcaccttcacgttctacgagctttgccaggacctgggctggtcgatcaatggccggtattacacgaaggccgaggaatgcctgtcgcgcctacaggcgacggcgatgggcttcacgtccgaccgcgttgggcacctggaatcggtgtcgctgctgcaccgcttccgcgtcctggaccgtggcaagaaaacgtcccgttgccaggtcctgatcgacgaggaaatcgtcgtgctgtttgctggcgaccactacacgaaattcatatgggagaagtaccgcaagctgtcgccgacggcccgacggatgttcgactatttcagctcgcaccgggagccgtacccgctcaagctggaaaccttccgcctcatgtgcggatcggattccacccgcgtgaagaagtggcgcgagcaggtcggcgaagcctgcgaagagttgcgaggcagcggcctggtggaacacgcctgggtcaatgatgacctggtgcattgcaaacgctagggccttgtggggtcagttccggctgggggttcagcagccagcgcctgatctggggaaccctgtggttggcacatacaaatggacgaacggataaaccttttcacgcccttttaaatatccgattattctaataaacgctcttttctcttaggtttacccgccaatatatcctgtcaaacactgatagtttaaactgaaggcgggaaacgacaatctgatctaagctaggcatgcctgcaggtcaacatggtggagcacgacacgcttgtctactccaaaaatatcaaagatacagtctcagaagaccaaagggcaattgagacttttcaacaaagggtaatatccggaaacctcctcggattccattgcccagctatctgtcactttattgtgaagatagtggaaaaggaaggtggctcctacaaatgccatcattgcgataaaggaaaggccatcgttgaagatgcctctgccgacagtggtcccaaagatggacccccacccacgaggagcatcgtggaaaaagaagacgttccaaccacgtcttcaaagcaagtggattgatgtgatatctccactgacgtaagggatgacgcacaatcccactatccttcgcaagacccttcctctatataaggaagttcatttcatttggagaggagaaaactaaaccatacaccaccaacacaaccaaacccaccacgcccaattgttacacacccgcttgaaaaagaaagtttaacaaatggccaaggtgcgcgaggtttaccaatcttttacagactccaccacaaaaactctcatccaagatgaggcttatagaaacattcgccccatcatggaaaaacacaaactagctaacccttacgctcaaacggttgaagcggctaatgatctagaggggttcggcatagccaccaatccctatagcattgaattgcatacacatgcagccgctaagaccatagagaataaacttctagaggtgcttggttccatcctaccacaagaacctgttacatttatgtttcttaaacccagaaagctaaactacatgagaagaaacccgcggatcaaggacattttccaaaatgttgccattgaaccaagagacgtagccaggtaccccaaggaaacaataattgacaaactcacagagatcacaacggaaacagcatacattagtgacactctgcacttcttggatccgagctacatagtggagacattccaaaactgcccaaaattgcaaacattgtatgcgaccttagttctccccgttgaggcagcctttaaaatggaaagcactcacccgaacatatacagcctcaaatacttcggagatggtttccagtatataccaggcaaccatggtggcggggcataccatcatgaattcgctcatctacaatggctcaaagtgggaaagatcaagtggagggaccccaaggatagctttctcggacatctcaattacacgactgagcaggttgagatgcacacagtgacagtacagttgcaggaatcgttcgcggcaaaccacttgtactgcatcaggagaggagacttgctcacaccggaggtgcgcactttcggccaacctgacaggtacgtgattccaccacagatcttcctcccaaaagttcacaactgcaagaagccgattctcaagaaaactatgatgcagctcttcttgtatgttaggacagtcaaggtcgcaaaaaattgtgacatttttgccaaagtcagacaattaattaaatcatctgacttggacaaatactctgctgtggaactggtttacttagtaagctacatggagttccttgccgatttacaagctaccacctgcttctcagacacactttctggtggcttgctaacaaagacccttgcaccggtgagggcttggatacaagagaaaaagatgcagctgtttggtcttgaggactacgcgaagttagtcaaagcagttgatttccacccggtggattttt
ctttcaaagtggaaacttgggacttcagattccaccccttgcaagcgtggaaagccttccgaccaagggaagtgtcggatgtagaggaaatggaaagtttgttctcagatggggacctgcttgattgcttcacaagaatgccagcttatgcggtaaacgcagaggaagatttagctgcaatcaggaaaacgcccgagatggatgtcggtcaagaagttaaagagcctgcaggagacagaaatcaatactcaaaccctgcagaaactttcctcaacaagctccacaggaaacacagtagggaggtgaaacaccaggccgcaaagaaagctaaacgcctagctgaaatccaggagtcaatgagagctgaaggtgatgccgaaccaaatgaaataagcgggacgatgggggcaatacccagcaacgccgaacttcctggcacgaatgatgccagacaagaactcacactcccaaccactaaacctgtccctgcaaggtgggaagatgcttcattcacagattctagtgtggaagaggagcaggttaaactccttggaaaagaaaccgttgaaacagcgacgcaacaagtcatcgaaggacttccttggaaacactggattcctcaattaaatgctgttggattcaaggcgctggaaattcagagggataggagtggaacaatgatcatgcccatcacagaaatggtgtccgggctggaaaaagaggacttccctgaaggaactccaaaagagttggcacgagaattgttcgctatgaacagaagccctgccaccatccctttggacctgcttagagccagagactacggcagtgatgtaaagaacaagagaattggtgccatcacaaagacacaggcaacgagttggggcgaatacttgacaggaaagatagaaagcttaactgagaggaaagttgcgacttgtgtcattcatggagctggaggttctggaaaaagtcatgccatccagaaggcattgagagaaattggcaagggctcggacatcactgtagtcctgccgaccaatgaactgcggctagattggagtaagaaagtgcctaacactgagccctatatgttcaagacctctgaaaaggcgttaattgggggaacaggcagcatagtcatctttgacgattactcaaaacttcctcccggttacatagaagccttagtctgtttctactctaaaatcaagctaatcattctaacaggagatagcagacaaagcgtctaccatgaaactgctgaggacgcctccatcaggcatttgggaccagcaacagagtacttctcaaaatactgccgatactatctcaatgccacacaccgcaacaagaaagatcttgcgaacatgcttggtgtctacagtgagagaacgggagtcaccgaaatcagcatgagcgccgagttcttagaaggaatcccaactttggtaccctcggatgagaagagaaagctgtacatgggcaccgggaggaatgacacgttcacatacgctggatgccaggggctaactaagccgaaggtacaaatagtgttggaccacaacacccaagtgtgtagcgcgaatgtgatgtacacggcactttctagagccaccgataggattcacttcgtgaacacaagtgcaaattcctctgccttctgggaaaagttggacagcaccccttacctcaagactttcctatcagtggtgagagaacaagcactcagggagtacgagccggcagaggcagagccaattcaagagcctgagccccagacacacatgtgtgtcgagaatgaggagtccgtgctagaagagtacaaagaggaactcttggaaaagtttgacagagagatccactctgaatcccatggtcattcaaactgtgtccaaactgaagacacaaccattcagttgttttcgcatcaacaagcaaaagatgagactctcctctgggcgactatagatgcgcggctcaagaccagcaatcaagaaacaaacttccgagaattcctgagcaagaaggacattggggacgttctgtttttaaactaccaaaaagctatgggtttacccaaagagcgtattcctttttcccaagaggtctgggaagcttgtgcccacgaagtacaaagcaagtacctcagcaagtcaaagtgcaacttgatcaatgggactgtgagacagagcccagacttcgatgaaaataagattatggtattcctcaagtcgcagtgggtcacaaaggtggaaaaactaggtctacccaagattaagccaggtcaaaccatagcagccttttaccagcagactgtgatgctttttggaactatggctaggtacatgcgatggttcagacaggctttccagccaaaagaagtcttcataaactgtgagacgacgccagatgacatgtctgcatgggccttgaacaactggaatttcagcagacctagcttggctaatgactacacagctttcgaccagtctcaggatggagccatgttgcaatttgaggtgctcaaagccaaacaccactgcataccagaggaaatcattcaggcatacatagatattaagactaatgcacagattttcctaggcacgttatcaattatgcgcctgactggtgaaggtcccacttttgatgcaaacactgagtgcaacatagcttacacccatacaaagtttgacatcccagccggaactgctcaagtttatgcaggagacgactccgcactggactgtgttccagaagtgaagcatagtttccacaggcttgaggacaaattactcctaaagtcaaagcctgtaatcacgcagcaaaagaagggcagttggcctgagttttgtggttggctgatcacaccaaaaggggtgatgaaagacccaattaagctccatgttagcttaaaattggctgaagctaagggtgaactcaagaaatgtcaagattcct
atgaaattgatctgagttatgcctatgaccacaaggactctctgcatgacttgttcgatgagaaacagtgtcaggcacacacactcacttgcagaacactaatcaagtcagggagaggcactgtctcactttcccgcctcagaaactttctttaaccgttaagttaccttagagatttgaataagatgtcagcaccagctagtacaacacagcccatagggtcaactacctcaactaccacaaaaactgcaggcgcaactcctgccacagcttcaggcctgttcactatcccggatggggatttctttagtacagcccgtgccatagtagccagcaatgctgtcgcaacaaatgaggacctcagcaagattgaggctatttggaaggacatgaaggtgcccacagacactatggcacaggctgcttgggacttagtcagacactgtgctgatgtaggatcatccgctcaaacagaaatgatagatacaggtccctattccaacggcatcagcagagctagactggcagcagcaattaaagaggtgtgcacacttaggcaattttgcatgaagtatgccccagtggtatggaactggatgttaactaacaacagtccacctgctaactggcaagcacaaggtttcaagcctgagcacaaattcgctgcattcgacttcttcaatggagtcaccaacccagctgccatcatgcccaaagaggggctcatccggccaccgtctgaagctgaaatgaatgctgcccaaactgctgcctttgtgaagattacaaaggccagggcacaatccaacgactttgccagcctagatgcagctgtcactcgaggaaggatcaccggaacgaccacagcagaggcagtcgttactctgcctcctccataacagaaactttctttaaccgttaagttaccttagagatttgaataagatggatattctcatcagtagtttgaaaagtttaggttattctaggacttccaaatctttagattcaggacctttggtagtacatgcagtagccggagccggtaagtccacagccctaaggaagttgatcctcagacacccaacattcaccgtgcatacactcggtgtccctgacaaggtgagtatcagaactagaggcatacagaagccaggacctattcctgagggcaacttcgcaatcctcgatgagtatactttggacaacaccacaaggaactcataccaggcactttttgctgacccttatcaggcaccggagtttagcctagagccccacttctacttggaaacatcatttcgagttccgaggaaagtggcagatttgatagctggctgtggcttcgatttcgagacgaactcaccggaagaagggcacttagagatcactggcatattcaaagggcccctactcggaaaggtgatagccattgatgaggagtctgagacaacactgtccaggcatggtgttgagtttgttaagccctgccaagtgacgggacttgagttcaaagtagtcactattgtgtctgccgcaccaatagaggaaattggccagtccacagctttctacaacgctatcaccaggtcaaagggattgacatatgtccgcgcagggccataggctgaccgctccggtcaattctgaaaaagtgtacatagtattaggtctatcatttgctttagtttcaattacctttctgctttctagaaatagcttaccccacgtcggtgacaacattcacagcttgccacacggaggagcttacagagacggcaccaaagcaatcttgtacaactccccaaatctagggtcacgagtgagtctacacaacggaaagaacgcagcatttgctgccgttttgctactgactttgctgatctatggaagtaaatacatatctcaacgcaatcatacttgtgcttgtggtaacaatcatagcagtcattagcacttccttagtgaggactgaaccttgtgtcatcaagattactggggaatcaatcacagtgttggcttgcaaactagatgcagaaaccataagggccattgccgatctcaagccactctccgttgaacggttaagtttccattgatactcgaaagaggtcagcaccagctagcaacaaacaagaacatggaactgaagaagtccatcagcgattacaccgaggctgagttcaagaagatcatcgaggctatcatcaactgcgagggtgatgagaaaacccaggatgataaccttgagttcttcatcagggtgaccgagtacccttctggtagcgatcttatctactaccctgagggtgataacgatggtagcaccgaggcaattatcaaagaaatcaaggaatggagggctgctaacggtaagcctggttttaagcaagcttaa
[0816]
seq id no:25沙门菌素scole1c的氨基酸序列
[0817]
msdntiayyedgvpysadgkvliiidgkmpvdtggtggggggkagvtsessaaihatakwskaqlqksleekaareretaaamaaakakrdaltqhlkdivndvlyhnahppavidlahannmamqaeaqrlgrakaeekarkeaeaaekslqeaerqceeaarqraeaerqlkqaeaeekrlaalseearaveiaqknlaaaqselskmdgeimslnvrlstsihardaemnslsgkrnelaqasakykeldelvkkleprandplqnrpffdaasrraragdtlaekqkevtasetrinelnteinqvqgaisqannnrnlnvqqvtetenalkvaidnlnssqmknavdatvsfyqtltekygekysliaqelaekskgkkignvdealaafekykdvldrkfskadrdaivnalksfnyddwakhldqfakylkitghvsfgydvvsdvl
karetgdwkplfitleqkaldtgmsylvvlmfsliagttlgifgvaiitailcsfvdkyilnalndalgi
[0818]
seq id no:26沙门菌素scole1d的氨基酸序列
[0819]
msdntiayyedgvpysadgkvliiidgkmpvdtggtggggggkagvtsessaaihatakwskaqlqksleekaareretaaamaaakakrdaltqhlkdivndvlyhnahppavidlahannmamqaeaqrlgrakaeekarkeaeaaekslqeaerqreeaarqraeaerqlkqaeaeekrlaalseearaveiaqknlaaaqselskmdgeimslnvrlstsihardaemnslsgkrnelaqasakykeldelvkkleprandplqnrpffdaasrraragdtlaekqkevtasetrinelnteinqvqgaisqannnrnlnvqqvtetenalkvaidnlnssqmknavdatvsfyqtltekygekysliaqelaekskgkkignvdealaafekykdvldrkfskadrdaivnalksfnyddwakhldqfakylkitghvsfgydvvsdvlkaretgdwkplfitleqkaldtgmsylvvlmfsliagttlgifgvaiitailcsfvdkyilnalndalgi
[0820]
seq id no:27沙门菌素scole1e的氨基酸序列
[0821]
msgsiayyedgvpysadgkvvivitgklpegtggsltadlgsagvsessaaihatakwstaqlqktkaeqavkvkeaavaqakakekrdaltqylkdivnqalshnsrppavtdlahannmamqaeaerlrlakaeakareeaeaaekafqlaeqqrlasereqaeterqlklaeaeekrlaalseearaveiaqknlaatqseltnmdgeiqnlnirlnnniherdaetsslsarrnelfqvseqykeidaqvkkleprandplqsrpffaamtrranvytvvqekqglvtasetrinqfnadisrlqeeivkanekrnmiithiheaeeqlkiakinlinsqikdatdsvigfyqtltekygqkysllaqelaekskgkkignvnealaafekyqdvlnkkfskadrdaifnalesvkyddwakhldqfakylkitgrvsfgydlvsdvlkvrdtgdwkplfltlekkaldtglsylvvlmfsliagttlgiwgvaivtgilcsfidksmlndlnealgi
[0822]
seq id no:28沙门菌素scolma的氨基酸序列
[0823]
mtdtitvvapvppsgsalagnysastmsagnrissgptflqfaypyyqspqlavncakwildfveshdmknannqkifsenvghfcfadknlvnypamkvldafggdrkfiysqdqisrlsgdvttpitawahflwgdgaartvnltdvglriqanqispvmdlvkggavgtfpvnakftrdtmldgiipasylgnitlqttgtltinslgawsydgvvkayndtydanpsthrgllgeystsvlghfsgtpyeiqmpgmipvkgngmr
[0824]
seq id no:29实施例中用于沙门菌素scole1c表达的核苷酸序列
[0825]
atgagcgacaacaccattgcctactacgaggatggtgtgccttacagcgctgatggtaaggtgctgatcatcatcgatggcaagatgcctgttgataccggtggtactggtggtggtggcggtggtaaggctggtgttacttctgaatctagcgctgctattcacgctaccgccaagtggtctaaggctcagcttcagaagtctctggaagagaaggctgctagagagagggaaactgctgctgctatggctgctgcaaaggctaagagagatgctcttacccagcacctgaaggacatcgtgaacgatgtgctttaccataacgctcaccctccagctgtgattgatcttgctcacgctaacaacatggctatgcaggctgaagctcagaggcttggtagagctaaggctgaggaaaaggctcgtaaagaagctgaggctgctgagaagtcacttcaagaggctgaaagacagtgcgaggaagctgctagacaaagagctgaagcagagaggcaacttaagcaggctgaggcagaagagaagaggcttgctgctctttctgaagaggctagggctgttgagatcgctcagaagaatcttgctgctgcacagagcgagctgtccaagatggatggtgagatcatgtctctgaacgtgcggctgtctacttctatccatgctagggatgccgagatgaacagcctttctggtaagaggaatgagctggctcaggctagcgccaagtacaaagaacttgatgagctggtgaagaagctcgagcctagggctaatgatccacttcagaacaggccattcttcgacgctgctagtagaagggctagagctggcgatactcttgccgagaagcagaaagaggttaccgcttctgagactcggatcaacgagcttaacaccgagattaaccaggtgcagggtgctatctcacaggccaacaacaataggaacctcaacgtgcagcaggtcaccgagactgagaacgctcttaaggtggcaatcgacaacctgaacagcagccagatgaagaacgctgtggatgctaccgtgagcttctaccagactttgaccgagaagtacggggagaagtacagccttattgctcaagagctggccgagaagtccaagggtaagaaaattggtaacgtggacgaggctctcgctgccttcgaaaagtacaaggatgtgctgga
ccggaagttcagcaaggctgatagggatgctattgtgaacgccctgaagtccttcaactacgacgattgggctaagcacctggaccagttcgctaagtaccttaagatcaccggccacgtgtccttcggttacgatgttgtttctgacgtgctgaaggctcgtgagactggtgattggaagcctctgttcattaccctcgagcagaaggctttggataccggcatgtcttaccttgtggtgctgatgttctctctgatcgctggtactacccttggcattttcggtgtggctatcatcaccgctatcctgtgctcattcgtggacaagtacatcctgaacgctctgaacgatgctctgggaatctaa
[0826]
seq id no:30实施例中用于沙门菌素scole1d表达的核苷酸序列
[0827]
atgagcgacaacaccattgcctactacgaggatggtgtgccttacagcgctgatggtaaggtgctgatcatcatcgatggcaagatgcctgttgataccggtggtactggtggtggtggcggtggtaaggctggtgttacttctgaatctagcgctgctattcacgctaccgccaagtggtctaaggctcagcttcagaagtctctggaagagaaggctgctagagagagggaaactgctgctgctatggctgctgcaaaggctaagagagatgctcttacccagcacctgaaggacatcgtgaacgatgtgctttaccataacgctcaccctccagctgtgattgatcttgctcacgctaacaacatggctatgcaggctgaagctcagaggcttggtagagctaaggctgaggaaaaggctcgtaaagaagctgaggctgctgagaagtcacttcaagaggctgaaaggcagagagaggaagctgcaagacaaagagcagaggctgagagacaacttaagcaggctgaggcagaagagaagaggcttgctgctctttctgaagaggctagggctgttgagatcgctcagaagaatcttgctgctgcacagagcgagctgtccaagatggatggtgagatcatgtctctgaacgtgcggctgtctacttctatccatgctagggatgccgagatgaacagcctttctggtaagaggaatgagctggctcaggctagcgccaagtacaaagaacttgatgagctggtgaagaagctcgagcctagggctaatgatccacttcagaacaggccattcttcgacgctgctagtagaagggctagagctggcgatactcttgccgagaagcagaaagaggttaccgcttctgagactcggatcaacgagcttaacaccgagattaaccaggtgcagggtgctatctcacaggccaacaacaataggaacctcaacgtgcagcaggtcaccgagactgagaacgctcttaaggtggcaatcgacaacctgaacagcagccagatgaagaacgctgtggatgctaccgtgagcttctaccagactttgaccgagaagtacggggagaagtacagccttattgctcaagagctggccgagaagtccaagggtaagaaaattggtaacgtggacgaggctctcgctgccttcgaaaagtacaaggatgtgctggaccggaagttcagcaaggctgatagggatgctattgtgaacgccctgaagtccttcaactacgacgattgggctaagcacctggaccagttcgctaagtaccttaagatcaccggccacgtgtccttcggttacgatgttgtttctgacgtgctgaaggctcgtgagactggtgattggaagcctctgttcattaccctcgagcagaaggctttggataccggcatgtcttaccttgtggtgctgatgttctctctgatcgctggtactacccttggcattttcggtgtggctatcatcaccgctatcctgtgctcattcgtggacaagtacatcctgaacgctctgaacgatgctctgggaatctaa
[0828]
seq id no:31实施例中用于沙门菌素scole1e表达的核苷酸序列
[0829]
atgagcggctctatcgcttactacgaggatggtgttccttacagcgctgatggtaaggtggtgatcgtgattaccggcaagcttcctgaaggtactggtggttctcttaccgctgatcttggatctgctggtgtgtctgaaagctctgctgctattcatgctaccgccaagtggtctactgctcagcttcaaaagactaaggctgagcaggccgtgaaggtgaaagaagctgctgttgctcaggccaaggccaaagaaaagagggatgctcttacccagtacctgaaggatatcgtgaaccaggctctgagccataactctagacctccagctgtgactgatctggctcacgctaacaatatggctatgcaggctgaggctgagaggcttagacttgctaaagctgaggctaaggctcgtgaagaagctgaagctgcagagaaggcttttcagcttgctgaacagcagaggcttgcttctgaaagagaacaggctgagacagagcggcagcttaagttggctgaagcagaggaaaagaggctggctgctctttctgaagaggctagggctgttgagatcgctcagaagaatcttgctgctacccagtctgagctgaccaacatggatggtgagatccagaacctgaacatccggctgaacaacaacatccatgagagggacgctgagacaagctctctgtctgctagacggaacgagcttttccaggttagcgagcagtacaaagagatcgacgcccaggttaagaagcttgagcctagggctaatgaccctcttcagtctaggcctttcttcgctgctatgaccaga
cgggctaatgtgtacactgtggtgcaagagaagcagggtcttgtgactgcttctgagactcggatcaaccagttcaacgctgacatctctaggctgcaagaagagatcgtcaaggccaacgagaagcggaacatgatcattacccacatccacgaggctgaggaacagctgaagatcgctaagatcaacctgatcaactcccagatcaaggacgctaccgatagcgtgatcggtttctaccagactctgaccgagaagtacggccagaagtactctttgcttgctcaagagctggccgagaagtccaagggtaagaaaatcggtaacgtgaacgaggcccttgctgcctttgagaagtaccaggatgtgctgaacaagaagttctccaaggctgacagggacgctatcttcaacgctcttgagtccgtgaagtacgacgattgggctaagcaccttgaccagttcgccaagtaccttaagatcaccggtagggtgagcttcggttacgatcttgtgtccgatgtcctcaaggtgagagatactggtgattggaagccgctgttcttgacccttgagaagaaggctcttgataccggcctgtcttaccttgtggtgctgatgttctctcttatcgccggtactacccttggcatttggggtgttgctattgtgaccggtatcctgtgctccttcatcgacaagagcatgctgaacgacctcaacgaggctcttggtatctaa
[0830]
seq id no:32实施例中用于沙门菌素scolma表达的核苷酸序列
[0831]
atgaccgacactattactgtggttgctcctgtgcctccttctggttctgctcttgctggtaactacagcgcctctactatgtctgctggcaacaggatttctagcggtcctacctttctgcagttcgcttacccttactaccagtctcctcagcttgctgtgaattgcgctaagtggatcctggacttcgttgagagccacgacatgaagaacgccaacaaccagaaaatcttcagcgagaacgtgggccacttctgcttcgctgataagaaccttgtgaactacccggccatgaaggtgttggatgcttttggtggtgaccggaagttcatctacagccaggatcagatctctcggctgtctggtgatgtgactactcctattactgcttgggctcacttcctgtggggtgatggtgctgctaggactgtgaatcttaccgatgttggtctgcggatccaggccaatcagatttctcctgtgatggacctggtgaaaggtggtgctgttggtactttccctgtgaacgctaagttcaccagggataccatgctggacggtatcatccctgctagctaccttggtaacattacccttcagaccaccggcaccttgaccatcaattctcttggtgcttggagctacgatggcgtggtgaaggcttacaacgatacctacgatgctaacccgtctactcacaggggtttgcttggtgagtacagcacttctgtgctcggtcatttctctggaaccccttacgagattcagatgcctggtatgatcccggtgaaaggcaatggtatgaggtaa
[0832]
seq id no:33沙门菌素scolmb的氨基酸序列
[0833][0834]
seq id no:34沙门菌素scolmc的氨基酸序列
[0835][0836]
seq id no:35实施例中用于沙门菌素scolmb表达的核苷酸序列
[0837]
atgaccgacactattaccgtggttgctcctgttcctccaagcggttctgctcttgctggtaactactc
tgcctctaccatgtctgctggcaacaggatttctagcggtcctacctttctgcagttcgcttacccttactaccagtctcctcagcttgctgtgaattgcgctaagtggatcctggacttcgttgagagccacgacatgaagaacgctaacaaccagcagatcttcagcgagaacgttggccagttttgcttcgctgataagaacctggtggactacccgaccatgaaggtgttggatgcttttggtggcgaccggaagttcatctactctcaggatcagatcagcaggctgtctggtgatgtgactactcctattactgcttgggctcacttcctgtggggtgatggtgctgctaggactgtgaatcttaccgatgttggtctgcggatccagccgaatcagatttctcctgtgatggacctggtgaaaggtggtgctgttggtactttccctgtgaacgctaagttcaccagggataccatgctggacggtatcatccctgctagctaccttggtaacattacccttcagaccaccggcaccttgaccatcaattctcttggtgcttggagctacgatggcgtggtgaaggcttacaacgatacctacgatgctaacccgtctactcacaggggtttgcttggtgagtactctacctctgtgctgaggcacttttctggaaccccttacgagattcagatgcctggtatgatcccggtgaaaggcaacggtatgaggtaa
[0838]
seq id no:36实施例中用于沙门菌素scolmc表达的核苷酸序列
[0839]
atgaccgacactattaccgtggttgctcctgttcctccaagcggttcttctcttgctggtaactacaccgccagcaccatgtcatctggtaacaggatttctagcggccctaccttccttcagttcgcttatccttactaccagtctcctcagctggctgtgaattgcgctaagtggattctggacttcgtcgagagccacgacatgaagaatgctaacaaccagcagatcttcagcgagaacgttggccagttttgcttcgctgataagaacctggtgaactacccggctatgaaggtgttggatgctttcggtggtgaccggaagttcatctactctcaggatcagatcagcaggctgtctggtgatgtgactactcctattactgcttgggctcacttcctgtggggtgatggtgctgctaggactgtgaatcttaccgatgttggtctgcggatccagccgaatcagatttctcctgtgatggatctggtgaaaggtggtgctgttggtactttccctgtgaacgctaagttcacccgggataccatgctggacggtattatccctgctagctacctgggtaacattacccttcagaccaccggtactctgaccatcaattctcttggcgcttggagctacgatggtgtggtgaaggcttacaacgatacctacgacgctaacccgtctactcacagaggtttgcttggtgagtacagcacctctgtgcttaggcacttttctggaaccccttacgagatccagatgcctggtatgattccggtgaaaggcaacggcatgaggtaa
[0840]
本专利申请要求于2019年9月20日提交的、公开号为us-2020-0010517-a1的美国专利申请16/577,484的优先权,该专利申请的内容通过引用并入本文,包括整个说明书、权利要求书、附图和序列表。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1