一种真姬菇Finc-B-7菌种的SSR标记指纹图谱及其构建方法与应用与流程
本发明属于真姬菇菌种的检测技术领域,具体涉及一种真姬菇finc-b-7菌种的ssr标记指纹图谱及其构建方法与应用。
背景技术:
真姬菇(拉丁文名hypsizygusmarmoreus(peck)h.e.bigelow)属担子菌门、伞菌纲、伞菌目、离褶伞科,因其菌盖表面的大理石斑纹而得名。其外形美观、质地脆嫩、具有独特的海鲜味且价格经济,受到广大消费者喜爱。真姬菇不仅含有丰富维生素和18种氨基酸,近年研究还发现真姬菇中所含的小分子化合物、多糖和多肽等成分具有溶血、抗氧化、抑制白血病和淋巴瘤细胞、降血脂、抗炎和抗氧化等功能,是一种经济价值很高的食药兼用菌。自从2001年我国第一家真姬菇工厂化栽培企业成立以来,真姬菇成为了我国市场发展较快的新型食用真菌之一。2019年中国真姬菇工厂化总产量为32.8万吨,较2018年同比增长58.63%,占食用菌工厂化总产量的9.6%,在我国工厂化栽培食用真菌生产总量中排名第三。
优质菌种在真姬菇的工厂化栽培中起着举足轻重的作用。目前,我国工厂化生产企业使用的真姬菇菌种主要为日本引入菌种或在其基础上进一步杂交选育而成,栽培后子实体为棕色的称为蟹味菇,子实体为白色的又称白玉菇或玉龙菇。尽管工厂化生产的产量逐年提升,但部分菌种仍存在栽培周期长、单产低、单根子实体外观整齐度差和易出现瘤盖等问题,这些缺陷都可能导致生产成本的增加,降低产品的市场竞争力。另一方面,由于真姬菇工厂化生产菌株的多样性不高,产品外观近似,无法满足市场消费者多样化的需求。我国有着丰富的真姬菇野生和自然栽培资源,高效利用这些优质的资源,拓展菌株的遗传基础,有利于国内高产、优质、特色真姬菇菌株的选育。
随着《国际植物新品种保护法》的颁布实施,建立成熟、快速、精准的分子生物学鉴定技术体系成为保护食用菌品种知识产权的有力手段。
技术实现要素:
本发明所要解决的技术问题是提供一种真姬菇“finc-b-7”菌种的ssr标记指纹图谱及其构建方法与应用,该指纹图谱与常规形态学检测、拮抗试验、出菇试验相比,具有检测时间短,准确性高,重复性好的优点。
真姬菇(hypsizygusmarmoreus)finc-b-7,于2016年11月28日保藏于中国微生物菌种保藏管理委员会普通微生物中心,地址为北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,保藏编号为cgmccno.13193。
本发明的一种真姬菇“finc-b-7”菌种的ssr标记指纹图谱,该指纹图谱由6对ssr标记组成,是基于真姬菇基因组简单重复序列片段开发ssr引物,扩增带型好,重复性高的ssr引物,标记详细信息如表1:
表1ssr标记详细信息列表
本发明的一种真姬菇“finc-b-7”菌种的ssr标记指纹图谱的构建方法,包括:
(1)菌丝培养:将真姬菇菌种转接到马铃薯葡萄糖琼脂固体培养基上,22℃培养10-14d后收集菌丝;
(2)基因组dna的提取:利用真菌dna提取试剂盒,按照试剂盒实验步骤提取基因组dna,并取2μldna进行1.2%琼脂糖凝胶电泳检测。紫外分光光度法检测总基因组dna浓度和纯度,调整样品dna的浓度一致;
(3)ssr分子标记引物开发:根据真姬菇菌株的基因组重测序结果,选取多态性序列设计ssr引物并合成;
(4)ssr分子标记的检测:对上述提取的dna进行基因ssr标记的pcr扩增;
(5)电泳检测:将上述pcr扩增得到的产物与甲酰胺加样缓冲液混匀,变性,上机检测;
(6)genemapper数据分析。
所述的真姬菇finc-b-7菌种的ssr标记指纹图谱的构建方法,其特征在于:所述步骤(2)基因组dna的提取,其具体方法包括:
(1)将真菌样本加入液氮充分研磨;
(2)向研磨好的粉末中迅速加入360μlbufferste和40μlbuffersds,迅速涡旋混匀后,将离心管放在65℃水浴15min,水浴过程中颠倒离心管以混合样本数次;
(3)加入5μlrnasesolution至裂解液中,涡旋混匀,室温静置15-30min;
(4)加入140μlbufferps,涡旋震荡30s,冰上放置10min;
(5)室温下,13000g离心5min,小心转移400μl上清液至新的离心管中;
(6)加入600μlbufferpbd至样品中,涡旋混匀30s;
(7)把dna结合柱装在收集管,转移一半混合液至柱子中,8000g离心1min;
(8)倒弃滤液把柱子装回收集管,转移剩余混合液至柱子中,8000g离心1min;
(9)倒弃滤液把柱子装回收集管,加入600μlbuffergw2至柱子中,8000g离心1min;
(10)重复步骤9;
(11)倒弃滤液把柱子装回收集管,10000g离心2min去除柱子中残留的乙醇;
(12)将柱子转移至新的1.5ml离心管中,加入30μl预热至65℃的bufferae至柱子的膜中央,室温静置2min,10000g离心1min;
(13)取2μldna用于1.2%琼脂糖凝胶电泳检测,取2μldna用于nanodrop分光光度计测浓度,剩余dna保存于-20℃。
所述步骤(3)中的pcr扩增体系为:总体积10μl,包括:10×pcrbuffer1μl,2.5mmol/ldntp0.8μl,5u/μltakarahstaq酶0.1μl,5μmol/ltp-m130.5μl,5μmol/lssr标记特异引物总体积各0.6μl,浓度20ng~30ng/μl提取的模板dna1.2μl,ddh2o5.2μl;
pcr反应条件:95℃5min;95℃30s,59℃30s,72℃30s,30个循环;95℃5min;95℃30s,53℃30s,72℃30s,10个循环;60℃30min。
所述步骤(5)中的加样缓冲液为分子量内标和甲酰胺混合液(0.5:8.5)9μl;pcr扩增产物加样量1μl。
所述步骤(5)中的变性的具体工艺为于95℃变性3min,结束后置于冰水混合物中冷却3min。
所述步骤(5)中的电泳的工艺参数为:变性聚丙烯酰胺凝胶为商用pop7胶,电泳缓冲液为3730bufferedta,注入电压2000v,运行电压15000v,进样时间10s,温度60℃,毛细管长度50cm,功率200w,电泳20min,电流和功率均为动态。
所述步骤(6)中的数据分析具体为:将检测得到的原始数据文件导入到分析软件genemapperid3.2中,运用popgene、ntsys等软件进行群体结构分析、聚类及杂合度的分析,核心种质资源计算分析。分析等位基因数(na,ne)、nei’s遗传多样性指数(he)、shannon’s多样性信息指数(i)和基因观测杂合度(ho)。
表2ssr引物扩增的等位片段信息汇总表
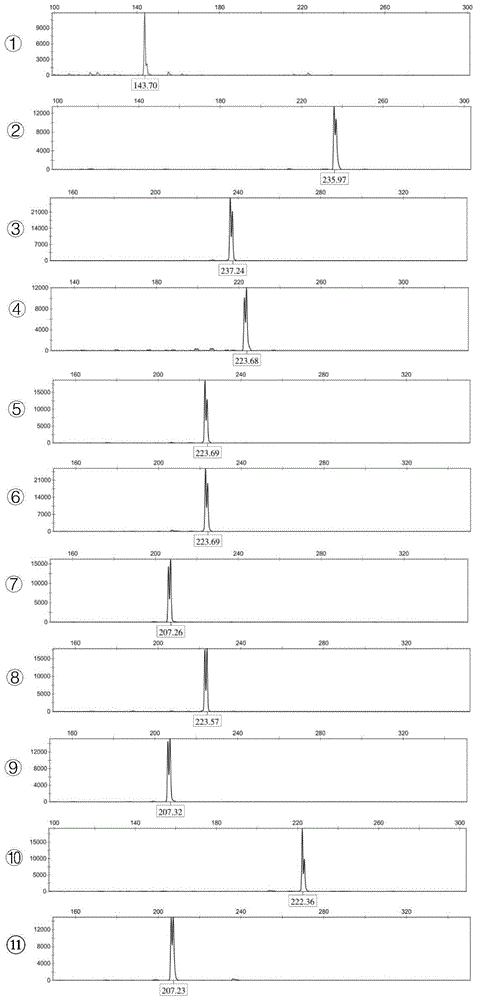
本发明的一种真姬菇“finc-b-7”菌种的ssr标记指纹图谱的应用,是利用真姬菇基因组简单重复序列片段开发的6对ssr引物,本发明对大量的ssr引物进行了筛选,通过对收集的56份真姬菇主要栽培品种的ssr引物带型扩增,确定了6对ssr引物在各个真姬菇栽培品种中扩增出的等位片段的数量并编号(表2),通过不同ssr等位位点的编号组合能够在所收集的56份主栽品种中有效识别“finc-b-7”菌种。通过毛细管电泳结合软件分析可确定各ssr引物扩增的等位位点的相对分子量,存在“finc-b-7”菌种特异ssr等位片段组合的菌种即是真姬菇“finc-b-7”菌种,该菌种的编号组合为:17/(9+8)/5/6/(13+16)/(7+9+11+4)。
本发明的有益效果:本发明与常规形态学检测、拮抗试验、出菇试验相比,具有检测时间短,准确性高,重复性好的优点。检测所需的操作时间在24h以内(包括提取基因组dna、pcr扩增、电泳分析、数据分析),而常规的拮抗试验所需时间至少需要两周时间,出菇试验则需要至少3个月的时间;本发明方法在所收集的56个市售真姬菇主栽培菌种中具有“finc-b-7”菌种的专一性,具有良好的应用前景。
附图说明
为了更清楚地说明本发明实施例的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动性的前提下,还可以根据这些附图获得其它的附图。其中:
图1为引物hmssr5分别在所选真姬菇栽培材料“finc-b-7”和几种主栽商业品种中依次检测得到的等位位点相对分子量峰图;
图2为引物hmssr26分别在所选真姬菇栽培材料“finc-b-7”和几种主栽商业品种中依次检测得到的等位位点相对分子量峰图;
图3为引物hmssr47分别在所选真姬菇栽培材料“finc-b-7”和几种主栽商业品种中依次检测得到的等位位点相对分子量峰图;
图4为引物hmssr48分别在所选真姬菇栽培材料“finc-b-7”和几种主栽商业品种中依次检测得到的等位位点相对分子量峰图;
图5为引物hmssr51分别在所选真姬菇栽培材料“finc-b-7”和几种主栽商业品种中依次检测得到的等位位点相对分子量峰图;
图6为引物hmssr59分别在所选真姬菇栽培材料“finc-b-7”和几种主栽商业品种中依次检测得到的等位位点相对分子量峰图。
具体实施方式
为使本发明的上述目的、特征和优点能够更加明显易懂,下面结合具体实施例对本发明的具体实施方式做详细的说明。
在下面的描述中阐述了很多具体细节以便于充分理解本发明,但是本发明还可以采用其他不同于在此描述的其它方式来实施,本领域技术人员可以在不违背本发明内涵的情况下做类似推广,因此本发明不受下面公开的具体实施例的限制。
其次,此处所称的“一个实施例”或“实施例”是指可包含于本发明至少一个实现方式中的特定特征、结构或特性。在本说明书中不同地方出现的“在一个实施例中”并非均指同一个实施例,也不是单独的或选择性的与其他实施例互相排斥的实施例。
实施例1:
(1)菌丝培养:将真姬菇菌种转接到马铃薯葡萄糖琼脂固体培养基上,22℃培养10d后收集菌丝;
(2)基因组dna的提取:利用真菌dna提取试剂盒,按照试剂盒实验步骤提取基因组dna,并取2μldna进行1.2%琼脂糖凝胶电泳检测。紫外分光光度法检测总基因组dna浓度和纯度,调整样品dna的浓度一致;
ctab法提取菌丝的基因组dna工艺包括:
①将真菌样本加入液氮充分研磨;
②向研磨好的粉末中迅速加入360μlbufferste和40μlbuffersds,迅速涡旋混匀后,将离心管放在65℃水浴15min,水浴过程中颠倒离心管以混合样本数次;
③加入5μlrnasesolution至裂解液中,涡旋混匀,室温静置15-30min;
④加入140μlbufferps,涡旋震荡30s,冰上放置10min;
⑤室温下,13000g离心5min,小心转移400μl上清液至新的离心管中;
⑥加入600μlbufferpbd(已用无水乙醇稀释)至样品中,,涡旋混匀30s;
⑦把dna结合柱装在收集管,转移一半混合液至柱子中,8000g离心1min;
⑧倒弃滤液把柱子装回收集管,转移剩余混合液至柱子中,8000g离心1min;
⑨倒弃滤液把柱子装回收集管,加入600μlbuffergw2(已用无水乙醇稀释)至柱子中,8000g离心1min;
⑩重复步骤9;
(3)ssr分子标记的检测:对上述提取的dna进行基因ssr标记的pcr扩增;
pcr扩增体系为:总体积10μl,包括:10×pcrbuffer1μl,2.5mmol/ldntp0.8μl,5u/μltakarahstaq酶0.1μl,5μmol/ltp-m130.5μl,5μmol/lssr标记特异引物总体积各0.6μl,浓度20ng~30ng/μl提取的模板dna1.2μl,ddh2o5.2μl;
pcr反应条件:95℃5min;95℃30s,59℃30s,72℃30s,30个循环;95℃5min;95℃30s,53℃30s,72℃30s,10个循环;60℃30min。
(4)电泳检测:将上述pcr扩增得到的产物1μl与9μl加样缓冲液混匀,95℃变性3min,结束后置于冰水混合物中冷却3min;点样3μl于变性聚丙烯酰胺凝胶上电泳,变性聚丙烯酰胺凝为商用pop7胶,电泳缓冲液为3730bufferedta,注入电压2000v,运行电压15000v,进样时间10s,温度60℃,毛细管长度50cm,功率200w,电泳20min,电流和功率均为动态,
(5)结果分析
采用6对ssr引物对真姬菇菌株进行pcr扩增和毛细管电泳,通过分析等位基因数(na,ne)、nei’s遗传多样性指数(he)、shannon’s多样性信息指数(i)和基因观测杂合度(ho),结合等位位点的相对分子量峰图,找到符合编号组合为:hmssr5、hmssr26、hmssr47、hmssr48、hmssr51、hmssr59,相应带型为:17/(9+8)/5/6/(13+16)/(7+9+11+4)的菌种,即可确定该菌种为真姬菇“finc-b-7”菌种。为保证鉴定的准确性,建议进行三次重复实验。
以几种主栽商业品种为例,给出6对引物依次检测得到的等位位点相对分子量峰图,如图1~6所示(依次为①finc-b-7②k③b2④15-x2⑤14-x1⑥13-x6⑦h2⑧bs027⑨x5⑩dtg
本发明所述指纹图谱,是指所述引物及其带型组合。
应说明的是,以上实施例仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的精神和范围,其均应涵盖在本发明的权利要求范围当中。
序列表
<110>上海丰科生物科技股份有限公司
<120>一种真姬菇finc-b-7菌种的ssr标记指纹图谱及其构建方法与应用
<130>2021020304-zf-wjn
<141>2021-02-03
<160>12
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>1
cacaccttacgaggtgagca20
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
tgtttgatgttagccgaacg20
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
gtgattgggttcgtgtcgtc20
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
acctcgagctcaacttctgc20
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
tctcatggaacgacctggag20
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
tccaaatccagactcttcgc20
<210>7
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>7
gccgtgtgacaccaaataca20
<210>8
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>8
acatctaaccgacccacgac20
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
caccccctcatcctacacat20
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
tggtcagaagaaacgtcgtg20
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
cactgttccagagtgggacc20
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
agggccttttcagagacgat20
- 还没有人留言评论。精彩留言会获得点赞!
- 2-仲丁基-5-羟基-4,8-二甲基-7,8-二氢-2H-吡喃[4,3,2-ij]异色原烯-3(9αH)-酮及其 ...的制作方法
- 7-羟基-8(2-羟基-3-甲基戊酰基)-3,7-二甲基-7,8-二氢-6h-异色原烯-6-酮及其制备方 ...的制作方法
- 拟茎点霉属内生真菌及其用途的制作方法
- 一株能产没食子酸的拟茎点霉菌的制作方法
- 一种草茎点霉的遗传转化方法
- 一种化合物及其制备方法和应用的制作方法
- 3-(2-羟基丙基)-1,5-二甲基-7-(2-甲基丁酰基)环戊烷[c]吡喃-6(7H)-酮及其制备方法 ...的制作方法
- 8-羟基-2-丙基色原烷-4-酮和5,8-二羟基-2-丙基色原烷-4-酮及其制备方法和应用的制作方法
- 一种化合物及其制备方法和应用的制作方法
- 桃溃疡病诊断方法