一种对硝基苄基酯酶突变体及应用
(一)技术领域
本发明涉及一种对硝基苄基酯酶突变体及其编码基因,以及其在酶催化dl-薄荷酯的手性拆分制备l-薄荷醇中的应用。
(二)
背景技术:
薄荷醇是目前需求量最大的环状单萜烯醇类香料,它一共有8种同分异构体,其中l-薄荷醇具有的独特薄荷香气和清凉效果,具有兴奋、杀菌镇痛、止痒、芳香清凉等功能,使得它在各产业中有非常广泛的应用,如食品,烟草,化妆品,口腔保健等产品。其也被广泛用于镇痛剂、局部麻醉剂等医药品以及作为手性化合物合成前体和辅助试剂等。
l-薄荷醇的生产方法包括植物提取法、化学合成法和生物酶手性拆分法等。植物提取法是目前l-薄荷醇的重要生产方法,因其具有天然来源的产品特性,备受消费者青睐。但是,其产品质量和价格受原料薄荷叶产地、光照的等多种因素影响。此外,提取液中富含原油杂质,难分离且能耗大,价格波动大。而化学法合成并进行光学拆分,生产成本高,工艺较为复杂,易带入毒性物质,不适合工业化应用。虽然已有化学不对称合成法,但技术难度较高。相较之下,生物酶法反应温和,原料价廉易得,对环境友好等因素推动研究者对生物法手性拆分的研究。
现有一个枯草芽孢杆菌来源的酯酶,在水解dl-乙酸薄荷酯手性拆分制备l-薄荷醇中具备良好的优点,包括反应速率快,底物投料量大等,但是其缺点是酶的立体选择性不高,且需要添加助溶剂如乙醇或丁醇等来提高酶的催化速率和催化的立体选择性。因此需要对该酯酶进行定向改造,提高其应用性能。
随着酶的晶体结构不断被解析,基于生物信息学的计算模拟技术的不断改进,酶的半理性设计并定向进化和筛选的成功显著提高。已使用的大量工业酶都经过人工改造,特别是在酯酶和脂肪酶性质改造领域取得很大的成功,主要集中在提高酶热稳定性,提高酶的催化反应活性,改进底物特异性以及提高对映体选择性等。
(三)
技术实现要素:
本发明目的是提供一种立体选择性好的对硝基苄基酯酶突变体及其编码基因,以及其在酶催化dl-乙酸薄荷酯的手性拆分制备l-薄荷醇中的应用。
本发明采用的技术方案是:
一种对硝基苄基酯酶突变体,由氨基酸序列如seqidno:2所示的野生型对硝基苄基酯酶在315位进行定点饱和突变获得。
酯酶的结构一般都存在两个活性口袋,包括疏水(体积较大)和亲水(体积较小)两个口袋。对于底物dl-薄荷酯,在催化机理中,底物六元环上的异丙基朝向酶催化口袋中的疏水口袋,底物六元环上的甲基则是与酶的亲水口袋相结合。这是一个针对于薄荷酯催化最有利的位置(holmquistmetal.2010.proteinscience,5:83-88),如图2所示(m表示中等取代基,l为大取代基)。通过改变活性口袋周围的残基,从而影响残基侧链效应,改变酶口袋的大小,使得不同底物的异构体与酶结合上造成差异,从而提高酶选择性。
seqidno:2所示的野生型对硝基苄基酯酶(pnb)来源于枯草芽孢杆菌(bacillussubtilis)cgmccno.17904(cn110373366a),可对dl-薄荷酯实现高效地手性拆分,用于制备l-薄荷醇,特别是其水解速率快,dl-薄荷酯底物投料量高。但是,该酶催化过程中需添加助溶剂如丁醇或乙醇,用来提高底物的立体选择性。若在无助溶剂体系下,酶催化的底物立体选择性较低。而在催化体系中添加有机溶剂,不但增加了生产成本,也增加了产物l-薄荷醇分离纯化的难度。本发明通过对野生型酶的突变改造,提高了酶在无助溶剂体系的催化速率,同时显著提高其在无助溶体系下对底物的立体选择性。
通过pcr扩增得到该对硝基苄基酯酶pnb基因,将其克隆至大肠杆菌bl21(de3)的表达载体pet28a,进行酶的过量表达。结合酶三维结构计算模拟,针对酯酶pnb底物结构口袋中对底物立体选择性起到关键作用的氨基酸位点进行饱和突变,再进行酶学比较分析后筛选得到本发明酯酶突变体。
优选的,所述对硝基苄基酯酶突变体氨基酸序列如seqidno:3所示。
本发明还涉及编码所述对硝基苄基酯酶突变体的基因。
所述的酯酶突变体pnb-f315e的编码基因,可以根据其氨基酸序列进行密码子优化后合成的基因序列,亦可从b.subtiliscgmccno.17904菌株基因组pcr扩增后突变改造的基因序列。优选的,所述编码基因核苷酸序列如seqidno:4所示。
本发明还涉及所述对硝基苄基酯酶突变体在酶催化dl-薄荷酯的手性拆分制备l-薄荷醇中的应用。
具体的,所述应用为:构建含有所述突变体编码基因的重组菌,以重组菌经发酵培养获得的湿菌体或菌体经破碎获得的含酶细胞为催化剂,对dl-薄荷酯进行水解,获得l-薄荷醇。
可将该酯酶突变体pnb-f315e的基因克隆到表达质粒,并转化到宿主细胞中,经过诱导发酵制作成酯酶制剂后,用于催化dl-薄荷酯选择性水解制备l-薄荷醇。所述的表达质粒和宿主细胞,优选pet28a质粒和大肠杆菌bl21(de3)宿主细胞,即构建重组菌e.colibl21(de3)(pet28a-pnb-f315e)。
所述水解可在无溶剂条件下进行。
优选的,水解反应体系中,细胞密度为5~30od(优选10od),dl-乙酸薄荷酯用量为1%~20%(w/w)(优选15%),水解过程控制ph6.5~8.5(优选ph8.0)、温度25℃~40℃(优选30℃)。
所述dl-薄荷酯优选为dl-乙酸薄荷酯。本发明酯酶突变体,在无助溶剂体系下,催化水解1%~20%的dl-乙酸薄荷酯制备l-薄荷醇,其l-乙酸薄荷酯底物转化率>70%,产物l-薄荷醇的eep值>95%。
本发明的有益效果主要体现在:①本发明酯酶突变体无需添加助溶剂,酶催化活性维持较高水平;②在无助溶剂体系下,突变体酶催化的立体选择性较野生型显著提高。将该突变体酶通过大肠杆菌过量表达后用于催化反应,并以10~200g/l的dl-乙酸薄荷酯为底物,进行水解拆分制备l-薄荷醇,结果表明l-乙酸薄荷酯底物转化率>70%,产物l-薄荷醇的eep值>95%,表现出良好的工业应用性能。
(四)附图说明
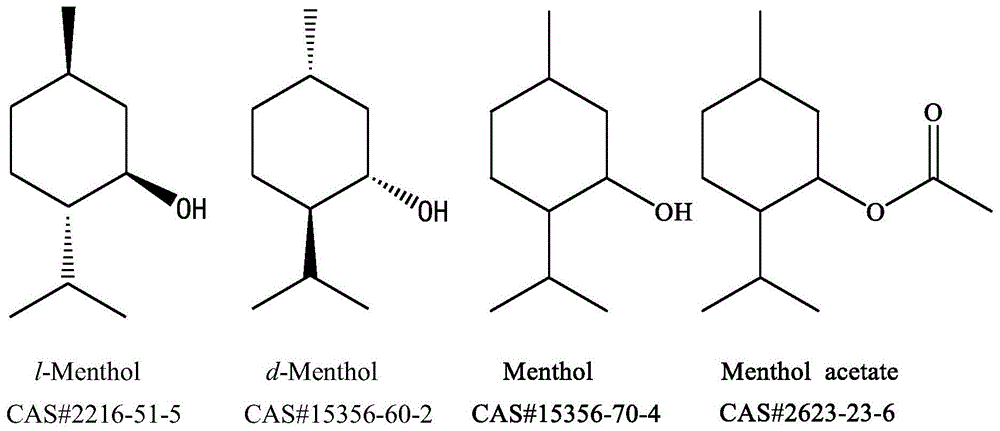
图1为l-薄荷醇、d-薄荷醇、dl-薄荷醇、dl-乙酸薄荷酯的结构示意图。
图2为酯酶pnb的底物结合口袋示意图。
图3为dl-乙酸薄荷酯、dl-薄荷醇标准样品的gc色谱图。
图4为酯酶突变体f315e催化dl-乙酸薄荷酯水解拆分制备l-薄荷醇的催化反应液的gc色谱图。
(五)具体实施方式
为了加深对本发明的理解,下面将结合具体实施例对本发明做进一步详细描述,该实施例仅用于解释本发明,并不对本发明的保护范围构成限定。
lb培养基:酵母粉5.0g/l、蛋白胨10.0g/l、nacl10.0g/l,溶剂为蒸馏水。
发酵培养基:酵母粉12.0g/l、蛋白胨15.0g/l、na2hpo4·12h2o8.9g/l、kh2po43.4g/l、nh4cl2.67g/l、na2so40.71g/l、mgso4·7h2o0.49g/l、卡那霉素50μg/l、ph7.0,溶剂为蒸馏水。
ph8.0磷酸缓冲溶液(200mm):na2hpo4·12h2o67.8g/l,nah2po4·2h2o0.82g/l,溶剂为蒸馏水。
实施例1:野生型酯酶pnb基因的克隆表达
利用pcr技术扩增b.subtiliscgmccno.17904基因组的酯酶pnb基因。扩增引物为pnb-f和pnb-r。
pnb-f:gaaggagatataccatgggcactcatcaaatagtaacgac
pnb-r:cggagctcgaattcggatccttattctccttttgaagggaatag
将pcr扩增得到的pnb基因片段,经琼脂糖凝胶电泳分离纯化后,用于连接反应。提取质粒pet-28a,用限制性核酸内切酶ncol/bamhi对质粒进行双酶切,琼脂糖凝胶电泳分离纯化后用于连接反应。采用诺维赞公司一步克隆试剂盒(clonexpressiionestepcloningkit),对纯化的pnb基因片段和纯化的线性化pet-28a质粒进行连接反应。取10μl连接产物,与100μl大肠杆菌e.colibl21(de3)感受态细胞混合,冰浴放置30min后,在42℃水浴中热击90s。将热击后的感受态细胞涂布于lb(含50μg/mlkana)平板。在37℃培养箱中生长12h后。将转化子用于菌落pcr验证,划线纯化后,接种lb液体培养基中培养,并提取质粒,测序验证,最终获得表达野生型酯酶pnb重组菌株e.colibl21(de3)(pet-28a-pnb),简称e.coliief-pnb。
野生型酯酶pnb的诱导表达。将菌株e.coliief-pnb从保藏的甘油管中划线培养过夜后,取单菌落接种到含有50μg/ml卡那霉素的lb液体培养基,37℃,200rpm过夜培养。按照0.5%(v/v)接种量接种到发酵培养基中,37℃,培养2.5h;加入终浓度为1.0mm的iptg,调整培养温度25℃,继续发酵5h,得到过量表达野生型酯酶pnb的菌剂。
实施例2:野生型酯酶pnb催化水解dl-乙酸薄荷酯制备l-薄荷醇的活性分析
取上述方法制备的野生型酯酶pnb菌剂2ml,10000×g,10min离心收集细胞,将细胞重新悬浮于1ml的ph8.0的200mm磷酸缓冲液中;取两份重悬菌剂,一份添加10%(v/v)正丁醇作为助溶剂(助溶剂种类已优化),另一份添加等量的纯水为对照;加入20μl的dl-乙酸薄荷酯,在30℃、1200rpm条件下金属浴振荡催化2h,催化液用于gc分析。
催化液的gc分析结果如表1所示,在有机溶剂正丁醇添加的催化体系下,其对dl-乙酸薄荷酯底物的立体选择性从70.13%提高到92.01%,所以,在助溶剂正丁醇体系中,野生型pnb酶对底物立体选择性显著提高。
表1:无溶剂与丁醇体系下pnb酶催化结果
催化液取样的gc分析方法:
1)样品前处理:500μl催化反应液,加入到同等比例的乙酸乙酯混合萃取;12000×g离心1min,取上清有机相用于色谱分析。
2)气相色谱检测条件:色谱柱:安捷伦cyclodex-b60m×0.250mm。升温程序:80℃,保持10min;5℃/min升温至140℃,保持5min;1℃/min升温至145℃,保持1min。检测器:fid检测器;产物l-薄荷醇出峰时间约36min,底物薄荷酯出峰时间约40min,dl-乙酸薄荷酯、dl-薄荷醇标准品的gc色谱图如图3所示,从左到右分别为d-薄荷醇、l-薄荷醇、l-乙酸薄荷酯、d-乙酸薄荷酯。
实施例3:酯酶pnb突变位点的设计和突变体的构建与筛选
为了提高野生型酯酶pnb在无助溶剂体系下的底物立体选择性,采用计算机辅助设计选择pnb的定点突变位点。根据已报道枯草芽孢杆菌来源的酯酶晶体结构(pdbid:1qe3),对菌株b.subtiliscgmccno.17904来源的酯酶pnb进行同源建模。利用蛋白质三维结构分析软件和分子对接软件,进行模拟分析。综合考虑酯酶与底物对接结果、酶的底物结合口袋特点、酶识辨底物立体选择的结构特点、酶的催化机制,最终对氨基酸序列seqidno:2的第315位点的苯丙氨酸残基功能预测:该位点处于酶催化疏水口袋入口处,疏水口袋与底物六元环上的较大基团(异丙基)的结合有密切的关联,这也是确定酶的立体选择性的关键位点。酶与底物l-薄荷乙酸酯结合关系如图2所示(黄色棍棒结构为靠近褐色疏水口袋的315位点苯丙氨酸)。因此,seqidno.2第315位的氨基酸被优先作为突变分析位点。
根据seqidno.2所示的氨基酸序列,设计第315位氨基酸的饱和突变引物,见表2所示。以载体pet28a-pnba为模板,进行pcr扩增整个质粒,经过0.8%琼脂糖凝胶电泳检测,得到大小正确的扩增条带。用限制性内切酶dpni酶消化处理pcr产物1h,消化甲基化的质粒模板。将消化后的pcr产物进行一步克隆法进行连接反应,随后转化至大肠杆菌bl21(de3)细胞,经过菌落pcr验证和测序验证,即获得了目标位点发生突变的酯酶突变体。
表2:基于多序列比对结果的突变引物设计表
实施例4:第315位氨基酸的饱和突变体库的筛选
在seqidno:2所述第315氨基酸位点通过定点突变获得19种突变体,且在相同催化条件下评价突变酶的催化活性和对底物立体选择性的差异。将平板上的转化子转移到含有50μg/ml卡那霉素的lb液体培养基中培养至对数生长中期。再按1%(v/v)的接种量转接至发酵培养基中,37℃,培养3h;加入终浓度为0.5mm的iptg,控制发酵温度25℃,继续发酵12h,得到诱导表达后的菌剂。
取培养好的含酯酶突变体的菌剂2ml,10000×g,10min离心收集细胞,将细胞重新悬浮于1ml的ph8.0的200mm磷酸缓冲液中;加入20μl的dl-乙酸薄荷酯,在30℃、1200rpm条件下金属浴振荡催化2h,催化液用于gc分析。
野生型pnb和19个pnb突变体的酶活和底物立体选择性的分析结果如表3所示。综合考虑底物选择性与酶的催化速率,f315e突变株为最优突变体。
表3:野生型pnb和19个pnb突变体的酶活和底物立体选择性比较
实施例5:突变株f315e在催化水解dl-乙酸薄荷酯制备l-薄荷醇中的应用
酯酶突变株f315e的诱导表达。将菌株e.coliief-pnb-f315e从保藏的甘油管中划线培养过夜后,取单菌落接种到含有50μg/ml卡那霉素的lb液体培养基,37℃,200rpm过夜培养。按照0.5%(v/v)接种量接种到发酵培养基中,37℃,培养2.5h;加入终浓度为1.0mm的iptg,调整培养温度25℃,继续发酵5h,得到酯酶突变株f315e的菌剂。
取酯酶突变株f315e发酵液菌剂,并离心得到湿菌体4g,用50ml的ph8.0的100mm磷酸钠缓冲液重悬,分别加入不同量的dl-乙酸薄荷酯,包括20g/l,150g/l,200g/l,在30℃,500rpm机械磁力搅拌下进行催化反应。在催化反应过程中,用2mnaoh溶液调控反应ph8.0。反应液用于气相色谱分析,如图4所示,从左到右分别为d-薄荷醇、l-薄荷醇、l-乙酸薄荷酯、d-乙酸薄荷酯。不同的投料量催化反应结果如表4所示,其中150g/l投料下,转化率>70%,eep>95%。
表4:突变株f315e在不同投料量下催化结果
序列表
<110>安徽丰乐香料有限责任公司
浙江工业大学
<120>一种对硝基苄基酯酶突变体及应用
<160>4
<170>siposequencelisting1.0
<210>1
<211>1473
<212>dna
<213>枯草芽孢杆菌(bacillussubtilis)
<400>1
atgggcactcatcaaatagtaacgactcaatacggcaaagtaaaaggcacaacggaaaac60
ggcgtacataagtggaaaggcatcccctatgccaagccgcctgtcggacaatggcgtttt120
aaagcacctgagccgcctgaagtgtgggaagatgtccttgatgccacagcgtacggccct180
atttgcccgcagccgtctgatttgctctcactgtcgtatgctgagttgccccgccagtcc240
gaggattgcttgtatgtcaatgtatttgcgcctgacactccaagccaaaacctgcctgtc300
atggtttggattcacggaggcgctttttatctcggcgcaggcagtgagccattgtatgac360
ggatcaaaacttgcggcgcagggagaggtcattgtcgttacactgaactatcggctgggg420
ccgtttggctttttgcacttgtcttcctttgatgaggcgtattccgataaccttgggctt480
ttagaccaagtcgccgcactgaaatgggtgcgggagaatatttcagcgtttggcggtgat540
cccgataacgtaacagtatttggagaatccgccggcgggatgagcattgccgcgcttctc600
gctatgcctgcggcaaaaggcctgttccagaaagcaatcatggaaagcggcgcttctcga660
acgatgacgaaagaacaagcggcgagcacctcggcagcctttttacaggtccttgggatt720
aacgagggccaactggataaattgcatacggtttctgcggaagatttgctaaaagcggct780
gatcagcttcggattgcagaaaaagaaaatatctttcagctgttcttccagcccgccctt840
gatccgaaaacgctgcctgaagaaccagaaaaagcgatcgcagaaggggctgcttctggc900
attccgctattgattggaacaacccgtgatgaaggatatttatttttcaccccggattca960
gacgttcattctcaggaaacgcttgatgcagcactcgagtatttactagggcagccgctg1020
gccaagaatgccgccgatttgtatccgcgttctctggaaagccaaattcaaatgatgact1080
gatttattattttggcgccctgccgtcgcctatgcatccgcacagtctcattacgcccct1140
gtctggatgtacaggttcgattggcacccggagaagccgccgtacaataaagcgtttcac1200
gcattagagcttccttttgtctttggaaatctggacggattggaacgaatggcacaagcg1260
gagattacggatgaggtgaaacagctttctcacacgatacaatcagcgtggatcacgttc1320
gctaaaacaggaaacccaagcaccgaagctgtgaattggccggcgtatcatgaagaaacg1380
agagagacgctgattttagactcagagattacgatcgaaaacgatcccgaatctgaaaaa1440
aggcagaagctattcccttcaaaaggagaataa1473
<210>2
<211>490
<212>prt
<213>枯草芽孢杆菌(bacillussubtilis)
<400>2
metglythrhisglnilevalthrthrglntyrglylysvallysgly
151015
thrthrgluasnglyvalhislystrplysglyileprotyralalys
202530
proprovalglyglntrpargphelysalaprogluproprogluval
354045
trpgluaspvalleuaspalathralatyrglyproilecysprogln
505560
proseraspleuleuserleusertyralagluleuproargglnser
65707580
gluaspcysleutyrvalasnvalphealaproaspthrprosergln
859095
asnleuprovalmetvaltrpilehisglyglyalaphetyrleugly
100105110
alaglysergluproleutyraspglyserlysleualaalaglngly
115120125
gluvalilevalvalthrleuasntyrargleuglypropheglyphe
130135140
leuhisleuserserpheaspglualatyrseraspasnleuglyleu
145150155160
leuaspglnvalalaalaleulystrpvalarggluasnileserala
165170175
pheglyglyaspproaspasnvalthrvalpheglygluseralagly
180185190
glymetserilealaalaleuleualametproalaalalysglyleu
195200205
pheglnlysalailemetgluserglyalaserargthrmetthrlys
210215220
gluglnalaalaserthrseralaalapheleuglnvalleuglyile
225230235240
asngluglyglnleuasplysleuhisthrvalseralagluaspleu
245250255
leulysalaalaaspglnleuargilealaglulysgluasnilephe
260265270
glnleuphepheglnproalaleuaspprolysthrleuprogluglu
275280285
proglulysalailealagluglyalaalaserglyileproleuleu
290295300
ileglythrthrargaspgluglytyrleuphephethrproaspser
305310315320
aspvalhisserglngluthrleuaspalaalaleuglutyrleuleu
325330335
glyglnproleualalysasnalaalaaspleutyrproargserleu
340345350
gluserglnileglnmetmetthraspleuleuphetrpargproala
355360365
valalatyralaseralaglnserhistyralaprovaltrpmettyr
370375380
argpheasptrphisproglulysproprotyrasnlysalaphehis
385390395400
alaleugluleuprophevalpheglyasnleuaspglyleugluarg
405410415
metalaglnalagluilethraspgluvallysglnleuserhisthr
420425430
ileglnseralatrpilethrphealalysthrglyasnproserthr
435440445
glualavalasntrpproalatyrhisglugluthrarggluthrleu
450455460
ileleuaspsergluilethrilegluasnaspprogluserglulys
465470475480
argglnlysleupheproserlysglyglu
485490
<210>3
<211>490
<212>prt
<213>未知(unknown)
<400>3
metglythrhisglnilevalthrthrglntyrglylysvallysgly
151015
thrthrgluasnglyvalhislystrplysglyileprotyralalys
202530
proprovalglyglntrpargphelysalaprogluproprogluval
354045
trpgluaspvalleuaspalathralatyrglyproilecysprogln
505560
proseraspleuleuserleusertyralagluleuproargglnser
65707580
gluaspcysleutyrvalasnvalphealaproaspthrprosergln
859095
asnleuprovalmetvaltrpilehisglyglyalaphetyrleugly
100105110
alaglysergluproleutyraspglyserlysleualaalaglngly
115120125
gluvalilevalvalthrleuasntyrargleuglypropheglyphe
130135140
leuhisleuserserpheaspglualatyrseraspasnleuglyleu
145150155160
leuaspglnvalalaalaleulystrpvalarggluasnileserala
165170175
pheglyglyaspproaspasnvalthrvalpheglygluseralagly
180185190
glymetserilealaalaleuleualametproalaalalysglyleu
195200205
pheglnlysalailemetgluserglyalaserargthrmetthrlys
210215220
gluglnalaalaserthrseralaalapheleuglnvalleuglyile
225230235240
asngluglyglnleuasplysleuhisthrvalseralagluaspleu
245250255
leulysalaalaaspglnleuargilealaglulysgluasnilephe
260265270
glnleuphepheglnproalaleuaspprolysthrleuprogluglu
275280285
proglulysalailealagluglyalaalaserglyileproleuleu
290295300
ileglythrthrargaspgluglytyrleugluphethrproaspser
305310315320
aspvalhisserglngluthrleuaspalaalaleuglutyrleuleu
325330335
glyglnproleualalysasnalaalaaspleutyrproargserleu
340345350
gluserglnileglnmetmetthraspleuleuphetrpargproala
355360365
valalatyralaseralaglnserhistyralaprovaltrpmettyr
370375380
argpheasptrphisproglulysproprotyrasnlysalaphehis
385390395400
alaleugluleuprophevalpheglyasnleuaspglyleugluarg
405410415
metalaglnalagluilethraspgluvallysglnleuserhisthr
420425430
ileglnseralatrpilethrphealalysthrglyasnproserthr
435440445
glualavalasntrpproalatyrhisglugluthrarggluthrleu
450455460
ileleuaspsergluilethrilegluasnaspprogluserglulys
465470475480
argglnlysleupheproserlysglyglu
485490
<210>4
<211>1473
<212>dna
<213>未知(unknown)
<400>4
atgggcactcatcaaatagtaacgactcaatacggcaaagtaaaaggcacaacggaaaac60
ggcgtacataagtggaaaggcatcccctatgccaagccgcctgtcggacaatggcgtttt120
aaagcacctgagccgcctgaagtgtgggaagatgtccttgatgccacagcgtacggccct180
atttgcccgcagccgtctgatttgctctcactgtcgtatgctgagttgccccgccagtcc240
gaggattgcttgtatgtcaatgtatttgcgcctgacactccaagccaaaacctgcctgtc300
atggtttggattcacggaggcgctttttatctcggcgcaggcagtgagccattgtatgac360
ggatcaaaacttgcggcgcagggagaggtcattgtcgttacactgaactatcggctgggg420
ccgtttggctttttgcacttgtcttcctttgatgaggcgtattccgataaccttgggctt480
ttagaccaagtcgccgcactgaaatgggtgcgggagaatatttcagcgtttggcggtgat540
cccgataacgtaacagtatttggagaatccgccggcgggatgagcattgccgcgcttctc600
gctatgcctgcggcaaaaggcctgttccagaaagcaatcatggaaagcggcgcttctcga660
acgatgacgaaagaacaagcggcgagcacctcggcagcctttttacaggtccttgggatt720
aacgagggccaactggataaattgcatacggtttctgcggaagatttgctaaaagcggct780
gatcagcttcggattgcagaaaaagaaaatatctttcagctgttcttccagcccgccctt840
gatccgaaaacgctgcctgaagaaccagaaaaagcgatcgcagaaggggctgcttctggc900
attccgctattgattggaacaacccgtgatgaaggatatttagagttcaccccggattca960
gacgttcattctcaggaaacgcttgatgcagcactcgagtatttactagggcagccgctg1020
gccaagaatgccgccgatttgtatccgcgttctctggaaagccaaattcaaatgatgact1080
gatttattattttggcgccctgccgtcgcctatgcatccgcacagtctcattacgcccct1140
gtctggatgtacaggttcgattggcacccggagaagccgccgtacaataaagcgtttcac1200
gcattagagcttccttttgtctttggaaatctggacggattggaacgaatggcacaagcg1260
gagattacggatgaggtgaaacagctttctcacacgatacaatcagcgtggatcacgttc1320
gctaaaacaggaaacccaagcaccgaagctgtgaattggccggcgtatcatgaagaaacg1380
agagagacgctgattttagactcagagattacgatcgaaaacgatcccgaatctgaaaaa1440
aggcagaagctattcccttcaaaaggagaataa1473
- 还没有人留言评论。精彩留言会获得点赞!