梨品种特异性分子标记及其筛选方法和应用与流程
1.本发明涉及12个梨品种特异性分子标记及其筛选方法和应用,属于生物技术领域。
背景技术:
2.梨为蔷薇科(rosaceae)梨亚科(pomaceae)梨属(pyrus l.)植物,是我国主栽果树之一,栽培面积和产量仅次于苹果。我国是世界梨的三大起源中心之一,也是梨生产第一大国,产量占全球总量的65%。梨属植物分为欧洲种群、地中海种群、中亚种群、东亚种群四大种群。秋子梨系统、西洋梨系统、白梨系统、砂梨系统是目前梨树的主要栽培品种。梨品种选育历史较长,目前在我国常见的品种有100多个,品种多、结构不合理,品种更新速度慢;良种苗木繁育体系不健全,苗木质量缺乏保障,梨园管理多采用传统模式,省力化栽培管理技术研发及应用不足是我国梨发展的制约因素。近年来,随着国外优良品种和栽培技术的引进,以及分子辅助育种技术应用,新优品种和栽培管理模式正在逐步进入和替代传统育种、繁育和种植模式。
3.本发明中的12个梨品种选自近年来我国主要梨种植产区推广的新优品种,性状优良,品质高端,占据梨产品的高端市场,是品种繁育部门和企业果园生产的主流产品。因此对这些品种品系的准确识别鉴定,对科研部门新品种选育、资源苗圃建立、企业苗木繁育销售、果园苗木种植生产等环节具有重大意义。
技术实现要素:
4.本发明提供12个梨品种的特异性分子标记位点及其筛选方法和应用,共筛选到4479个品种特异性的snp标记,基于这些标记开发出两种简便、快速、可靠鉴定12个梨品种的方法,为从源头上准确控制梨品种奠定技术基础。
5.本发明采用下述技术方案予以实现:
6.提供一种利用简化基因组测序技术super
‑
gbs筛选12个梨品种的特异性分子标记的方法,包括如下步骤:
7.(1)利用品种已知且准确的秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水梨品种为样品,取叶片提取基因组dna;
8.(2)按照简化基因组测序技术super
‑
gbs方法构建文库,文库质检合格后上机测序;
9.(3)对测序数据进行过滤后,使用gatk获得snp位点;
10.(4)对snp进行过滤,过滤条件为:snp测序深度不低于4;剔除maf小于0.01的位点;剔除snp分型缺失率高于20%的位点;剔除所有样品中分型一致的位点;
11.(5)根据每个品种的分型结果,筛选每个品种内所有个体分型完全一致、分型一致位点没有个体缺失,且与其他品种不同的位点;最终筛选到4479个snp位点,具体见表1;
12.(6)利用4479个snp位点或者其中的部分snp位点构建进化树和聚类分析。
13.表1中,表头部分的数字1
‑
12分别表示的品种是秋月梨、豊水、jumbo新高、幸水、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金。
14.其次,本发明提供一种利用pcr方法鉴定12个梨品种的方法,包括如下步骤:
15.(1)从表1的snp位点中筛选几个snp位点组合成能够鉴定12个梨品种的位点群;
16.(2)根据位点所在的基因组位置设计特异性的pcr扩增引物;
17.(3)提取12个梨品种的叶片基因组dna;
18.(4)依据权利要求2所述的snp标记群筛选方法进行筛选,筛选出的三个位点群及位点对应的特异性引物,如下表2
‑
7;
19.(5)利用能够扩增出snp位点的引物进行pcr扩增;
20.(6)将获得的pcr产物进行一代测序;
21.(7)根据序列信息对照snp位点信息进行梨品种的分析和品种鉴定。
22.本发明能够筛选出几千组准确鉴定12个梨品种的snp位点标记群,其中本发明列举三组能够准确鉴定12个梨品种的snp位点标记群信息及标记扩增引物信息如下:
23.表2能够准确鉴定12个梨品种的一组snp位点信息
[0024][0025]
表3表2中snp位点鉴定引物信息
[0026]
[0027][0028]
表4能够准确鉴定12个梨品种的一组snp位点信息
[0029][0030]
表5表4中snp位点鉴定引物信息
[0031][0032]
表6能够准确鉴定12个梨品种的一组snp位点信息
[0033][0034][0035]
表7表6中snp位点鉴定引物信息
[0036][0037]
本发明的有益效果:梨品种基因组差异性小,在筛选过程中需要过滤掉大量干扰信息最终才得到能够准确鉴定12个梨品种的snp位点,这些位点可以一起使用,也可以合理组合分组使用,都能够准确鉴定秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水的组培苗、嫁接苗、成品苗,确保育苗企业对品种的把控,减少繁育过程中误差造成的经济损失。
附图说明
[0038]
下面结合附图对本发明的具体实施方式作进一步详细的说明。
[0039]
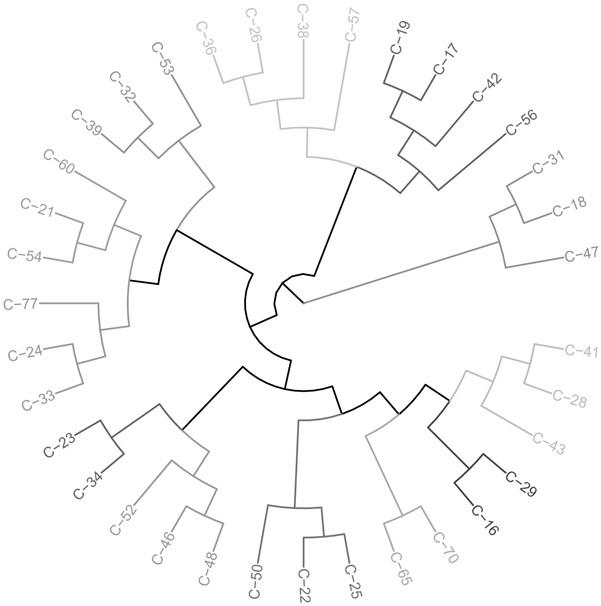
图1附图为利用筛选到的秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水共12个梨品种的4479个特异snp标记(snp标记见表1)构建进化树,能够准确的区分12个梨品种。
[0040]
图2附图为从利用筛选到的4479个snp位点中90%以上的位点进行12个梨品种的分类,能够准确的区分12个梨品种。
[0041]
说明,本发明的图1和图2中,样品编号对应的品种如下表所示:
[0042]
序号品种样品数量样品编号1秋月梨3c
‑
16、c
‑
29、c
‑
452黄金梨4c
‑
61、c
‑
50、c
‑
22、c
‑
253皇冠4c
‑
39、c
‑
32、c
‑
53、c
‑
644早酥5c
‑
19、c
‑
17、c
‑
42、c
‑
56、c
‑
665锦丰4c
‑
31、c
‑
18、c
‑
47、c
‑
716新梨7号2c
‑
23、c
‑
347玉露香4c
‑
54、c
‑
21、c
‑
60、c
‑
698香梨4c
‑
24、c
‑
33、c
‑
77、c
‑
809华金5c
‑
36、c
‑
26、c
‑
38、c
‑
57、c
‑
7510豊水4c
‑
41、c
‑
28、c
‑
43、c
‑
6311jumbo新高4c
‑
55、c
‑
46、c
‑
48、c
‑
5212幸水3c
‑
78、c
‑
65、c
‑
70
具体实施方式
[0043]
实施例1
[0044]
本实施例提供一种利用简化基因组测序技术super
‑
gbs筛选秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水共12个梨品种特异性snp分子标记的方法,包括如下步骤:
[0045]
(1)利用品种准确的2株秋月梨、3株黄金梨、3株皇冠、4株早酥、3株锦丰、2株新梨7号、3株玉露香、3株香梨、4株华金、3株豊水、3株jumbo新高和2株幸水梨品种样品,取叶片提取基因组dna;
[0046]
(2)按照简化基因组测序技术super
‑
gbs方法构建文库,文库质检合格后上机测序;
[0047]
(3)对测序数据进行过滤后,使用gatk获得snp位点;
[0048]
(4)对snp进行过滤,过滤条件为:snp测序深度不低于4;剔除maf小于0.01的位点;剔除snp分型缺失率高于20%的位点;剔除所有样品中分型一致的位点;
[0049]
(5)根据每个品种的分型结果,筛选每个品种内所有个体分型完全一致、分型一致位点没有个体缺失,且与其他11个梨品种中至少一个品种位点不同的snp位点,最终筛选到4479个snp位点;
[0050]
(6)利用4479个snp位点构建进化树和聚类分析。
[0051]
具体的操作步骤如下:
[0052]
本实施例主要包含以下步骤,即酶切、连接、纯化、扩增、混库和分析。
[0053]
1.酶切:
[0054]
对本公司购买的权威机构提供的品种准确的2株秋月梨、3株黄金梨、3株皇冠、4株早酥、3株锦丰、2株新梨7号、3株玉露香、3株香梨、4株华金、3株豊水、3株jumbo新高和2株幸水梨品种进行super
‑
gbs建库,具体过程如下(以下为每个样品酶切试剂使用量):
[0055][0056]
所有成分混匀后37℃酶切2h,然后75℃保温20min使酶灭活。
[0057]
2.连接:
[0058]
在40μl体系中连接adapter,barcode及酶切片段。
[0059][0060]
所有成分混匀后22℃酶切2h,然后65℃保温20min使酶灭活。
[0061]
3.纯化:
[0062]
取连接产物35μl加入0.7倍体积的sera
‑
mag beads(ge healthcare life sciences),室温静置5min,以去除300bp以下小片段。从上清中回收磁珠,并用200μl 70%酒精洗脱3遍。最后用40μl 10mm tris.hcl(ph 8.0)从磁珠中回收dna。
[0063]
4.扩增:
[0064][0065]
所有成分混匀后置于pcr仪中,反应条件为95℃预变性30s,扩增16个循环,每个循环95℃变性30s,62℃退火20s,68℃延伸15s,最后在68℃延伸5min,4℃保存。
[0066]
5.混库:
[0067]
使用qubit测定每个样品的文库浓度,浓度大于5ng/μl的样品用于混库测序。通过加入0.7倍体积的sera
‑
mag beads去除文库中的引物和小片段,然后根据测序量需求进行混样测序,测序平台为illumina nova pe150。建库过程所用的接头和引物序列详见下表8。
[0068]
表8 super
‑
gbs测序文库构建接头及引物序列
[0069]
名称序列(5
’‑3’
)common adaptor topgatcggtctcggcattcctgctgaaccgctcttccgatctcommon adaptor botcgagatcggaagagcggggactttaagcpsti adaptor topcacgacgctcttccgatctaacxxxxxxtgcapsti adaptor botyyyyyyagatcggaagagcgtcgtgprimer1aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatctprimer2caagcagaagacggcatacgagatcggtctcggcattcctgctgaa
[0070]
6.分析:
[0071]
对12个梨品种,共35个样品进行super
‑
gbs测序,一共得到96m高质量reads。将高质量reads比对到参考基因组上,比对率为77.37%~8693%,所有样品的平均测序深度为37.52
×
。利用gatk(v3.8
‑
1)软件得到snp位点,然后筛选12个品种间至少有一个品种与其它品种不同的snp位点,最终获得4479个snp位点,采用treebest软件分析,利用r包ggtree绘图,这些位点可用于12个梨品种的准确鉴定,鉴定结果见图1。
[0072]
实施例2
[0073]
本实施例提供按照本发明获得的4479个snp标记中的一部分对山东大丰园农业有限公司品种圃随机采集的11个梨品种进行品种鉴定,同时加入已知品种的12种梨树苗样品(实施例1中样品)的测序数据作为对照进行试验和验证。包括如下步骤:
[0074]
(1)从山东大丰园农业有限公司品种圃随机采集秋月梨、黄金梨、皇冠、早酥、锦丰、玉露香、香梨、华金、豊水、jumbo新高和幸水各1株,提取基因组dna;
[0075]
(2)按照简化基因组测序技术super
‑
gbs方法构建文库,文库质检合格后上机测序;
[0076]
(3)对测序数据进行过滤后,使用gatk获得snp位点;
[0077]
(4)对snp进行过滤,过滤条件为:snp测序深度不低于4;剔除maf小于0.01的位点;剔除snp分型缺失率高于20%的位点;剔除所有样品中分型一致的位点。保留与表1中的4479个位点重合的snp位点,最终获得4255个snp位点;
[0078]
(5)利用最终获得的4255个snp位点构建进化树,确定采集的品种圃中梨样品的品种。
[0079]
具体的操作步骤如下:
[0080]
本实施例主要包含以下步骤,即酶切、连接、纯化、扩增、混库和分析。
[0081]
1.酶切:
[0082]
对随机采集来自山东大丰园农业有限公品种圃的秋月梨、黄金梨、皇冠、早酥、锦丰、玉露香、香梨、华金、豊水、jumbo新高和幸水各1株进行super
‑
gbs建库,具体过程如下(以下为每个样品使用量):
[0083][0084]
所有成分混匀后37℃酶切2h,然后75℃保温20min使酶灭活。
[0085]
2.连接:
[0086]
在40μl体系中连接adapter,barcode及酶切片段。
[0087][0088]
所有成分混匀后22℃酶切2h,然后65℃保温20min使酶灭活。
[0089]
3.纯化:
[0090]
取连接产物35μl加入0.7倍体积的sera
‑
mag beads(ge healthcare life sciences),室温静置5min,以去除300bp以下小片段。从上清中回收磁珠,并用200μl 70%酒精洗脱3遍。最后用40μl 10mm tris.hcl(ph 8.0)从磁珠中回收dna。
[0091]
4.扩增:
[0092][0093]
所有成分混匀后置于pcr仪中,反应条件为95℃预变性30s,扩增16个循环,每个循环95℃变性30s,62℃退火20s,68℃延伸15s,最后在68℃延伸5min,4℃保存。
[0094]
5.混库:
[0095]
使用qubit测定每个样品的文库浓度,浓度大于5ng/μl的样品用于混库测序。通过加入0.7倍体积的sera
‑
mag beads去除文库中的引物和小片段,然后根据测序量需求进行混样测序,测序平台为illumina nova pe150。建库过程所用的接头和引物序列详见下实施
例1中的表8。
[0096]
6.分析:
[0097]
对11个梨样品进行super
‑
gbs测序,一共得到27.9m高质量reads。将高质量reads比对到参考基因组上,比对率为78.25
‑
82.43,所有样品的平均测序深度为38.2。利用gatk(v3.8
‑
1)软件得到大量snp位点,与表1中的4479个snp位点进行比对,最终获得4479个snp位点中的4255个snp位点,采用treebest软件分析,利用r包ggtree绘图,这些位点可用于12个梨品种的准确鉴定,鉴定结果见图2。
[0098]
实施例3
[0099]
本实施例提供按照本发明的4479个snp位点筛选少数几个snp位点,并根据位点所在基因组的位置设计相对应的pcr检测引物,对山东大丰园农业有限公司品种圃随机采集的多份梨品种基因组dna进行pcr扩增,并通过一代测序的方法进行12个梨品种鉴定,同时加入已知品种的12个梨品种作为阳性对照进行试验和验证。包括如下步骤:
[0100]
(1)本公司购买的标准机构提供的品种准确的秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水树苗各2株,提取基因组dna;
[0101]
(2)从山东大丰园农业有限公司品种圃随机采集秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水树苗各2株,提取基因组dna;
[0102]
(3)从4479个snp位点中挑取位点组成能够准确鉴定12个梨品种的的一组snp位点群,见表2;
[0103]
(4)根据挑取的snp位点所在的基因组位置设计pcr扩增的上下游引物,见表3;
[0104]
(5)利用表3中12个梨品种的通用引物进行pcr扩增;
[0105]
(6)对扩增的序列进行一代测序;
[0106]
(7)参照所用的snp位点信息,根据测序结果中对应的位点的序列进行梨品种的分型判读。
[0107]
具体的操作步骤如下:
[0108]
本实施例主要包含以下步骤,即pcr、测序、比对和分析。
[0109]
1、pcr扩增
[0110]
根据表2中的位点所在位置设计pcr扩增引物,引物序列见表3。扩增条件为94℃3min,94℃30sec,55℃45sec,72℃45sec,37个循环,72℃7min,12℃30min。扩增体系如下。
[0111][0112]
2.测序
[0113]
将获得的pcr扩增产物利用1%琼脂糖凝胶电泳进行检测,在预计位置获得特异扩增条带的样品寄送上海生工生物有限公司进行测序。
[0114]
3.序列比对
[0115]
测序获得的结果利用dnaman软件或者snapgene进行序列比对,利用本发明4479个snp位点中筛选的一个snp位点标记群(见表2)进行12个梨品种的分型。
[0116]
4.分析
[0117]
序列比对鉴定结果发现,12个梨品种秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水共计48株鉴定结果与实际品种情况相符。
[0118]
实施例4
[0119]
本实施例提供按照本发明的4479个snp位点筛选少数几个snp位点,并根据位点所在基因组的位置设计相对应的pcr检测引物,对山东大丰园农业有限公司品种圃随机采集的多份梨品种基因组dna进行pcr扩增,并通过一代测序的方法进行12个梨品种鉴定,同时加入已知品种的12个梨品种作为阳性对照进行试验和验证。包括如下步骤:
[0120]
(1)本公司购买的标准机构提供的品种准确的秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水树苗各2株,提取基因组dna;
[0121]
(2)从山东大丰园农业有限公司品种圃随机采集秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水树苗各2株,提取基因组dna;
[0122]
(3)从4479个snp位点中挑取位点组成能够准确鉴定12个梨品种的的一组snp位点群,见表4;
[0123]
(4)根据挑取的snp位点所在的基因组位置设计pcr扩增的上下游引物,见表5;
[0124]
(5)利用表5中12个梨品种的通用引物进行pcr扩增;
[0125]
(6)对扩增的序列进行一代测序;
[0126]
(7)参照所用的snp位点信息,根据测序结果中对应的位点的序列进行梨品种的分型判读。
[0127]
具体的操作步骤如下:
[0128]
本实施例主要包含以下步骤,即pcr、测序、比对和分析。
[0129]
1、pcr扩增
[0130]
根据表4中的位点所在位置设计pcr扩增引物,引物序列见表5。扩增条件为94℃3min,94℃30sec,55℃45sec,72℃45sec,37个循环,72℃7min,12℃30min。扩增体系如下。
[0131][0132]
2.测序
[0133]
将获得的pcr扩增产物利用1%琼脂糖凝胶电泳进行检测,在预计位置获得特异扩增条带的样品寄送上海生工生物有限公司进行测序。
[0134]
3.序列比对
[0135]
测序获得的结果利用dnaman软件或者snapgene进行序列比对,利用本发明4479个snp位点中筛选的一个snp位点标记群(见表4)进行12个梨品种的分型。
[0136]
4.分析
[0137]
序列比对鉴定结果发现,12个梨品种秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水共计48株鉴定结果与实际品种情况相符。
[0138]
实施例5
[0139]
本实施例提供按照本发明的4479个snp位点筛选少数几个snp位点,并根据位点所在基因组的位置设计相对应的pcr检测引物,对山东大丰园农业有限公司品种圃随机采集的多份梨品种基因组dna进行pcr扩增,并通过一代测序的方法进行12个梨品种鉴定,同时加入已知品种的12个梨品种作为阳性对照进行试验和验证。包括如下步骤:
[0140]
(1)本公司购买的标准机构提供的品种准确的秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水树苗各2株,提取基因组dna;
[0141]
(2)从山东大丰园农业有限公司品种圃随机采集秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水树苗各2株,提取基因组dna;
[0142]
(3)从4479个snp位点中挑取位点组成能够准确鉴定12个梨品种的的一组snp位点群,见表6;
[0143]
(4)根据挑取的snp位点所在的基因组位置设计pcr扩增的上下游引物,见表7;
[0144]
(5)利用表7中12个梨品种的通用引物进行pcr扩增;
[0145]
(6)对扩增的序列进行一代测序;
[0146]
(7)参照所用的snp位点信息,根据测序结果中对应的位点的序列进行梨品种的分型判读。
[0147]
具体的操作步骤如下:
[0148]
本实施例主要包含以下步骤,即pcr、测序、比对和分析。
[0149]
1、pcr扩增
[0150]
根据表6中的位点所在位置设计pcr扩增引物,引物序列见表7。扩增条件为94℃3min,94℃30sec,55℃45sec,72℃45sec,37个循环,72℃7min,12℃30min。扩增体系如下。
[0151][0152][0153]
2.测序
[0154]
将获得的pcr扩增产物利用1%琼脂糖凝胶电泳进行检测,在预计位置获得特异扩增条带的样品寄送上海生工生物有限公司进行测序。
[0155]
3.序列比对
[0156]
测序获得的结果利用dnaman软件或者snapgene进行序列比对,利用本发明4479个snp位点中筛选的一个snp位点标记群(见表6)进行12个梨品种的分型。
[0157]
4.分析
[0158]
序列比对鉴定结果发现,12个梨品种秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、
玉露香、香梨、华金、豊水、jumbo新高和幸水共计48株鉴定结果与实际品种情况相符。从4479个snp位点中挑取其他snp位点标记群,同样可以准确鉴定秋月梨、黄金梨、皇冠、早酥、锦丰、新梨7号、玉露香、香梨、华金、豊水、jumbo新高和幸水这12个梨品种。
[0159]
表1 4479个snp位点如下:
[0160]
[0161]
[0162]
[0163]
[0164]
[0165]
[0166]
[0167]
[0168]
[0169]
[0170]
[0171]
[0172]
[0173]
[0174]
[0175]
[0176]
[0177]
[0178]
[0179]
[0180]
[0181]
[0182]
[0183]
[0184]
[0185]
[0186]
[0187]
[0188]
[0189]
[0190]
[0191]
[0192]
[0193]
[0194]
[0195]
[0196]
[0197]
[0198]
[0199]
[0200]
[0201]
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1