基因组编辑系统和方法
基因组编辑系统和方法
1.本技术是申请日为2017年12月27日的第201780035094.8号发明名称为“基因组编辑系统和方法”的中国专利申请的分案申请。
技术领域
2.本发明涉及基因工程领域。具体而言,本发明涉及新的基因组编辑系统和方法。更具体而言,本发明涉及新的能够对细胞基因组进行高效编辑的crispr
‑
c2c1系统及其用途。
背景技术:
3.crispr(clustered regularly interspaced short palindromic repeats,成簇的规律间隔的短回文重复序列)系统是细菌在进化过程中产生的用于防御外来基因入侵的免疫系统。其中,ii型crispr
‑
cas9系统是通过两个小rna(crrna和tracrrna)或者一个人工合成小rna(sgrna)介导一个cas9蛋白进行dna切割的系统,也是最早发现的三种(i、ii、iii型)crispr系统中最简单的系统。由于该系统简单易操作,在2013年被改造并成功实现了真核生物基因组的编辑。crispr/cas9系统迅速成为了生命科学领域最热门的技术。
4.2015年,zhang et al.通过序列比对和系统分析的方法又发现了区别于crispr
‑
cas9系统之外的新的v
‑
a型基因组编辑系统,即crispr
‑
cpf1系统。该系统只需要一个小rna(crrna)的介导即可实现基因组的编辑。
5.2015年,shmakov等人还鉴定出新的基因组编辑系统(molecular cell 60,385
‑
397,november 5,2015):c2c1(v
‑
b)、c2c2(vi)和c2c3(v
‑
c)系统。其中来自alicyclobacillus acidoterrestris的aacc2c1被证实可以实现dna切割,然而其活性受到例如温度的限制。aacc2c1系统在低于40℃无法切割dna。并且,没有证明aacc2c1系统能够在真核生物中实现基因组编辑
6.为了更便利地进行基因编辑,本领域仍然需要更多能够实现高效基因组编辑的系统。
7.发明简述
8.本发明人鉴定出一种新的crispr
‑
c2c1系统,其可进行哺乳动物细胞的基因组编辑。本发明所鉴定的c2c1核酸酶在体外实验中具有耐高温、耐酸碱的特性。并且,本发明对所鉴定的crispr
‑
c2c1系统的sgrna进行了优化,使其长度大大缩短,而不影响其打靶效率。最后,本发明人还对所鉴定的c2c1蛋白本身进行改造,使其从核酸内切酶变成死亡的c2c1(dc2c1),扩展了其用途。
9.在一方面,本发明提供了一种用于对细胞基因组中的靶序列进行定点修饰的基因组编辑系统,其包含以下i)至v)中至少一项:
10.i)c2c1蛋白或其变体,和向导rna;
11.ii)包含编码c2c1蛋白或其变体的核苷酸序列的表达构建体,和向导rna;
12.iii)c2c1蛋白或其变体,和包含编码向导rna的核苷酸序列的表达构建体;
13.iv)包含编码c2c1蛋白或其变体的核苷酸序列的表达构建体,和包含编码向导rna
的核苷酸序列的表达构建体;
14.v)包含编码c2c1蛋白或其变体的核苷酸序列和编码向导rna的核苷酸序列的表达构建体;
15.其中所述向导rna能够与所述c2c1蛋白或其变体形成复合物,将所述c2c1蛋白或其变体靶向所述细胞基因组中的靶序列,导致所述靶序列中的一或多个核苷酸的取代、缺失和/或添加。在一些实施方案中,所述c2c1蛋白是来自alicyclobacillus acidiphilus或alicyclobacillus kakegawensis的c2c1蛋白。
16.在另一方面,本发明提供了一种对细胞基因组中的靶序列进行定点修饰的方法,包括将本发明的基因组编辑系统导入所述细胞。
17.在另一方面,本发明提供了一种治疗有需要的对象中的疾病的方法,包括向所述对象递送有效量的本发明的基因组编辑系统以修饰所述对象中与所述疾病相关的基因。
18.在另一方面,本发明提供了本发明的基因组编辑系统在制备用于治疗有需要的对象中的疾病的药物组合物中的用途,其中所述基因组编辑系统用于修饰所述对象中与所述疾病相关的基因。
19.在另一方面,本发明提供了一种用于治疗有需要的对象中的疾病的药物组合物,其包含本发明的基因组编辑系统和药学可接受的载体,其中所述基因组编辑系统用于修饰所述对象中与所述疾病相关的基因。
20.附图描述
21.图1和图2示出了aac2c1核酸酶活性的体外分析结果。
22.图3和图4示出了aac2c1和akc2c1在哺乳动物细胞中的基因组编辑活性。
23.图5和图6示出了对指导aac2c1基因组编辑的单向导rna(sgrna)进行优化。
24.图7示出了靶序列长度和错配度对aac2c1编辑活性的影响。
25.图8示出了对aac2c1的脱靶效应分析。
26.图9示出了aac2c1的关键催化残基的鉴定和突变分析。
27.图10示出不同物种来源c2c1蛋白的序列比对和结构分析。
28.发明详述
29.在本发明中,除非另有说明,否则本文中使用的科学和技术名词具有本领域技术人员所通常理解的含义。并且,本文中所用的蛋白质和核酸化学、分子生物学、细胞和组织培养、微生物学、免疫学相关术语和实验室操作步骤均为相应领域内广泛使用的术语和常规步骤。例如,本发明中使用的标准重组dna和分子克隆技术为本领域技术人员熟知,并且在如下文献中有更全面的描述:sambrook,j.,fritsch,e.f.和maniatis,t.,molecular cloning:a laboratory manual;cold spring harbor laboratory press:cold spring harbor,1989(下文称为“sambrook”)。
30.在一方面,本发明提供了一种用于对细胞基因组中的靶序列进行定点修饰的基因组编辑系统,其包含以下i)至v)中至少一项:
31.i)c2c1蛋白或其变体,和向导rna;
32.ii)包含编码c2c1蛋白或其变体的核苷酸序列的表达构建体,和向导rna;
33.iii)c2c1蛋白或其变体,和包含编码向导rna的核苷酸序列的表达构建体;
34.iv)包含编码c2c1蛋白或其变体的核苷酸序列的表达构建体,和包含编码向导rna
的核苷酸序列的表达构建体;
35.v)包含编码c2c1蛋白或其变体的核苷酸序列和编码向导rna的核苷酸序列的表达构建体;
36.其中所述向导rna能够与所述c2c1蛋白或其变体形成复合物,将所述c2c1蛋白或其变体靶向所述细胞基因组中的靶序列,导致所述靶序列中的一或多个核苷酸的取代、缺失和/或添加。
[0037]“基因组”如本文所用不仅涵盖存在于细胞核中的染色体dna,而且还包括存在于细胞的亚细胞组分(如线粒体、质体)中的细胞器dna。
[0038]“c2c1核酸酶”、“c2c1蛋白”和“c2c1”在本文中可互换使用,指的是包括c2c1蛋白或其片段的rna指导的核酸酶。c2c1具有向导rna介导的dna结合活性以及dna切割活性,能在向导rna的指导下靶向并切割dna靶序列形成dna双链断裂(dsb)。dsb能够激活细胞内固有的修复机制非同源末端连接(non
‑
homologous end joining,nhej)和同源重组(homologous recombination,hr)对细胞中的dna损伤进行修复,在修复过程中,对该特定的dna序列进行定点编辑。
[0039]
在一些实施方案中,所述c2c1蛋白是来自alicyclobacillus acidiphilus的c2c1蛋白(aac2c1)。例如,所述c2c1蛋白是来自alicyclobacillus acidiphilus nbrc 100859的aac2c1蛋白。在一些实施方案中,所述aac2c1蛋白包含seq id no:1所示的氨基酸序列。
[0040]
本发明人令人惊奇地发现,aac2c1蛋白在约4℃
‑
约100℃的宽温度范围内均具有rna指导的dna切割活性,而在约30℃
‑
约60℃具有最佳活性。此外,aac2c1蛋白在约ph 1.0
‑
约ph 12.0的宽ph范围内均具有rna指导的dna切割活性,在约ph 1.0
‑
约ph 8.0具有最佳活性。因此,本发明的基因组编辑系统可以在多种温度和酸碱性条件下工作。
[0041]
在一些实施方案中,所述c2c1蛋白的变体包含与seq id no:1所示的野生型aac2c1蛋白具有至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%序列相同性的氨基酸序列,并且具有野生型aac2c1蛋白的rna介导的dna结合活性和/或dna切割活性。
[0042]
在一些实施方案中,所述c2c1蛋白的变体相对于seq id no:1具有一或多个氨基酸残基取代、缺失或添加的氨基酸序列,并且具有野生型aac2c1蛋白的rna介导的dna结合活性和/或dna切割活性。例如,所述c2c1蛋白的变体包含相对于seq id no:1具有1个、2个、3个、4个、5个、6个、7个、8个、9个或10个氨基酸残基取代、缺失或添加的氨基酸序列。在一些实施方案中,所述氨基酸取代是保守型取代。
[0043]
在另一些实施方案中,所述c2c1蛋白是来自alicyclobacillus kakegawensis的c2c1蛋白(akc2c1)。例如,所述akc2c1蛋白来自alicyclobacillus kakegawensis nbrc 103104。在一些实施方案中,所述akc2c1蛋白包含seq id no:5所示的氨基酸序列。
[0044]
在一些实施方案中,所述c2c1蛋白的变体包含与seq id no:5所示的野生型akc2c1蛋白具有至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%序列相同性的氨基酸序列,并且具有野生型akc2c1蛋白的rna介导的dna结合活性和/或dna切割活性。
[0045]
在一些实施方案中,所述c2c1蛋白的变体相对于seq id no:5具有一或多个氨基酸残基取代、缺失或添加的氨基酸序列,并且具有野生型akc2c1蛋白的rna介导的dna结合
活性和/或dna切割活性。例如,所述c2c1蛋白的变体包含相对于seq id no:1具有1个、2个、3个、4个、5个、6个、7个、8个、9个或10个氨基酸残基取代、缺失或添加的氨基酸序列。在一些实施方案中,所述氨基酸取代是保守型取代。
[0046]“多肽”、“肽”、和“蛋白质”在本发明中可互换使用,指氨基酸残基的聚合物。该术语适用于其中一个或多个氨基酸残基是相应的天然存在的氨基酸的人工化学类似物的氨基酸聚合物,以及适用于天然存在的氨基酸聚合物。术语“多肽”、“肽”、“氨基酸序列”和“蛋白质”还可包括修饰形式,包括但不限于糖基化、脂质连接、硫酸盐化、谷氨酸残基的γ羧化、羟化和adp
‑
核糖基化。
[0047]
序列“相同性”具有本领域公认的含义,并且可以利用公开的技术计算两个核酸或多肽分子或区域之间序列相同性的百分比。可以沿着多核苷酸或多肽的全长或者沿着该分子的区域测量序列相同性。(参见,例如:computational molecular biology,lesk,a.m.,ed.,oxford university press,new york,1988;biocomputing:informatics and genome projects,smith,d.w.,ed.,academic press,new york,1993;computer analysis of sequence data,part i,griffin,a.m.,and griffin,h.g.,eds.,humana press,new jersey,1994;sequence analysis in molecular biology,von heinje,g.,academic press,1987;and sequence analysis primer,gribskov,m.and devereux,j.,eds.,m stockton press,new york,1991)。虽然存在许多测量两个多核苷酸或多肽之间的相同性的方法,但是术语“相同性”是技术人员公知的(carrillo,h.&lipman,d.,siam j applied math 48:1073(1988))。
[0048]
在肽或蛋白中,合适的保守型氨基酸取代是本领域技术人员已知的,并且一般可以进行而不改变所得分子的生物活性。通常,本领域技术人员认识到多肽的非必需区中的单个氨基酸取代基本上不改变生物活性(参见,例如,watson et al.,molecular biology of the gene,4th edition,1987,the benjamin/cummings pub.co.,p.224)。
[0049]
在一些实施方案中,所述c2c1蛋白的变体包含核酸酶死亡的c2c1蛋白(dc2c1)。核酸酶死亡的c2c1蛋白指的是保留向导rna介导的dna结合活性但是不具备dna切割活性的c2c1蛋白。
[0050]
在一些实施方案中,所述dc2c1中对应于野生型aac2c1蛋白第785位的氨基酸被取代。在一些具体实施方案中,所述dc2c1相对于野生型aac2c1蛋白包含氨基酸取代r785a。在一些具体实施方案中,所述dc2c1包含seq id no:4所示氨基酸序列。
[0051]
在一些实施方案中,所述c2c1蛋白的变体是dc2c1与脱氨酶的融合蛋白。例如,所述融合蛋白中的dc2c1与脱氨酶可以通过接头例如肽接头连接。
[0052]
如本发明所用,“脱氨酶”是指催化脱氨基反应的酶。在本发明一些实施方式中,所述脱氨酶指的是胞嘧啶脱氨酶,其能够接受单链dna作为底物并能够催化胞苷或脱氧胞苷分别脱氨化为尿嘧啶或脱氧尿嘧啶。在本发明一些实施方式中,所述脱氨酶指的是腺嘌呤脱氨酶,其能够接受单链dna作为底物并能够催化腺苷或脱氧腺苷(a)形成肌苷(i)。通过使用c2c1切口酶变体与脱氨酶的融合蛋白,可以实现靶dna序列中的碱基编辑,例如c至t的转换或a至g的转换。本领域已知多种合适的接受单链dna作为底物的胞嘧啶脱氨酶或腺嘌呤脱氨酶。
[0053]
在本发明的一些实施方案中,本发明的基因组编辑系统中的c2c1蛋白或其变体还
可以包含核定位序列(nls)。一般而言,所述c2c1蛋白或其变体中的一个或多个nls应具有足够的强度,以便在细胞核中驱动所述c2c1蛋白或其变体以可实现其基因编辑功能的量积聚。一般而言,核定位活性的强度由所述c2c1蛋白或其变体中nls的数目、位置、所使用的一个或多个特定的nls、或这些因素的组合决定。
[0054]
在本发明的一些实施方案中,本发明的基因组编辑系统中的c2c1蛋白或其变体的nls可以位于n端和/或c端。在一些实施方案中,所述c2c1蛋白或其变体包含约1、2、3、4、5、6、7、8、9、10个或更多个nls。在一些实施方案中,所述c2c1蛋白或其变体包含在或接近于n端的约1、2、3、4、5、6、7、8、9、10个或更多个nls。在一些实施方案中,所述c2c1蛋白或其变体包含在或接近于c端约1、2、3、4、5、6、7、8、9、10个或更多个nls。在一些实施方案中,所述c2c1蛋白或其变体包含这些的组合,如包含在n端的一个或多个nls以及在c端的一个或多个nls。当存在多于一个nls时,每一个可以被选择为不依赖于其他nls。在本发明的一些实施方式中,所述c2c1蛋白或其变体包含2个nls,例如所述2个nls分别位于n端和c端。
[0055]
一般而言,nls由暴露于蛋白表面上的带正电的赖氨酸或精氨酸的一个或多个短序列组成,但其他类型的nls也是已知的。nls的非限制性实例包括:kkrkv、pkkkrkv,或sggspkkkrkv。
[0056]
此外,根据所需要编辑的dna位置,本发明的c2c1蛋白或其变体还可以包括其他的定位序列,例如细胞质定位序列、叶绿体定位序列、线粒体定位序列等。
[0057]
在本发明的一些实施方案中,所述靶序列长度为18
‑
35个核苷酸,优选20个核苷酸。在本发明的一些实施方案中,所述靶序列在其5’端侧翼为选自:5’tttn
‑3’
、5’attn
‑3’
、5’gttn
‑3’
、5’cttn
‑3’
、5’ttc
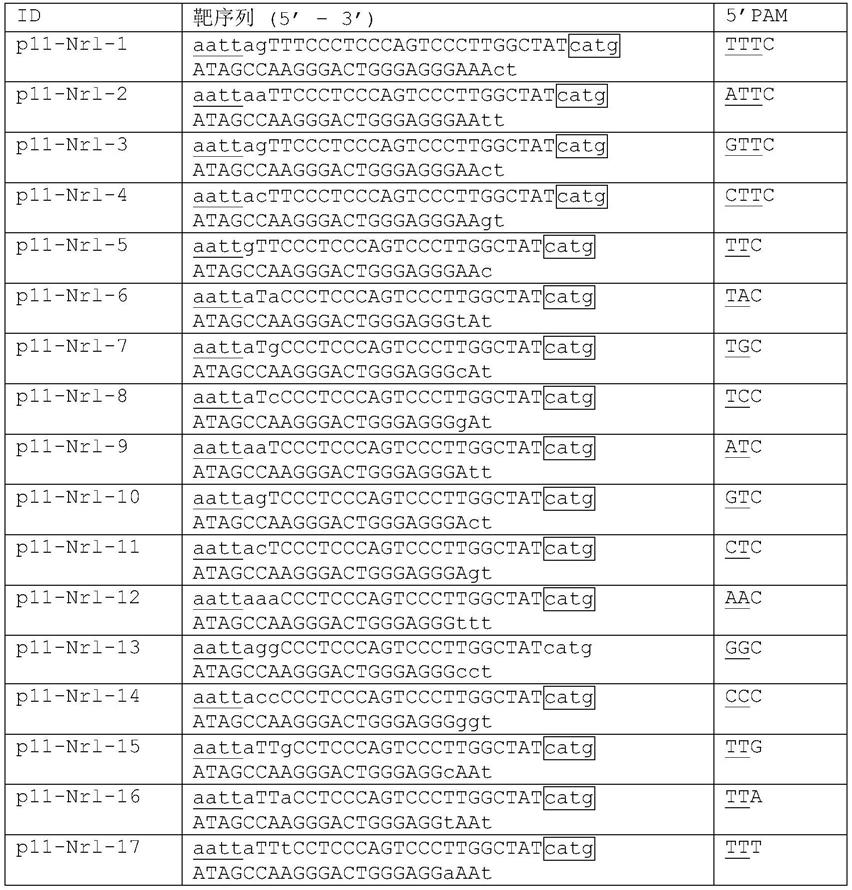
‑3’
、5’ttg
‑3’
、5’tta
‑3’
、5’ttt
‑3’
、5’tan
‑3’
、5’tgn
‑3’
、5’tcn
‑3’
和5’atc
‑3’
的pam(前间区邻近基序)序列,优选5’tttn
‑3’
,其中n选自a、g、c和t。
[0058]
在本发明中,待进行修饰的靶序列可以位于基因组的任何位置,例如位于功能基因如蛋白编码基因内,或者例如可以位于基因表达调控区如启动子区或增强子区,从而实现对所述基因功能修饰或对基因表达的修饰。可以通过t7ei、pcr/re或测序方法检测基因组靶序列中的取代、缺失和/或添加
[0059]“向导rna”和“grna”在本文中可互换使用,通常由部分互补形成复合物的crrna和tracrrna分子构成,其中crrna包含与靶序列具有足够相同性以便与靶序列的互补序列杂交并且指导crispr复合物(c2c1+crrna+tracrrna)与该靶序列以序列特异性方式结合的序列。然而,可以设计并使用单向导rna(sgrna),其同时包含crrna和tracrrna的特征。
[0060]
在本发明的一些实施方案中,所述向导rna是由crrna和tracrrna部分互补形成的复合物。在一些实施方案中,所述tracrrna由以下的核酸序列编码:5
’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggc aaagcccgttgaacttctcaaaaagaacgctcgctcagtgttctgac
‑3’
。在一些实施方案中,所述crrna由以下的核酸序列编码:5
’‑
gtcggatcactgagcgagcgatctgagaagtggcac
‑
n
x
‑3’
,其中n
x
表示x个连续的核苷酸组成的核苷酸序列,n各自独立地选自a、g、c和t;x为18≤x≤35的整数。优选地,x=20。在一些实施方案中,序列n
x
(spacer序列)能够与靶序列的互补序列特异性杂交。
[0061]
在本发明的一些实施方案中,所述向导rna是sgrna。在一些具体实施方案中,所述sgrna由选自以下之一的核酸序列编码:
[0062]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttg
aacttctcaaaaagaacgctcgctcagtgttctgacgtcggatcactgagcgagcgatctgagaagtggcac
‑
n
x
‑3’
;
[0063]5’‑
aactgtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaaaaagaacgctcgctcagtgttctgacgtcggatcactgagcgagcgatctgagaagtggcac
‑
n
x
‑3’
;
[0064]5’‑
ctgtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaaaaagaacgctcgctcagtgttctgacgtcggatcactgagcgagcgatctgagaagtggcac
‑
n
x
‑3’
;
[0065]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaaaaagaacgctcgctcagtgttatcactgagcgagcgatctgagaagtggcac
‑
n
x
‑3’
;
[0066]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaaaaagaacgatctgagaagtggcac
‑
n
x
‑3’
;
[0067]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaaaaagctgagaagtggcac
‑
n
x
‑3’
;
[0068]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaaagctgagaagtggcac
‑
n
x
‑3’
;
[0069]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaaaactgagaagtggcac
‑
n
x
‑3’
;
[0070]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaagcgagaagtggcac
‑
n
x
‑3’
;
[0071]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctaagcagaagtggcac
‑
n
x
‑3’
;和
[0072]5’‑
gtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttcaagcgaagtggcac
‑
n
x
‑3’
;
[0073]
其中n
x
表示x个连续的核苷酸组成的核苷酸序列(spacer序列),n各自独立地选自a、g、c和t;x为18≤x≤35的整数。优选地,x=20。在一些实施方案中,序列n
x
(spacer序列)能够与靶序列的互补序列特异性杂交。所述sgrna中除n
x
之外的序列为sgrna的scaffold序列。
[0074]“多核苷酸”、“核酸序列”、“核苷酸序列”或“核酸片段”在本文可互换使用并且是单链或双链rna或dna聚合物,任选地可含有合成的、非天然的或改变的核苷酸碱基。核苷酸通过如下它们的单个字母名称来指代:“a”为腺苷或脱氧腺苷(分别对应rna或dna),“c”表示胞苷或脱氧胞苷,“g”表示鸟苷或脱氧鸟苷,“u”表示尿苷,“t”表示脱氧胸苷,“r”表示嘌呤(a或g),“y”表示嘧啶(c或t),“k”表示g或t,“h”表示a或c或t,“i”表示肌苷,并且“n”表示任何核苷酸。
[0075]
为了在靶细胞中获得有效表达,在本发明的一些实施方式中,所述编码c2c1蛋白或其变体的核苷酸序列针对待进行基因组编辑的细胞所来自的生物体进行密码子优化。
[0076]
密码子优化是指通过用在宿主细胞的基因中更频繁地或者最频繁地使用的密码子代替天然序列的至少一个密码子(例如约或多于约1、2、3、4、5、10、15、20、25、50个或更多个密码子同时维持该天然氨基酸序列而修饰核酸序列以便增强在感兴趣宿主细胞中的表
达的方法。不同的物种对于特定氨基酸的某些密码子展示出特定的偏好。密码子偏好性(在生物之间的密码子使用的差异)经常与信使rna(mrna)的翻译效率相关,而该翻译效率则被认为依赖于被翻译的密码子的性质和特定的转运rna(trna)分子的可用性。细胞内选定的trna的优势一般反映了最频繁用于肽合成的密码子。因此,可以将基因定制为基于密码子优化在给定生物中的最佳基因表达。密码子利用率表可以容易地获得,例如在www.kazusa.orjp/codon/上可获得的密码子使用数据库(“codon usage database”)中,并且这些表可以通过不同的方式调整适用。参见,nakamura y.等,“codon usage tabulated from the international dna sequence databases:status for the year2000.nucl.acids res.,28:292(2000)。
[0077]
在本发明的一些具体实施方式中,所述编码c2c1蛋白或其变体的核苷酸序列针对人进行密码子优化。在一些具体实施方式中,所述密码子优化的编码c2c1蛋白的核苷酸序列选自seq id no:3或7。
[0078]
根据本发明的一些实施方式,本发明所述系统的表达构建体中所述编码c2c1蛋白或其变体的核苷酸序列和/或所述编码向导rna的核苷酸序列与表达调控元件如启动子可操作地连接。
[0079]
如本发明所用,“表达构建体”是指适于感兴趣的核苷酸序列在生物体中表达的载体如重组载体。“表达”指功能产物的产生。例如,核苷酸序列的表达可指核苷酸序列的转录(如转录生成mrna或功能rna)和/或rna翻译成前体或成熟蛋白质。本发明的“表达构建体”可以是线性的核酸片段、环状质粒、病毒载体,或者,在一些实施方式中,可以是能够翻译的rna(如mrna)。
[0080]
本发明的“表达构建体”可包含不同来源的调控序列和感兴趣的核苷酸序列,或相同来源但以不同于通常天然存在的方式排列的调控序列和感兴趣的核苷酸序列。
[0081]“调控序列”和“调控元件”可互换使用,指位于编码序列的上游(5'非编码序列)、中间或下游(3'非编码序列),并且影响相关编码序列的转录、rna加工或稳定性或者翻译的核苷酸序列。调控序列可包括但不限于启动子、翻译前导序列、内含子和多腺苷酸化识别序列。
[0082]“启动子”指能够控制另一核酸片段转录的核酸片段。在本发明的一些实施方案中,启动子是能够控制细胞中基因转录的启动子,无论其是否来源于所述细胞。启动子可以是组成型启动子或组织特异性启动子或发育调控启动子或诱导型启动子。
[0083]“组成型启动子”指一般将引起基因在多数细胞类型中在多数情况下表达的启动子。“组织特异性启动子”和“组织优选启动子”可互换使用,并且指主要但非必须专一地在一种组织或器官中表达,而且也可在一种特定细胞或细胞型中表达的启动子。“发育调控启动子”指其活性由发育事件决定的启动子。“诱导型启动子”响应内源性或外源性刺激(环境、激素、化学信号等)而选择性表达可操纵连接的dna序列。
[0084]
如本文中所用,术语“可操作地连接”指调控元件(例如但不限于,启动子序列、转录终止序列等)与核酸序列(例如,编码序列或开放读码框)连接,使得核苷酸序列的转录被所述转录调控元件控制和调节。用于将调控元件区域可操作地连接于核酸分子的技术为本领域已知的。
[0085]
本发明可使用的启动子的实例包括但不限于聚合酶(pol)i、pol ii或pol iii启
动子。pol i启动子的实例包括鸡rna pol i启动子。pol ii启动子的实例包括但不限于巨细胞病毒立即早期(cmv)启动子、劳斯肉瘤病毒长末端重复(rsv
‑
ltr)启动子和猿猴病毒40(sv40)立即早期启动子。pol iii启动子的实例包括u6和h1启动子。可以使用诱导型启动子如金属硫蛋白启动子。启动子的其他实例包括t7噬菌体启动子、t3噬菌体启动子、β
‑
半乳糖苷酶启动子和sp6噬菌体启动子。当用于植物时,启动子可以是花椰菜花叶病毒35s启动子、玉米ubi
‑
1启动子、小麦u6启动子、水稻u3启动子、玉米u3启动子、水稻肌动蛋白启动子。
[0086]
可通过本发明的系统进行基因组编辑的细胞优选是真核生物细胞,包括但不限于,哺乳动物细胞如人、小鼠、大鼠、猴、犬、猪、羊、牛、猫;家禽如鸡、鸭、鹅的细胞;植物细胞包括单子叶植物细胞和双子叶植物细胞,例如水稻、玉米、小麦、高粱、大麦、大豆、花生、拟南芥等的细胞。在本发明的一些实施方案中,所述细胞是真核生物细胞,优选哺乳动物细胞,更优选是人细胞。
[0087]
在另一方面,本发明提供了一种修饰细胞基因组中靶序列的方法,包括将本发明的基因组编辑系统导入所述细胞,由此所述向导rna将所述c2c1蛋白或其变体靶向所述细胞基因组中的靶序列,导致所述靶序列中的一或多个核苷酸的取代、缺失和/或添加。
[0088]
将本发明的基因组编辑系统的核酸分子(例如质粒、线性核酸片段、rna等)或蛋白质“导入”细胞是指用所述核酸或蛋白质转化细胞,使得所述核酸或蛋白质在细胞中能够发挥功能。本发明所用的“转化”包括稳定转化和瞬时转化。“稳定转化”指将外源核苷酸序列导入基因组中,导致外源基因稳定遗传。一旦稳定转化,外源核酸序列稳定地整合进所述生物体和其任何连续世代的基因组中。“瞬时转化”指将核酸分子或蛋白质导入细胞中,执行功能而没有外源基因稳定遗传。瞬时转化中,外源核酸序列不整合进基因组中。
[0089]
可用于将本发明的基因组编辑系统导入细胞的方法包括但不限于:磷酸钙转染、原生质融合、电穿孔、脂质体转染、微注射、病毒感染(如杆状病毒、痘苗病毒、腺病毒、腺相关病毒、慢病毒和其他病毒)、基因枪法、peg介导的原生质体转化、土壤农杆菌介导的转化。
[0090]
在一些实施方式中,所述方法在体外进行。例如,所述细胞是分离的细胞。在一些实施方式中,所述细胞是car
‑
t细胞。在一些实施方式中,所述细胞是诱导的胚胎干细胞。
[0091]
在另一些实施方式中,所述方法还可以在体内进行。例如,所述细胞是生物体内的细胞,可以通过例如病毒介导的方法将本发明的系统体内导入所述细胞。例如,所述细胞可以是患者体内的肿瘤细胞。
[0092]
在另一方面,本发明还提一种产生经遗传修饰的细胞的方法,包括将本发明的基因组编辑系统导入细胞中,由此所述向导rna将所述c2c1蛋白或其变体靶向所述细胞基因组中的靶序列,导致所述靶序列中的一或多个核苷酸取代、缺失和/或添加。
[0093]
在另一方面,本发明还提供经遗传修饰的生物体,其包含通过本发明的方法产生的经遗传修饰的细胞或其后代。
[0094]
如本文所用,“生物体”包括适于基因组编辑的任何生物体,优选真核生物。生物体的实例包括但不限于,哺乳动物如人、小鼠、大鼠、猴、犬、猪、羊、牛、猫;家禽如鸡、鸭、鹅;植物包括单子叶植物和双子叶植物,例如水稻、玉米、小麦、高粱、大麦、大豆、花生、拟南芥等。在本发明的一些实施方案中,所述生物体是真核生物,优选哺乳动物,更优选人。
[0095]
如本文所用,“经遗传修饰的生物体”或“经遗传修饰的细胞”意指在其基因组内包含外源多核苷酸或修饰的基因或表达调控序列的生物体或细胞。例如外源多核苷酸能够稳
定地整合进生物体或细胞的基因组中,并遗传连续的世代。外源多核苷酸可单独地或作为重组dna构建体的部分整合进基因组中。修饰的基因或表达调控序列为在生物体或细胞基因组中所述序列包含单个或多个脱氧核苷酸取代、缺失和添加。针对序列而言的“外源”意指来自外来物种的序列,或者如果来自相同物种,则指通过蓄意的人为干预而从其天然形式发生了组成和/或基因座的显著改变的序列。
[0096]
在另一方面,本发明提供了一种基因表达调控系统,其基于本发明的核酸酶死亡的c2c1蛋白。此系统尽管并没有改变靶基因的序列,在本文范围内也定义为基因组编辑系统。
[0097]
在一些实施方案中,本发明的基因表达调控系统是基因抑制或沉默系统,其可以包含以下之一:
[0098]
i)核酸酶死亡的c2c1蛋白或其与转录阻遏蛋白的融合蛋白,和向导rna;
[0099]
ii)包含编码核酸酶死亡的c2c1蛋白或其与转录阻遏蛋白的融合蛋白的核苷酸序列的表达构建体,和向导rna;
[0100]
iii)核酸酶死亡的c2c1蛋白或其与转录阻遏蛋白的融合蛋白,和包含编码向导rna的核苷酸序列的表达构建体;
[0101]
iv)包含编码核酸酶死亡的c2c1蛋白或其与转录阻遏蛋白的融合蛋白的核苷酸序列的表达构建体,和包含编码向导rna的核苷酸序列的表达构建体;或
[0102]
v)包含编码核酸酶死亡的c2c1蛋白或其与转录阻遏蛋白的融合蛋白的核苷酸序列和编码向导rna的核苷酸序列的表达构建体。
[0103]
所述核酸酶死亡的c2c1蛋白或向导rna的定义如上所述。所述转录阻遏蛋白的选择属于本领域技术人员的技能范围。
[0104]
如本文所用,基因抑制或沉默是指基因表达水平的下调或消除,优选在转录水平。
[0105]
然而,本发明的基因表达调控系统还可以使用核酸酶死亡的c2c1蛋白和转录激活蛋白的融合蛋白。在此种情况下,所述基因表达调控系统是基因表达激活系统。例如,本发明的基因表达激活系统可以包含以下之一:
[0106]
i)核酸酶死亡的c2c1蛋白和转录激活蛋白的融合蛋白,和向导rna;
[0107]
ii)包含编码核酸酶死亡的c2c1蛋白和转录激活蛋白的融合蛋白的核苷酸序列的表达构建体,和向导rna;
[0108]
iii)核酸酶死亡的c2c1蛋白和转录激活蛋白的融合蛋白,和包含编码向导rna的核苷酸序列的表达构建体;
[0109]
iv)包含编码核酸酶死亡的c2c1蛋白和转录激活蛋白的融合蛋白的核苷酸序列的表达构建体,和包含编码向导rna的核苷酸序列的表达构建体;或
[0110]
v)包含编码核酸酶死亡的c2c1蛋白和转录激活蛋白的融合蛋白的核苷酸序列和编码向导rna的核苷酸序列的表达构建体。
[0111]
所述核酸酶死亡的c2c1蛋白或向导rna的定义如上所述。所述转录激活蛋白的选择属于本领域技术人员的技能范围。
[0112]
如本文所用,基因激活是指基因表达水平的上调,优选在转录水平。
[0113]
在另一方面,本发明还涵盖本发明的基因组编辑系统在疾病治疗中的应用。
[0114]
通过本发明的基因组编辑系统对疾病相关基因进行修饰,可以实现疾病相关基因
的上调、下调、失活、激活或者突变纠正等,从而实现疾病的预防和/或治疗。例如,本发明中靶序列可以位于疾病相关基因的蛋白编码区内,或者例如可以位于基因表达调控区如启动子区或增强子区,从而可以实现对所述疾病相关基因功能修饰或对疾病相关基因表达的修饰。
[0115]“疾病相关”基因是指与非疾病对照的组织或细胞相比,在来源于疾病影响的组织的细胞中以异常水平或以异常形式产生转录或翻译产物的任何基因。在改变的表达与疾病的出现和/或进展相关的情况下,它可以是以异常高的水平被表达的基因;它可以是以异常低的水平被表达的基因。疾病相关基因还指具有一个或多个突变或直接负责或与一个或多个负责疾病的病因学的基因连锁不平衡的遗传变异的基因。转录的或翻译的产物可以是已知的或未知的,并且可以处于正常或异常水平。
[0116]
因此,在另一方面,本发明还提供一种治疗有需要的对象中的疾病的方法,包括向所述对象递送有效量的本发明的基因组编辑系统以修饰与所述疾病相关的基因。
[0117]
在仍另一方面,本发明还提供本发明的基因组编辑系统在制备用于治疗有需要的对象中的疾病的药物组合物中的用途,其中所述基因组编辑系统用于修饰与所述疾病相关的基因。
[0118]
在仍另一方面,本发明还提供用于治疗有需要的对象中的疾病的药物组合物,其包含本发明的基因组编辑系统和药学可接受的载体,其中所述基因组编辑系统用于修饰与所述疾病相关的基因。
[0119]
在一些实施方式中,所述对象是哺乳动物,例如人。
[0120]
所述疾病的实例包括但不限于肿瘤、炎症、帕金森病、心血管疾病、阿尔茨海默病、自闭症、药物成瘾、年龄相关性黄斑变性、精神分裂症、遗传性疾病等。
[0121]
在仍另一方面,本发明还包括用于本发明的方法的试剂盒,该试剂盒包括本发明的基因组编辑系统,以及使用说明。试剂盒一般包括表明试剂盒内容物的预期用途和/或使用方法的标签。术语标签包括在试剂盒上或与试剂盒一起提供的或以其他方式随试剂盒提供的任何书面的或记录的材料。
实施例
[0122]
材料与方法
[0123]
dna操作
[0124]
根据molecular cloning:a laboratory manual(分子克隆:实验室手册)通过一定程度的修改进行包括dna制备、消化、连接、扩增、纯化、琼脂糖凝胶电泳等dna操作。
[0125]
简而言之,通过在p11
‑
lacy
‑
wtx1载体的ecori和sphi位点之间连接退火的寡核苷酸(表1)构建pam序列测定质粒,并且pcr产生相应的携带不同pam序列的dsdna片段。
[0126]
通过将退火的寡核苷酸连接到经basi消化的puc19
‑
u6
‑
sgrna载体中来构建用于细胞转染测定的靶向sgrna。
[0127]
使用含有t7启动子序列的引物对sgrna体外转录的模板进行pcr扩增。
[0128]
从头基因合成和质粒构建。
[0129]
通过psi
‑
blast程序鉴定的新型v
‑
b crispr
‑
c2c1蛋白编码序列被人源化(密码子优化),并全长合成。分别应用pcag
‑
2aegfp载体和bpk2014
‑
ccdb载体用于c2c1哺乳动物细
胞表达和大肠杆菌表达。在用于哺乳动物细胞表达的puc19
‑
u6载体中构建向导rna。
[0130]
蛋白质纯化
[0131]
将合成的c2c1编码序列构建到bpk2014
‑
ccdb表达载体中。所得到的融合构建体含有c末端融合的his
10
标签。蛋白在大肠杆菌bl21(λde3)(transgen biotech)中表达,在cm
r
+lb培养基中于37℃生长至od600约0.4,用0.5mm iptg在16℃诱导16小时。收集300ml诱导的细胞用于蛋白质纯化,并且所有随后的步骤在4℃下进行。在裂解之前,将细胞沉淀物在补充有1x蛋白酶抑制剂(roche complete,不含edta)的30ml裂解缓冲液(npi
‑
10:50mm nah2po4,300mm nacl,10mm咪唑,5%甘油,ph8.0)超声。在4℃下离心8,000转/分离心10分钟使裂解物澄清,上清液与his60 ni superflow resin(takara)在4℃下分批孵育2小时。用20ml洗涤缓冲液1(npi
‑
20:50mm nah2po4,300mm nacl,20mm咪唑,5%甘油,ph8.0),清洗缓冲液2(npi
‑
40:50mm nah
2 po4,300mm nacl,40mm咪唑,5%甘油,ph8.0)和洗涤缓冲液3(npi
‑
100:50mm nah 2po4,300mm nacl,100mm咪唑,5%甘油,ph8.0)用5ml洗脱缓冲液(npi
‑
300:50mm nah2po4,300mm nacl,300mm咪唑,5%甘油,ph8.0)洗涤。使用100kda透析器,使用储存缓冲液(tris
‑
hcl,ph8.0,200mm kcl,0.1mm edta ph8.0,1mm dtt,20%甘油)过夜透析纯化的c2c1蛋白。合并级分并用100kda离心过滤装置(millipore)浓缩。通过sds
‑
page和考马斯染色分析富集的蛋白质的纯度,并使用bca蛋白质测定试剂盒(thermo fisher)定量浓度。
[0132]
体外rna转录。
[0133]
使用hisribetm t7 quick high yield rna合成试剂盒(neb)和携带t7启动子序列的pcr扩增的dna模板体外转录rna。使用oligo clean&concentrator tm(zymo research)纯化转录的rna并在nanodrop tm 2000(thermo fisher)上定量。
[0134]
pam序列在体外确定。
[0135]
为了测定aac2c1的pam序列,将100nm aac2c1蛋白、400ng体外转录的sgrna和具有不同pam序列(表1)的200ng pcr产生的双链dna(dsdna)在切割缓冲液(50mm tris
‑
hcl,100mm nacl,10mm mgcl2,ph8.0)中37℃温育1小时。加入rnase a终止反应,37℃消化20min,75℃消化5min后用~3%琼脂糖凝胶电泳和溴化乙锭染色分离。
[0136]
双链dna切割测定。
[0137]
对于双链dna切割测定,如果没有特别指出,将100nm c2c1蛋白、400ng体外转录的sgrna和200ng pcr产生的含有5'tttn
‑
pam序列的双链dna(dsdna)在37℃下在切割缓冲液(50mm tris
‑
hcl,100mm nacl,10mm mgcl2,ph8.0)孵育1小时。
[0138]
为了确定aac2c1的热稳定性,在裂解缓冲液(50mm tris
‑
hcl,100mm nacl,10mm mgcl2,ph8.0)中在大温度范围(4℃~100℃)下反应1小时。
[0139]
对于ph耐受性测定,在ph值范围为1.0至13.0的切割缓冲液(50mm tris
‑
hcl,100mm nacl,10mm mgcl 2)中在37℃下进行切割反应1h。
[0140]
在mg
2+
依赖性测定中,向切割缓冲液(50mm tris
‑
hcl,100mm nacl,ph8.0)中添加edta(0mm,1mm,5mm,10mm,20mm和40mm)或mg
2+
1mm,5mm,10mm,20mm和40mm),37℃保温1h。
[0141]
进一步的金属依赖性切割反应在补充有1或5mm的cacl2,mncl2、srcl 2
,nicl 2
,fecl 2
,cocl 2
,zncl2或cucl2的裂解缓冲液(50mm tris
‑
hcl,100mm nacl,10mm mgcl2,1mm edta,ph8.0)中于37℃进行1小时。加入rnase a于37℃20min消化sgrna终止反应,随后75℃
5min失活rnase a,用~3%琼脂糖凝胶电泳和溴化乙锭染色分离。
[0142]
细胞培养、转染和荧光激活细胞分选(facs)。
[0143]
将人胚胎肾(hek)细胞系hk293t维持在补充有10%胎牛血清和1%antibiotic
‑
antimycotic(gibco)的dulbecco改良的eagle培养基(dmem)中,37℃,5%co2培养。在具有activin a(20ng/ml,r&d)和fgf2(12.5ng/ml,r&d)的n2b27培养基中,将小鼠外胚层干细胞(episc)系维持在纤连蛋白上。在转染前一天将hk293t或episc细胞接种到24孔板(corning)中。按照制造商推荐的方案,使用lipofectamine ltx(invitrogen)转染细胞。对于24孔板的每个孔,使用总共750ng质粒。然后在转染后48小时,使用moflo xdp(beckman coulter)分选gfp阳性细胞。
[0144]
t7核酸内切酶i(t7ei)测定和基因组修饰的测序分析。
[0145]
将用质粒dna转染48小时后收集的或facs分选的gfp
‑
阳性hk293t或episc细胞进行基因组dna提取。简而言之,用缓冲液l(bimake)直接裂解细胞,并在55℃下孵育3小时,在95℃下孵育10分钟。对每个基因,pcr扩增crispr
‑
c2c1靶位点周围的基因组区域。将200
‑
400ng pcr产物与ddh2o混合至终体积10μl,进行再退火处理以使异源双链体形成。再退火后,将产物用1/10体积的nebuffer
tm 2.1和0.2μl的t7ei(neb)在37℃下处理30分钟,并在3%琼脂糖凝胶上分析。indel基于相对条带强度进行定量。
[0146]
t7ei分析鉴定出的突变产物进克隆ta载体peasy
‑
t1(transgen biotech)并转化感受态大肠杆菌菌株trans1
‑
t1(transgen biotech)。过夜培养后,随机挑出菌落并测序。
[0147]
脱靶预测和检测。
[0148]
由于v
‑
b型crispr
‑
c2c1系统尚未被用于编辑哺乳动物基因组,因此没有预测脱靶的指导原则。在图7中的初步数据提供了一些参考,即种子区域可能是间区序列5'末端的前17个核苷酸(nt),因为当间区(spacer)长度截短到18nt时检测到最小的脱靶切割活性。由于间区序列的5'末端的第7个错配可以容许脱靶,因此用含有5'ttn
‑
pam序列的5'末端的14nt的种子序列来搜索人基因组。仍然包括14nt种子序列中的一个错配或两个不连续的错配。应用t7ei分析确定是否存在脱靶。
[0149]
定点c2c1基因诱变。
[0150]
将含有所需定点突变和5'端重叠的两对引物用于基因扩增。使用dna assembly master mix(neb)按照制造商推荐的方案将两个琼脂糖凝胶纯化的基因片段无缝地组装到xmai和nhei双消化的哺乳动物表达载体中。大肠杆菌表达载体构建使用消化和连接依赖的方法。
[0151]
实施例1、aac2c1核酸酶活性的体外分析。
[0152]
首先,通过体外核酸切割鉴定本发明来自a.acidiphilus的c2c1的pam序列。图1a示出了aac2c1和sgrna靶向带有各种pam的基因座的切割。图下方的符号“+”表示强体外切割活性。结果显示aac2c1的pam可以是5’tttn
‑
、5’attn
‑
、5’gttn
‑
、5’cttn
‑
、5’ttc
‑
、5’ttg
‑
、5’tta
‑
、5’ttt
‑
、5’tan
‑
、5’tgn
‑
、5’tcn
‑
、5’atc
‑
。
[0153]
其次,测试了aac2c1的温度和酸碱性耐受。图1b示出了在宽温度范围(4℃
‑
100℃)下aac2c1的切割活性。图1c分析在宽范围ph值(ph1.0
‑
ph13.0)下aac2c1切割活性。图下方的符号“+”表示强体外切割活性。结果显示aac2c1在4℃
‑
100℃都可以工作,且在大约30℃
‑
60℃切割效率较高。aac2c1在ph1.0
‑
ph12.0都可以工作,且在ph1.0
‑
ph8.0切割效率较高。
[0154]
图2a示出了本发明鉴定的来自a.acidiphilus的c2c1的细菌基因组基因座的图谱。由于含有c2c1基因的a.acidiphilus的基因组基因座没有已经测序的直接重复(dr)阵列,因此本研究采用来自已报道的a.acidoterrestris的假定crrna。
[0155]
图2b显示出本实施例使用的大肠杆菌表达的aac2c1
‑
his
10
的逐步纯化。
[0156]
图2c通过来自图1a的切割产物的sanger测序所确定的切割位点。
[0157]
图2d显示在不同浓度edta和mg
2+
存在下的体外aac2c1切割测定。表明aac2c1是mg
2+
依赖性核酸内切酶。
[0158]
图2e通过aac2c1在选定的金属ca
2+
,mn
2+
,sr
2+
,ni
2+
,fe
2+
,co
2+
,zn
2+
,cu
2+
存在下进行dna切割测定。图下方的符号“+”表示体外强烈的切割活性。
[0159]
表1、用于体外dna切割测定的带有各种5'pam序列的靶序列。用于pam序列的体外dna切割分析的靶序列是商业合成的(bgi),其中分别用下划线和方框背景突出显示ecori 5'和sphi 3'突出端。将退火的寡核苷酸构建到ecori和sphi双重消化的p11
‑
lacy
‑
wtx1载体中。
[0160][0161]
实施例2 aac2c1在哺乳动物细胞中的基因组编辑活性
[0162]
本实施例检测了aac2c1在哺乳动物细胞中的基因组编辑活性。所述使用的靶序列
见下表3。
[0163]
图3a是aac2c1 sgrna
‑
dna
‑
靶向复合物的示意图。
[0164]
图3b显示的是在人类rnf2靶位点处产生的indel的t7ei分析。具有突变的泳道下的数字显示indel比例。三角形表示切割片段。
[0165]
图3c显示来自图3b的切割产物的sanger测序结果。红色字体突出显示pam序列。
[0166]
t7ei实验显示aac2c1在小鼠nr1基因座处诱导插入缺失(图3d)。图3e示出来自图3d的nrl基因靶点1切割造成的等位基因序列。
[0167]
可见,aac2c1在哺乳动物细胞中介导稳健的基因组编辑。图4的数据进一步证实这一结论。图4a通过t7ei分析显示aac2c1在小鼠apob基因座上诱导插入缺失。三角形表示切割片段。图4b为相对应sanger测序结果。图4c通过t7ei分析显示aac2c1靶向人cd34基因。图4d为相对应sanger测序结果。图4e显示aac2c1靶向人内源性rnf2基因的额外靶点。图4f为相对应sanger测序结果。图4g显示aac2c1靶向人内源性rnf2基因的额外靶点。图4h为相对应sanger测序结果。
[0168]
表2、用于靶基因扩增和t7ei测定的引物序列。
[0169][0170]
表3、哺乳动物基因组靶前间隔区序列。
[0171]
[0172][0173]
实施例3、sgrna优化
[0174]
本实施例对指导aac2c1基因组编辑的单向导rna(sgrna)进行了优化。原始sgrna为基于aac2c1基因座中的tracrrna以及a.acidoterrestris的假定crrna构建的sgrna1。
[0175]
图6a示出了在sgrna1的茎环3进行5’截短的不同版本的sgrna支架序列结构。图5a示出sgrna的茎环3的截短和破坏在体内消除了aac2c1对人内源基因rnf2靶位点8的靶向活性。图6b示出sgrna的茎环3的截短和破坏在体外消除了aac2c1靶向活性。图6c示出sgrna的茎环3的截短和破坏在体内消除了aac2c1对小鼠内源基因nrl靶位点1的靶向活性。
[0176]
图6d示出了在sgrna1的茎环2和1进行截短和优化的不同版本的sgrna支架序列结构。图5b示出sgrna1的茎环2的截短破坏体内aac2c1活性,而茎环1的截短保留aac2c1内切核酸酶活性。图5c示出进一步优化茎环1上的aac2c1 sgrna和体内功能验证。图6e和f为对应的体外和小鼠实验获得的类似结果。
[0177]
图5d和e示出使用不同直系同源sgrna(图6g)的aac2c1靶向内源性人rnf2基因的结果。t7ei分析表明aac2c1能够与a.acidiphilus、a.kakegawensis和a.macrosporangiidus的sgrna在体内发挥功能。图5e右幅的结果显示来自a.kakegawensis的akc2c1蛋白也能在哺乳动物细胞中实现基因组编辑。图6j
‑
m为对应的体外和小鼠实验获得的结果。
[0178]
图6h和i分别示出alicyclobacillus acidiphilus(nbrc 100859)、
alicyclobacillus kakegawensis(nbrc 103104)、alicyclobacillus macrosporangiidus(菌株dsm 17980)、bacillus sp.(nsp2.1)的sgrna和c2c1蛋白质序列的进化关系。
[0179]
表4、aac2c1单向导rna(sgrna)的序列优化。改造了来自alicyclobacillus acidiphilus的v
‑
b型crispr基因座的单嵌合向导rna(sgrna)。斜体和粗体分别突出了tracrrna和crrna序列。3’端的连续的n代表20nt前间区序列(靶序列)。由于a.acidiphilus crispr基因座中没有直接重复序列(dr)阵列,我们采用了直接a.acidoterrestris的v
‑
b crisrp基因座的crrna序列,并用a.acidiphilus v
‑
b crispr基因座的tracrrna进行工程化改造。列出了来自原始sgrna1的优化的sgrna的序列。n独立地选自a、t、g、c。
[0180]
[0181][0182]
实施例4、脱靶分析和预测
[0183]
图7a的实验结果显示了前间区长度对体内aac2c1靶向活性的影响。结果显示18个核苷酸以下的前间区序列无法有效切割。
[0184]
图7b显示了sgrna
‑
靶dna单个错配对体内aac2c1靶向活性的影响。结果显示对靶序列5’端第7位的错配耐受。
[0185]
图7c显示了sgrna
‑
靶dna连续错配对体内aac2c1靶向活性的影响。结果显示aac2c1在体内对sgrna
‑
靶dna连续错配不耐受。
[0186]
图7d显示了由aac2c1和携带不同前间区序列长度的sgrna介导的内源性人rnf2基因破坏的效率。误差线表示平均值的标准误差(s.e.m),n=3。
[0187]
基于图7的实验结果,对内源脱靶点进行了预测和分析。图8a示出了由人内源性rnf2基因靶位点8诱导的脱靶位点的t7ei分析。三角形标记预测的切割条带。注意,脱靶位点1在rnf2假基因座上,并且具有完全相同的间区序列和pam序列。泳道下的符号“*”表示具有不一致的切割条带的脱靶位点。图8b示出脱靶位点1、18、26、27的代表性序列。似乎“*”表示的切割条带是由于pcr扩增造成。
[0188]
表5、脱靶点分析。列出了人内源性rnf2基因靶位点8的预测的基因组脱靶位点。脱靶位点的pam序列用下划线表示,用斜体突出显示错配。
[0189]
[0190][0191]
实施例5 aac2c1中dna切割所必需的催化残基
[0192]
图9a是具有催化残基突变的aac2c1结构域的示意图。基于a.acidoterrestris c2c1(aacc2c1)(pdb:5wqe)的序列同源性鉴定催化残基。
[0193]
图9b示出催化残基突变的aac2c1变体的体外dna切割分析。图9c示出通过t7ei分析aac2c1催化残基突变对体内dna靶向的影响。结果显示r785a突变在体内或体外均消除了dna切割活性。
[0194]
图9d(上图)使用定点突变的aac2c1体外切割nb.btsi
‑
切口的dsdna片段的示意图。(下图)aac2c1变体(r785a)的体外无法切割切口dsdna。可见,包含r785a的aac2c1变体为无内切酶活性变体。此类dc2c1变体可显著扩展aac2c1的用途。
[0195]
图10示出了aacc2c1、aac2c1、akc2c1、amc2c1和bsc2c1的蛋白序列比对。aacc2c1、aac2c1、akc2c1、amc2c1和bsc2c1氨基酸序列的多重序列比对显示高度保守的残基。严格相同的残基用红色背景突出显示,保守突变框出和红色字体突出显示。二级结构预测在对齐上方突出显示。α螺旋显示为卷曲符号,β链显示为箭头。严格的α转角显示为ttt和严格的β转角显示为tt。
[0196]
序列信息:
[0197]
seq in no:1aac2c1 protein sequence
[0198]
mavksmkvklrldnmpeiraglwklhtevnagvryytewlsllrqenlyrrspngdgeqecyktaeeckaellerlrarqvenghcgpagsddellqlarqlyellvpqaigakgdaqqiarkflspladkdavgglgiakagnkprwvrmreagepgweeekakaearkstdrtadvlraladfglkplmrvytdsdmssvqwkplrkgqavrtwdrdmfqqaiermmsweswnqrvgeayaklveqksrfeqknfvgqehlvqlvnqlqqdmkeashgleskeqtahyltgralrgsdkvfekwekldpdapfdlydteiknvqrrntrrfgshdlfaklaepkyqalwredasfltryavynsivrklnhakmfatftlpdatahpiwtrfdklggnlhqytflfnefgegrhairfqklltvedgvakevddvtvpismsaqlddllprdphelvalyfqdygaeqhlagefggakiqyrrdqlnhlharrgardvylnlsvrvqsqseargerrppyaavfrlvgdnhrafvhfdklsdylaehpddgklgsegllsglrvmsvdlglrtsasisvfrvarkdelkpnsegrvpfcfpiegnenlvavhersqllklpgeteskdlraireerqrtlrqlrtqlaylrllvrcgsedvgrrerswaklieqpmdanqmtpdwreafedelqklkslygicgdrewteavyesvrrvwrhmgkqvrdwrkdvrsgerpkirgyqkdvvggnsieqieylerqykflkswsffgkvsgqviraekgsrfaitlrehidhakedrlkkladriimealgyvyalddergkgkwvakyppcqlilleelseyqfnndrppsennqlmqwshrgvfqellnqaqvhdllvgtmyaafssrfdartgapgircrrvparcareqnpepfpwwlnkfvaehkldgcplraddliptgegeffvspfsaeegdfhqihadlnaaqnlqrrlwsdfdisqirlrcdwgevdgepvliprttgkrtadsygnkvfytktgvtyyerergkkrrkvfaqeelseeeaellveadeareksvvlmrdpsgiinrgdwtrqkefwsmvnqriegylvkqirsrvrlqesacentgdi
[0199]
seq in no:2 c2c1 coding sequence from alicyclobacillus acidiphilus nbrc 100859(genebank id:nz_bcqi01000053.1)
[0200]
atggccgttaaatccatgaaagtgaaacttcgcctcgataatatgccggagattcgggctggtttatggaaactccatacggaggtcaacgcgggggttcgatattacacggaatggctgagtcttctgcgtcaagagaatttgtatcgaagaagtccgaatggggacggagagcaagaatgttataagactgcagaagaatgcaaagccgaattgttggagcggctgcgcgcgcgtcaagtggagaatggacactgtggtccggcgggatcggacgatgaattgctgcagttggctcgtcaactttatgaactgttggttccgcaggcgataggtgcgaaaggcgatgcgcagcaaattgcgcgcaagtttttgagccccttagccgacaaggatgcagtgggtgggcttggaatcgcgaaggcggggaacaaaccgcggtgggttcgcatgcgcgaagcgggagaacctggctgggaagaggagaaggcgaaggctgaggcgaggaaatctacggatcgaactgcggatgttttgcgcgcgctcgcggattttgggttaaagccactgatgcgcgtgtacaccgattctgacatgtcatctgttcagtggaaaccgcttcggaagggccaagcggttcggacgtgggacagggatatgttccaacaggccatcgagcggatgatgtcgtgggagtcgtggaatcagcgcgttggcgaagcgtacgcgaaactggtagagcaaaaaagtcgatttgagcagaagaacttcgtcggccaggaacatttggttcaactcgtcaatcagttgcaacaagatatgaaagaagcatcgcacgggctcgaatcgaaagagcaaaccgcacattatctgacgggacgggcattgcgcggatcggacaaagtgtttgagaagtgggagaaactcgaccctgatgcgccattcgatttgtacgacaccgaaatcaagaacgtgcagagacgtaacacgaggcgattcggctcacacgacttgttcgcgaaattggcggaaccgaagtatcaggccctgtggcgcgaagatgcttcgtttctcacgcgttacgcggtgtacaacagcatcgttcgcaaactgaatcacgccaaaatgttcgcgacgtttactttaccggatgcaactgcgcatccgatttggactcgctttgataaattgggcgggaatttgcaccagtacacctttttgttcaacgaattcggagaaggcaggcacgcgattcgttttcaaaagctgttgaccgtcgaagatggtgtcgcaaaagaagttgatgatgtaacggtgcccatttccatgtcagcgcaattggatgatctgctgccaagagatccccatgaactggttgcactatattttcaagattatggagccgaacagcatttggcgggtgaattcggtggcgcgaagattcagtaccgtcgggatcaactaaatcatttgcacgcacgcagaggggcgagggatgtttatctcaatctcagcgtacgtgtgcagagccagtctgaggcacggggagaacgccgcccgccgtatgccgcagtattccgcctggtcggggacaaccatcgtgcgtttgtccattttgataaattatcggattatcttgcggaacatccggatgatgggaagcttggatcggaggggctgctttccgggctacgggtgatgagtgtcgatctcggccttcgcacatcggcatcgatttccgtttttcgcgttgcccggaaggacgagttgaagccgaactcggaagggcgtgtcccattctgttttccgattgaagggaatgaaaatctcgtcgcggttcatgaacgatctcaacttttgaagctgcctggcgaaacagagtcaaaggacctgcgggctatccgagaagagcgccaacggaccctgcggcagctgcggacgcaactggcgtatttgcggctgctcgtgcggtgtgggtcggaagatgtgggacggcgtgaacggagttgggcaaagcttattgagcagcccatggatgccaatcagatgacaccggattggcgcgaagcctttgaagacgaacttcagaagcttaagtcactctatggtatctgtggcgacagggaatggacggaggctgtctacgagagcgttcgccgcgtgtggcgccatatgggcaaacaggttcgcgattggcgaaaggacgtacggagtggagagcggccgaagattcgcggctatcaaaaagatgtggtcggcggaaattcgattgagcaaattgagtatcttgaacggcagtacaagtttctcaagagttggagcttttttggcaaggtatcgggacaagtgattcgtgcggagaagggatcccgatttgcgatcacgctgcgtgaacacattgatcacgcgaaggaagaccggctgaagaaattggcggatcgcatcattatggaggcgctcggttatgtgtacgcgttggatgatgagcgcggcaaaggaaagtgggttgcgaagtatccgccgtgccagctcatcctgctggaggaattgagcgagtaccagttcaataacgacaggcctccgagtgaaaacaatcagttgatgcaatggagccatcgcggcgtgttccaggagttgttgaatcaggcccaagtccacgatttactcgttgggacgatgtatgcagcgttctcgtcgcgattcgacgcgcgaaccggggcaccgggtatccgctgtcgcagggtaccggcgcgttgcgctcgggagcagaatccagaaccatttccttggtggctgaacaagtttgtggcggaacacaagttggatggttgtcccttacgg
gcagacgacctcatccccacgggtgaaggagagttttttgtctcgccgttcagtgcggaggaaggggactttcatcagattcatgccgacctgaatgcggcgcaaaacctgcagcggcgactctggtctgattttgatatcagtcaaattcggttgcggtgtgattggggtgaagtggacggtgaacccgttctgatcccaaggaccacaggaaagcgaacggcggattcatatggcaacaaggtgttttataccaaaacaggtgtcacctattatgagcgagagcgggggaagaagcggagaaaggttttcgcgcaagaggaattgtcggaggaagaggcggagttgcttgtggaagcagacgaggcaagggagaaatcggtcgttttgatgcgtgatccgtccggcattatcaatcgtggcgactggaccaggcaaaaggagttttggtcgatggtgaaccagcggattgaaggatacttggtcaagcagattcgctcgcgcgttcgcttacaagaaagtgcgtgtgaaaacacgggggatatt
[0201]
seq in no:3 humanized aac2c1 coding sequence
[0202]
atggccgtgaagagcatgaaggtgaagctgcgcctggacaacatgcccgagatccgcgccggcctgtggaagctgcacaccgaggtgaacgccggcgtgcgctactacaccgagtggctgagcctgctgcgccaggagaacctgtaccgccgcagccccaacggcgacggcgagcaggagtgctacaagaccgccgaggagtgcaaggccgagctgctggagcgcctgcgcgcccgccaggtggagaacggccactgcggccccgccggcagcgacgacgagctgctgcagctggcccgccagctgtacgagctgctggtgccccaggccatcggcgccaagggcgacgcccagcagatcgcccgcaagttcctgagccccctggccgacaaggacgccgtgggcggcctgggcatcgccaaggccggcaacaagccccgctgggtgcgcatgcgcgaggccggcgagcccggctgggaggaggagaaggccaaggccgaggcccgcaagagcaccgaccgcaccgccgacgtgctgcgcgccctggccgacttcggcctgaagcccctgatgcgcgtgtacaccgacagcgacatgagcagcgtgcagtggaagcccctgcgcaagggccaggccgtgcgcacctgggaccgcgacatgttccagcaggccatcgagcgcatgatgagctgggagagctggaaccagcgcgtgggcgaggcctacgccaagctggtggagcagaagagccgcttcgagcagaagaacttcgtgggccaggagcacctggtgcagctggtgaaccagctgcagcaggacatgaaggaggccagccacggcctggagagcaaggagcagaccgcccactacctgaccggccgcgccctgcgcggcagcgacaaggtgttcgagaagtgggagaagctggaccccgacgcccccttcgacctgtacgacaccgagatcaagaacgtgcagcgccgcaacacccgccgcttcggcagccacgacctgttcgccaagctggccgagcccaagtaccaggccctgtggcgcgaggacgccagcttcctgacccgctacgccgtgtacaacagcatcgtgcgcaagctgaaccacgccaagatgttcgccaccttcaccctgcccgacgccaccgcccaccccatctggacccgcttcgacaagctgggcggcaacctgcaccagtacaccttcctgttcaacgagttcggcgagggccgccacgccatccgcttccagaagctgctgaccgtggaggacggcgtggccaaggaggtggacgacgtgaccgtgcccatcagcatgagcgcccagctggacgacctgctgccccgcgacccccacgagctggtggccctgtacttccaggactacggcgccgagcagcacctggccggcgagttcggcggcgccaagatccagtaccgccgcgaccagctgaaccacctgcacgcccgccgcggcgcccgcgacgtgtacctgaacctgagcgtgcgcgtgcagagccagagcgaggcccgcggcgagcgccgccccccctacgccgccgtgttccgcctggtgggcgacaaccaccgcgccttcgtgcacttcgacaagctgagcgactacctggccgagcaccccgacgacggcaagctgggcagcgagggcctgctgagcggcctgcgcgtgatgagcgtggacctgggcctgcgcaccagcgccagcatcagcgtgttccgcgtggcccgcaaggacgagctgaagcccaacagcgagggccgcgtgcccttctgcttccccatcgagggcaacgagaacctggtggccgtgcacgagcgcagccagctgctgaagctgcccggcgagaccgagagcaaggacctgcgcgccatccgcgaggagcgccagcgcaccctgcgccagctgcgcacccagctggcctacctgcgcctgctggtgcgctgcggcagcgaggacgtgggccgccgcgagcgcagctgggccaagctgatcgagcagcccatggacgccaaccagatgacccccgactggcgcgaggccttcgaggacgagctgcagaagctgaagagcctgtacggcatctgcggcgaccgcgagtggaccgaggccgtgtacgagagcgtgcgccgcgtgtggcgccacatgggcaagcaggtgcgcgactggcgcaaggacgtgcgcagcggcgagcgccccaagatccgcggctaccagaaggacgtggtgggcggcaacagcatcgagcagatcgagtacctggagcgccagtaca
agttcctgaagagctggagcttcttcggcaaggtgagcggccaggtgatccgcgccgagaagggcagccgcttcgccatcaccctgcgcgagcacatcgaccacgccaaggaggaccgcctgaagaagctggccgaccgcatcatcatggaggccctgggctacgtgtacgccctggacgacgagcgcggcaagggcaagtgggtggccaagtaccccccctgccagctgatcctgctggaggagctgagcgagtaccagttcaacaacgaccgcccccccagcgagaacaaccagctgatgcagtggagccaccgcggcgtgttccaggagctgctgaaccaggcccaggtgcacgacctgctggtgggcaccatgtacgccgccttcagcagccgcttcgacgcccgcaccggcgcccccggcatccgctgccgccgcgtgcccgcccgctgcgcccgcgagcagaaccccgagcccttcccctggtggctgaacaagttcgtggccgagcacaagctggacggctgccccctgcgcgccgacgacctgatccccaccggcgagggcgagttcttcgtgagccccttcagcgccgaggagggcgacttccaccagatccacgccgacctgaacgccgcccagaacctgcagcgccgcctgtggagcgacttcgacatcagccagatccgcctgcgctgcgactggggcgaggtggacggcgagcccgtgctgatcccccgcaccaccggcaagcgcaccgccgacagctacggcaacaaggtgttctacaccaagaccggcgtgacctactacgagcgcgagcgcggcaagaagcgccgcaaggtgttcgcccaggaggagctgagcgaggaggaggccgagctgctggtggaggccgacgaggcccgcgagaagagcgtggtgctgatgcgcgaccccagcggcatcatcaaccgcggcgactggacccgccagaaggagttctggagcatggtgaaccagcgcatcgagggctacctggtgaagcagatccgcagccgcgtgcgcctgcaggagagcgcctgcgagaacaccggcgacatc
[0203]
seq in no:4 daac2c1 protein sequence
[0204]
mavksmkvklrldnmpeiraglwklhtevnagvryytewlsllrqenlyrrspngdgeqecyktaeeckaellerlrarqvenghcgpagsddellqlarqlyellvpqaigakgdaqqiarkflspladkdavgglgiakagnkprwvrmreagepgweeekakaearkstdrtadvlraladfglkplmrvytdsdmssvqwkplrkgqavrtwdrdmfqqaiermmsweswnqrvgeayaklveqksrfeqknfvgqehlvqlvnqlqqdmkeashgleskeqtahyltgralrgsdkvfekwekldpdapfdlydteiknvqrrntrrfgshdlfaklaepkyqalwredasfltryavynsivrklnhakmfatftlpdatahpiwtrfdklggnlhqytflfnefgegrhairfqklltvedgvakevddvtvpismsaqlddllprdphelvalyfqdygaeqhlagefggakiqyrrdqlnhlharrgardvylnlsvrvqsqseargerrppyaavfrlvgdnhrafvhfdklsdylaehpddgklgsegllsglrvmsvdlglrtsasisvfrvarkdelkpnsegrvpfcfpiegnenlvavhersqllklpgeteskdlraireerqrtlrqlrtqlaylrllvrcgsedvgrrerswaklieqpmdanqmtpdwreafedelqklkslygicgdrewteavyesvrrvwrhmgkqvrdwrkdvrsgerpkirgyqkdvvggnsieqieylerqykflkswsffgkvsgqviaaekgsrfaitlrehidhakedrlkkladriimealgyvyalddergkgkwvakyppcqlilleelseyqfnndrppsennqlmqwshrgvfqellnqaqvhdllvgtmyaafssrfdartgapgircrrvparcareqnpepfpwwlnkfvaehkldgcplraddliptgegeffvspfsaeegdfhqihadlnaaqnlqrrlwsdfdisqirlrcdwgevdgepvliprttgkrtadsygnkvfytktgvtyyerergkkrrkvfaqeelseeeaellveadeareksvvlmrdpsgiinrgdwtrqkefwsmvnqriegylvkqirsrvrlqesacentgdi
[0205]
seq in no:5 akc2c1 protein sequence
[0206]
mavksikvklrlsecpdilagmwqlhratnagvryytewvslmrqeilysrgpdggqqcymtaedcqrellrrlrnrqlhngrqdqpgtdadllaisrrlyeilvlqsigkrgdaqqiassflsplvdpnskggrgeaksgrkpawqkmrdqgdprwvaarekyeqrkavdpskeilnsldalglrplfavftetyrsgvdwkplgksqgvrtwdrdmfqqalerlmsweswnrrvgeeyarlfqqkmkfeqehfaeqshlvklaraleadmraasqgfeakrgtahqitrralrgadrvfeiwksipeealfsqydevirqvqaekrrdfgshdlfaklaepkyqplwradetfltryalyngvlrdlekarqfatftlpdacvnpiwtrfessqgsnlhkyeflfdhlgpgrhavrfqrllvvesegakerdsvvvpvapsgqldklvlreeekssvalhlhdtarpdgfmaewagaklqyerstlarkarrdkqgmrswrrqpsmlmsaaqmledakqagdvylnisv
rvkspsevrgqrrppyaalfriddkqrrvtvnynklsayleehpdkqipgapgllsglrvmsvdlglrtsasisvfrvakkeevealgdgrpphyypihgtddlvavhershliqmpgetetkqlrklreerqavlrplfaqlallrllvrcgaaderirtrswqrltkqgreftkrltpswrealeleltrleaycgrvpddewsrivdrtvialwrrmgkqvrdwrkqvksgakvkvkgyqldvvggnslaqidyleqqykflrrwsffarasglvvradreshfavalrqhienakrdrlkkladrilmealgyvyeasgpregqwtaqhppcqliileelsayrfsddrppsensklmawghrgileelvnqaqvhdvlvgtvyaafssrfdartgapgvrcrrvparfvgatvddslplwltefldkhrldknllrpddviptgegeflvspcgeeaarvrqvhadinaaqnlqrrlwqnfditelrlrcdvkmggegtvlvprvnnarakqlfgkkvlvsqdgvtffersqtggkphsekqtdltdkeleliaeadearaksvvlfrdpsghigkghwirqrefwslvkqrieshtaerirvrgvgssld
[0207]
seq in no:6 c2c1 coding sequence from alicyclobacilluskakegawensis nbrc 103104(genebank id:nz_bcrp01000027.1)
[0208]
atggctgtaaaatctattaaggtcaagttgcggttgtcagagtgcccagacatcctggctggcatgtggcagctccaccgggcgacaaacgcgggggttcgatactacacagaatgggtgagcttgatgcgccaggagatcctctactcgcgcgggccggacggcggtcagcagtgctacatgaccgcggaggattgccaacgcgagctgctgcggcggctgcgcaatcgccagctccataatggccgccaggaccagcccggtacagatgcagacctactggcaatcagtaggagactctatgaaattctggtcctgcaatccatcggcaagaggggggacgcccagcagatagcgagcagcttcctcagccctctggtcgatccgaactccaaaggtgggcggggtgaagccaagtccggtcgaaagcctgcgtggcagaagatgcgcgatcaaggtgatcctcgttgggttgcggcaagggaaaagtacgagcaacgcaaggcggttgatccatctaaagaaatcctgaattcattggacgccctgggtctcaggccgctatttgcggtcttcacggagacctacaggtcgggagtcgattggaagccgctcggcaaaagccaaggtgtgcgcacatgggaccgtgacatgttccagcaggccctcgagcgcctgatgtcctgggagtcttggaaccgccgcgtgggcgaggagtacgcccgtcttttccaacagaagatgaagttcgagcaggaacacttcgcggaacagtctcatctggttaaactggcgcgcgcgttggaggcggacatgcgcgccgcttcacagggcttcgaagccaaacgcggcactgcgcaccagatcacaagacgggcgctgcgcggggcggatcgggtatttgagatatggaagagtattccagaggaagctttgttctcccaatatgatgaagtgattcgacaggtccaggcggagaaaagacgggactttgggtcccatgatctgttcgccaagttggcggaaccgaagtatcagcccctgtggcgcgccgacgagacctttttgacgcgctacgccctgtacaatggagtcttgcgggatttagagaaagcgagacagttcgccacgttcacgctgccggatgcctgcgtcaatccaatttggacgcgttttgaaagcagccaggggagcaatctgcataaatatgaatttctctttgaccacctgggacccggacggcacgcggtgcgttttcagaggctgctggtggtagagagcgaaggtgcgaaggagagggactcggtggtggtgccagtcgcgccatccgggcaactggacaagcttgtcctgcgtgaagaagagaaatcaagcgttgccttacaccttcatgacacagcccggccggacggtttcatggcagaatgggcgggggcgaagctgcaatatgaacgcagtaccttggcacgcaaggcgcgccgtgataagcaagggatgcggtcgtggcgtaggcagccgtctatgctgatgtctgcggcacagatgttggaagacgcaaagcaagccggagacgtgtatctgaacatcagtgtgcgtgtgaagagccccagtgaagtccgcggccagaggcggcctccttacgcggccctgtttcggatagacgataaacagcggcgtgtgaccgtaaattacaacaaactgtcggcttacctagaggaacatccggataaacagattccaggcgcacctgggctcctttccggtcttcgggtaatgagcgtcgaccttgggttgcgcacctccgcttccatcagtgtgttccgtgtggcaaagaaggaagaggtggaagcgctgggcgacggtcgtccccctcattattatcccatccatggcactgacgacctggtggcggtgcacgagcgctcacatttgattcaaatgccaggcgaaaccgaaacgaaacagctgcgcaagttgcgtgaggaacggcaggctgtcttgcgtccactgttcgctcaactggccctgctacggttgctggtccggtgtggtgcagccgacgagcggattcgtacacgcagttggcagcgcttgacgaagcaggggcgtgagtttacgaagcgattgacgccgtcctggcgggaggcgttggaattggagttaactcgcttggaggcgtattgcggtagggttccagacgacgaatggagccgcatcgttgatag
aacggtaatcgctttgtggcgtcgcatgggaaaacaggtgcgcgattggcgtaaacaggtgaaatccggtgcgaaagtcaaggtcaaggggtaccagctggatgtagtcggcggcaactcgctggcgcaaatcgattatctcgaacagcagtacaagtttctgcggcgctggagcttctttgcgcgggccagcggtctggttgtgcgggcggatcgcgaatcgcatttcgcagtcgctttacgccagcacattgaaaatgccaagcgggatcggctgaaaaagttggcggaccgcatcctgatggaggcgctgggctacgtgtatgaagcttccgggccgcgcgaaggacagtggacggcgcagcatccgccgtgccagttgattatcttggaggaattaagcgcgtaccggttcagtgacgaccgtccgccgagcgagaacagtaaattgatggcttgggggcatcggggaattttggaggagttggtcaaccaagcacaggttcacgacgtgttagtggggacggtgtacgccgctttttcgtcccgcttcgatgcccgcacaggcgcccctggagtgcgctgccgccgggtacccgcacgttttgtcggcgcgacggtggatgattcactgccgctttggctcacagagtttctggacaagcacaggctggataaaaacctcctgcggcctgacgatgtgattccgaccggagagggtgagtttttggtttctccgtgtggcgaggaagcggctcgggttcggcaggtgcacgccgacatcaacgcggcgcaaaacctgcagcggaggctgtggcagaattttgacattacagagctgcgtctgcgctgcgatgtgaagatgggtggcgaaggaacggtgctggtaccaagggtcaacaacgcccgcgccaaacaactgtttggaaagaaggtgttggtttcgcaagatggcgtgacgttctttgaacgcagtcaaacaggtgggaaaccgcacagcgagaagcagacggatttgaccgacaaggaactagaactaattgcggaggcggacgaggcgcgcgccaagtcggtcgtcctctttcgcgatccgtccgggcacatcggcaagggccactggattcgccaaagggagttttggtcgttggtgaagcaaaggattgaatcgcacacggcggaaaggatacgggttcgcggcgtcggtagctcgctggat
[0209]
seq in no:7 humanized akc2c1 coding sequence
[0210]
atggccgtgaagagcatcaaggtgaagctgcgcctgagcgagtgccccgacatcctggccggcatgtggcagctgcaccgcgccaccaacgccggcgtgcgctactacaccgagtgggtgagcctgatgcgccaggagatcctgtacagccgcggccccgacggcggccagcagtgctacatgaccgccgaggactgccagcgcgagctgctgcgccgcctgcgcaaccgccagctgcacaacggccgccaggaccagcccggcaccgacgccgacctgctggccatcagccgccgcctgtacgagatcctggtgctgcagagcatcggcaagcgcggcgacgcccagcagatcgccagcagcttcctgagccccctggtggaccccaacagcaagggcggccgcggcgaggccaagagcggccgcaagcccgcctggcagaagatgcgcgaccagggcgacccccgctgggtggccgcccgcgagaagtacgagcagcgcaaggccgtggaccccagcaaggagatcctgaacagcctggacgccctgggcctgcgccccctgttcgccgtgttcaccgagacctaccgcagcggcgtggactggaagcccctgggcaagagccagggcgtgcgcacctgggaccgcgacatgttccagcaggccctggagcgcctgatgagctgggagagctggaaccgccgcgtgggcgaggagtacgcccgcctgttccagcagaagatgaagttcgagcaggagcacttcgccgagcagagccacctggtgaagctggcccgcgccctggaggccgacatgcgcgccgccagccagggcttcgaggccaagcgcggcaccgcccaccagatcacccgccgcgccctgcgcggcgccgaccgcgtgttcgagatctggaagagcatccccgaggaggccctgttcagccagtacgacgaggtgatccgccaggtgcaggccgagaagcgccgcgacttcggcagccacgacctgttcgccaagctggccgagcccaagtaccagcccctgtggcgcgccgacgagaccttcctgacccgctacgccctgtacaacggcgtgctgcgcgacctggagaaggcccgccagttcgccaccttcaccctgcccgacgcctgcgtgaaccccatctggacccgcttcgagagcagccagggcagcaacctgcacaagtacgagttcctgttcgaccacctgggccccggccgccacgccgtgcgcttccagcgcctgctggtggtggagagcgagggcgccaaggagcgcgacagcgtggtggtgcccgtggcccccagcggccagctggacaagctggtgctgcgcgaggaggagaagagcagcgtggccctgcacctgcacgacaccgcccgccccgacggcttcatggccgagtgggccggcgccaagctgcagtacgagcgcagcaccctggcccgcaaggcccgccgcgacaagcagggcatgcgcagctggcgccgccagcccagcatgctgatgagcgccgcccagatgctggaggacgccaagcaggccggcgacgtgtacctgaacatcagcgtgcgcgtgaagagccccagcgaggtgcgcggccagcgccgccccccctacgccgccctgttccgcatcgacga
caagcagcgccgcgtgaccgtgaactacaacaagctgagcgcctacctggaggagcaccccgacaagcagatccccggcgcccccggcctgctgagcggcctgcgcgtgatgagcgtggacctgggcctgcgcaccagcgccagcatcagcgtgttccgcgtggccaagaaggaggaggtggaggccctgggcgacggccgccccccccactactaccccatccacggcaccgacgacctggtggccgtgcacgagcgcagccacctgatccagatgcccggcgagaccgagaccaagcagctgcgcaagctgcgcgaggagcgccaggccgtgctgcgccccctgttcgcccagctggccctgctgcgcctgctggtgcgctgcggcgccgccgacgagcgcatccgcacccgcagctggcagcgcctgaccaagcagggccgcgagttcaccaagcgcctgacccccagctggcgcgaggccctggagctggagctgacccgcctggaggcctactgcggccgcgtgcccgacgacgagtggagccgcatcgtggaccgcaccgtgatcgccctgtggcgccgcatgggcaagcaggtgcgcgactggcgcaagcaggtgaagagcggcgccaaggtgaaggtgaagggctaccagctggacgtggtgggcggcaacagcctggcccagatcgactacctggagcagcagtacaagttcctgcgccgctggagcttcttcgcccgcgccagcggcctggtggtgcgcgccgaccgcgagagccacttcgccgtggccctgcgccagcacatcgagaacgccaagcgcgaccgcctgaagaagctggccgaccgcatcctgatggaggccctgggctacgtgtacgaggccagcggcccccgcgagggccagtggaccgcccagcaccccccctgccagctgatcatcctggaggagctgagcgcctaccgcttcagcgacgaccgcccccccagcgagaacagcaagctgatggcctggggccaccgcggcatcctggaggagctggtgaaccaggcccaggtgcacgacgtgctggtgggcaccgtgtacgccgccttcagcagccgcttcgacgcccgcaccggcgcccccggcgtgcgctgccgccgcgtgcccgcccgcttcgtgggcgccaccgtggacgacagcctgcccctgtggctgaccgagttcctggacaagcaccgcctggacaagaacctgctgcgccccgacgacgtgatccccaccggcgagggcgagttcctggtgagcccctgcggcgaggaggccgcccgcgtgcgccaggtgcacgccgacatcaacgccgcccagaacctgcagcgccgcctgtggcagaacttcgacatcaccgagctgcgcctgcgctgcgacgtgaagatgggcggcgagggcaccgtgctggtgccccgcgtgaacaacgcccgcgccaagcagctgttcggcaagaaggtgctggtgagccaggacggcgtgaccttcttcgagcgcagccagaccggcggcaagccccacagcgagaagcagaccgacctgaccgacaaggagctggagctgatcgccgaggccgacgaggcccgcgccaagagcgtggtgctgttccgcgaccccagcggccacatcggcaagggccactggatccgccagcgcgagttctggagcctggtgaagcagcgcatcgagagccacaccgccgagcgcatccgcgtgcgcggcgtgggcagcagcctggac
[0211]
seq in no:8 pcag
‑
2aegfp partial sequence
[0212]
(cag
‑
nls
‑
xmai
‑
nhei
‑
nls
‑
t2a
‑
egfp
‑
sv40)
[0213]
gacattgattattgactagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatgggtcgaggtgagccccacgttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttattttttaattattttgtgcagcgatgggggcggggggggggggggcgcgcgccaggcggggcggggcggggcgaggggcggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccgaaagtttccttttatggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcggcgggcgggagtcgctgcgttgccttcgccccgtgccccgctccgcgccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccacaggtgagcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctcgtttcttttctgtggctgcgtgaaagccttaaagggctccgggagggccctttgtgcgggggggagcggctcggggggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggcccgcgctgcccggcggctgtgagcgctgcgggcgcggcgcggggctttgtg
cgctccgcgtgtgcgcgaggggagcgcggccgggggcggtgccccgcggtgcgggggggctgcgaggggaacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcagggggtgtgggcgcggcggtcgggctgtaacccccccctgcacccccctccccgagttgctgagcacggcccggcttcgggtgcggggctccgtacggggcgtggcgcggggctcgccgtgccgggcggggggtggcggcaggtgggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggaggggcgcggcggcccccggagcgccggcggctgtcgaggcgcggcgagccgcagccattgccttttatggtaatcgtgcgagagggcgcagggacttcctttgtcccaaatctgtgcggagccgaaatctgggaggcgccgccgcaccccctctagcgggcgcggggcgaagcggtgcggcgccggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccgccgtccccttctccatctccagcctcggggctgtccgcagggggacggctgccttcgggggggacggggcagggcggggttcggcttctggcgtgtgaccggcggctctagcgcctctgctaaccatgttcatgccttcttctttttcctacagctcctgggcaacgtgctggttattgtgctgtctcatcattttggcaaagctagtgaattctaatacgactcactataggccgccaccatgcccaagaagaagaggaaggttcccggggctagcccaaagaagaagaggaaagtctctagatacccttatgatgttccagattatgccggatacccatacgatgtccctgactatgcaggctcctacccttatgacgtcccagactacgccggatccaggtccggcggcggagagggcagaggaagtcttctaacatgcggtgacgtggaggagaatcccggcccaatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagtaactgcagcgcggggatctcatgctggagttcttcgcccaccccaacttgtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgtatctta
[0214]
seq in no:9 bpk2104
‑
ccdb partial sequence
[0215]
(laci
‑
t7
‑
laco
‑
nls
‑
xmai
‑
spei
‑
his
10
‑
terminator)
[0216]
tcactgcccgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgccagggtggtttttcttttcaccagtgagacgggcaacagctgattgcccttcaccgcctggccctgagagagttgcagcaagcggtccacgctggtttgccccagcaggcgaaaatcctgtttgatggtggttaacggcgggatataacatgagctgtcttcggtatcgtcgtatcccactaccgagatgtccgcaccaacgcgcagcccggactcggtaatggcgcgcattgcgcccagcgccatctgatcgttggcaaccagcatcgcagtgggaacgatgccctcattcagcatttgcatggtttgttgaaaaccggacatggcactccagtcgccttcccgttccgctatcggctgaatttgattgcgagtgagatatttatgccagccagccagacgcagacgcgccgagacagaacttaatgggcccgctaacagcgcgatttgctggtgacccaatgcgaccagatgctccacgcccagtcgcgtaccgtcttcatgggagaaaataatactgttgatgggtgtctggtcagagacatcaagaaataacgccggaacattagtgcaggcagcttccacagcaatggcatcctggtcatccagcggatagttaatgatcagcccactgacgcgttgcgcgagaagattgtgcaccgccgctttacaggcttcgacgccgcttcgttctaccatcgacaccaccacgctggcacccagttgatcggcgcgagatttaatcgccgcgacaatttgcgacggcgcgtgcagggccagactggaggtggcaacgccaatcagcaacgac
tgtttgcccgccagttgttgtgccacgcggttgggaatgtaattcagctccgccatcgccgcttccactttttcccgcgttttcgcagaaacgtggctggcctggttcaccacgcgggaaacggtctgataagagacaccggcatactctgcgacatcgtataacgttactggtttcacattcaccaccctgaattgactctcttccgggcgctatcatgccataccgcgaaaggttttgcgccattcgatggtgtccgggatctcgacgctctcccttatgcgactcctgcattaggaaattaatacgactcactataggggaattgtgagcggataacaattcccctgtagaaataattttgtttaactttaataaggagatatcatatgcccaagaagaagaggaaggttcccggggctagtcatcaccatcaccaccatcatcaccatcactaggcggccgcataatgcttaagtcgaacagaaagtaatcgtattgtacacggccgcataatcgaaattaatacgactcactatagggaattcggtacctgagaataactagcataaccccttggggcctctaaacgggtcttgaggggttttttgctgaaacctcaggcattt
[0217]
seq in no:10 puc19
‑
u6 partial sequence
[0218]
(u6
‑
basi
‑
hindiii)
[0219]
tgtaaaacgacggccagtgaattcgagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggagagaccnnnnnnnggtctcannnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnaagcttggcgtaatcatggtcatagctgtttcctg
[0220]
seq in no:11 puc19
‑
u6
‑
aasgrna1 partial sequence
[0221]
(u6
‑
aasgrna1_scaffold
‑
basi
‑
basi
‑
terminator)
[0222]
tgtaaaacgacggccagtgaattcgagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccgggtctaaaggacagaatttttcaacgggtgtgccaatggccactttccaggtggcaaagcccgttgaacttctcaaaaagaacgctcgctcagtgttctgacgtcggatcactgagcgagcgatctgagaagtggcacagagaccgagagagggtctcattttttttaagcttggcgtaatcatggtcatagctgtttcctg
[0223]
seq in no:12 puc19
‑
u6
‑
aksgrna partial sequence
[0224]
(u6
‑
aksgrna1_scaffold
‑
basi
‑
basi
‑
terminator)
[0225]
tgtaaaacgacggccagtgaattcgagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggtcgtctataggacggcgaggacaacgggaagtgccaatgtgctctttccaagagcaaacaccccgttggcttcaagatgaccgctcgctcagcgatctgacaacggatcgctgagcgagcggtctgagaagtggcacagagaccgagagagggtctcattttttttaagcttggcgtaatcatggtcatagctgtttcctg
[0226]
seq in no:13 puc19
‑
u6
‑
amsgrna partial sequence
[0227]
(u6
‑
amsgrna1_scaffold
‑
basi
‑
basi
‑
terminator)
[0228]
tgtaaaacgacggccagtgaattcgagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtaga
aagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggggaattgccgatctataggacggcagattcaacgggatgtgccaatgcactctttccaggagtgaacaccccgttggcttcaacatgatcgcccgctcaacggtccgatgtcggatcgttgagcgggcgatctgagaagtggcacagagaccgagagagggtctcattttttttaagcttggcgtaatcatggtcatagctgtttcctg
[0229]
seq in no:14 puc19
‑
u6
‑
bssgrna partial sequence
[0230]
(u6
‑
bssgrna1_scaffold
‑
basi
‑
basi
‑
terminator)
[0231]
tgtaaaacgacggccagtgaattcgagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccggccataagtcgacttacatatccgtgcgtgtgcattatgggcccatccacaggtctattcccacggataatcacgactttccactaagctttcgaatgttcgaaagcttagtggaaagcttcgtggttagcacagagaccgagagagggtctcattttttttaagcttggcgtaatcatggtcatagctgtttcctg
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1