一种利用长穗偃麦草特异序列开发的寡聚核苷酸探针
1.本发明涉及一种用长穗偃麦草特异序列开发的寡聚核苷酸探针,属于细胞生物学领域。
技术背景
2.小麦是三大粮食作物之一,为全世界20%以上的人口提供了所需要的能量和蛋白质。但是,随着世界人口总数的逐年增长,可耕地面积的缩小,加上全球气候变化,小麦种植面积逐年下降,而且育种中亲本利用单一,缺乏具有突破性的品种。面对小麦育种的现状,丰富和拓展小麦的遗传基础对小麦遗传育种工作首当其冲,十分重要。实践证明,小麦近缘野生种携带许多优良农艺性状,将这些优良基因导入普通小麦,可拓宽小麦的遗传基础,是小麦遗传育种研究中常用的途径之一。
3.长穗偃麦草是小麦近缘野生种,具有赤霉病高抗特性,是小麦遗传改良的优良基因资源,在小麦遗传和育种工作中得到了广泛的应用。通常我们会利用染色体工程手段,将所需要的染色体片段导入到栽培品种中。因此,如何准确识别长穗偃麦草染色体是获得优良种质的基础。目前,鉴定长穗偃麦草染色体身份最常用的方法是fish分子核型。近年来,随着原位杂交技术的不断进步,寡聚核苷酸探针在外源染色体的鉴定中得到广泛应用。相比于质粒探针,寡聚核苷酸探针具有更短的杂交时间,鉴定效率高,实验结果重复性好,成本低等优势。目前寡聚核苷酸探针oligo
‑
psc119.2、oligo
‑
pas1等已在小麦中广泛应用,并且信号较为丰富,可用于fish核型分析。但这些探针并非来自于长穗偃麦草特异的序列,分析的染色体核型也比较局限。因此,为了丰富长穗偃麦草染色体fish核型,本发明专利公开一个利用长穗偃麦草特异序列开发的寡聚核苷酸探针。
技术实现要素:
4.本发明的目的就是针对上述现有技术的缺陷,提供一种用长穗偃麦草特异序列开发的寡聚核苷酸探针。
5.本发明的目的是这样实现的:一种利用长穗偃麦草特异序列开发的寡聚核苷酸探针,其特征是,包括以下步骤:
6.(1)提取长穗偃麦草dna,得到dna样品,将dna样品完成整个文库制备,完成整个文库制备的步骤包括酶切、加测序接头、纯化、pcr扩增;构建好的文库通过illumina测序仪进行测序,将获取的原始序列进行质控分析,获得高质量序列;
7.(2)使用seqtk程序对获得的高质量序列进行随机选择,所选的序列为测序深度的0.25倍;
8.(3)利用repeatexplorer网站中repeatexplorer utilities工具下的preprocessing of fastq paired
‑
end reads程序对所选的序列进行整合,具体参数为read sampling:no,quality cutoff:10,percent above cutoff:95,trim reads:yes,start position:1,end position:101,rename reads:yes;
9.(4)利用repeatexplorer网站中repeatexplorer2工具下的repeatexplorer2 clustering程序对整合过的数据进行分析;
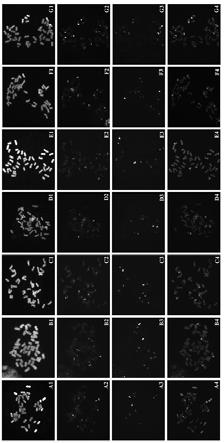
10.(5)下载分析后的数据,打开文件中tarean报告,选择报告中putative satellites(high confidence)列出的序列:
11.agcctagttcaaataattttacactagagttgaactagctctat,将该序列合成,并在序列的5’端进行tamra修饰,获得oligo
‑
pth1探针。
12.还包括步骤(6):利用荧光原位杂交技术对合成的oligo
‑
pth1探针进行验证,观察该oligo
‑
pth1探针在中国春
‑
长穗偃麦草附加系中的信号位置,并根据荧光信号绘制染色体fish核型图。
13.当前用于小麦近缘野生种染色体鉴定的方法主要是fish,即将重复序列制作成探针,利用其在不同染色体上出现的位置情况绘制成fish核型来识别染色体身份。但目前常用的探针序列并非来自于长穗偃麦草,分析的染色体核型比较局限。因此,针对已有的技术缺点,本发明提供一种利用长穗偃麦草特异序列开发的寡聚核苷酸探针,该探针可以用来丰富染色体的fish核型图,有利于准确鉴定远缘杂交种质中结构变异的染色体。
14.本发明方法先进科学,通过本发明,一种利用长穗偃麦草特异序列开发的寡聚核苷酸探针,利用测序技术获取长穗偃麦草基因组序列信息并进行质控分析,得到高质量长穗偃麦草基因组序列;使用seqtk程序对获得的高质量序列进行随机选择,所选的序列为测序深度的0.25倍;利用repeatexplorer网站中repeatexplorer utilities工具下的preprocessing of fastq paired
‑
end reads程序对所选的序列进行整合;整合后的序列利用repeatexplorer网站中repeatexplorer2工具下的repeatexplorer2 clustering程序进行分析;下载分析后的数据,打开文件中tarean报告,选择报告中putative satellites(high confidence)列出的序列:agcctagttcaaataattttacactagagttgaactagctctat,将该序列提交公司合成,并在序列的5’端进行tamra修饰,获得oligo
‑
pth1探针;利用该探针在中国春
‑
长穗偃麦草附加系中进行验证,观察荧光信号的位置,并绘制染色体fish核型图。
15.本发明的有益效果为:获得oligo
‑
pth1探针序列来自于长穗偃麦草,不同于目前常用的探针,杂交信号稳定,位置丰富,与其他探针组合使用可以丰富染色体fish核型图,不仅可以准确识别染色体的身份,还能为物种间的进化关系提供一种鉴定方式。
16.综上,本发明公开了一种利用长穗偃麦草特异序列开发的寡聚核苷酸探针,主要是通过基因组测序的手段,获取长穗偃麦草中高度重复的序列,设计成寡核酸探针oligo
‑
pth1,实验中可与其他探针组合使用,可以丰富染色体fish核型图,用于识别远缘杂交种质中染色体的结构变异。
17.小麦远缘杂交材料常常作为中间种质在小麦育种中得以应用,oligo
‑
pth1探针可以辅助鉴定出小麦背景中的染色体的结构变异,能够显著提外源染色体的鉴定准确性,便于培育出优质的小麦新种质。
附图说明
18.图1为中国春
‑
长穗偃麦草附加系荧光原位杂交结果;
19.图中:
20.a
‑
g:中国春
‑
长穗偃麦草附加da1e
‑
da7e;
21.a1
‑
g1:dapi染色体后的染色体;
22.a2
‑
g2:寡核酸探针oligo
‑
pth1的荧光信号;
23.a3
‑
g3:寡核酸探针oligo
‑
pth2的荧光信号;
24.a4
‑
g4:寡核酸探针oligo
‑
pth1和oligo
‑
pth2组合的荧光信号。
具体实施方式
25.下面通过实施例的方式进一步说明本发明。
26.实施例1
27.为了验证寡核酸探针oligo
‑
pth1的可行性,明确其荧光信号强度,利用oligo
‑
pth1与oligo
‑
pth2组合,在中国春
‑
长穗偃麦草附加系进行荧光原位杂交实验,具体操作如下:
28.(1)将oligo
‑
pth1和oligo
‑
pth2的序列送公司合成,并在序列的5’端分别进行tamra和fam修饰;
29.(2)合成后的探针根据浓度需求稀释,并根据表1中的体系配置杂交液,配置好的杂交液置于103℃的金属浴中变性13min,变性完成后立即置于
‑
20℃预冷的酒精中,放置在
‑
20℃的冰箱中以上;
30.(3)将制好的中国春
‑
长穗偃麦草附加系的染色体片子放在0.15mol/l的naoh的70%酒精溶液中变性5min;制片变性后在70%、90%和100%的三个梯度的酒精中分别脱水,每次各5min,吹干备用;
31.(4)在每张变性后的片子上加40μl杂交液,盖上盖玻片置于37℃的恒温箱中6h以上(或者杂交过夜);
32.(5)将杂交后的片子于1
×
pbs中室温洗脱3次,每次5min,洗脱后吹干;
33.(6)在吹干后的片子上加8μl的dapi(vector公司,h
‑
1200),盖上盖玻片复染10min,在nikon(ni
‑
u)荧光显微镜下观察,并用nikon ds
‑
qi1mc拍照。
34.(7)拍摄的照片利用photoshop处理后利用nis
‑
elements d4.30.00合成,用于分析oligo
‑
pth1探针在染色体上的信号。从图1中可以清楚的看到,oligo
‑
pth1探针在染色体上有明显的荧光信号,结合其它探针的荧光信号,可以绘制各染色体的fish核型图,同时也可以识别染色体的结构变异情况。
35.以上实验结果表明,寡核酸探针oligo
‑
pth1在远缘杂交种质的染色体上能产生荧光信号,可用于染色体fish核型图的绘制,以及染色体结构变异的分析。
36.表1荧光原位杂交杂交液体系
37.[0038][0039]
所用序列
[0040]
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1