肠道微生物组合及其作为系统性红斑狼疮标志物的应用的制作方法
1.本发明涉及微生物学,具体地涉及肠道微生物组合及其作为系统性红斑狼疮标志物的应用。
背景技术:
2.系统性红斑狼疮(systemic lupus erythematosus,sle)是一种多系统受累的常见慢性自身免疫性结缔组织病。sle患者主要以年轻女性为主,女性与男性比例为9:1,高峰发病年龄为20~40岁。目前普遍认为其发病原因与t淋巴细胞减少、t
‑
reg功能降低、b细胞过度活化以及功能异常有关,同时产生了大量的自身抗体从而导致多器官和多系统损害。目前系统性红斑狼疮临床诊断主要采用临床症状以及血清学的实验室自身抗体等检查手段,缺乏特异性的辅助病情判断方法及相关检测试剂盒。
3.肠道微生态体系是人体最大的生态系统,肠道中定植着超过10
14
数量级的细菌,肠道菌群基因组的基因数约为人类基因组基因总数的150倍。肠道菌群与人体相互依存,影响着包括肠道神经系统、肠道内分泌系统、免疫系统、肠道通透性等功能,在对食物消化、维生素营养、抵御外来病原微生物入侵、刺激免疫等方面起着重要作用。肠道微生态系统的平衡受到包括食物、药物等一系列外界因素的影响,抗生素、病原体等会打破肠道菌群的平衡,进而引起一系列相关病症,包括各种胃肠道疾病、肝脏性疾病、炎症性疾病、风湿性关节炎、多发性硬化症等免疫系统疾病、糖尿病、代谢综合征、肥胖等代谢系统疾病等。近年来研究表明,特定的肠道菌群可通过免疫系统影响宿主的健康状态,具有调节免疫、提高防御功能、抑制肿瘤形成、促进正常的细胞代谢、促进骨质健康、抑制骨钙流失、延缓衰老等作用。随着“肠
‑
肝轴”与“脑
‑
肠轴”概念的提出,越来越多的研究开始集中在肠道菌群的种类与功能上。通过使用益生菌操纵肠细菌组成和局部代谢物已被探索为用于疾病治疗干预的有希望的途径。
4.针对系统性红斑狼疮,目前临床尚未有十分有效的早期检查方法,因此亟需建立一种具有高灵敏性、和经济性的方法,以满足临床诊断、预防或治疗系统性红斑狼疮疾病的需求。
技术实现要素:
5.本发明的目的在于提供一种能够作为系统性红斑狼疮疾病标志物的肠道微生物组合。
6.本发明的第一方面,提供了一种肠道微生物组合,所述肠道微生物组合包括选自下组的一种或多种微生物:孢子杆菌(sporobacter)、丁酸弧菌(butyricimonas)、考拉杆菌(phascolarctobacterium)。
7.在另一优选例中,所述肠道微生物组合至少包括:孢子杆菌(sporobacter)、丁酸弧菌(butyricimonas)、和考拉杆菌(phascolarctobacterium)。
8.在另一优选例中,所述肠道微生物组合还包括选自下组的一种或多种微生物:理
炎菌(alistipes)、拟普雷沃菌(alloprevotella)、臭气杆菌(odoribacter)、和普雷沃氏菌(prevotella)。
9.在另一优选例中,所述肠道微生物组合用于系统性红斑狼疮的诊断(包括早期诊断和/或辅助诊断)和治疗(包括辅助治疗);或用于制备一试剂盒或试剂,所述的试剂盒或试剂用于系统性红斑狼疮的诊断(包括早期诊断和/或辅助诊断)和治疗(包括辅助治疗)。
10.在另一优选例中,通过pcr对各个微生物生物标志物进行检测;优选地,所述pcr包括qpcr、或rt-qpcr。
11.在另一优选例中,所述肠道微生物组合为基因组合,所述基因组合包含各微生物的特异性基因核酸片段。
12.本发明的第二方面,提供了一种用于诊断(包括早期诊断和/或辅助诊断)系统性红斑狼疮的试剂组,所述试剂组包括用于检测如本发明第一方面所述的肠道微生物组合中各微生物的试剂。
13.在另一优选例中,各所述试剂为各微生物特异的引物、探针、反义寡核苷酸、适体或抗体。
14.在另一优选例中,所述试剂为pcr检测试剂;优选地包括特异性扩增各微生物基因的引物;更优选地,还包括特异性结合各微生物基因的探针。
15.在另一优选例中,所述pcr检测试剂包括多重pcr检测体系,所述多重pcr检测体系包括引物对组,所述引物对组包括:
16.第一引物对,所述第一引物对包括如seq id no.:4所示的正向引物;和,如seq id no.:5所示的反向引物;
17.第二引物对,所述第二引物对包括如seq id no.:8所示的正向引物;和,如seq id no.:9所示的反向引物;和
18.第三引物对,所述第三引物对包括如seq id no.:13所示的正向引物;和,如seq id no.:14所示的反向引物。
19.在另一优选例中,所述引物对组包括:
20.第四引物对,所述第四引物对包括如seq id no.:10所示的正向引物;和,如seq id no.:11所示的反向引物。
21.在另一优选例中,所述多重pcr检测体系还包括探针组,所述探针组包括:
22.第一探针,所述第一探针序列如seq id no.:12所示;
23.第二探针,所述第二探针序列如seq id no.:15所示;
24.第三探针,所述第三探针序列如seq id no.:16所示;和/或
25.第四探针,所述第四探针序列如seq id no.:17所示。
26.本发明的第三方面,提供了一种试剂盒,所述的试剂盒包括如本发明第一方面所述的肠道微生物组合,和/或如本发明第二方面所述的试剂组。
27.在另一优选例中,如本发明第一方面所述的肠道微生物组合中各微生物用作标准品。
28.在另一优选例中,所述试剂盒还包括pcr缓冲液、和/或pcr反应酶系。
29.本发明的第四方面,提供了肠道微生物组合或其检测试剂的用途,用于制备一试剂盒,所述的试剂盒用于诊断(包括早期诊断和/或辅助诊断)系统性红斑狼疮,其中,所述
肠道微生物组合包括选自下组的一种或多种微生物:孢子杆菌(sporobacter)、丁酸弧菌(butyricimonas)、考拉杆菌(phascolarctobacterium)。
30.在另一优选例中,所述肠道微生物组合还包括选自下组的一种或多种微生物:理炎菌(alistipes)、拟普雷沃菌(alloprevotella)、臭气杆菌(odoribacter)、和普雷沃氏菌(prevotella)。
31.在另一优选例中,所述的诊断包括步骤:
32.(1)提供一来源于待测对象的样品,用pcr方法对样品中所述肠道微生物组合中各微生物的水平进行检测;
33.(2)将步骤(1)测得的水平与正常人群中的对应微生物水平进行比较。
34.在另一优选例中,步骤(1)中使用pcr方法对各微生物的16srdna进行检测。
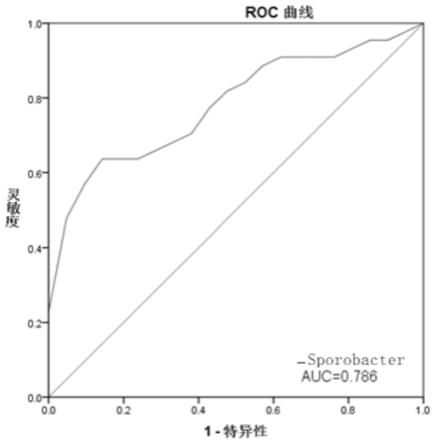
35.在另一优选例中,所述的样品选自粪便。
36.本发明的第五方面,提供了一种用于治疗(包括辅助治疗)系统性红斑狼疮的药物,所述药物具有调节患者肠道内本发明第一方面所述的肠道微生物组合的能力。
37.本发明的第六方面,提供了一种筛选用于治疗(包括辅助治疗)系统性红斑狼疮的药物的方法,所述方法包括步骤:
38.(1)在测试组中,向待测对象施用测试化合物,检测测试组中来源于所述对象的样品中所述的肠道微生物组合中各微生物的水平l1;在对照组中,向待测对象施用空白对照(包括溶媒),检测对照组中来源于所述对象的样品中所述肠道微生物组合中各微生物的水平l2;
39.(2)对上一步骤检测得到的水平l1和水平l2进行比较,从而确定所述测试化合物是否是治疗系统性红斑狼疮的候选化合物;
40.其中,所述肠道微生物组合包括选自下组的一种或多种:孢子杆菌(sporobacter)、丁酸弧菌(butyricimonas)、考拉杆菌(phascolarctobacterium)。
41.在另一优选例中,所述肠道微生物组合还包括选自下组的一种或多种微生物:理炎菌(alistipes)、拟普雷沃菌(alloprevotella)、臭气杆菌(odoribacter)、和普雷沃氏菌(prevotella)。
42.应理解,在本发明范围内,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
43.图1显示了sle患者和健康人群肠道菌群丰富度和多样性比较结果:
44.(a)采用chao1指数评估α多样性;(b)采用shannon指数评估α多样性;(***p<0.001,kruskal
‑
wallis检验)。
45.图2显示评估了sle患者和健康人群肠道菌群结构多样性的结果:
46.(a)基于bray
‑
curtis距离的pcoa可视化图;(b)pca分析图。
47.图3显示了sporobacter菌的roc曲线。
48.图4显示了butyricimonas菌的roc曲线。
49.图5显示了phascolarctobacterium菌的roc曲线。
具体实施方式
50.本发明的发明人进行了深入的研究,通过分别对来自系统性红斑狼疮个体和健康个体的肠微生物样品进行16s rrna测序,确定sle患者中存在的多种肠道微生物水平差异,确定了这些水平差异与系统性红斑狼疮的关系。这些具有水平差异的肠道微生物能够用于预防、诊断/治疗系统性红斑狼疮疾病的组合物或试剂盒。
51.本发明确认了sle患者在肠道菌物种分类上明显不同于健康人群,在多种微生物水平上存在差异(p<0.05),患者的孢子杆菌(sporobacter)、丁酸弧菌(butyricimonas)、考拉杆菌(phascolarctobacterium)表达量显著低于健康人群,差异具有显著的统计学意义,提示这些指标的表达量与系统性红斑狼疮的发病密切相关。
[0052]“16s rrna”指构成原核生物核糖体的30s小亚单位的rrna,其一方面碱基序列的大部分被高度保留,另一方面部分区域显示出高的碱基序列多样性。特别是在同种之间几乎不存在多样性而异种间显示出多样性,因此通过比较16srrna的序列,能够有效鉴定原核生物。
[0053]
在本发明优选地实施方式中,各菌株16srrna的靶基因的序列如下:
[0054]
sporobacter菌代表性序列(seq id no.:1):
[0055]
gacgaacgctggcggcgtgcctaacacatgcaagtcgaacggagacaattggttcgctgattgtcttagtggcggacgggtgagtaacgcgtgagcaatctgcccttcggagggggacaacagctggaaacggctgctaataccgcataatgtatattcaaggcatcttggatataccaaagatttatcgccgaaggatgagctcgcgtctgattagctagttggtgaggtaaaggctcaccaaggctgcgatcagtagccggactgagaggttgaacggccacattggaactgagatacgggccagactcctacgggagggagcagtggggaattttggncaatgggggaaagccntacccagcaacgccgcgtgaaggaagaaggccttcgggttgtaaacttctttgaccagggacgaaacaaatgacggtacctggaaaacaagccacggctaactacgtgccagcagccgcggtattacgtaggtggcaagcgttgtccggatttactgggtgtaaagggcgcgtaggcgggagtacaagtcagatgtgaaatctgggggcttaaccctcaaactgcatttgaaactgtatttcttgagtatcggagaggcaggcggaattcctagtgtagcggtgaaatgcgttgatattaggaggaacaccagtggcgaaggcggcctgctggacgacaactgactctgaggcgcgaaagcgtggggagcaaacaggattagataccctggtagtccacgctgtaaacgatgaatactaggtgtggggggactgaccccctccgtgccggagttaacacaataagtattccacctggggagtacgnccgcaaggttgaaactcaaaggaattgacgggggcccgcacaagcagtggattatgtggtttaattcgaagcaacgcgaagaaccttaccagggcttgacatcgtactaacgaagcagagatgcattaggtgcccttccggggaaagtatagacaggtggtgcatggttgtcgtcagctcgtgtcgtgagatgttgggttaagtcccgcaacgagcgcaacccctattgtnatttgctacncgagancactctagcgaggctgccgatgacaaaccggaggaaggtggggacgacgtcaaatcatcatgccccttatgtcctgggctacacacgtaatacaatgtctctcacagagggaagcaagaccgcgaggtggagcaaatccctaaaatgcgtctcagttcagattgcaggctgcaactcgcctgcatgaagtcggaattgctagtaatcgcggatcagcatgccgcggtgaatacgttcccgggccttgtacacaccgcccgtcacaccatgagagccgggaacacccgaagtccgtagtctaaccgcaagggggacgcggccgaaggtgggtttggtaattggggtg
[0056]
butyricimonas菌代表性序列如下(seq id no.:2):
[0057]
agagtttgatcctggctcaggatgaacgctagcgacaggcttaacacatgcaagtcgaggggcagcacggtgtagcaatacactggtggcgaccggcgcacgggtgagtaacacgtgtgcaaccaaccccgtaccgggagataacccgcggaaacgtggactaacatcccatgatactcgagagccgcatggctctcgatttaaaattccggtggtacgggacgggcacgcgcgacattaggtagttggcggggtaacggcccaccaagccgacgatgtctaggggttctgagaggaagg
tcccccacactggaactgagacacggtccagactcctacgggaggcagcagtgaggaatattggtcaatgggcgagagcctgaaccagccaagtcgcgtgagggaagaatggtctatggcctgtaaacctcttttgtcagggaagaataaggatgacgagtcattcgatgccagtacttgacgaataagcatcggctaactccgtgccagcagccgcggtaatacgggggatgcgagcgttatccggatttattgggtttaaagggcgcgtaggcgggacgtcaagtcagcggtaaaagactgcagctaaactgtagcacgccgttgaaactggcgccctggagacgagacgagggaggcggaacaagtgaagtagcggtgaaatgcatagatatcacttggaaccccgatagcgaaggcagcttcccaggctcgttctgacgctgatgcgcgagagcgtgggtagcgaacaggattagataccctggtagtccacgccgtaaacgatgctcactggatcttggcgatacactgccagggttcaagcgaaagtattaagtgagccacctggggagtacgtcggcaacgatgaaactcaaaggaattgacgggggcccgcacaagcggaggaacatgtggtttaattcgatgatacgcgaggaaccttacccgggtttaaatgtagattgcatgaggtggaaacgcttcttcccttcggggctatttacaaggtgctgcatggttgtcgtcagctcgtgccgtgaggtgtcgggttaagtcccataacgagcgcaacccctatcgccagttgccatcggttgaagccgggcactctgtcgagactgccaccgtaaggtgcgaggaaggcggggatgacgtcaaatcagcacggcccttacacccggggcgacacacgtgttacaatggccggtacagagggcagccacggggtgacccggagcgaatctctaaagccggtcgtagttcggactggagtctgcaacccgactccacgaagttggattcgctagtaatcgcgcatcagccatggcgcggtgaatacgttcccgggccttgtacacaccgcccgtcaagccatggaagccgggagtacctgaagatcgtgaccgcgaggaacgggctagggtaataccggtaactggggctaagtcgtaacaaggtaacc
[0058]
phascolarctobacterium代表性序列如下(seq id no.:3):
[0059]
gacgaacgctggcggcgtgcctaacacatgcaagtcgnacggagaattttatttcggtagaattcttagtggcgaacgggtgagtaacgcgtaggcaacctaccctttagacggggacaacattccgaaaggagtgctaataccggatgtgatcatcgtgccgcatggcaggatgaagaaagatggcctctacaagtaagctatcgctaaaggatgggcctgcgtctgattagctagttggtagtgtaacggactaccaaggcgatgatcagtagccggtctgagaggatgaacggccacattgggactgagacacggcccaaactcctacgggaggcagcagtggggaatcttccgcaatggacgaaagtctgacggagcaacgccgcgtgagtgatgaaggatttcggtctgtaaagctctgttgtttatgacgaacgtgcagtgtgtgaacaatgcattgcaatgacggtagtaaacgaggaagccacggctaactacgtgccagcagccgcggtaatacgtaggtggcgagcgttgtccggaattattgggcgtaaagagcatgtaggcggcttaataagtcgagcgtgaaaatgcggggctcaaccccgtatggcgctggaaactgttaggcttgagtgcaggagaggaaaggggaattcccagtgtagcggtgaaatgcgtagatattgggaggaacaccagtggcgaaggcgcctttctggactgtgtctgacgctgagatgcgaaagccagggtagcgaacgggattagataccccggtagtcctggccgtaaacgatgggtactaggtgtaggaggtatcgaccccttctgtgccggagttaacgcaataagtaccccgcctggggagtacggccgcaaggttgaaactcaaaggaattgacgggggcccgcacaagcggtggagtatgtggtttaattcgacgcaacgcgaagaaccttaccaaggcttgacattgattgaacgctctagagatagagatttcccttcggggacaagaaaacaggtggtgcatggctgtcgtcagctcgtgtcgtgagatgttgggttaagtcccgcaacgagcgcaacccctatcctatgttaccagcaagtaaagttggggactcatgggagactgccagggacaacctggaggaaggcggggatgacgtcaagtcatcatgccccttatgtcttgggctacacacgtactacaatggtcggaaacagagggaagcgaagccgcgaggcagagcaaaccccagaaacccgatctcagttcggatcgcaggctgcaacccgcctgcgtgaagtcggaatcgctagtaatcgcaggtcagcatactgcggtgaatacgtncccgggccttgtacacaccgcccgtcacaccacgaaagttggtaacacccgaagccggtgaggtaacctattaggagccagccgtctaaggtggngccgatgattggggtg
[0060]
本发明提供肠道微生物组合用于制备或筛选系统性红斑狼疮检测产品的用途,所述肠道微生物组合包括选自下组的一种或多种微生物:孢子杆菌(sporobacter)、丁酸弧菌
nucleic acids)、gna(glycerolnucleic acid,甘油核酸)、tna(threose nucleic acid,苏糖核酸)等人工核酸置换得到的多核苷酸。
[0073]
下面结合具体实施例,进一步详陈本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明详细条件的实验方法,通常按照常规条件如美国sambrook.j等著《分子克隆实验室指南》(黄培堂等译,北京:科学出版社,2002年)中所述的条件,或按照制造厂商所建议的条件。除非另外说明,否则百分比和份数按重量计算。以下实施例中所用的实验材料和试剂如无特别说明均可从市售渠道获得。
[0074]
实施例1研究受试者和样品收集
[0075]
在知情同意的情况下,在南方医科大学南方医院收集来自300名受试者的粪便样品,包括120个sle患者和180个健康对照。
[0076]
为了有资格纳入本研究,个人必须符合以下粪便样品收集标准:
[0077]
1)没有服用抗生素或免疫调节剂治疗,没有特定饮食(糖尿病患者、素食者等)和正常生活方式(没有额外的压力)至少3个月;
[0078]
2)任何医疗干预后至少3个月;
[0079]
3)无怀孕、肿瘤、感染或自身免疫性疾病或风湿性疾病
[0080]
为研究受试者派发粪便样本采集管,并告知具体的收集方法,受试者取10
‑
15g新鲜粪便放于带有粪便基因组保护液的采集管内,常温快递到医院实验室,4℃冰箱保存直到使用。
[0081]
实施例2 16s rrna分析肠道微生物群落
[0082]
2.1dna提取
[0083]
使用粪便微生物基因组提取试剂盒(广东南芯医疗科技有限公司)根据制造商的说明书进行dna提取。
[0084]
提取物用无dna酶的rna酶处理以消除rna污染。使用nanodrop分光光度计,qubit荧光计测定dna含量。
[0085]
2.2dna文库构建和测序
[0086]
用细菌通用引物对纯化后的基因组dna进行两轮16s rrna基因pcr扩增,完成测序样本文库构建,使用2.0fluorometer(invitrogen,usa)检测浓度,agilent2100(agilent,usa)检测文库的大小;应用illumina miseq(illumina,san diego,usa)测序仪对扩增的16s rrna高变区序列进行测序,测序区域为v3
‑
v4区。
[0087]
2.3数据分析
[0088]
使用trimmomatic v.0.32软件对原始测序数据(fastq)进行修剪和过滤,将优质序列文件通过97%相似度的标准聚类,得到操作分类单元(otu)。然后使用qiime2 v.2017.12软件和r软件进行α和β多样性分析。
[0089]
使用chao1、shannon多样性指数评估肠道菌群α多样性,使用主坐标分析(pcoa)和pca分析以评估肠道菌群的β多样性。使用基于线性判别分析效应量的分析方法(lefse)分析各组样本显著性差异的类群。
[0090]
2.4结果
[0091]
通过基于16s rrna的分析获得了健康人群、sle患者的肠道微生物群落信息,证实了,sle患者的肠道菌群多样性和丰富度较健康人群明显降低(图1)。
[0092]
pcoa分析以及pca分析评估肠道菌群β多样性,结果,sle患者和健康人群有明显的分离趋势,提示sle患者的肠道菌群结构不同与hc组(图2)。
[0093]
此外,确认了sle患者在肠道菌物种分类上明显不同于健康人群,在多种肠道微生物水平上存在差异(p<0.05),患者的sporobacter菌、butyricimonas菌、phascolarctobacterium菌表达量显著低于健康人群,差异具有显著的统计学意义,提示这些指标的表达量与系统性红斑狼疮的发病密切相关(表1)。
[0094]
表1差异物种
[0095][0096][0097]
实施例3 sle患者肠道菌群的临床检测验证
[0098]
上述研究表明,sle患者在肠道菌物种分类上明显不同于健康人群,在多重肠道微生物水平上存在差异,患者的sporobacter菌、butyricimonas菌、phascolarctobacterium菌表达量显著低于健康人群(p<0.05)。
[0099]
根据相关菌群与sle的相关性,可以通过检测患者样本中相关菌群的丰度对sle进行诊断。
[0100]
因此,本实施例收集了健康和sle患者的粪便样本各100例,将样本编号后,进行测试。针对目标微生物的16s rrna基因序列,设计特异性荧光定量pcr引物探针,并设置内标。
[0101]
检测样本中sporobacter菌、butyricimonas菌、phascolarctobacterium菌丰度的实时荧光定量pcr(quantitative real
‑
time pcr)特异性引物序列如表2所示。
[0102]
表2
[0103][0104]
荧光定量pcr扩增检测体系:各菌株上下游引物各0.5μl,模板dna 5μl,sybr green 6μl。荧光定量pcr反应条件为:95℃10min,95℃15s,60℃2min,共40个循环。
[0105]
每个样本分别检测9种细菌,设置一个内标基因和一个纯水对照样本。
[0106]
在abi7500荧光实时定量pcr仪上进行pcr反应,采用δδct法(ct值比较法)来计算样本中各目的基因的表达丰度。
[0107]
使用logistics回归模型进行分析,并绘制roc曲线来考量菌种的诊断性能。roc曲
线是根据一系列不同的二分类方式(分界值或决定阈),以真阳性率(灵敏度)为纵坐标,假阳性率(1
‑
特异度)为横坐标绘制的曲线。roc曲线越靠近左上角,标志物的诊断准确性就越高,最靠近左上角的roc曲线的点是错误最少的最好阈值,其假阳性和假阴性的总数最少。计算各个潜在标志物的roc曲线下的面积(auc)可判断潜在标志物诊断价值,auc越大其诊断价值就越大。
[0108]
表3.诊断潜力分析
[0109][0110]
检测结果显示,相对于健康个体,sle患者的肠道菌群中sporobacter菌、butyricimonas菌、phascolarctobacterium菌呈现显著性差异(p<0.05)。该结果与16srrna文库测序结果一致。
[0111]
图3至图5显示了各菌群的roc曲线,结果表明检测的菌群均具有较高的灵敏度和特异性,因此能够用于sle患者的辅助诊断。
[0112]
因此,当受测人群的肠道菌群中sporobacter菌、butyricimonas菌、phascolarctobacterium菌的其中一个或多个指标低于正常人时,则患系统性红斑狼疮的几率较大,由此有助于协助疾病病情的判断,同时也是治疗系统性红斑狼疮的潜在靶点。
[0113]
实施例4检测试剂盒的制备
[0114]
本实施例提供了一种基于检测样本中sporobacter菌、butyricimonas菌、和phascolarctobacterium菌丰度的实时荧光定量pcr(quantitative real
‑
time pcr)检测试剂盒。
[0115]
为了提高样本检测效率,本发明人在研究过程中,对上述各菌株的pcr扩增引物进行了多轮设计及实验验证,以获得能够对上述多个菌株进行多重检测的多重检测体系。经过人工设计多对引物和探针,再对其进行优化选择并验证,最后将筛选出的针对各菌群的不同引物、探针进行组合测试,最终很好地解决多重荧光pcr体系引物间互相干扰抑制的难题,获得了具有较佳特异性和灵敏度的多重检测体系,体系中各引物探针序列信息如下,其中,f为正向引物,r为反向引物,p为探针。
[0116]
表4.多重检测体系
[0117][0118]
使用过程中pcr反应体系中各组分的含量如下:
[0119]5×
pcr buffer5μl各引物
‑
f(50μmol/l)0.3μl各引物
‑
r(50μmol/l)0.3μl各探针
‑
p(50μmol/l)0.1μl20mmol/l dntps(含dutp)0.3μlpcr反应酶系3μl模板核酸5μldepc水补足至25μl
[0120]
在实时荧光pcr仪中进行扩增反应,荧光通道选择fam、vic、texas red和cy5,pcr扩增程序如下;
[0121]
50℃,2min,95℃,15min;1个循环
[0122]
94℃,15sec,55℃,45sec(搜集荧光);40个循环。
[0123]
pcr结束后通过不同荧光通道曲线及ct值,采用δδct法(ct值比较法)来计算样本中各目的基因的表达丰度。
[0124]
分别使用上述多重检测体系,对实施例3中的各100例健康和sle患者样本进行检测。检测结果与实施例3中单重检测检测结果进行比对,判断临床符合率。结果表明,上述三个多重检测体系的检测结果与实施例3的单重检测结果能够完全对应,临床符合率达到100%。
[0125]
对比例1
[0126]
本发明人对目标菌的16srna基因序列,进行深入比对分析后,针对各目标序列设计了数对引物和探针,由于多重反应体系引物之间的竞争抑制、引物特异性差异、退火温度不一致、以及引物二聚体等原因,很难获得有效的多重pcr扩增引物以及探针序列。通过大量的实验,对设计的引物和探针进行优化选择并验证,最终确定了可以用于多重pcr扩增的
引物和探针序列及其组合。
[0127]
实验中发现,即使在已经基本确定针对各目标核酸的引物对和探针序列的情况下,不同引物对组合进行多重扩增的效果也存在显著地差异。
[0128]
例如,在多重pcr步骤中,将上述多重反应体系中的butyricimonas菌引物序列替换为对照引物对1:
[0129]
f1:ggtccagactcctacgggag(seq id no.:6)
[0130]
r1:gcagcaccttgtaaatagcc(seq id no.:7)
[0131]
其他组分不变。
[0132]
上述对照引物对1在单重pcr检测中能够正常检测出butyricimonas菌靶核酸序列。
[0133]
在多重检测体系中,20例样本的检测结果表明,包含该对照引物对1的多重检测体系,仅在13例样本中检测出目标butyricimonas菌,准确性较差,无法满足临床检测要求。
[0134]
将上述多重反应体系中的sporobacter菌引物序列替换为对照引物对2:
[0135]
f2:gcggcgtgcctaacacatg(seq id no.:18)
[0136]
r2:cataatccactgcttgtgcg(seq id no.:19)
[0137]
其他组分不变。
[0138]
上述对照引物对2在单重pcr检测中能够正常检测出sporobacter菌靶核酸序列。
[0139]
在多重检测体系中,20例样本的检测结果表明,包含该对照引物对2的多重检测体系,仅在16例样本中检测出目标sporobacter菌,准确性无法满足临床检测要求。
[0140]
在本发明提及的所有文献都在本技术中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本技术所附权利要求书所限定的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1