一种逆向跨多级神经示踪方法
1.本发明涉及采用病毒进行神经示踪研究技术领域,具体涉及一种利用g蛋白缺陷型狂犬病毒进行逆向跨多级神经示踪的方法。
背景技术:
2.狂犬病(rabies)是全世界已知最致命的神经性病毒性疾病之一。绝大部分病例的产生与欧洲、亚洲和非洲的狗咬伤以及美洲的蝙蝠咬伤离不开关系。不同亚种的狂犬病毒(rabies virus,以下简称为rv)导致的狂犬病表型有所差异,大致分为狂躁型狂犬病与瘫痪型狂犬病,被咬伤后根据rv亚种不同以及伤口大小,发病率也不尽相同,相同的是其发病后接近百分之一百的死亡率,全球每年有数以万计的人因狂犬病死亡。
3.rv归类于弹状病毒科(rhabdoviridae)狂犬病毒属(lyssavirus)。病毒外壳呈弹状,有包膜包裹,核衣壳螺旋对称,内含负义单链rna,编码5种病毒蛋白,分别为g蛋白(glycoprotein,糖蛋白,以下简称为g蛋白),n蛋白(nucleoprotein,核蛋白),p蛋白(phosphoprotein,磷蛋白),m蛋白(matrix protein,基质蛋白)和l蛋白(large protein,大蛋白,本质是rna导向的rna聚合酶)。其中g蛋白与rv的感染与传播息息相关,在失去g蛋白的情况下,rv无法形成感染性病毒颗粒,也无法感染与传播,从另一个意义上来说,g蛋白也定义了rv的感染与传播特性。不同亚种的rv携带的g蛋白的序列与特性不尽相同,在神经系统中的感染与传播效率也有所不同。根据rv的致死与否,rv目前的亚株又被分为不致死的疫苗株以及致死的街毒株。相对来说,街毒株g蛋白的感染与传播效率强于疫苗株,以实验室常用的街毒株以及疫苗株来说,作为街毒株的cvs株g蛋白的感染与传播效率就要显著强于作为疫苗株的sad株g蛋白。
4.目前rv因为其嗜神经以及失去g蛋白后没有感染与传播能力的特性,被广泛应用于神经系统的环路标记上。神经系统的环路标记根据其电信号的传播方向分为两大类,一个是顺着电信号方向传播的顺向神经示踪标记,另一个是逆着电信号方向传播的逆向神经示踪标记。根据其跨神经元级数的标记模式,又被分为点对点精准标记的跨单级神经示踪标记,以及接连不断感染,并标记所有感染神经元的跨多级神经示踪标记。可以说rv是目前最为成功的逆向跨单级神经示踪病毒工具之一。其做法是将rv基因组上的g蛋白基因敲除之后,原位添加了一个gfp(绿色荧光蛋白,以下简称gfp)基因用于表达gfp,从而对它感染上的神经元进行绿色荧光标记,人为控制这个被感染的神经元a为rv提供g蛋白,就可以让rv完成生活周期跨一级出去,利用神经元之间的突触连接进行感染,感染所有和这个神经元a有关系的神经元b并进行标记,而神经元b不能为rv提供g蛋白,这样rv的感染和传播就会到此为止,一轮标记流程下来就只会标记神经元a和所有与神经元a相关的神经元b。这样人们就可以通过荧光显微镜来观察被绿色荧光标记的神经元,从而对大脑以及神经元之间的链接进行研究。
5.之所以要敲除掉g蛋白基因,一是由于携带g蛋白基因的rv可以完成它完整的病毒感染生活周期,这样就和感染狂犬病毒没有任何区别,有相当程度的生物安全危险性。二是
由于实验目标的需要,即对脑内神经环路的研究需要相对精准的点对点关系,如果一个病毒可以在神经元之间不断地感染复制,那么当我们观察时,根本无法判断这些后续被rv感染和标记的神经元究竟是不是和所要研究的目标脑区有着直接联系,这种跨多级的标记方式并不是脑内研究的主流需求,所以rv作为神经示踪工具使用时,基因组里基本都是没有g蛋白的。
6.但是,用去除g蛋白基因的rv进行神经示踪也造成了一个空白,在脑内研究中跨多级的标记方式不是主流并不代表这个示踪方式不被人需要,研究外周神经环路与中枢神经系统的关系时,采用点对点也就是跨单级标记方式的工具根本无法从外周回到脑内,所以必须采用跨多级的标记方式进行标记。而rv如上所述,由于其特性,如果能够跨多级标记,那就和感染rv没有多少区别,有相当程度的生物安全危险性。所以目前这项任务主要由标记效果与标记效率比rv相对差很多的病毒工具,假狂犬病毒,也就是pseudorabies virus(以下简称prv)示踪工具来担当。让prv基因组携带gfp基因或者其他颜色的荧光蛋白基因来对每一个感染的神经元进行标记,因为prv不感染灵长类动物,所以生物安全性上相对rv来说安全不少,也是现在最主流的,用于逆向跨多级神经示踪的病毒工具。
7.prv示踪工具作为逆向示踪病毒工具与rv示踪工具相比,在常用的小鼠模型动物上的标记效率(指感染神经元的数量)以及标记效果(指神经元标记亮度)均较弱,并且由于更大的神经元毒性,被标记的神经元往往呈现出不正常的形态,并在非常短的标记周期中失去生物活性,不利于进行下一步的研究。而且作为它优点之一的生物安全性是因为它不感染灵长类动物,这又带来另外一个问题:后续脑连接组的研究需要利用神经示踪工具来研究更接近人类的灵长类模式动物比如猴子,那么毫无疑问prv示踪工具根本无法胜任——因为它感染不了猴子——并影响后续的脑连接组计划。
技术实现要素:
8.本发明的目的是提供一种逆向跨多级神经示踪方法,以解决rv标记所带来的生物安全性问题,以及prv标记效率和标记效果弱、不能感染灵长类动物的问题。
9.本发明技术方案详述如下:
10.一种逆向跨多级神经示踪方法,是构建能够表达狂犬病病毒g蛋白的转基因动物,再用失去g蛋白的缺陷型狂犬病病毒株感染转基因动物,利用转基因动物被感染的细胞为失去g蛋白的缺陷型狂犬病病毒株提供g蛋白,恢复感染传播活性,实现逆向跨多级神经示踪目的。
11.可选或优选的,上述逆向跨多级神经示踪方法中,所述转基因动物构建方法如下:构建含有外源g蛋白编码基因的打靶载体,通过同源重组的方式插入到实验动物胚胎干细胞的rosa26位点两外显子之间,胚胎干细胞发育获得外源全身性表达g蛋白的转基因动物。
12.可选或优选的,上述逆向跨多级神经示踪方法中,所述打靶载体为rosa26-tdtomato-g-tva-vector,基因组包括5’同源臂、启动子cag、lsl表达盒、tdtomato序列、g蛋白编码基因序列、ires序列、tva序列、wpre序列、pa序列、frt序列、neo抗性基因、3’同源臂、pgk启动子、dta序列、polya signal、cole1质粒复制起点、ampr抗性基因、f1单链dna复制起点。
13.上述打靶载体中,各基因序列的功能说明如下:
[0014]5’
同源臂和3’同源臂是用于同源重组到rosa26位点的两端序列。
[0015]
启动子cag,是高效无差别表达的启动子。
[0016]
lsl表达框:loxp-stop-loxp.两端的loxp是cre重组酶的识别位点,中间的stop是polya,用来终止表达的序列。有lsl表达框在,后续的基因都没法表达。在有cre重组酶的情况下,由cre重组酶识别两端的loxp位点,把中间的stop切除,从而让后续的基因能够表达。
[0017]
tdtomato序列:第一个字母t小写,第三个字母t大写,是表达红色荧光蛋白的基因序列。
[0018]
g蛋白编码序列:表达街毒g蛋白的基因序列。该g蛋白编码序列可以来源于cvs-n2c株rv、drv株、cvs-11株等,本发明并不做限定。
[0019]
ires序列:内部核糖体进入位点,使连接的基因能够独立地起始翻译。
[0020]
tva序列:表达特异性识别禽类肉瘤病毒(aslv)的外膜蛋白(enva)受体(tva)的基因序列。
[0021]
wpre序列:用于增强与稳定表达的辅助序列。
[0022]
pa序列:同下述的polya signal,都是终止表达的序列,lsl表达框里的stop即是该序列。
[0023]
frt序列:flpo重组酶的识别位点。在有flpo重组酶的情况下,将整个表达框进行切割。用于终止后代品系继续表达该插入序列。
[0024]
neo抗性基因:新霉素抗性基因,用于在真核细胞中筛选目的序列是否转入到es细胞中的筛选标记。
[0025]
pgk启动子、dta序列、polya signal、cole1质粒复制起点、ampr抗性基因、f1单链dna复制起点,均是cole1质粒自带的序列。
[0026]
可选或优选的,上述逆向跨多级神经示踪方法中,所述打靶载体添加有酶切位点,包括salⅰ、smaⅰ、ecor
ⅴ
、xhoⅰ,其中,
[0027]
salⅰ分别添加在启动子cag与lsl表达盒之间、tdtomato序列和g蛋白编码基因序列之间、wpre序列和pa序列之间、neo抗性基因和3’同源臂之间、3’同源臂和pgk启动子之间,ampr抗性基因和f1单链dna复制起点之间;
[0028]
smaⅰ分别添加在g蛋白编码基因序列和ires序列之间、pa序列和frt序列之间;
[0029]
ecor
ⅴ
分别添加在tva序列和wpre序列之间、neo抗性基因和3’同源臂之间、3’同源臂和pgk启动子之间;
[0030]
xhoⅰ添加在polya signal和cole1质粒复制起点之间。
[0031]
可选或优选的,上述逆向跨多级神经示踪方法中,所述插入外源g蛋白编码基因后的rosa26位点所在基因核苷酸序列如seq id no.1所示。
[0032]
可选或优选的,上述逆向跨多级神经示踪方法中,所述转基因动物为鼠、狗、或非人的灵长类动物。
[0033]
可选或优选的,上述逆向跨多级神经示踪方法中,所述失去g蛋白的缺陷型狂犬病病毒株为sad-b19或cvs-n2c。
[0034]
与现有技术相比,本发明具有如下有益效果:
[0035]
用失去g蛋白的缺陷型狂犬病毒株rv与补充g蛋白的辅助病毒如aav(腺相关病毒)配合使用的策略,目前广泛应用于逆向跨单级神经示踪中用绿色荧光标记神经元。而本发
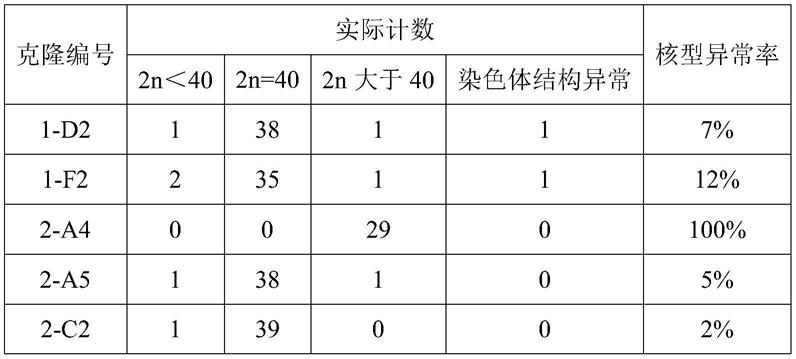
明的示踪方法,是采用能够表达g蛋白的转基因动物体内被感染的任何一个细胞为失去g蛋白的狂犬病毒rv示踪工具提供g蛋白,恢复rv的感染传播活性,让rv示踪工具能够连续不断地感染神经元,从而实现逆向跨多级标记的目的。这种方式由于表达g蛋白的转基因动物和缺乏g蛋白的狂犬病毒可以分开制备和运输保存,各自独立存在时没有感染致病的能力,可视作无感染性材料,因此生物安全性更有保证。
[0036]
本发明方法,对神经元的标记效率以及效果都更加优秀;因为rv的生活周期以及感染特性的原因,rv的神经元毒性要比prv小得多,更有利于后续对神经元形态以及功能上的研究;另外,因为rv的基因组相比prv来说长度更短,研究更透彻,所以更利于基因操作,方便后续改造并提供不同功能的病毒工具方便后续研究的跟进。最后,rv能感染灵长类动物而prv不能,所以rv更适合后续脑连接组研究的需求。
附图说明
[0037]
图1为打靶前后es细胞rosa26位点基因结构示意图;
[0038]
图2为打靶载体结构示意图;
[0039]
图3为打靶载体酶切验证结果电泳图,m为marker,1为xhol酶切结果,2为scal和xhol双酶切结果,3为salⅰ酶切结果,4为smaⅰ和ecor
ⅴ
双酶切结果,ck为对照组;
[0040]
图4为对重组的es细胞克隆鉴定电泳结果图;
[0041]
图5为f1代小鼠的基因检测策略示意图;
[0042]
图6为f1代小鼠基因检测验证结果扩增产物电泳图;
[0043]
图7为primitive line与纯合dppa3-cre小鼠杂交前后基因变化示意图;
[0044]
图8为三只f1小鼠经过绿光进行激发后的荧光结果,左一左二均未产生激发荧光,右一身体被激发出红色荧光;
[0045]
图9为g蛋白缺陷型cvs-n2c株rv的基因型和原始基因型对照;
[0046]
图10为使用g蛋白缺陷型rv进行神经示踪实验所得gt鼠组织切片荧光照片,上排为蓝色和绿色两种荧光,蓝色标识细胞核,绿色标识神经元;下排为单一绿色荧光标识的神经元。
具体实施方式
[0047]
下面结合具体实施例对本发明作进一步说明,以使本领域的技术人员可以更好的理解本发明并能予以实施,但所举实施例不作为对本发明的限定。
[0048]
在本说明书中,除非特别指明,否则所用技术术语为本领域中的普通技术人员常用术语;本说明书中未注明具体条件的实验方法是按常规实验方法;本说明书中所用的试验材料如无特别说明均为市售购买产品,各种试剂及培养基的成分和配制方法可参见常规实验手册中的操作。
[0049]
实施例1打靶载体构建
[0050]
请参考图1结合图2,以cole1质粒作为出发载体,将图1中2号所示序列(包括启动子cag、lsl表达盒、tdtomato序列、cvs-g序列(表达rv cvs-11株型中的g蛋白的基因序列)、ires序列、tva序列、wpre序列、pa序列、frt序列、neo抗性基因)插入到了该质粒中,用于表达tdtomato、g蛋白以及tva。获得打靶载体,打靶载体结构如图2所示。
[0051]
该载体在外源g蛋白编码基因上游克隆有lsl表达盒,通过cre介导实现g蛋白的条件性表达。lsl表达盒即loxp-stop-loxp表达盒,包含3
×
stop元件,即3个重复的sv40polya,序列两端为loxp位点,设置在外源g蛋白编码基因上游,能够限制外源g蛋白编码基因的表达。cre重组酶不存在的情况下,该lsl表达盒能够完全阻止外源g蛋白编码基因的正常表达,当将cre重组酶引入后,外源g蛋白编码基因上游的lsl表达盒将会被删除,使得lsl外源g蛋白编码基因能够在启动子下被激活表达。通过这种条件性表达的限制,可以有效避免全身性表达g蛋白引起动物死亡导致实验失败的情况发生,提高实验成功率。
[0052]
对打靶载体进行酶切验证的策略如下:
[0053]
1、用限制性内切酶xhol进行酶切,酶切产物大小应为19554bp;
[0054]
2、用限制性内切酶scal和xhol进行双酶切,酶切产物大小应为554bp、1784bp、5577bp和11639bp四种;
[0055]
3、用限制性内切酶salⅰ进行酶切,酶切产物大小应为2409bp、3244bp、6529bp和7372bp四种;
[0056]
4、用限制性内切酶smaⅰ和ecor
ⅴ
进行酶切,酶切产物大小应为866bp、981bp、1623bp、4677bp和11407bp五种。
[0057]
酶切验证实验结果如图3所示,酶切产物长度符合预期,证明打靶载体构建成功。
[0058]
实施例2转基因小鼠构建
[0059]
以c57bl/6小鼠作为实验动物。
[0060]
将打靶载体线性化,然后电转c57bl/6小鼠的es细胞(embryonic stem cell,胚胎干细胞,以下简称es细胞)。筛选正确重组的es细胞克隆(即打靶成功的es细胞克隆)。
[0061]
筛选策略:选用southern blot方法,同时利用5’probe(探针),3’probe和neo probe进行es细胞筛选,在5’端loxp位点和3
×
stop元件之间引入5’端southern酶切位点,在neo cassette的下游有3’端southern酶切位点,若发生正确重组,将会出现野生型和突变型两个条带;若未正确重组,将只会出现野生型条带。
[0062]
限制性内切酶探针(probe)野生型(wt)扩增结果突变型(targeted)扩增结果hindⅲ5’4.4kb5.5kbecor
ⅴ3’
11.5kb9.5kbxbaⅰ3’neo-5.7kb
[0063]
当neo基因被整合进真核细胞dna后,则能启动neo基因编码的序列转录为mrna,从而获得抗性产物氨基糖苷磷酸转移酶的高效表达,使细胞获得抗性而能在含有g418的选择性培养基中生长。因此我们利用g418进行初筛,获得了200个重组克隆。
[0064]
经5’probe(探针),3’probe和3’neo probe对初筛获得的200个重组克隆进行es细胞筛选后,共筛选到8个两端均正确的重组es细胞克隆1-a2,1-a3,1-b2,1-d2,1-f2,2a4,2-a5,2-c2。结果如图4所示。
[0065]
由于在es细胞传代过程中,随着传代次数的增加,二倍体核型比率会逐渐减小,而具有正常二倍体核型的es细胞才能发挥正常功能,因此,需要对上述重组es细胞克隆进行核型检测,以验证哪些组别的es细胞克隆能够正常使用。结果如下表所示:
[0066][0067]
结果显示,2-a4组的es细胞克隆核型异常率过高,不能使用。而1-d2,2-a5,2-c2核型异常率在10%以下,可以使用,拟作为实验候选克隆。
[0068]
我们选择编号1-d2的克隆进行es细胞显微注射,并获得f1代小鼠。具体是将打靶成功的编号1-d2的es细胞显微注射到小鼠囊胚中,将注射后成活的囊胚移植入假孕母鼠子宫内,获得f0代嵌合体小鼠,将f0代嵌合体小鼠和野生型小鼠交配,获得来源于打靶es的基因修饰小鼠,即f1代小鼠,其基因组中含有tdtomato-g-tva表达框。
[0069]
由于使用含插入有外源g蛋白编码基因的胚胎干细胞通常是采用显微注射方式注射到小鼠囊胚中,发育后获得的f0代小鼠身上只有一部分细胞是由被注射的外源g蛋白编码基因的胚胎干细胞长成的,即f0代小鼠是嵌合鼠。如果该嵌合鼠的生殖系统不是由被注射的外源g蛋白编码基因的胚胎干细胞长成的,则用该f0代小鼠与cre小鼠进行杂交,后代不会出现携带g蛋白编码基因的小鼠,杂交没有意义。所以为了提高方法的稳定性,获得更稳定的、全身都表达g蛋白的小鼠,会使用f0代嵌合体小鼠先经过一轮自交或与野生型小鼠杂交,挑选能够全身表达g蛋白的子代即f1代小鼠,用该f1代小鼠与cre小鼠交配,会更加稳定可靠。
[0070]
我们需要对该f1代小鼠进行基因检测,以验证外源g蛋白编码基因成功插入到了f1代小鼠基因中,基因检测策略如图5所示,方法如下:提取f1代小鼠基因组dna作为模板,分别用以下两对引物进行pcr扩增,如果同时出现469bp和296bp的扩增产物,则验证成功。
[0071][0072]
备注:wt:野生型,mut:含有外源g蛋白编码基因的突变型。
[0073]
pcr实验条件如下:
[0074]
95℃ 5min;
[0075]
95℃ 30sec,63℃ 30sec,72℃ 30sec,35次循环;
[0076]
72℃ 10min;
[0077]
4℃ 10min。
[0078]
验证结果扩增产物电泳如图6所示,图中1,2,3,5号f1代小鼠为阳性杂合子,是含有外源g蛋白编码基因的,以primitive line作为其代称。
[0079]
得到该primitive line后,我们拿它与纯合dppa3-cre小鼠(标记为cre line)进行杂交,杂交策略如图7所示。primitive line基因组中含有tdtomato-g-tva表达框,其中启动子cag后跟随lsl表达盒,之后有tdt(即tdtomato,红色荧光蛋白)基因和cvs-g(表达rv cvs-11株型中的g蛋白的基因序列)以及ires和tva;cre line基因组中含有cre基因(即cre重组酶基因),二者杂交的子代(标记为gt line)则能够删除lsl表达盒而表达tdt基因和cvs-g基因。子代gt line在理论上,tdtomato-g-tva表达框的表达不受限制,并会在全身表达红色荧光。
[0080]
我们以该理论为依据,通过观察新生小鼠是否能观察到全身自发荧光来筛选目标转基因小鼠gt line,简称gt鼠。
[0081]
结果如图8所示,以其中三只f1子代小鼠为例,用绿光进行激发,我们发现左边两只小鼠都没有产生激发荧光,而右一可以看见身体被激发出红色荧光,即是gt line中tdtomato-g-tva表达框正常表达的表现,以此证明右一这种子代鼠即是我们所最终构建完成的gt鼠。
[0082]
实施例3转基因动物与g蛋白缺陷狂犬病毒结合进行神经示踪
[0083]
以实施例2构建的能够表达g蛋白的gt鼠作为被感染动物,利用g蛋白缺陷型cvs-n2c株rv作为感染毒株,对gt鼠进行接种感染(滴鼻或者肌肉注射)。
[0084]
如图9所示,1是该毒株的原始基因型,携带n,p,m,g,l五种病毒蛋白的编码基因。2是该毒株缺失g蛋白之后的基因型,g蛋白被替换成了gfp(绿色荧光蛋白)。
[0085]
感染以后,由gt鼠身体内被感染的任何一个细胞为g蛋白缺陷型cvs-n2c株rv提供g蛋白,恢复街毒rv的感染传播活性,模拟rv街毒的自然感染进程,即可通过g蛋白缺陷型毒株自身所携带的荧光基因表达而观察其对提供g蛋白的转基因动物的神经元的感染和传播情况。
[0086]
实施例4使用g蛋白缺陷型rv进行神经示踪
[0087]
使用sad-b19
‑△
g-gfp这个g蛋白缺陷型(
△
g)的rv作为示踪病毒工具,注射在实施例2方法所述的gt鼠的股直肌中,病毒滴度为2*10^8pfu/ml,注射体积为1微升。
[0088]
等待24天后,对该gt鼠进行心脏灌流,多聚甲醛固定后取大脑组织,脱水,最后对处理好的大脑样品进行冠状切片,通过荧光显微镜观察大脑是否已经被g蛋白缺陷型的rv示踪病毒感染和标记。
[0089]
因为该病毒工具基因组中携带绿色荧光蛋白的表达基因gfp,所以被感染的神经元中会被绿色荧光标记。根据实验结果,我们发现,本来不能在野生鼠体内传播和感染的g蛋白缺陷型的rv示踪病毒工具能够在gt鼠中从肌肉感染并传播回到大脑内,感染和标记一系列的脑区和神经元,证明我们的gt鼠确实能够帮助g蛋白缺陷型的rv示踪病毒工具重新获得传播与感染的能力,并完成标记任务,实现逆向跨多级神经示踪。
[0090]
实验结果参见图10,其中上排的1、2、3、4图是脑组织冠状切片后,某一个切面的具体脑区展示,从1到4分别是后扣带回皮层(retro-splenial granular cortex,rsg),感觉皮层(sensory cortex,s),纹状体(striatum,str),以及位于小脑的三叉神经束。呈现绿色荧光的区域为g蛋白缺陷型的rv示踪病毒工具sad-b19
‑△
g-gfp标记的神经元;绿色荧光区
域外周的部分则呈现蓝色荧光,是dapi(4’,6-diamidino-2
’‑
phenylindole,4’,6-联脒-2
’‑
苯基吲哚)标记的细胞核。下排的5-8对应1-4中单纯的绿色荧光标记。
[0091]
以上所述实施例仅是为充分说明本发明而所举的较佳的实施例,本发明的保护范围不限于此。本技术领域的技术人员在本发明基础上所作的等同替代或变换,均在本发明的保护范围之内。本发明的保护范围以权利要求书为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1