与陆地棉亚麻酸关联的SNP分子标记及其应用
与陆地棉亚麻酸关联的snp分子标记及其应用
技术领域
1.本发明涉及与陆地棉亚麻酸关联的snp分子标记及其应用,属于分子生物学、生物信息学领域。
背景技术:
2.植物油是一种重要的资源,是膳食营养得来源之一,也是生物燃料和润滑剂、动物饲料等产品的重要原材料。棉花(gossypium spp.)不仅是一种重要的纤维作物,同时还是重要的食用油料作物,是全球第六大植物油料来源,在保障我国食用油安全方面具有重要的作用。近年来人们发现亚麻酸对于心脑血管疾病有着很好的疗效,由于亚麻酸存在于特定水生生物和植物中,人们摄入的并不多,使得亚麻酸的开发成为了一个新的热点。而在棉花的研究及生产应用过程中往往只重视产量和品质的提升,忽视棉籽中亚麻酸相关的研究,导致棉籽油的产量和品质低下,市场认可度不高。因此,开展棉花亚麻酸的研究,对于改良棉籽油的营养成分比例和商品货架期具有重要的意义。
3.全基因组关联分析(genome-wide association study,gwas)是一种对多个个体在全基因组范围的遗传变异多态性与目标性状表型的多样性结合起来分析,可直接鉴定出与表型变异密切相关且具有特定功能的基因位点或标记位点。在全基因组范围内进行整体研究,能够一次性对优良性状进行轮廓性概览,适用于挖掘优良性状等的研究。
4.分子标记技术可以快速找到和亚麻酸含量qtl紧密连锁的标记,通过分子标记对亚麻酸含量进行辅助选择,可以加快棉籽油品质的育种进程。研究人员采用slaf、ssr等分子标记技术对棉亚麻酸候选基因初定位,发现候选基因与分子标记的遗传距离普遍较远,说明亚麻酸形成分子机制非常复杂,有待于深入研究和探索,充分挖掘和利用这些控制亚麻酸的基因,将丰富纤维品质改良的基因资源,为培育出满足各种需求的棉花新品种提供重要基础。
5.近年来,随着高通量dna测序技术的迅猛发展,本发明人成功完成了1279份棉花nam群体资源的重测序。通过生物信息学进行数据分析比对,获得大量高质量的snps,这些snps可用于单体型图谱、遗传图谱、关联性图谱、指纹图谱的构建,为分子育种、系统进化、种质资源鉴定提供重要保障。本发明利用全基因组关联分析发掘了与陆地棉亚麻酸关联的一批snp分子标记,为分子标记辅助选择及聚合育种改良棉籽油品质奠定了基础。
技术实现要素:
6.针对现有技术的不足,本发明的目的是提供一批与陆地棉亚麻酸关联的snp分子标记及其应用。
7.为了实现上述目的,本发明的技术方案是:
8.与陆地棉亚麻酸关联的snp分子标记,所述snp分子标记为如seq id no.1-seq id no.28所示核苷酸序列中至少一个。
9.所述snp分子位点于序列第51bp处发生突变,所述snp分子标记的突变形式如下所
示:
[0010][0011]
一种所述snp分子标记在棉花亚麻酸含量早期预测和筛选中的应用。
[0012]
一种所述snp分子标记在高产棉花分子标记辅助育种中的应用。
[0013]
一种所述snp分子标记在棉花种质资源改良中的应用。
[0014]
一种所述snp分子标记在鉴定高产棉花品种中的应用。
[0015]
一种检测所述snp分子标记的引物或试剂。
[0016]
一种检测所述snp分子标记的试剂盒。
[0017]
一种含有所述snp分子标记的基因芯片。
[0018]
一种利用所述snp分子标记分析陆地棉亚麻酸的方法,包括以下步骤:
[0019]
(1)提取待测样品的基因组dna;
[0020]
(2)以提取的dna为模板,根据snp分子标记设计引物,分别进行pcr扩增;
[0021]
(3)根据pcr扩增产物,分析陆地棉亚麻酸含量。
[0022]
本发明的有益效果:
[0023]
本发明提供的与陆地棉亚麻酸关联的snp分子标记可以用于棉花亚麻酸性状的早期预测和筛选。其直接以dna的形式表现,在棉花的各个组织、各个发育阶段均可检测到,不
受季节、环境限制、不存在表达与否等问题;表现为中性,不影响目标性状的表达;snp适于快速、规模化筛查。基因组筛选中snps往往只需+/-的分析,而不用分析片段的长度,利于发展自动化技术筛选或检测snps。
具体实施方式
[0024]
以下结合实施例对本发明的具体实施方式作进一步详细说明。
[0025]
实施例1:snp分子标记的获得
[0026]
(1)亚麻酸测定:
[0027]
群体于2017、2018年进行1点2重复试验。1271个子代和8个亲本(3个母本、5个父本)在亚群体内及亚群体间进行随机排列。亚群体内随机加入该亚群体的双亲。群体整体设置3个对照,分别为该群体的母本中植棉3号、鲁棉研28、锦科178。3个对照在群体中每隔15个材料依次出现,并最终均匀覆盖到整个群体中。试验点为:河南安阳(ay)。试验点按单行区、2m行长种植,每行株数在10-30株之间(具体依照当地栽培模式),取样时间为9月20日至10月20日不等(具体依照当地霜期及栽培模式),除两端2株外,每小区取其余株中部靠主茎铃,每株取1-2个铃不等,共取20铃。利用本实验室的气相色谱仪(gc-2030,shimadzu,japan)对收获的各小区棉籽亚麻酸成分含量进行测定。亚麻酸成分含量测定采用沸腾的石油醚萃取,koh-甲醇溶液进行甲酯化处理。色谱柱选用sh-rtx-65(30m
×
0.25mm
×
0.50μm),检测器为fid(氢火焰离子化检侧器)。高纯度氮气(99.999%)作为载气,进样体积为1μl(包含正己烷溶剂和种子脂肪酸甲酯样品),分流比为39:1。升温程序设定为:(1)维持100℃柱温、保留时间为1min,(2)100℃升温到210℃,升温速率为4℃/min,(3)维持210℃柱温、保留时间为4min,(4)210℃升温到230℃,升温速率为4℃/min,(5)维持230℃柱温、保留时间为5min。试验中最高测定温度设定为280℃,进样口温度设定为250℃。
[0028]
(2)snp的检测:
[0029]
共采集陆地棉样本1279份进行基因组重测序。采集样品时,将各品系的种子播种在培养箱中,采集棉株幼叶。用ctab法提取所有样本高质量的棉花基因组dna各5μg。上述提取的基因组dna送到深圳华大基因科技有限公司,用于基因组重测序。测序获得高质量的clean data,数据量为20.47tb,亲本平均测序深度35x,子代平均测序深度4x以上。以优质四倍体棉(g.hirsutum’texas marker 1’)基因组为参考基因组进行序列定位。在映射之前,将所有未组装的contigs连接到一个伪染色体(命名为“chrun”)。使用bwa(v.0.7.12)软件分别将1279份样本的短序列映射到参考基因组,剔除所有未对齐的读段和低质量(映射质量小于20)的读段。然后,采用gatk unifiedgenotyper(v.3.8.0)分别对每个样本进行变异鉴定,并将所有样本(n=1279)的变异文件合并为一个总的vcf文件。最后,分别鉴定出11,856,129个和4,543,742个高品质snp和indel。基于次等位基因频率大于0.05和缺失率小于0.2,使用vcftools进一步过滤变异位点筛选出高质量的snp和indel分别1,855,955个和l,309,084个,用作后续的全基因组关联分析。所有变异的影响由annovar进行注解。
[0030]
(3)陆地棉亚麻酸性状全基因组关联分析:
[0031]
陆地棉亚麻酸性状全基因组扫描(gwas)定位,对步骤(1)所得的陆地棉亚麻酸性状结果和步骤(2)所得的基因型数据,采用(efficient mixed-model association expedited)(emmax)统计分析软件的混合线性模型进行统计分析,具体可参考(http://
csg.sph.umich.edu/kang/emmax/download/index.html)。统计模型为:
[0032]
y=xα+zβ+wμ+e
[0033]
y为表型性状,x为固定效应的指示矩阵,α为固定效应的估计参数;z为snp的指示矩阵,β为snp的效应;w为随机效应的指示矩阵,μ为预测的随机个体,e是随机残差,服从e~(0,δ
e2
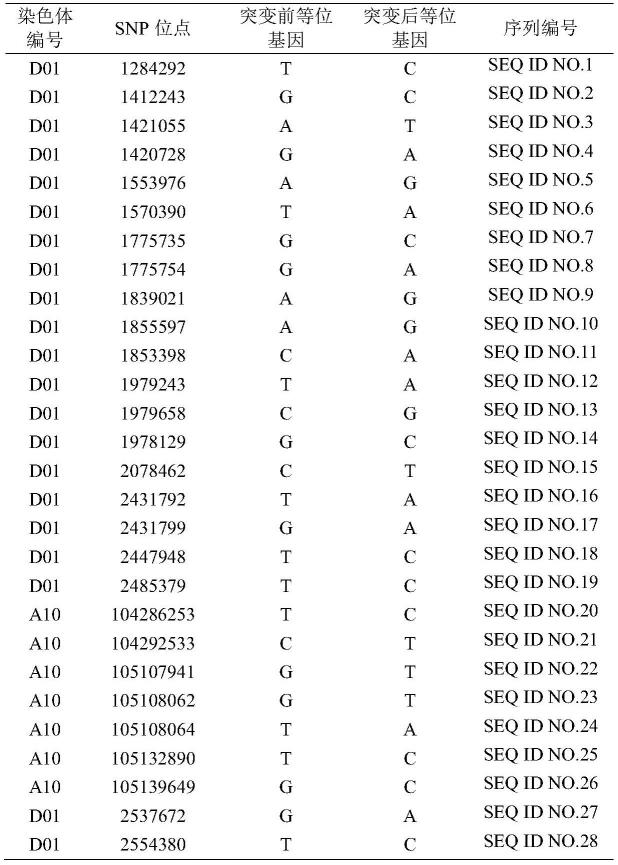
)。该模型中,通过在μ中加入亲缘关系矩阵来校正群体分析。分析发现共计有28个snps与陆地棉亚麻酸性状显著相关,所述snp标记的等位基因位点信息如表1所示。参考序列为陆地棉栽培品种tm-1,参考基因组版本号g.hirsutum_tm-1_icr(http://grand.cricaas.com.cn/page/download/download)。这些snp位点上下游50bp的核苷酸序列如seq id no.1-seq id no.28所示。
[0034]
表1与陆地棉亚麻酸关联的snp分子标记
[0035][0036][0037]
(4)验证:利用1279份棉花多亲本群体2年5点共计10个环境下的亚麻酸含量blup值(最佳线性无偏预测值)对上述snp的效应进行了验证,结果显示100%的snp表现出对陆地棉亚麻酸含量性状变异具有显著的影响。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1