基于差分进化的谷氨酰胺太赫兹谱波长选择方法及装置与流程
本发明涉及一种基于差分进化的谷氨酰胺太赫兹谱波长选择方法及装置,属于太赫兹光谱检测技术领域。
背景技术:
在对谷氨酰胺样品进行太赫兹吸收谱定量分析中,通过实验得到的谷氨酰胺样品的原始太赫兹吸收谱通常涵盖一段较宽的频段,包含大量的波长点数据,其中不仅包括信噪比较高的有用数据,也包含信噪比较低的噪声数据以及不属于任一组分特征的冗余数据,若直接将原始吸收谱用于定量分析势必导致较高误差,因此需要进行适当选择。由于吸收谱是由一系列波长点数据组成的,对吸收谱数据的选择实际上就是对波长的选择,因而在光谱学中被定义为波长选择(Wavelength selection)。对于太赫兹光谱定量分析领域而言,波长选择对定量分析的准确度至关重要,若选择不恰当,会导致较大误差。但是目前在太赫兹光谱定量分析中,波长选择常用的做法是人为地依据经验从原始光谱中选取某一波段数据用于定量计算,而对太赫兹光谱波长选择的机理及方法缺乏系统性的深入研究。
中国计量学院的王强教授等人分别利用偏最小二乘法(partial least squares,PLS)、区间偏最小二乘法(interval PLS,iPLS)、向后区间偏最小二乘法(backward iPLS,biPLS)以及移动窗口偏最小二乘法(moving window PLS,mwPLS)对噻苯咪唑位于0.3-1.6THz频段内的太赫兹特征光谱进行了波长选择,并对四种算法的性能进行了细致的比较。桂林电子科技大学的陈涛等人就太赫兹光谱定量分析中的特征谱区筛选进行了相关研究。除上述王强等人提出的波长选择方法外,又采用了联合区间偏最小二乘法(siPLS)并进行了一系列对比。但是基于偏最小二乘的波长选择方法,是通过将原始光谱分割成若干区间加以筛选,难免会将部分无意义数据含入其中,甚至将一些有意义数据错误地抛弃,以至于所选择的波长不合适,从而导致定量分析的误差比较大。
技术实现要素:
本发明的目的是提供一种基于差分进化的谷氨酰胺太赫兹谱波长选择方法,以解决目前谷氨酰胺太赫兹吸收谱波长选择不合适而导致定量分析的误差比较大的问题。同时本发明还提供了一种基于差分进化的谷氨酰胺太赫兹谱波长选择装置。
本发明为解决上述技术问题而提供一种基于差分进化的谷氨酰胺太赫兹谱波长选择方法,该选择方法包括以下步骤:
1)随机生成一个大小为S的初始种群X;
2)对初始种群X进行基于差分的变异操作,以得到变异种群V,并对变异种群V进行交叉操作,以得到交叉种群U;
3)分别利用初始种群X和交叉种群U从谷氨酰胺样品的太赫兹吸收谱中进行选取,以得到初始种群X和交叉种群U中每个个体相对应的经过波长选择的谷氨酰胺样品的重构太赫兹吸收谱;
4)构建适应度函数,利用所构造的适应度函数分别计算初始种群X和交叉种群U中每个个体的适应度;
5)对初始种群X和交叉种群U中相对应个体的适应度值进行比较,将适应度值较大的个体保留下来,从而得到新一代种群Xnext;
6)将新一代种群Xnext作为新的初始种群,重复步骤2)-5),直至进化代数达到设定阈值,并将最终代种群中适应度值最高的个体作为所选择的谷氨酰胺太赫兹吸收谱波长的最优解。
进一步地,所述步骤2)中的变异操作如下::
其中Vi,j表示变异种群V中第i个个体的第j个二进制元素,Xi,a、Xi,b和Xi,c分别表示初始种群X中第i个个体的第a,b和c个二进制元素,并且a,b和c是3个互不相同的正整数。
进一步地,所述步骤2)中的交叉操作为:
其中Ui,j表示交叉种群U中第i个个体的第j个二进制元素,rand为0至1区间内的随机数,CR为预先设定的交叉概率,jrand为1至L区间内的随机整数,Xi,j表示初始种群X中第i个个体的第j个二进制元素。
进一步地,所述步骤1)中的初始种群X由S个长度为L的二进制字符串组成,该二进制字符串与谷氨酰胺样品的太赫兹吸收谱中的L个频率点一一对应。
进一步地,所述步骤3)中的波长选择过程如下:
对于初始种群X中的各个个体,若其某个二进制元素为“1”,则对应谷氨酰胺太赫兹吸收谱的频率点数据被保留,否则该频率点数据则被抛弃,将所有保留下的频率点数据整合在一起,组成经过波长选择的谷氨酰胺样品的重构太赫兹吸收谱;对于交叉种群U中的各个个体,若其某个二进制元素为“1”,则对应谷氨酰胺太赫兹吸收谱的频率点数据被保留,否则该频率点数据则被抛弃,将所有保留下的频率点数据整合在一起,组成经过波长选择的谷氨酰胺样品的重构太赫兹吸收谱。
进一步地,所述步骤4)中构建的适应度函数为:
其中F是适应度值,m是校正集中谷氨酰胺样品的总数量,qe是每个谷氨酰胺样品对应的定量分析误差,n代表校正集中谷氨酰胺样品的某一个,其中ccal和creal分别是谷氨酰胺样品的计算浓度和真实浓度。
进一步地,所述新一代种群Xnext满足下式
其中Xinext为新一代种群Xnext的第i个个体,F(Ui)为交叉种群U中第i个个体的适应度值,F(Xi)为初始种群X中第i个个体的适应度值。
本发明还提供了一种基于差分进化的谷氨酰胺太赫兹谱波长选择装置,该选择装置包括初始种群生成模块、变异交叉操作模块、太赫兹吸收谱重构模块、适应度计算模块、比较模块和选择模块,
所述初始种群生成模块用于随机生成一个大小为S的初始种群X;
所述的变异交叉操作模块用于对初始种群X进行基于差分的变异操作,以得到变异种群V,并对变异种群V进行交叉操作,以得到交叉种群U;
所述的太赫兹吸收谱重构模块用于分别利用初始种群X和交叉种群U从谷氨酰胺样品的太赫兹吸收谱中进行选取,以得到初始种群X和交叉种群U中每个个体相对应的经过波长选择的谷氨酰胺样品的重构太赫兹吸收谱;
所述的适应度计算模块用于构建适应度函数,并利用所构造的适应度函数分别计算初始种群X和交叉种群U中每个个体的适应度;
所述的比较模块用于对初始种群X和交叉种群U中相对应个体的适应度值进行比较,将适应度值较大的个体保留下来,从而得到新一代种群Xnext;
所述的选择模块用于将新一代种群Xnext作为新的初始种群,重复执行变异交叉操作模块、选取模块、适应度计算模块和比较模块,直至进化代数达到设定阈值,并将最终代种群中适应度值最高的个体作为所选择的谷氨酰胺太赫兹吸收谱波长的最优解。
进一步地,所述变异交叉操作模块所采用变异操作如下:
其中Vi,j表示变异种群V中第i个个体的第j个二进制元素,Xi,a、Xi,b和Xi,c分别表示初始种群X中第i个个体的第a,b和c个二进制元素,并且a,b和c是3个互不相同的正整数。
进一步地,所述变异交叉操作模块所采用交叉操作如下:
其中Ui,j表示交叉种群U中第i个个体的第j个二进制元素,rand为0至1区间内的随机数,CR为预先设定的交叉概率,jrand为1至L区间内的随机整数,Xi,j表示初始种群X中第i个个体的第j个二进制元素。
本发明的有益效果是:本发明首先对初始种群X进行基于差分的变异操作和交叉操作,以得到交叉种群U;然后分别利用初始种群X和交叉种群U从谷氨酰胺样品的太赫兹吸收谱中进行选取,利用所构造的适应度函数分别计算初始种群X和交叉种群U中每个个体的适应度;并将适应度值较大的个体保留下来,从而得到新一代种群Xnext;最后将新一代种群Xnext作为新的初始种群进行进化迭代,直至进化代数达到设定阈值,并将最终代种群中适应度值最高的个体作为所选择的谷氨酰胺太赫兹吸收谱波长的最优解。本发明通过对谷氨酰胺样品的太赫兹吸收谱进行逐点深度选择,挑选有用信息,从而显著提高了定量分析的精度,取得了良好的应用效果。
附图说明
图1是基于差分进化的谷氨酰胺太赫兹谱波长选择方法的流程图;
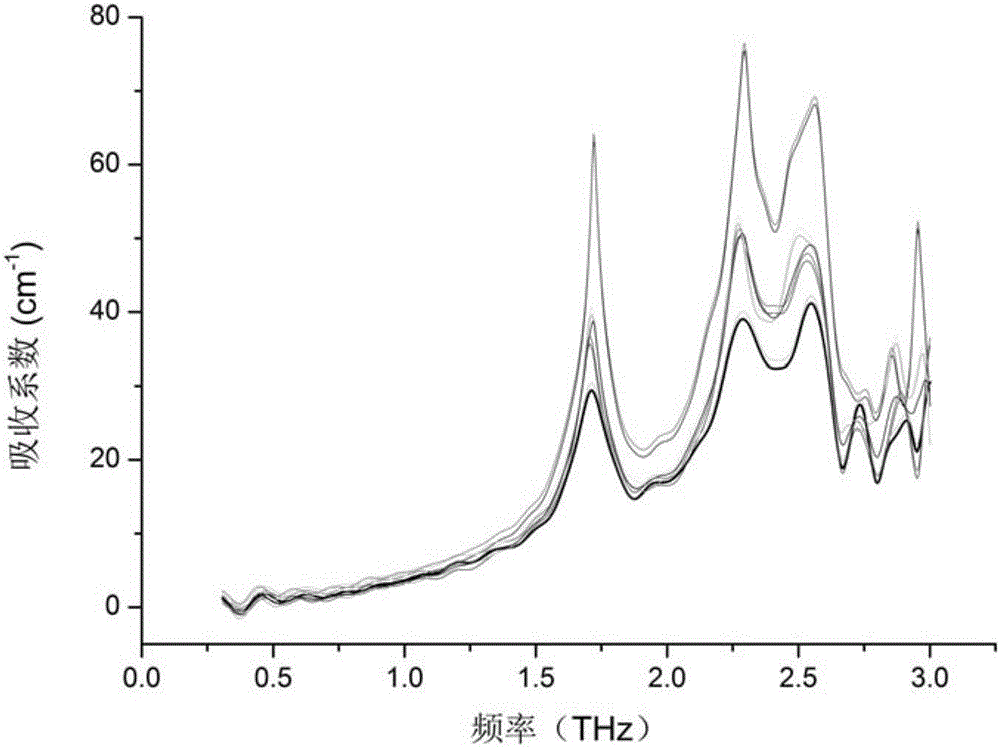
图2是未经波长选择的谷氨酰胺样品的太赫兹吸收谱图;
图3是波长选择后的重构谷氨酰胺太赫兹吸收谱图。
具体实施方式
下面结合附图对本发明的具体实施方式做进一步的说明。
本发明基于差分进化的谷氨酰胺太赫兹谱波长选择方法的实施例
本发明首先对初始种群X进行基于差分的变异操作和交叉操作,以得到交叉种群U;然后分别利用初始种群X和交叉种群U从谷氨酰胺样品的太赫兹吸收谱中进行选取,利用所构造的适应度函数分别计算初始种群X和交叉种群U中每个个体的适应度;并将适应度值较大的个体保留下来,从而得到新一代种群Xnext;最后将新一代种群Xnext作为新的初始种群进行进化迭代,直至进化代数达到设定阈值,并将最终代种群中适应度值最高的个体作为所选择的谷氨酰胺太赫兹吸收谱波长的最优解。该方法的流程如图1所示,具体实施过程如下。
1.随机生成一个大小为S的初始种群X。
本实施例中生成的初始种群X由S个长度为L的二进制字符串组成,该二进制字符串与谷氨酰胺样品的太赫兹吸收谱中的L个频率点一一对应。
2.对初始种群x执行基于差分思想的变异操作,从而得到变异种群V。
本实施例中的变异操作后得到的种群V为:
其中Vi,j表示变异种群V中第i个个体的第j个二进制元素,Xi,a、Xi,b和Xi,c分别表示初始种群X中第i个个体的第a,b和c个二进制元素,并且a,b和c是3个互不相同的正整数。
3.对变异种群进行交叉操作,得到交叉种群U。
交叉操作得到的交叉种群U为:
其中Ui,j表示交叉种群U中第i个个体的第j个二进制元素,rand为0至1区间内的随机数,CR为预先设定的交叉概率,jrand为1至L区间内的随机整数,Xi,j表示初始种群X中第i个个体的第j个二进制元素。
4.分别利用初始种群X和交叉种群U中的个体对谷氨酰胺太赫兹吸收谱进行波长选择,得到重构吸收谱。
波长选择通过以下方式进行,对于初始种群X中的各个个体,若其某个二进制元素为“1”,则对应谷氨酰胺太赫兹吸收谱的频率点数据被保留,否则该频率点数据则被抛弃,然后将所有保留下的频率点数据整合在一起,组成经过波长选择的谷氨酰胺样品的重构太赫兹吸收谱;;对于交叉种群U中的各个个体,若其某个二进制元素为“1”,则对应谷氨酰胺太赫兹吸收谱的频率点数据被保留,否则该频率点数据则被抛弃,然后将所有保留下的频率点数据整合在一起,组成经过波长选择的谷氨酰胺样品的重构太赫兹吸收谱。
5.构建适应度函数F。
构建的适应度函数用下列公式表示:
其中F是适应度值,m是校正集中谷氨酰胺样品的总数量(校正集是由若干个成分浓度信息已知的谷氨酰胺样品组成的),qe是每个谷氨酰胺样品对应的定量分析误差,n代表校正集中谷氨酰胺样品的某一个:
其中ccal和creal分别是谷氨酰胺样品的计算浓度和真实浓度;谷氨酰胺样品的计算浓度ccal是利用经过波长选择后的重构太赫兹吸收谱,通过最小二乘线性回归得到,谷氨酰胺样品的真实浓度creal是预先配制的。
6.利用构建的适应度函数F分别对初始种群X和交叉种群U中的个体进行评价,得到它们各自的适应度值。
这里的适应度值是分别利用初始种群X和交叉种群U中的各个个体对谷氨酰胺样品的太赫兹吸收谱进行波长选择,而后通过适应度函数计算种群中各个个体的适应度值。
7.对初始种群X和交叉种群U中相对应个体的适应度值进行比较,将适应度值较大的个体保留下来,从而得到新一代种群Xnext。
本实施例中新一代种群Xnext需满足下式:
其中Xinext为新一代种群Xnext的第i个个体,F(Ui)为交叉种群U中第i个个体的适应度值,F(Xi)为初始种群X中第i个个体的适应度值。
8.将新一代种群Xnext作为新的初始种群,重复步骤2至7,直至进化代数达到上限G,终止进化,并将最终代种群中适应度值最高的个体作为问题的最优解输出。
本发明基于差分进化的谷氨酰胺太赫兹谱波长选择装置的实施例
本实施例中的选择装置包括初始种群生成模块、变异交叉操作模块、太赫兹吸收谱重构模块、适应度计算模块、比较模块和选择模块;初始种群生成模块用于随机生成一个大小为S的初始种群X;变异交叉操作模块用于对初始种群X进行基于差分的变异操作,以得到变异种群V,并对变异种群V进行交叉操作,以得到交叉种群U;太赫兹吸收谱重构模块用于分别利用初始种群X和交叉种群U从谷氨酰胺样品的太赫兹吸收谱中进行选取,以得到初始种群X和交叉种群U中每个个体相对应的经过波长选择的谷氨酰胺样品的重构太赫兹吸收谱;适应度计算模块用于构建适应度函数,并利用所构造的适应度函数分别计算初始种群X和交叉种群U中每个个体的适应度;比较模块用于对初始种群X和交叉种群U中相对应个体的适应度值进行比较,将适应度值较大的个体保留下来,从而得到新一代种群Xnext;选择模块用于将新一代种群Xnext作为新的初始种群,重复执行变异交叉操作模块、选取模块、适应度计算模块和比较模块,直至进化代数达到设定阈值,并将最终代种群中适应度值最高的个体作为所选择的谷氨酰胺太赫兹吸收谱波长的最优解。
这里的波长选择装置可以采用单片机、DSP、PLC或MCU等实现,波长选择装置执行有上述六个模块,这里的模块可以位于RAM存储器、闪存、ROM存储器、EPROM存储器、EEPROM存储器、寄存器、硬盘、移动磁盘、CD-ROM或者本领域已知的任何其他形式的存储介质,可以将该存储介质耦接至波长选择装置,使波长选择装置能够从该存储介质读取信息,或者该存储介质可以是波长选择装置的组成部分。各模块的具体实现手段已在方法的实施例中进行了详细说明,这里不再赘述。
为了验证本发明的效果,下面设计了一系列定量分析的实验。实验选取了9个不同含量的谷氨酰胺样品的太赫兹吸收谱(其中前6个为校正集,后3个为验证集),分别利用不经选择的谷氨酰胺全吸收谱以及经过本发明提出的波长选择方法选择后的谷氨酰胺重构太赫兹吸收谱对谷氨酰胺样品进行定量分析,谷氨酰胺样品含量以及定量分析的误差如表1所示。本实验中,谷氨酰胺样品(具体包括谷氨酸和谷氨酰胺)的原始太赫兹吸收谱范围为0.3-3THz,分辨率约为4.5GHz,共有590个频率点,所以种群中二进制字符串个体的长度为590,种群大小为100,交叉概率为0.9,进化代数上限为30000。
表1
未经波长选择的谷氨酰胺样品的太赫兹吸收谱如图2所示,经本发明波长选择后的重构谷氨酰胺太赫兹吸收谱如图3所示,可见利用本发明提出的波长选择方法,能够有效降低对谷氨酰胺样品太赫兹吸收谱进行定量分析的误差,误差大致在3%以下,取得了优异的效果。
- 还没有人留言评论。精彩留言会获得点赞!