基于定量生物标志物的癌症诊断方法及其数据库与流程
基于定量生物标志物的癌症诊断方法及其数据库
相关申请的交叉引用
1.本技术主张基于2019年11月1号递交的美国临时专利申请62/929,396的所有权益并且在此将该公开的全部内容通过参照而并入本技术中。
技术领域
2.本发明涉及基于一组生物标志物(biomarker)的定量水平对癌症患者进行诊断(diagnostics)、预估(prediction)和预后(prognosis)的方法、系统和软件。具体而言,本发明涉及参照数据库中同一组生物标志物的定量水平对癌症患者进行诊断、预估和预后。
背景技术:
3.对于大多数癌症患者来说,它们的肿瘤组织经手术移除后会以福尔马林固定-石蜡包埋(ffpe)的形式保存在医院或其它医疗机构中。因此,储存的数百万ffpe样本以及与之相关联的包括治疗方案和临床结局的详细病例,作为数量庞大且尚未得到充分利用的医疗资源积累起来。这些ffpe样本的巨大数量足以涵盖个体水平上的不同分子特征(molecular identity)。结合其已知的临床结局,这些样本成为面向临床个性化治疗研究的无与伦比的资源。在这里,我们可以为世界上任何一个癌症患者识别出具有相似分子特征的已知病例。
4.在临床实践中,出于临床诊断和预后的目的,免疫组化分析(ihc)被广泛用于在蛋白质水平上评估生物标志物。典型的生物标志物的ihc报告表述为“+”或
“‑”
或者进而分类成“0、1+、2+、3+”。例如,通过ihc方法分析常用于乳腺癌诊断的生物标志物——人表皮生长因子受体2(her2)的表达水平,以确定是否在治疗过程中包括her2针对性疗法(her2-dependent therapy)。ihc的结果被分成三组:0和1+、2+和3+。0组和1+组的结果被认为是阴性,3+组被认为是阳性,而2+组被认为是两可。
5.结合ihc结果进行诊断和预后是临床实践中的常规操作。例如,对于乳腺癌患者来说,4个肿瘤生物标志物,包括雌激素受体(er)、孕激素受体(pr)、ki67和her2被用于将患者区分为管腔a(luminal a)型、管腔b(lumina b)型、her2型和三阴型4个亚型。her2和er/pr的ihc结果被用于将患者分为管腔型、her2型和三阴型,而ki67的表达水平进一步将管腔型分为管腔a型和管腔b型。
6.ihc的分类结果也难以用于进一步临床实践。例如,虽然在阳性的个体患者中存在显著差异,它们在临床实践中被归结为同一类别。因而,来自ihc分析的结果难以用来进行充分的数据分析以提供更准确、更有预见性的诊断和预后。
7.ihc方法也严重受制于内在的主观性和不一致性。肿瘤组织的异质性也使诊断过程更为复杂化。
8.很多科学家致力于在组织水平将生物标志物测定为绝对的且连续的变量。例如,酶联免疫法(elisa)可以用来测定新鲜和冰冻组织中生物标志物的表达水平。然而,这种方法无法用于测定ffpe样本中生物标志物的表达水平。因此也严重限制了其在临床诊断和预
后中的应用。
9.量化斑点免疫印迹分析(qdb)法能够以高通量的形式测定新鲜、冰冻和ffpe样本中的生物标志物表达水平作为绝对的且连续的变量。当引入标准蛋白时,不管以重组蛋白形式还是以纯化蛋白形式,都可以将这种方法非常方便地转化为绝对定量的方法以对某特定蛋白的含量在细胞或组织水平上进行绝对定量测定。
10.在qdb方法应用之前,数百万计的储存ffpe样本无法通过现有可用的蛋白技术进行处理,因为现有技术无法在群体水平上有效区分单个ffpe样本。目前常用的蛋白分析方法,包括免疫组化(ihc)、西式印迹分析、反相蛋白质阵列分析(rppa)、以及质谱分析均已用于分析ffpe样本。然而,这些方法都不足以用于分析数量庞大的ffpe样本。
11.对于ihc和西式印迹分析来说,它们的定性的内在特征会使群体水平上的个体间差异变得模糊。
12.其它方法能够定量测定蛋白质表达水平以揭示群体水平上的个体差异,但提供了相对结果从而限制研究规模。我们可以通过举例来更好地说明这种限制。蛋白质的表达水平可以表示为绝对值(例如nmole/g)或者表示为相对值(参考蛋白质b的百分比%)。虽然可以很容易在多个(本文中的“多个(a plurality of)”均特指“两个以上”)分析中比较以绝对形式表达的蛋白含量,但很难比较在多个分析中比较基于各不相同的参考蛋白b含量的结果。
13.ms和rppa都属于这种情形。其结果都表述为相对于一个参考蛋白质的值,而该参考蛋白质的含量在每次研究中都可能各不相同[boellner,et al,microarrays,4(2):98-114,2015,desouza,et al,clin.biochem.46:421-431,2013]。因此,基于这些方法的研究规模也就受限于每次研究中样本的数量,无法通过合并其它研究结果来扩大研究规模。实时荧光定量pcr(rt-pcr)方法产生的数据集也存在同样的问题。
[0014]
另一方面,qdb方法可以提供一种能处理庞大的具有绝对性和连续性的ffpe样本数据集的方法。关于连续性,需要量化的结果来体现在群体水平上个体间的细微差异。关于绝对性,无论时间、地点等如何变化,每个蛋白的定量应该一致,由此确保数据可以共享、相互印证及合并以保证数据集的急需增长来应对数量庞大的ffpe样本。
[0015]
本发明提供了方法、系统和软件以利用世界上广泛储存的ffpe样本来辅助患者的诊断。在临床中采用该方法可以显著提高治疗的有效性以实现个性化治疗的目的。
技术实现要素:
[0016]
本发明提供了利用作为连续变量的三个或者更多的生物标志物对癌症进行诊断、预估和预后的方法。本发明对生物标志物的评估是通过定量而不是现行的通用定性的测量,然后表达为绝对单元从而可以很容易合并到现有的数据库中。
[0017]
实验样品可以是来自个体(subject)的组织。在本发明的一实施方式中,组织是指活检组织。在本发明的另一实施方式中,组织是指经过福尔马林固定
·
石蜡包埋的样本(ffpe样本)。
[0018]
个体可以是患者。更具体而言,个体可以是癌症患者。在本发明一实施方式中,个体可以是乳腺癌患者。
[0019]
利用绝对定量的蛋白生物标志物含量可以用来开发一个回顾性癌症概况
(profile)数据库(rc)或者更准确地说不同癌症类型的数据库(包括乳腺癌、结直肠癌或者前列腺癌)以充分利用巨量的储存的ffpe样品(在本文中的“癌症概况”对应英文表达为“cancer profile”,有时单独称其为“癌症概况信息”或者根据语境简称为“概况信息”或“信息”)。
[0020]
通过组合多个绝对定量的蛋白标志物,足以从数百万储存的ffpe样本中区分单个ffpe样本。在某种意义上,这些蛋白标志物的组合成为数据库中每一个ffpe样本独特的“指纹”。
[0021]
以这个独特的“指纹”为核心,可以结合与之匹配的临床记录,包括传统临床病理参数、治疗方案和相应的临床结局,为每一个ffpe样本提供全方位的信息。
[0022]
来自这些不同方面的所有信息构成了数据库中每个ffpe样本的个体癌症概况信息(icp)。任何其它临床相关的性状表现(trait)均可包括在这些癌症概况信息中。例如,包括单核苷酸变异(snv)、染色体错位以及各种基因预测分析的评分在内的遗传信息均可以包括在癌症概况信息中。
[0023]
数据库的绝对特征保证数据库可以不断成长。虽然icp的来源不同,但基于其数据的绝对特征可以有效结合在一起。新的癌症简况也可以随时间不断增加、填补。在未来可预期该数据库会容纳相当多的储存的ffpe样品的简况来支持基于“大数据”的临床诊断。
[0024]
此外,上述方法进一步还包括构建一个回顾性癌症(rc)数据库以提供癌症的诊断、预估和预后的方法,该方法包括:提供患有已知临床结局的癌症的多个个体;为所述多个个体中的每个生成icp,其中所述icp包括:i)经过绝对定量测定的多个蛋白标志物、以及ii)癌症的已知临床结局,以及将生成的所述多个个体的icp存储于数据库中。
[0025]
在本发明的一实施方式中,生物标志物的表达水平可以被测定为绝对的且连续的变量。
[0026]
在本发明的一实施方式中,生物标志物的表达水平可以通过质谱分析法进行测定。
[0027]
在本发明的一实施方式中,生物标志物的表达水平可以通过酶联免疫法(elisa)进行测定。
[0028]
在本发明的一实施方式中,生物标志物的表达水平可以通过量化斑点免疫印迹分析(qdb)法进行测定。
[0029]
在本发明的一实施方式中,三个以上(包括三个)的生物标志物的蛋白表达水平可以通过elisa、qdb和质谱分析任意组合的方法进行测定。
[0030]
数据库中icp的生物标志物定量表达量水平可以与其相关的临床信息结合以用于医学用途的数学分析。例如可以探索生物标志物的绝对量与无病生存期(dfs)的潜在关系,为患者提供预测性的临床预后。
[0031]
在本发明的一实施方式中,来自icp的以连续变量形式表示的多种生物标志物的量可以与相关临床信息(包括但不限于无病生存期、总生存期(overall survival:os)、副作用、年龄、疾病的不同发展期)结合以便寻找潜在的因果关系,并且该因果关系可以用于癌症诊断和预后的目的。
[0032]
在本发明的一实施方式中,来自icp的三个以上的生物标志物的绝对量可以作为(x,y,z)的坐标,将个体定位在由x、y、z轴决定的空间里(定位点)。样品的定位点与相关临
床信息(包括但不限于无病生存期、总生存期、副作用、年龄、疾病的不同发展期)结合以寻找空间相关性,并且该相关性可以用于诊断和预后的目的。
[0033]
在本发明的一实施方式中,在x、y、z轴决定的空间中多于一个icp的定位点可以被分成与临床诊断、预估和预后相关的临床亚群。
[0034]
本发明的另一方面涉及一种参考数据库,该数据库用于根据患者活检样品中多于一种标志物的定量分析来用于癌症的诊断。参考数据库包括多个icp,其中每个icp均通过以下步骤构建:(a)从癌症患者中获得已知临床诊断的活检样品;(b)在活检样品中测定三个以上的所述生物标志物水平作为绝对的且连续的变量;(c)利用三种生物标志物水平作为x、y、z坐标点,在空间中定位每个icp(定位点);(d)将每个icp按照定位点与该癌症患者已知的临床诊断、预估和预后相关联,从而基于空间定位获得参考概况信息(reference profile)。
[0035]
另一方面,本发明提供了对患者进行癌症诊断的方法。该方法包括以下步骤:(i)提供上述参考空间数据库;(ii)从患者中获得活检样品;(iii)对所述活检样品中三种生物标志物进行测定,其结果表达为绝对的且连续的变量,测定结果是所述活检样品中三种标志物的连续变量;(iv)将所述三种生物标志物水平分别作为(x,y,z)坐标将样品定位于参考空间数据库中;以及(v)在参考空间数据库中识别与患者样品最匹配的参考空间概况信息,输出与识别的参考概况信息相关联的已知临床诊断。
[0036]
本发明的另一方面涉及一种用于提供癌症的诊断、预估或预后的数据库,所述数据库含有多个由已知癌症临床结局的个体生成的icp。此外,icp包括:i)从来自储存的个体的ffpe样本的定量测定的多个临床参数,以及ii)癌症的已知临床结局。此外,多个临床参数中的每个临床参数代表生物标志物的定量测定结果。此外,定量测定结果是连续的而且是样本中的生物标志物的绝对的量。
[0037]
本发明的另一方面涉及提供患者癌症的诊断、预估或预后的方法。该方法包括:1)采集患者的ffpe样本;2)从上述数据库中获得i)存储的icp以及ii)数据库中使用的临床参数集(即一组临床参数);3)将数据库中每个icp中所述临床参数集的定量水平与患者的ffpe样本中的同一临床参数集的定量水平进行比较;4)通过比较来识别出数据库中与患者最相配的icp;5)将数据库中识别出的icp的临床结局输出。
[0038]
在上述方法中,所述比较旨在确定icp中的所述临床参数集与从患者的ffpe样本中测定的相同临床参数集之间的最大相似度。
[0039]
在本发明的一实施方式中,相似度可以通过比较一组蛋白标志物的绝对水平来判断。通过使用患者的生物标志物的定量水平来识别鉴定在同一生物标志物的预设范围内的icp。对于一组生物标志物中每种生物标志物的水平都处于患者的该同一组生物标志物中相应的生物标志物的预设范围内的icp而言,其被认为是与该患者相似的。
[0040]
在本发明的一实施方式中,一组生物标志物中每种生物标志物的预设范围可以相同。
[0041]
在本发明的另一实施方式中,用于评估icp与患者的相似度的一组蛋白标志物中的不同生物标志物的预设范围可以不同。
[0042]
在本发明的一实施方式中,icp与患者的相似度可以基于两组定量的临床参数集之间的欧氏距离(euclidean distance)来计算。
[0043]
可以基于相似度从数据库中识别出多个icp。并且通过对其临床结局进行数学分析来为患者提供个性化预后。
[0044]
可以基于相似度从数据库中识别出多个icp。并且通过对其治疗方案及临床结局进行数学分析来为患者提供预后最好的治疗方案。
[0045]
在本发明的另一实施方式中,其它的临床表现(包括如年龄、肿瘤大小、肿瘤等级以及淋巴结状态在内的传统的临床参数)可以用于进一步提高icp与患者的相似度。
[0046]
本发明的详情也反映于附图以及在下面的描述中阐述。本发明的其它特征、目的和优势在本领域技术人员阅读这些附图和描述以及附加的权利要求书时应显而易见。附图简要说明
[0047]
图1示出了利用来自1049个乳腺癌样本的pr、er和her2的表达水平作为坐标构建的三维散点图。er、pr和her2的表达水平是通过qdb方法进行测定,其数值被用于利用origin软件构建三维散点图,以x轴表示pr、y轴表示er、z轴表示her2。来自每个患者的定位点的分布将空间分成不同的区域,包括激素区(激素组:hormone group)样品完全分布在x和y轴所呈现的平面上、her2区(her2组:her2 group)围绕在z轴上,以及角落区(角落组:corner group)(样品堆积在x、y和z轴的交界处。角落区包括了三阴型(三阴组:triple negative group)和正常样型(正常样组:normal like group,也称类正常组)。
[0048]
图2示出了使用kaplan-meier生存分析法针对基于er、pr、her2和ki67的绝对水平识别出的5名假定患者的相似组(similarity groups)的总生存期(os)与相应临床亚型的总生存期进行的比较。基于ihc的替代分析将数据库中的概况信息分为管腔a样亚型、管腔b样亚型、her2阳性亚型和三阴性(tnbc)亚型,它们的总生存期被用作为参考,使用log-rank检验与5名假定患者的相似组进行比较,p《0.05被认为具有统计显著性。(a)#1388和#1843患者的相似组与tnbc亚型的总生存期比较;(b)#1445患者的相似组与her2阳性亚型的总生存期比较;(c)##1807患者的相似组与管腔a样亚型的总生存期比较;以及(d)#1519*患者的相似组与管腔b样亚型的总生存期比较。作为低于两倍定量限[2
×
limit of quantitation(loq)]的生物标志物的水平被认为没有区别,以增加可供分析的概况信息数量。
[0049]
图3示出了使用kaplan-meier生存分析法针对基于er、pr、her2、ki67和cyclind1的绝对水平识别的5名假定患者的相似组的总生存期与相应临床亚型的总生存期进行的比较。基于ihc的替代分析将数据库中的概况信息分成管腔a样亚型、管腔b样亚型、her2阳性亚型和三阴性(tnbc)亚型,它们的总生存期被作为参考,使用log-rank检验与5名假定患者的相似组进行比较,p<0.05被认为具有统计显著性。(a)#1388和#1843患者的相似组与tnbc亚型的总生存期进行比较;(b)#1445患者的相似组与her2阳性亚型的总生存期进行比较;(c)#1807患者的相似组与管腔a样亚型的总生存期进行比较;以及(d)#1519*患者的相似组与管腔b样亚型的总生存期进行比较。在两倍定量限(loq)以内的生物标志物的量被认为没有区别以增加可供分析的概况信息数量。
[0050]
图4示出了使用kaplan-meier生存分析法针对基于er、pr、her2、ki67的绝对量识别的5名假定患者的相似组内接受不同的临床治疗的概况信息的总生存期进行的比较。治疗方案不清的概况信息未包括在分析内。(a)#1388患者的相似组内接受化疗(chemo)、内分泌治疗(et)以及化疗和内分泌联合治疗(cet:本说明书及其附图中也记作“c&e”或“c+e”,下同)的概况信息的总生存期分析;(b)#1843患者的相似组内接受化疗(chemo)、内分泌治
疗(et)以及化疗和内分泌联合治疗(cet)的概况信息的总生存期分析;(c)#1807患者的相似组内接受化疗(chemo)的概况信息的总生存期分析;(d)#1519*患者的相似组内接受化疗(chemo)以及化疗和内分泌联合治疗(cet)的概况信息的总生存期分析。在两倍定量限(loq)以内的生物标志物的水平被认为相同以增加可供分析的概况信息数量
具体实施方式
[0051]
在描述本发明的方法之前,需要声明的是,本发明在具体应用中会存在变化而不仅仅局限于在此所述的这些方法和装置。本说明书中所记载的术语仅仅是出于描述特定实施方式的目的,而并非限制这些实施方式,本发明的保护范围仅受附加的权利要求书的限制。
[0052]
除非另有说明,本说明书中使用的所有科技术语与本领域技术人员的理解相同。
[0053]
本发明涉及的内容主要出于临床诊断的目的。因而,在本说明书中,“决定或鉴定或测定(determining)”、“测量或检测(measuring)”、“评估(assessing)”和“分析(assaying)”可混用,并且包括了定量和定性两种测量方式。这些术语既可以指定量也可以指半定量,因此,“决定或鉴定或测定(determining)”和“分析(assaying)”、“测量或检测(measuring)”及其类似的描述可以混用。当要说明定量测量时,则会用到“测定分析物的量”或类似的描述。当要说明定量或半定量时,则会用到“测定分析物的水平”或者“检定(detecting)分析物”的描述。
[0054]
本说明书中描述的“量化”分析通常提供样品中的分析物与参比物(对照)的相对水平的信息,一般以数字呈现,其中“0”值可指定为分析物的量低于检测极限(lod)。
[0055]“个体(subject)”、“宿主(host)”、“患者”和“个人(individual)”在本说明书可以混用,指任何需要诊断或治疗的哺乳类(特别是人类)对象。
[0056]“空间”和“三维”(3d)可以互换地描述在三维空间中对代表样品的定位点(斑点:spot)的定位,其中定位点的强度代表了作为连续变量的第四种生物标志物的量。
[0057]
在组织水平对生物标志物的蛋白表达水平的定量测定可以通过任何方法来实现。该方法应该在其最广泛的文脉(语境)下考虑,只要生物标志物的表达水平可以被定量为连续变量的方法都包含其中。这些方法包括但不限于质谱分析或免疫分析以及两种方法的组合。
[0058]
本发明的测定结果可以是相对的结果,也可以是利用标准蛋白而表达为绝对的结果。术语“相对”和“绝对”是指两种测定方式,应该包含其最广泛的文脉。相对测定的是一种物质与另一种物质的比较结果,绝对测定利用标准单位测定一种物质的已知水平。也许,这两种方法最显著的区别在于其应用范围。相对结果只有在同样的实验条件下才有意义,而绝对结果可以在许多不同的分析中进行比较,即使分析是在完全不同的时间和地点进行的。
[0059]
在本说明书中混用的“样品(sample)”、“患者样品(patient sample)”或“样本(specimen)”和“生物样品(biological sample)”一般是指可用于测量特定分子的样品,优选诸如下文中所述的生物标志物等的与生物特征相关联的特定的标志物分子。样品可以包括但不限于外周血细胞、cns液、血清、血浆、口腔拭子(swab)、尿液、唾液、泪液、胸水及其类似物。在本发明中的样品一般指组织。
[0060]
术语“标志物”和“生物标志物”应定义为其最广泛的文脉。在此可互换混用的“标志物”和“生物标志物”一般是指,在来自一种表型(例如患者)的样品中与来自另一种表型(例如没有患病或患有不同疾病)的相比是存在不同的分子(例如多肽)。一种生物标志物的确立基于其在两种不同的表型中表达不同,也就是当其在第一种表型中的平均或者中位水平同其在第二种表型中经过计算呈现具有统计学意义的不同。
[0061]
在本发明中,生物标志物是指与生物或者疾病状态相关联的可测量的分子。它可以是已充分确立的临床诊断用生物标志物(例如免疫组化分析用临床生物标志物)或者是新发现的体外诊断用生物标志物。
[0062]
在本说明书中“参考”或“对照”是可被互换性地来指可用来针对观察的数据相比较的已知数据或已知数据组。已知数据代表两个参数之间的已知相互关系,例如生物标志物的表达水平与其相关的表型之间的相互关系。在此,已知数据构成了参考数据库中的参考概况信息。
[0063]
相应地,参考数据库可以通过存储多个参考概况信息以用于诊断目的,每个参考概况信息包括从已知诊断结果或治疗后已知临床疗效结果的对象个体获得的样品中生物标志物的水平。
[0064]
在一实施方式中,本发明涉及构建一种为癌症患者提供诊断、预估和预后的rc数据库的方法,该方法包括:提供多个已知癌症临床结局的个体;为多个个体中的每个个体构建icp,该icp包括i)多个绝对定量的蛋白标志物以及ii)癌症的已知临床结局;以及将生成的多个个体的icp存储于数据库中。
[0065]
在每个icp中不仅仅包括多个蛋白标志物,还可以包括其它的临床表现(包括年龄、肿瘤大小、肿瘤等级和淋巴结状态)。其它来自临床分析的结果(包括血液生物标志物的水平以及各种酶的水平)都可以包括在icp内。
[0066]
本发明还涉及如何从回顾性癌症数据库中识别与患者的概况信息最匹配的一个以上的参考icp的方法。该方法包括:(a)在合适的程序控制计算机上,将患者的一组蛋白标志物的表达水平与数据库中每个icp的表达水平进行比较;(b)在合适的程序控制计算机上,识别与患者高度相似的icp;以及(c)将同患者的概况信息最匹配的参考数据库中的icp的最大相似度或其相关表型输出到用户界面设备、计算机可读存储介质或者区域性或可远程接入的计算机系统,或者直接显示。
[0067]
有多种用于比较患者与数据库中icp的相似度的方法,其包括:基于针对预设的蛋白标志物的数学分析的评估,或者通过一组生物标志物的表达量逐步筛选icp。
[0068]
生物标志物的表达水平在通过数学分析比较icp与患者的相似度的过程中可以进行校正,也可以在这一过程中不进行校正。
[0069]
生物标志物的表达水平在通过数学分析比较icp与患者的相似度的过程中可以进行加权,也可以在这一过程中不进行加权。
[0070]
在一实施方式中,相似度是通过基于一组生物标志物计算icp与患者的欧氏距离来实现。
[0071]
与患者相似的icp也可以通过基于一组生物标志物表达水平逐步筛选来识别。这种方法包括:a)筛选其生物标志物a在患者的生物标志物a预设表达范围内的所有icp;b)从筛选的icp中进一步筛选其生物标志物b在患者的生物标志物b的预设表达范围内该的所有
icp;c)在进一步筛选的icp中,进一步筛选其生物标志物c在患者的生物标志物c的预设表达范围的所有icp;
……
n)在进一步筛选的icp中进一步筛选其生物标志物n在患者的生物标志物n的预设表达范围内的所有icp。
[0072]
在预先设定的生物标志物中,每种生物标志物的预设范围可以相同,也可以不同。
[0073]
在本发明的一实施方式中,经过上述过程筛选后的icp可以进一步通过其它临床表现,包括年龄、性别、肿瘤大小、肿瘤等级等进行筛选。例如,对于一个59岁的男性肺癌患者,肿瘤大小为2级、肿瘤等级为三级、淋巴结状态为n2的患者,基于蛋白标志物的相似icp可以进一步缩小限制至相似的年龄(55到60岁)、男性、肿瘤大小为2级、肿瘤等级为三级、淋巴结状态为n2的icp来提高对患者的临床诊断的准确性。
[0074]
一种基于空间关系的方法可以包括:(a)测定作为连续变量的三个以上的生物标志物的样品;(b)利用三种生物标志物(a,b,c)的值作为坐标(x,y,z),在由x、y、z轴确立的空间中定位一个代表样品的点(定位点);(c)根据患者在空间的定位点对患者进行评估,特别是步骤(c)包括了对患者癌症的诊断和预后。对患者癌症的诊断和预后的例子包括无病生存期、总生存期或癌症治疗预测。
[0075]
在上述方法中还可以包括:(d)第四种生物标志物(d)可以用来替代上述三种生物标志物中的一个,例如(a,b,d)来确立样品在新空间的定位点;然后(e)基于新空间的定位点进一步评估患者,特别是步骤(e)包括了对患者癌症的诊断和预后。对患者癌症的诊断和预后的例子包括无病生存期、总生存期或癌症治疗预测。
[0076]
此外,在上述方法中还可以是:(d)第四和第五种生物标志物(d和e)可以用来替代上述三种生物标志物中的一个,例如(a,d,e)来确立样品在新空间的定位点;以及(e)基于新空间的定位点进一步评估患者,特别是在步骤(e)中包括了对患者癌症的诊断和预后。对患者癌症的诊断和预后的例子包括无病生存期、总生存期或癌症治疗预测。
[0077]
此外,在上述的方法中还可以是:(d)第四、第五和第六种生物标志物(d、e和f)可以用来将样品在新空间中定位;以及(e)基于新空间的定位点进一步评估患者,特别是步骤(e)中包括了对患者癌症的诊断和预后。对患者癌症的诊断和预后的例子包括无病生存期、总生存期或癌症治疗预测。
[0078]
由a、b、c决定的空间定位点位置以及由a、b、d决定的新的定位点可以依次用于进一步评估患者,包括对患者癌症的诊断和预后。对患者癌症的诊断和预后的例子包括无病生存期、总生存期或癌症治疗预测。
[0079]
由a、b、c决定的空间定位点位置以及由a、b、d决定的新的定位点可以同步用于进一步评估患者,包括对患者癌症的诊断和预后。对患者癌症的诊断和预后包括无病生存期、总生存期或癌症治疗预测。
[0080]
由a、b、c决定的空间定位点位置以及由a、d、e决定的新的定位点可以依次用于进一步评估患者,包括对患者癌症进行诊断和预后。对患者癌症的诊断和预后包括无病生存期、总生存期或癌症治疗预测。
[0081]
由a、b、c决定的空间定位点位置以及由a、d、e决定的新的定位点可以同步用于进一步评估患者,包括对患者癌症的诊断和预后。对患者癌症的诊断和预后包括无病生存期、总生存期或癌症治疗预测。
[0082]
由a、b、c决定的空间定位点位置以及由d、e、f决定的新的定位点可以依次用来于
进一步评估患者,包括对患者癌症的诊断和预后。对患者癌症的诊断和预后包括无病生存期、总生存期或癌症治疗预测。
[0083]
由a、b、c决定的空间定位点位置以及由d、e、f决定的新的定位点可以同步用于进一步评估患者,包括对患者癌症的诊断和预后。对患者癌症的诊断和预后包括无病生存期、总生存期或癌症治疗预测。
[0084]
在一实施方式中,本发明还包括确定与患者概况信息最匹配的参考空间数据库中的一个亚组(sub-group)的参考空间概况信息(reference spatial profile)(sub-group)的方法。这个方法包括如下步骤:(a)在合适的程序控制计算机上,使用三种生物标志物的水平作为坐标,比较来自患者样品在空间中的定位(定位点);以及(b)与在参考空间数据库中的参考空间概况信息的亚组进行比较,以确定与参考空间概况信息的亚组的接近度(closeness);(b)在合适的程序控制计算机上,识别在参考数据库中与定位点最近的参考空间概况信息的亚组;以及(c)将同患者的空间概况信息最匹配的、参考数据库中的参考空间概况信息的亚组的最大相似度或其相关表型输出到用户界面设备、计算机可读存储介质或者区域性或可远程接入的计算机系统,或者直接显示。
[0085]
使用数学方法来探究患者的定位点与一个临床表现的推定关系。作为例子包括但不限于在由er、pr和her2的表达水平构成的三维散点图中定位点与无病生存期的关系。这些信息可以在同类分析中为其它患者提供预后。
[0086]
上述rc数据库可用于探索icp与临床结局之间的关系。使用数学分析来探究同每个icp相关的已知临床结局与生物标志物的表达水平之间的因果关系。
[0087]
临床“表现(trait)”和临床“信息(information)”可互换并且应该在其最广泛的文脉下考虑。临床表现(clinical trait)包括但不限于年龄、性别、血压、葡萄糖水平、癌症分级、无病生存期或者任何与患者的诊断、预防和治疗相关的信息。
[0088]
数据库的多个icp的临床结局可以集合起来用于统计分析来为患者提供个性化诊断、预估和预后。统计方法包括了单因素生存分析、多因素生存分析、c指数分析、kaplan-meier生存分析以及log-rank生存分析。
[0089]
不同于现在流行的用于乳腺癌和前列腺癌诊断的分型方法,本发明利用世界上广泛存在的大量储存的ffpe样本以单个患者为中心,以识别与该患者高度相似的一组icp,并分析其临床结局从而为每个癌症患者提供个性化的诊断、预估和预后。
[0090]
显而易见,可供使用的储存的ffpe样本越多,为患者提供的诊断、预估和预后越准确。
[0091]
简而言之,现有的诊断方法是画几个圆,然后把每个癌症患者放入这些圆里,由此为癌症患者提供精准治疗。相反,本发明用每个患者作为圆心以包含与其相似的icp来画出其个性化的圆。结果,有多少位癌症患者就会有多少个圆来利用rc数据库为每个癌症患者提供个性化的诊断、预估和预后。需要说明的是,在这里描述的示例性实施方式是当前优选的实施方式,因此应该仅作为描述而不局限于此。在每个实施方式中的特征或方案的描述通常应该视为也可用于其它实施方式中的其它类似特征或方案中。实施例1材料和方法
[0092]
个体及人源细胞系福尔马林固定石蜡包埋(ffpe)的乳腺癌组织切片及其临床信息取自当地医院(位于山东烟台的烟台毓璜顶医院和滨州医学院附属医院)。
[0093]
通用试剂用于细胞培养的所有通用试剂均购自thermo fisher scientifics(美国马萨诸塞州沃尔瑟姆),包括细胞培养基和培养皿。蛋白酶抑制剂购自sigma aldrich公司(美国密苏里州圣路易斯市)。所有其它化学药品购自国药集团化学试剂有限公司(中国北京)。
[0094]
裂解液的制备将两个2
×
15μm的福尔马林固定石蜡包埋的组织块切片收集到eppendorf离心管中。将切片脱石蜡,在加有蛋白酶抑制剂(2μg/ml亮抑酶肽、2μg/ml抑肽酶、1μg/ml胃酶抑素、2mm pmsf、2mm naf)的300μl的裂解缓冲液(50mm hepes、137mm nacl、5mm edta、1mm mgcl2、10mm na2p2o7、1%tritonx-100、10%glycerol)中进行超声处理2分钟,然后以12000
×
g进行离心5分钟。收集上清液进行免疫印迹分析。总蛋白质浓度根据制造商的说明使用pierce bca蛋白质测定试剂盒进行测量。
[0095]
qdb分析特异性抗体(针对her2的ep3或4b5克隆,针对ki67的mib1克隆,针对雌激素受体(er)的sp1和孕激素受体的1e2、针对cyclind1的sa38-08)各自的线性范围是用来自几个分别对这些生物标志物呈阳性反应的患者的裂解物加以混合的混合液来确定的。首先混合等量的3至4个来自乳腺癌组织的组织裂解液,将混合的裂解液从0至2μg进行梯度稀释以确定qdb分析的线性范围。对于作为商品购入的或者由公司内部自行表达并纯化的标准蛋白也按0pg至500pg的梯度稀释以用于确定qdb分析的线性范围。
[0096]
将样品按一式三份以2μl/单元加到qdb板上,并如现有技术中所述进行处理。在每孔中加入100μl第一抗体,在4℃下孵育过夜。再在室温下将驴抗兔或驴抗小鼠第二抗体与qdb板孵育4小时。先用tbst简单冲洗qdb板两次,再清洗5次,每次10分钟,然后将它们放入白色96孔板中3分钟,该白色96孔板预先加有100μl/孔根据制造商的说明书制备的ecl溶液。然后使用tecan infiniti 200pro酶标仪,通过在用户界面上选择“带盖的板”来定量测量来自组合板(recombinant plate)的每个孔的化学发光信号。
[0097]
使用所获得的读数通过对照标准蛋白(蛋白标准品)来测定ffpe样本中生物标志物水平。将测得的pr、er、her2和ki67的生物标志物水平输入数据库。将样本分为三组用于qdb分析。从每组中选取6个样本(2个强表达、2个弱表达和2个中等表达)在相同实验中进行测定以验证结果的一致性。
[0098]
将这些结果使用originpro 9.1软件绘制3d散点图。
[0099]
实施例1介绍了如何使用er、pr和her2的蛋白水平来创建3d散点图,并使用该散点图来确定患者的治疗计划。
[0100]
利用qdb方法将pr、er和her2的蛋白质水平测定为绝对的且连续的变量,将结果输入qdb数据库。
[0101]
将更多的样品的结果输入该qdb数据库以确保数据库的增长。
[0102]
使用qdb数据库中er、pr和her2水平创建3d散点图,并不断调整该散点图以确保其准确性和全面性(图1)。
[0103]
每个定位点代表一个样本的3d散点图定义为参考空间数据库。
[0104]
使用数学分析将临床信息(包括dfs和os)与参考空间数据库中的每个定位点相关联。
[0105]
使用qdb方法测定患者ffpe样本的er、pr和her2的水平。
[0106]
将代表该患者的定位点定位于参考空间数据库中。
[0107]
根据患者在参考空间分布图中定位点来识别参考空间概况信息,并对来自该参考空间概况信息的临床信息进行分析,以用于该患者的诊断、预估和预后。
[0108]
或者,根据空间定位可将参考空间概况信息划分为不同的亚组。
[0109]
将临床信息(包括dfs和os)与参考空间概况信息的每个亚组相关联。在此情况下是指激素组、her2组和角落组。
[0110]
利用qdb方法测定患者的ffpe样本中er、pr和her2的水平,通过空间定位将其定位点定位于某亚组中。
[0111]
根据定位点所在的亚组为患者提供临床诊断、预估和预后。实施例2
[0112]
材料和方法的详细描述见实施例1。
[0113]
本实施例介绍了如何利用基于临床研究的3d模型为患者提供诊断、预估和预后。
[0114]
通过分析大量具有匹配临床信息的研究空间信息,构建了一个将空间定位与临床信息(包括dfs和os)相关联的3d模型。
[0115]
将患者的三种生物标志物的蛋白水平测定为绝对的且连续的变量。
[0116]
在由仪器或软件支持的3d模型中,根据三种生物标志物的表达水平对患者进行空间定位。
[0117]
通过3d模型,基于三种生物标志物的水平对病人进行的空间定位,为患者提供诊断、预估或预后。实施例3
[0118]
材料和方法的详细描述见实施例1。
[0119]
本实施例介绍了如何针对亚亚组(sub-sub-group)患者依次使用两个3d散点图以进行临床诊断、预估和预后。
[0120]
使用qdb方法测定两例患者ffpe样本中6种生物标志物er、pr、her2、ki67、pcna以及p53的水平。
[0121]
在以er、pr、her2表达水平为x、y、z轴的参考空间数据库3d散点图中,根据这两例患者er、pr及her2表达水平确定的定位点的空间位置,将其划分到同一亚组。
[0122]
将基于这两例患者ki67、pcna、p53表达水平确定的两个定位点,定位于以ki67、pcna、p53为x、y、z轴的3d散点图中。
[0123]
定位点201位于由ki67和pcna构建的侧壁上,并且p53不表达,并且定位点202因ki67、pcna和p53的强表达而导致其漂浮在空间中。
[0124]
因此,尽管患者201和202在er、pr、her2的3d散点图中属于同一亚组(管腔a组),但在以ki67、pcna、p53的3d散点图中属于不同的亚组。实施例4
[0125]
使用从本地采集的427例ffpe样本开发的乳腺癌概况数据库进行研究。临床结局仅限于本研究中患者的总生存期(os)。虽然用于评估相似度的最优选的一组蛋白标志物尚待探索,但已经利用qdb方法完成了对这些ffpe样本中乳腺癌常用的几种生物标志物(er、pr、her2、ki67以及cyclind1)的绝对定量测定。这些蛋白标志物的测定结果,结合记录在案
的临床病理参数(年龄、肿瘤大小、肿瘤分级、淋巴结状态)、治疗方案以及得到的临床结局,对这个初级的癌症概况数据库中的每个样本创建了icp。
[0126]
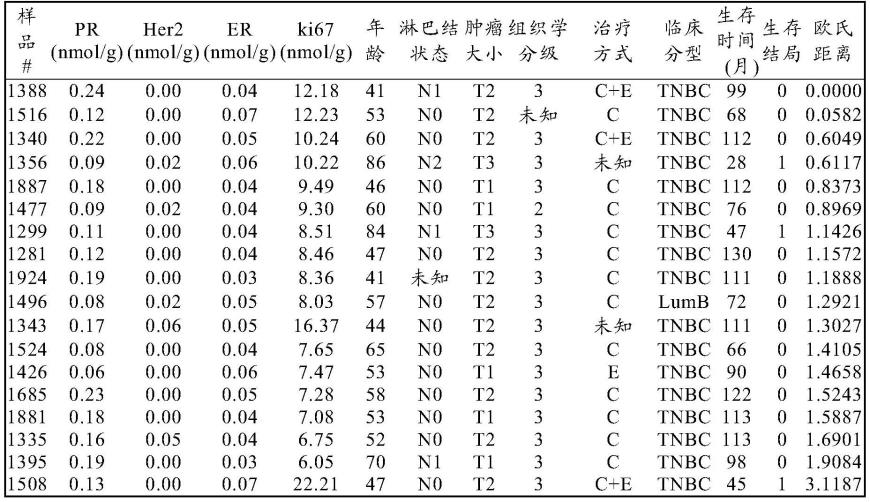
从癌症概况数据库中随机选取至少一种生物标志物水平相互存在显著差异的5个ffpe样本(#1388、#1843、#1445、#1807、#1519)的概况信息被用来作为假定的新增患者(表1)。另外,这些样本还代表基于ihc替代分型(分析)的四种临床亚型,其中#1388和#1843为三阴性(tnbc)亚型、#1445为her2阳性亚型、#1807为管腔a样亚型、#1519为管腔b样亚型。
[0127]
在临床实践中,er、pr、her2及ki67这4种生物标志物最先被用于定义患者的临床亚型。在本研究中,这4种标志物也首先被用来评估不同的假定患者与数据库中每个癌症概况信息的相似度。计算每名假定患者与数据库中每个癌症概况信息的欧氏距离并按照从低到高进行排序。如果生物标志物的水平都低于最低定量限(loq),则认为它们的表达水平相同。此外,由于数据库的规模较小,若某癌症概况信息的一种或多种生物标志物水平低于假定患者的50%或高于假定患者的2倍,则该癌症概况信息不予采用。例如#1519假定患者的er水平为2nmole/g,则认为数据库中任何er小于1nmole/g或大于4nmole/g的癌症概况信息都与#1519患者不相似而拒绝。
[0128]
在这个小型数据库中,分别为假定患者#1388、#1843、#1445、#1807找到了18、35、10、14例相似癌症概况信息,与患者#1519相似的只有3个癌症概况信息(表1)。显然,数据库的规模极大地限制了使用者或研究者的分析能力,这进一步强调了需要将数据库扩大到成千上万甚至数百万的癌症概况信息,才能充分发挥这种方法的潜力。
[0129]
将每一组的相似的癌症概况信息(相似组)的os与假定患者相应的临床亚型的os进行比较(图2)。如图2中a部分所示,#1388的相似组(本说明书及其附图中也称“#1388相似组”或“#1388组”,其它类推)和#1843的相似组的10年生存率(简称:10y sp)均略高于tnbc亚型。然而,这些差异尚未达到统计显著性(p=0.057)。对于作为her2阳性亚型的#1445的相似组的os较整个her2阳性组略有提高,10y sp从72%提高到88%(p=0.27)。对于与作为管腔a样亚型的#1807相似的14个癌症概况信息,其10y sp与整个管腔a样亚型高度相似(p=0.91)。
[0130]
由于数据库规模较小,只有3个癌症概况信息与作为管腔b样亚型的#1519相似。为了从该数据集中提取潜在的指示信息以供参考,略微放宽了针对相似度的限制,即把生物标志物水平低于2
×
loq的癌症概况信息也纳入其中。例如,若her2的loq为0.15nmole/g,则任何her2小于0.3nmole/g的概况信息都被认为有相同的her2水平。在该放宽的条件下,16个癌症概况信息被确认为与#1519相似,并且该相似组与整个管腔b样亚型相比10y sp显著变差(p=0.0096)。
[0131]
可想而知,评估中包含的生物标志物越多,概况信息与新患者之间的相似度就越能达到更高的水平。因此,鉴于cyclind1在预测管腔样亚型患者的总生存期方面是独立于ki67之外的生物标志物,将其纳入评估中以识别5名假定患者各自的相似组。分别计算数据库中每个概况信息与这5名假定患者之间的欧氏距离,并按照距离进行排序(表2)。任何与假定患者的生物标志物水平相比低于其50%或高于其2倍的癌症概况信息,都不被纳入相似组。
[0132]
不出所料,在所有病例中能发现与假定患者相似的癌症概况信息更少。#1388相似组由18例降为7例、#1843相似组由35例降为20例、#1445相似组由10例降为7例、#1807相似
组由14例降为5例、#1519相似组由16例降为9例。采用log rank检验,将各相似组的os与假定患者相应临床亚型的os进行比较(图3)。出乎意料的是,cyclind1的加入能够使#1843组、#1388组与tnbc组的差异均达到统计显著性(p=0.023),#1843组的10y sp(100%)显著高于,而#1388组的10y sp(57%)显著低于tnbc亚型的10y sp(75%)(图3的a)。另一方面,cyclind1的加入对#1445组的预后帮助不大(图3的b)。cyclind1的加入使#1807组的预后比管腔a样亚型更差,但这种差异未达到统计学差异(p=0.095)。对于#1519组,其os仍然比管腔b样亚型差,其log rank检验值为p=0.034。
[0133]
该方法最大的用处是为新患者提供量身定制的治疗建议。因此,每个相似组内的概况信息根据其所接受的治疗被进一步分组,并进行os分析以确定疗效最佳的治疗方案。由于数据库规模较小,治疗方式被简单分为化疗(ct)、内分泌治疗(et)以及化疗内分泌联合治疗(cet)。该研究使用了基于er、pr、her2和ki67四种生物标志物确定的相似组,以引入更多的概况信息以供分析。
[0134]
对于#1388相似组(图4的a),仅接受ct的患者(n=12)的5年生存率(简称:5y sp)为92%,而接受cet治疗的患者(n=3)的5y sp为67%。对于#1843组,无论接受何种治疗,所有患者均存活。对于#1445组,由于该组所有患者都仅接受了ct,因此无法提取到任何相关信息。对于#1807组,仅接受ct的患者(n=4)的10y sp为100%,而接受cet的患者(n=8)的10y sp为71%。对于#1519组,虽然接受cet的患者(n=6)的5y sp比仅接受ct的患者(n=9)表现出优势,但这种优势在后期消失了。
[0135]
因此,本文显示了基于相邻原则的诊断方法为5名假定患者提供个性化os预测的可行性。#1388、#1843和#1519的10y sp与相应临床亚型的10y sp存在显著差异。通过os分析,还对5名假定患者的相似组进行了各种治疗方式的有效性评估。然而,由于数据库规模的限制,他们的差异没有统计显著性,无法为这些假定患者提供指导。
[0136]
值得注意的是,尽管#1843属于预后最差的临床亚型tnbc,但其相似组的所有患者不论接受何种治疗也仍然在研究结束时存活(图3的a以及图4的b)。这令人怀疑#1843属于最初分子分型研究中描述的正常样亚型(normal-like subtype,也称“类正常亚型”)。
[0137]
显然,随着概念和技术的成熟,在日常临床实践中应用这种新型诊断方法的限速步骤是开发一个比本研究中使用的数据库大得多的癌症概况数据库。例如,若有大于10000例乳腺癌概况信息(breast cancer profile),基于5种生物标志物可以确定大约500个(10000/400
×
20)概况信息与#1843高度相似,125个概况信息与#1807高度相似,175个概况信息与#1388和#1445高度相似,从而为这5名假定患者提供可靠的指导。
[0138]
然而,即使在这种规模下,仍然难以识别出足够数量的与#1519相似的癌症概况信息,因为基于4种生物标志物只能识别75个概况信息,如果评估相似度时使用5种生物标志物,则能识别的概况信息就更少了。可想而知,像#1519这样的患者在目前的临床实践中更有可能被过度治疗或治疗不足,这是由于在涉及成百上千病例的临床试验中这些患者不太可能得到充分应对。
[0139]
同样,由于数据库规模有限,本研究将治疗方式分为ct、et和cet。然而,单就化疗而言,至少有4种类型的药物:烷化剂、抗肿瘤抗生素、抗代谢药和有丝分裂抑制剂。在每种类型中,还有几种不同的药物可供选择。显然,只有通过扩展数据库才能找出对新患者具有最佳疗效结局的确切药物。
[0140]
所有这些考虑都强调了广泛扩展数据库的必要性。幸运的是,由于qdb技术绝对定量的特性,前面所述的癌症概况数据库是一个不断成长的数据库。随着这种方法在全球范围内被接受,预计在不久的将来该数据库将呈指数级增长。
[0141]
本研究中使用的5种生物标志物不一定是评估乳腺癌患者相似度的最佳选择。然而,它们可以作为从所有现有生物标志物和候选生物标志物中识别最优选数量和组合的基础,以评估乳腺癌患者之间的相似度。能够对患者提供可靠指导所需的相似的概况信息最低数量还需要通过全球肿瘤学家和统计学家的合作努力来确定。
[0142]
不可否认,欧氏距离在本研究中的作用有限,因为所有生物标志物水平与假定患者相比都大于其50%且小于其200%的癌症概况信息数量有限。然而,可以想象其对于显著扩张的数据库的巨大作用,可以确立一个基于欧氏距离的切断(cut)值以识别那些与新患者具有最高相似度的癌症概况信息。
[0143]
同样值得一提的是,尽管在本研究中,相似度评估是完全基于一组蛋白标志物的表达水平,但其它临床病理参数(如年龄、肿瘤大小、淋巴结状态、基于oncotype测定的rs分数、基于pam50测定的ror评分)也可以结合到评估中以提高相似度的水平。同样,虽然本研究仅限于os分析,但包括复发在内的其它临床结局也可在需要的时候用于评估。材料和方法
[0144]
个体及人源细胞系:由滨州医学院烟台附属医院和青岛大学附属烟台毓璜顶医院(中国烟台市)共同提供490例福尔马林固定石蜡包埋(ffpe)的乳腺癌组织的切片(2
×
15μm/例),其中427例具有总生存期(os)数据。包括样品采集和研究方案在内的所有研究均符合《赫尔辛基宣言》,并经滨州医学院烟台附属医院伦理委员会(批准号:#20191127001
–
郝俊梅)和毓璜顶医院伦理委员会(批准号:[2017]76
–
于国华)批准。由于这两项研究中的个体是医院大量保存的匿名的、具有回顾性临床数据的ffpe样本,因此豁免了提供知情同意书的要求。
[0145]
除了生物标志物水平是采用qdb方法测定外,所有临床信息均来自病历。qdb流程在其它文献中也有详细描述。
[0146]
通用试剂:所有的通用试剂均在别处描述过,er(sp1)兔单克隆抗体购自abcam inc,pr(1e2)兔单克隆抗体购自roche diagnostics gmbh,her2(ep3)兔单抗、ki67(mib1)鼠单抗及cyclind1(ep12)兔单抗均购自中杉金桥生物技术有限公司(中国北京,www.zsbio.com)。hrp标记的驴抗兔igg二抗购自jackson immunoresearch实验室(美国pa,pike west grove)。qdb板由quanticision diagnostics inc提供(美国nc,rtp)。
[0147]
欧氏距离的计算:用以下公式分别计算样品a(er、pr、her2、ki67)与样品b(er’、pr’、her2’、ki67’)以及样品c(er、pr、her2、ki67和cyclind1)与样品d(er’、pr’、her2’、ki67’、cyclind1’)之间的距离:d_ab=√((er-er')^2+(pr-pr')^2+(her2-her2')^2+(ki67-ki67')^2)d_cd=√((er-er')^2+(pr-pr')^2+(her2-her2')^2+(ki67-ki67')^2+(cyclind1-cyclind1')^2)。
[0148]
预先对所有样品中每种生物标志物的qdb结果标准化(通过z-score转化)。定量限(loq):her2为0.15nmol/g,er为0.1nmol/g,pr为0.25nmol/g,ki67为1.3nmol/g。
[0149]
生存分析:不同亚组的总生存期采用kaplan-meier法显示,并通过log-rank检验
进行比较。p<0.05被认为有统计显著性。所有的统计分析均使用r4.0.1进行(http://www.r-project.org)。
[0150]
相信所附权利要求书中特别指出了来自于上述披露的本发明中某些特征组合及子组合,具备新颖性和创造性。作为体现在以特征、功能、元件或属性的其它组合和子组合的发明,可以通过修改本权利要求或在本技术或相关申请中提出新的权利要求而得到保护。这些修改的或新的权利要求,无论它们是针对不同的发明还是针对同一发明,无论它们在范围上与原权利要求不同、更宽、更窄或相同,都被视为包括在本发明的所披露的对象范围内。表1:基于er、pr、her2及ki67蛋白绝对定量水平的相似癌症概况信息组(相似组)表1-1#1388相似组表1-2#1843相似组
表1-3#1445相似组表1-4#1807相似组
表1-5#1519*相似组缩略词:所有临床病理因子均符合美国癌症联合委员会的定义(ajcc)。c:化疗;e:内分泌治疗;luma与lumb:管腔a样与管腔b样临床亚型;tnbc:三阴型乳腺癌;her2:her2阳性亚型;生存结局:0-活,1-死。注意:所有在定量限(loq)范围内的生物标志物水平均被视为是相同的。至少有一种生物标志物水平量与假定患者有显著差异(《50%或》200%)的那些样品被淘汰。*:在2
×
loq范围内的生物标志物水平被认为是相同的。至少有一种生物标志物水平量与假定患者有显著差异(《50%或》200%)的那些样品被淘汰。表2:基于er、pr、her2、ki67及cyclind1蛋白绝对定量水平的相似癌症概括信息组(相似组)表2-1#1388相似组
表2-2#1843相似组表2-3#1445相似组表2-4#1807相似组
表2-5#1519*相似组缩略词:所有临床病理因子均符合美国癌症联合委员会的定义(ajcc)。c:化疗;e:内分泌治疗;luma与lumb:管腔a样与管腔b样临床亚型;tnbc:三阴型乳腺癌;her2:her2阳性亚型;生存结局:0-活,1-死。注意:所有在定量限(loq)范围内的生物标志物水平均被视为是相同的。至少有一种生物标志物水平与假定患者有显著差异(《50%或》200%)的那些样品被淘汰。*:在2
×
loq范围内的生物标志物水平被认为是相同的。至少有一种生物标志物水平与假定患者有显著差异(《50%或》200%)的那些样品被淘汰。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1