淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法与流程
本发明涉及一种去除单核细胞干扰的方法。特别是涉及一种淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法。
背景技术:
流式细胞技术中,待测样本细胞经液流系统传送,形成单细胞流,在激光照射区域,细胞上标记的荧光染料受到激光的激发,产生荧光信号。在不同的实验体系中,根据细胞标记的荧光素不同,在不同波长激发下,发射出不同波长的荧光,这些荧光信号可以反映不同的细胞生物学特性。细胞的组分可以用光度学定量测量,对同一细胞可以获得有关不同组分的多方面信息,用作鉴别细胞的依据。淋巴细胞亚群的表型分析对了解淋巴细胞的分化、功能和鉴别新的淋巴细胞亚群具有重要的价值。
在6色抗体组合淋巴细胞亚群识别中,现有的自动和人工淋巴细胞亚群分析都不能比较精准的将单核细胞和淋巴细胞分离。
在缺少cd45-fifc/cd14-pe作为淋巴细胞的反向设门的情况下,现有的人工细胞亚群分析,是通过ssc/cd45对淋巴进行设门,由于缺少单核细胞特异性cd14荧光标记,在淋巴门中,无法识别混入的单核细胞;仪器模拟人工的分析,减少了因个人偏好或其他因素导致的主观错误,但并未完善单核细胞的分离。表1给出了单核细胞正常分化抗原表达。
表1正常单核细胞的分化分期和抗原表达特点
+:阳性;-:阴性。
如表1所示,cd14为成熟单核细胞特异性表达,cd4、cd16部分表达;t淋巴细胞部分强表达,部分不表达;nk淋巴细胞表达cd16。因此cd4可作为算法分离单核细胞和淋巴细胞的依据。
技术实现要素:
本发明所要解决的技术问题是,提供一种能够有效地将单核细胞和淋巴细胞中区分开来的淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法。
本发明所采用的技术方案是:一种淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法,通过cd3、ssc-a、cd45和基于密度的聚类算法,加上核密度估计算法确定cd3的荧光强度的强弱,识别t淋巴细胞,以此确定淋巴细胞在cd45和ssc-a上二维分布的区域及相应位置参数,并结合核密度估计算法分别拟合细胞在cd45和ss上分布的密度曲线,由此确定单核细胞与淋巴细胞在ss上分布的密度曲线阈值,及t淋巴细胞cd45分布的分位数,进而利用核密度估计拟合淋巴t细胞在cd4上的分布,以确定cd4不表达与弱表达阈值;在确定阈值的基础上,对ssc-a、cd4、cd45、cd16和cd19多维数据是用k-means聚类算法进行细胞分群,根据ssc-a、cd45、cd4各个阈值区分非t淋巴细胞与单核细胞。
淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法,具体包括如下步骤:
1)去除黏连细胞
在数据包含fsc-a和fsc-h项的情况下,将fsc-h减fsc-a得到差,对所述的差计算60%的分位数,从原始文件的全部细胞数据中选取差值大于分位数的细胞数据,此细胞数据即为大概率属于非黏连细胞的细胞数据,以fsc-a作为x轴,以fsc-h作为y轴,进行线性拟合获得斜率,根据斜率计算拟合直线与y轴的夹角,作为旋转角度,将fcs-a和fsc-h数据转化为极坐标数据,并进行数据旋转,对旋转后的数据进行核密度估计,去除黏连细胞,得到有效细胞数据;
2)识别粒细胞ss+cd45+相对单核细胞和淋巴细胞的位置
通过cd3、ssc-a和cd45确定t淋巴细胞cd3+ss-cd45+的对照位置,将t淋巴细胞的ssc-a、cd45和fsc-a作为内参照,层次化聚类分群,识别粒细胞ss+cd45+相对单核细胞和淋巴细胞的位置,其中,所述单核细胞和淋巴细胞的位置是细胞群ss-cd45++;
3)去除单核细胞
通过ssc-a、cd3、cd45和基于密度的聚类算法,以及核密度估计算法确定细胞群ss-cd45++的各聚类分群的cd3荧光强度均值和细胞荧光强度的密度分布曲线,将cd3荧光强度符合正态分布的ss-cd45++细胞识别为t淋巴细胞,以被识别出的t淋巴细胞来圈定淋巴细胞区域,并获得相应的位置参数,结合核密度估计算法拟合的密度曲线及t淋巴细胞在核密度估计算法拟合的密度曲线中的位置调整参数,对cd4、ssc-a和cd45数据使用k-means聚类算法,根据调整后的参数识别非t淋巴细胞与单核细胞,去除单核细胞得到非t淋巴细胞。
步骤1)中对所述的差计算60%的分位数,是将差值的概率分布范围分为一百个等份的数值点,取得60等分位置的数值。
步骤1)所述的根据斜率计算拟合直线与y轴的夹角,是采用如下公式:
拟合直线与y轴的夹角=90-arctan(k)
其中,k为斜率,arctan(k)为斜率k的反正切函数值。
步骤3)中,当步骤1)提到的去除黏连的有效细胞数据中包含有cd16和cd19数据时,则还要对cd16和cd19数据使用k-means聚类算法,根据调整后的参数识别非t淋巴细胞与单核细胞,去除单核细胞得到非t淋巴细胞。
步骤3)中,所述的调整参数,包括调整如下参数:t淋巴细胞ssc-a主体最大值、单核细胞和淋巴细胞密度曲线阈值、cd4不表达与弱表达阈值和t淋巴细胞cd45主体中位数值。
本发明的淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法,在缺少cd45-fifc/cd14-pe作为淋巴细胞的反向设门的情况下,通过聚类等算法能够通过cd3-fitc/cd4-pe准确分离出单核细胞群簇与淋巴细胞;其次对所有的数据进行更加客观性的分析,不会使细胞分群受到人为主观因素的影响;最后能够有效去除噪音干扰。
附图说明
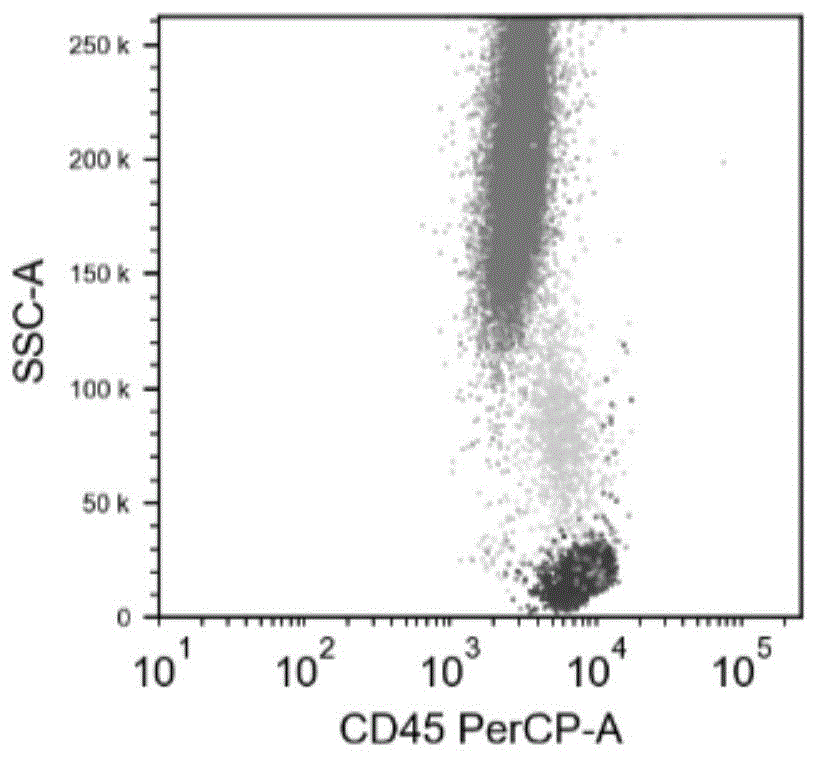
图1a是淋巴细胞,单核细胞和粒细胞在cd45与ssc-a上二维分布图;
图1b是淋巴细胞和单核细胞在ssc-a与cd4上二维分布图;
图2是去除黏连细胞的流程图。
具体实施方式
下面结合实施例和附图对本发明的淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法做出详细说明。
在单核细胞、淋巴细胞、淋巴细胞和nk细胞中,淋巴细胞具有cd3特异性表达,非t淋巴细胞与通过cd3识别的t淋巴细胞组成淋巴细胞。
本发明的淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法,包括通过cd3、ssc-a、cd45和基于密度的聚类算法,加上核密度估计算法确定cd3的荧光强度的强弱,识别t淋巴细胞,以此确定淋巴细胞在cd45和ssc-a上二维分布的区域及相应位置参数,并结合核密度估计算法分别拟合细胞在cd45和ss上分布的密度曲线,由此确定单核细胞与淋巴细胞在ss上分布的密度曲线阈值,及t淋巴细胞cd45分布的分位数,进而利用核密度估计拟合淋巴t细胞在cd4上的分布,以确定cd4不表达与弱表达阈值;在确定阈值的基础上,对ssc-a、cd4、cd45、cd16和cd19多维数据是用k-means聚类算法进行细胞分群,根据ssc-a、cd45、cd4各个阈值区分非t淋巴细胞与单核细胞。
如图1所示,本发明的淋巴细胞亚群自动分析中有效去除单核细胞干扰的方法,具体包括如下步骤:
1)去除黏连细胞
在数据包含fsc-a(forwardscatterarea)和fsc-h(forwardscatterheight)项的情况下,将fsc-h减fsc-a得到差,对所述的差计算60%的分位数,从原始文件的全部细胞数据中选取差值大于分位数的细胞数据,此细胞数据即为大概率属于非黏连细胞的细胞数据,以fsc-a作为x轴,以fsc-h作为y轴,进行线性拟合获得斜率,根据斜率计算拟合直线与y轴的夹角,作为旋转角度,将fcs-a和fsc-h数据转化为极坐标数据,并进行数据旋转,对旋转后的数据进行核密度估计,去除黏连细胞,得到有效细胞数据;其中,
所述的差计算60%的分位数,是将差值的概率分布范围分为一百个等份的数值点,取得60等分位置的数值。
所述的根据斜率计算拟合直线与y轴的夹角,是采用如下公式:
拟合直线与y轴的夹角=90-arctan(k)
其中,k为斜率,arctan(k)为斜率k的反正切函数值。
如图2所示,在数据包含forwardscatterarea(fsc-a)和forwardscatterheight(fsc-h)项的情况下,通过fsc-h减fsc-a的数据60%分位数,选取40%极大概率属于非黏连细胞的细胞数据。进行线性拟合获得斜率。根据计算旋转角度,进行核密度估计,去除黏连细胞。
2)识别粒细胞ss+cd45+相对单核细胞和淋巴细胞的位置
粒系在淋巴细胞亚群分析中,可作为阴性参照,但由于占cd45阳性比例通常大于50%,容易影响结果,需先识别粒细胞,减少其影响。通过cd3、ssc-a和cd45确定t淋巴细胞cd3+ss-cd45+的对照位置,将t淋巴细胞的ssc-a、cd45和fsc-a作为内参照,层次化聚类分群,识别粒细胞ss+cd45+相对单核细胞和淋巴细胞的位置,其中,所述单核细胞和淋巴细胞的位置是细胞群ss-cd45++;
3)去除单核细胞
通过ssc-a、cd3、cd45和基于密度的聚类算法,以及核密度估计算法(kde)确定细胞群ss-cd45++的各聚类分群的cd3荧光强度均值和细胞荧光强度的密度分布曲线,将cd3荧光强度符合正态分布的ss-cd45++细胞识别为t淋巴细胞,以被识别出的t淋巴细胞来圈定淋巴细胞区域,并获得相应的位置参数,结合核密度估计算法拟合的密度曲线及t淋巴细胞在核密度估计算法拟合的密度曲线中的位置调整参数,对cd4、ssc-a和cd45数据使用k-means聚类算法,根据调整后的参数识别非t淋巴细胞与单核细胞,去除单核细胞得到非t淋巴细胞。其中,
当步骤1)提到的去除黏连的有效细胞数据中包含有cd16和cd19数据时,则还要对cd16和cd19数据使用k-means聚类算法,根据调整后的参数识别非t淋巴细胞与单核细胞,去除单核细胞得到非t淋巴细胞。
所述的调整参数,包括调整如下参数:t淋巴细胞ssc-a主体最大值、单核细胞和淋巴细胞密度曲线阈值、cd4不表达与弱表达阈值和t淋巴细胞cd45主体中位数值。
- 还没有人留言评论。精彩留言会获得点赞!