一种基于UPLC-MS技术推断大鼠死亡时间的方法
本发明涉及法医学领域,具体是一种基于uplc-ms技术推断大鼠死亡时间的方法。
背景技术:
死亡时间(postmorteminterval,pmi)是指发现尸体或检验尸体的时间点与发生死亡的时间点之间的时间间隔。推断死亡时间是法医尸检的一项重要任务,对确定作案时间、调查犯罪现场、筛查犯罪嫌疑人有重要意义。死亡时间的推断方法包括观察死后变化和实验室的生化检测等检测手段,目前对于较早期死亡时间推断的研究较为全面(死亡时间≤24小时),但是对于较晚期死亡时间推断的研究相对匮乏(尤其是死亡时间≥15天)。这是由于随着死亡时间的延长,组织细胞发生自溶,蛋白质和核酸逐渐降解,难以检测。但是,代谢产物相对不易降解,并且代谢组学对于被检样本质量的要求也比较宽松。所以,代谢组学技术或许更适用于研究较晚期死亡时间推断。
起初,用于推断pmi的金标准是基于温度的列线图方法,除此之外,还会参考一些死后变化,如尸斑、尸僵、角膜浑浊等,但是根据死后变化推断pmi主要是依靠丰富的知识和检案经验等主观因素。因此,寻找更加有效的技术方法或客观的生物指标来推断死亡时间就显得尤为重要。后来有研究发现对滑液和玻璃体液进行生化检测可用于推断pmi,蛋白质的降解也被证明与死亡时间存在一定的关系。而死后尸体在微生物作用下逐渐发生腐败,各种小分子化合物在组织中消耗、生成、积累,腐败微生物和腐败产物的量随着死亡时间的延长,而不断变化。代谢组学技术可以通过单次检测对成百上千的小分子进行定性定量研究,在筛选标志物上有着一定优势。另外,生物体内小分子种类极为相似,构建动物模型有利于推广应用到实践中。因此,我们认为检测死后小分子物质的变化可能有助于推断pmi。
近年来,代谢组学发展成为研究生命科学的热门领域,可以通过对肌肉、体液及其他检材进行高通量分析,提供非常全面的小分子化合物信息。目前,法医学者已应用代谢组学技术进行不同方向的研究:研究生物死后组织或体液中代谢物的变化以推断pmi;检测滥用药物及其代谢物;法医相关病理损伤后组织或体液可能出现的代谢变化。随着人工智能的快速发展,机器学习算法被广泛地应用于科学研究的各个领域。机器学习算法主要是指通过数学及统计学方法求解最优化问题的过程,针对不同的数据和不同的模型需求,选择合适的算法可以更高效地解决实际问题。代谢组学技术作为一种高通量、高灵敏度的检测方法,可以获得庞大的原始数据,应用机器学习算法可以更全面地挖掘出数据背后隐藏的生物学意义,建立更加准确、可靠的预测模型。
在当前的法医学死亡时间推断的研究中,不论是从基因层面、蛋白质层面还是代谢物层面来看,对于相对较短死亡时间的推断研究都已经较为成熟且建立了具有较高预测准确度的数学模型,但是由于个体差异、环境等影响因素的存在,目前的研究尚未应用于实践。此外,目前关于相对较晚期死亡时间的推断研究仍然较为缺乏。
技术实现要素:
本发明为了解决较晚期死亡时间推断的关键科学问题和实践难点,提供了一种基于uplc-ms技术推断大鼠死亡时间的方法。本发明采用uplc-ms技术,检测不同死亡时间的大鼠骨骼肌组织,筛选出与死亡时间相关的差异小分子化合物,结合神经网络算法,构建三级死亡时间推断模型,最后把三级死亡时间推断模型串联起来对样本进行预测。三级串联预测模型进行分段分步鉴别死亡时间,提高死亡时间预测的准确性和缩短死亡时间鉴别的时间窗口。
本发明是通过以下技术方案实现的:大鼠死亡时间推断模型的建立方法,包括以下步骤:
㈠收集死亡后不同时间点收集的大鼠骨骼肌样本,提取的小分子化合物,通过uplc-ms技术进行代谢组学检测;对色谱峰识别、峰对齐,获得化合物名称、保留时间、精确质荷比和峰面积,峰面积归一化,获得随死亡时间变化的化合物数据集;
㈡依据化合物在不同死亡时间样本中含量不同,通过主成份分析获得大鼠死亡时间分段信息,根据正交偏最小二乘判别针对每一个死亡时间段筛选差异化合物以变量权重重要性排序和t检验中p<0.05为原则,筛选用于死亡时间推断数学模型的潜在差异化合物集合,并根据和数据库中二级质谱图比对,共确定了25种内源性化合物作为差异小分子化合物集合;
㈢打开clementine12.0,分步建立三级串联预测模型:
(1)建立一级预测模型a
第一步:选择源选项里的excel选项,将用于预测0天、1-3天、5-7天、9-15天和18-30天死亡时间段的19个差异小分子化合物含量导入,19个差异小分子化合物为dl-色氨酸、异戊胺、油酸酰胺、邻羟基肉桂酸、n-乙酰-l-苯基丙氨酸、脯氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、l-苯丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、泛酸、尿苷、2-巯基苯并噻唑、8(s)-羟基-(5z,9e,11z,14z)-二十碳四烯酸、l-酪氨酸、l-丝氨酸;
第二步:选择字段选项里的类型选项,判别变量为19个差异小分子化合物,死亡时间分组变量包括a:0天,b:1-3天,c:5-7天,d:9-15天,e:18-30天,死亡时间分组为输出变量,依次读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样大部分用作训练集,少部分用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络一级预测模型a的超参数,得到最优预测模型以及内部验证评价模型;
(2)建立二级预测模型
①建立二级预测模型b
第一步:选择源选项里的excel选项,将用于鉴别1天、2天、3天死亡时间点的14个差异小分子化合物含量导入,14个差异小分子化合物为dl-色氨酸、5-氨基戊酸、异戊胺、油酸酰胺、n-乙酰-l-苯基丙氨酸、脯氨酸、甲硫氨酸、吲哚-3-乳酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、缬氨酸、尿苷、l-酪氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括1天、2天和3天;判别变量为上述14个差异化合物;死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样大部分用作训练集,少部分用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络二级预测模型b的超参数,得到最优预测模型以及内部验证评价模型;
②建立二级预测模型c的步骤为:
第一步:选择源选项里的excel选项,将用于鉴别5天、7天死亡时间点的15个差异小分子化合物含量导入,15个差异小分子化合物为dl-色氨酸、5-氨基戊酸、油酸酰胺、邻羟基肉桂酸、n-乙酰-l-苯基丙氨酸、脯氨酸、甲硫氨酸、吲哚-3-乳酸、l-苯基丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、2-巯基苯并噻唑、l-丝氨酸、l-酪氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括5天和7天,判别变量为上述15个差异小分子化合物,死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样大部分用作训练集,少部分用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络二级预测模型c的超参数,得到最优预测模型以及内部验证评价模型;
③建立二级预测模型d
第一步:选择源选项里的excel选项,将用于鉴别的9天、12天和15天的21个差异小分子化合物含量导入,21个差异小分子化合物为dl-色氨酸、异戊胺、5-氨基戊酸、油酸酰胺、苯乙胺、邻羟基肉桂酸、n-乙酰-l-苯基丙氨酸、脯氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、胸腺嘧啶、l-苯基丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、甲氨酰胺、2-巯基苯并噻唑、8(s)-羟基-(5z,9e,11z,14z)-二十碳四烯酸、l-丝氨酸、l-酪氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括9天、12天和15天,判别变量为上述21个差异小分子化合物,死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样大部分用作训练集,少部分用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络二级预测模型d的超参数,得到最优预测模型以及内部验证评价模型;
④建立二级预测模型e
第一步:选择源选项里的excel选项,将用于鉴别的18-24天和27-30天的15个差异小分子化合物含量导入,15个差异小分子化合物为dl-色氨酸、异戊胺、5-氨基戊酸、n-乙酰-l-苯基丙氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、缬氨酸、dl-高丝氨酸、尿苷、2-巯基苯并噻唑、l-丝氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量18-24天和27-30天,判别变量为上述15个差异小分子化合物,死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样大部分用作训练集,少部分用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络二级预测模型e的超参数,得到最优预测模型以及内部验证评价模型;
(3)建立第三级预测模型
①建立三级预测模型f:
第一步:选择源选项里的excel选项,将用于鉴别18天、21天、24天死亡时间点的21个差异小分子化合物含量导入,21个差异小分子化合物为dl-色氨酸、缬氨酸、异戊胺、5-氨基戊酸、油酸酰胺、n-乙酰-l-苯基丙氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、胸腺嘧啶、l-苯丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、dl-高丝氨酸、尿苷、2-巯基苯并噻唑、l-丝氨酸、邻羟基肉桂酸、脯氨酸、甲氨酰胺;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括18天、21天和24天;判别变量为上述21个差异小分子化合物;死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样大部分用作训练集,少部分用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络三级预测模型f的超参数,得到最优预测模型以及内部验证评价模型;
②建立三级预测模型g:
第一步:选择源选项里的excel选项,将用于鉴别27天、30天死亡时间点的16个差异小分子化合物含量导入,16个差异小分子化合物为dl-色氨酸、异戊胺、n-乙酰-l-苯基丙氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、胸腺嘧啶、l-苯丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、缬氨酸、dl-高丝氨酸、尿苷、2-巯基苯并噻唑、l-丝氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括27天和30天;判别变量为上述16个差异小分子化合物;死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样大部分用作训练集,少部分用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络三级预测模型g的超参数,得到最优预测模型以及内部验证评价模型;
完成三级串联预测模型的建立。
作为本发明的大鼠死亡时间推断模型的建立方法的技术方案的进一步改进,所述数据库为mzcloud数据库。
作为本发明的大鼠死亡时间推断模型的建立方法的技术方案的进一步改进,在步骤㈠中,在步骤㈠中,在收集完所有死亡后不同时间点小分子化合物后混合制成质控样本;在样本通过uplc-ms技术分析过程中,每10个实验样本后,将空白样本和质控样本各进样一次。
作为本发明的大鼠死亡时间推断模型的建立方法的技术方案的进一步改进,所有通过uplc-ms技术分析的组织样本随机排序进样。
作为本发明的大鼠死亡时间推断模型的建立方法的技术方案的进一步改进,在步骤㈠中,对色谱峰识别、峰对齐是将在compounddiscoverer2.0软件内完成的。
作为本发明的大鼠死亡时间推断模型的建立方法的技术方案的进一步改进,在步骤㈠中,大鼠骨骼肌样本的处理方法为:脏器组织于冰上解冻后,称取200mg±5mg置于800微升的冷乙腈溶液中,利用氧化锆珠进行组织匀浆,放置10分钟后4℃下13000转离心30分钟,取上清液400微升,冻干;冻干物中加入200微升80%乙腈-水后,涡旋振荡1分钟,4℃下13000转离心30分钟,滤过膜过滤后得到的小分子化合物待uplc-ms检测。
作为本发明的大鼠死亡时间推断模型的建立方法的技术方案的进一步改进,所述uplc-ms技术分析的条件为:
色谱条件:采用acquityuplctmhsst32.1mm×100mm色谱柱;色谱柱柱温45℃,流动相组成:a液为0.1%甲酸水,b液为0.1%甲酸乙腈;流速为0.3ml/min;每针进样量为5微升;样品室温度10℃;
质谱条件:采用hesi离子化方式;喷雾电压:正极,3.0kv;负极,2.7kv;毛细管温度320℃;加热器温度300℃;鞘气流速:30abr,辅助气流速:15abr;扫描模式为fullscan/dd-ms2,采集范围为80-1200m/z,正负离子切换采集模式:分辨率采用msfullscan35000fwhm,ms/ms17500fwhm,nce为12.5ev,25ev和37.5ev。
本发明进一步提供了一种关于大鼠死亡时间的差异小分子化合物集合,包括以下差异化合物,dl-色氨酸、缬氨酸、异戊胺、5-氨基戊酸、油酸酰胺、苯乙胺、n-乙酰-l-苯基丙氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、胸腺嘧啶、l-苯丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、dl-高丝氨酸、泛酸、尿苷、2-巯基苯并噻唑、l-酪氨酸、l-丝氨酸、邻羟基肉桂酸、脯氨酸、甲氨酰胺、8(s)-羟基-(5z,9e,11z,14z)-二十碳四烯酸。
本发明进一步提供了一种基于uplc-ms技术推断大鼠死亡时间的方法,是在上述三级串联预测模型内完成的。
作为本发明基于uplc-ms技术推断大鼠死亡时间的方法技术方案的进一步改进,该方法包括以下步骤:
(ⅰ)收集死亡后大鼠骨骼肌样本,提取的小分子化合物,通过uplc-ms技术进行代谢组学检测;对色谱峰识别、峰对齐,获得化合物名称、保留时间、精确质荷比和峰面积,峰面积归一化,获得差异小分子化合物集合;
(ⅱ)将差异小分子化合物的含量导入三级串联预测模型中,进行逐级预测,得到一级预测结果后,导入相应的二级预测模型中,然后导入对应的三级模型中,最后得出预测的死亡时间。
本发明采用uplc-ms技术,探讨了不同死亡时间大鼠骨骼肌中化合物时序性变化,将检测并筛选出的差异小分子化合物用于建立大鼠死亡时间预测模型,能够提高死亡时间模型的准确性和普适性,但是由于多个指标之间具有潜在的复杂相关关系,化合物之间在某一个死亡时间段所蕴含的信息在一定程度上存在重叠,使用不同的化合物针对不同的死亡时间段分段逐步建立预测模型,能够有效的提高死亡时间预测的精准性。涉及了0天至30天内的死亡时间且时间点划分地也较为紧密,这是在代谢组学用于死亡时间推断的研究中未有的。此外,本发明有力地补充了晚期死亡时间(死亡时间≥9天)推断方向的研究,并为晚期死亡时间推断提供了重要参考。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
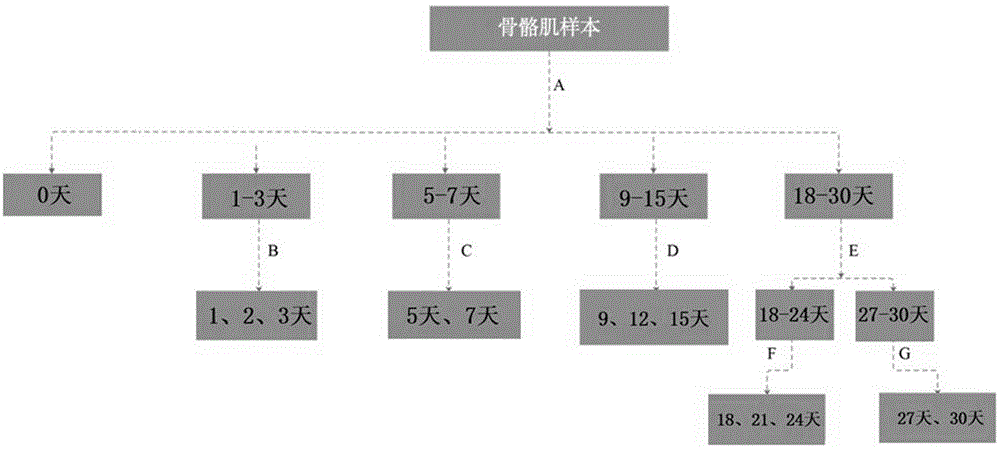
图1为本发明所述三级串联预测模型的流程图。
具体实施方式
为使本发明的目的、技术方案和优点更加清楚,下面将对本发明的技术方案进行详细的描述。显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动的前提下所得到的所有其它实施方式,都属于本发明所保护的范围。
1、不同死亡时间骨骼肌样本制备:
健康成年sprague-dawley大鼠(10~12周龄,体重250g~300g)随机分为1个对照组(死亡即时组)、13个死亡组,每组动物各9只,共14个时间点。3﹪戊巴比妥钠腹腔注射(0.13ml/100g体重)麻醉,将大鼠颈椎脱臼致死,置于气候箱内,气候箱条件模拟太原当地春秋季情况,即温度16℃,湿度50%,光照条件为昼夜12小时交替。在预定时间点(死亡即时(0天)、死亡1、2、3、5、7、9、12、15、18、21、24、27、30天),分别取每组9只大鼠的骨骼肌,锡纸包裹后液氮速冻,之后置于-80℃冰箱用于下一步检测。
2、不同死亡时间骨骼肌样本小分子化合物的提取:
每只大鼠的肌肉组织于冰上解冻后,分别称取200mg±5mg置于800微升的冷乙腈溶液ep管中,管内加入两粒氧化锆珠进行组织匀浆(30次/秒×30秒×5次),放置10分钟后4℃下13000转离心30分钟,取上清液400微升,冻干4小时。冻干物中加入200微升80%乙腈-水(v/v)后,涡旋振荡1分钟,4℃下13000转离心30分钟,滤过膜过滤后得到的小分子化合物待uplc-ms检测。
3、质控样本的制备
从步骤2中每一样本小分子化合物溶液中吸取10ul液体混合后制成质控样本;在样本通过uplc-ms技术分析过程中,每10个实验样本(对照组和死亡组)后,将空白样本(纯色谱乙腈)和质控样本各进样一次,以评价uplc-ms条件及仪器的稳定性,可以减少在整个分析过程中可能产生的仪器系统误差,所有被检测的组织样本随机排序进样。
4、不同死亡时间骨骼肌样本小分子化合物检测分析uplc-ms条件:
色谱条件:本发明采用acquityuplctmhsst3色谱柱(2.1mm×100mm)(美国waters公司);色谱柱柱温45℃,流动相组成:a液为0.1%甲酸水,b液为0.1%甲酸乙腈;流速为0.3ml/min;每针进样量为5微升。样品室温度10℃。梯度洗脱程序见表1。
表1梯度洗脱程序
质谱条件:采用hesi离子化方式:喷雾电压:正极,3.0kv;负极,2.7kv。毛细管温度320℃;加热器温度300℃;鞘气流速:30abr,辅助气流速:15abr;扫描模式为fullscan/dd-ms2,采集范围为80-1200m/z,正负离子切换采集模式:分辨率采用msfullscan35000fwhm,ms/ms17500fwhm,nce为12.5ev,25ev和37.5ev。
5、不同死亡时间骨骼肌样本小分子化合物uplc-ms检测数据预处理:
经uplc-ms分析检测后获得的原始数据.raw文件导入compounddiscoverer2.0软件(美国thermo公司),进行数据提取,并自动完成色谱峰识别、峰对齐等前处理过程。最后输出的数据包括化合物名称、保留时间、精确质荷比和峰面积等。为消除样本之间的差异以及仪器所造成的误差,将上述所得到的数据导入excel中进行峰面积归一化。
6、不同死亡时间骨骼肌特征差异小分子化合物的筛选:
将从compounddiscover2.0导出的数据归一化处理,而后导入simca-p(14.0)中进行多元统计学分析,包括pca、opls-da和置换检验。对数据进行偏最小二乘法(opls-da)回归分析,观察不同死亡时间之间存在一定线性关系。采用置换检验(n=200)来检查模型是否过拟合。对归一化数据进行t检验,vip值大于1且p<0.05的化合物视为有统计学意义,筛选用于死亡时间推断数学模型的潜在差异化合物集合,并根据和mzcloud数据库中二级质谱图比对,共确定了25种内源性化合物作为差异小分子化合物集合,差异小分子化合物的具体信息如下:
表1差异小分子化合物信息
7、大鼠死亡时间预测模型的建立:
打开clementine12.0,分步建立三级串联预测模型:
(1)建立一级预测模型a
第一步:选择源选项里的excel选项,将用于预测0天、1-3天、5-7天、9-15天和18-30天死亡时间段的19个差异小分子化合物含量导入,19个差异小分子化合物为dl-色氨酸、异戊胺、油酸酰胺、邻羟基肉桂酸、n-乙酰-l-苯基丙氨酸、脯氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、l-苯丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、泛酸、尿苷、2-巯基苯并噻唑、8(s)-羟基-(5z,9e,11z,14z)-二十碳四烯酸、l-酪氨酸、l-丝氨酸;
第二步:选择字段选项里的类型选项,判别变量为19个差异小分子化合物,死亡时间分组变量包括a:0天,b:1-3天,c:5-7天,d:9-15天,e:18-30天,死亡时间分组为输出变量,依次读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样80%用作训练集,20%用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络一级预测模型a的超参数,得到最优预测模型以及内部验证评价模型;
(2)建立二级预测模型
①建立二级预测模型b
第一步:选择源选项里的excel选项,将用于鉴别1天、2天、3天死亡时间点的14个差异小分子化合物含量导入,14个差异小分子化合物为dl-色氨酸、5-氨基戊酸、异戊胺、油酸酰胺、n-乙酰-l-苯基丙氨酸、脯氨酸、甲硫氨酸、吲哚-3-乳酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、缬氨酸、尿苷、l-酪氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括1天、2天和3天;判别变量为上述14个差异化合物;死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样80%用作训练集,20%用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络二级预测模型b的超参数,得到最优预测模型以及内部验证评价模型;
②建立二级预测模型c的步骤为:
第一步:选择源选项里的excel选项,将用于鉴别5天、7天死亡时间点的15个差异小分子化合物含量导入,15个差异小分子化合物为dl-色氨酸、5-氨基戊酸、油酸酰胺、邻羟基肉桂酸、n-乙酰-l-苯基丙氨酸、脯氨酸、甲硫氨酸、吲哚-3-乳酸、l-苯基丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、2-巯基苯并噻唑、l-丝氨酸、l-酪氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括5天和7天,判别变量为上述15个差异小分子化合物,死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样80%用作训练集,20%用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络二级预测模型c的超参数,得到最优预测模型以及内部验证评价模型;
③建立二级预测模型d
第一步:选择源选项里的excel选项,将用于鉴别的9天、12天和15天的21个差异小分子化合物含量导入,21个差异小分子化合物为dl-色氨酸、异戊胺、5-氨基戊酸、油酸酰胺、苯乙胺、邻羟基肉桂酸、n-乙酰-l-苯基丙氨酸、脯氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、胸腺嘧啶、l-苯基丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、甲氨酰胺、2-巯基苯并噻唑、8(s)-羟基-(5z,9e,11z,14z)-二十碳四烯酸、l-丝氨酸、l-酪氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括9天、12天和15天,判别变量为上述21个差异小分子化合物,死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样80%用作训练集,20%用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络二级预测模型d的超参数,得到最优预测模型以及内部验证评价模型;
④建立二级预测模型e
第一步:选择源选项里的excel选项,将用于鉴别的18-24天和27-30天的15个差异小分子化合物含量导入,15个差异小分子化合物为dl-色氨酸、异戊胺、5-氨基戊酸、n-乙酰-l-苯基丙氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、缬氨酸、dl-高丝氨酸、尿苷、2-巯基苯并噻唑、l-丝氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量18-24天和27-30天,判别变量为上述15个差异小分子化合物,死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样80%用作训练集,20%用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络二级预测模型e的超参数,得到最优预测模型以及内部验证评价模型;
(3)建立第三级预测模型
①建立三级预测模型f:
第一步:选择源选项里的excel选项,将用于鉴别18天、21天、24天死亡时间点的21个差异小分子化合物含量导入,21个差异小分子化合物为dl-色氨酸、缬氨酸、异戊胺、5-氨基戊酸、油酸酰胺、n-乙酰-l-苯基丙氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、胸腺嘧啶、l-苯丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、dl-高丝氨酸、尿苷、2-巯基苯并噻唑、l-丝氨酸、邻羟基肉桂酸、脯氨酸、甲氨酰胺;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括18天、21天和24天;判别变量为上述21个差异小分子化合物;死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样80%用作训练集,20%用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络三级预测模型f的超参数,得到最优预测模型以及内部验证评价模型;
②建立三级预测模型g:
第一步:选择源选项里的excel选项,将用于鉴别27天、30天死亡时间点的16个差异小分子化合物含量导入,16个差异小分子化合物为dl-色氨酸、异戊胺、n-乙酰-l-苯基丙氨酸、甲硫氨酸、吲哚-3-乳酸、(+/-)12(13)-dihome、胸腺嘧啶、l-苯丙氨酸、dl-苹果酸、还原型l-谷胱甘肽、n-乙酰-dl-色氨酸、缬氨酸、dl-高丝氨酸、尿苷、2-巯基苯并噻唑、l-丝氨酸;
第二步:选择字段选项里的类型选项,死亡时间分组变量包括27天和30天;判别变量为上述16个差异小分子化合物;死亡时间分组设置为输出变量,读入数据;
第三步:选择记录选项里的样本选项,将原始数据随机抽样80%用作训练集,20%用作内部验证集;
第四步:选择建模选项里的神经网络选项,然后运行,训练神经网络三级预测模型g的超参数,得到最优预测模型以及内部验证评价模型;
完成三级串联预测模型的建立。
8、预测模型的结果及评价
内部验证集能够验证评价所建立的预测模型的准确率,当准确率较高时,则认为该模型为最优预测模型;当准确率较低时,可通过不断调整超参数来获得准确率较高的最优预测模型。在本发明中,该准确率并不统一或固定,可能不同模型的准确率不同。
另外,在本发明中每次随机抽样大部分作为训练集,剩余少部分作为内部验证集,且训练集和内部验证集的百分率占比并不作特定要求,可以是80%和20%,也可以是70%和30%。
预测模型在骨骼肌样本数据集上运行10次(每次随机选择样本的80%为训练集,20%为内部验证集),最终选择预测准确率评价模型。不同模型的内部验证的准确率分别为:预测模型a为85.71%;预测模型b为75%;预测模型c为50%;预测模型d为100%;预测模型f为60%;预测模型g为100%。
9、采用外部验证数据评价三级串联预测模型
将28个已知死亡时间的大鼠骨骼肌样本,提取小分子化合物,获得各个样本的25个差异小分子化合物的含量(获取方法同上述1至5步),将差异小分子化合物的含量导入三级串联预测模型中,进行逐级预测,得到一级预测结果后,导入相应的二级预测模型中,然后导入对应的三级模型中,最后得出预测的死亡时间。
结果显示,共有28个外部验证样本,预测错误7个样本,预测正确21个样本。结果表明在预测死亡时间小于等于9天的样本时,模型表现出了良好的预测能力(89%);在预测死亡时间大于9天的样本时,模型的预测准确性有所下降(64.3%)。虽然死亡时间大于9天时外部验证准确率不高,但是死亡时间小于9天时,预测准确率能够达到目前要求的结果。
10、特殊情况的考量
以死亡时间为6天的大鼠骨骼肌样本为例,将其25个差异小分子化合物的含量导入三级串联预测模型中,进行逐级预测,将其中19个差异小分子化合物的含量导入至一级预测模型a预测后,输出结果为c,然后将15个差异小分子化合物的含量导入至二级预测模型c预测后,输出结果为5天。由于三级串联预测模型输出的结果与实际死亡时间相差不大,因此该模型的输出结果为可接受的。
以上所述,仅为本发明的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,可轻易想到变化或替换,都应涵盖在本发明的保护范围之内。因此,本发明的保护范围应以所述权利要求的保护范围为准。
- 还没有人留言评论。精彩留言会获得点赞!