使用母体血清DNA预测胎儿单基因遗传变异的方法与流程
本发明涉及一种通过对母体血清DNA测序来预测胎儿单基因遗传变异的方法。更具体地,本发明涉及一种基于母体血清DNA测序而非侵入性地预测胎儿单基因遗传变异及其导致的单基因疾病的方法。
背景技术:
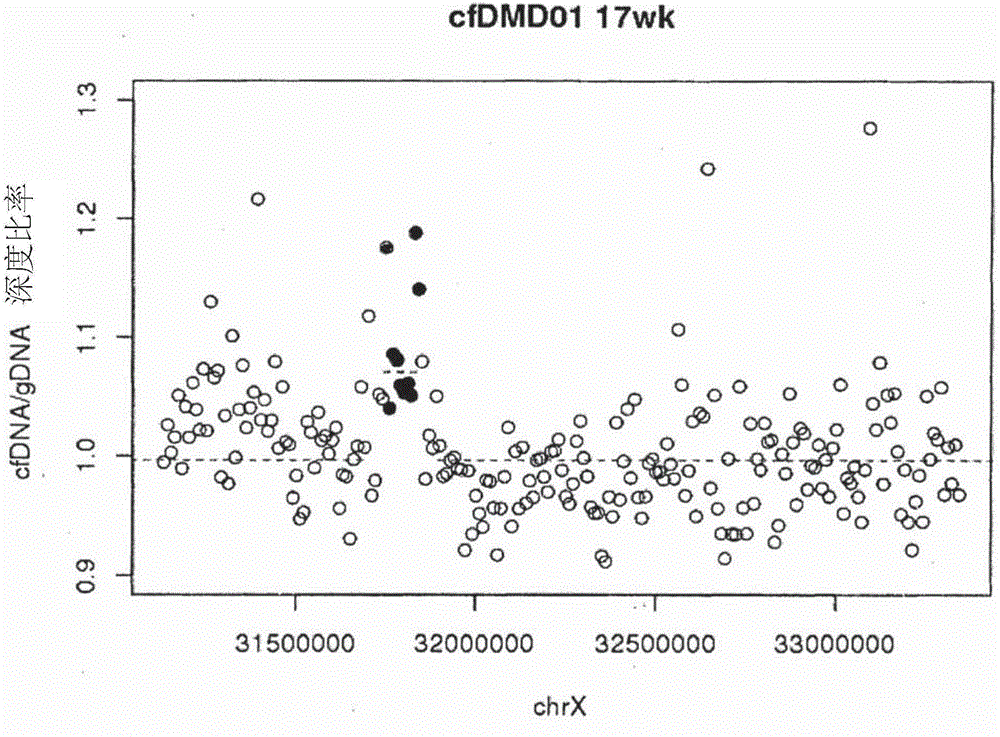
:最近,由于畸形胎儿出生率的增加和各种产前诊断设备的开发,人们对产前诊断的关心与日俱增。特别地,推荐这样的孕妇接受产前诊断:其年龄在35岁以上、其分娩过染色体异常的胎儿、其出生时具有染色体结构异常或其胎儿是与具有染色体结构异常的男性所怀的、其具有家族遗传疾病史、其具有神经管缺陷的风险、或其在母体血清筛查和超声波检查中被怀疑具有胎儿畸形。产前诊断方法可大致分为侵入性诊断方法和非侵入性诊断方法。由于侵入性诊断方法在诊断过程中对胎儿施加影响而有可能引起流产、疾病或畸形等,因此,已开发了非侵入性诊断方法作为替代方法来克服此类问题。在母体血浆的游离DNA(cfDNA)中的游离胎儿DNA(cffDNA)的发现为开发非侵入性的产前遗传诊断方法提供了强有力的工具。大规模平行测序(也称为新一代测序)的引入进一步促进了cffDNA在产前诊断中的应用。此外,一些研究通过全基因组测序(WGS)和cffDNA目标富集后的测序证实了胎儿和母体DNA在整个基因组上是均匀分布的(LoYMetal.,ScienceTranslationalMedicine2010;2:61ra91;LiaoGJetal.,ClinicalChemistry2011;57:92-101;KitzmanJOetal.,ScienceTranslationalMedicine2012;4:137ra76)。这些结果提供了可将检测扩展至单基因疾病的基础,单基因疾病比染色体非整倍性更为常见。然而,相对于非整倍性检测在临床实践中的便利的应用,应用于单基因疾病还有很多复杂的障碍要克服。cffDNA在母体血浆中的含量很低而且其在不同母体血浆中的含量变化较大,因而很难在单核苷酸的水平上可靠地检测出胎儿变体。技术实现要素:技术问题本发明旨在提供一种通过使用母体血清DNA测序而简便地检验、检测或诊断胎儿的单基因变异的方法,或预测或诊断与所述单基因变异相关的单基因疾病的方法、或提供与所述单基因变异相关的单基因疾病的预测/诊断信息的方法。技术方案本发明涉及一种通过母体血清DNA测序来预测胎儿单基因遗传变异的方法。更具体地,本发明涉及一种通过母体血清DNA测序来非侵入性地预测、检测或诊断胎儿单基因遗传变异及其导致的单基因疾病的方法。本发明的一个实施方案涉及一种使用母体血清RNA检测胎儿遗传变异的方法,所述方法包括:通过母体血清DNA的测序分析,在母体单基因中定义基因缺失或重复型遗传突变类型和遗传变异区域,并在具有所述遗传变异的母体血清DNA的变异区域中使用杂合SNP的等位基因频率来确定胎儿是否在单基因疾病相关的单基因中具有基因缺失或重复型遗传变异的步骤。本发明的另一个实施方案涉及一种使用母体血清RNA来检测胎儿遗传变异的方法,所述方法包括:通过母体血清DNA的测序分析,在母体单基因中定义基因缺失或重复型遗传突变类型和遗传变异区域,并使用通过新一代测序法分析具有所述遗传变异的母体的血清DNA和血球(corpuscle)DNA而获得的读码深度比(readdepthratio)(血清读码深度/血球读码深度),并应用该读码深度比来确定胎儿是否在单基因疾病相关的单基因中具有基因缺失或重复型遗传变异的步骤。随着DNA测序技术的发展,可解读大量的遗传信息,并且基于新一代测序(NGS)技术的基因组分析方法被应用于产前分析领域中。特别地,基于母体血液包含一定水平的胎儿基因组这一事实,母体血液样品可用于分析胎儿基因组信息。该方法简便,对母体或胎儿没有影响,并可用作早期预测胎儿遗传变异的产前诊断方法。有益效果本发明的方法能够以非侵入性的方式,通过对携带者(carrier)母体的血清DNA测序而在胎儿出生前(从怀孕后6周以后)预测胎儿单基因遗传变异,该方法有效且简便,因而能够代替传统的非侵入性方法(如绒毛膜绒毛取样和羊膜穿刺术)而应用于产前诊断。附图说明图1示出了从本发明具体实施方式中获得的等位基因频率相对胎儿DNA分数的图,其中上面的线是指在单基因中具有突变的胎儿的等位基因频率,下面的线是指在单基因中不具有突变的胎儿的等位基因频率。图2示出了从本发明具体实施方式中获得的单基因中变异和非变异区域中的血清读码深度与血球读码深度的比率(血清读码深度/血球读码深度,γ),其中X轴为X-染色体上的核苷酸位置,Y轴表示变异区域(实心圆)和非变异区域(空心圆)的相对读码深度比(γ)和各区域的代表值(虚线)。具体实施方式在下文中,将更详细地描述本发明。本发明涉及一种通过母体血清DNA测序来检验、检测或诊断胎儿单基因遗传变异的方法。本方法简便,因为它不需要父系或雄性系DNA。具体地,使用母体血清DNA检测胎儿单基因遗传变异的方法包括:根据母体血清DNA的测序分析数据,在单基因疾病相关联的母体单基因中定义基因缺失或重复型遗传突变类型及变异发生的区域(步骤A),使用具有遗传变异的母体血清DNA,依据所述变异区域或非变异区域(正常区域)中的胎儿遗传变异来分析有区别的遗传性状(步骤B),以及确定胎儿是否在单基因疾病相关的单基因中具有基因缺失或重复型遗传变异(步骤C)。根据本发明的一些实施方案,所述使用具有遗传变异的母体血清DNA来分析有区别的遗传性状的步骤B可通过两种方式进行。第一种,对具有遗传变异的母体血清DNA测量变异区域中杂合SNP的等位基因频率,得到等位基因频率的测量分布。通过使用母体血清DNA中胎儿DNA分数(f)和孟德尔定律来计算并获得对于存在及不存在胎儿遗传变异的单基因的变异区域中杂合SNP的等位基因频率的预测值。然后,使用所述测量分布和预测值来确定胎儿是否具有遗传突变。第二种,通过新一代测序法来分析母体血清DNA和母体血球DNA(母体基因组DNA)的序列信息,从各母体血清DNA和母体血球DNA中获得变异区域和非变异区域中的读码深度,并通过获取并比较血清读码深度与血球读码深度的比率(血清读码深度/血球读码深度),来确定是否存在胎儿遗传突变。在本发明的一些实施方案中,使用遗传变异区域中的杂合SNP的等位基因频率的方法可如下进行:分析母体血清DNA以获得母体测序数据;通过使用所述测序数据来定义在母体单基因疾病相关联的单基因中的缺失或重复型遗传突变及变异区域;通过在具有所述遗传变异的母体血清DNA中测量变异区域中杂合SNP的等位基因频率,而得到等位基因频率的测量分布;获得母体血清DNA中的胎儿DNA分数(f)并使用孟德尔定律来计算单基因的变异区域中杂合SNP的等位基因频率的预测值,当胎儿具有单基因的遗传变异时将其作为第一预测值(θexp|aff),当胎儿不具有单基因的遗传变异时将其作为第二预测值(θexp|unaff);以及基于所述第一预测值(θexp|aff)和第二预测值(θexp|unaff)是否落入等位基因频率的测量分布的统计学显著区间来确定胎儿是否在单基因疾病相关的单基因上具有基因缺失或重复型遗传突变。在下文中将分步对本发明的检测方法分进行更详细的说明。(步骤A)此步骤为通过使用母体血清DNA的测序分析数据来定义在单基因疾病相关的母体单基因中的基因缺失或重复型遗传突变及所述遗传变异发生区域的步骤。由于本发明的方法基于对母体血清中X染色体连锁(X-linked)疾病即由单基因遗传变异引起的疾病的等位基因频率的分析,本领域技术人员可容易地理解本发明的方法可用于X染色体连锁的隐性疾病或常染色体的隐性疾病。所述由单基因遗传变异引起的疾病的实例可以是单基因疾病,包括但不限于杜氏肌营养不良、佩利措伊斯-梅茨巴赫病、肌管性肌病、眼脑肾综合征、门克斯综合征、X-连锁肾上腺脑白质营养不良、Hoyeraal-Hreidarsson综合征、脊髓性肌肉萎缩症、异染性脑白质病变和克拉伯病。在本发明的方法中,术语“携带者母体”或“目标母体”是指在X染色体中具有本发明的方法所要确认的单基因遗传突变的母体,并且由于所述携带者母体仅在其两条X(XX)染色体中的一条中具有隐性单基因突变(X’),所以其不出现所述单基因疾病。在本发明中,携带者母体是怀有胎儿的母体,优选怀孕6周以上的母体。所述携带者母体的游离DNA包含游离胎儿DNA。可通过调查相应单基因疾病的家族史或对基因组DNA测序并将序列与参照DNA序列比对来确定所述携带者母体是否为携带者,所述参照DNA序列是正常DNA序列或具有相应的单基因突变。本身为携带者并且可在其怀孕期间获得游离DNA的母体是本发明的实验对象。可通过新一代测序方法进行所述血清DNA的测序,该方法的实例包括但不限于目标富集和大规模平行测序。可使用许多熟知的结构变异检测程序来获知所述遗传突变的类型(插入或缺失)和区域。这些结构变异检测程序的实例包括但不限于Delly、Pindell、BreakDancer、GASV、Hydra和CNVnator。在一个具体的实施方案中,优选可通过以下的步骤来确定所述遗传突变的类型和区域:大规模平行测序后,对窗口尺寸为10kb读码点的重叠滑动窗(overlappingslidingwindow)计算读码深度的移动平均值;以及应用CBS(循环二元分割)算法。例如,单基因中基因缺失或重复型遗传突变及变异区域的确定可如下进行:通过新一代测序法分析母体血清DNA而获得的读码深度来计算移动平均值,并应用CBS(循环二元分割)算法。任选地,所述方法还可包括以下步骤:使用已知方法预先消除读码深度数据中由基因区域的GC含量差异而导致的变异。(步骤B和步骤C)步骤B利用母体血清DNA的遗传变异区域中杂合SNP的等位基因频率,包含使用SNP等位基因频率的方法(B-1)和使用单基因的变异和非变异区域的血清读码深度与血球读码深度的比率(血清读码深度/血球读码深度)的方法(B-2)。在一个实施方案中,用于获得等位基因频率的测量分布和预测值的等位基因是相同的。例如,可在获得等位基因频率的测量分布以及获得所述等位基因频率的预测值的两个步骤中都使用等位基因频率小于0.5的次要等位基因或等位基因频率大于0.5的主要等位基因。当将次要等位基因和主要等位基因一起使用来获得等位基因频率的测量分布和预测值时,不论是否存在胎儿遗传变异,测量分布和预测值都集中在0.5,因此,这不但使基于假设的差异变小,而且还使预测值的计算复杂化。适于使用SNP等位基因频率的步骤B的第一子步骤(B-1)可包含如下步骤:(B-11)通过测量具有遗传变异的母体血清DNA在变异区域中的杂合单核苷酸多态性(SNP)的等位基因频率,以得到等位基因频率的测量分布,(B-12)获得母体血清DNA中的胎儿DNA分数(f)并使用孟德尔定律来计算单基因的变异区域中杂合SNP的等位基因频率的预测值,当胎儿在单基因中具有遗传变异时,将其作为第一预测值(θexp|aff),当胎儿在单基因中不具有遗传变异时,将其作为第二预测值(θexp|unaff),以及(B-13)基于第一预测值(θexp|aff)和第二预测值(θexp|unaff)是否落入等位基因频率的测量分布的统计学显著区间的事实,来确定胎儿是否在单基因疾病相关的单基因上具有基因缺失或重复型遗传突变。任选地,所述子步骤B-11还可包括使用已知方法消除等位基因频率值中的异常值的步骤。所述子步骤B-11被设计为从母体血清DNA测序数据中测量具有单基因遗传变异(重复/缺失遗传变异)的DNA区域中杂合SNP的平均等位基因频率(θobs)。可使用如Samtools的常规程序通过对新一代测序数据中各等位基因的数目进行计数来计算等位基因频率。在所述子步骤B-12中,获得携带者母体的血清DNA中的胎儿DNA分数(f),并可使用多种常规方法之一来测定所述胎儿DNA分数。例如,可如下获得所述母体血清DNA中的男性胎儿DNA分数(f):采用捕获探针捕获ZFX和ZFY基因,所述捕获探针可分别捕获X连锁的锌指(Zfx)基因区域和Y连锁的锌指(Zfy)基因区域。对于男性胎儿,更详细地,可使用靶向ZFX(X连锁的锌指蛋白)和ZFY(Y连锁的锌指蛋白)基因区域的常见捕获探针来捕获ZFX和ZFY基因,然后根据以下方程计算:胎儿DNA分数:其中,ZFX′和ZFY′是总的匹配的读码数目除以探针数目的商。可使用在非变异区域中的中位值为0.1、范围为0.02至0.3的等位基因频率上形成的SNP等位基因频率的分布来获得所述母体血清DNA中的女性胎儿DNA分数(f)。在所述子步骤B-12中,获得母体血清DNA中的胎儿DNA分数(f)并应用孟德尔定律来获得所述单基因的变异区域中杂合SNP的等位基因频率的预测值,当胎儿在单基因中具有遗传变异时,将其作为第一预测值(θexp|aff),当胎儿在单基因中不具有遗传变异时,将其作为第二预测值(θexp|unaff)。可根据染色体的类型(如常染色体和异染色体)、孟德尔氏遗传的类型以及在异染色体的情况下根据胎儿的性别,并在母体为携带者的假设下,建立方程,根据该方程计算所述第一预测值和第二预测值。例如,当胎儿是具有X染色体连锁的隐性疾病的男性时,可根据以下方程1和2来计算第一预测值(θexp|aff)和第二预测值(θexp|unaff):<方程1>θexp|aff=2/(3-f)<方程2>θexp|unaff=2(1-f)/3-2f所述子步骤B-13被设计为使用第一预测值(θexp|aff)和第二预测值(θexp|unaff)是否落入等位基因频率的测量分布的统计学显著区间内,来确定胎儿是否在单基因疾病的单基因中具有基因缺失或重复的遗传突变。例如,当测得的平均等位基因频率(θobs)更接近具有单基因遗传变异的胎儿的等位基因频率的预测值(θexp|aff)时,预测携带者母体中的胎儿具有单基因遗传变异。另一方面,当测得的平均等位基因频率(θobs)更接近不具有单基因遗传变异的胎儿的等位基因频率的预测值(θexp|unaff)时,预测携带者母体中的胎儿不具有单基因遗传变异。对于统计学显著性,测得的等位基因频率(θobs)的显著性区域是在其为二项式分布的假设下计算的。如果两个预测值都在显著性区域内,则认为该预测不具有统计学显著性。在本发明的另一些实施方案中,步骤B的第二子步骤B-2——其被设计为使用单基因的变异区域和非变异区域中的血清读码深度与血球读码深度的比率(血清读码深度/血球读码深度)——可如下进行:使用母体血清DNA的测序分析数据来定义母体单基因疾病相关的单基因中关于基因突变(缺失或重复)类型的遗传变异及该遗传变异发生的区域;从具有遗传变异的母体的血清DNA测序数据中获得单基因中变异区域和非变异区域中的各血清读码深度;从具有遗传变异的母体的血球DNA测序数据中获得单基因中变异区域和非变异区域中的各血球读码深度;计算单基因中变异区域中的血清读码深度与血球读码深度的第一比率(血清读码深度/血球读码深度)及单基因中非变异区域中血清读码深度与血球读码深度的第二比率(血清读码深度/血球读码深度);以及将第一比率和第二比率进行比较,确定胎儿是否在单基因中具有单基因疾病相关的基因缺失或重复型遗传突变。任选地,获得所述第一和第二比率的子步骤还可包括使用已知方法消除读码深度数据中由基因区域的GC含量差异而导致的变异,并使用已知方法消除读码深度数据中等位基因频率值的异常值。在使用母体血清DNA的测序分析数据来定义关于遗传变异的类型和区域的步骤中,可通过分析母体血清DNA的测序信息并将其用于在单基因疾病相关的单基因中定义遗传变异的类型(缺失或重复)及遗传变异的区域。所述母体血清DNA的测序分析可通过获得母体血清DNA并对所述血清DNA进行测序而进行。可使用与步骤A中相同的方式进行所述血清DNA的测序分析及遗传变异类型和区域的确定。使用新一代测序(NGS)技术来分析具有单基因遗传变异的携带者母体的血清DNA和基因组DNA,以确定如上所描述的单基因遗传变异的类型和区域。该方法可应用于重复和缺失型突变两种类型。可采用与步骤A中相同的方式处理步骤B-2中所使用的疾病、母体和单基因。详细地,使用单基因的变异和非变异区域中血清读码深度与血球读码深度的比率(血清读码深度/血球读码深度)的方法可通过以下步骤来实施:(B-21)从具有遗传变异的母体的血清DNA测序数据中获得单基因中变异区域和非变异区域中的各血清读码深度;(B-22)从具有遗传变异的母体的血球DNA测序数据中获得单基因中变异区域和非变异区域中的各血球读码深度;(B-23)计算所述单基因变异区域中血清读码深度与血球读码深度的第一比率(血清读码深度/血球读码深度)以及所述单基因非变异区域中血清读码深度与血球读码深度的第二比率(血清读码深度/血球读码深度);以及(B-24)将所述第一比率和第二比率进行比较,确定胎儿是否在单基因疾病相关的单基因中具有基因缺失或重复型遗传突变。为了用于子步骤B-21——从具有遗传变异的母体的血清DNA测序数据中获得母体血清DNA的单基因中变异区域和非变异区域中各血清读码深度——中,可使用如步骤A中所描述的新一代测序技术来获得测序信息和读码深度。例如,可使用包括目标富集和大规模平行测序的新一代测序方法。可采用与所述子步骤B-21相同的方式进行子步骤B-22的从具有遗传变异的母体的血球DNA测序数据中获得母体血球DNA的单基因中变异区域和非变异区域的各血球读码深度,不同之处是使用血球DNA代替血清DNA。例如,获得母体血球DNA然后使用新一代测序方法分析其序列和读码深度。可通过将所述母体血液样品分离为血清和血球并从所述血清和血球中分离出各自的DNA来获得母体血清DNA和血球DNA。可通过将母体的血液样品进行离心分离来获得所述血球,但不限于此,并可通过溶解所述血球以提取基因组DNA。可使用本领域熟知的常规方法进行DNA分离。将母体血液样品分离为血清和血球使得能够仅从母体取一次血液样品就同时获得血清和血球,因而本方法非常简便。此外,本发明的方法具有不需要父系DNA且对于检测胎儿遗传变异具有高灵敏性和准确性的优点。根据优选的实施方案,可在相同的条件下使用新一代测序方法采用相同的常规捕获探针来分析母体血清DNA和血球DNA,以使测量误差降至最低。当确定了目标单基因时,可容易地选择常规捕获探针,因此不作特别限定。在子步骤B-23和B-24中,通过计算并比较所述单基因的变异区域中血清读码深度与血球读码深度的第一比率(血清读码深度/血球读码深度)和所述单基因的非变异区域中血清读码深度与血球读码深度的第二比率(血清读码深度/血球读码深度),以确定胎儿是否在单基因疾病相关的单基因中具有基因缺失或重复型遗传突变。详细地,采用变异区域中的母体血清DNA读码深度除以母体基因组DNA的读码深度来计算相对血清DNA的读码深度。同样地,采用在不非遗传变异区域中的母体血清DNA的读码深度除以母体基因组DNA的读码深度来计算相对血清DNA读码深度。可使用如Samtools的常规程序来计算读码深度,并根据碱基序列位置计算相对读码深度。根据本发明的一些实施方案,所述第一比率可以是从单基因变异区域中的单个核苷酸中获得的两次或更多次测量的平均值或中位值,而所述第二比率可以是从单基因非变异区域中的单个核苷酸中获得的两次或更多次测量的平均值或中位值。或者,所述第一比率可以是从单基因变异区域中的5至100,000个碱基的单个核苷酸库(bin)中获得的读码深度的移动平均值的两次或更多次测量的平均值或中位值,而所述第二比率可以是从单基因非变异区域中的5至100,000个碱基的单个核苷酸库中获得的读码深度的移动平均值的两次或更多次测量的平均值或中位值。为计算所述读码深度的移动平均值,可将所述核苷酸库设定为重叠的或不重叠的。将所述第一比率和第二比率进行比较来确定胎儿是否在单基因疾病相关的单基因中具有基因缺失或重复型遗传突变。对此,当所述母体遗传变异是缺失型且变异区域的第一比率小于非变异区域的第二比率时,可确定胎儿在单基因中具有缺失型遗传变异。另一方面,当所述母体遗传变异是重复型且变异区域的第一比率大于非变异区域的第二比率时,可确定胎儿在单基因中具有重复型遗传变异。例如,当具有单基因遗传变异的妇女怀有男性胎儿时,血清DNA读码深度与基因组DNA读码深度的比率(相对读码深度:γ)随胎儿是否具有单基因遗传变异而不同,且随胎儿单基因遗传变异类型(如果存在)的不同而不同(参见表5)。从表5可见,当胎儿具有缺失型突变时,变异区域中的γ比正常区域中的小。当胎儿不具有缺失型突变时,变异区域中的γ比正常区域中的大。当胎儿具有重复型突变时,变异区域中的γ比正常区域中的大。另一方面,当胎儿不具有重复型突变时,变异区域中的γ比正常区域中的小。对于具有缺失型遗传突变的母体,当变异区域的第一比率小于非变异区域的第二比率时,确定胎儿在单基因中具有缺失型遗传突变。当母体具有重复型遗传突变,并且变异区域的第一比率大于非变异区域的第二比率时,可确定胎儿在单基因中具有重复型遗传突变。更详细地,当将具有缺失型突变的母体的第一比率与第二比率进行比较时,可从单基因变异区域中的相对读码深度小于单基因非变异区域中的相对读码深度的比较结果,预测胎儿可能具有单基因遗传变异。另一方面,可从单基因变异区域中相对读码深度大于单基因中非变异区域中的读码深度的比较结果,预测胎儿可能不具有单基因遗传变异。对于具有重复型突变的母体,当将所述第一比率和第二比率进行比较时,可从单基因变异区域中的相对读码深度大于单基因非变异区域中的相对读码深度的比较结果,预测胎儿具有单基因遗传变异。相反,可从单基因变异区域中相对读码深度小于单基因非变异区域中的相对读码深度的比较结果,预测胎儿不具有单基因遗传变异。如上文所述,本发明的方法是通过确定母体是否具有单基因缺失或重复型突变之后,比较正常区域中的γ和变异区域中的γ进行的,从而可容易地确定所述单基因遗传突变是否遗传给胎儿,即确定胎儿是否具有单基因遗传突变。如此以来,根据本发明的一个实施方案的方法被设计为通过使用新一代测序方法分析母体血清DNA和基因组DNA来精确地预测胎儿的单基因突变。因此,本发明可通过使用母体血清DNA确定胎儿在单基因中的缺失或重复型遗传突变而提供有关胎儿单基因疾病的诊断信息。通过以下实施例可获得对本发明更好的理解,提供以下实施例是为了举例说明,不应理解为是对本发明的限制。实施例1:胎儿单基因遗传变异的预测<1-1>母体血清DNA测序选择X染色体连锁的隐性疾病作为本实验中处理的单基因疾病。假设怀孕中的母体血清DNA在某些区域具有会引起遗传疾病的重复型突变。假设母体的目标测序覆盖范围约为95%,平均血清DNA读码深度约为100,平均基因组DNA读码深度约为100。<1-2>遗传变异类型和区域的鉴定为鉴定单基因突变的类型和区域,使用CBS算法计算并处理读码深度的移动平均值。由此,确定区域并发现母体具有重复型突变。<1-3>胎儿DNA分数的测量如实施例<1-1>中所述测量母体DNA的胎儿DNA含量。为此,使用含有捕获探针的AgilentSureSelectCustom试剂盒来捕获ZFX和ZFY基因,所述捕获探针靶向ZFX(X连锁的锌指蛋白)和ZFY(Y连锁的锌指蛋白)区域。胎儿DNA分数:其中,ZFX′和ZFY′是总的匹配读码的数目除以探针数目的商。使用ZFX和ZFY基因预测胎儿DNA分数(f)的结果示于下表1中。表1从表1可见,母体I-1的血清中的胎儿DNA分数是8.6%,母体I-2的血清中的胎儿DNA分数是6.4%。<1-4>杂合单核苷酸多态性的等位基因频率的预测假设各母体为重复型突变携带者且怀有男性胎儿,可使用胎儿DNA分数f根据以下方程1获得单基因变异区域中杂合SNP的等位基因频率(θexp/aff)并根据以下方程2获得单基因非变异区域中杂合SNP的等位基因频率(θexp/unaff):<方程1>θexp/aff=2/(3-f)<方程2>θexp/unaff=2(1-f)/(3-2f)图1中绘制了等位基因频率相对胎儿DNA分数的图。上面的直线表示具有单基因突变的胎儿而下面的直线表示不具有单基因突变的胎儿。沿上面直线的灰色区域表示由变异区域中12个SNP的等位基因频率而计算的置信区间和由连续的6和3个SNP的随机样品计算的100个循环(round)而获得的标准误差。将实施例1-3中计算的胎儿DNA分数代入方程,预测的等位基因频率如下表2所示。表2项目母体I-1母体1-2胎儿DNA分数(f,%)8.604%6.484%θexp/aff(具有基因突变的胎儿)0.68635120.6813939θexp/unaff(不具有基因突变的胎儿)0.64638320.6516068<1-5>具有单基因突变的DNA区域中杂合SNP的平均等位基因频率(θobs)的测量可从母体血清DNA的新一代测序数据中,通过对各等位基因的数目进行计数来计算具有单基因突变的DNA区域中杂合SNP的平均等位基因频率(θobs)。来自实施例的数据的计算结果示于下表3中。表3由于表3中计算的SNP的平均等位基因频率(θobs)更接近表2中计算的突变区域的θexp/aff值,因而可预测所述母体怀有的胎儿具有单基因遗传突变。实施例2:胎儿单基因遗传变异的预测<2-1>母体血清DNA的测序假设母体具有会引起隐性遗传疾病的缺失型突变且怀有男性胎儿。使用与实施例1-1中相同的方式,使用大规模平行测序(一种新一代测序)分析所述母体的血清DNA和基因组DNA。母体的目标测序覆盖范围为约97.7%,平均血清DNA读码深度为约465至约530,平均基因组DNA读码深度约为1210。<2-2>遗传变异类型和区域的鉴定为鉴定单基因突变的类型和区域,计算读码深度的移动平均值并应用CBS算法。<2-3>胎儿DNA分数的测量根据记载于实施例<1-3>中的方法测量母体DNA的胎儿DNA含量。将所述测量结果示于下表4中。表4从表4可见,母体II-1的血清中的胎儿DNA分数是5.4%,母体II-2的血清中的胎儿DNA分数是7.3%。<2-4>正常(非变异)和变异区域中血清DNA和血球DNA(基因组DNA)之间的读码深度的比率假设各母体为单基因突变携带者且怀有男性胎儿,根据胎儿是否具有单基因突变,可预测血清DNA读码深度与基因组DNA读码深度的比率,如表5所示。表5f:胎儿DNA分数从表5可见,当胎儿具有缺失型突变时,变异区域中的γ比正常区域中的小。另一方面,当胎儿不具有缺失型突变时,变异区域中的γ比正常区域中的大。因此,当母体单基因突变是缺失型时,可通过比较正常区域和变异区域之间的γ来确定从母体至胎儿的单基因突变的遗传性,即确定胎儿是否具有单基因突变。此外,当胎儿具有重复型突变时,变异区域中的γ比正常区域中的γ大。另一方面,胎儿不具有重复型突变时,变异区域中的γ比正常区域中的γ小。因此,当母体单基因突变是重复型时,可通过比较正常区域和变异区域之间的γ来确定从母体至胎儿的单基因突变的遗传性,即确定胎儿是否具有单基因突变。基于所述预测,对正常和变异区域中血清DNA读码深度与血球DNA读码深度的比率进行了比较。实验结果是,母体II-1的正常区域和变异区域中的相对读码深度(γ)分别是0.99632和1.06031。母体II-2的正常区域中的相对读码深度(γ)是0.99647,变异区域中的相对读码深度(γ)是1.07075。将母体II-2的相对读码深度(γ)示于图2中。在图2中,X轴表示X染色体上的核苷酸位置而Y轴表示相对读码深度(γ)。示出了变异区域(实心圆)和非变异区域(空心圆)中的读码深度比率,并给出了各区域的代表值(虚线)。对于具有缺失型突变的母体II-1,预测胎儿是正常的,因为变异区域中的相对读码深度比正常区域中的相对读码深度大。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1