复杂核酸序列数据的处理和分析的制作方法
本申请是申请日为2012年04月13日、申请号为“201280029331.7”(pct申请号pct/us2012/033686)、发明名称为“复杂核酸序列数据的处理和分析”的分案申请。
对相关申请的交叉引用
本申请要求2011年4月14日提交的美国临时专利申请no.61/517,196的优先权权益,其在此通过提及完整并入。
本申请要求2011年8月25日提交的美国临时专利申请no.61/527,428的优先权权益,其在此通过提及完整并入。
本申请要求2011年10月12日提交的美国临时专利申请no.61/546,516的优先权权益,其在此通过提及完整并入。
发明背景
需要用于分析复杂核酸的改善的技术,例如特别是用于改善序列准确度及用于分析具有经由核酸扩增引入的大量误差的序列的方法。
此外,需要用于测定对高等生物体基因组的亲本贡献,即人基因组的单元型定相(phasing)的改善的技术。用于单元型定相的方法,包括计算方法和实验定相综述于browningandbrowning,naturereviewsgenetics12:703-7014,2011。
发明概述
本发明提供了用于分析源自复杂核酸测序的序列信息的技术(如本文中定义的),其导致单元型定相、误差降低和其它特征,基于算法和分析技术,与长片段读取结果(lfr)技术结合开发。
依照本发明的一个方面,提供了用于测定一种或多种生物体(也就是说,个别生物体或生物体群体)的复杂核酸(例如全基因组)序列的方法。此类方法包括:(a)在一个或多个计算装置接收复杂核酸的多个读取结果;并(b)用计算装置从所述读取结果产生复杂核酸的装配序列,所述装配序列在70,75,80,85,90或95%或更高的响应率时每兆碱基包含小于1.0,0.8,0.7,0.6,0.5,0.4,0.3,0.2,0.1,0.08,0.07,0.06,0.05或0.04假单核苷酸变体,其中通过一种或多种计算装置实施所述方法。在一些方面,计算机可读的非短暂存储介质存储一种或多种顺序的指令,所述指令包含在由一种或多种计算装置执行时引起所述一种或多种计算装置实施此类方法的步骤的指令。
依照一个实施方案(其中此类方法牵涉单元型定相),所述方法进一步包括鉴定装配序列中的多个序列变体,并对序列变体定相(例如70,75,80,85,90,95%或更多的序列变体)以产生定相序列,即对序列变体定相的序列。可以在误差校正背景中使用此类定相信息。例如,依照一个实施方案,此类方法包括将与至少两个(或三个或更多个)定相序列变体的定相不一致的序列变体鉴定为误差。
依照另一个此类实施方案,在此类方法中,接收复杂核酸的多个读取结果的步骤包括从多个等分试样之每个接收多个读取结果的计算装置和/或其计算机逻辑,每个等分试样包含复杂核酸的一个或多个片段。关于提供此类片段的等分试样的信息可用于校正误差或响应碱基,该碱基在其它情况中会是“无响应”。依照一个此类实施方案,此类方法包括计算装置和/或其计算机逻辑,其基于来自两个或更多个等分试样的位置的初步碱基响应(preliminarybasecall),在所述装配序列的所述位置处响应碱基。例如,方法可以包括基于来自至少两个、至少三个、至少四个或超过四个等分试样的初步碱基响应,在所述装配序列的某个位置处响应碱基。在一些实施方案中,此类方法可以包括若碱基响应在至少两个、至少三个、至少四个等分试样或超过四个等分试样中存在,则将其鉴定为真的。在一些实施方案中,此类方法可以包括若碱基响应在至少大多数(或至少60%、至少75%或至少80%)对装配序列中所述位置做出初步碱基响应的等分试样中存在,则将其鉴定为真的。依照另一个此类实施方案,此类方法包括计算装置和/或其计算机逻辑,其在碱基响应在来自两个或更多个等分试样的读取结果中存在三次或更多次时将该碱基响应鉴定为真的。
依照另一个此类实施方案,通过鉴定与每个片段附接的等分试样特异性标签(或等分试样特异性标签组)测定读取结果起源的等分试样。任选地,此类等分试样特异性标签包含误差校正或误差检测代码(例如reed-solomon误差校正码)。依照本发明的一个实施方案,在对片段和附接的等分试样特异性标签测序后,所得的读取结果包含标签序列数据和片段序列数据。若标签序列数据是正确的,即若标签序列匹配用于等分试样鉴定的标签序列,或备选地若标签序列数据具有可以使用误差校正代码校正的一个或多个误差,则可以使用包括此类标签序列数据的读取结果用于所有目的,特别是用于第一计算机方法(例如在一个或多个计算装置上执行),其需要标签序列数据并产生第一输出,包括但不限于单元型定相、样品多路复用、库多路复用、定相或基于正确标签序列数据的任何误差校正方法(例如基于鉴定特定读取结果的起源等分试样的误差校正方法)。若标签序列是不正确的并且不能校正,则不将包含此类不正确标签序列数据的读取结果弃去,而且在第二计算机方法(例如由一个或多个计算装置执行)中使用,所述第二计算机方法不需要标签序列数据,包括但不限于定位、装配和基于集合的统计学,并且产生第二输出。
依照另一个实施方案,此类方法进一步包括:提供复杂核酸的某个区域的第一定相序列的计算装置和/或其计算机逻辑,所述区域包含短串联重复;比较所述区域的第一定相序列的读取结果(例如规则(regular)或配偶-对(mate-pair)读取结果)与所述区域的第二定相序列的读取结果(例如使用序列覆盖)的计算装置和/或其计算机逻辑;和基于所述比较鉴定第一定相序列或第二定相序列之一中短串联重复扩充的计算装置和/或其计算机逻辑。
依照另一个实施方案,所述方法进一步包括计算装置和/或其计算机逻辑,其从生物体的至少一个亲本获得基因型数据,并从读取结果和基因型数据产生复杂核酸的装配序列。
依照另一个实施方案,所述方法进一步包含实施步骤的计算装置和/或其计算机逻辑,所述步骤包括:对所述复杂核酸的第一区比对多个所述读取结果,由此创建比对读取结果间的重叠;鉴定所述重叠内的n个杂合候选物;聚簇2n至4n种可能性的空间或其选定子空间,由此创建多个簇;鉴定两个具有最高密度的簇,每个鉴定的簇包含基本上无噪音的中心;并对所述复杂核酸的一个或多个别的区域重复前述步骤。对每个区域鉴定的簇可以限定重叠群,并且这些重叠群可以彼此匹配以形成重叠群组,一个代表每个单元型。
依照另一个实施方案,此类方法进一步包括提供一定量的复杂核酸,并对复杂核酸测序以产生读取结果。
依照另一个实施方案,在此类方法中,复杂核酸选自下组:基因组、外显子组(exome)、转录物组、甲基化组(methylome)、不同生物体基因组的混合物、和生物体的不同细胞类型的基因组的混合物。
依照本发明的另一个方面,提供了通过任何上述方法产生的装配的人基因组序列。例如,一个或多个计算机可读的非短暂存储介质存储通过任何上述方法产生的装配的人基因组序列。依照另一个方面,计算机可读的非短暂存储介质存储一种或多种顺序的指令,所述指令包含在由一种或多种计算装置执行时引起所述一种或多种计算装置实施任何、一些或所有上述方法的指令。
依照本发明的另一个方面,提供了用于测定人全基因组序列的方法,此类方法包括:(a)在一个或多个计算装置接收所述基因组的多个读取结果;并(b)用所述一个或多个计算装置从所述读取结果产生所述基因组的装配序列,所述装配序列在70%或更大的基因组响应率时包含每千兆碱基小于600个假杂合单核苷酸变体;依照一个实施方案,基因组的装配序列具有70%或更多的基因组响应率和70%或更大的外显子组响应率。在一些方面,计算机可读的非短暂存储介质存储一种或多种顺序的指令,该指令包含在由一种或多种计算装置执行时引起所述一种或多种计算装置实施本文中描述的任何发明方法的指令。
依照本发明的另一个方面,提供了用于测定人全基因组序列的方法,此类方法包括:(a)在一个或多个计算装置接收来自多个等分试样之每个的多个读取结果,每个等分试样包含基因组的一个或多个片段;并(b)用所述一种或多种计算装置从所述读取结果产生所述基因组的定相装配序列,所述装配序列在70%或更大的基因组响应率时包含每千兆碱基小于1000个假单核苷酸变体。在一些方面,计算机可读的非短暂存储介质存储一种或多种顺序的指令,该指令包含在由一种或多种计算装置执行时引起所述一种或多种计算装置实施此类方法的指令。
附图简述
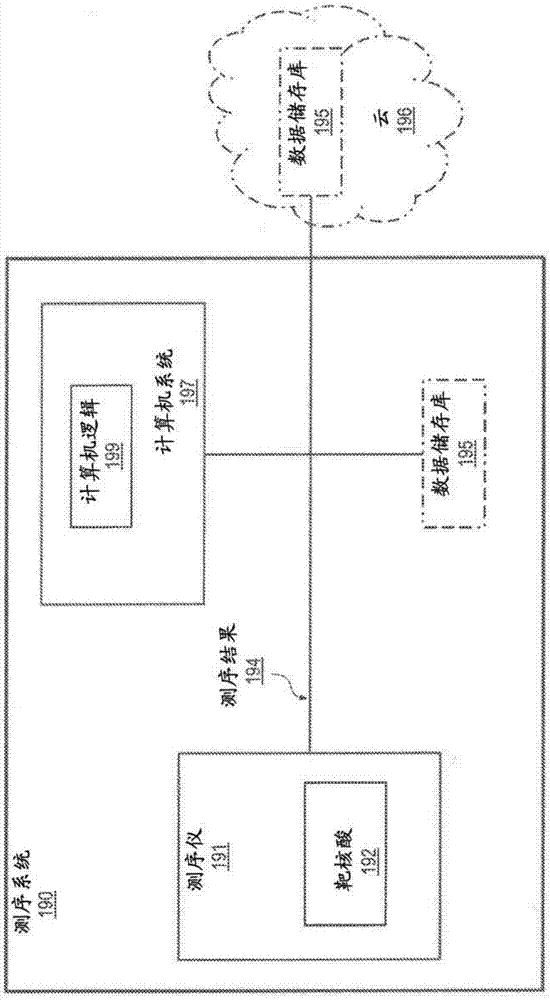
图1a和1b显示了测序系统的例子。
图2显示了可以在测序仪和/或计算机系统中使用或与测序仪和/或计算机系统结合使用的计算装置的例子。
图3显示了lfr算法的一般体系结构。
图4显示了对邻近杂合snp的成对分析。
图5显示了选择假设和将得分归入假设的例子。
图6显示了图构建。
图7显示了图优化。
图8显示了重叠群比对。
图9显示了亲本辅助通用定相。
图10显示了天然的重叠群分离。
图11显示通用定相。
图12显示使用lfr进行的误差检测。
图13显示了降低假阴性数目的方法的例子,其中,不管读取结果的数目是如何的小,也能够产生可信的杂合snp响应。
图14显示了用解析单元型的克隆覆盖法检测人胚胎中ctg重复的扩充(expansion)。
图15的图显示了用多重置换扩增(mda)方案对纯化的基因组dna标准品(1.031、8.25和66皮克[pg])和1或10个pvp40细胞进行的扩增,如实施例1中描述的。
图16显示了用两种mda方案扩增得出的与gc偏爱有关的数据。测定跨整个板的平均循环数目,并将其从每个个别标志物扣除以计算“△循环”数目。相对于每个标志物周围的1000个碱基对的gc含量对△循环绘图以指示每个样品的相对gc偏爱(未显示)。将每个△循环的绝对值求和以创建“△和”量度。较低的△和、以及数据相对于gc含量的相对平坦曲线产生了被较好呈现的全基因组序列。△和是61(对于我们的mda方法)和287(对于sureplex扩增的dna),这指示我们的方案比sureplex方案产生小得多的gc偏爱。
图17显示了样品7c和10c的基因组覆盖。使用相对于单倍体基因组覆盖标准化的100千碱基覆盖窗的10兆碱基移动平均值对覆盖绘图。拷贝数目1和3处的虚线分别代表单倍体和三倍体拷贝数目。这两个胚胎是男性的,并且对于x和y染色体具有单倍体拷贝数目。在这些样品中未发现全染色体或染色体大区段的其它丧失或获得。
图18是用于本发明方法的条形码衔接头设计的实施方案的示意图。lfr衔接头由独特的5’条形码衔接头、共同的5’衔接头和共同的3’衔接头组成。共同的衔接头均设计为具有不能与3’片段连接的3’双脱氧核苷酸,这消除衔接头二聚体的形成。在连接后,将衔接头的封闭部分除去,并用未封闭的寡核苷酸替换。通过随后用taq聚合物进行切口平移并用t4连接酶进行连接来解决剩余的切口。
图19显示了累积gc覆盖图。对lfr和标准库绘制gc的累积覆盖以比较gc偏爱差异。对于样品na19240(a和b),对整个基因组(c)和仅编码部分(d)两者绘制3个lfr库(重复1、重复2、和10个细胞)和1个标准库。在所有lfr库中,高gc区中的覆盖丧失是明显的,其在含有更高比例的富含gc的区域的编码区(b和d)中是更明显的。
图20显示了基因组装配物之间的单元型分型表现的比较。将标准装配库和lfr装配库的变体响应组合,并作为用于定相的基因座来使用,除了规定的情况外。lfr定相率基于亲本定相杂合snp的计算。*对于那些没有亲本基因组数据的个体(na12891、na12892和na20431),通过用定相杂合snp的数目除以预期为真的杂合snp数目(尝试要进行定相的snp的数目–50,000个预期误差),来计算定相率。n50计算基于相对于ncbi构件36(在na1924010个细胞和高覆盖及na20431高覆盖的情况中为构件37)人参照基因组的所有重叠群总装配长度。由于所有dna变性成单链后在384孔板上被分散,致使单倍体片段覆盖比细胞数目大4倍。起始dna不足量解释了na20431基因组中较低的定相效率。#10个细胞的样品用含有超过10个细胞的各个孔(其可能是这些细胞在收集过程中处在细胞周期各个阶段的结果)的覆盖来衡量。定相率范围为84%至97%。
图21显示了lfr单元分型算法。(a)变量提取:从加标签的等分试样的读取结果提取变量。10碱基reed-solomon码确保能经由误差校正而实现标签恢复。(b)杂合snp对的连接性评估:针对某个邻域内的每个杂合snp对,计算共享等分试样的矩阵。环1是一条染色体上的总体杂合snp。环2是染色体上位于环1杂合snp的邻域中的总体杂合snp。此邻域由预期的杂合snp数目和预期的片段长度来限制。(c)图的生成:产生无向图,其中结点对应于杂合snp,而连接(connections)对应于那些snp间关系的最佳假设的方向(orientation)和强度。(如本文中使用的,“结点”是可以具有一个或多个数值的数据[数据项或数据对象],所述数值代表多核苷酸序列中的碱基响应或其它序列变异(例如杂合性或indel(插入缺失))。)方向是二元的(binary)。图21分别描绘了杂合snp对之间的翻转和未翻转的关系。强度通过对共享等分试样矩阵的要素采用模糊逻辑操作来限定。(d)图优化:经由最小跨度树操作来优化所述图。(e)重叠群产生:将每个子树简化成重叠群,这通过使第一杂合snp保持未改变、并使所述子树上的其它杂合snp基于其通向第一杂合snp的路径而翻转或不翻转来进行。对每个重叠群指派亲本1(p1)和亲本2(p2)是任意的。全染色体树中的缺口限定所述染色体上不同子树/重叠群的边界。(f)将lfr重叠群定位到亲本染色体:使用亲本信息,将母亲或父亲标签置于每个重叠群的p1和p2单元型上。
图22显示了重复lfr文库间的单元型不一致性。在所有共享的定相杂合snp基因座处比较来自样品na12877和na19240的两个重复文库。这是全面比较,因为大多数定相基因座在两个文库间是共享的。
图23显示了通过lfr实现的误差降低。单独的标准文库杂合snp响应、以及与lfr响应的组合都独立地通过重复lfr文库来定相。一般地,lfr引入约10倍以上的假阳性变体响应。这最可能因在基于phi29的多重置换扩增期间随机掺入不正确碱基而发生。重要的是,若要求杂合snp响应必须要定相,并且要可见于三个或更多个独立孔中,则误差的降低是显著的,结果也好于没有误差校正的标准文库。lfr也可以从标准文库除去误差,这将响应准确度改善约10倍。
图24显示了无响应的位置的lfr再响应。为了证明lfr挽救无响应的位置的潜力,在染色体18上选择通过标准软件未响应(没有响应)的三个实例位置。通过将它们用作为lfr重叠群一部分的c/t杂合snp定相,可以部分或完全响应这些位置。共享孔(针对成对碱基中每个碱基都有至少一个读取结果的那些孔;一对接受评估的基因座有16对碱基)的分布容许将三个n/n位置再响应到a/n、c/c和t/c响应中,并将c-a-c-t和t-n-c-c限定为单元型。使用孔的信息容许lfr精确响应那些在2-3个预期孔中只有少到2-3个读取结果(比无孔信息的情况少约3倍)的等位基因。
图25显示了在每个分析样品中具有多个不利变异的基因的数目。
图26显示了在na20431中具有等位表达差异、并具有能改变tfbs的snp的基因。在被证明有显著的等位表达差异的非穷尽基因列表中,发现6个基因具有能改变tfbs的snp,其与观察到的等位基因之间的表达差异相关联。相对于ncbi构件37给出所有位置。“cds”代表编码序列,且“utr3”代表3’非翻译区。
发明详述
如本文中及所附权利要求书中使用的,单数形式“一个”、“一种”、和“所述/该”包括复数提及物,除非上下文另有明确规定。如此,例如提及“聚合酶”指一种试剂或此类试剂的混合物,并且提及“所述方法”包括提及本领域技术人员已知的等同步骤和/或方法,等等。
除非另有定义,本文中使用的所有技术和科学术语与本发明所属领域普通技术人员的通常理解具有相同的意义。为了描述及公开出版物中描述并且可以与目前描述的方法结合使用的装置、组合物、配制剂和方法,本文中提及的所有出版物通过提及并入本文。
在提供数值范围的情况下,应当理解本发明内涵盖每个居间数值,除非上下文另有明确规定,至下限单位的十分之一,介于所述范围的上限和下限之间以及该陈述范围中的任何其它陈述或居间数值。这些较小范围的上限和下限可以独立包括在内,较小范围也涵盖在本发明内,服从陈述范围中的任何明确排除界限。在陈述范围包括界限之一或两者的情况中,本发明中还包括排除那些包括的界限两者之任一的范围。
在以下描述中,列出大量具体详情以提供对本发明的更彻底理解。然而,本领域技术人员会显而易见的是,可以在没有一个或多个这些具体详情的情况下实施本发明。在其它情况中,尚未描述公知的特征和本领域技术人员公知的规程以避免使本发明难理解。
虽然本发明主要参照具体实施方案描述,还涵盖的是,在读取结果本公开内容后,其它实施方案对于本领域技术人员会变得显而易见,并且意图此类实施方案包含在本发明方法内。
测序系统和数据分析
在一些实施方案中,可以通过测序系统实施dna样品(例如诸如代表全人基因组的样品)的测序。图1中显示了测序系统的两个例子。
图1a和1b是实例测序系统190的框图,所述测序系统190配置为实施依照本文中描述的实施方案的用于核酸序列分析的技术和/或方法。测序系统190可以包含多个子系统或者与多个子系统联系,所述子系统诸如例如一个或多个测序仪诸如测序仪191、一个或多个计算机系统诸如计算机系统197和一个或多个数据储存库诸如数据储存库195。在图1a中显示的实施方案中,系统190的多个子系统可以通过一个或多个网络193通信连接,所述网络193可以包括包交换或其它类型的网络基础设施装置(例如路由器、开关等),其配置为促成远程系统间的信息交换。在图1b中显示的实施方案中,测序系统190是测序装置,其中多个子系统(例如诸如测序仪191、计算机系统197和可能数据储存库195)是通信和/或操作偶联并在测序装置内集成的组件。
在一些操作背景中,图1a和1b中显示的实施方案的数据储存库195和/或计算机系统197可以在云计算环境196内配置。在云计算环境中,可以将包含数据储存库的存储装置和/或包含计算机系统的计算装置分配并例示,作为效用且按需要使用;如此,云计算环境作为服务提供基础设施(例如物理和虚拟机器、原始/块存储、防火墙、负载均衡器、聚合器(aggregator)、网络、存储簇(storagecluster),等等)、平台(例如可以包含操作系统的计算装置和/或解决办法栈(solutionstack)、编程语言执行环境、数据库服务器、网络服务器、应用服务器,等等)和实施任何存储相关和/或计算任务必需的软件(例如应用、应用编程界面或api,等等)。
注意到在多个实施方案中,本文中描述的技术可以通过包含各种构造和形式因素的一些或所有上述子系统和组件(例如诸如测序仪、计算机系统和数据储存库)的多种系统和装置实施;如此,应当以例示性而非限制性意义看待图1a和1b中显示的实例实施方案和构造。
测序仪191配置并可操作为接受源自生物学样品碎片的靶核酸192,并且对靶核酸实施测序。可以使用可以实施测序的任何合适的机器,其中此类机器可以使用各种测序技术,其包括但不限于通过杂交测序、通过连接测序、通过合成测序、单分子测序、光学序列检测、电磁序列检测、电压变化序列检测和适合于从dna产生读取结果测序结果的任何其它现在已知或以后开发的技术。在多个实施方案中,测序仪可以测序靶核酸,并且可以产生读取结果测序结果,其可以包含或不包含缺口,并且可以是或不是配对-对(或成对末端)读取结果。如图1a和1b中显示的,测序仪191测序靶核酸192,并获得读取结果测序结果194,其得到传送以(暂时和/或持久)存储于一个或多个数据储存库195和/或通过一个或多个计算机系统197处理。
数据储存库195可以在一个或多个存储装置(例如硬盘驱动器、光盘、固态驱动器等)上执行,所述存储装置可以配置为盘阵列(例如诸如scsi阵列)、存储簇或任何其它合适的存储装置构造。数据储存库的存储装置可以配置为系统190的内部/集成组件或与系统190可附接的外部组件(例如诸如外部硬驱动机或盘阵列)(例如如图1b中显示的),和/或可以以合适的方式通信互连,所述合适的方式诸如例如网格、存储簇、存储区网络(san)和/或网络附接存储(nas)(例如如图1a中显示的)。在多个实施方案和实现中,数据储存库可以在存储装置上以一个或多个以文件存储信息的文件系统、以一个或多个以数据记录存储信息的数据库和/或以任何其它合适的数据存储构造执行。
计算机系统197可以包含一个或多个计算装置,其包含通用处理器(例如中央处理单元或cpu)、存储器和计算机逻辑199,其与配置数据和/或操作系统(os)软件一起可以实施本文中描述的一些或所有技术和方法,和/或可以控制测序仪191的操作。例如,本文中描述的任何方法(例如用于误差校正、单元型定相,等等)可以完全或部分由计算装置实施,所述计算装置包含处理器,该处理器可以配置为执行逻辑199,用于实施方法的各个方法。此外,虽然方法步骤可以以编号步骤呈现,但是应当理解本文中描述的方法的步骤可以同时(例如通过计算装置簇平行进行)或以不同次序实施。计算机逻辑199的功能性可以以单一集成模块(例如在集成逻辑中)执行或者可以在两个或更多个软件模块中组合,所述软件模块可以提供一些别的功能性。
在一些实施方案中,计算机系统197可以是单一计算装置。在其它实施方案中,计算机系统197可以包含多个计算装置,其可以在网格、簇或在云计算环境中通信和/或可操作互连。此类多个计算装置可以在不同形状因子(formfactor)诸如计算结点、刀片(blade),或任何其它合适的硬件构造中配置。出于这些原因,应当以例示性而非限制性意义看待图1a和1b中的计算机系统197。
图2是作为测序仪和/或计算机系统的一部分的实例计算装置200的框图,所述计算装置200可以配置为执行用于实施各种数据处理和/或控制功能性的指令。
在图2中,计算装置200包含直接或经由一个或多个系统总线诸如总线275间接互连的几个组件。此类组件可以包含但不限于键盘278、持久性存储装置279(例如诸如固定盘、固态盘、光盘等等)和显示适配器282,一个或多个显示装置(例如诸如lcd监视器、平板监视器、等离子屏等)可以与所述显示适配器282偶联。外围设备和输入/输出(i/o)装置(其与i/o控制器271偶联)可以通过本领域中已知的多种手段(包括但不限于一个或多个串行端口、一个或多个并行端口和一个或多个通用串行总线(usb)与计算装置200连接。外部接口281(其可以包括网络接口卡和/或串行端口)可以用于连接计算装置200与网络(例如诸如因特网或局域网络(lan))。外部接口281还可以包括许多输入接口,其可以从各种外部装置诸如例如测序仪或其任何组件接受信息。经由系统总线275的互连容许一个或多个处理器(例如cpu)273与每个连接的组件通信并且执行来自系统存储器272和/或来自存储装置279的指令(和/或控制其执行)及各个组件间的信息交换。系统存储器272和/或存储装置279可以作为一个或多个计算机可读的非短暂存储介质体现,所述计算机可读的非短暂存储介质存储由处理器273执行的指令序列及其它数据。此类计算机可读的非短暂存储介质包括但不限于随机存取存储器(ram)、只读存储器(rom)、电磁介质(例如诸如硬盘驱动器、固态驱动器、拇指存储器(thumbdrive)、软盘等)、光学介质诸如紧致磁盘(cd)或数字通用盘(dvd)、闪速存储器等。各种数据值和其它结构化或未结构化信息可以从一个组件或子系统输出到另一个组件或子系统,可以经由显示适配器282和合适的显示装置对用户呈现,可以通过网络经由外部接口281发送到远程装置或远程数据储存库,或者(暂时和/或永久)存储于存储装置279上。
由计算装置200实施的任何方法和功能性可以以模块或集成方式使用硬件和/或计算机软件以逻辑形式执行。如本文中使用的,“逻辑”指在由一个或多个计算装置的一个或多个处理器(例如cpu)执行时可操作为实施一个或多个功能性和/或返回一个或多个结果形式的数据或由其它逻辑元件使用的数据的一组指令。在多个实施方案和实现中,任何给定的逻辑可以作为由一个或多个处理器(例如cpu)可执行的一个或多个软件组件、作为一个或多个硬件组件诸如专用集成电路(application-specificintegratedcircuit,asic)和/或现场可编程门阵列(field-programmablegatearray,fpga),或者作为一个或多个软件组件和一个或多个硬件组件的任何组合执行。任何特定逻辑的软件组件可以不限于作为独立软件应用、作为客户机-服务器系统中的客户机、作为客户机-服务器系统中的服务器、作为一个或多个软件模块、作为一个或多个功能库以及作为一个或多个静态和/或动态连接库执行。在执行期间,任何特定逻辑的指令可以作为一个或多个计算机过程、线程、纤维和任何其它合适的运行时间实体体现,所述运行时间实体可以在一个或多个计算装置的硬件上例示,并且可以是分配的计算资源,其可以包括但不限于存储器、cpu时间、存储空间和网络带宽。
用于lfr过程的技术和算法
碱基响应
使用本发明的组合物和方法对靶核酸测序的总体方法记载于本文中及例如美国专利申请公开文本2010/0105052-a1;公布的专利申请号wo2007120208,wo2006073504,wo2007133831和us2007099208以及美国专利申请no.11/679,124;11/981,761;11/981,661;11/981,605;11/981,793;11/981,804;11/451,691;11/981,607;11/981,767;11/982,467;11/451,692;11/541,225;11/927,356;11/927,388;11/938,096;11/938,106;10/547,214;11/981,730;11/981,685;11/981,797;11/934,695;11/934,697;11/934,703;12/265,593;11/938,213;11/938,221;12/325,922;12/252,280;12/266,385;12/329,365;12/335,168;12/335,188;和12/361,507,其通过提及完整并入本文用于所有目的。还可见drmanacetal.,science327,78-81,2010。长片段读取(lfr)方法已经披露于美国专利申请no.12/816,365,12/329,365,12/266,385,和12/265,593及美国专利no.7,906,285,7,901,891和7,709,197,其在此通过提及完整收入。本文中提供了进一步的详细和改进。
在一些实施方案中,数据提取会依赖于两类图像数据:划分表面上所有dnb位置的明视场图像,和在每个测序循环期间获得的荧光图像组。数据提取软件可以用于鉴定具有明视场图像的所有对象,然后对于每个此类对象,软件可以用于计算每个测序循环的平均荧光值。对于任何给定的循环,有四个数据点,其对应于以不同波长拍摄的四个图像,用以询问所述碱基是否是a、g、c或t。合并这些原始数据点(在本文中又称为“碱基响应”),对每个dnb产生不连续的读取结果测序结果。
计算装置可以装配鉴定碱基群体以提供关于靶核酸的序列信息和/或鉴定靶核酸中特定序列的存在。例如,计算装置可以通过执行各种逻辑依照本文中描述的技术和算法装配鉴定碱基群体;此类逻辑的例子是以任何合适的编程语言诸如java、c++、perl、python和任何其它合适的常规和/或面向对象的编程语言书写的软件代码。在以一个或多个计算机过程形式执行时,此类逻辑可以读取结果、书写和/或以其它方式处理结构化和未结构化的数据,该数据可以以各种结构在持久性存储上和/或在易失性存储器中存储;此类存储结构的例子包括但不限于文件、表、数据库记录、阵列、列表、向量、变数、存储器和/或处理器寄存器、从面向对象类别例示的持久性和/或存储器数据对象和任何其它合适的数据结构。在一些实施方案中,通过比对从对多个dnb实施的多个测序循环获得的重叠序列将鉴定的碱基装配成完整序列。如本文中使用的,术语“完整序列”指部分或整个基因组及部分或整个靶核酸的序列。在别的实施方案中,由一个或多个计算装置或其计算机逻辑实施的装配方法利用可以用于“拼凑”重叠序列以提供完整序列的算法。在又一些实施方案中,参照表用于辅助将鉴定的序列装配成完整序列。可以使用关于选择生物体的现有测序数据编译参照表。例如人基因组数据可以经由国立生物技术信息中心于ftp.ncbi.nih.gov/refseq/release,或者经由j.craigventerinstitute于www.jcvi.org/researchhuref/访问。整个人基因组信息或人基因组信息的子集可以用于创建用于特定测序询问的参照表。另外,特定参照表可以从源自特定群体的经验数据,包括来自具有特定种族性、地理传统、宗教或文化限定群体的人类的遗传序列构建,因为人基因组内的变异可以随其中含有的信息起源而使参照数据倾斜。,例如美国专利公开文本no.2011-0004413,名称为“methodandsystemforcallingvariationsinasamplepolynucleotidesequencewithrespecttoareferencepolynucleotidesequence”(其通过提及并入本文用于所有目的)中提供了用于响应与参照多核苷酸序列相比多核苷酸序列中的变异及用于多核苷酸序列装配(或重新装配)的例示性方法。
在本文中讨论的发明的任何实施方案中,核酸模板和/或dnb群体可以包含许多靶核酸以基本上覆盖整个基因组或整个靶多核苷酸。如本文中使用的,“基本上覆盖”意指分析的核苷酸(即靶序列)量含有当量至少两个拷贝的靶多核苷酸,或在另一个方面,至少10个拷贝,或在另一个方面,至少20个拷贝,或在另一个方面,至少100个拷贝。靶多核苷酸可以包含dna片段,其包含基因组dna片段和cdna片段及rna片段。用于重新构建靶多核苷酸序列的步骤的指导可以参见以下参考文献,其通过提及并入:landeretal,genomics,2:231-239(1988);vingronetal,j.mol.biol.,235:1-12(1994);及类似的参考文献。
在一些实施方案中,对测序的复杂核苷酸的每个询问位置产生四幅图像,一幅用于每种颜色染料。通过调节染料和背景强度之间的串扰测定图像中每个点的位置和四种颜色中每种的所得强度。定量模型可以拟合至所得的四维数据集。以质量得分对给定点响应碱基,所述质量得分反映四个强度多么好地拟合所述模型。
可以通过一个或多个计算装置或其计算机逻辑在几个步骤中实施每个视场的四幅图像的碱基响应。首先,使用修改的形态学“图像打开”操作针对背景校正图像强度。由于dnb的位置与照相机像素位置排在一起,强度提取作为来自经背景校正的图像的像素强度的简单读取结果完成。然后,针对光学和生物学信号串扰两者的几个来源校正这些强度,如下文描述的。然后,将经校正的强度通到概率模型,其最终对每个dnb产生四种可能碱基响应结果的四种可能性的组。然后,使用预先拟合的逻辑回归将几种度量组合以计算碱基响应得分。
强度校正:使用作为由一个或多个计算装置执行的计算机逻辑执行的线性回归模型校正生物学和光学串扰的几个来源。线性回归优于解卷积方法,该解卷积方法在计算上更昂贵的并且产生具有相似质量的结果。光学串扰的来源包括四个荧光染料谱间的过滤带重叠,和邻近dnb间由于其极其接近处的光衍射所致的侧面串扰。串扰的生物学来源包括先前循环的不完全清洗、探针合成误差和污染邻近位置信号的探针“滑动”、在询问锚定(anchor)“外部”(与锚定更远离的)碱基时不完全锚定延伸。线性回归用于测定dnb强度中可以使用任一邻近dnb的强度或来自先前循环或其它dnb位置的强度预测的部分。然后,从初始提取强度扣除可以通过串扰的这些来源解释的强度部分。为了测定回归系数,线性回归模型的左侧的强度需要主要仅由“背景”强度,即正在实施回归的给定碱基不会响应的dnb的强度组成。这需要使用初始强度进行的预响应(pre-calling)步骤。一旦选择没有特定碱基响应(具有合理置信度)的dnb,计算装置或其计算机逻辑实施串扰来源的同时回归:
近邻dnb串扰都使用上述回归校正。还有,使用涉及所有可用dnb位置里所有近邻的线性模型对每个dnb校正其特定的邻域。
碱基响应概率:使用最大强度响应碱基不造成四种碱基的背景强度分布的不同形状。为了解决此类可能的差异,基于背景强度的经验概率分布开发概率模型。一旦校正强度,计算装置或其计算机逻辑预响应使用最大强度的一些dnb(通过某个置信度阈值的dnb),并且使用这些预响应的dnb来驱动背景强度分布(给定碱基不响应的dna的强度分布)。在获得此类分布后,计算装置可以对每个dnb计算所述分布下的尾概率,其描述所述强度是背景强度的经验概率。因此,对于每个dnb和四种强度中的每种,计算装置或其逻辑可以获得并存储其作为背景的概率
然后,可以组合这16种概率以获得四种可能碱基响应的四种概率的缩减集。也就是说:
得分计算:逻辑回归用于得到得分计算公式。计算装置或其计算机逻辑将逻辑回归拟合到使用几种度量作为输入的碱基响应的定位结果。所述度量包括响应碱基和下一最高碱基之间的概率比、响应碱基的强度、响应碱基身份的指示变量和描述域(field)的总体聚簇质量的度量。所有度量转化为与协调的和不协调的响应之间的对数优势率(log-odds-ratio)为共线。使用交叉验证改进所述模型。具有最终逻辑回归系数的分对数(logit)函数用于计算产生的得分。
定位和装配
在别的实施方案中,读取结果数据以压缩二进制形式编码,并且包括响应的碱基和质量得分两者。质量得分与碱基准确度相关联。分析软件逻辑,包括序列装配软件可以使用得分来从具有读取结果的各个碱基确定证据的贡献。
读取结果由于dnb结构而可以是“有缺口的”。缺口大小随酶消化固有的变化性而有所变化(通常+/-1个碱基)。由于cpal的随机存取性质,读取结果在其它情况下高质量的dnb中偶尔可以具有未读取结果的碱基(“无响应”)。使读取结果对配对。
能够比对读取结果数据与参照序列的定位软件逻辑可以用于将通过本文中描述的测序方法产生的数据定位。在由一个或多个计算装置执行时,此类定位逻辑一般会容许相对于参照序列的小变化,诸如由各个基因组变异、读取结果误差或未读取结果碱基引起的那些变化。此特性经常容许直接再建snp。为了支持转配较大的变异,包括大规模结构变化或密集变异区,可以分开定位dnb的每个臂,在比对后应用配偶(mate)配对约束。
如本文中使用的,术语“序列变体”或仅“变体”包括任何变体,包括但不限于一个或多个碱基的取代或替换;一个或多个碱基的插入或缺失(又称为“indel”);倒位;转变;重复或拷贝数变化(cnv);三核苷酸重复扩充;结构变化(sv;例如染色体内或染色体间重排,例如易位);等等。在二倍体基因组中,“杂合性”或“het”是基因对中特定基因的两个不同等位基因。两个等位基因可以是不同突变体或与突变体成对的野生型等位基因。本方法也可以在分析非二倍体生物体中使用,无论此类生物体是单倍体/一倍体(n=1,其中n=染色体的单倍体数目)还是多倍体或非整倍体。
在一些实施方案中,序列读取结果的装配可以利用支持dnb读取结果结构(具有非响应碱基的配对的、有缺口的读取结果)的软件逻辑来产生二倍体基因组装配,其在一些实施方案中可以被产生用于对杂合子位点定相的本发明lfr方法的序列信息利用。
本发明的方法可以用于重建不存在于参照序列中的新区段。在一些实施方案中可以使用如下的算法,其利用证据(贝叶斯)推理和基于debruijin图的算法的组合。在一些实施方案中,可以使用针对每个数据集凭经验校正的统计学模型,容许所有读取结果数据在没有预过滤或数据修整的情况下使用。也可以通过调节配对读取结果来检测大规模结构变化(包括但不限于缺失、易位等)和拷贝数变化。
对lfr数据定相
图3描述了lfr数据定相中的主要步骤。这些步骤如下:
(1)使用lfr数据进行的图构建:一个或多个计算装置或其计算机逻辑产生无向图,其中顶点代表杂合snp,而边缘代表那些杂合snp间的连接。边缘由方向和连接强度构成。一个或多个计算装置可以在存储结构中存储此类图,所述存储结构包括但不限于文件、表、数据库记录、阵列、列表、向量、变量、存储器和/或处理器寄存器、持久性和/或从面向对象的种类例示的存储器数据对象、和任何其它合适的短暂和/或持久性数据结构。
(2)使用配偶对数据进行的图构建:步骤2与步骤1类似,其中与lfr数据相反,基于配偶对数据进行连接。为了进行连接,必须在相同读取结果(相同臂或配偶臂)中用两个感兴趣的杂合snp找到dnb。
(3)图组合:上述每幅图的计算装置或其计算机逻辑表示经由nxn稀疏矩阵进行,其中n是所述染色体上候选杂合snp的数目。两个结点在上述每种方法中可以仅具有一个连接。在组合两个方法的情况中,两个结点可以有多至两个连接。因此,计算装置或其计算机逻辑可以使用选择算法来选择一个连接作为选择的连接。对于这些研究,发现配偶对数据的质量显著次于lfr数据的质量。因此,仅使用lfr导出的连接。
(4)图修整:针对存储的图数据通过计算装置设计并应用一系列试探法以除去一些误差的连接。更精确地,结点必须满足一个方向上至少两个连接和另一个方向上一个连接的条件;否则,将其消除。
(5)图优化:计算装置或其计算机逻辑通过产生最小跨度树(mst)来优化图。幂函数设置为-|强度|。在此过程期间,在可能的情况下,由于与较强路径的竞争而使较低的强度边缘消除。因此,mst提供了最强且最可靠的连接的自然选择。
(6)重叠群建立:一旦在计算机可读介质中产生和/或存储最小跨度树,计算装置或其逻辑可以使所有结点再取向,期间取得一个结点(在这里,第一结点)常数。此第一结点是锚结点。对于每个结点,计算装置然后寻找至锚结点的路径。测试结点的方向是路径上边缘方向的聚集体。
(7)通用定相:在上述步骤后,计算装置或其逻辑对在先前步骤中建立的每个重叠群定相。在这里,与定相形成对比,这部分的结果称为预定相的,指示这不是最终的定相。由于第一结点任意选择为锚结点,整个重叠群的定相不必与亲本染色体一致。对于通用定相,使用重叠群上可获得三人一组信息的几个杂合snp。然后,使用这些三人一组杂合snp来鉴定重叠群的比对。在通用定相步骤结束时,所有重叠群都已经适当标记,并且因此可以认为是全染色体重叠群。
重叠群产生
为了产生重叠群,对于每个杂合snp对,计算装置或其计算机逻辑测试两个假设:正向方向和反向方向。正向方向意味着两个杂合snp以它们最初列出(最初按字母表顺序)的相同方向连接。反向方向意味着两个杂合snp以其最初列表的相反次序连接。图4描绘了对邻近杂合snp的成对分析,其涉及将正向和反向方向归入杂合snp对。
每个方向会具有数字支持,显示了相应假设的有效性。此支持是图5中显示的连接矩阵的16个单元的函数,该图5显示了假设选择的例子,及对其分配得分。为了简化函数,将16个变量简化成3个:幂(power)1、幂2和杂质(impurity)。幂1和幂2是对应于每个假设的两个最高值单元。杂质是所有其它单元(而不是与假设对应的2个)的总和与矩阵中单元的总和的比率。基于相应单元的总和进行两个假设间的选择。具有较高和的假设是获胜假设。以下计算仅用于分配所述假设的强度。强假设是对于幂1和幂2具有高数值及对于杂质具有低数值的假设。
将三个量度幂1、幂2和杂质馈给到模糊推理系统(图6)中,以将其效应简化成0和1之间(包括端点)的单一数值-得分。模糊推理系统(fis)作为计算机逻辑实施,所述计算机逻辑可以由一个或多个计算装置执行。
对多至预期重叠群长度(例如20-50kb)的合理距离内的每个杂合snp对进行连接操作。图6显示了图构建,描绘了三个邻近杂合snp的一些例示性连接和强度。
模糊推理引擎的规则如下定义:
(1)若幂1较小且幂2较小,则得分是非常小的。
(2)若幂1是中等的且幂2较小,则得分是小的。
(3)若幂1是中等的且幂2是中等的,则得分是中等的。
(4)若幂1较大且幂2较小,则得分是中等的。
(5)若幂1较大且幂2是中等的,则得分较大。
(6)若幂1较大且幂2较大,则得分是非常大的。
(7)若杂质较小,则得分较大。
(8)若杂质是中等的,则得分是小的。
(9)若杂质较大,则得分是非常小的。
对于每个变量,较小、中等和较大的定义是不同的,并且由其特定隶属函数决定。在将模糊推理系统(fis)暴露于每个变量集后,将输入集对规则的贡献传播到模糊逻辑系统,并且产生输出的单一(去模糊化)数字:得分。此得分限于0和1之间,1显示最高质量。
在对每个结点对应用fis后,计算装置或其计算机逻辑构建整幅图。图7显示了此图的例子。根据获胜假设的方向给结点着色。每个连接的强度通过对感兴趣的杂合snp对应用fis导出。一旦构建初步图(图7的顶部图),计算装置或其计算机逻辑优化该图(图7的底部图),并将其简化成树。此优化过程通过从初始图产生最小跨度树(mst)完成。mst保证从每个结点到任何另一结点的独特路径。
图7显示了图优化。在此应用中,每个重叠群上的第一结点用作锚结点,并且所有其它结点相对于所述结点取向。根据方向,每个命中将必须翻转或不然,以匹配锚结点的方向。图8显示了给定例子的重叠群比对方法。在此方法结束时,可得到定相的重叠群。
在定量方法中的此点时,将两个单元型分开。虽然已知这些单元型之一来自模板,而一个来自父本,但是完全不知道哪一个来自哪个亲本。在定相的下一步中,计算装置或其计算机逻辑尝试将正确的亲本标签(母本/父本)归入每个单元型。此过程称为通用定相。为了这样做,需要知道至少几个杂合snp(在重叠群上)与亲本的联系。此信息可以通过进行三人一组(母本-父本-后代)定相获得。使用三重测序基因组,鉴定出具有已知亲本联系的一些基因座,更具体地在至少一个亲本是纯合时。然后,计算装置或其计算机逻辑使用这些联系以将正确的亲本标签(母本/父本)归入整个重叠群,也就是说,实施亲本辅助通用定相(图9)。
为了保证高准确度,可以实施下列各项:(1)在可能时(例如在na19240的情况中),从多个来源(例如内部和1000个基因组)获得三重信息,并使用此类资源的组合;(2)需要重叠群包含至少两个已知的三重定相基因座;(3)消除在一行中具有一系列三重错配(指示区段误差)的重叠群;并(4)消除在三重基因座末端具有单一三人一组错配(指示潜在的区段误差)的重叠群。
图10显示了自然重叠群分离。不论使用亲本数据与否,重叠群在天然情况下经常不连续下去超出某个点。重叠群分离的原因是:(1)某些区域中超过平常的dna片段化或缺乏扩增,(2)低杂合snp密度,(3)参照基因组上的多聚n序列,和(4)dna重复区(倾向于误差定位)。
图11显示了通用定相。通用定相的主要优点之一是获得完整染色体“重叠群”的能力。这是有可能的,因为每个重叠群(在通用定相后)携带具有正确亲本标签的单元型。因此,所有携带标签母本的重叠群可以放在相同单元型上;并且可以对父本重叠群完成相似操作。
lfr方法的另一个主要优点是显著提高杂合snp响应的准确度的能力。图12显示了源自使用lfr方法的误差检测的两个例子。图12(左侧)中显示了第一个例子,其中连接矩阵不支持任何预期的假设。这指示杂合snp之一实际上不是杂合snp。在此例子中,a/c杂合snp实际上是纯合基因座(a/a),其被装配器误差标记为杂合基因座。可以鉴定此误差,并且将其消除或(在此情况中)校正。图13(右侧)中显示了第二个例子,其中此情况的连接矩阵同时支持这两种假设。这是杂合snperozygous响应不真实的迹象。
“健康”杂合snp连接矩阵是仅具有两个高单元(在预期的杂合snp位置,即不在直线上)的连接矩阵。所有其它可能性指向潜在的问题,并且可以消除或用于对感兴趣的基因座产生交替的碱基响应。
lfr方法的另一个优点是以较弱的支持响应杂合snp的能力(例如其中由于偏爱或错配率而难以定位dnb)。由于lfr方法需要对杂合snp的额外约束,可以降低杂合snp响应在非lfr装配器中需要的阈值。图13表明此情况的例子,其中可以进行确信的杂合snp响应,尽管有少量读取结果。在图13(右侧)中,在正常情况下,低数目的支持性读取结果会阻止任何装配器确信地响应相应的杂合snp。然而,由于连接矩阵是“干净的”,可以更确信地将杂合snp响应归入这些基因座。
注释剪接位点中的snp
转录rna中的内含子需要在它们变为mrna前剪接出去。关于剪接的信息在这些rna的序列内体现,并且基于一致性的。剪接位点共有序列中的突变是许多人类疾病的原因(faustinoandcooper,genesdev.17:419-437,2011)。大多数剪接位点符合外显子周围的固定位置处的简单共有序列。在这点上,开发出注释剪接位点突变的程序。在此程序中,使用共有剪接位置模型(www.life.umd.edu/labs/mount/rnainfo)。对样式:外显子5’端区中的cag|g(“|”表示外显子开始)和相同外显子3’端区中的mag|gtrag(“|”表示外显子的结束)实施查找。这里,m={a,c},r={a,g}。此外,将剪接共有位置分类成两类:i型,其中与模型的一致性是100%需要的;和ii型,其中与模型的一致性在大于50%情况中保持。据推测,i型位置中的snp突变会引起错过剪接,而ii型位置中的snp仅会降低剪接事件的效率。
用于注释剪接位点突变的程序逻辑包括两个部分。在部分i中,产生含有来自输入参照基因组的模型位置序列的文件。在部分2中,将来自测序项目的snp与这些模型位置序列比较,并报告任何i型和ii型突变。程序逻辑是外显子中心的,代替内含子中心的(为了便于分析基因组)。对于给定的外显子,在其5’端中,我们查找共有“cagg”(对于位置-3,-2,-1,0。0意味着外显子的开始)。大写字母意味着i型位置,而小写字母意味着ii型位置)。在外显子的3’端中,对共有“maggtrag”(对于位置序列-3,-2,-1,0,1,2,3,4)实施查找。仅忽略不符合这些要求的基因组释放的外显子(占所有情况的约5%)。这些外显子落入其它次要种类的共有剪接位点,并且不通过程序逻辑调查。将来自测序的基因组的任何snp与这些基因组位置处的模型序列比较。会报告i型中的任何错配。若突变偏离一致性,则报告ii型位置中的错配。
上述程序逻辑检测大多数坏的剪接位点突变。报告的坏的snp无疑是成问题的。但是有许多其它坏的snp,其引起通过此程序检测不到的剪接问题。例如,人基因组内有不符合上文提及的一致性的许多内含子。还有,内含子中间的分支点突变也可以引起剪接问题。没有报告这些剪接位点突变。
注释影响转录因子结合位点(tfbs)的snp。jaspar模型用于从释放的人基因组序列(构件36或构件37)寻找tfbs。jasparcore是以矩阵建模的针对脊椎动物的130个tfbs位置频率数据的集合(bryneetal.,nucl.acidsres.36:d102-d106,2008;sandelinetal.,nucl.acidsres.23:d91-d94,2004)。这些模型从jaspar网址(http://jaspar.genereg.net/cgi-bin/jaspar_db.pl?rm=browse&db=core&tax_group=vertebrates)下载。使用以下公式将这些模型转化成位置权重矩阵(pwm):wi=log2[(fi+pni1/2)/(ni+ni1/2)/p],其中:fi是对位置i处特定碱基观察到的频率;ni是所述位置处的总体观察;且p是目前核苷酸的背景频率,其默认为0.25(bogdan.org.ua/2006/09/11/position-frequency-matrix-to-position-weight-matrix-pfm2pwm.html;wassermanandsandelin,naturereviews,genetics5:p276-287,2004)。一种特定的程序mast(meme.sdsc.edu/meme/mast-intro.html)用于对基因组内的序列区段搜索tfbs位点。运行程序以提取参照基因组中的tfbs位点。步骤的概要如下:(i)对于具有mrna的每个基因,从基因组提取[-5000,1000]推定的含有tfbs的区域,0是mrna起始位置。(ii)对所有pwm模型运行推定的含有tfbs的序列的mast搜索。(iii)选择高于给定阈值的那些命中。(iv)对于具有多个或重叠命中的区域,仅选择1-命中,即具有最高mast搜索得分的命中。
凭借来自合适计算机可读介质中产生和/或存储的参照基因组的tfbs模型命中,计算装置或其计算机逻辑可以鉴定位于命中区内的snp。这些snp会影响模型,和命中得分变化。书写第二种程序以计算命中得分的此类变化,因为含有snp的区段两次运行到pwm模型中,一次对于参照,及第二次对于具有snp取代的区段。引起区段命中得分下降超过3的snp鉴定为坏的snp。
具有两个坏的snp的基因的选择。将具有坏的snp的基因分类成两个种类:(1)那些影响转录的aa序列的;和(2)那些影响转录结合位点的。对于aa序列影响,包括以下snp亚类:
(1)无义或无终止变异。这些突变引起截短的蛋白质或延伸的蛋白质。在任一情况中,蛋白质产物的功能是完全丧失的或不太有效的。
(2)剪接位点变异。这些突变引起内含子的剪接位点被破坏(对于那些根据所述模型需要是100%的某个核苷酸的那些位置)或严重减少(对于那些根据所述模型对于某个核苷酸需要是大于50%的位点。snp引起剪接位点核苷酸突变为另一种核苷酸,其低于50%一致性,如通过剪接位点共有序列模型预测的)。这些突变有可能会生成截短的、缺少外显子、或在蛋白质产物数量上严重减少的蛋白质。
(3)aa变异的polyphen2注释。对于引起蛋白质氨基酸序列而非其长度变化的snp,使用polyphen2(adzhubeietal.,nat.methods7:248-249,2010)作为主要的注释工具。polyphen2将snp注释为“良性的”、“未知的”、“可能损害性的”和“大概损害性的”。“可能损害性的”和“大概损害性的”两者鉴定为坏的snp。polyphen2的这些种类分配基于polyphen2软件的结构预测。
对于转录结合位点突变,基于参照基因组作为tfbs结合位点的筛选,使用75%的模型最大得分(maxscore)。除去所述区域中<=75%最大得分的任何模型命中。对于那些剩余的命中,若snp引起命中得分下降3以上,则认为它是有害的snp。
报告了两类基因。1类基因是那些具有至少2个坏aa影响性突变的基因。这些突变可以全在单一等位基因上(1.1类),或在2个独特等位基因上散布(1.2类)。2类基因是1类集的超集。2类基因是含有至少2个坏的snp的基因,不论它是aa影响性的还是tfbs位点影响性的。但是,要求是至少1个snp是aa影响性的。2类基因是那些在1类中的基因,或那些具有1处有害aa突变和1处以上有害tfbs影响性变异的基因。2.1类意味着所有这些有害突变来自单一等位基因,而2.2类意味着有害snp来自两个独特等位基因。
前述技术和算法适用于用于对复杂核酸测序的方法,任选地与测序前的lfr处理结合(与测序结合的lfr可以称为“lfr测序”),其如下详细描述。用于对复杂核酸测序的此类方法可以通过一个或多个执行计算机逻辑的计算装置实施。此类逻辑的一个例子是以任何合适的编程语言诸如java、c++、perl、python和任何其它合适的常规的和/或面向对象的编程语言书写的软件代码。在以一个或多个计算机过程形式执行时,此类逻辑可以读取结果、书写和/或以其它方式处理结构化的和非结构化的数据,该数据可以在持久性存储器上和/或易失性存储器中在多个结构中存储;此类存储结构的例子包括但不限于文件、表、数据库记录、阵列、列表、向量、变数、存储器和/或处理器寄存器、从面向对象类别例示的持久性和/或存储器数据对象和任何其它合适的数据结构。
改善长读取结果测序中的准确度
在使用某些长读取结果技术的dna测序中(例如纳米孔测序),长(例如10-100kb)读取结果长度是可用的,但是一般具有较高的假阴性和假阳性率。来自此类长读取结果技术的序列的最终准确度可以依照以下一般方法使用单元型信息(完全或部分定相)显著增强。
首先,计算装置或其计算机逻辑将读取结果彼此比对。预期大量杂合响应存在于重叠中。例如,若2个至5个100kb片段重叠最少10%,则这导致>10kb重叠,其可以粗略转变成10个杂合基因座。或者,将每个长读取结果与参照基因组比对,通过参照基因组,会隐含获得读取结果的多重比对。
一旦实现了多重读取结果比对,可以考虑重叠区。可以调节重叠会包括大量(例如n=10)杂合基因座的实情以考虑杂合的组合。此组合形式导致单元型概率的较大空间(4n或4^n;若n=10,则4n=约100万)。在n维空间中的所有这4n个点中,预期仅两个点含有生物学可行的信息,即,那些对应于两个单元型的。换言之,存在有4n/2(在这里为1e6/2或约500,000)的噪音抑制率。实际上,大部分的此4n空间是退化的,特别是因为序列已经比对(并且因此相似),而且还因为每个基因座通常不携带超过2个可能的碱基(若它是真的杂合的话)。因此,此空间的下界实际上是2n(若n=10,则2n=约1000)。因此,噪音抑制率可以仅是2n/2(在这里为1000/2=500),其仍然是相当给人印象深刻的。随着假阳性和假阴性数目增加,空间的大小从2n扩充到4n,其继而导致较高的噪音抑制率。换言之,随着噪音增加,它会自动地受到更多抑制。因此,预期输出积仅保留非常小的(且相当恒定的)噪音量,几乎不依赖于输入噪音。(权衡(tradeoff)是更有噪声条件中的产率(yield)损失)。当然,在下述情况下改变这些抑制率:(1)误差是系统性的(或其它数据特质),(2)算法不是最佳的,(3)重叠部分是较短的,或者(4)覆盖冗余是较小的。n可以是大于1的任何整数,诸如2,3,5,10或更多。
以下方法可用于提高长读取结果测序方法的准确度,所述长读取结果测序方法可以具有较大的初始误差率。
首先,计算装置或其计算机逻辑比对几个读取结果,例如5个读取结果或更多,诸如10-20个读取结果。假设读取结果是约100kb,且共享重叠是10%,这导致5个读取结果中的10kb重叠。还假设每1kb中有杂合性。因此,在此共同区域中会有总共10个杂合性。
接着,计算装置或其计算机逻辑填写上述10个候选杂合性的alpha10个可能性(其中alpha介于2和4之间)的部分(例如仅非零元素)或整个矩阵。在一个实现中,此矩阵的alpha10个单元中仅2个应当是高密度(例如如通过阈值测量的,所述阈值可以是预先确定的或动态的)。这些是对应于真正杂合性的单元。这两个单元可以认为是基本上无噪音的中心。剩余部分应当含有几乎0且偶而1个隶属关系,尤其在误差不是系统性的时。若误差是系统性的,可以有聚簇事件(例如具有超过仅0或1的第三个单元),其使任务更加困难。然而,即使在此情况中,假簇的簇隶属关系应当显著弱于(例如如通过绝对或相对量测量的)两个预期簇的簇隶属关系。此情况中的权衡是起始点应当包括比对的更多多重序列,这与具有较长的读取结果或较大的覆盖冗余直接相关。
上述步骤假设在重叠读取结果间观察到两个可行簇。对于大量假阳性,情况不会如此。若情况如此,在alpha维空间中,会使预期的两个簇变模糊,即代替作为具有高密度的单一点,它们会是感兴趣单元周围的m个点的变模糊的簇,其中感兴趣的这些单元是在簇中心的无噪音中心。这使聚簇方法能够捕捉预期点的位置,尽管有精确的序列不在每个读取结果中呈现的实情。簇事件也可以在簇模糊(即可以有超过两个中心)时,但是与上文的描述类似的方式发生,对于二倍体生物体,得分(例如簇单元的总计数)可以用于区分较弱的簇与两个真实的簇。两个真实的簇可以用于对多个区域创建重叠群,如本文中所描述的,并且重叠群可以匹配到两组中以对复杂核酸的较大区域形成单元型。
最终,计算装置或其计算机逻辑可以使用基于群体的(已知的)单元型来提高置信度和/或在寻找真实簇中提供额外的指引。一种实现此方法的方式是给每个观察到的单元型提供权重,并且对未观察到的单元型提供较小的但非零的数值。通过这样做,实现对天然单元型的偏爱,所述天然单元型已经在感兴趣的群体中观察到。
使用具有含未校正误差的标签序列数据的读取结果
如本文中讨论的,依照本发明的一个实施方案,将复杂核酸的样品分成多份等分试样(例如多孔板中的孔),扩增,并片段化。然后,将等分试样特异性标签与片段连接以鉴定复杂核酸的特定片段起源的等分试样。任选地,标签包含误差校正代码,例如reed-solomon误差校正(或误差检测)代码。在对片段测序时,对标签和复杂核酸序列的片段两者测序。若标签序列中有误差,且不可能鉴定片段起源的等分试样,或者使用误差校正代码校正序列,则可以放弃整个读取结果,导致大量序列数据的损失。应当注意到,包含正确的和经校正的标签序列数据的读取结果是高准确度的,但是低产率的,而包含不能校正的标签序列数据的读取结果是低准确度的,但高产率的。取而代之,此类序列数据用于与那些需要此类数据以依靠特定标签与特定等分试样的联合的身份鉴定起源等分试样的那些方法不同的方法。需要具有正确的(或经校正的)标签序列数据的读取结果的方法的例子包括但不限于样品或库多路复用、定相或误差校正或任何其它需要正确的(或经校正的)标签序列的方法。可以采用具有不能校正的标签序列数据的读取结果的方法的例子包括任何其它方法,包括但不限于定位、基于参照的且局部的重新装配、基于集合的统计学(例如等位基因频率、重新突变的位置,等等)。
将长读取结果转化成虚(virtual)lfr
设计用于lfr的算法(包括定相算法)可以通过将随机虚标签(具有一致分布)归入每个(10-100kb)长片段用于长读取结果。虚标签具有使真正一致的分布能够用于每个代码的益处。由于合并代码的差异和代码的解码效率差异,lfr不能实现此一致性水平。可以在lfr中的任何两个代码的表示中容易地观察到3:1(和多至10:1)的比率。然而,虚lfr方法导致任何两个代码间的真正1:1比率。
用于对复杂核酸测序的方法
概述
依照本发明的一个方面,提供了用于对复杂核酸测序的方法。依照本发明的某些实施方案,提供了用于对非常少量的此类复杂核酸(例如1pg至10ng)测序的方法。即使在扩增后,此类方法产生以高响应率和准确度为特征的装配序列。依照其它实施方案,使用等分取样来鉴定并消除复杂核酸测序中的误差。依照另一个实施方案,lfr与复杂核酸测序结合使用。
除非另有指示,本发明的实践可以采用有机化学、聚合物技术、分子生物学(包括重组技术)、细胞生物学、生物化学和免疫学的常规技术和描述,其在本领域技术内。此类常规技术包括聚合物阵列合成、杂交、连接和使用标记物检测杂交。可以通过参考下文的例子具有合适技术的具体例示。然而,当然也可以使用其它等同的常规方法。此类常规技术和描述可以参见标准实验室手册,诸如genomeanalysis:alaboratorymanualseries(vols.i-iv),usingantibodies:alaboratorymanual,cells:alaboratorymanual,pcrprimer:alaboratorymanual,andmolecularcloning:alaboratorymanual(allfromcoldspringharborlaboratorypress),stryer,l.(1995)biochemistry(4thed.)freeman,newyork,gait,“oligonucleotidesynthesis:apracticalapproach”1984,irlpress,london,nelsonandcox(2000),lehninger,principlesofbiochemistry3rded.,w.h.freemanpub.,newyork,n.y.andbergetal.(2002)biochemistry,5thed.,w.h.freemanpub.,newyork,n.y.,其全部通过提及完整并入本文用于所有目的。
使用本发明的组合物和方法对靶核酸测序的总体方法记载于本文及例如美国专利公开文本2010/0105052和us2007099208及美国专利申请no.11/679,124(以us2009/0264299公布);11/981,761(us2009/0155781);11/981,661(us2009/0005252);11/981,605(us2009/0011943);11/981,793(us2009-0118488);11/451,691(us2007/0099208);11/981,607(us2008/0234136);11/981,767(us2009/0137404);11/982,467(us2009/0137414);11/451,692(us2007/0072208);11/541,225(us2010/0081128;11/927,356(us2008/0318796);11/927,388(us2009/0143235);11/938,096(us2008/0213771);11/938,106(us2008/0171331);10/547,214(us2007/0037152);11/981,730(us2009/0005259);11/981,685(us2009/0036316);11/981,797(us2009/0011416);11/934,695(us2009/0075343);11/934,697(us2009/0111705);11/934,703(us2009/0111706);12/265,593(us2009/0203551);11/938,213(us2009/0105961);11/938,221(us2008/0221832);12/325,922(us2009/0318304);12/252,280(us2009/0111115);12/266,385(us2009/0176652);12/335,168(us2009/0311691);12/335,188(us2009/0176234);12/361,507(us2009/0263802),11/981,804(us2011/0004413);和12/329,365;公布的国际专利申请号wo2007120208,wo2006073504和wo2007133831,其全部通过提及完整并入本文用于所有目的。用于响应与参照多核苷酸序列相比多核苷酸序列中的变异及用于多核苷酸序列装配(或再装配)的例示性方法例如在美国专利公开文本no.2011-0004413,(app.no.12/770,089)中提供,其通过提及完整并入本文用于所有目的。还可见drmanacetal.,science327,78-81,2010。还通过并且完整并为了所有目的并入共同悬而未决的相关申请nos.61/623,876,标题为“identificationofdnafragmentsandstructuralvariations“。
此方法包括将靶核酸从样品提取并片段化。片段化的核酸用于生成靶核酸模板,其一般会包含一个或多个衔接头。将靶核酸模板进行扩增方法以形成核酸纳米球,该核酸纳米球通常在表面上布置。对本发明的核酸纳米球实施测序应用,通常经由通过连接技术的测序进行,所述连接技术包括组合探针锚定连接(“cpal”)方法,其在下文更为详细描述。cpal和其它测序方法也可以用于检测特定序列,诸如包括本发明核酸构建体(其包括核酸纳米球及线性和环状核酸模板)中的单核苷酸多态性(“snps”)。上文提及的专利申请和drmanac等的引用文章提供了关于下列各项的额外的详细信息:例如制备核酸模板,包括衔接头设计、将衔接头插入基因组dna片段中以生成环状库构建体;扩增此类库构建体以生成dna纳米球(dnb);在固体支持物上生成dnb的阵列;cpal测序;等等,其与本文中公开的方法结合使用。
如本文中使用的,术语“复杂核酸”指不同核酸或多核苷酸的大群体。在某些实施方案中,靶核酸是基因组dna;外显子组dna(针对转录序列富集的全基因组dna的子集,其含有基因组中外显子的集合);转录物组(即细胞或细胞群体中生成的所有mrna转录物的集合,或由此类mrna生成的cdna)、甲基化组(methylome)(即基因组中甲基化位点的群体和甲基化样式);微生物组(microbiome);不同生物体基因组的混合物、生物体的不同细胞类型的基因组的混合物;和包含大量不同核酸分子的其它复杂核酸混合物(例子包括但不限于微生物组、异种移植物、包括正常细胞和肿瘤细胞两者的实体瘤活组织检查,等等),包括前述类型的复杂核酸的子集。在一个实施方案中,此类复杂核酸具有包含至少一千兆碱基(gb)的整个序列(二倍体人基因组包含约6gb序列)。
复杂核酸的非限制性例子包括“循环核酸”(cna),其是在人血液或其它体液(例如包括但不限于淋巴液、液体、腹水、乳液、尿液、粪和支气管灌洗)中循环,并且可以作为无细胞的(cf)或细胞关联的核酸(综述见pinzanietal.,methods50:302-307,2010)(例如预期母本血流中的循环胎儿细胞(见例如kavanaghetal.,j.chromatol.b878:1905-1911,2010)或来自癌症患者血流的循环肿瘤细胞(ctc)(见例如allardetal.,clincancerres.10:6897-6904,2004))区分的核酸。另一个例子是单细胞或少量细胞,诸如例如来自活组织检查(例如从胚泡滋养外胚层活组织检查得到的胎儿细胞;来自实体瘤的针吸出的癌细胞;等等)的少量细胞的基因组dna。另一个例子是组织中、血液或其它体液中的病原体,例如细菌细胞、病毒或其它病原体,等等。
如本文中使用的,术语“靶核酸”(或多核苷酸)或“感兴趣的核酸”指适合于通过本文中描述的方法处理和测序的任何核酸(或多核苷酸)。核酸可以是单链的或双链的,并且可以包括dna、rna或其它已知的核酸。靶核酸可以是任何生物体的那些靶核酸,所述生物体包括但不限于病毒、细菌、酵母、植物、鱼、爬行类、两栖类、鸟类和哺乳动物(包括但不限于小鼠、大鼠、犬、猫、山羊、绵羊、牛、马、猪、兔、猴和其它非人灵长类及人)。靶核酸可以获自个体或多个个体(即群体)。获得核酸的样品可以含有来自细胞或甚至生物体的混合物的核酸,诸如:包含人细胞和细菌细胞的人唾液样品;包含小鼠细胞和来自移植的人肿瘤的细胞的小鼠异种移植物;等等。
靶核酸可以是未扩增的或者可以通过本领域中已知的任何合适的核酸扩增方法扩增。可以依照本领域中已知的方法纯化靶核酸以除去细胞和亚细胞杂质(脂质、蛋白质、碳水化合物、与要测序的那些核酸不同的核酸,等等),或者它们可以是未纯化的,即包括至少一些细胞和亚细胞杂质,包括但不限于受到破坏以释放其核酸用于处理和测序的完整细胞。可以使用本领域中已知的方法从任何合适的样品获得靶核酸。此类样品包括但不限于:组织、分离的细胞或细胞培养物、体液(包括但不限于血液、尿液、血清、淋巴、唾液、肛门和阴道分泌物、汗液和精液);空气、农业、水和土壤样品,等等。在一个方面,自基因组dna形成本发明的核酸构建体。
鸟枪测序的高覆盖是期望的,因为它可以克服碱基响应和装配中的误差。如本文中使用的,对于装配序列(assembledsequence)中的任何给定位置,术语“序列覆盖丰余”、“序列覆盖”或仅“覆盖”意指代表位置的读取结果的数量。它可以从初始基因组的长度(g)、读取结果数(n)和平均读取结果长度(l)以nxl/g计算。覆盖也可以通过对每个参照位置进行碱基的计数来直接计算。对于全基因组序列,覆盖以装配序列中的所有碱基的平均值表示。序列覆盖是碱基被读出的平均次数(如上文描述的)。它经常以“倍数覆盖”表示,例如“40倍覆盖”,这意味着每个碱基在最终装配序列中以平均40个读取结果来代表。
如本文中使用的,术语“响应率(callrate)”意指复杂核酸中完全响应的碱基的百分比比较,通常参考合适的参照序列,诸如,例如参照基因组。因此,对于全人基因组,“基因组响应率”(或简单地说“响应率”)是相对于全人基因组参照而言在人基因组中完全响应的碱基的百分比。“外显子组响应率”是相对于外显子组参照而言在外显子组中完全响应的碱基的百分比。外显子组序列可以通过用从dna样品选择性捕获目标基因组区的多个已知方法富集的基因组部分测序获得。或者,外显子组序列可以通过对包括外显子组序列的全人基因组测序获得。如此,全人基因组序列可以具有“基因组响应率”和“外显子组响应率”两者。还有“原始读取结果响应率”,其反映的是被分配了a/c/g/t的碱基的数目,而不是所尝试的碱基的总数。(偶而地,术语“覆盖”代替“响应率”使用,但是意义从上下文看会是显而易见的)。
制备复杂核酸的片段
核酸分离。使用常规技术分离靶基因组dna,例如如上文引用的sambrookandrussell,molecularcloning:alaboratorymanual中公开的。在一些情况中,特别是如果在特定步骤中采用少量dna,那么有利的是每当仅可用少量样品dna且经由例如对容器壁等的非特异性结合而有损失危险时提供要与样品dna混合并一起使用的载体dna,例如无关环状合成双链dna。
依照本发明的一些实施方案,在进行或不进行纯化的情况中从单个细胞或少量细胞获得基因组dna或其它复杂核酸。
长片段对于lfr是期望的。可以通过许多不同方法从细胞分离基因组核酸的长片段。在一个实施方案中,将细胞裂解,并用温和的离心步骤将完整的核沉淀。然后,经由蛋白酶k和rna酶消化几小时释放基因组dna。可以处理材料以降低剩余细胞废物的浓度,例如通过透析一段时间(即2-16小时)和/或稀释进行。由于此类方法不需要采用许多破坏性方法(诸如乙醇沉淀、离心和涡旋振荡),基因组核酸很大程度上保持完整,产生具有超过150千碱基的长度的大多数片段。在一些实施方案中,片段的长度是约5至约750千碱基。在别的实施方案中,片段的长度是约150至约600、约200至约500、约250至约400和约300至约350千碱基。可以用于lfr的最小片段是含有至少两个杂合性的片段(约2-5kb),并且没有最大理论大小,尽管片段长度可以由于源自起始核酸制备物操作的剪切而受到限制。产生较大片段的技术导致需要较少的等分试样,并且产生较短片段的那些技术可能需要较多的等分试样。
一旦分离dna且在将其等分取样到单个孔中前,将其仔细片段化以避免材料的损失,特别是来自每个片段末端的序列,因为此类材料的损失可以导致最终基因组装配中的缺口。在一个实施方案中,通过使用罕见的切口酶避免序列损失,所述切口酶在彼此约100kb的距离处创建聚合酶,诸如phi29聚合酶的起始位点。由于聚合酶创建新的dna链,它置换旧的链,这创建聚合酶起始位点附近的重叠序列。因此,有非常少的序列缺失。
5’外切核酸酶的受控使用(在例如通过mda的扩增之前或期间)可以促进初始dna从单细胞的多重复制,如此使经由拷贝复制得到的早期误差的增长最小化。
在其它实施方案中,以使剪切或dna对容器的吸附最小化的方式分离并操作长dna片段,包括例如在琼脂糖凝胶塞中的琼脂糖或油中分离细胞,或者使用特殊包被的管和板。
在一些实施方案中,在等分取样前从单细胞进一步复制片段化dna可以如下实现,即将衔接头与单链引发突出物连接并使用衔接头特异性引物和phi29聚合酶以从每个长片段生成两个拷贝。这可以从单细胞生成相当于4个细胞的dna。
片段化。然后,通过常规技术将靶基因组dna分级或片段化至期望的大小,所述常规技术包括酶促消化、剪切或超声处理,其中后两种在本发明中特别有用。
靶核酸的片段大小可以随来源靶核酸和使用的库构建方法而变化,但是对于标准的全基因组测序,此类片段的长度范围通常是50至600个核苷酸。在另一个实施方案中,片段的长度是300至600或200至2000个核苷酸。在又一个实施方案中,片段的长度是10-100,50-100,50-300,100-200,200-300,50-400,100-400,200-400,300-400,400-500,400-600,500-600,50-1000,100-1000,200-1000,300-1000,400-1000,500-1000,600-1000,700-1000,700-900,700-800,800-1000,900-1000,1500-2000,1750-2000和50-2000个核苷酸。较长的片段可用于lfr。
在别的实施方案中,分离特定大小或在特定大小范围中的片段。此类方法是本领域中公知的。例如,凝胶分级可以用于生成一定碱基对范围内的特定大小的片段群体,例如对于500个碱基对+50个碱基对。
在许多情况中,不需要对提取的dna的酶促消化,因为裂解和提取过程中产生的剪切力会生成期望范围中的片段。在别的实施方案中,可以使用限制性内切核酸酶通过酶促片段化生成较短的片段(1-5kb)。在又一个实施方案中,约10至约1,000,000个基因组当量(equivalent)的dna确保片段群体覆盖整个基因组。含有从重叠片段的此类群体生成的核酸模板的文库如此会包含靶核酸,该靶核酸的序列一旦得到鉴定并装配便会提供整个基因组的大部分或整个序列。
在本发明的一些实施方案中,利用受控随机酶促(“core”)片段化方法来制备片段。core片段化是一种酶促端点测定法,并且具有酶促片段化的优点(诸如对较低量和/或体积的dna使用它的能力)且没有其许多缺点(包括对底物或酶浓度变化的敏感性和对消化时间的敏感性)。
在一个方面,本发明提供了在本文中称为受控随机酶促(core)片段化的片段化方法,其可以单独或与本领域中已知的其它机械和酶促片段化方法组合使用。core片段化涉及三个酶促步骤系列。首先,将核酸进行扩增方法处理,所述扩增方法在存在掺有一定比例的脱氧尿嘧啶(“du”)或尿嘧啶(“u”)的dntp的情况中进行以导致扩增产物的两条链中在限定的且可控制比例的t位置处的dutp或utp取代。任何合适的扩增方法可以在本发明的此步骤中使用。在某些实施方案中,在存在以与dttp的限定比率掺有dutp或utp的dntp的情况中的多重置换扩增(mda)用于产生具有取代到两条链上的某些点中的dutp或utp的扩增产物。
扩增和尿嘧啶模块插入后,然后,通常经由udg、endoviii和t4pnk的组合切除尿嘧啶,以创建具有官能性5’磷酸根和3’羟基末端的单一碱基缺口。单一碱基缺口会以由mda产物中u频率限定的平均间隔创建。也就是说,dutp量越高,所得的片段越短。如本领域技术人员会领会的,也可以使用会导致核苷酸用可类似产生切割的经修饰的核苷酸选择性替换的其它技术,诸如化学或其它酶促易感性核苷酸。
用具有外切核酸酶活性的聚合酶处理有缺口的核酸导致切口沿着核酸的长度“平移”或“移位”,直到相反链上的切口会聚,由此创建双链断裂,这产生相对同质大小的双链片段的相对群体。聚合酶(诸如taq聚合酶)的外切核酸酶活性会切割靠近切口的短dna链,而聚合酶活性会“填充”切口及随后所述链中的核苷酸(实际上,taq沿着链移动,使用外切核酸酶活性切除碱基并且添加相同的碱基,结果是切口沿着链移位,直到酶达到末端)。
由于双链片段的大小分布是mda反应中使用的dttp与dutp或utp的比率的结果,而不是由于酶促处理的持续时间或程度,此core片段化方法产生高度的片段化再现性,这生成均为相似大小的双链核酸片段群体。
片段末端修复和修饰。在某些实施方案中,在片段化后,将靶核酸进一步修饰以使它们制备好依照本发明方法插入多个衔接头。
在物理片段化后,靶核酸通常具有平端和突出端的组合以及末端的磷酸根和羟基化学的组合。在此实施方案中,用几种酶处理靶核酸以创建具有特定化学的平端。在一个实施方案中,聚合酶和dntp用于填充突出物的任何5’单链以创建平端。具有3’外切核酸酶活性的聚合酶(一般但不总是与5’活性酶相同的酶,诸如t4聚合酶)用于除去3’突出物。合适的聚合酶包括但不限于t4聚合酶、taq聚合酶、大肠杆菌dna聚合酶1、klenow片段、逆转录酶、phi29相关聚合酶,包括野生型phi29聚合酶和此类聚合酶的衍生物、t7dna聚合酶、t5dna聚合酶、rna聚合酶。可以使用这些技术来生成平端,其可用于多种应用。
在别的任选实施方案中,改变末端化学以避免靶核酸彼此连接。例如,在聚合酶外,蛋白质激酶也可以用于创建平端的过程,这通过利用其3’磷酸酶活性来将3’磷酸根基团转化成羟基基团进行。此类激酶可以包括但不限于商品化激酶诸如t4激酶,及非商品化但是具有期望活性的激酶。
类似地,可以使用磷酸酶来将末端磷酸根基团转化成羟基基团。合适的磷酸酶包括但不限于碱性磷酸酶(包括小牛肠磷酸酶)、南极磷酸酶、腺苷三磷酸双磷酸酶、焦磷酸酶、无机(酵母)热稳定性无机焦磷酸酶等,其是本领域中已知的。
这些修饰防止靶核酸在本发明方法的随后步骤中彼此连接,如此确保衔接头(和/或衔接头臂)与靶核酸末端连接的步骤期间,靶核酸会与衔接头而不与其它靶核酸连接。可以以期望的方向将靶核酸与衔接头连接。修饰末端避免不想要的构造,其中靶核酸彼此连接和/或衔接头彼此连接。也可以经由控制衔接头和靶核酸两者的末端化学来控制每个衔接头-靶核酸连接的方向。此类修饰可以防止含有以未知构造连接的不同片段的核酸模板的创建,如此降低和/或消除可源自此类不想要模板的序列鉴定和装配中的误差。
可以在片段化后使dna变性以生成单链片段。
扩增。在一个实施方案中,在片段化后(且实际上在本文中概述的任何步骤之前或之后),可以对片段化核酸群体应用扩增步骤以确保足够大浓度的所有片段可用于后续步骤。依照本发明的一个实施方案,提供了用于对少量复杂核酸,包括高等生物体的那些复杂核酸测序的方法,其中扩增此类复杂核酸以生成足够的核酸,用于通过本文中描述的方法测序。本文中描述的测序方法在充分扩增的情况下即使用一份基因当量作为起始材料以高响应率提供高精确序列。注意细胞包含约6.6皮克(pg)基因组dna。可以通过本发明的方法实施来自单细胞或生物体(包括高等生物体诸如人)的少量细胞的全基因组或其它复杂核酸。可以使用1pg,5pg,10pg,30pg,50pg,100pg或1ng复杂核酸作为起始材料实现高等生物体的复杂核酸的测序,所述起始材料通过本领域中已知的任何核酸扩增方法扩增,以生成例如200ng,400ng,600ng,800ng,1μg,2μg,3μg,4μg,5μg,10μg或更大量的复杂核酸。我们还公开了使gc偏爱最小化的核酸扩增方案。然而,可以仅通过分离一个细胞或少量细胞,在本领域中已知的合适培养条件下将它们培养足够的时间,并使用一个或多个起始细胞的后代进行测序来进一步降低对扩增的需要及随后的gc偏爱。
此类扩增方法包括但不限于:多重置换扩增(mda)、聚合酶链式反应(pcr)、连接链式反应(有时称为寡核苷酸连接酶扩增ola)、循环探针技术(cpt)、链置换测定法(sda)、转录介导的扩增(tma)、基于核酸序列的扩增(nasba)、滚环扩增(rca)(对于环化片段)和侵入性切割技术。
可以在片段化后或在本文中概述的任何步骤之前或之后实施扩增。
具有降低的gc偏爱的mda扩增方案。在一个方面,本发明提供了制备样品的方法,其中在库构建和测序前,如实扩增每等分试样约10mbdna,例如根据起始dna量为约30,000倍。
依照本发明的lfr方法的一个实施方案,lfr以用5’外切核酸酶处理基因组核酸,通常是基因组dna开始,以创建3’单链突出物。此类单链突出物充当mda起始位点。使用外切核酸酶还消除对扩增前热或碱变性步骤的需要且不对片段群体引入偏爱。在另一个实施方案中,将碱变性与5’外切核酸酶处理组合,这导致偏爱的降低大于用任一单独处理看到的情况。然后,将用5’外切核酸酶并任选地用碱变性处理的dna稀释至亚基因组浓度,并在多个等分试样间分散,如上文讨论的。在分成等分试样后,例如在多个孔间,将每个等分试样中的片段扩增。
在一个实施方案中,使用基于phi29的多重置换扩增(mda)。许多研究已经检查了不想要的扩增偏爱的范围、背景产物形成和经由基于phi29的mda引入的嵌合矫作物,但是这些缺点中的许多已经在极端扩增条件(大于100万倍)下发生。通常,lfr采用实质上较低的扩增水平,并且以长dna片段(例如约100kb)开始,这产生有效的mda和更可接受的扩增偏爱水平及其它扩增相关问题。
我们已经开发出改进的mda方案以克服与使用各种添加物(例如dna修饰酶、糖和/或化学品,如dmso)的mda有关的问题,和/或降低、提高或取代mda反应条件的不同组分以进一步改进方案。为了使嵌合物最小化,也可以包含如下的试剂,其用以降低起不正确模板作用用于延伸的dna链(其是嵌合物形成的常见机制)的置换单链dna的利用度。由mda引入的覆盖偏爱的主要来源是由富含gc的区域对富含at的区域之间的扩增差异引起。这可以通过使用mda反应中的不同试剂和/或通过调节引物浓度以产生在基因组的所有%gc区间均匀引发的环境来校正。在一些实施方案中,在引发mda中使用随机六聚体。在其它实施方案中,利用其它引物设计来降低偏爱。在别的实施方案中,在mda之前或期间使用5’外切核酸酶可以帮助启动低偏爱成功引发,特别是用较长(即200kb至1mb)片段进行,所述较长片段可用于测序以长区段复制(即在一些癌细胞中)和复杂重复为特征的区域。
在一些实施方案中,使用改进的、更有效的片段化和连接步骤,其将制备样品需要的mda扩增轮次数目降低多达10,000倍,这进一步降低源自mda的偏爱和嵌合物形成。
在一些实施方案中,mda反应设计为将尿嘧啶引入扩增产物中以准备好进行core片段化。在一些实施方案中,利用随机六聚体的标准mda反应用于扩增每孔中的片段;或者,可以使用随机8聚体引物来降低片段群体中的扩增偏爱(例如gc偏爱)。在别的实施方案中,也可以对mda反应添加几种不同酶以降低扩增偏爱。例如,可以使用低浓度的非进行性5’外切核酸酶和/或单链结合蛋白来创建8聚体的结合位点。也可以使用化学剂诸如甜菜碱、dmso和海藻糖来降低偏爱。
在扩增每个等分试样中的片段后,任选地,可以将扩增产物进行另一轮片段化处理。在一些实施方案中,core方法用于进一步片段化扩增后每个等分试样中的片段。在此类实施方案中,每个等分试样中的片段的mda扩增设计为将尿嘧啶掺入mda产物中。用尿嘧啶dna糖基化酶(udg),dna糖基化酶-裂合酶内切核酸酶viii和t4多核苷酸激酶的混合物处理含有mda产物的每个等分试样以切除尿嘧啶碱基,并创建具有官能性5’磷酸根和3’羟基基团的单碱基缺口。经由使用聚合酶诸如taq聚合酶的切口平移导致双链平端断裂,这生成依赖于mda反应中添加的dutp浓度的大小范围的可连接片段。在一些实施方案中,使用的core方法涉及通过phi29聚合和链置换除去尿嘧啶。也可以经由超声处理或酶促处理实现mda产物的片段化。可以在此实施方案中使用的酶促处理包括但不限于dna酶i、t7内切核酸酶i、微球菌核酸酶,等等。
在mda产物片段化后,可以修复所得片段的末端。许多片段化技术可以生成具有突出端的末端和具有不可用于后来的连接反应的官能团,诸如3’和5’羟基基团和/或3’和5’磷酸根基团的末端。具有修复成具有平端的片段可以是有用的。也可以期望修饰末端以添加或除去磷酸根和羟基基团,从而阻止靶序列的“聚合”。例如,磷酸酶可以用于消除磷酸根基团,使得所有末端含有羟基基团。然后,可以将每个末端选择性改变以容许期望组分间的连接。然后,可以通过碱性磷酸酶处理“活化”片段的一个末端。然后,可以用衔接头使片段加标签以鉴定来自lfr方法中相同等分试样的片段。
给每个等分试样中的片段加标签。扩增后,使每个等分试样中的dna加标签,从而鉴定每个片段起源的等分试样。在别的实施方案中,可以在用衔接头加标签前进一步片段化每个等分试样中的扩增dna,使得来自相同等分试样的片段均会包含相同标签;见例如us2007/0072208,其在此通过提及并入。
依照一个实施方案,在两个区段中设计衔接头:一个区段对于所有孔而言是共同的,并且平端使用本文中进一步描述的方法直接连接片段。“共同”衔接头作为两个衔接头臂添加:一个臂是与片段的5’端连接的平端,而另一个臂是与片段的3’端连接的平端。加标签衔接头的第二区段是对于每孔独特的“条形码”区段。此条形码一般是独特的核苷酸序列,并且对特定孔中的每个片段给予相同条形码。如此,在将来自所有孔的加标签片段重新组合以进行测序应用时,可以经由鉴定条形码衔接头鉴定来自同一孔的片段。将条形码与共同衔接头臂的5’端连接。可以将共同衔接头和条形码衔接头与片段序贯或同时连接。如本文中会更为详细描述的,共同衔接头和条形码衔接头的末端可以修饰为使得每个衔接头区段会以正确方向且与正确的分子连接。此类修饰通过确保片段不能彼此连接,且衔接头区段仅能够以例示的方向连接来防止衔接头区段或片段的“聚合”。
在别的实施方案中,对用于使每孔中的片段加标签的衔接头利用三区段设计。此实施方案与上文描述的条形码衔接头设计类似,只是条形码衔接头区段分成两个区段。此设计容许一大批可能的条形码,其通过容许组合条形码衔接头区段通过将不同条形码区段连接在一起以形成完全条形码区段生成来实现。此组合设计在减少需要生成的完全大小条形码衔接头数目的情况下提供可能的条形码衔接头的较大全集。在别的实施方案中,用8-12个碱基对误差校正条形码实现每个等分试样的独特鉴定。在一些实施方案中,使用与孔相同数目的衔接头(上述非限制性例子中为384和1536)。在别的实施方案中,通过基于两组40个半条形码衔接头的新组合加标签方法降低与生成衔接头有关的成本。
在一个实施方案中,库构建涉及使用两个不同衔接头。a和b衔接头容易修饰为各含有不同半条形码序列以产生数千个组合。在别的实施方案中,在相同衔接头上掺入条形码序列。这可以通过将b衔接头分成两个部分来实现,所述两个部分各具有以用于连接的共同突出序列分开的半条形码序列。两个标签组分各具有4-6个碱基。8碱基(2x4个碱基)标签组能够独特地使65,000个等分试样加标签。一个额外的碱基(2x5个碱基)会容许误差检测,并且12个碱基标签(2x6个碱基,1200万个独特的条形码序列)可以设计为容许在10,000或更多个等分试样中使用reed-solomon设计的实质性误差检测和校正(美国专利申请12/697,995,以us2010/0199155公布,其通过提及并入本文)。2x5碱基和2x6碱基标签两者都可以包括使用简并碱基(即“百搭”)来实现最佳解码效率。
在使每孔中的片段加标签后,将所有片段组合或合并以形成单一群体。然后,这些片段可以用于生成核酸模板或库构建体,用于测序。从这些加标签的片段生成的核酸模板根据与每个片段附接的条形码标签衔接头会可鉴定为属于特定孔。
长片段读取(lfr)技术
概述
个体人基因组在自然界中是二倍体的,半数的同源染色体源自每个亲本。在每个单个染色体上发生变异的背景对基因组的基因和其它转录区的表达和调节可以具有深远的影响。此外,测定两个潜在有害的突变是否在基因的一个或两个等位基因内发生具有极为重要的临床意义。
用于全基因组测序的本方法缺乏以划算的方式分开装配亲本染色体并描述变异共同发生的背景(单元型)的能力。模拟实验显示了染色体水平单元型测定需要至少70-100kb范围间的等位基因连锁信息。这用使用扩增dna的现有技术不能实现,所述现有技术由于难以一致扩增长dna分子和测序中连锁信息损失而限于小于1000个碱基的读取结果。配对技术可以提供延长的读取结果长度的等值,但是由于生成此类dna库的低效率(由于长度长于几kb的环状dna的困难所致)而限于小于10kb。此方法还需要极端的读取结果覆盖以联系所有杂合子。
对大于100kb的dna片段进行单分子测序如果是可行的,则当单分子测序的准确度较高、且检测/仪器成本较低时,该种测序可用于单元型测定。这非常难以以高产率对短分子实现,更别说对100kb片段实现。
已经在短读取结果长度(<200bp)、高度并行化系统上实施新近的人基因组测序,以几百纳克dna开始。这些技术在快速且经济地产生大量数据方面是卓越的。不幸地,经常与小配对缺口大小(500bp-10kb)成对的短读取结果消除超出几千碱基的大部分snp相信息(mckernanetal.,genomeres.19:1527,2009)。此外,在没有由于剪切所致的片段化的多个处理步骤中非常难以维持较长的dna片段。
目前,三个个人基因组,即j.craigventer(levyetal.,plosbiol.5:e254,2007)的三个个人基因组(一个印度古吉特拉裔(hapmap样品na20847;kitzmanetal.,nat.biotechnol.29:59,2011)和两个欧洲裔(maxplanckone[mp1];suketal.,genomeres.,2011;genome.cshlp.org/content/early/2011/09/02/gr.125047.111.full.pdf;andhapmapsamplena12878;duitamaetal.,nucl.acidsres.40:2041-2053,2012))已经进行了测序并且以二倍体装配。全部均涉及了以与构建人参照基因组期间使用的细菌人工染色体(bac)测序类似的方法将长dna片段克隆到构建体中(venteretal.,science291:1304,2001;landeretal.,nature409:860,2001)。虽然这些方法生成较长的定相重叠群(350kb[levyetal.,plosbiol.5:e254,2007]、386kb[kitzmanetal.,nat.biotechnol.29:59-63,2011]和1mb[suketal.,genomeres.21:1672-1685,2011]的n50s),它们需要大量初始dna、广泛的库处理,并且太昂贵以致不能用于常规的临床环境。
另外,全染色体单元型测定已经经由直接分离中期染色体得到证明(zhangetal.,nat.genet.38:382-387,2006;maetal.,nat.methods7:299-301,2010;fanetal.,nat.biotechnol.29:51-57,2011;yangetal.,proc.natl.acad.sci.usa108:12-17,2011)。这些方法对于远程单元型测定是卓越的,但是尚未用于全基因组测序,并且需要制备和分离全中期染色体,其对于一些临床样品可以是挑战性的。
lfr方法克服这些限制。lfr包括与相关算法和软件一起的dna制备和加标签,从而以显著降低的实验和计算成本在二倍体基因组中实现亲本染色体的分开序列的精确装配(即完全单元型测定)。
lfr基于多个不同等分试样间基因组dna(或其它核酸)的长片段的物理分离,从而存在有在相同等分试样中呈现的母本和父本组分两者的基因组的任何给定区的低几率。通过在每个等分试样中放置独特的标识符并总计分析多个等分试样,dna序列数据可以装配成二倍体基因组,例如可以测定每个亲本染色体的序列。lfr不需要将复杂核酸的片段克隆到载体中,如在使用大片段(例如bac)库的单元型测定方法中一样。lfr也不需要直接分离生物体的各个染色体。最后,lfr可以对个体生物体实施,并且不需要生物体群体来实现单元型定相。
如本文中使用的,术语“载体”意指插入外来dna片段的质粒或病毒载体。载体用于将外来dna导入合适的宿主细胞中,其中载体和插入的外来dna由于载体中存在例如功能性复制起点或自主复制序列而复制。如本文中使用的,术语“克隆”指dna片段对载体的插入及合适宿主细胞中具有插入的外来dna的载体的复制。
lfr可以与本文中详细讨论的测序方法一起使用,且更一般地作为与本领域中已知的任何测序技术,包括短读取结果和较长读取结果方法两者一起的预处理方法使用。lfr也可以与各种类型的分析结合使用,所述分析包括例如分析转录物组、甲基化组,等等。由于它需要非常少的输入dna,lfr可以用于对一个或少量细胞测序并测定单元型,这对于癌症、产前诊断学、和个人化医学可以是特别重要的。这可以促进家族型遗传病的鉴定,等等。通过使得有可能区别来自二倍体样品中两组染色体的响应,lfr也容许低覆盖的变体和非变体位置的较高置信度响应。lfr的其它应用包括解析癌症基因组中的广泛重排和可变剪接转录物的全长测序。
lfr可以用于处理并分析复杂核酸,包括但不限于基因组dna,其是纯化的或未纯化的,包括受到温和破坏以释放此类复杂核酸而不剪切和多度片段化此类复杂核酸的细胞和组织。
在一个方面,lfr产生长度约100-1000kb的虚读取结果长度。
另外,lfr也可以显著降低计算需要和任何短读取结果技术的关联成本。重要地,lfr消除对延长读取结果测序结果长度(若其降低总体产率)的需要。lfr的别的益处是可以源自目前的测序技术的误差或有疑问碱基响应的实质性(10至1000倍)降低,通常每100kb为1个,或每个人染色体基因组为30,000个假阳性响应,以及每个人基因组相似数目的未检出变体。误差的此显著降低使对追踪检测变体的构造的需要最小化,并且便于采用人基因组测序进行诊断应用。
除可适用于所有测序平台外,基于lfr的测序可以适用于任何应用,包括但不限于癌症基因组中结构重排的研究、全甲基化组分析,包括甲基化位点的单元型,和甚至对复杂多倍体基因组,如植物中存在的基因组的宏基因组学或新基因组测序的重新装配应用。
与仅亲本或相关染色体的共有序列形成对比,lfr提供了获得各个染色体的真实序列的能力(尽管其高相似性和长重复和区段复制的存在)。为了产生此类数据,一般在长dna范围,诸如100kb至1mb里建立序列的连续性。
本发明的又一个方面包括用于有效利用lfr数据进行全染色体单元型和结构变异定位及假阳性/阴性误差校正到少于每个人染色体300个误差的软件和算法。
在又一个方面,本发明的lfr技术根据使用的等分试样和细胞数目将每个等分试样中的dna复杂性降低100-1000倍。大于100kb的长dna中的复杂性降低和单元型分离可以有助于更有效且划算地(成本降低多至100倍)装配,并且检测人和其它二倍体基因组中的所有变异。
本文中描述的lfr方法可以作为预处理步骤使用,用于使用本领域中已知的任何测序方法对二倍体基因组测序。在其它实施方案中,本文中描述的lfr方法可以在许多测序平台上使用,所述测序平台包括例如但不限于基于聚合酶的合成测序(例如hiseq2500系统,illumina,sandiego,ca)、基于连接的测序(例如solid5500,lifetechnologiescorporation,carlsbad,ca)、离子半导体测序(例如离子pgm或离子质子测序仪,lifetechnologiescorporation,carlsbad,ca)、零模波导(例如pacbiors测序仪,pacificbiosciences,menlopark,ca)、纳米孔测序(例如oxfordnanoporetechnologiesltd.,oxford,unitedkingdom)、焦磷酸测序(例如454lifesciences,branford,ct)或其它测序技术。这些中的一些测序技术是短读取结果技术,但是其它技术产生较长的读取结果,例如gsflx+(454lifesciences;多至1000bp)、pacbiors(pacificbiosciences;约1000bp)和纳米孔测序(oxfordnanoporetechnologiesltd.;100kb)。对于单元型定相,较长的读取结果是有利的,需要少得多的计算,尽管它们趋于具有较高的误差率,并且可能需要在单元型定相前依照本文中列出的方法鉴定并校正此类长读取结果中的误差。
依照本发明的一个实施方案,lfr的基本步骤包括:(1)将复杂核酸(例如基因组dna)的长片段分成等分试样,每个等分试样含有一份基因组当量的dna;(2)扩增每个等分试样中的基因组片段;(3)片段化扩增的基因组片段以创建适合于库构建的大小的短片段(例如,在一个实施方案中长度约500个碱基);(4)使短片段加标签以容许鉴定短片段起源的等分试样;(5)合并加标签的片段;(6)对合并的、加标签的片段测序;并(7)分析所得序列数据以定位并装配数据并获得单元型信息。依照一个实施方案,lfr使用在每孔中具有10-20%单倍体基因组的384孔板,产生每个片段的母本和父本等位基因两者的理论19-38x物理覆盖。初始dna冗余19-38x确保完整的基因组覆盖及较高的变体响应和定相准确度。lfr避免复杂核酸片段对载体的亚克隆或者对分离各个染色体(例如中期染色体)的需要,并且它可以是完全自动化的,使得它适合于高通量、划算的应用。
为了误差降低和本文中详述的其它目的,我们还已经开发出使用lfr的技术。lfr方法已经披露于美国专利申请no.12/816,365,12/329,365,12/266,385,和12/265,593,及美国专利no.7,906,285,7,901,891,和7,709,197,它们全部在此通过提及完整并入。
如本文中使用的,术语“单元型”意指染色体上邻近位置(基因座)处一起传递的等位基因组合,或备选地,染色体对的单一染色体上统计学关联的一组序列变体。每个人个体具有两组染色体,即一个父本和另一个母本。通常,dna测序仅产生基因型信息,即沿着dna区段的无序等位基因的序列。为基因型推断单元型将每个无序对中的等位基因分成两个各称作单元型的不同序列。单元型信息对于许多不同类型的遗传分析(包括疾病关联研究和对群体祖先进行推断)是必要的。
如本文中使用的,术语“定相(phasing)”(或解析(resolution))意指将序列数据分类成两组亲本染色体或单元型。单元型定相指接受一个个体或一个群体(即不止一个个体)的一组基因型作为输入,并输出每个个体的一对单元型(一个是父本的,而另一个是母本的)的问题。定相可以涉及解析基因组的区域的序列数据,或少到仅仅是读取结果或重叠群中的两个序列变体,其可以称为局部定相或微定相。它还可以涉及较大重叠群(一般包括约10个以上序列变体)或甚至全基因组序列的定相,其可以称为“通用定相(universalphasing)”。任选地,在基因组装配期间对序列变体进行定相。
等分取样多份基因组当量的复杂核酸
lfr方法基于将长片段中的基因组随机物理分成多个等分试样,使得每个等分试样含有一份单倍体基因组。随着每个集合中基因组的分数降低,具有来自相同集合中的两个亲本染色体的相应片段的统计学概率显著减小。
在一些实施方案中,将10%的基因组当量等分取样到多孔板的每孔中。在其它实施方案中,将1%至50%的基因组当量的复杂核酸等分取样每孔中。如上文记录的,等分试样和基因组当量的数目可以取决于等分试样的数目、初始片段大小或其它因素。任选地,在等分取样前使双链核酸(例如人基因组)变性;如此,可以将单链互补物分配成不同等分试样。依照一个实施方案,每个等分试样包含复杂核酸的大多数链的2、4、6或更多个拷贝(或互补物)(或2、4、6或更多个互补物,若在等分取样前使双链核酸变性)。
例如,在每个等分试样0.1个基因组当量时(在每份人类基因组约6.6pg时,约0.66皮克或pgdna),两个片段会重叠有10%概率,且那些片段会源自不同亲本染色体有50%概率;这产生等分试样中的95%碱基对是非重叠的,即特定等分试样对于给定片段会不提供信息的5%总体概率,因为等分试样含有源自母本和父本染色体两者的片段。可以鉴定不提供信息的等分试样,因为源自此类等分试样的序列数据含有增加的“噪音”量,也就是说,杂合性对间连接矩阵的杂质。模糊干扰系统(fis)容许针对某种程度杂质的稳健性,即,尽管有杂质(多至某个程度),它可以进行正确的连接。甚至可以使用较小量的基因组dna,特别是在微滴或纳米滴或乳剂的背景中,其中每滴可以包含一个dna片段(例如基因组dna的单一50kb片段或约1.5x10-5个基因组当量)。甚至在50%的基因组当量,大多数等分试样会是提供信息的。在较高的水平,例如70%基因组当量,可以鉴定并使用提供信息的孔。依照本发明的一个方面,0.000015,0.0001,0.001,0.01,0.1,1,5,10,15,20,25,40,50,60,或70%基因组当量的复杂核酸存在于每个等分试样中。
应当领会,稀释因子可以取决于片段的初始大小。也就是说,使用温和技术来分离基因组dna,可以获得大约100kb的片段,然后,将该片段等分取样。容许较大片段的技术导致需要较少的等分试样,并且生成较短片段的技术可以需要更多稀释。
我们已经在没有dna纯化的同一反应中成功实施所有6个酶促步骤,这促进小型化和自动化,而且使得使lfr适用于极其多种平台和样品制备方法变得可行。
依照一个实施方案,多孔板(例如384孔板)的分开孔中含有每个等分试样。然而,本领域中已知的任何合适类型的容器或系统可以用于容纳等分试样,或者可以使用微滴或乳剂实施lfr方法,如本文中描述的。依照本发明的一个实施方案,将体积降低至亚微升水平。在一个实施方案中,可以在1536孔形式中使用自动化移液方法。
一般地,随着等分试样数目增加,例如增加至1536,且基因组的百分比下降到约1%单倍体基因组,单元型的统计学支持显著增加,因为同一孔中的母本和父本单元型两者的偶发存在减少。因此,每个等分试样具有忽略的混合单元型频率的大量小等分试样容许使用较少的细胞。类似地,较长的片段(例如300kb或更长)有助于桥接缺乏杂合基因座的区段。
提供50-100nl无触点移液的纳升(nl)分配工具(例如hamiltonroboticsnano移液头,ttplabtechmosquito,等等)可以用于快速且低成本移液以平行产生数十个基因组库。等分试样数目增加(与384孔板相比)导致每孔内基因组复杂性的较大降低,这使总体计算成本降低超过10倍并且提高数据质量。另外,此方法的自动化提高通量,并且降低产生库的动手成本。
使用较小等分试样体积(包括微滴和乳剂)的lfr
甚至可以使用微滴实现进一步的成本降低和其它优点。在一些实施方案中,在乳剂或微流控装置中用组合加标签实施lfr。在10,000个等分试样中体积下降至皮升水平可以由于较低的试剂和计算成本而实现甚至更大的成本降低。
在一个实施方案中,lfr在384孔形式中对每孔使用10微升(μl)体积的试剂。例如,可以通过在1536孔形式中使用商品化自动化移液方法降低至此类体积。进一步的体积降低可以使用提供50-100nl无触点移液的纳升(nl)分配工具(例如hamiltonroboticsnano移液头,ttplabtechmosquito,等等)实现,该纳升(nl)分配工具可以用于快速且低成本移液以平行产生数十个基因组库。增加等分试样数目导致每孔内基因组复杂性的较大降低,这使总体计算成本降低并且提高数据质量。另外,此方法的自动化提高通量,并且降低产生库的成本。
在别的实施方案中,用8-12个碱基对误差校正条形码实现每个等分试样的独特鉴定。在一些实施方案中,使用与孔相同数目的衔接头。
在别的实施方案中,使用一种新颖的组合加标签方法,其基于两组40个半条形码衔接头。在一个实施方案中,库构建涉及使用两个不同衔接头。a和b衔接头容易修饰为各含有不同半条形码序列以产生数千个组合。在别的实施方案中,在相同衔接头上掺入条形码序列。这可以通过将b衔接头分成两个部分来实现,所述两个部分各具有以用于连接的共同突出序列分开的半条形码序列。两个标签组分各具有4-6个碱基。8碱基(2x4个碱基)标签组能够独特地使65,000个等分试样加标签。一个额外的碱基(2x5个碱基)会容许误差检测,并且12个碱基标签(2x6个碱基,1200万个独特的条形码序列)可以设计为容许在10,000或更多个等分试样中使用reed-solomon设计的实质性误差检测和校正。在例示性的实施方案中,采用2x5碱基和2x6碱基标签两者,包括使用简并碱基(即“百搭(wild-cards)”)来实现最佳解码效率。
体积下降至皮升水平(例如在10,000个等分试样中)可以实现甚至更大的试剂和计算成本降低。在一些实施方案中,此水平的成本降低和广泛等分取样经由将lfr方法与组合加标签组合到乳剂或微流控型装置来实现。在没有dna纯化的同一反应中实施所有酶促步骤的能力促进使此方法小型化和自动化的能力,而且导致对极其多种平台和样品制备方法的适应性。
在一个实施方案中,lfr方法与乳剂型装置结合使用。使lfr适合于乳剂型装置的第一步是制备每滴具有单一独特条形码的有条形码标签的组合衔接头的乳剂试剂。两组100个半条形码足以独特鉴定10,000个等分试样。然而,将半条形码衔接头的数目增加至超过300可以容许以任何两个等分试样含有相同条形码组合的低概率随机添加要与样品dna组合的条形码液滴。可以生成组合条形码衔接头液滴,并在单管中以试剂贮存,用于数千个lfr库。
在一个实施方案中,将本发明从10,000扩大至100,000或更多个等分试样库。在别的实施方案中,通过增加初始半条形码衔接头的数目使lfr方法适合于进行此类扩大。然后,将这些组合衔接头液滴与含有代表小于1%单倍体基因组的准备好连接的dna的液滴一对一融合。使用保守估值每个液滴1nl和10,000个液滴,这代表对于整个lfr库为总体积10μl。
最近的研究还已经提出通过将反应体积下降至纳升大小得到的扩增(例如通过mda进行)后gc偏爱改善和背景扩增降低。
目前有几类微流控装置(例如由advancedliquidlogic,morrisville,nc出售的装置)或皮/纳滴(例如raindancetechnologies,lexington,ma),其具有皮/纳滴生成、融合(3000/秒)和收集功能,并且可以在lfr的此类实施方案中使用。在其它实施方案中,使用改善的纳移液或声学液滴喷出技术(例如labcyteinc.,sunnyvale,ca)或使用能够处理多至9216个单个反应孔的微流控装置(例如由fluidigm,southsanfrancisco,ca生产的装置),将约10-20纳升滴在3072-6144以上形式(仍然是划算的总mda体积60μl,且不丧失计算成本节约或对来自少量细胞的基因组dna测序的能力)中在板中或在玻璃载玻片上放置。增加等分试样数目导致每孔内基因组复杂性的较大降低,这使总体计算成本降低并且提高数据质量。另外,此方法的自动化提高通量,并且降低产生库的成本。
扩增
依照一个实施方案,lfr方法以用5’外切核酸酶对基因组dna的短处理开始以创建充当mda起始位点的3’单链突出。外切核酸酶的使用消除对扩增前热或碱变性步骤的需要且不将偏爱引入片段群体中。碱变性可以与5’外切核酸酶处理组合,这导致偏爱的进一步降低。然后,将dna稀释至亚基因组浓度并等分取样。在等分取样后,例如,使用mda方法扩增每孔中的片段。在某些实施方案中,mda反应是一种改良的基于phi29聚合酶的扩增反应,尽管可以使用另一种已知的扩增方法。
在一些实施方案中,mda反应设计为将尿嘧啶引入扩增产物中。在一些实施方案中,利用随机六聚体的标准mda反应用于扩增每孔中的片段。在许多实施方案中,与随机六聚体不同,使用随机8聚体引物来降低片段群体中的扩增偏爱。在其它实施方案中,也可以将几种不同酶添加至mda反应以降低扩增偏爱。例如,可以使用低浓度的非进行性5’外切核酸酶和/或单链结合蛋白来创建8聚体的结合位点。化学剂诸如甜菜碱、dmso和海藻糖也可以用于经由相似的机制降低偏爱。
片段化
依照一个实施方案,在每孔中的dna扩增后,将扩增产物进行一轮片段化处理。在一些实施方案中,在扩增后使用上文描述的core方法将每孔中的片段进一步片段化。为了使用core方法,用于扩增每孔中的片段的mda反应设计为将尿嘧啶掺入mda产物中。也可以经由超声处理或酶促处理实现mda产物的片段化。
若使用core方法来片段化mda产物,则用尿嘧啶dna糖基化酶(udg)、dna糖基化酶-裂合酶内切核酸酶viii和t4多核苷酸激酶的混合物处理含有扩增dna的每孔以切除尿嘧啶碱基并创建具有官能性5’磷酸根和3’羟基基团的单一碱基缺口。经由使用聚合酶诸如taq聚合酶的切口平移导致双链平端断裂,这生成依赖于mda反应中添加的dutp浓度的大小范围的可连接片段。在一些实施方案中,使用的core方法涉及通过phi29聚合和链置换除去尿嘧啶。
mda产物的片段化后,可以修复所得片段的末端。此类修复可以是必要的,因为许多片段化技术可以生成具有突出端的末端和具有不可用于后来的连接反应的官能团,诸如3’和5’羟基基团和/或3’和5’磷酸根基团的末端。在本发明的许多方面,具有修复成具有平端的片段可以是有用的,且在一些情况中,可以期望改变末端光化学,使得不存在正确的磷酸根和羟基基团方向,从而阻止靶序列的“聚合”。可以使用本领域中已知的方法提供对末端化学的控制。例如,在一些情况中,磷酸酶的使用消除所有磷酸根基团,使得所有末端含有羟基基团。然后,可以通过碱性磷酸酶处理“活化”片段的一个末端。然后,可以将每个末端选择性改变以容许期望组分间的连接。然后,可以“活化”片段的一个末端,在一些实施方案中通过用碱性磷酸酶处理进行。
在片段化及任选地末端修复后,用衔接头使片段加标签。
加标签
一般地,在两个区段中设计标签衔接头臂:一个区段对于所有孔而言是共同的,并且平端使用本文中进一步描述的方法直接连接片段。第二区段对于每个孔而言是独特的,并且含有“条形码”序列,使得在组合每孔的内容物时,可以鉴定来自每孔的片段。
依照一个实施方案,“共同”衔接头作为两个衔接头臂添加:一个臂是与片段的5’端连接的平端,而另一个臂是与片段的3’端连接的平端。加标签衔接头的第二区段是对于每孔独特的“条形码”区段。此条形码一般是独特的核苷酸序列,并且对特定孔中的每个片段给予相同条形码。如此,在将来自所有孔的加标签片段重新组合以进行测序应用时,可以经由鉴定条形码衔接头鉴定来自同一孔的片段。将条形码与共同衔接头臂的5’端连接。可以将共同衔接头和条形码衔接头与片段序贯或同时连接。共同衔接头和条形码衔接头的末端可以修饰为使得每个衔接头区段会以正确方向且与正确的分子连接。此类修饰通过确保片段不能彼此连接,且衔接头区段仅能够以例示的方向连接来防止衔接头区段或片段的“聚合”。
在别的实施方案中,对用于使每孔中的片段加标签的衔接头利用三区段设计。此实施方案与上文描述的条形码衔接头设计类似,只是条形码衔接头区段分成两个区段。此设计容许一大批可能的条形码,其通过容许组合条形码衔接头区段通过将不同条形码区段连接在一起以形成完全条形码区段生成来实现。此组合设计在减少需要生成的完全大小条形码衔接头数目的情况下提供可能的条形码衔接头的较大全集。
依照一个实施方案,在使每孔中的片段加标签后,将所有片段组合以形成单一群体。然后,这些片段可以用于生成本发明的核酸模板,用于测序。从这些加标签的片段生成的核酸模板根据与每个片段附接的条形码标签衔接头可鉴定为源自特定孔。类似地,在对标签测序后,与其附接的基因组序列也可鉴定为源自该孔。
在一些实施方案中,本文中描述的lfr方法不包括多个水平或层级的片段化/等分取样,如记载于2006年6月13日提交的美国专利申请no.11/451,692的,其通过提及完整并入本文用于所有目的。也就是说,一些实施方案仅利用一轮等分取样,并且也容许重新合并用于单一阵列的等分试样,而不是对每个等分试样使用不同阵列。
使用一个或少量细胞作为复杂核酸的来源的lfr
依照一个实施方案,lfr方法用于分析单个细胞或少量细胞的基因组。在此情况中用于分离dna的方法与上文描述的方法类似,但是可以在更小的体积中发生。
如上文讨论的,可以通过多种不同方法实现从细胞分离基因组核酸的长片段。在一个实施方案中,将细胞裂解,并用温和的离心步骤将完整的核沉淀。然后,经由蛋白酶k和rna酶消化几小时释放基因组dna。在一些实施方案中,可以处理材料以降低剩余细胞废物的浓度,此类处理是本领域中公知的,并且可以包括但不限于透析一段时间(即2-16小时)和/或稀释。由于分离核酸的此类方法不涉及许多破坏性方法(诸如乙醇沉淀、离心和涡旋振荡),基因组核酸很大程度上保持完整,产生具有超过150千碱基的长度的大多数片段。在一些实施方案中,片段的长度是约100至约750千碱基。在别的实施方案中,片段的长度是约150至约600、约200至约500、约250至约400和约300至约350千碱基。
一旦分离dna且在将其等分取样到单个孔中前,必须将基因组dna仔细片段化以避免材料的损失,特别是避免来自每个片段末端的序列的损失,因为此类材料的损失可以导致最终基因组装配中的缺口。在一个情况中,通过使用罕见的切口酶避免序列损失,所述切口酶在彼此约100kb的距离处创建聚合酶,诸如phi29聚合酶的起始位点。由于聚合酶创建新的dna链,它置换旧的链,最终结果是在聚合酶起始位点附近有重叠序列,导致非常少的序列缺失。
在一些实施方案中,5’外切核酸酶的受控使用(在mda反应之前或期间)可以促进初始dna从单细胞的多重复制,如此使经由拷贝复制得到的早期误差的增长最小化。
在一个方面,本发明的方法产生来自单细胞的质量基因组数据。假设没有dna损失,有以少量细胞(10个或更少)代替使用来自大量制备的等同量dna开始的益处。以小于10个细胞开始且对基本上所有dna精确等分取样确保基因组的任何给定区域的长片段中的一致覆盖。以5个以下细胞开始容许每个等分试样中每100kbdna片段的4倍或更大的覆盖且不使总读取结果数目增加得高于120gb(6gb二倍体基因组的20倍覆盖)。然而,大量等分试样(10,000或更多)和较长的dna片段(>200kb)对于从少数细胞测序甚至更为重要,因为对于任何给定的序列,仅有与重叠片段一样多的起始细胞数目,并且来自一个等分试样中两个亲本染色体的重叠片段的出现可以是毁灭性的信息损失。
lfr完全适合于此问题,因为它以相当于起始输入基因组dna的仅约10个细胞开始产生卓越的结果,且即使一个单细胞会提供足够的dna来实施lfr。一般地,lfr中的第一步是低偏爱全基因组扩增,其可以特别用于单细胞基因组分析。由于处理中的dna链断裂和dna损失,即使单分子测序方法也有可能会需要从单细胞的一定水平dna扩增。对单细胞测序的困难来自尝试扩增整个基因组。使用mda对细菌实施的研究已经遭受最终装配序列中大致一半基因组的损失及那些测序区间覆盖的相当大量的变化。这可以部分解释为是由于具有切口和链断裂的初始基因组dna,其不能在末端复制,如此在mda方法期间损失。lfr提供了针对此问题的解决办法,其经由在mda前创建基因组的长重叠片段进行。依照本发明的一个实施方案,为了实现这点,使用温和的方法从细胞分离基因组dna。然后,用常见的切口酶轻度处理很大程度上完整的基因组dna,从而生成半随机切口的基因组。然后,使用phi29的链置换能力来从切口聚合,创建非常长的(>200kb)重叠片段。然后,这些片段用作lfr的起始模板。
使用lfr的甲基化分析
在又一个方面,本发明的方法和组合物用于基因组甲基化分析。目前有几种方法可用于全局基因组甲基化分析。一种方法涉及基因组dna的硫酸氢盐处理及对重复元件或通过甲基化特异性限制酶片段化获得的基因组部分测序。此技术产生关于总体甲基化的信息,但是不提供基因座特异性数据。下一更高的分辨率水平使用dna阵列,并且受限于芯片上特征的数目。最后,最高分辨率且最昂贵的方法需要硫酸氢盐处理,接着对整个基因组测序。使用lfr,有可能对基因组的所有碱基测序,并且用关于人基因组中每个胞嘧啶位置的甲基化水平(即5-碱基测序)的数字信息装配完整的二倍体基因组。此外,lfr容许将100kb或更大的甲基化序列区组连接以对单元型测序,提供甲基化单元型测定,即不可能用任何目前可用的方法实现的信息。
在一个非限制性的例示性实施方案中,在如下的方法中获得甲基化状态,其中首先将基因组dna等分取样并变性以进行mda。接着,用亚硫酸氢盐处理dna(即需要变性的dna的步骤)。剩余的制备遵循那些记载于例如6/13/2006提交的美国申请流水号11/451,692和12/15/2008提交的12/335,168的方法,每篇在此通过提及完整并入用于所有目的且特别是用于所有涉及依照长片段读取结果技术对片段混合物的核酸分析的教导。
在一个方面,mda会扩增特定片段的每条链,其对于任何给定胞嘧啶位置独立产生50%读取结果为不受亚硫酸氢盐影响(即胞嘧啶相对的碱基鸟嘌呤不受硫酸氢盐影响)和50%提供甲基化状态。每个等分试样降低的dna复杂性有助于精确定位和装配不太提供信息的、通常3-碱基(a,t,g)读取结果。
已经报告了亚硫酸氢盐处理使dna片段化。然而,变性和硫酸氢盐缓冲液的小心滴定可以避免基因组dna的广泛片段化。在lfr中可以容许胞嘧啶对尿嘧啶的50%转变,这容许dna对亚硫酸氢盐的暴露降低以使片段化最小化。在一些实施方案中,等分取样后某个程度的片段化是可接受的,因为它不会影响单元型测定。
使用lfr来分析癌症基因组
已经提出了超过90%的癌症含有人基因组区中的显著损失或获得,称作异倍体性,已经观察到一些个别癌症含有一些染色体的超过4个拷贝。染色体和染色体内区域的拷贝数的此升高的复杂性使对癌症基因组测序实质上变得更加困难。lfr技术对非常长的(>100kb)基因组片段测序和装配的能力使得其完全适合于完整癌症基因组的测序。
通过对多个等分试样中的靶核酸测序进行的误差降低
依照一个实施方案,即使不实施基于lfr的定相并且使用标准测序方法,也将靶核酸分成多个等分试样,其各含有一定量的靶核酸。在每个等分试样中,将靶核酸片段化(若需要片段化),并且在扩增前用等分试样特异性标签(或等分试样特异性标签组)使片段加标签。或者,在处理组织样品时,可以将一个或多个细胞分配到多个等分试样之每个,之后进行细胞破坏,片段化,用等分试样特异性标签使片段加标签,并扩增。在任一情况中,可以将自每个等分试样扩增的dna分开测序或者合并并在合并后测序。此方法的一个优点是可以鉴定并校正由于扩增(或每个等分试样中发生的其它步骤)引入的误差。例如,若碱基响应存在于来自两个或更多个等分试样(或其它阈值数目)的序列数据中,或在实质上大多数预期等分试样中(例如在至少51、70或80%中),其中分母可以限于在特定位置处具有碱基响应的等分试样,则序列数据的特定位置(例如相对于参照物)处的碱基响应(例如鉴定特定碱基,诸如a,c,g或t)可以接受为真的。碱基响应可以包括改变杂合性或潜在杂合性的一个等位基因。若特定位置处的碱基响应仅存在于一个等分试样(或其它阈值数目的等分试样)中,或者在实质上少数等分试样(例如,小于10、5或3个等分试样或如用相对数目测量,诸如20或10%)中,则它可以接受为假的。阈值数值可以基于测序数据预先确定或动态确定。若特定位置处的碱基响应不存在于实质上少数中和在实质上大多数预期等分试样中(例如在40-60%中),则它可以转化/接受为“无响应”。在一些实施方案和实现中,可以使用多个参数(例如在分布、概率和/或其它函数或统计学中)来表征什么可以认为是实质上少数或实质上大多数等分试样。此类参数的例子包括但不限于下列一项或多项:鉴定特定碱基的碱基响应的数目;特定位置处响应碱基的覆盖或总数;产生包括特定碱基响应的序列数据的独特等分试样的数目和/或身份;产生包含特定位置处的至少一个碱基响应的序列数据的独特等分试样的总数;特定位置处的参照碱基;等等。在一个实施方案中,用于特定碱基响应的上述参数的组合可以输入到函数以测定特定碱基响应的得分(例如概率)。然后,可以将得分与一个或多个阈值数值比较作为测定碱基响应是否是接受的(例如高于阈值)、错误的(例如低于阈值)、还是无响应(例如若碱基响应的所有得分低于阈值)的一部分。碱基响应的确定可以依赖于其它碱基响应的得分。
作为一个基本的例子,若碱基响应a存在于超过35%(得分的例子)的含有感兴趣位置读取结果的等分试样中,且碱基响应c存在于超过35%的这些等分试样中,且其它碱基响应各具有小于20%的得分,则可以认为该位置是由a和c构成的杂合性,可能服从其它标准(例如最小数目的含有感兴趣位置处的读取结果的等分试样)。如此,每个得分可以输入到另一个函数(例如试探法,其可以使用比较或模糊逻辑)中以提供所述位置的碱基响应的最终测定。
作为另一个例子,可以使用特定数目的含有碱基响应的等分试样作为阈值。例如,在分析癌症样品时,可以有低流行体细胞突变。在此类情况中,碱基响应可以在小于10%的覆盖所述位置的等分试样中出现,但是仍然可以认为碱基响应是正确的,可能服从其它标准。如此,多个实施方案可以使用绝对数或相对数,或两者(例如作为对比较或模糊逻辑的输入)。并且,等分试样的此类数目可以输入到函数(如上文提及的),以及与每个数目对应的阈值,并且函数可以提供得分,该得分也可以与一个或多个阈值比较以做出关于特定位置处的碱基响应的最终测定。
误差校正函数的别的例子涉及原始读取结果中的序列误差,该序列误差导致与其它变体响应及其单元型不一致的推定变体响应。若变体a的20个读取结果存在于9和8个属于相应单元型的等分试样中,且变体g的7个读取结果存在于6孔(其中的5或6个与具有a读取结果的等分试样共享)中,则逻辑可以拒绝变体g为测序误差,因为对于二倍体基因组,仅一个变体可以驻留于每个单元型中的一个位置。变体a得到实质上更多阅读支持,而g读取结果实质上遵循a读取结果的等分试样,指示它们最可能是由于错误读取结果g而不是a而产生。若g读取结果几乎专门在与a分开的等分试样中,则这可以指示g读取结果错误定位或者它们来自污染性dna。
鉴定具有短串联重复的区域中的扩充(expansions)
dna中的短串联重复(str)是具有强周期性样式的dna区段。在两个或更多个核苷酸的样式重复且重复的序列彼此直接相邻时发生str;重复可以是完全的或不完全的,即可以有不匹配周期性基序的几个碱基对。一般地,所述样式的长度范围为2至5个碱基对(bp)。str通常位于非编码区中,例如在内含子中。在同源str基因座在个体间的重复数目上有所不同时发生短串联重复多态性(strp)。str分析经常用于测定遗传概况,用于法庭目的。存在于基因外显子中的str可以代表与人疾病关联的超突变区(madsenetal,bmcgenomics9:410,2008)。
在人基因组(和其它生物体的基因组)中,str包括三核苷酸重复,例如ctg或cag重复。三核苷酸重复扩充,又称为三联体重复扩充,是由dna复制期间的滑动引起的,并且与分类为三核苷酸重复病症诸如亨廷顿病的某些疾病有关。一般地,扩充越大,越有可能引起疾病或提高疾病的严重性。此特性导致三核苷酸重复病症中看到的“早现”特征,也就是说,由于这些重复的扩充经过受累家族的连续世代疾病发作的年龄减小和症状严重性升高的趋势。鉴定三核苷酸重复的扩充可以用于对三核苷酸重复病症精确预测发作年龄和疾病进展。
使用下一代测序方法,str诸如三核苷酸重复的扩充可以是难以鉴定的。此类扩充不能定位,并且在库中可以是缺少或呈现不足的。使用lfr,有可能看到str区中序列覆盖的显著下降。例如,具有str的区域与没有此类重复的区域相比在特征上会具有更低的覆盖水平,并且若存在有所述区域的扩充,则会有所述区域中覆盖的实质性降低,其在覆盖对基因组中位置的图中可观察到。
图14显示了受影响胚胎中ctg重复扩充的检测的例子。lfr用于测定胚胎的亲本单元型。在均值标准化的克隆覆盖对位置的图中,具有扩充ctg重复的单元型没有或具有非常少量的穿过扩充区的dnb,导致区域中覆盖的降低。降低也可以在两个单元型的组合序列覆盖中检出;然而,一个单元型的下降可能更难以鉴定。例如,若序列覆盖是平均约20,则具有扩充区的区域会具有显著下降,例如若受影响单元型在扩充区中具有0覆盖,则下降至10。如此,会发生50%下降。然而,若比较两个单元型的序列覆盖,则覆盖在正常单元型中是10,而在受影响单元型中是0,这是下降10,但是总体百分比下降100%。或者,可以分析相对量,其对于组合序列覆盖是2:1(正常对扩充区中的覆盖),但是是10:0(单元型1对单元型2),这是无穷大或0(取决于如何形成比率),如此是较大的区别。
序列数据的诊断用途
使用本发明方法产生的序列数据可用于极其多种目的。依照一个实施方案,本发明的测序方法用于鉴定复杂核酸序列(例如全基因组序列)中的序列变异,例如其提供关于患者或胚胎或胎儿的特征性或医学状态,诸如胚胎或胎儿的性别或具有遗传组分的疾病(包括例如囊性纤维化病、镰状细胞贫血、马方综合征、亨延顿氏病和血色素沉着病或多种癌症,诸如乳腺癌)的存在或预后的信息。依照另一个实施方案,本发明的测序方法用于提供序列信息,其以来自患者(包括但不限于胎儿或胚胎)的1-20个细胞开始并且基于序列评估患者的特征。
癌症诊断学
全基因组测序在评估疾病的遗传基础中是一种有价值的工具。许多有遗传基础的疾病(例如囊性纤维化病)是已知的。
全基因组测序的一个应用是了解癌症。下一代测序对癌症基因组学的最重要影响是对单一患者及给定癌症类型的多个患者样品的匹配肿瘤和正常基因组再测序、分析和比较的能力。使用全基因组测序,可以考虑整个范围的序列变异,包括种系易感性基因座、体细胞单核苷酸多态性(snp)、小插入和缺失(indel)突变、拷贝数变化(cnv)和结构变体(sv)。
一般地,癌症基因组由患者的种系dna构成,所述种系dna上已经叠加体细胞基因组改变。通过测序鉴定的体细胞突变可以分类为“驱动(driver)”或“乘客”突变。所谓的驱动突变是那些通过对细胞赋予生长或存活优势而直接促成肿瘤进展的突变。乘客突变涵盖已经在细胞分裂、dna复制和修复中的误差期间获得的中性体细胞突变;这些突变可以在细胞在表型上正常时或者在新生物变化明显后获得。
在历史上,已经尝试阐明癌症的分子机制,并且已经鉴定出几个“驱动”突变或生物标志物,诸如her2/neu2。基于此类基因,已经开发出治疗性方案来特异性靶向具有已知遗传变化的肿瘤。此方法的最佳限定例子是曲妥单抗(trastuzumab)(herceptin)对乳腺癌细胞中her2/neu的靶向。然而,癌症不是简单的单成因疾病,取而代之,而是以个体间可以有所不同的遗传变化组合为特征。因此,这些对基因组的其它干扰可以使一些药物方案对某些个体变得无效。
用于全基因组测序的癌细胞可以获自全肿瘤活检(包括少量细胞的微活检),自患者的血流或其它体液分离的癌细胞,或本领域中已知的任何其它来源。
植入前遗传诊断学
本发明方法的一个应用是用于植入前遗传诊断学。约2至3%出生婴儿具有某些类型的主要出生缺陷。由于遗传物质(染色体)的遗传分离所致的一些问题的风险随母亲年龄而升高。这些类型的问题的约50%机会是由于唐氏综合征,其是染色体21的第三个拷贝(三体性21)。另一半源自其它类型的染色体异常,包括三体性、点突变、结构变异、拷贝数变化,等等。许多这些染色体问题导致严重受累的婴儿或者甚至没有存活到分娩的。
在医学和(临床)遗传学中,植入前遗传诊断学(pgd或pigd)(又称为胚胎筛选)指在植入前对胚胎,有时甚至在受精前对卵母细胞实施的规程。pgd可以容许父母避免选择性妊娠终止。术语植入前遗传筛选(pgs)用于指不寻找特定疾病,但是使用pgd技术来鉴定由于例如可以导致疾病的遗传状况而有风险的胚胎的规程。在受精前对性细胞实施的规程可以取而代之称为卵母细胞选择或精子选择的方法,尽管该方法和目的与pgd部分重叠。
植入前遗传序型分析(pgp)是一种辅助生殖技术以实施胚胎选择的方法,所述胚胎似乎具有成功妊娠的最大机会。在用于晚期母体年龄的女性及用于反复体外受精(ivf)失败的患者时,主要实施pgp作为用于检测染色体异常诸如非整倍性、相互易位和罗伯逊易位、和其它异常诸如染色体倒位或缺失的筛选。另外,pgp可以对遗传标志物检查特征,包括多种疾病状态。pgp使用后面的原则是,由于已知许多染色体遗传解释大多数妊娠丧失病例,并且较大比例的人胚胎是非整倍体,整倍体胚胎的选择性替换应当提高成功ivf治疗的机会。全基因组测序提供了全面染色体分析方法,诸如阵列全面基因组杂交(acgh)、定量pcr和snp微阵列等方法的备选。例如,整个全基因组测序可以提供information关于单碱基变化、插入、缺失、结构变化和拷贝数变化的信息。
由于可以对来自不同发育阶段的细胞实施pgd,活检规程相应变化。可以在所有植入前阶段,包括但不限于未受精的和经受精的卵母细胞(对于极体,pb),对三天卵裂阶段胚胎(对于卵裂球)和对胚泡(对于滋养外胚层细胞)实施活检。
鉴于本发明的上述详细描述,依照本发明的一个方面,提供了用于对生物体(例如哺乳动物诸如人,无论是单一单个生物体或包含超过一个个体的群体)的复杂核酸测序的方法,此类方法包括:(a)等分取样复杂核酸的样品以生成多个等分试样,每个等分试样包含一定量的复杂核酸;(b)对来自每个等分试样的所述量的复杂核酸测序以从每个等分试样产生一个或多个读取结果;并(c)装配来自每个等分试样的读取结果,从而产生复杂核酸的装配序列,其在响应率70,75,80,85,90或95%或更大时每兆碱基包含不超过1,0.8,0.7,0.6,0.5,0.4,0.3,0.2,0.1,0.08,0.06,0.04或更小的假单核苷酸变体。若复杂核酸是哺乳动物(例如人)基因组,任选地,装配序列具有70%或更大的基因组响应率和70,75,80,85,90或95%或更大的外显子组响应率。依照一个实施方案,复杂核酸包含至少1千兆碱基。
依照此类方法的一个实施方案,复杂核酸是双链的,并且所述方法包括在等分取样前分开双链复杂核酸的单链。
依照另一个实施方案,此类方法包括使每个等分试样中的所述量的复杂核酸片段化,以生成复杂核酸的片段。依照一个实施方案,此类方法进一步包括用等分试样特异性标签(或等分试样特异性标签组)使每个等分试样中复杂核酸的片段加标签,通过等分试样特异性标签(或等分试样特异性标签组),可确定加标签片段起源的等分试样。在一个实施方案中,此类标签是多核苷酸,包括例如包含误差校正代码或误差校正代码,包括但不限于reed-solomon误差校正代码的标签。
依照另一个实施方案,此类方法包括在测序前合并等分试样。
依照此类方法的另一个实施方案,序列包含序列位置处的碱基响应,并且此类方法包括若碱基响应源自两个或更多个等分试样,或来自源自两个或更多个等分试样的三个或更多个读取结果,则将其鉴定为真的。
依照另一个实施方案,此类方法包括鉴定装配序列中的多个序列变体并对序列变体定相。
依照此类方法的另一个实施方案,复杂核酸的样品包含生物体的1至20个细胞或自细胞分离的基因组dna,其可以是纯化的或未纯化的。依照另一个实施方案,样品包含1pg-100ng,例如1pg,6pg,10pg,100pg,1ng,10ng或100ng基因组dna,或1pg至1ng、或1pg至100pg、或6pg至100pg。对于参照目的,单一人细胞含有约6.6pg基因组dna。
依照另一个实施方案,此类方法包括扩增每个等分试样中所述量的复杂核酸。
依照此类方法的另一个实施方案,复杂核酸选自下组:基因组、外显子组、转录物组、甲基化组、不同生物体的基因组的混合物、生物体的不同细胞类型的基因组的混合物及其亚组。
依照此类方法的另一个实施方案,装配序列具有80x,70x,60x,50x,40x,30x,20x,10x,或5x的覆盖。较低的覆盖可以与较长的读取结果一起使用。
依照本发明的另一个方面,提供了哺乳动物复杂核酸的装配序列,其在响应率70%或更大时每兆碱基包含少于1的假单核苷酸变体。
依照本发明的另一个方面,提供了对生物体的复杂核酸测序的方法,该方法包括:(a)提供包含1pg至10ng复杂核酸的样品;(b)扩增复杂核酸以生成扩增的核酸;并(c)对扩增的核酸测序以生成具有至少70%复杂核酸响应率的序列。依照一个此类方法,复杂核酸是未纯化的。依照另一个实施方案,此类方法包括通过多重置换扩增来扩增复杂核酸。依照另一个实施方案,此类方法包括将复杂核酸扩增至少10,100,1000,10,000或100,000倍或更多。依照此类方法的另一个实施方案,样品包含1至20个包含复杂核酸的细胞(或细胞核)。依照另一个实施方案,此类方法包括裂解细胞(或核),所述细胞包含复杂核酸和细胞杂质,并在存在细胞杂质的情况下扩增复杂核酸。依照此类方法的另一个实施方案,细胞是来自高等生物体的血液的循环非血细胞。依照此类方法的另一个实施方案,装配序列具有70,75,80,85,90或95%或更多的响应率。依照此类方法的另一个实施方案,序列每兆碱基包含2,1,0.8,0.7,0.6,0.5,0.4,0.3,0.2,0.1,0.08,0.06,0.04或更小的假单核苷酸变体。依照另一个实施方案,此类方法进一步包括:对样品等分取样以生成多个等分试样,每个等分试样包含一定量的复杂核酸;扩增每个等分试样中所述量的复杂核酸以在每个等分试样中生成扩增的核酸;对来自每个等分试样的扩增核酸测序以从每个等分试样产生一个或多个读取结果;并装配读取结果以产生序列。依照另一个实施方案,此类方法进一步包括:使每个等分试样中的扩增核酸片段化以在每个等分试样中生成扩增核酸的片段;并用等分试样特异性标签将每个等分试样中的扩增核酸的片段加标签以在每个等分试样中生成加标签的片段。依照此类方法的另一个实施方案,若序列位置处的碱基响应存在于来自两个或更多个等分试样的读取结果中,或更严格地,在来自两个或更多个等分试样的读取结果中出现3次或更多次,则其接受为真的。依照另一个实施方案,此类方法进一步包括鉴定序列中的序列变异,其提供关于生物体特征(例如医学状态)的信息。依照另一个实施方案,细胞是来自高等生物体的血液(或其它样品)的循环非血细胞,包括但不限于来自母亲血液的胎儿细胞和来自患有癌症的患者的血液的癌细胞。依照本发明的另一个实施方案,复杂核酸是循环核酸(cna)。如此,要评估的生物体的特征可以包括但不限于癌症的存在和关于癌症的信息(无论生物体是否是妊娠的),和关于由妊娠个体携带的胎儿的性别或遗传信息。例如,此类方法可用于鉴定与疾病可能性、医学诊断或预后等相关联的单碱基变异、插入、缺失、拷贝数变化、结构变化或重排,等等。依照本发明的另一个实施方案,提供了评估胚胎的遗传状态(例如性别、亲子关系、遗传异常的存在或缺乏或与疾病素因有关的基因型,等等)的方法,其包括:(a)提供约1-20个胚胎细胞;(b)获得通过对所述细胞的基因组dna测序产生的装配序列,其中所述装配序列具有至少80%的响应率;并(c)比较装配序列与参照序列以评估胚胎的遗传状态。例如,此类方法可用于鉴定与疾病可能性、医学诊断或预后等相关联的单碱基变异、插入、缺失、拷贝数变化、结构变化或重排,等等。依照另一个实施方案,提供了评估胚胎的遗传状态(例如性别、亲子关系、遗传异常的存在或缺乏或与疾病素因有关的基因型,等等)的方法,其包括:(a)提供约1-20个胚胎细胞;(b)获得通过对所述细胞的基因组dna测序产生的装配序列,其中所述装配序列具有至少80%胚胎基因组的响应率;并(c)比较装配序列与参照序列以评估胚胎的遗传状态。
依照本发明的另一个方面,提供了装配的全人基因组序列,该序列每兆碱基包含不超过1个假单核苷酸变体和至少70%的响应率,其中通过对1pg-10ng人基因组dna测序产生所述序列。
依照本发明的另一个方面,提供了用于对包含多个染色体的个体生物体的基因组序列变体定相的方法,该方法包括:(a)提供包含所述多个染色体中每个的无载体片段的混合物的样品;(b)对无载体片段测序以产生包含多个序列变体的基因组序列;并(c)对序列变体定相。依照一个实施方案,此类方法包括对至少70,75,80,85,90,或95%或更多的序列变体定相。依照此类方法的另一个实施方案,基因组序列具有至少70%基因组的响应率。依照此类方法的另一个实施方案,样品包含1pg至10ng基因组,或个体生物体的1至20个细胞。依照此类方法的另一个实施方案,基因组序列具有每兆碱基少于1个假单核苷酸变体。
依照本发明的另一个方面,提供了用于对包含多个染色体的个体生物体的基因组序列变体定相的方法,该方法包括:提供包含所述多个染色体的片段的样品;在没有在载体中克隆片段的情况下对片段测序以产生全基因组序列,其中全基因组序列包含多个序列变体;并对序列变体定相。依照此类方法的一个实施方案,在全基因组序列的装配期间发生对序列变体定相。
实施例
实施例1:dna扩增方法的比较
植入前遗传诊断学(pgd)是一种产前诊断学形式,其由遗传筛选体外受精(ivf)产生的胚胎(通常每个循环平均为10个),之后将它们转移到未来的母体构成。它通常适用于晚期母体年龄(大于34岁)的女性或者有传递遗传病风险的夫妇。目前用于遗传筛选的技术是荧光原位杂交(fish)、比较基因组杂交(cgh)、用于检测染色体异常的snp阵列和阵列cgh、和用于检测基因缺陷的snp阵列和pcr。用于单基因缺陷的pgd目前由对于每个患者而言独特的定制设计测定法组成,该测定法经常组合特定的突变检测与连锁分析作为备份并控制和监测污染。通常,在发育的第3天从每个胚胎活检获得1个细胞,并在第5天(其是可以转移胚胎的最近日)给出结果。开始应用胚泡活检,其由从胚泡(第5天胚胎)滋养外胚层的3-15个细胞的活检,接着是胚胎冷冻组成。胚胎可以在不显著丧失潜力的情况下无限期保持冷冻,其适合于全基因组测序,容许在一个部位获得活检,然后转移至另一个部位,用于全基因组测序。胚泡活检的全基因组测序会使得用于单一基因缺陷和可以通过此技术鉴定的其它遗传异常的“通用”pgd测试变得可能。
在常规的卵巢刺激和取卵后,通过细胞浆内精子注射(icsi)使卵受精以避免pgd测试中的精子污染。在生长到第3天后,使用细玻璃针活检取得胚胎,并从每个胚胎取出一个细胞。将每个卵裂球单独添加到干净的管,用分子级油覆盖,并在冰上运到pgd实验室。在到达后立即使用设计用于扩增基因dmpk中ctg重复扩充的突变和两个连锁标志物的测试处理样品。
在临床pgd测试和胚胎转移后,将未使用的胚胎捐赠给ivf诊所,并在开发新的pgd测试形式中使用。捐赠8个胚泡,并在这些实验中使用。
胚泡活检提供每个细胞约6.6皮克(pg)基因组dna。扩增提供足够用于全基因组测序的dna。图15显示了使用我们的方案(如下文描述的)通过mda扩增1.031pg、8.25pg和66pg纯化的基因组dna标准品和1或10个pvp40细胞的结果。可以运行mda反应,长达对于获得特定测序方法需要的dna量必要(例如30分钟至120分钟)。预期扩增程度越大,会产生越多gc偏爱。
比较两种dna扩增方法以鉴定在使gc偏爱引入最小化的情况下生成对于全基因组序列分析足够质量的模板dna的方法。我们比较我们的方案与通常用于阵列cgh的sureplex扩增(rubicongenomicsinc.,annarbor,michigan)和修改的mda。
从受到强直性肌营养不良的r-1mt突变影响的胚胎获得10-20个细胞的活检。将样品裂解,并在单一管中使dna变性,然后,使用我们的方案和sureplex试剂盒依照制造商的说明书通过mda扩增。通过这两种扩增方法生成约2ugdna。在全基因组序列分析前,用分散在基因组间的96个独立的qpcr标志物筛选扩增的样品以选择具有最低量偏爱的样品。图16显示了结果。简言之,我们测定跨整个板的平均循环数目,并将该数目从每个单独的标志物扣除以计算“△循环”数目。相对于每个标志物周围的1000个碱基对的gc含量将△循环绘图,以指示每个样品的相对gc偏爱。为了弄清样品的总体“噪音”,将每个△循环的绝对值求和以产生“△和”量度。较低的△和和相对于gc含量的相对平坦的数据绘图产生我们的经验中的呈现良好的全基因组序列。△和是61(对于我们的mda方法)和287(对于sureplex扩增的dna),指示我们的方案比sureplex方案产生少得多的gc偏爱。
实施例2:用于植入前遗传诊断学(pgd)的胚泡活检的完全基因组测序
采用修改的多重置换扩增(mda)(deanetal.(2002)procnatlacadsciusa99,5261-5266)来生成足够用于全基因组序列分析的模板dna(约1μg),如本文中描述的。简言之,将5-20个来自每个5天龄胚胞的细胞分离,冷冻,并在干冰上从分离它们的实验室运输。将样品融化,并裂解以释放基因组dna。在不纯化基因组dna使其远离细胞杂质的情况中,通过添加1μl400mmkoh/10mmedta使dna碱变性。使用基于phi29聚合酶的多重置换扩增(mda)反应对胚胎基因组dna进行全基因组扩增以生成足够量的dna(约1μg)以进行测序。碱变性后1分钟,对变性dna添加受硫醇保护的随机8聚体。在2分钟后中和混合物,并添加含有终浓度50mmtris-hcl(ph7.5),10mmmgcl2,10mm(nh4)2so4,4mmdtt,250μmdntps(usb,cleveland,oh)和12个单位的phi29聚合酶(enzymatics,beverly,ma)的主混合物以产生总反应体积100ul。将mda反应于37℃温育45分钟,并于65℃灭活5分钟。通过mda反应生成约2μgdna。然后,将此扩增的dna片段化,并用于文库构建和测序,如上文描述的。
强直性肌营养不良1型(dm1)是一种由编码强直性肌营养不良蛋白激酶(dmpk)的基因的3'非翻译区中的三核苷酸重复扩充,即胞嘧啶-胸腺嘧啶-鸟嘌呤(ctg)n引起的常染色体显性疾病。我们检查了dmpkctg重复区间的克隆覆盖。本文中描述的测序技术导致35bp配对末端读取结果,其通常跨越约400bp。对于未受累的个体和一份未知样品,400bp足以跨越两个等位基因的此ctg重复区,导致约2的拷贝数。在受累个体和一份未知样品中,观察到约1的拷贝数,提示了重复扩充对于400bp配对末端太大以致于不能跨越;仅未受累的等位基因在此区域中具有覆盖。
下文表1提供了用于定位和装配pgd胚胎样品的汇总信息。所有变异和定位统计学相对于国立生物技术信息中心(ncbi)第37版人基因组参照装配而言。样品2a、5b和5c的扩增质量较差,导致基因组的响应较少,且鉴定的snp总数降低。样品5b和5c是来自同一胚胎的不同活检。样品na20502在文库制备前按照标准规程处理且不扩增。
图17显示了两个样品(7c和10c)的基因组覆盖。使用相对于单倍体基因组覆盖标准化的100千碱基覆盖窗的10兆碱基移动平均值对覆盖绘图。拷贝数目1和3的虚线分别代表单倍体和三倍体拷贝数目。这两个胚胎是男性的,并且对于x和y染色体具有单倍体拷贝数目。没有全染色体或染色体大区段的其它丧失或获得在这些样品中是明显的。
表现最差的样品实现基因组覆盖85%,而最佳样品覆盖95%的基因组,即与通过使用几微克纯化的、未扩增的人基因组dna的上文描述的方法进行的标准全基因组测序方法(“标准测序”)相似的水平。一般地,与标准测序相比,覆盖是“嘈杂的”,但是使用10兆碱基的移动平均值容许精确检测全基因组和染色体臂扩增和缺失。我们还证明了可以检测许多多态性,并且除dmpk突变外,形成某些疾病的风险可以用于胚泡植入选择。
在此实施例中,将起始基因组dna广泛扩增(超过必要约10倍)以确保足够量的基因组dna可用于测序。预期降低扩增程度会改善序列覆盖和测序质量。也可以通过容许活检得到的组织(或其它起始材料,诸如癌症活检或针吸出物、自血流分离的胎儿或癌细胞,等等)在培养物中生长来降低扩增。此方法稍微增加方法的总体周转时间。然而,培养少量可用细胞导致染色体复制的细胞过程中基因组dna的高保真性“扩增”。
因为dmpk突变是一种三核苷酸重复疾病,使用采用长约400bp的配偶对读取结果的目前测序方法难以分析突变。较长的配偶对读取结果(例如1千碱基或更长)可以用于跨越这些区域并且因此在这些区域间测序,这导致重复大小的精确测定。
实施例3:来自10-20个人细胞的临床精确基因组测序和单元型测定
在此实施例中,将65-130pg(10-20个细胞)长人基因组dna(50%长度60-500kb)分成384个等分试样,在每个等分试样中扩增,片段化,并加标签。测序后,在没有dna克隆或中期染色体的分离的情况下装配二倍体(定相)基因组。使用10个lfr库来从7个独特基因组产生约3.3太碱基(tb)定位读取结果。将多至97%杂合单核苷酸变体(snv)装配成重叠群,其中50%覆盖碱基(n50)在长于约500kb(对于欧洲种族性样品)和约1mb(对于非洲样品)的重叠群中。在重复文库间的广泛比较中,发现lfr单元型是高度精确的,每10兆碱基(mb)具有1个假阳性snv。尽管以100皮克(pg)dna开始和10,000倍体外扩增,实现与非lfr基因组相比准确度的此20-30倍升高(drmanacetal.,science327:78,2010;roachetal.,am.j.hum.genet.89:382-397,2011),因为大多数误差与真实单元型不一致。我们已经证明了来自10-20个人细胞的划算且临床精确的基因组测序和单元型测定。
lfr技术是一种没有克隆或全中期染色体分离的划算的dna预处理步骤,其容许以临床相关成本和规模完全测序和装配不同亲本染色体。lfr可以适合于用作任何测序方法前的预处理步骤,尽管我们采用短读取结果测序技术,如上文详述描述的。
lfr可以产生长范围定相snp,因为它与长度为10-1000kb的片段的单分子测序在概念上相似。这通过在没有任何dna克隆步骤的情况下将相应的亲本dna片段随机分成物理上独特的集合,接着片段化以生成较短的片段(这与fosmid克隆的等分取样(kitzmanetal.,nat.biotechnol.29:59-63,2011;suketal.,genomeres.21:1672-1685,2011)相似)实现。由于每个集合中基因组的分数降低至小于单倍体基因组,具有来自相同集合中的两个亲本染色体的相应片段的统计学概率显著降低。同样地,询问的单个集合越多,来自母本和父本同系物的片段会在不同集合中得到分析的次数越大。
例如,在每孔中具有0.1个基因组当量的384孔板产生每个片段的母本和父本等位基因两者的理论19x覆盖。约19x的此类高初始dna冗余比使用采用fosmid集合的策略(其导致范围为约3x(kitzmanetal.,nat.biotechnol29:59-63,2011)至约6x(suketal.,genomeres.21:1672-1685,2011)的覆盖)的实现产生更完整的基因组覆盖和更高的变体响应和定相准确度。
为了以高通量方式制备lfr库,我们开发出一种自动化方法,其在同一384孔板中实施所有lfr特定步骤。以下是方法的概述。首先,使用经修改的基于phi29的多重置换扩增(mda;deanetal.,proc.natl.acad.sci.u.s.a.99:5261,2002)实施高度一致的扩增以将每个片段复制约10,000倍。接着,经由在没有居间纯化步骤的情况下在每孔内的酶促步骤过程,将dna片段化并与条形码衔接头连接。简言之,通过受控随机酶促片段化(core)将长dna分子加工成平端300-1,500bp片段。core经由除去尿苷碱基使dna片段化,所述去尿苷碱基通过尿嘧啶dna糖基化酶和内切核酸酶iv在mda过程中以预先确定的频率掺入。用大肠杆菌聚合酶1从所得的单碱基缺口进行的切口平移解决片段,并产生平端。然后,将独特的10碱基reed-solomon误差校正条形码衔接头(pct/us2010/023083,以wo2010/091107公布,其通过提及并入本文)(其设计为降低由每个条形码的序列和浓度差异引起的任何偏爱(图18))连接以使用高产率、低嵌合物形成方案(drmanacetal.,science327:78,2010)使每孔中的dna片段化。最后,将所有384孔组合,并使用与连接衔接头共同的引物采用不饱和的聚合酶链式反应以生成对于短读取结果测序平台足够的模板。以下提供了关于我们采用的lfr方案的更多详情。
使用recovereasedna分离试剂盒(agilent,lajolla,ca)遵循制造商的方案从细胞系gm12877,gm12878,gm12885,gm12886,gm12891,gm12892gm19240,和gm20431(coriellinstituteformedicalresearch,camden,nj)纯化高分子量dna。将高分子量dna部分剪切以使其更适合于使用raininp1000移液器通过移液20-40次进行的操作。使用bioradchef-drii用以下参数在具有0.5xtbe缓冲液的1%琼脂糖凝胶上分析200ng基因组dna:6v/cm,50-90秒渐变转换时间和20小时总运行。使用500ng酵母染色体pfg标志物(newenglandbiolabs,ipswich,ma)和lambdaladderpfg标志物(newenglandbiolabs,ipswich,ma)来测定纯化的基因组dna的长度。
另外,将永生化细胞系gm19240(coriellinstituteformedicalresearch,camden,nj)在细胞培养的标准环境条件下在补充有10%fbs的rpmi中培养。将单个细胞在200倍放大率下用显微操作器(eppendorf,hamburg,germany)分离,并放到1.5ml具有10uldh2o的微型管中。将细胞用1ul20mmkoh和0.5mmedta变性。然后,让变性的细胞进入lfr过程中。
将来自多个细胞系中每个的dna稀释,并在20mmkoh和0.5mmedta溶液中以50pg/ul的浓度变性。于室温温育1分钟后,将120pg变性的dna取出,并添加到32ul1mm3’受硫醇保护的随机八聚体(idt,coralville,ia)。在2分钟后,用dh2o将混合物达到体积400ul,并将1ul分配到384孔板的每孔。将1μl2x基于phi29聚合酶(enzymaticsinc.,beverly,ma)的多重置换扩增(mda)混合物添加到每孔以生成约3-10纳克dna(10,000至25,000倍扩增)。mda反应由50mmtris-hcl(ph7.5),10mmmgcl2,10mm(nh4)2so4,4mmdtt,250umdntp(usb,cleveland,oh),10um2'-脱氧尿苷5'-三磷酸(dutp)(usb,cleveland,oh),和0.25个单位的phi29聚合酶组成。
然后,实施受控随机酶促片段化(core)。使过量的核苷酸失活,并通过于37℃将mda反应与0.031个单位的虾碱性磷酸酶(sap)(usb,cleveland,oh)、0.039个单位的尿嘧啶dna糖基化酶(newenglandbiolabs,ipswich,ma)和0.078个单位的内切核酸酶iv(newenglandbiolabs,ipswich,ma)的混合物一起温育120分钟除去尿嘧啶碱基。将sap于65℃热灭活15分钟。在具有0.1纳摩尔dntp(usb,cleveland,oh)添加的相同缓冲液中用0.1个单位大肠杆菌dna聚合酶1(newenglandbiolabs,ipswich,ma)的60分钟室温切口平移解决缺口,并使dna片段化成300-1,300个碱基对片段。将大肠杆菌dna聚合酶1于65℃热灭活10分钟。通过于37℃与0.031个单位的sap(usb,cleveland,oh)一起温育60分钟除去剩余的5’磷酸根。将sap于65℃热灭活15分钟。
然后,实施加标签衔接头连接和切口平移。使用两部分定向连接方法将10个碱基的dna条形码衔接头(对于每个孔而言独特的)附着于片段化的dna。将约0.03pmol片段化mda产物于室温在总体积7ul中的反应中温育4小时,所述反应含有50mmtris‐hcl(ph7.8),2.5%peg8000,10mmmgcl2,1mmratp,100倍摩尔过量的5’‐磷酸化(5’po4)且3’双脱氧末端的(3’dd)共同ad1(图18)和75个单位的t4dna连接酶(enzymatics,beverly,ma)。ad1含有用于与独特条形码衔接头连接和杂交的共同突出区。在4小时后,将200倍摩尔过量的独特5’磷酸化的加标签衔接头添加至每孔,并容许温育16小时。将384孔组合成总体积约2.5ml,并通过添加2.5mlampure珠(beckman-coulter,brea,ca)纯化。实施一轮pcr以创建具有一侧的5’衔接头和标签和另一侧的3’平端的分子。如上文所描述的,在与5’衔接头相似的连接反应中添加3’衔接头。为了密封通过连接创建的切口,将dna在含有0.33umad1pcr1引物、10mmtris‐hcl(ph78.3)、50mmkcl、1.5mmmgcl2、1mmratp、100umdntp的反应中于60℃温育5分钟以用以3’-oh为末端的ad1pcr1引物交换3’双脱氧末端的ad1寡聚物。然后,将反应冷却至37℃,并在添加90个单位的taqdna聚合酶(newenglandbiolabs,ipswich,ma)和21600个单位的t4dna连接酶后,于37℃再温育30分钟,以通过taq催化的切口平移从ad1pcr1引物3’-oh端创建官能性5’-po4gdna端,且以通过t4dna连接密封所得的修复切口。在此点时,将材料掺入标准dna纳米阵列测序方法中。
从总rna开始,使用ovationrna-seq试剂盒(nugen,sancarlos,ca)和spriwork(beckman-coulter,brea,ca)得到rna-seq数据以制备具有平均插入物大小150-200bp的测序库。在个性化遗传医学中心(centerforpersonalizedgeneticmedicine)(harvardmedicalschool,boston,ma)在hiseq2000(illumina,sandiego,ca)上实施75bp配对末端测序反应。使用bowtiev0.12.7(langmeadetal.,genomebiol.10:r25,2009)用tophatv1.2.0(trapnelletal.,bioinformatics25:1105-1111,2009)装配配对末端读取结果,并用参照用hg19和注释已知snp的dbsnp第132版使用gatkunifiedgenotyperv1.1(http://www.broadinstitute.org/gsa/wiki/index.php/gatk_release_1.1)响应单核苷酸变体(snv)。同时将snv定位到来自refseq的基因及如cufflinksv1.0.3(http://cufflinks.cbcb.umd.edu/tutorial.html)鉴定的转录物组中的同等型。
为了鉴定共表达等位基因的单元型,过滤关于杂合snv的数据,所述杂合snv同时在相同lfr重叠群上及在具有至少一个另一杂合snv的相同基因上发生。在转录物展现出等位基因特异性表达的情况中,lfr定相单元型上表达的杂合等位基因应当比另一单元型上的其对应物均具有更高的,或均具有更低的读取结果计数。在这里,我们将较高表达的单元型鉴定为大多数其杂合等位基因比其对应物展现出更高表达的单元型。若其表达与其含有的单元型一致,则杂合计算为“一致的”。在平分的情况中(其中没有单元型多数),半数的杂合snv计算为一致的。另外,为了被完全考虑,需要杂合snv具有至少20倍rna-seq读取结果覆盖。通过随机使用二项检验与选择ase和覆盖的概率比较对杂合snv进一步过滤来自gatk基因型测定器(genotyper)的噪音。
出于误差校正目的,用具有用于未知误差位置的1碱基误差校正能力或在已知误差位置时的2碱基误差校正能力(美国专利申请12/697,995,以us2010/0199155公布,其通过提及并入本文)的10个碱基的reed-solomon码加标签每个dnb。这384个代码选自全面的一组4096个具有上述特性的reed-solomon码(美国专利申请12/697,995,其通过提及并入本文)。来自此组的每个代码具有距该组中的任何另一代码的最小汉明距离3。对于此研究,假设误差位置是未知的。
结果。为了证明lfr测定精确二倍体基因组序列的能力,我们产生约鲁巴人女性hapmap样品na19240的三个库。广泛询问na19240作为hapmapproject(consortium,nature437:1299-1320,2005;frazeretal.,nature449:851-861,2007)、1,000genomesproject(nature467:1061-1073,2010)及我们自身努力(www.completegenomics.com/sequence-data/download-data/)中三个一组的一部分(na19240是样品na19238和na19239的子代)。因此,基于亲本样品na19238和na19239的冗余序列数据,可以产生关于170万个杂合snp的高度精确单元型信息。以相应的永生化b细胞系的10个细胞(65pgdna)开始,生成1个na19240lfr库。基于总有效读取结果覆盖60x及使用384个独特的片段等分试样或集合,我们估计若将dna在分配到孔(20个细胞当量的dsdna;下文表1)中前变性,则起始细胞的最佳数目会是10。从估计的100-130pg(15-20个细胞当量)变性高分子量基因组dna产生2个重复文库。测定了在从变性的分离的dna开始时,每个库的最佳量会是约100pg。此量选择为通过使样品的随机取样最小化实现较为一致的基因组覆盖。
使用dna纳米阵列测序(drmanacetal.,science327:78-81,2010)分析所有三个库。使用定制比对算法(drmanacetal.,science327:78-81,2010;carnevalietal.,j.computationalbiol.,19,2011)将35个碱基的配偶对读取结果定位到参照基因组,以大于80x的平均基因组覆盖平均产生超过230gb定位数据(下文表1)。对定位lfr数据的分析显示了可归因于mda的2个独特特征:富含gc的序列的轻微呈现不足(图19)和嵌合序列的增加。另外,100kb窗间标准化的覆盖的可变性多约2倍。不过,几乎所有基因组区覆盖有足够的读取结果(5或更多),表明通过我们的优化方案进行的10,000倍mda扩增可以用于全面的基因组测序。
使用条形码以基于其在每个文库内的物理孔位置(其显示覆盖的脉冲,即几乎没有读取结果覆盖的长跨越间散布的覆盖的稀疏区)图形分组定位读取结果。平均每个孔含有长度范围为10kb至超过300kb的片段中10-20%的单倍体基因组(300-600mb),n50为约60kb(图20)。初始片段覆盖在染色体间是非常一致的。如从所有检测片段评估的,从提取的dna产生两个文库实际使用的dna总量是约62pg和84pg(9.4和12.7个细胞当量,图20)。这小于预期的100-130pg,指示一些损失或检测不到的dna或dna定量的不精确。令人感兴趣地,10个细胞的文库表现为从约90pg(13.6个细胞)的dna生成,最可能是由于一些细胞在分离期间处于s期所致(图20)。
使用设计为从约40个单个孔询问低覆盖读取结果数据(小于2x覆盖)的两步定制基因型测定算法,来自位于不同孔中的同一亲本染色体的片段的重叠杂合snp装配为单元型重叠群(图21)。与其他实验方法(kitzmanetal.,nat.biotechnol.29:59-63,2011;suketal.,genomeres.21:1672-1685,2011;duitamaetal.,nucl.acidsres.40:2041-2053,2012)不同,lfr并不限定每个初始片段的单元型。取而代之,lfr通过在等分试样的数目和给定读取结果覆盖方面使dna片段输入最大化来确保基因组的完全呈现。
在第一步中,将来自未定相na19240基因组装配(www.completegenomics.com/sequence-data/download-data/)的杂合snp与每个lfr库组合以创建全面的snp组用于定相。接着,对每个染色体构建网络,其中节点对应于杂合snp响应,而连接涉及每对snp间的连接性得分。与连接得分一起,还获得方向作为搜索关于每对杂合snp的最佳假设的一部分。然后,使用域知识修整此高度冗余的稀疏连接网络,随后使用kruskal的最小跨度树(mst)算法优化。这产生较长的重叠群,来自950-1200kb的n50获自这些文库(图20)。
通过lfr在每个文库中定相总共约240万个杂合snp(图20)。lfr定相预期会逐步采用这些文库的杂合snp的约90%。10个细胞的文库定相由自分离的dna生成的两个文库定相的变体的超过98%,证明lfr通过少量分离的细胞起作用的潜力。使读取结果数目倍增至约160x覆盖将定相杂合snp的数目进一步增加到超过258万,由此将定相率增加到96%(图20)。组合重复1和2(总共768个独立孔)(各具有80x覆盖)产生超过265万个定相的杂合snp,且产生97%的定相率。仅使用定相用的lfr文库中响应的snp基因座(省略lfr算法的步骤1)通常导致定相snp总数降低5-15%(图20)。
重要地,仅通过lfr(仅从10-20个细胞的dna开始)得到的定相snp数目略高于通过目前的fosmid方法(kitzmanetal.,nat.biotechnol.29:59-63,2011;suketal.,genomeres.21:1672-1685,2011;duitamaetal.,nucl.acidsres.40:2041-2053,2012)定相的snp数目。由于双亲共享儿童中的较大分数的变体,这是可以通过使用标准亲本序列(roachetal.,am.j.hum.genet.89:382-397,2011)定相的杂合snp的实质上超过81%。将亲本衍生的单元型数据添加到768孔文库将定相率改善至98%。约115,000(约4%)定相杂合snp来自高覆盖lfr文库,并且在标准文库中没有被响应,指示mda扩增和160x覆盖有助于一些区域得到足以正确响应的读取结果(5个或更多个)。可以调节高覆盖lfr定相率以平衡单元型完整性对定相误差。
欧洲裔谱系的单元型测定。为了使我们进一步了解lfr的性能,我们从欧洲人祖先谱系生成额外的文库。选择ceph家族1463,因为它具有三个世代的个体,容许全面研究遗传性。此家族先前已经作为公共数据释放(www.completegenomics.com/sequence-data/download-data/)的一部分研究。从每个世代的个体产生文库。对na12877,na12885,na12886,na12891,和na12892产生总共超过1.6tb序列数据。一般地,定相在具有定相到重叠群中的约92%的尝试snp的所有样品间是非常高的(图20)。组合两个lfr文库(图20)或lfr与基于亲本的定相将定相snp的总体比率改善到97%。所有分析家族成员间的n50重叠群长度是500-600kb。此长度限于低于na19240的长度。snp在几个不同族群的基因组间分布的调查解释此差异。
非非洲裔群体中低杂合性区域的起源和影响。在欧洲裔谱系样品中比在na19240中具有多约两倍的30kb-3mb的低杂合性区域(rlh,定义为每10kb具有小于1.4个杂合snp的30kb基因组区域,比种植密度低约7倍),澄清非非洲裔中纯合子的先前报告的相对过量(gibsonetal.,hum.mol.genet.15:789-795,2006;lohmuelleretal.,nature451:994-997,2008)并且通过分析52个完整基因组(nicholasschork,个人通信)得到进一步支持。这些区域是定相的障碍,导致小两倍的n50重叠群长度。欧洲裔基因组中的超过90%重叠群以无关个体间有所变化的这些rlh结束。
非非洲裔基因组中约3%的所有杂合snp(30-60%的所有非定相杂合snp)属于这些rlh,其覆盖非常大分数(30-40%)的这些基因组。在华裔和欧洲裔基因组中,较长的rlh对每mb聚簇45个左右的杂合snp(基因组覆盖是在rlh外部每mb的约1000个),指示它们在37,000-43,000年前左右共享共同的祖先(基于每20年世代的60-70个snp的突变率;roachetal.,science328:636-639,2010;conradetal.,nat.genet.43:712-714,2011)。这可能是由于人类离开非洲时或之后且在10,000-65,000年前的先前确定的范围内的强瓶颈所致(lianddurbin,nature475:493-496,2011)。此外,在欧洲裔和印度裔女性(na12885,na12892和na20847)中在与非洲裔女性(na19240)相比时在x染色体上观察到过量的rlh,分别涵盖此染色体的约50%对17%(对于这些相同个体中的整个基因组为30%对14%)。这指示甚至x染色体方面更强的离开非洲(out-of-africa)瓶颈。可能的解释是实质上较少的女性留在非洲,并且与多个男性具有后代。
这些观察提示了数千个多种多样的基因组中的全基因组变异分析,包括单元型测定会提供对人群体遗传学的深刻了解和这些广泛“近亲繁殖”区(其通常各包含大于100个纯合子变体)对人疾病和其他极端表型的影响。另外,它显示了长度大于100kb的约2,000个rlh会存在于所有非非洲裔个体中。具有有限数目的高频率单元型的群体(其可以源自新近的瓶颈或近亲繁殖(gibsonetal.,hum.mol.genet.15:789-795,2006))也可以具有双亲中存在的相同杂合snp的长运行,这限制亲本用于定相或分配较短的lfr重叠群。如此,群体史和一些生殖方式可以使定相变成挑战性的,如由非非洲裔女性的x染色体展现的。不管这些因素,lfr定相性能是大致等同的,在欧洲裔和非洲裔个体两者中定相多至97%的杂合snp,即应当在所有群体间转化的结果。如下文描述的组合lfr与一个亲本的标准基因型测定(会更限于一些家族的策略,如上文讨论的)外,使用长于300kb的初始dna片段(例如通过在凝胶块中俘获细胞或预纯化的dna(cook,emboj.3:1837-1842,1984))会跨越约95%的所有rlh,并对这些区域中发生的大多数重新突变测定单元型。这用限于40kb片段的目前fosmid克隆策略(kitzmanetal.,nat.biotechnol.29:59-63,2011;suketal.,genomeres.21:1672-1685,2011)会是不可行的。
lfr再现性和定相误差率分析。致力于了解lfr的再现性,我们比较两个na19240重复文库间的单元型数据。一般地,文库是非常一致的,这两个文库仅定相约220万个杂合snp中每个文库的64个差异(图22)。这代表定相误差率0.003%或44mb中的1个误差。在与自先前通过多个方法测序的亲本基因组na19238和na19239产生的保守但精确的全染色体定相相比时,lfr也是高度精确的。仅发现157万个相当的单个基因座中的约60个例子,其中lfr定相与亲本单元型测定的变体不一致的变体(若半数的不一致性是由于亲本基因组中的测序误差所致,则假定相率为0.002%)。lfr数据还含有每个文库约135个重叠群(2.2%),其具有一个或多个翻转的单元型块(图22)。将这些分析延伸到样品na12877的欧洲裔重复文库(图22)并将它们与最近使用na12877的4个儿童及其母亲na12878进行的基于家庭的高质量分析(roachetal.,am.j.hum.genet.89:382-397,2011)比较产生相似的结果,其假设每个方法贡献观察到的不一致性的一半。在na19240和na12877文库两者中,几个重叠群具有许多的翻转区段。大多数这些重叠群趋向于位于低杂合性区域(rlh)、低读取结果覆盖区、或在数目大得出乎意料的孔中观察到的重复区(例如亚端粒(subtelomeric)或着丝粒区)。
将单元型重叠群归入亲本染色体。可以通过对这些区域中的末端重叠群强加lfr定相算法校正大多数翻转误差。或者,可以通过将来自至少一个亲本的标准高密度阵列基因型数据(约100万个或更大的snp)对lfr装配的简单、低成本添加来除去这些误差。另外,我们发现了亲本基因型可以连接全染色体间98%的lfr定相的杂合snp。另外,此数据容许将单元型归入母本和父本谱系,即可用于在遗传诊断中掺入亲本印记的信息。若得不到亲本数据,也可以使用群体基因型数据来连接全染色体间的lfr重叠群,尽管此方法可以增加定相误差(browningandbrowning,nat.rev.genet.12:703-714,2011)。即使技术上有挑战的办法诸如中期染色体分离(其已经证明全染色体单元型测定)在没有某种形式的亲本基因型数据的情况下不能分配亲本起源(fanetal.,nat.biotechnol.29:51-57,2011)。两个简单技术(即lfr和亲本基因型测定)的此组合以低成本提供精确的、完全的、且注释的单元型。
定相重新突变。作为我们二倍体基因组测序的完全性和准确度的证明,我们评估最近在na19240基因组中报告的35个重新突变的定相(conradetal.,nat.genet.43:712-714,2011)。这些突变中的34个在标准基因组或lfr文库之一中响应。那些之中,在两个重复lfr文库的至少一个中定相32个重新突变(16个来自每个亲本)。不令人惊讶地,两个非定相变体驻留于rlh中。这32个变体中,通过conradetal.(同上)定相21个,并且18个与lfr定相结果一致。三个不一致性可能是由于先前研究中的误差(matthewhurles个人通信),确认lfr准确度,而不影响报告的本质结论。
仅使用lfr文库从100pgdna的基因组测序和单元型测定。上文描述的分析掺入来自标准和lfr文库两者的杂合snp。然而,鉴于由于以与10-20个细胞中存在的dna量等同的dna量开始而预期基因组的完全呈现,有可能仅使用lfr文库。我们已经证明了mda提供足够一致的扩增,且凭借高(80x)总体读取结果覆盖,单独采用的lfr文库容许在不对我们的标准文库变异-响应算法的任何修改的情况中检测多至93%的杂合snp。为了证明仅使用lfr文库的潜力,我们定相na19240重复1以及额外的250gb的来自同一文库的读取结果(总共500gb)。我们观察到定相的snp总数分别降低15%和5%(图20)。鉴于从60pgdna,代替最佳量的200pg生成此文库(下文表1)且还鉴于在通过mda的体外扩增期间掺入的先前提及的gc偏爱,此结果不是令人惊讶的。另一个285gblfr文库从组合的标准和lfr文库响应并定相仅90%的所有变体(图20)。尽管定相的总snp减少,重叠群长度很大程度上不受影响(n50>1mb)。
通过用于从10个细胞的精确基因组测序的lfr实现的误差降低。实质性误差校正(100-1,000个响应的千碱基中的约1个snv)是所有目前大规模并行化测序技术的共同属性。这些比率对于诊断用途可能是太高的,并且它们使搜索新突变的许多研究变得复杂。绝大多数假阳性变异不再可能在母本或父本染色体上发生。lfr可以利用这种缺乏与周围的真变异的一致连接性以从最终装配的单元型消除这些误差。约鲁巴人三人一组和欧洲裔谱系两者提供了用于证明lfr的误差降低能力的卓越平台。我们限定了na19240和na12877中的一组杂合snp(大于85%的所有杂合snp),其以高置信度在个人父母的每位中报告为在两个等位基因上匹配人参照基因组。存在着满足此标准的na19240中的约44,000个杂合snp和na12877中的30,000的。依靠其在亲本基因组中的不存在性,这些变异是重新突变、细胞系特异性体细胞突变、或假阳性变体。在两个来自样品na19240和na12877的重复文库中的每个可再现定相这些变体中的约1,000-1,500个(图23)。这些数目与那些对na19240中的重新和细胞系特异性突变报告的数目相似(conradetal.,nat.genet.43:712-714,2011)。剩余的变体可能是初始的假阳性,其中每个文库定相仅约500个。这代表定相的那些变异中假阳性率的60倍降低。这些假变体中仅约2,400个存在于标准文库中,其中仅定相约260个(20mb中小于1个假阳性snv;5700个单倍体mb/260个误差)。与通过标准方法测序的基因组相比,每个lfr文库展现出定相前文库特异性假阳性响应的15倍增加。大多数这些假阳性snv可能已经被mda引入;罕见的细胞系变体的取样可以造成较小的百分比。尽管从100pgdna生成lfr文库并经由mda扩增引入大量误差,应用lfr定相算法将总体测序误差率降低至99.99999%(约600个假杂合snv/6gb),即比使用相同的基于连接的测序化学观察到的误差率低约10倍(roachetal.,am.j.humangenet.89:382-397,2011)。
改善具有lfr信息的碱基响应。在定相和消除假阳性杂合snv外,lfr可以通过评估支持每个碱基响应的读取结果的孔起源来“挽救”“无响应”位置或验证其它响应(例如纯合参照或纯合变体)。作为证明,我们发现na19240重复1的基因组中没有响应,但是与邻近定相杂合snp相邻的位置。在这些例子中,所述位置能够被“再响应”,因为定相的杂合snp的确针对相邻定相snp和无响应位置之间的共享孔的存在(图24)。虽然lfr可以不能挽救所有无响应位置,但是此简单的演示突出显示lfr在更精确响应所有基因组位置以降低无响应中的有用性。
非洲裔和非非洲裔基因组中存在的高度趋异的单元型。通过大规模基因型测定研究诸如hapmap项目实现的单元型分析对于了解群体遗传学是非常重要的。然而,个体的完整单元型的解析很大程度上是难处理的或昂贵得惊人的。高度精确的单元型(过滤掉由于重复区的假定位而积累的聚簇假杂合子)(lianddurbin,nature475:493-496,2011;roachetal.,science328:636-639,2010)会有助于了解个体基因组内找到的许多群体现象。作为证明,我们对na19240的lfr重叠群扫描母本和父本拷贝之间的高趋异区。鉴定7000个含有大于33个snv的10-kb区;比预期的10个snv增加3倍。假设每100万年为0.1%持续变异(standingvariation)和0.15%碱基差异(基于从共同祖先进化约600万年的人和黑猩猩基因组的1%趋异性),我们的计算提示了此非洲裔基因组中找到的这些区域中的约50mb(约2.0%的“非近亲繁殖”基因组)可能已经分开进化超过150万年。若黑猩猩-人分开小于500万年前,此估值更接近1myr(hobolthetal.,genomeres.21:349-356,2011)。此全基因组分析与hammer等对非洲裔群体中几个靶定基因组区域(假设非洲的不同人种的可能的杂种繁殖(interbreeding))的目前研究(proc.natl.acad.sci.u.s.a.108:15123-15128,2011)一致。我们的分析显示了2.1%的欧洲裔非近亲繁殖基因组也具有类似趋异的序列,通常在不同基因组位置处。这些中的大多数可能在人类离开非洲前引入。
单个基因组含有多个在两个等位基因中都具有失活变异的基因。高度精确的二倍体基因组对于使人基因组测序对临床背景有价值是一种必需。为了证明lfr可以如何用于诊断/预后环境,我们na19240的编码snp数据分析无义和剪接位点破坏变异。我们使用polyphen2(adzhubeietal.,nat.methods7:248-249,2010)进一步分析所有错义变异以仅选择那些编码不利变化的变异。认为“可能损害”和“大概损害”两者对于蛋白质功能是不利的,因为都是无义突变。3485个变体匹配这些标准。定相和除去假阳性后,仅保留1252个变体;即潜在误导性信息的重要降低。我们进一步降低该列表以仅检查那316个杂合变体,其中至少两个在同一基因中共发生。使用定相数据,我们能够鉴定79个基因内同一等位基因中存在的189个变体。发现剩余的127个snp在47个在每个等位基因中具有至少一个不利变异的基因间分散(图25)。通过组合两个lfr文库对na19240测定单元型将此数目增加到65个基因。将此分析延伸到欧洲裔谱系证明相似数目的基因(在两个等位基因中都具有编码突变的32-49个)潜在改变至表达很少至没有有效蛋白质产物的点(图25)。将此分析延伸到破坏转录因子结合位点(tfbs)的变体引入每个个体额外的约100个基因。这些中的许多有可能是功能变化的部分损失或无损失。由于lfr的高准确度,不太可能的是,这些变体是测序误差的结果。许多发现的不利突变可以已经在这些细胞系的增殖中引入。在无关个体中发现少数这些基因,提示了它们可以是不正确注释的或者系统性定位或参照误差的结果。na19240的基因组在功能种类的完全丧失中含有额外的约10个基因;这最可能是由于通过使用欧洲裔参照基因组注释非洲裔基因组引入的偏爱所致。不过,这些数目与几个目前关于定相个别基因组的研究中找到的数目一致(suketal.,genomeres.21:1672-1685,2011;lohmuelleretal.,nature451:994-997,2008),并且提示了大多数一般健康个体可能具有正常生活不绝对需要的少量基因,其编码无效的蛋白质产物。我们已经证明了lfr能够将snp放入较大基因组距离里的单元型,其中那些snp的相可以引起潜在完全的功能丧失发生。此类信息对于患者基因组的有效临床解读及对于携带者筛选会是至关重要的。
与等位表达差异联系的tfbs破坏。涵盖顺式调节区和编码序列两者的长单元型对于了解和预测基因的每个等位基因的表达水平是至关重要的。通过分析来自对na20431的淋巴细胞的rna测序的5.6gb非穷尽表达数据,我们鉴定少量在等位基因表达方面具有显著差异的基因。在这些基因的每个中,对转录起始稳点上游的5kb调节区和下游的1kb扫描snv,该snv显著改变超过300个不同转录因子的结合位点(sandelinetal.,32:d91-d94,2004)。在六个例子中(图26),发现两个等位基因间的1-3个碱基在每个基因中存在不同,对一个或多个推定的结合位点引起显著的影响并潜在解释等位基因间观察的差异表达。虽然这仅是一个数据集并且目前并不清楚这些变化对转录因子结合具有多大的影响,但是这些结果证明凭借此类型的大规模研究(rozowskyetal.,mol.syst.biol.7:522,2011),使用lfr单元型测定变得可行的是,可以阐明对转录因子结合位点的序列变化的后果。
讨论。我们已经证明lfr将基因组中多至97%的所有检测的杂合snp精确定相到dna的长连续区段(长度为400-1500kb的n50)中的能力。即使在没有来自标准文库的候选杂合snp的情况下且如此仅使用10-20个人细胞定相的lfr库能够定相85-94%的可用snp,尽管目前的实现有限制。在几个例子中,此文章中使用的lfr文库具有小于最佳的起始输入dna(例如na20431)。通过组合两个重复文库(样品na19240和na12877)或以更多dna(na12892)开始看到的定相率改善与此结论一致。另外,富含gc的序列的呈现不足导致响应的较少基因组(90-93%对大于96%(对于标准文库))。对mda方法(例如通过添加区域特异性引物或通过改善其它步骤中的产率使用较少扩增进行)或我们实施lfr文库中碱基和变体响应的方式(可能通过使用读取结果对孔的分配进行)的改进会有助于提高这些区域中的覆盖。此外,随着全基因组测序的成本不断下降,较高覆盖的文库(其显著改善响应率和定相)会变得更加负担得起。
共有单倍体序列对于许多应用是足够的;然而,它缺乏两个非常重要的关于个人化基因组的数据的部分:定相杂合变体和假阳性和阴性变体响应的鉴定。个人基因组的目的之一是检测引起变体的疾病及极端确信测定个体是否携带此类变体或者具有一个或两个未受影响的等位基因。通过独立提供来自母本和父本染色体两者的序列信息,lfr能够检测基因组装配中仅已经覆盖一个等位基因的区域。同样地,避免假阳性响应,因为lfr在不同等分试样中将母本和父本染色体两者独立测序10-20次。结果是随机序列误差会在一个亲本等位基因上的相同碱基位置处几个等分试样中重复出现的统计学低概率。如此,lfr第一次容许对来自少数(优选是10-20个)人细胞的基因组的既精确又划算的测序,尽管使用体外dna扩增和所致的大量不可避免的聚合酶误差。此外,通过在几百个千碱基至多个兆碱基里定相snp(或在整个染色体里通过整合lfr与一个或两个亲本的常规基因型测定进行),lfr能够更精确预测复合调节变体和亲本印记对多个组织类型中等位基因特异性基因表达和功能的影响。总之,这提供了关于可以引起蛋白质功能获得或丧失的潜在基因组变化的高度精确报告。便宜地对每个患者获得的此种信息低于基因组数据的临床使用会是至关重要的。此外,从10个细胞开始的人基因组的成功且负担得起的二倍体测序打开来自多种多样的组织来源,诸如循环肿瘤细胞或经由体外受精生成的植入前胚胎的微活检的全面且精确的遗传筛选的可能性。
虽然多种不同形式的实施方案满足本发明,如结合本发明的优选实施方案详细描述的,但是应当理解,应当认为本公开内容是本发明原理例示性的,而并不意图将本发明限于本文中例示和描述的具体实施方案。本领域技术人员可以在不偏离本发明精神的前提下做出许多变化。本发明的范围会以所附权利要求书及其等同方案测量。摘要和发明名称不应解释为限制本发明的范围,因为其目的是使合适的权力机构及一般公众能够快速确定本发明的一般性质。在所附权利要求书中,除非使用术语“手段”,其中叙述的特征或要素无一应当解释为属于35u.s.c.§112,
本发明提供了以下各项:
1.测定一种或多种生物体的复杂核酸的序列的方法,该方法包括:
(a)在一个或多个计算装置上接收所述复杂核酸的多个读取结果;并
(b)用所述一个或多个计算装置从所述读取结果产生所述复杂核酸的装配序列(assembledsequence),所述装配序列在70%或更大的响应率(callrate)时每兆碱基包含不到1个假单核苷酸变体。
2.项1的方法,其进一步包括鉴定所述装配序列中的多个序列变体,并对所述多个序列变体定相(phase)以产生定相序列。
3.项2的方法,其包括对至少三个所述序列变体定相,并将与至少两个序列变体的定相不一致的序列变体鉴定为误差(error)。
4.项2的方法,其中所述装配序列是全基因组序列,所述方法包括对至少70%的所述序列变体定相。
5.项2的方法,其中所述装配序列是全基因组序列,所述方法包括对至少80%的所述序列变体定相。
6.项2的方法,其中所述装配序列是全基因组序列,所述方法包括对至少85%的所述序列变体定相。
7.项2的方法,其中所述装配序列是全基因组序列,所述方法包括对至少90%的所述序列变体定相。
8.项2的方法,其中所述装配序列是全基因组序列,所述方法包括对至少95%的所述序列变体定相。
9.项1的方法,其中接收所述复杂核酸的多个读取结果的步骤是,接收多个等分试样之每个的多个读取结果,每个等分试样包含所述复杂核酸的一个或多个片段。
10.项9的方法,其包括响应所述装配序列一个位置处的碱基,是基于两个或更多个等分试样在该位置处的初步碱基响应来进行。
11.项9的方法,其包括将两个或更多个等分试样的读取结果中出现3次或更多次的碱基响应鉴定为真的。
12.项9的方法,其中将等分试样特异性标签附着于每个所述片段,所述方法还包括,通过鉴定所述等分试样特异性标签,来确定是哪个等分试样给出了所述读取结果。
13.项12的方法,其中所述等分试样特异性标签包含误差校正代码,并且每个读取结果包含标签序列数据和片段序列数据,其中所述标签序列数据是正确的标签序列数据或包含一个或多个误差的不正确标签序列数据;该方法进一步包括:
(c)使用所述误差校正代码来校正所述不正确标签序列数据,由此产生经校正的标签序列数据和不能校正的标签序列数据;
(d)在要求标签序列数据的第一计算机方法中,使用包含所述正确标签序列数据和所述经校正的标签序列数据的读取结果,并且产生第一输出;并
(e)在不要求标签序列数据的第二计算机方法中,使用包含所述不能校正的标签序列数据的读取结果,并且产生第二输出。
14.项13的方法,其中所述第一计算机方法选自:样品多路复用、库多路复用、定相、和采用标签序列数据的误差校正方法。
15.项13的方法,其中所述第二计算机方法包括定位、装配和基于集合的统计学。
16.项13的方法,其中所述误差校正代码是reed-solomon码。
17.项1的方法,其中所述方法进一步包括:
(c)提供所述复杂核酸的一个区域的第一定相序列,所述区域包含短串联重复;
(d)将所述区域的第一定相序列的读取结果与所述区域的第二定相序列的读取结果进行比较;并
(e)基于所述比较,鉴定所述短串联重复在所述第一定相序列或所述第二定相序列之一中的扩充。
18.项1的方法,其进一步包括从所述生物体的至少一个亲本获得基因型数据,并从所述读取结果和所述至少一个亲本的基因型数据产生所述复杂核酸的装配序列。
19.项1的方法,其进一步包括添加群体基因型数据,并从所述读取结果和所述群体基因型数据产生所述复杂核酸的装配序列。
20.项1的方法,其进一步包括:
(c)比对所述复杂核酸的第一区的多个读取结果,由此在被比对的读取结果之间创建重叠;
(d)鉴定所述重叠内的n个杂合候选物,其中n是大于2的整数;
(e)聚簇所述n个杂合候选物的2n至4n种可能性的空间或所述空间的选定子空间,由此创建多个簇;
(f)鉴定两个具有最高密度的簇,每个被鉴定的簇包含基本上无噪音的中心;并
(g)对所述复杂核酸的一个或多个别的区域重复步骤(a)-(d)。
21.项1的方法,其中所述装配序列每兆碱基包含少于0.8个假单核苷酸变体。
22.项1的方法,其中所述装配序列每兆碱基包含少于0.6个假单核苷酸变体。
23.项1的方法,其中所述装配序列每兆碱基包含少于0.4个假单核苷酸变体。
24.项1的方法,其中所述装配序列每兆碱基包含少于0.2个假单核苷酸变体。
25.项1的方法,其中所述装配序列每兆碱基包含少于0.1个假单核苷酸变体。
26.项1的方法,其中所述装配序列有所述复杂核酸至少80%的响应率。
27.项1的方法,其中所述装配序列具有至少85%的响应率。
28.项1的方法,其中所述装配序列具有至少90%的响应率。
29.项1的方法,其进一步包括:(a)提供一定量的所述复杂核酸,并(b)对所述量的所述复杂核酸测序以产生所述多个读取结果。
30.项1的方法,其中所述复杂核酸选自下组:基因组、外显子组(exome)、转录物组、甲基化组(methylome)、不同生物体基因组的混合物、一个生物体的不同细胞类型的基因组的混合物、及它们的子集。
31.项1的方法,其中所述生物体是哺乳动物。
32.项1的方法,其中所述生物体是人。
33.一种或多种计算机可读的非短暂存储介质,其存储通过项1的方法产生的装配人基因组序列。
34.计算机可读的非短暂存储介质存储指令,其在由一种或多种计算装置执行时引起所述一种或多种计算装置实施项1的方法。
35.一种测定人基因组序列的方法,该方法包括:
(a)在一个或多个计算装置上接收所述基因组的多个读取结果;并
(b)用所述一个或多个计算装置从所述读取结果产生所述基因组的装配序列,所述装配序列在70%或更大的基因组响应率时包含每千兆碱基小于600个假单核苷酸变体。
36.项34的方法,其中所述人基因组的装配序列包含70%的基因组响应率和70%或更大的外显子组响应率。
37.计算机可读的非短暂存储介质存储指令,其在由一种或多种计算装置执行时引起所述一种或多种计算装置实施项35的方法。
38.一种测定人基因组序列的方法,该方法包括:
(a)在一个或多个计算装置上接收来自多个等分试样之每个的多个读取结果,每个等分试样包含所述人基因组的片段;并
(b)用所述一种或多种计算装置从所述读取结果产生所述基因组的定相装配序列,所述装配序列在70%或更大的基因组响应率时包含每千兆碱基小于1000个的假单核苷酸变体。
39.计算机可读的非短暂存储介质存储指令,其在由一种或多种计算装置执行时引起所述一种或多种计算装置实施项38的方法。
序列表
<110>考利达基因组股份有限公司(completegenomics,inc.)
drmanac,radoje
peters,brocka.
kermani,bahramg.
<120>复杂核酸序列数据的处理和分析
<130>92171-836153(5039-us)
<140>us13/448,279
<141>2012-04-16
<150>us61/546,516
<151>2011-10-12
<150>us61/527,428
<151>2011-08-25
<150>us61/517,196
<151>2011-04-14
<160>10
<170>patentinversion3.5
<210>1
<211>20
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>1
ccgcagtagcttacgaatcg20
<210>2
<211>20
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>2
gatttaactgagcacttggc20
<210>3
<211>10
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>3
aacgagtatt10
<210>4
<211>10
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>4
tttggcgttc10
<210>5
<211>10
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>5
gtagtaccgg10
<210>6
<211>10
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>6
aactgagcgg10
<210>7
<211>12
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>7
cagtcaagtgat12
<210>8
<211>12
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>8
catgatgaggac12
<210>9
<211>12
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>9
tcttagcatgta12
<210>10
<211>12
<212>dna
<213>人工序列
<220>
<223>合成的多核苷酸
<400>10
gtaactattcag12
- 还没有人留言评论。精彩留言会获得点赞!