基于云平台的生物信息分析系统的制作方法
本发明涉及一种基于云平台的生物信息分析系统,属于生物信息学分析技术领域。
背景技术:
随着测序技术的快速发展,基因研究机构、医学科研机构和公司产生了海量的生物学测序数据。但是众所周知,实验测序得到的原始数据并不能直接提供有价值的科学研究信息或疾病治疗药物的关联信息,需要利用生物信息学分析技术对这些数据进行计算挖掘,从而给出清晰且易于导出结论的结果信息。生物信息学是在生命科学的研究中以计算机为主要研究工具对生物学数据进行存储和计算分析,面对高通量测序所产生的海量数据,越来越多的研究人员或公司基于高性能计算机集群通过安装各种生物信息分析软件来进行计算分析。生物信息分析过程也是很多软件和程序相互关联,通过不同的步骤方法处理数据后得到最终的分析结果,由于生物信息软件层出不断、更新迭代,每个分析程序参数、文件格式、运行方式各异,研究人员不断的要去研究新的软件方法并在计算机或集群上安装部署都变得十分复杂。海量数据的存储、计算和传输共享也是研究者们快速访问和分析数据中的难点。所以设计构建一种自动化扩展性好的生物信息云计算系统十分必要。
技术实现要素:
本发明的目的是解决目前生物信息分析软件品类繁多,分析效率低下,自动扩展性差,大规模数据难于存储共享和多样化软件程序构建部署分析流程复杂的技术问题。
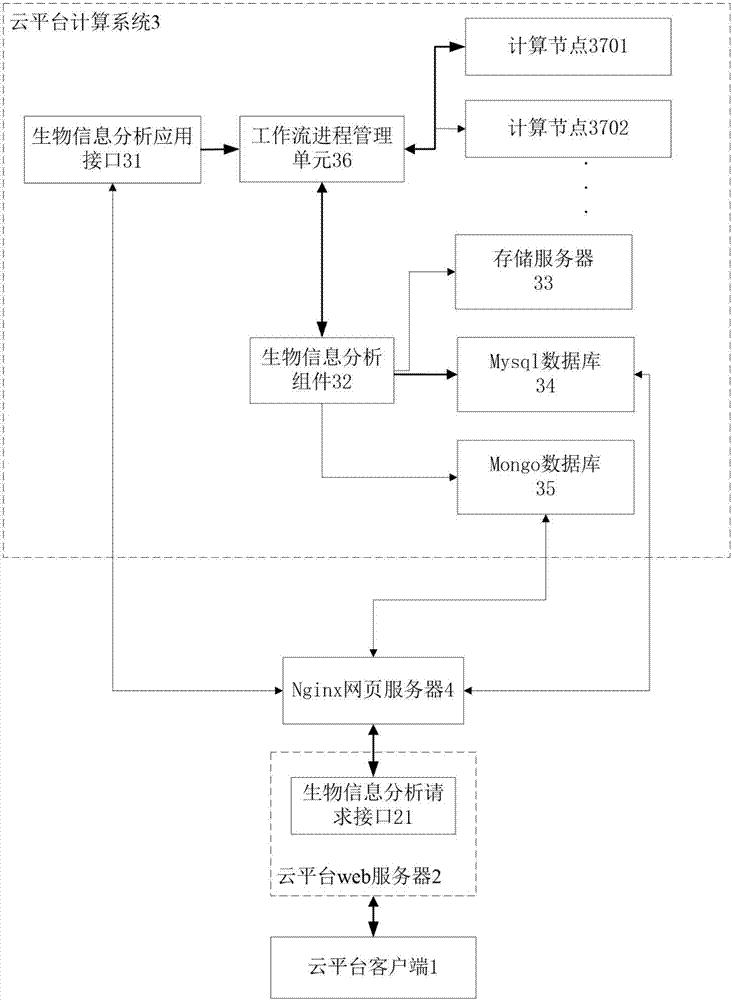
为实现以上发明目的,一方面,本发明提供一种基于云平台的生物信息分析系统,包括云平台客户端、云平台web服务器和云平台计算系统;
所述云平台客户端和所述云平台web服务器之间通过网络收发信息,所述云平台web服务器和所述云平台计算系统之间通过nginx网页服务器交换数据;
所述云平台web服务器包括生物信息分析请求接口;
所述云平台计算系统包括生物信息分析应用接口、生物信息分析组件、存储服务器、mysql数据库和mongo数据库;
所述云平台计算系统还包括若干计算节点,用于对生物信息进行分析计算;
所述生物信息分析请求接口通过所述nginx网页服务器连接至所述生物信息分析应用接口,所述生物信息分析应用接口的输出端通过工作流进程管理单元连接至所述生物信息分析组件的输入端,所述生物信息分析组件的输出端分别连接至所述存储服务器、mysql数据库和mongo数据库;
所述生物信息分析请求接口向所述生物信息分析应用接口发出生物信息分析请求并发送参数;
所述存储服务器用于存储分析结果数据,所述mysql数据库用于存储分析记录、状态和日志信息,所述mongo数据库用于存储供所述云平台客户端展示的图表数据。
进一步地,所述生物信息分析组件为即时模式分析组件或提交模式分析组件。
进一步地,所述提交模式分析组件包括工作流进程管理单元、分析模块和文件组件;
所述分析模块包括若干分析工具,各所述分析工具通过对应的工具代理与所述工作流进程管理单元进行通信;
所述工作流进程管理单元用于将不同的分析任务调度给不同的所述分析模块;
所述文件组件用于使分析结果形成通用的生物信息数据格式。
进一步地,所述工作流进程管理单元通过slurm系统进行任务调度。
另一方面,本发明提供一种基于云平台的生物信息分析方法,包括如下步骤:
在客户端输入用于分析的参数并向web服务器发出分析请求消息;
通过nginx网页服务器接收请求消息后转发至生物信息分析应用接口,解析参数;
判读所述参数为即时计算型还是投递计算型;
若为即时计算型,则直接在专用即时计算服务器上运行生物信息分析工作流;
若为投递计算型,则将参数传递至计算节点服务器上运行生物信息分析工作流;
将生成的结果数据上传到云平台的存储服务器和mongo数据库;
将生物信息分析工作流的运行状态实时存入mysql数据库;
向web服务器返回分析成功的消息,web服务器从mongo数据库获取结果提供给客户端。
进一步地,所述投递计算型的分析工作流如下:
工作流进程管理单元获取参数后,按照内部预先定义的逻辑关系,将参数传递到生物信息分析组件的各分析模块,触发各分析模块的运行,各分析模块之间通过协程监听互相有依赖关系的分析模块的运行状态,以各分析模块中的各分析工具为最小级别分析组件,将各分析工具通过slurm投递到计算节点开始计算分析,同时不断监听和接收分析的状态。
与现有技术相比,本发明的有益效果是:
本发明的分析程序模块化,可组合复用,具有扩展性好的以自动化工作流运行的系统框架,解决了大规模数据难于存储共享和多样化软件程序构建部署分析流程复杂的技术问题,分析效率大为提高,部署方便。
附图说明
图1是本发明系统的原理框图;
图2是生物信息分析组件的一个实施例原理框图;
图3是本发明方法的流程图。
图中,云平台客户端1;云平台web服务器2;生物信息分析请求接口21;云平台计算系统3;生物信息分析应用接口31;生物信息分析组件32;分析模块322;工具代理3221;分析工具3222;文件组件323;存储服务器33;mysql数据库34;mongo数据库35;工作流进程管理单元36;计算节点3701、3702…;nginx网页服务器4。
具体实施方式
下面结合附图和具体实施例对本发明作进一步说明。
实施例1
如图1所示,本发明的基于云平台的生物信息分析系统,包括云平台客户端1、云平台web服务器2和云平台计算系统3;
云平台客户端1和所述云平台web服务器2之间通过网络收发信息,云平台web服务器2和所述云平台计算系统3之间通过nginx网页服务器4交换数据;
云平台web服务器2包括生物信息分析请求接口21;
云平台计算系统3包括生物信息分析应用接口31、生物信息分析组件32、存储服务器33、mysql数据库34和mongo数据库35;
云平台计算系统3还包括若干计算节点3701、3702等,用于对生物信息进行分析计算;云平台计算系统3包含有分析各种不同生物信息的大规模计算节点,不同的分析组件被投递到计算节点对生物信息进行分析计算;
生物信息分析请求接口21通过nginx网页服务器4连接至生物信息分析应用接口31,生物信息分析应用接口31的输出端通过工作流进程管理单元(wpm)36连接至生物信息分析组件32的输入端,生物信息分析组件32的输出端分别连接至存储服务器33、mysql数据库34和mongo数据库35,mysql数据库34和mongo数据库35的输出端分别通过nginx网页服务器4连接至云平台web服务器2;
生物信息分析请求接口21向生物信息分析应用接口31发出生物信息分析请求并发送参数;
存储服务器33用于存储分析结果数据,mysql数据库34用于存储分析记录、状态和日志信息,mongo数据库35用于存储供云平台客户端1展示的图表数据。
优选地,生物信息分析组件32为即时模式分析组件或提交模式分析组件。
优选地,如图2所示,提交模式分析组件32包括分析模块322和文件组件323;
分析模块322包括若干分析工具3222,各分析工具3222通过对应的工具代理3221与工作流进程管理单元36进行通信;不同的生物信息分析任务由不同功能的分析工具被投递到单个或多个计算节点完成分析计算;
工作流进程管理单元36用于将不同的分析任务调度给不同的分析模块222;
文件组件323用于使分析结果形成通用的生物信息数据格式并进行格式检查。
优选地,工作流进程管理单元36通过slurm系统进行任务调度。
实施例2
如图3所示,本发明的基于云平台的生物信息分析方法,包括如下步骤:
s100:在客户端向web服务器发出分析请求消息并输入用于分析的参数;
s110:通过nginx网页服务器接收请求消息后转发至生物信息分析应用接口,解析参数;
s120:判读参数为即时计算型还是投递计算型;
s125:若为即时计算型,则直接在专用即时计算服务器上运行生物信息分析工作流;
s126:若为投递计算型,则将参数传递至计算节点服务器上运行生物信息分析工作流;
s130:将生成的结果数据上传到云平台的存储服务器和mongo数据库;
s140:将生物信息分析工作流的运行状态实时存入mysql数据库;
s150:向web服务器返回分析成功的消息,web服务器从mongo数据库获取结果提供给客户端。
优选地,投递计算型的分析工作流如下:
工作流进程管理单元获取参数后,按照内部预先定义的逻辑关系,将参数传递到生物信息分析组件的各分析模块,触发各分析模块的运行,各分析模块之间通过协程监听互相有依赖关系的分析模块的运行状态,以各分析模块中的各分析工具为最小级别分析组件,将各分析工具通过slurm投递到计算节点开始计算分析,同时不断监听和接收分析的状态。
本系统搭建在高性能集群中,是云平台的后端数据存储和分析计算系统,并通过nginx网页服务器与云平台的网络端进行数据通讯,包括接收分析任务id和参数,发送分析计算状态。
系统采用lustre可扩展并行文件系统存储数据文件,为所有客户机提供统一的命名空间,支持大规模高通量测序数据采集存储、分析中间文件和结果文件存储访问,以及大量生物信息数据库的存放,所有生物信息分析程序软件可以统一安装部署和特定的环境配置,数据和程序均可支持所有计算节点的统一访问和调用。
系统中核心部分为自动化计算框架,包括以下部分:
nginx网页服务器使用uwsgi协议接口进行客户端与web框架应用之间的信息交换,响应云平台网页端的请求,本系统中采用web.py网络框架来创建编写生物信息分析计算应用接口。按照生物信息分析组件的模式组合分析计算工作流,实现后端数据在高性能集群上的自动化运算。
生物信息分析组件,包括流程(workflow)、模块(moudule)、工具及其代理(tool,toolagent),以及定义生物信息数据格式的文件组件(file)。
工具是一个单一功能的生物信息分析组件,可调用一个或多个分析软件、或自定义程序包,可以重复利用,实现一个分析功能。生物信息分析计算都是由工具为最小单位在计算节点上完成。由于计算节点属于远程服务器,因而需要一个工具代理负责与工具进行通信,工具与工具代理一一对应,通过网络通信实现信息互通。
分析模块完成一个特定的较复杂的分析功能,分析模块中包含多个工具的组合,通过定义分析工具之间的运行逻辑来实现一个特定的分析功能。
流程完成一个完整的生信分析过程,通过定义运行逻辑,调用组合一系列的模块和工具实现一套的分析流程方案。工具和模块可以根据用户的分析设计被复用在不同的流程中,从而实现一个分析应用一次打包反复使用的目的。
文件定义一种通用的生物信息数据格式,包含数据属性和通用处理方法函数,工具、模块之间通过在参数中设置输入、输出的文件对象传递数据,并进行格式验证和文件处理通用方法的调用。
wpm工作流进程管理单元,系统通过wpm管理分析工作流的自动化运作。wpm监听接收到的任务请求和参数,开启一个进程运行工作流组件,并将参数传递给对应的工作流组件,流程组件中按照定义的逻辑关系,传递参数到其中的模块和工具,触发子组件的运行,并将工具投递到计算节点开始计算分析,同时不断监听和接收运行的状态。
slurm任务调度,系统中计算节点的任务运行调度采用了slurm系统来管理往计算节点的任务投递,slurm是一种可用于大型计算节点集群的高度可伸缩容错的集群管理器和作业调度系统。
数据库系统,系统中将分析运行的状态实时存入mysql数据库,将分析计算结果数据按预先定义好的数据结构存入mongo数据库,供网页端服务器快速有效的获取和查询,实现云平台的客户端用户快速直接的访问数据。
为了提高用户与数据快速、即时访问的交互效率和体验,本系统实现了两种分析计算模式,一种是对于大规模数据分析运算耗时较长的任务采用提交模式,用户不用等在客户端,在客户端点击提交即可,运行完成前可随时查看运行的进度。一种是对于小型数据或已处理后数据表进行耗时较短的分析时,系统自动判断并使用即时模式,无需任务投递,直接在专门配置的多个计算节点运行计算,并快速返回结果。
系统运行步骤如下:
1.云端web服务器通过客户端浏览器提供给用户网络访问平台系统的方式,用户在浏览器云平台网站选择生物信息分析应用,填写参数提交后,客户端将提交的数据通过网络传输协议发送给web服务器,由nginx网页服务器接收消息,nginx是一款面向性能设计的http服务器,支持高并发和高性能负载均衡,可并行接收数据请求,处理请求和返回响应。
2.nginx服务器接收到请求消息后,通过uwsgi接口将消息和参数传递给web.py框架中预先开发创建好的生物信息分析应用接口,由应用接口处理消息,对传递的参数进行解析和检查后,开始启动生物信息数据分析计算工作流;
3.wpm工作流进程管理启动和管理分析工作流运行,wpm监听接收到接口应用发起的任务请求和参数,通过应用类型参数判断分析计算类型,包括两种类型,一种是即时计算型,一种是投递计算型,同时开启一个进程运行工作流api,并将参数传递给对应的工作流;
4.如果接口应用是即时计算类型,计算耗时较短,工作流分析组件将直接在专用即时计算服务器上运行,生成的结果数据会上传到平台存储服务器位置,同时将需要展示在客户端网站的图表数据存入生物信息分析mongo数据库,供网页端获取展示,然后返回消息给web服务器,web服务器从数据库获取结果数据后展示给客户端。用户即可实时通过网络查看分析结果。
5.如果接口应用是投递计算类型,生物学数据需要经过较长时间(几十分钟到几个小时不等),流程组件获取参数后,按照内部预先定义的逻辑关系,传递参数到其中的模块和工具,触发子组件的运行,组件之间的通过协程监听互相有依赖关系的模块的运行状态,到最小级别工具组件运行时,将工具通过slurm投递到计算节点开始计算分析,同时不断监听和接收运行的状态。工具的运行状态通过工具代理与同一工作流中其他组件交换数据,运行当中会按照各组件自身的特性动态配置计算资源,并记录运行状态和进度、报错等日志信息,如若出现连接或运行失败,系统会根据预设的情况判断是否调整配置重新计算,同时会不断将运行进度状态在数据库中进行更新,web服务器端利用定时器获取状态后实时在页面刷新进度,用户可随时跟踪运行情况。计算运行结束后,结果数据会上传到存储服务器位置,同时将需要的数据存入生物信息分析mongo数据库,返回消息给web服务器端,web服务器返回新的结果页面,供用户查看。
生物信息分析组件包括流程(workflow)、模块(moudule)、工具及其代理(tool,toolagent),以及定义生物信息数据格式的文件组件(file)。流程是由模块和工具根据具体的分析产品运行逻辑组合而成,模块和工具之间具有依赖关联关系,模块或工具组件之间通过参数传递数据,包括输入输出文件参数和字段参数,输入输出文件参数需要预先定义一个该文件格式的文件组件,文件组件中会定义文件的属性和方法,并对传递的文件格式进行检查。组件之间的输入输出参数传递的是文件组件对象。在任务管理节点,一个流程中的组件包括模块和工具代理,工具代理在启动运行时通过slurm将任务投递到计算节点对应的工具进行计算,并通过工具代理实时交换数据,工具代理给工具发送运行指令和计算所需参数,工具返回运行状态和日志。一个组件如果依赖多个组件的计算结果,则需等待多个组件运行成功结束后自动激活运行。整个过程通过wpm流程管理进行监控和控制,从而实现分析系统的自动化运作。
以上述依据本发明的理想实施例为启示,通过上述的说明内容,相关工作人员完全可以在不偏离本项发明技术思想的范围内,进行多样的变更以及修改。本项发明的技术性范围并不局限于说明书上的内容,必须要根据权利要求范围来确定其技术性范围。
- 还没有人留言评论。精彩留言会获得点赞!