面向生物医学文献的跨模态图像模式识别方法与流程
本发明涉及图像识别和自然语言处理领域,尤其涉及一种面向生物医学文献的跨模态图像模式识别方法。
背景技术
随着互联网的发展,数字化生物医学文献的数量与日俱增,全球用户可以通过检索文献,掌握自己领域内的最新动态,做出新的发明或发现。数字医疗影像和医学数据重绘的示意图,作为生物医学文献的重要组成部分,对医学研究和教育,具有不可或缺的作用。
利用大量现存医学术语资源,如mesh、irma和radlex等,为医学概念分类,然后结合医学领域本体知识和图像低级视觉特征,将概念影射到不同的生物医学图像类别,进而提出一种精炼的层次化的生物医学图像分类法,能够清楚将生物医学文献中不同外观和语义的图像,划分为不同的生物医学模式。
要建立针对生物医学领域的图像模式识别算法,首先应该了解该领域的特点。第一,在生物医学文献中,有40%以上的图像属于复合图像,为了有效利用丰富的生物医学图像资源,如果首先探测复合图像,再针对复合图像和简单图像分别进行模式识别,将会对生物医学文献的图像模式识别性能产生积极作用。第二,为生物医学文献的图像往往会配备规范的说明文本,对图像进行解释说明,在说明文本中,也蕴含着丰富的生物医学模式信息,如果结合图像内容和文本信息,将有助于提高生物医学文献的图像模式识别性能。第三,生物医学领域的数据标注成本和数据特点,影响了训练数据的规模和质量,限制了深度卷积神经网络优势作用的发挥,而通用领域已有大规模(百万级)的已标注数据,生物医学领域也存在小规模的已标注数据,如果综合利用通用领域和生物医学领域的已标注数据,将会对生物医学文献的图像模式识别性能带来巨大的提升。
生物医学文献的图像模式识别算法是一系列对生物医学文献的图像进行生物医学模式分类算法的总称,它的主要特点是应用机器学习技术解决图像和文本分类问题。在文献检索系统中,利用生物医学图像模式产生元数据,提供有效归档文献中图像的手段,可以辅助科研工作者或教育者更快理解医学主题,更准确地获取感兴趣的文献。
然而,传统的生物医学文献的图像模式识别技术,根据专家的经验,人工拟合特征,训练分类器,识别图像的生物医学模式。这种特征工程的方法,对词典和规则依赖性强,而生物医学图像模式差异性有的显著、有的细微,面对多样性样本泛化性能差,很难取得较好的分类性能。
技术实现要素:
本发明的目的是提供一种能更准确识别图像的生物医学模式,提供更有效的归档文献中图像的手段,更好地满足用户检索信息需求的生物医学文献图像模式识别方法。
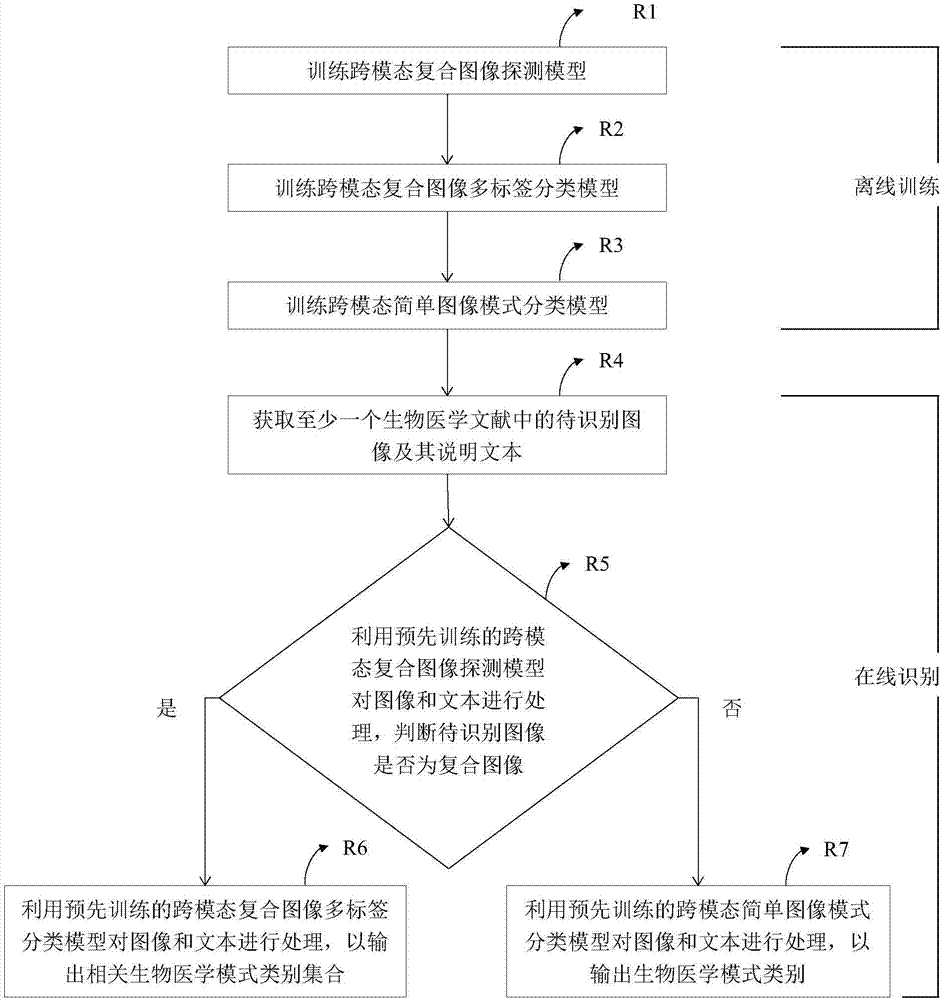
本发明解决现有技术问题所采用的技术方案:一种面向生物医学文献的图像模式识别方法,包括以下离线训练训练阶段和在线识别阶段,其中,离线训练阶段包括以下步骤:
r1、训练跨模态复合图像探测模型:
搭建基于图像内容的复合图像探测模型。视觉深度卷积神经网络包含四个具有不同数量卷积核的卷积层,用于捕获图像多种多样的局部特征,统一采用小尺寸卷积核。第二、四个卷积层后面各有一池化层,依次从卷积输出小范围内选择最大值。将池化后的多维数据一维化后,连接两个全连接层。
搭建文本复合图像探测模型。基于文本信息的卷积神经网络模型包含词嵌入层、卷积层、池化层、全连接层。首先,从大规模生物医学文献中,抽取图像说明文本,使用词向量训练工具,将所有说明文本中出现的单词处理简化为v维向量空间中的向量词典,结合已标注说明文本,生成嵌入层速查表词典。其次,在词嵌入层后面,连接一维卷积层,抽取输入序列的局部特征。然后,卷积层后面连接池化层,选择全局最大值操作,捕获最有用的局部特征,获得固定大小的全局特征向量。最后,连接两个全连接层。
搭建跨模态复合图像探测模型。使用已标注复合图像和简单图像从头训练两种模态的网络后,进行决策级融合,得到更符合样本真实类别的置信值,为探测复合图像提供更加可靠的依据。
r2、训练跨模态复合图像多标签分类模型:
搭建视觉复合图像多标签分类模型。基于图像内容的极深卷积神经网络,最后一层全连接层包含n个神经元,其激活函数为sigmoid函数,用来预测n个标签类别的相关后验概率。
搭建基于文本信息的卷积神经网络模型,与r1中复合图像探测文本模型结构相似,不同之处是,最后一层全连接层包含n个神经元,其激活函数为sigmoid函数,用来预测n种标签的相关后验概率。
训练视觉网络时,采用混合迁移学习方法。首先,在大规模自然图像数据上训练网络,实现异质迁移学习,通过迁移海量自然图像信息,缓解生物医学数据集规模过小引起的过拟合问题,使模型能够对图像的通用特征保持敏感性;然后,引入生物医学领域生物医学图像,进行同质迁移学习。
训练文本网络时,采用同质迁移学习方法。首先,初始化整个文本卷积神经网络的权重后,使用生物医学领域图像的说明文本更新网络;然后,使用多标签复合图像说明文本,根据已标注数据的真实标签,有监督地微调上一步获得的预训练网络,适应生物医学复合图像领域数据分布规律。
搭建跨模态复合图像多标签分类模型。采用全局择优法和均值法相结合的分步融合方式,为当前样本标定标签。具体来说,首先依照阈值函数,根据图像模型输出的后验概率,将高于阈值的标签加入到相关标签集;如果某样本的相关标签集为空,计算图像模型和文本模型输出的所有标签的概率平均值,取最大k个平均概率的标签为相关标签。
r3、训练跨模态简单图像模式分类模型:
搭建基于图像内容的简单图像模式分类模型。视觉模型集成具有显著多样性的若干子模型,包括d种深度差异较大的卷积神经网络。视觉集成模型的子模型采用不同的训练方式。首先,极深的深度卷积神经网络在归纳式迁移学习基础上完成训练。具体来说,使用通用领域的大规模自然图像训练网络后,使得模型对图像类型的输入能够收敛,以此为基础再从医学图像中学习领域专有特征,实现跨领域异质迁移学习,以提高模型性能。然后,对于稍浅的深度卷积神经网络,采用从头训练的方法,捕获更多的生物医学领域特征。
搭建基于文本信息的简单图像模式分类模型。基于文本信息的卷积神经网络模型,与r2中复合图像多标签分类模型结构相似,最后一层全连接层包含n个神经元,不同之处是其激活函数为softmax函数,用来预测n种标签的后验概率。
搭建跨模态简单图像模式分类模型。利用已标注简单图像训练网络后,进行决策级融合,通过投票系统合并子模型对每个模式的置信值,提高融合系统的置信度,进而确定输入样本的模式类别。
在线识别阶段包括以下步骤:
r4、获取至少一个生物医学文献中的待识别图像及其说明文本:
从生物医学文献中提取待识别的图像及其说明文本。
r5、利用预先训练的跨模态复合图像探测模型对图像和文本进行处理,判断待识别图像是否为复合图像:
将r4中获取的图像和说明文本,分别输入到r1的视觉和文本复合图像探测模型,得到两种模态的复合图像预测后验概率,计算概率向量平均值后,根据向量最高值元素位置,输出待识别图像为复合图像或者简单图像。
r6、利用预先训练的跨模态复合图像多标签分类模型对图像和文本进行处理,以输出复合图像子图的生物医学模式类别:
若r5的输出为复合图像,则将r4中获取的图像和文本,分别输入到r2的视觉和文本复合图像多标签分类模型,根据分段式融合策略,按照预测的后验概率向量和阈值函数,输出相关生物医学模式类别集合。
r7、利用预先训练的跨模态简单图像模式分类模型对图像和文本进行处理,以输出简单图像的生物医学模式类别:
若r5的输出为简单图像,则将r4中获取的图像和文本,分别输入到r3的视觉和文本模式分类模型,得到两种模态的生物医学模式预测后验概率,计算概率向量的平均值后,根据向量最高值元素位置,输出生物医学模式类别。
本发明的有益效果在于:本发明从跨模态融合的角度出发,通过运用特征学习和混合迁移学习等技术手段在图像和文本两个模态的信息中学习生物医学模式特征,能够更准确地完成生物医学模式识别任务,从而更有效地生成图像元数据,本发明利用通用领域和生物医学领域资源,能够提高训练样本的多样性,改善识别的性能。当使用imageclef医学任务数据集作为数据集合,采用人工拟合特征探测复合图像时,可以获得90.74%的识别准确率,而采用本发明所涉及的特征学习方法识别时,可以获得96.33%的识别准确率;采用人工拟合特征进行模式分类时,可以获得87.56%的准确率,而采用本发明所涉及的特征学习方法,可以获得的准确率87.88%;采用基于异质迁移学习的复合图像多标签分类模型,可以获得0.320宏平均f1值,而采用本所涉及的跨模态混合迁移学习方法,可以获得0.488的宏平均f1值,在三个子任务上,生物医学模式识别的性能都得到了显著提升。
附图说明
图1为本发明识别方法的流程示意图;
图2为本发明识别方法与基准方法的视觉(visual)、文本(textual)和跨模态(cross-media)模型识别准确率的对比图;
图3为本发明识别方法与基准方法的复合图像多标签分类结果的对比图;
图4为本发明识别方法的简单图像模式分类混淆矩阵热图。
具体实施方式
为了使本发明的目的、技术方案和优点更加清楚,下面结合附图及具体实施方式对本发明进行详细描述。
图1是本发明一种面向生物医学文献的图像模式识别方法的流程示意图,一种面向生物医学文献的图像模式识别方法,包括以下离线训练阶段和在线识别阶段,其中,离线训练阶段包括以下步骤:
r1、训练跨模态复合图像探测模型:
a1:首先,需要搭建基于图像内容的复合图像探测模型。视觉深度卷积神经网络的四个具有不同数量卷积核的卷积层,在本实施例中,前两个包含32个卷积核,后两个包含64个卷积核,用于捕获图像多种多样的局部特征,统一采用小尺寸卷积核,在本实施例中,卷积核尺寸为3×3。第二、四个卷积层后面各有一池化层,依次从卷积输出的小范围内选择最大值,在本实施例中,池化操作尺寸为2×2。将池化后的多维数据一维化后,连接两个全连接层,在本实施例中,分别包含512和2个神经元。在本实施例中,视觉网络的卷积层和第一个全连接层神经元采用relu激活函数,第一层全连接层使用dropout技术,随机舍弃部分连接,减少权重参数数量,缓解过拟合问题,最后一层全连接层采用softmax激活函数输出复合图像的预测概率。
a2:其次,搭建基于本文信息的复合图像探测模型。文本卷积神经网络包含词嵌入层、卷积层、池化层、全连接层。首先,从n篇生物医学文献中,抽取图像说明文本,使用词向量训练工具,将所有说明文本中出现的单词处理简化为v维向量空间中的向量词典,根据训练集和测试集说明文本,生成嵌入层速查表词典,在本实施例中,n=30万,v=300,词向量训练工具选用word2vec。其次,在词嵌入层后面,连接一维卷积层,抽取输入序列的局部特征,在本实施例中,卷积核数量为250个。然后,卷积层后面连接池化层,选择全局最大值操作,捕获最有用的局部特征,获得固定大小的全局特征向量。最后,连接两个全连接层,在本实施例中,分别包含250个和2个神经元。在本实施例中,文本网络的卷积层和第一个全连接层使用relu激活函数抽取非线性特征,最后一个全连接层使用softmax激活函数输出复合图像预测概率。
在本实施例中,使用二元交叉熵损失函数,评估r1中a1和a2所述视觉和文本两种模态的网络输出概率分布与真实分布的差异情况。对于样本训练集x,目标是以迭代方式更新权重参数w,最小化二元交叉熵损失函数:
其中,x(i)是训练集x中的第i个样本,n为样本训练集x中包含的样本数量;权重为w的情况下,函数f(x(i),w)得到样本x(i)相关类别的预测概率向量,为方便表示,记为y(i);y'(i)是第i个样本真实的类别向量,采用one-hot表示;l(y(i),y'(i)是计算的输出为y(i)而不是真实类别y'(i)的惩罚函数:
l(y(i),y'(i))=-y'(i)log(y(i))+(1-y'(i))log(1-y(i)))
最后,搭建跨模态复合图像探测模型。使用已标注复合图像和简单图像以及相关说明文本从头训练两种模态的网络后,进行决策级融合,根据两个子模型预测概率的平均值,得到更符合样本真实类别的置信值,为探测复合图像提供更加可靠的依据。
在本实施例中,均值策略的融合公式如下所示:
y=argmax(σ(p(k)))
其中,y是预测的类别标签,复合图像探测可以转换为两类,y的结果为0时,表示当前样本是单模式图像,反之,结果为1时,表示输入样本为复合图像;σ(·)函数计算k=2个子模型预测概率向量p(k)的平均值;argmax(·)函数返回概率均值向量中具有最大值的元素下标。
r2、训练跨模态复合图像多标签分类模型:
a1:首先,需要搭建基于图像内容的复合图像多标签分类模型。视觉极深卷积神经网络,在本实施例中,搭建50层深度残差网络resnet-50。最后一层全连接层包含n个神经元,其激活函数为sigmoid函数,用来预测n个标签类别的相关后验概率。
训练视觉网络时,提出混合迁移学习方法。在本实施例中,首先,在百万级imagenet自然图像数据上训练网络,实现异质迁移学习,通过迁移海量自然图像信息,缓解生物医学数据集规模过小引起的过拟合问题,使模型能够对图像的通用特征保持敏感性;然后,引入生物医学领域单标签简单图像,进行同质迁移学习,获得图像内容与标签的强关联信息,丰富了训练数据每个类别的样式,削弱了复合图像数据集中标签分布不均衡造成的负面影响。a2:其次,搭建基于文本信息的复合图像多标签分类模型。文本卷积神经网络模型,与r1中基于文本信息的复合图像探测模型结构相似,不同之处是,前者最后一层全连接层包含n个神经元,其激活函数为sigmoid函数,用来预测n个标签的相关后验概率。
训练文本网络时,采用同质迁移学习方法。在本实施例中,首先,使用均匀分布初始化整个文本卷积神经网络的权重后,使用生物医学领域单标签的简单图像的说明文本更新网络权重;然后,使用多标签复合图像说明文本,根据已标注数据的真实标签,有监督地微调上一步获得的预训练网络,适应生物医学复合图像领域数据分布规律。
在本实施例中,n=30,使用二元交叉熵损失函数评估r2中a1和a2所述视觉和文本两种模态网络输出概率分布与真实分布的差异情况。
a3:最后,搭建跨模态复合图像多标签分类模型。采用全局择优法和均值法相结合的分步融合方式,为当前样本标定标签。在本实施例中,给定分类器输出测试集实例x(i)(1<i<m)的后验概率向量
r3、训练跨模态简单图像模式分类模型:
a1:首先,需要搭建基于图像内容的简单图像模式分类模型,所述视觉模型集成具有显著多样性的d个子模型,集成d种深度差异较大的卷积神经网络,在本实施例中,d=3,即集成6层卷积神经网络cnn-6、16层卷积神经网络vgg-16和50层深度残差网络resnet-50。三种卷积神经网络的最后一层全连接层包含n个神经元,均选择softmax函数作为激活函数,由softmax输出n种生物医学模式的后验概率。
在本实施例中,视觉集成模型的三个子模型采用不同的训练方式。其中,卷积神经网络vgg-16和resnet-50在归纳式迁移学习基础上完成训练。具体来说,使用imagenet自然图像训练两个网络后,使得模型对图像类型的输入能够收敛,以此为基础,使用生物医学简单图像微调顶层网络特征,保留通用领域特征后,学习领域专有特征,实现跨领域异质迁移学习,以提高模型性能。而且,为融入更多的生物医学图像领域特征,视觉模型集成了从头训练的卷积神经网络cnn-6。
a2:然后,搭建基于文本信息的简单图像模式分类模型。基于文本信息的卷积神经网络模型,与r2中复合图像多标签分类模型结构相似,最后一层全连接层包含n个神经元,不同之处是其激活函数为softmax函数,用来预测n种标签的后验概率。
在本实施例中,n=30,使用分类交叉熵损失函数,评估r3中a1和a2所述视觉和文本两种模态的网络输出概率分布与真实分布的差异情况。假定训练集x包含n个样本,训练网络就是更新网络权重w的迭代过程,使得分类交叉熵损失函数l最小化。
其中,x(i)是训练集x中的第i个样本;网络权重为w的情况下,f(x(i),w)得出样本x(i)的类别预测概率向量,为方便表示,记为y(i);y'(i)是第i个样本真实类别向量,采用one-hot表示,真实类别元素值为1,其他类别为0;l(y(i),y'(i))是按元素计算的预测类别为
其中,
最后,搭建跨模态简单图像模式分类模型。利用已标注简单图像训练网络后,进行决策级融合,利用不同子模型之间提供的信息冗余性、相关性,通过投票系统合并子模型对每个模式的置信值,提高融合系统的置信度,进而确定输入样本的模式类别。在本实施例中,首先,投票系统赋予视觉集成模型的子模型不同的权重,采用加权平均的策略得出视觉集成模型的预测概率;然后,再根据两种模态子模型预测概率的平均值,预测当前样本类别,得到更符合样本真实类别的置信值,为识别简单图像的生物医学模式提供更加可靠的依据。
在本实施例中,投票系统的视觉模型融合公式如下所示:
y=argmax(σ(wk·p(k)))
其中,y是当前样本的预测模式类别标签,p(k)是第k个子模型模式的预测概率向量,每个元素对应三十个模式类别,σ(·)函数返回k个子模型输入结果的平均值,第k个子模型预测概率的权重为wk,argmax(·)函数返回使得函数σ取得最大值的元素位置下标,对应模式类别标签。跨模态融合时,wk为二维向量,向量元素值均为0.5。
在线识别阶段包括以下步骤:
r4、获取至少一个生物医学文献中的待识别图像及其说明文本:
从生物医学文献中提取待识别的图像及其说明文本。在本实施例中,从pubmed检索系统中,解析出文献的待识别图像及其说明文本。
r5、利用预先训练的跨模态复合图像探测模型对图像和文本进行处理,判断待识别图像是否为复合图像:
将r4中获取的图像和说明文本,分别输入到r1的视觉和文本复合图像探测模型,得到两种模态的复合图像预测后验概率,计算概率向量平均值后,根据向量最高值元素位置,输出待识别图像为复合图像或者简单图像。
r6、利用预先训练的跨模态复合图像多标签分类模型对图像和文本进行处理,以输出复合图像子图的生物医学模式类别:
若r5的输出为复合图像,则将r4中获取的图像和文本,分别输入到r2的视觉和文本复合图像多标签分类模型,根据分段式融合策略,按照预测的后验概率向量和阈值函数,输出相关生物医学模式类别集合。
r7、利用预先训练的跨模态简单图像模式分类模型对图像和文本进行处理,以输出简单图像的生物医学模式类别:
若r5的输出为简单图像,则将r4中获取的图像和文本,分别输入到r3的视觉和文本模式分类模型,得到两种模态的生物医学模式预测后验概率,计算概率向量的平均值后,根据向量最高值元素位置,输出生物医学模式类别。
根据上述针对于本发明所涉及方法具体实施方式的描述,结合具体实施例进行说明。本实施例中,假定已经通过已标注数据完成了三种识别模型的训练。
首先,针对3456个已标注生物医学图像及相关说明文本,分别采用本发明所述方法和基准方法进行复合图像探测。基准方法人工拟合图像分界线和连通域特征搭建视觉模型,人工拟合文本分隔符特征搭建文本模型,采用均值策略进行结果融合。图2是本发明方法与基准方法的视觉(visual)、文本(textual)和跨模态(cross-media)模型识别准确率,可以看出本发明所述方法,单模态和跨模态模型能够自动学习图像的复合特征,识别准确率有显著性提高,。
然后,针对1083个已标注复合图像及相关说明文本,分别采用本发明所述方法和基准方法,进行复合图像多标签学习,识别复合图像的子图模式类别。基线方法包括标准的分离复合图像后识别子图模式方法(standard)、利用模式分类模型自动标注简单图像进行扩展多标签数据的方法(extendedmulti-label)以及抽取alexnet的decaf特征训练svm分类器的方法(alexnet_svm)。图3是本发明所述方法与基准方法的复合图像多标签分类的结果,可以看出本发明所述方法,宏平均f1值(f1-macro)超过三种基准算法,能够保证较高识别精度的情况下,获取较高召回率,有效地缓解过拟合占多数类的问题,从而提高多标签分类性能。
最后,针对6776个已标注简单图像及相关说明文本,采用本发明所述方法,进行简单图像模式分类,识别简单图像的生物医学模式类别。图4是本发明方法的简单图像模式分类混淆矩阵热图,由于测试样本类比分布存在偏差,因此这里将样本数量标准化为[0,1]区间,颜色从浅到深渐变,便于比较模型在每个类别上的分类性能。纵坐标是样本真实的生物医学图像模式类别,横坐标是本发明所述方法预测的生物医学模式类别。在混淆矩阵图中,对角线上的点表示预测的模式类别与真实的模式类别相同的样本数量,颜色越深,表示预测正确的样本数量越多;非对角线上的点表示分类器误分类的样本数量,颜色越浅表示误分类的样本数量越少,以此反映误分类样本的类别分布情况。从图4可以看出本发明方法能够比较准确地区分生物医学图像的模式类别。
本发明方法能够利用通用领域和生物医学领域的资源,提高训练数据的多样性,搭建卷积神经网络从图像内容和文本信息中提取生物医学模式信息,保证了图像模式识别结果的准确性,进而满足生物医学文献的图像归档和检索的需要。
以上内容是结合具体的优选技术方案对本发明所作的进一步详细说明,不能认定本发明的具体实施只局限于这些说明。对于本发明所属技术领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干简单推演或替换,都应当视为属于本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!