基于神经网络的肺部病理切片癌细胞分割算法
1.本发明属于深度学习技术领域,具体涉及一种基于神经网络的肺部病理切片癌细胞分割算法。
背景技术:
2.随着计算机技术的高速发展,计算机医学图像处理在特征提取和分析的研究中发挥着重要作用,计算机在医疗辅助诊断中也发挥着越来越大的作用。如何将模式识别,图像分割与临床病理组织切片的诊断更好的结合在一起已经成为当前研究的主要方向之一。肺肿瘤的病理组织切片诊断作为临床诊断的难点之一,自然也被列为研究的热点数据类型之一。
3.不同于ct和mri影像,肺肿瘤病理组织切片图像的特征提取难度更大,近几年,相比其他人体器官和组织的医学影像分析比如肝癌分割,乳腺癌分割等,针对肺肿瘤病理切片图像分割的研究较少。在算法方面,仍然存在很多不足,比如特征提取缺少针对性目的性,卷积神经网络的特征向量维度过高,分割或者分类的精度还不足以达到临床应用的要求,研究过程中缺少病理专家的诊断学指导等等。
技术实现要素:
4.为了解决现有技术中的问题,本发明提供一种基于神经网络的肺部病理切片癌细胞分割算法,解决现有技术中特征提取缺少针对性目的性,卷积神经网络的特征向量维度过高,分割或者分类的精度还不足以达到临床应用的要求问题。
5.本发明的技术方案为:
6.一种基于神经网络的肺部病理切片癌细胞分割算法,包括以下步骤:
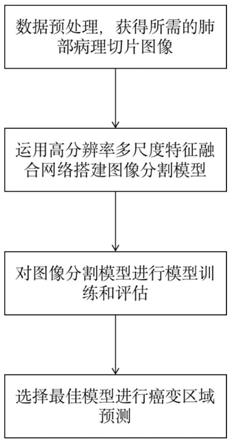
7.s1、数据预处理,获得所需的肺部病理切片图像;
8.s2、运用高分辨率多尺度特征融合网络构建分割模型;具体包括:
9.s2-1、将病理切片图像的patch输入高分辨率多尺度特征融合网络模型;
10.s2-2、依据构建的分割模型,生成预测的普通细胞区域和癌变细胞区域;
11.s3、对所述分割模型进行模型训练和评估。
12.所述s1步骤包括:
13.s1-1、采用病理学切片图像数据作为数据源;
14.s1-2、将xml格式的标注文件转换为与图像大小相同的二值png图像;
15.s1-3、使用改进的基于vahadane方法的颜色归一化方法来处理数据;
16.s1-4、使用数据增强技术,如镜像、翻转、旋转和偏移来扩充数据量。
17.所述s2高分辨率多尺度特征融合网络模型包括卷积层、归一化层、relu激活函数和反卷积层。
18.所述s3步骤包括:
19.s3-1、使用cross-entropy loss和dice score loss之和作为损失函数;其中
cross-entropy loss的计算公式为:
[0020][0021]
dice score loss的计算公式为:
[0022][0023]
s3-2、使用sgd对模型进行优化;将mini-batch大小设置为12,学习率设置为0.01,采用聚学习率策略调整学习率,学习率下降公式为:
[0024][0025]
其中lr
initial
=0.01,power=0.9,iter
max
=500;然后使用he初始化策略来初始化权重;最后,选择miou(平均交并比)最高的模型作为最佳模型。
[0026]
本发明有益效果:本发明提出了一种基于多尺度融合网络的小细胞肺癌病理切片图像自动分割模型。这是一个重要的问题,因为u型网络的发展与医学图像分割的发展密切相关,多尺度特征的提取有利于高分辨率医学图像分割算法的发展。本发明提出了一种基于多尺度特征融合的u形网络。我们使用miou作为分割效果的主要评价标准。结果表明,本发明提出的网络性能更优。我们可以得出结论,在基于肺病理切片图像的小细胞肺癌分割中,相比于传统方法,本技术的方法更加清晰明了,得到的预测结果更加准确。
附图说明
[0027]
图1为本发明方法的流程图;
[0028]
图2为本发明的高分辨率多尺度特征融合网络的结构示意图;
[0029]
图3为本发明的预测结果与标注结果的示意图。
具体实施方式
[0030]
以下结合附图1~3和具体实施例对本发明作进一步详细说明,但不作为对本发明的限定。
[0031]
一种基于多尺度特征融合的肺部病理切片癌细胞分割方法,如图1所示,包括以下步骤:
[0032]
s1、数据预处理,获得所需的肺部病理切片图像;
[0033]
s1-1、采用病理学切片图像数据作为数据源;
[0034]
s1-2、将xml格式的标注文件转换为与图像大小相同的二值png图像;
[0035]
s1-3、使用改进的基于vahadane方法的颜色归一化方法来处理数据。
[0036]
s1-4、使用数据增强技术,如镜像、翻转、旋转和偏移来扩充数据量。
[0037]
s2、运用高分辨率多尺度特征融合网络构建分割模型;
[0038]
s2-1、将病理切片图像的patch输入高分辨率多尺度特征融合网络模型,高分辨率多尺度特征融合网络模型包括卷积层、归一化层、relu激活函数和反卷积层。;
[0039]
s2-2、依据构建的分割模型,生成预测的普通细胞区域和癌变细胞区域。
[0040]
s3、对所述分割模型进行模型训练和评估。
[0041]
s3-1、使用cross-entropy loss和dice score loss之和作为损失函数;其中cross-entropy loss的计算公式为:
[0042][0043]
dice score loss的计算公式为:
[0044][0045]
s3-2、使用sgd对模型进行优化。将mini-batch大小设置为12,学习率设置为0.01,采用聚学习率策略调整学习率,学习率下降公式为:
[0046][0047]
其中lr
initial
=0.01,power=0.9,iter
max
=500。然后使用he初始化策略来初始化权重。最后,选择miou(平均交并比)最高的模型作为最佳模型。
[0048]
在具体的预测方法中,使用数字幻灯片扫描仪(hamamatsu nano zoomer digital pathology)对31例患者的肺组织切片进行了h&e染色,客观放大倍数为20倍。形态学诊断依据世界卫生组织(who)肺、胸膜、胸腺、心脏肿瘤分类标准(第四版)。一位胸外科医生将肿瘤细胞所在区域圈起来。
[0049]
并且,在模型训练之前,首先采用python软件中的openslide工具包对数据进行预处理。对于收集到的所有病理切片图像数据,数据的预处理操作包括:
[0050]
(一)格式转换,将xml格式的标注文件转换为与图像大小相同的二值png图像;
[0051]
(二)归一化,使用改进的基于vahadane方法的颜色归一化方法来处理数据。
[0052]
(三)数据增强,使用数据增强技术,如镜像、翻转、旋转和偏移来扩充数据量。
[0053]
在数据处理好后,采用高分辨率多尺度特征融合网络来搭建图像分割模型。高分辨率多尺度特征融合网络构建了一种新型网络架构,在整个处理过程中,能够保持高分辨率表示。通过并行多个分辨率的分支,加上不断进行不同分支之间的信息交互,同时达到提取高级特征和保留精准位置信息的目的。
[0054]
模型的输入是维度为256x256x3的肺部病理切片图像,输出是维度为256x256x1的预测mask,其中每个像素点的取值为0或1,代表预测结果为正常细胞或者癌变细胞。
[0055]
具体来说,如图2所示,左侧为编码路径,右侧为解码路径,中间实线部分表示的是跳跃链接。该网络的编码路径分为5个阶段,第一阶段将输入的图像经过convblock运算得到通道数为64的高分辨率feature map。每个convblock由一个卷积核大小为3,步长为1的卷积层和一个relu激活函数构成,得到的feature map的高和宽的计算公式分别为:
[0056][0057][0058]
第二阶段将第一阶段得到的高分辨率feature map经过三个convblock运算得到,并将得到的feature map与输入的feature map经过一个核大小为1,步长为1的卷积后的结
果相加并通过relu函数激活得到一个通道数为256,大小与输入相同的feature map。将该feature map经过一个transition layer得到其下采样结果。
[0059]
transition layer由n-1个步长为1的convblock和一个步长为2的convblock构成,其中n为可配置的参数。
[0060]
第三、四阶段均由fuse layer和transition layer构成,每个layer都有n个convblock。第五阶段将分辨率最低但最高阶的feature map再经过一次卷积核大小为3,步长为2的下采样运算得到通道数为1024的feature map。经过五个阶段的特征提取,我得到了5个不同分辨率的feature map,这些feature map的通道数分别为64、128、256、512、1024。
[0061]
为了控制每层卷积后特征图谱的尺寸大小,卷积操作过程中,先在图像外围填充0,这么做的优点有两个:
[0062]
1.可以充分提取到图像的边缘信息。
[0063]
2.卷积操作后的输出图像尺寸大小与输入图像相同。
[0064]
编码路径中输入feature map尺寸变小是通过步长为2的卷积运算完成的。这与u-net的构大池化操作不同,目的是为了能够在保证模型输入输出的图片尺寸仸致的前提下尽可能的保留更多的原始特征,达到端到端分割的目的,从而实现精准的像素级别的分割。
[0065]
在具体的预测方法中,还可使用paddlepaddle来实现算法。在训练过程中,使用cross-entropy loss与dice score loss之和作为损失函数。其中cross-entropy loss的计算公式为
[0066][0067]
dice score loss的计算公式为
[0068][0069]
使用sgd对模型进行优化。我们将mini-batch大小设置为12,学习率设置为0.01,采用聚学习率策略调整学习率,学习率下降公式为
[0070][0071]
其中lr
initial
=0.01,power=0.9,iter
max
=500。然后使用he初始化策略来初始化权重。最后,选择miou(平均交并比)最高的模型作为最佳模型。在验证集中,最优的模型实现了平均交并比miou=0.8875,kappa系数=0.8813。最佳模型的分割结果如图3所示。其中(a)、(b)、(c)、(d)为验证集中随机抽取的四张图像,(e)、(f)、(g)、(h)分别为(a)、(b)、(c)、(d)的标注掩码图像,(i)、(j)、(k)、(l)为最佳模型的预测结果。黑色区域为正常区域,白色区域为病变区域。根据分割结果,可以直观地观察到生成的图像与掩码图像非常相似。此外,在未标记的部分,发现了一些白色的小区域,这意味着未标记的病理细胞也被分离出来了。因此,可以认为神经网络通过标记信息学习到的特征具有良好的泛化性,能够对未标记区域的散发性病理细胞进行分割。结果表明,该网络在多尺度特征信息学习方面优于预期,证明了神经网络在医学影像领域的强大应用。
[0072]
因此,在本技术中,根据实验需要收集一定数量的肺部病理切片图像数据,完成数
据的采集;之后使用预处理技术对数据进行初步处理得到符合标准的数据,并将数据集按照8:1的比例分为训练集和验证集;接下来根据算法设计逐步搭建图像分割模型,在模型搭建好后,使用训练集对模型进行训练,并使用验证集对模型效果进行评估。相比于传统方法,本技术的方法更加清晰明了,得到的预测癌变区域更加准确。
[0073]
根据上述说明书的揭示和教导,本发明所属领域的技术人员还能够对上述实施方式进行变更和修改。因此,本发明并不局限于上述的具体实施方式,凡是本领域技术人员在本发明的基础上所作出的任何显而易见的改进、替换或变型均属于本发明的保护范围。此外,尽管本说明书中使用了一些特定的术语,但这些术语只是为了方便说明,并不对本发明构成任何限制。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1