一种基于深度学习的图像中肾间质炎症细胞确定方法、计算机设备及计算机可读存储介质与流程
1.本发明具体涉及一种基于深度学习的图像中肾间质炎症细胞确定方法、计算机设备及计算机可读存储介质。
背景技术:
2.肾间质是位于肾单位及集合管之间的结缔组织,由间质细胞、网状纤维、少量弹力纤维、成纤维细胞和巨噬细胞等组成。因炎症、遗传或其他疾病导致肾间质受损,可累及肾小管、肾盂、肾盏,发生排尿障碍和机体电解质以及酸碱平衡紊乱。
3.现有技术中,主要通过实际经验来进行确定,该方法诊断效率慢,误检率高,花费成本高。
技术实现要素:
4.针对上述情况,为克服现有技术的缺陷,本发明提供一种基于深度学习的图像中肾间质炎症细胞确定方法、计算机设备及计算机可读存储介质。
5.为了实现上述目的,本发明提供以下技术方案:
6.一种基于深度学习的图像中肾间质炎症细胞确定方法,包括以下步骤:
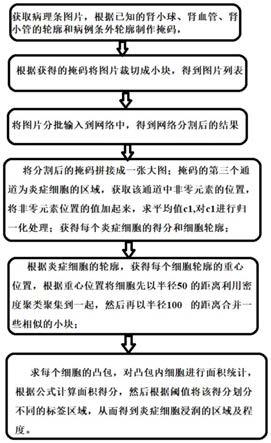
7.(1)获取一张病理条图片,然后根据已知的肾小球、肾血管、肾小管的轮廓和病理条外轮廓制作掩码,将病理条外轮廓填充为1,将肾小球、肾血管、肾小管轮廓填充为0;
8.(2)根据获得的掩码将图片裁切成小块,得到图片列表;
9.(3)将图片列表按照要求的batch_size大小,将图片分批输入到训练好的分割模型中,得到分割后的结果;
10.(4)根据裁切窗口坐标位置,将分割后的掩码拼接成一张大图;掩码的第三个通道为炎症细胞的区域,然后获取该通道中非零元素的位置,将非零元素位置的值加起来,求平均值得到c1,然后对c1进行归一化处理,得到score=c1/255;这样就获得每个炎症细胞的得分和细胞轮廓。
11.进一步地,还包括:
12.步骤(5)根据得到的炎症细胞的轮廓,获得每个炎症细胞轮廓的重心位置,然后根据重心位置将细胞先以半径50的距离利用密度聚类聚集到一起,然后再以半径100的距离合并一些相似的小块,得到一个相对大的炎症细胞区域。
13.进一步地,还包括:
14.步骤(6)求每个炎症细胞的凸包,对凸包内炎症细胞进行面积统计,根据公式计算面积得分score=1.0*(非浸润区域细胞面积+浸润区域细胞面积)/(肾小球、肾血管、肾小管区域面积+非细胞区域面积+非浸润区域细胞面积+浸润区域细胞面积),然后根据[0.05,0.1,0.2,0.3,1.0]阈值将该得分划分不同的标签区域,从而得到炎症细胞浸润的区域及程度。
[0015]
进一步地,步骤(1)中,病理条外轮廓的检测是利用函数findcontours提取得到:
[0016]
(1.1)首先将bgr格式的图片转化为灰度图片,然后对病理条进行缩放,获得缩放图片中阈值小于200大于10的像素区域,转为uint8格式,得到图片掩码;
[0017]
(1.2)然后利用闭操作进行处理,将掩码中的空洞进行填充,然后使用轮廓检测函数,获得图片中的轮廓,取轮廓面积大于设定阈值的轮廓,最终获得病理条图片的外轮廓。
[0018]
进一步地,步骤(2)中,裁切过程中边界像素记为boundary_pixels,图片缩放尺寸为img_scale,缩放后的边界像素点bp=boundary_pixels/img_scale,窗口大小为win_size=640/img_scale也和图片保持一样的缩放比;大图图片的宽高分别填充2*bp大小,然后以win_size大小的窗口进行裁切,重叠区域overlap=2*bp进行裁切。
[0019]
进一步地,步骤(4)中,利用findcontours函数检测出所有炎症细胞的细胞轮廓。
[0020]
进一步地,步骤(3)中,分割模型采用的是unet网络模型,unet网络模型的下采样过程用efficientnet_b3代替。
[0021]
进一步地,步骤(3)中,分割模型训练过程中设置的参数:
[0022]
学习轮次为200次,初始学习率为0.01,batch_size大小为24;weight_decay=0.0001,使用的优化器为adam。
[0023]
一种计算机设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,处理器执行计算机程序时能够实现以上所述的基于深度学习的图像中肾间质炎症细胞确定方法中的步骤。
[0024]
一种计算机可读存储介质,其上存储有计算机程序,计算机程序被处理器执行时能够实现以上所述的基于深度学习的图像中肾间质炎症细胞确定方法中的步骤。
[0025]
本发明的有益效果是:
[0026]
本发明方法能够用于确定肾间质炎症细胞、炎症细胞浸润的区域及程度,为后续的诊断与治疗提供了依据。本发明所提出的方法,还可以用于解决目前传统图像处理时效性差的问题。本技术基于深度学习的方法,使得模型具有更好的鲁棒性和泛化能力,相比现有的检测法能够获得较好的准确性,并能提高就检测效率。
附图说明
[0027]
图1是肾间质炎症细胞浸润确定方法的流程图。
[0028]
图2是计算机设备的示意图。
[0029]
图3是实施例1中,步骤(1)处理前的图片与处理后得到的图片示意图,其中(1)是处理前的病理条图片,(2)是处理后得到的图片。
[0030]
图4是实施例1中,裁切后得到的小图示例,其中(1)是裁切得到的小图,(2)是模型输出的小图掩码图片。
[0031]
图5是实施例1中,将所有分割出来的掩码拼接到一起得到的输出掩码图。
[0032]
图6是实施例1中,浸润区域凸包的示意图,其中(1)是凸包,(2)是从原图中裁切下来的炎症细胞聚集的区域。
[0033]
图7是实施例1中,得到的最终结果图。
具体实施方式
[0034]
以下结合附图对本发明的技术方案做进一步详细说明,应当指出的是,具体实施方式只是对本发明的详细说明,不应视为对本发明的限定。
[0035]
一种基于深度学习的图像中肾间质炎症细胞确定方法,包括以下步骤:
[0036]
(1)获取一张病理条图片,然后根据已知的肾小球、肾血管、肾小管的轮廓和病理条外轮廓制作掩码,将病理条外轮廓填充为1,将肾小球、肾血管、肾小管轮廓填充为0。由步骤(1)可以得到肾间质区域。
[0037]
病理图片采集设备(比如病理切片扫描仪)中获得的正常的图片都可以适用于本发明的方法。
[0038]
所述病理条指的是:有效的病理条,即包含皮质区域的病理条,皮质区域中包含肾小球、肾血管、肾小管。
[0039]
其中病理条外轮廓的检测是利用opencv提取得到:
[0040]
(1.1)首先将bgr格式的图片转化为灰度图片,然后对病理条进行缩放(一般缩放1/4或者1/8),获得缩放图片中阈值小于200大于10的像素区域,转为uint8格式,得到图片掩码。
[0041]
(1.2)然后利用闭操作进行处理,将掩码中的空洞进行填充,然后使用轮廓检测函数,获得图片中的轮廓,取轮廓面积大于设定阈值的轮廓,最终获得病理条图片的外轮廓。因为图片灰度化后,转为二值图可能会出现留白的情况,用闭操作函数之后,会将这些留白填充,这样就能得到一个完整的二值图,提取轮廓时误差较小。
[0042]
具体地,轮廓检测调用的是opencv中定义的findcontours函数,该函数能够检测图片最外面的轮廓,也可以检测所有的轮廓,建立等级关系而且使用起来方便。本实施例中,调用findcontours函数,用于检测外轮廓。
[0043]
此处,阈值的设定是根据获得的最大轮廓的面积乘以0.5进行处理。但也根据实际情况适当地调整这个系数。
[0044]
(2)根据获得的掩码将图片裁切成小块,裁切过程中边界像素记为boundary_pixels,图片缩放尺寸为img_scale,缩放后的边界像素点bp=boundary_pixels/img_scale,窗口大小为win_size=640/img_scale也和图片保持一样的缩放比。大图图片(即裁切前的图)的宽、高分别填充2*bp大小,然后以win_size大小的窗口进行裁切,重叠区域overlap=2*bp进行裁切,得到图片列表。所述图片列表是一个维度为(n,c,h,w)的数组,其中n表述图片的张数,c表示图片的通道,h和w分表表示图片的高与宽,形式如np.array([[[[100,255,
…
]],[[255,120,
…
]],[[100,90,
…
]]],[[[0,255,...]],[[3,200,
…
]],[[4,10,
…
]]],
…
])。
[0045]
此处,大图图片指的是病理条图片;图片一般比较大,千万像素甚至更大,在处理的时候要将大图切成小块,裁切图片的窗口和重叠区域可以按照具体任务来设定。
[0046]
(3)将图片列表按照要求的batch_size大小,将图片分批输入到训练好的分割模型中,得到分割后的结果。batch_size是根据网络所能接受的最大batch_size设定的;在一些实施方式中,batch_size可以设定为24或者32,在其他实施方式中,也可以设置设定为其他的数值,本技术不对此不做限制。对图片批量处理,处理速度更快、工作效率较高;比如,一次输入32张就会出来32张图片的结果,如果单独输入图片,那么就要32次才能得到32张
图片的结果。
[0047]
分割模型的训练主要包括数据集构建阶段、模型的训练阶段,
[0048]
输入模型的数据集为:上文提到的图片列表中的图片。模型的训练阶段:采用的是unet网络模型。unet网络模型的下采样过程用efficientnet_b3代替,使得更有效的提取图片中的边缘特征和语义特征。unet网络模型结构较为简单,参数量和计算量较低,对该任务来说unet模型较为合适。
[0049]
训练过程中输入模型的是n,c,h,w维度的数组图片,模型输出的是n,c,h,w的得分映射图(即预测掩码图)。
[0050]
本实施例中,模型训练过程中需要设置的参数:
[0051]
学习轮次为200次,初始学习率为0.01,batch_size大小为24;weight_decay=0.0001,使用的优化器为adam,模型训练至收敛即可;
[0052]
(4)根据裁切窗口坐标位置,将分割后的掩码拼接成一张大图。
[0053]
因为步骤(2)将一张大图裁切成小图时,会获得左上角和右下角的坐标,所以分割后的掩码根据当前图片的左上角右下角坐标拼接。
[0054]
掩码的第三个通道为炎症细胞的区域,然后获取该通道中非零元素的位置,将非零元素位置的值加起来,求平均值得到c1,然后对c1进行归一化处理,得到score=c1/255;这样就可以获得每个炎症细胞的得分score和细胞轮廓。炎症细胞轮廓根据掩码图片得到,每个细胞在掩码中都表现为0,1的两个值,利用findcontours函数就可以检测出所有的细胞轮廓。
[0055]
(5)根据得到的炎症细胞的轮廓,获得每个炎症细胞轮廓的重心位置,然后根据重心位置将细胞先以半径50的距离利用密度聚类聚集到一起,然后再以半径100的距离合并一些相似的小块,得到一个相对大的炎症细胞区域;
[0056]
此处采用的是现有技术中常规的密度聚类方法,本发明不对其进行改进。
[0057]
所述半径50,100并不是常规数学中所描述的半径,此处表示两个样本之间的最大距离,可以理解为像素之间的距离。所述半径指的是一种相对关系,作为密度聚类函数的参数。本实施例中,选择半径50,100;主要是先将附近的一些炎症细胞合并在一起,认为50的距离内,这样的炎症细胞是一样的,对其进行合并(即聚类)。然后再选择一个更大的距离100,再做一次聚类,这样小块炎症细胞区域又聚类为更大的块,这样方便统计炎症细胞的面积,进而来判断是否炎症细胞浸润了。本实施例中,选择半径为50,100后聚类得到的炎症细胞的区域比较符合临床诊断中的情况。在实际中可以根据实际情况进行选择。
[0058]
(6)最后求每个炎症细胞的凸包,对凸包内炎症细胞进行面积统计,根据公式计算面积得分score=1.0*(非浸润区域炎症细胞面积+浸润区域炎症细胞面积)/(肾小球、肾血管、肾小管区域面积+非细胞区域面积+非浸润区域炎症细胞面积+浸润区域炎症细胞面积),然后根据阈值将该得分划分不同的标签区域,从而得到炎症细胞浸润的区域及程度。
[0059]
所述浸润区域指的是炎症细胞聚集的区域;非浸润区域炎症细胞面积与浸润区域炎症细胞面积之和等于步骤(5)中得到的炎症细胞区域细胞面积。肾小球、肾血管、肾小管区域面积+非细胞区域面积+非浸润区域细胞面积+浸润区域细胞面积=整个病理条外轮廓面积。面积计算可以采用opencv函数contourarea()统计得到。
[0060]
在一些优选的方式中,可以通过opencv中函数convexhull得到凸包。
[0061]
实施例1
[0062]
具体地,首先获取病人张三的肾脏病理图片如图3(1)所示,获得肾小球、肾小管、肾血管的轮廓(在判断肾间质炎症细胞的时候,肾小球、肾小管、肾血管的轮廓是已知的,已经保存在了json文件中,此处只需要读取相应病理条的json文件即可),然后获得图片外轮廓,根据外轮廓生成一张与病理条图片大小一致的掩码图片,将掩码中外轮廓部分填充为1,将肾小球、肾血管、肾小管的位置填充为0。这样就得到了病理图片最终的掩码图,如图3(2)所示。
[0063]
然后将病理图片按预设窗口大小进行裁切,从而获得分割模型的输入图片列表,将裁切后的图片列表按照分割模型要求的batch_size大小进行批量输入,输入后会得到模型预测结果,然后对预测结果进行后处理,后处理过程中将预测得到的掩码图片,按照裁切时得到的图片左上角和右下角坐标,放到一张尺寸与大图一致的掩码图上(这里是将小图合并成大图的过程)。
[0064]
本实施例中,预设窗口是800*800的大小。在其他实施方式中,可以采用其他大小的预设窗口,比如1024*1024的大小,这个大小根据最终输入网络模型的图片大小确定,如果输入网络的图片大小为512*512,那么这两个窗口大小都可以使用,如果输入图片小于512那么就采用800*800的窗口;另外这个窗口的大小也要尽可能多的裁切到肾间质的部分,一般,先对整体病理图片缩小1/4或者1/8,然后选择1024*1024或者800*800的窗口,或者选择更大的窗口如2048*2048大小。
[0065]
最终得到大图的预测掩码,如图5所示;进一步地得到肾间质区域的炎症细胞的轮廓,根据预测掩码图,利用opencv的findcontours函数即可得到炎症细胞轮廓。
[0066]
得到炎症细胞轮廓后,先以半径50的距离合并炎症细胞,合并之后就得到小块的炎症细胞区域,然后再以100的距离合并炎症细胞的小块区域,得到一个相对大的炎症细胞区域;
[0067]
求每个炎症细胞的凸包,如图6所示,图6(1)为一种凸包的示例(在一些实施例中,可能会有很多凸包),对凸包内细胞进行面积统计,然后根据公式score=1.0*(非浸润区域细胞面积+浸润区域细胞面积)/(肾小球、肾血管、肾小管区域面积+非细胞区域面积+非浸润区域细胞面积+浸润区域细胞面积),根据阈值[0.05,0.1,0.2,0.3,1.0],即可得到炎症细胞浸润程度,分为无炎症细胞浸润,局灶性、多灶性、大片状、弥漫性。
[0068]
所述阈值可以采用常规的kmeans算法进行聚类得到。
[0069]
本实施例中,得分score小于0.05,则判断没有炎症细胞浸润,得分大于等于0.05,小于0.1,属于局灶炎症细胞浸润;得分大于等于0.1,小于0.2属于多灶炎症细胞浸润;得分大于等于0.2,小于0.3,属于大片状炎症细胞浸润;得分大于等于0.3,小于1.0属于弥漫性炎症细胞浸润。
[0070]
本实施例中所选取的阈值是当前比较合适的阈值。将得分与阈值进行比较来判定炎症细胞浸润的程度(无浸润、局灶性、多灶性、大片状、弥漫性)。
[0071]
本实施例中,采用本发明方法处理具体的图片,平均每张图片处理的时间为0.43ms,准确率是98.52%。图7为最终得到图片的一种示例,图7中,从图片中可以看到炎症细胞浸润的严重程度;正红色点代表炎症细胞浸润区域,从中可以看出哪些区域炎症细胞浸润比较严重(颜色深的区域表示炎症细胞浸润比较严重),哪些区域比较轻微。
[0072]
实施例2,参照附图2。
[0073]
在本实施例中,提供了一种计算机设备100,包括存储器102、处理器101及存储在存储器102上并可在处理器101上运行的计算机程序103,处理器101执行计算机程序103时能够实现上述实施例1所提供的一种基于深度学习的图像中肾间质炎症细胞确定方法中的步骤。
[0074]
实施例3
[0075]
在本实施例中,提供了一种计算机可读存储介质,其上存储有计算机程序,计算机程序被处理器执行时能够实现上述各个实施例所提供的一种基于深度学习的图像中肾间质炎症细胞确定方法中的步骤。
[0076]
在本实施例中,所述计算机程序可以是实施例2中的计算机程序。
[0077]
在本实施例中,所述计算机可读存储介质可以被实施例2中的计算机设备所运行。
[0078]
本领域普通技术人员可以理解实现上述实施例中的全部或部分流程,是可以通过计算机程序来指令相关的硬件来完成,该计算机程序可存储于一非易失性计算机可读取存储介质中,该计算机程序在执行时,可包括如上述各方法的实施例的流程。其中,本发明所提供的各实施例中所使用的对存储器、存储、数据库或其它介质的任何引用,均可包括非易失性和/或易失性存储器。非易失性存储器可包括只读存储器(rom)、可编程rom(prom)、电可编程rom(eprom)、电可擦除可编程rom(eeprom)或闪存。易失性存储器可包括随机存取存储器(ram)或者外部高速缓冲存储器。作为说明而非局限,ram以多种形式可得,诸如静态ram(sram)、动态ram(dram)、同步dram(sdram)、双数据率sdram(ddrsdram)、增强型sdram(esdram)、同步链路(synchlink)dram(sldram)、存储器总线(rambus)直接ram(rdram)、直接存储器总线动态ram(drdram)、以及存储器总线动态ram(rdram)等。
[0079]
以上所述实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例的所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。
[0080]
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对保护范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1