一种在单细胞水平上同时可视化和定量细胞累积金属纳米颗粒的方法
1.本发明属于纳米材料检测技术领域,具体涉及一种在单细胞水平上同时可视化和绝对定量细胞累积金属纳米颗粒的方法。
背景技术:
2.aunps颗粒(aunps)由于其独特的光学特性、优异的生物相容性和表面易修饰等特性被广泛应用于生物光学成像、生物传感器和药物递送等生物医学领域。在体外实验研究中,量化和可视化细胞中累积的aunps在评估药物递送效率、阐明纳米药物与细胞相互作用的机理以及aunps的内化机制等方面起到重要的作用。尤其是,在单细胞水平上研究细胞累积aunps的异质性能够提供更深层次的信息。
3.结合了暗场显微成像系统的高光谱技术是一种原位识别生物中aunps的新型方法。这是因为aunps具有独特的表面等离子体共振效应使其在400-1000nm的可见-近红外波段展现出很强的光谱吸收,而装配有独特分光光度计的高光谱成像系统中同时配备电荷耦合器件(ccd)相机,可记录这一波段aunps独特的光谱曲线。但是由于aunps的表面等离子体共振效应的敏感性,使得对aunps识别结果的可重复性较低,导致不同实验室或者不同技术人员所得到的定量的结果可能不一致。此外,在定量细胞中的aunps时,面对大量的高光谱图片数据,也没有方便省时的方法去处理。这些限制使得高光谱技术无法像其他技术那样成为定量细胞累积aunps的主流技术。
技术实现要素:
4.发明目的:本发明所要解决的技术问题是针对现有技术的不足,提供一种更加全面准确且简单的方法,在单细胞水平上同时可视化和绝对定量细胞累积金属纳米颗粒。
5.为了实现上述目的,本发明采取的技术方案如下:
6.一种在单细胞水平上同时可视化和定量细胞累积金属纳米颗粒的方法,包括如下步骤:
7.步骤一:将累积了一系列浓度金属纳米颗粒的细胞样品分别分为两组,一组用icp-ms定量细胞累积的金属纳米颗粒,另一组用高光谱技术定量细胞累积的金属纳米颗粒;
8.步骤二:根据步骤一两种定量结果之间的对应关系建立绝对定量的标准曲线;
9.步骤三:计算高光谱图片中单个细胞内金属纳米颗粒面积和细胞面积的比值,根据步骤二中的绝对定量的标准曲线,绝对定量单个细胞累积的金属纳米颗粒。
10.具体地,所述的金属纳米颗粒为在暗场高光谱成像系统下能够建立相应的参考库并特异性识别的纳米颗粒。
11.优选地,所述的金属纳米颗粒为含金属元素的纳米颗粒。
12.更优选地,所述的金属纳米颗粒包括纳米金、纳米银或纳米氧化铁。
13.具体地,步骤一中,用高光谱技术定量细胞累积的金属纳米颗粒包括以下步骤:
14.(1)准备待成像的细胞样品;
15.(2)利用步骤(1)准备的样品,获取相应的高光谱图片;
16.(3)将步骤(2)中获取的高光谱图片进行平滑处理,再使用获取的光源光谱将平滑后的高光谱图像进行光谱校正,随后收集金属纳米颗粒的光谱库,利用该光谱库识别细胞内化的金属纳米颗粒;
17.(4)计算每张图片中金属纳米颗粒面积和细胞面积的比值,统计每个处理组所有的高光谱图片中该比值的平均值去定量细胞累积的金属纳米颗粒。
18.其中,步骤(2)中,对于每个样品,随机获取不同视野的6到8张高光谱图片,这些高光谱图片中共含有》50个细胞。
19.步骤(3)中,利用envi 4.8软件中的adjacent band averaging功能将高光谱图像进行平滑处理。
20.步骤(4)中,细胞面积的计算包括如下步骤:
21.s1:先通过人工处理一定数量细胞的高光谱图片,获得细胞面积;
22.s2:对步骤s1的数据进行机器学习建模,用于建模的数据-标签对为细胞的高光谱图片-人工处理后的细胞图片;
23.s3:用步骤s2得到的机器学习模型自动识别新的细胞高光谱图片,得到处理后的细胞图片,再以像素个数表示细胞面积。
24.具体地,步骤s1中,使用imagej人工处理细胞的高光谱图片,获得细胞面积,用该方法处理至少30张图片。
25.具体地,步骤s2中,基于python 3.6的sklearn包对步骤s1的数据进行机器学习建模;使用的机器学习方法为决策树decisiontree。
26.有益效果:
27.本发明建立了一种同时定量和可视化单细胞累积无标记纳米金的方法,利用机器学习的方法高效准确处理大量的高光谱图片数据,通过icp-ms绝对定量的结果与高光谱定量结果的标准曲线,将高光谱技术定量细胞累积纳米金的结果绝对化。
附图说明
28.下面结合附图和具体实施方式对本发明做更进一步的具体说明,本发明的上述和/或其他方面的优点将会变得更加清楚。
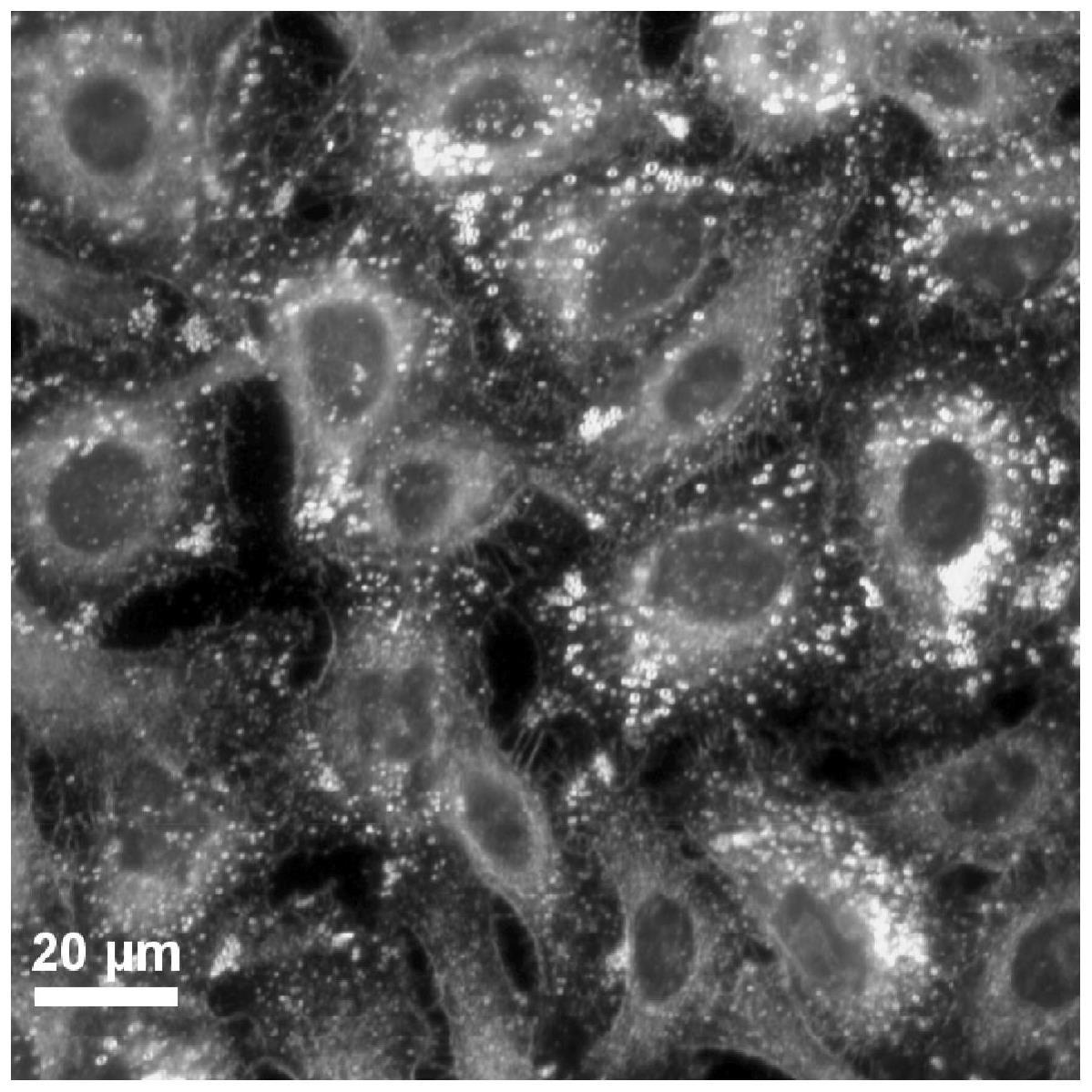
29.图1是细胞的高光谱图片。
30.图2是imagej手绘的细胞面积。
31.图3是机器学习识别的细胞面积。
32.图4是机器学习识别误差的正态分布。
33.图5是icp-ms和高光谱定量结果的标准曲线。
34.图6是imagej绘制的细胞轮廓。
35.图7是统计分析单个细胞之间累积aunps的差异分布。
具体实施方式
36.根据下述实施例,可以更好地理解本发明。
37.实施例1
38.本实施例提供了在单细胞水平上同时可视化和绝对定量细胞累积纳米金(aunps)的方法,包括以下步骤:
39.步骤一:六孔板中放入5张无菌盖玻片,将约40万个hela细胞接种在这5个孔中,并在盖玻片上生长24h以贴壁,随后更换含浓度分别为0.3、1、3mg/l的60nm柠檬酸修饰aunps的培养基,暴露时间为2、4、6和8h。细胞吸收aunps后用2ml pbs缓冲液清洗5遍。将5个孔的细胞分为两组,其中3个孔中的细胞为一组用icp-ms定量细胞累积的aunps;另2个孔的细胞为一组用高光谱技术定量细胞累积的aunps;
40.对于高光谱技术定量细胞累积的aunps,具体步骤为:
41.(1)在用于成像的2个孔中,加入含2%多聚乙醛的pbs溶液将细胞固定20min,随后用pbs清洗2遍,带有细胞的盖玻片覆盖在载玻片上,用指甲油密封以保持细胞处于液体状态。
42.(2)利用该样品,使用暗场高光谱显微镜,对此吸收了aunps的细胞进行成像。对于每张盖玻片,随机获取不同视野的6-8张高光谱图片,这些高光谱图片一共含有》50个细胞。
43.(3)获取高光谱图片后,利用envi 4.8软件中的adjacent band averaging功能将所有高光谱图像进行平滑处理,再使用获取的光源光谱将平滑后的高光谱图像进行光谱校正。随后收集aunps的光谱库,利用该光谱库识别细胞内化的aunps。
44.(4)计算每张图片中识别出的aunps的面积和细胞面积的比值,取每个处理组所有的高光谱图片中该比值的平均值去定量细胞累积的aunps。其中细胞面积的计算过程为:
45.s1使用imagej,将高光谱图片(如图1)调为8bit,手动描出细胞的轮廓。随后通过人工描边的方式,描画出细胞的精细轮廓,最后将细胞部分调为黑色,其他部分调为白色,如图2所示。再统计黑色像素个数表示的细胞面积。用该方法处理40张图片。
46.s2基于python 3.6的sklearn包对步骤一的数据进行机器学习建模。用于建模的数据-标签对为细胞的高光谱图片-人工处理后细胞图片,使用的机器学习方法为决策树(decisiontree)。其中75%的数据-标签对作为训练集,其余25%作为测试集。训练得到的模型在训练集上准确率为95.7%,测试集上准确率95.6%。对比imagej加人工处理识别得到的细胞面积和机器学习模型得到的细胞面积(如图3),两者误差分布符合正态分布(如图4),误差率在15%内。
47.s3用得到的机器学习模型自动识别新的细胞高光谱图片,快速得到处理后的细胞图片,再以像素个数表示细胞面积。
48.步骤二:根据步骤一两种定量结果之间的对应关系建立绝对定量的标准曲线如图5所示。
49.步骤三:如图6所示,使用imagej软件,半自动描出单个细胞的轮廓,并同时计算高光谱图片中单个细胞中识别出的aunps面积和细胞面积的比值,在2h,4h和6h三个时间点分别统计分析25张照片的细胞。根据步骤二中的绝对定量的标准曲线,将高光谱定量的结果转为绝对定量的结果,如图7所示。此结果说明了细胞之间累积aunps的能力有一定的异质性。
50.本发明提供了一种在单细胞水平上同时可视化和定量细胞累积金属纳米颗粒的方法的思路及方法,具体实现该技术方案的方法和途径很多,以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。本实施例中未明确的各组成部分均可用现有技术加以实现。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1