指纹谱图数据库的构建方法、装置以及可读存储介质
1.本发明涉及生物技术领域,尤其涉及指纹谱图数据库的构建方法、指纹谱图数据库的构建装置和计算机可读存储介质。
背景技术:
2.在微生物病原菌鉴定过程中,一般通过检测样本的蛋白组成获得特征峰,与数据库中细菌指纹图谱进行比较,达到鉴定细菌属、种的目的。其中,在数据库建库的方法是不同菌种的菌株进行纯培养,再通过标准化的前处理后上机采集谱图,将采集的谱图经过一定的质量筛选后进行预建库,验证合格可作为菌株的标准数据库图谱进行入库。
3.目前复合群在建库的过程中,不同菌种是进行单独建库的,也就是,每个菌种会分别建立一张标准谱图进行入库。然而,单独建库存在复合群中不同菌种的谱图的相似性较高导致入库数据出现混淆、建库过程容易出现入库菌株谱图信息不全的结果、数据库仅能包含复合群中部分已知菌种的谱图等问题,这容易导致数据库应用于复合群鉴定时存在鉴定准确性和鉴定成功率不佳的问题。
技术实现要素:
4.本发明的主要目的在于提供一种指纹谱图数据库的构建方法、指纹谱图数据库的构建装置以及计算机可读存储介质,旨在提高复合群鉴定的准确性和鉴定成功率。
5.为实现上述目的,本发明提供一种指纹谱图数据库的构建方法,所述指纹谱图数据库的构建方法包括以下步骤:
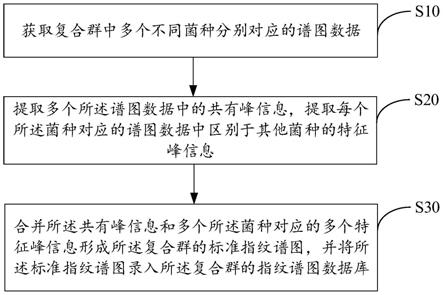
6.获取复合群中多个不同菌种分别对应的谱图数据;
7.提取多个所述谱图数据中的共有峰信息,提取每个所述菌种对应的谱图数据中区别于其他菌种的特征峰信息;
8.合并所述共有峰信息和多个所述菌种对应的多个特征峰信息形成所述复合群的标准指纹谱图,并将所述标准指纹谱图录入所述复合群的指纹谱图数据库。
9.可选地,所述提取多个所述谱图数据中的共有峰信息,提取每个所述菌种对应的谱图数据中区别于其他菌种的特征峰信息的步骤包括:
10.对所述多个不同菌种对应的全部谱图数据进行聚类分析,获得聚类分析结果;
11.根据所述聚类分析结果确定每个所述菌种对应的所述特征峰信息的目标数量;
12.根据每个所述菌种对应的所述特征峰信息的目标数量提取每个所述菌种对应的所述特征峰信息。
13.可选地,所述聚类分析结果包括每个所述菌种的聚类距离,所述根据所述聚类分析结果确定每个所述菌种对应的所述特征峰信息的目标数量的步骤包括:
14.根据每个所述菌种的聚类距离对应确定每个菌种对应的所述特征峰信息的目标数量。
15.可选地,所述根据每个所述菌种的聚类距离对应确定每个菌种对应的所述特征峰
信息的目标数量的步骤包括:
16.根据每个所述菌种的聚类距离与特征距离之间的数量关系,将每个所述菌种的聚类距离转换成每个菌种对应的距离表征值,所述距离表征值为大于1的数值;
17.根据每个菌种对应的距离表征值的对数值确定每个菌种对应的所述特征峰信息的目标数量。
18.可选地,所述根据每个菌种对应的距离表征值的对数值确定每个菌种对应的所述特征峰信息的目标数量的步骤包括:
19.按照以下公式计算每个菌种对应的目标数量:
20.mi=a*logdi+b;
21.其中,mi为每个菌种对应的所述特征峰信息的目标数量,a和b均为预设值,di为每个菌种对应的距离表征值,log di为每个菌种对应的距离表征值的对数值。
22.可选地,所述聚类分析结果还包括所述复合群中的菌种数量,所述根据所述聚类分析结果确定每个所述菌种对应的所述特征峰信息的目标数量的步骤包括:
23.当所述菌种数量多于两种时,执行所述根据每个所述菌种的聚类距离对应确定每个菌种对应的所述特征峰信息的目标数量的步骤;
24.当所述菌种数量为两种时,确定每个所述菌种对应的谱图数据中区别于其他菌种的信号峰的总数,根据两个所述总数确定每个所述菌种对应的所述特征峰信息的目标数量。
25.可选地,所述根据两个所述总数确定每个所述菌种对应的所述特征峰信息的目标数量的步骤包括:
26.确定两个所述总数中的最小数值为每个所述菌种对应的目标数量。
27.可选地,所述获取复合群中多个不同菌种分别对应的谱图数据的步骤包括:
28.按照标准提取法提取所述复合群的样本获得每个所述菌种分别对应的菌株,并采集每个菌种的菌株的多个谱图;
29.在每个菌种对应的多个谱图中删除满足预设条件的预设个谱图,获得每个菌种对应的多个目标谱图,所述预设条件包括主峰的基线漂移量小于预设漂移量、且谱图内多个特征峰的强度偏差值大于预设差值;
30.将每个菌种的多个目标谱图与对应的菌种的属性信息关联,获得所复合群中每个菌种对应的谱图数据;
31.且/或,所述合并所述共有峰信息和多个所述菌种对应的多个特征峰信息形成所述复合群的标准指纹谱图的步骤之后,还包括:
32.根据每个所述谱图数据与所述标准指纹谱图的比对结果,确定每个谱图数据对应的鉴定分值;
33.若每个所述鉴定分值均大于设定分值,则执行所述将所述标准指纹谱图录入所述复合群的指纹谱图数据库的步骤。
34.此外,为了实现上述目的,本技术还提出一种指纹谱图数据库的构建装置,所述指纹谱图数据库的构建装置包括:存储器、处理器及存储在所述存储器上并可在所述处理器上运行的指纹谱图数据库的构建程序,所述指纹谱图数据库的构建程序被所述处理器执行时实现如上任一项所述的指纹谱图数据库的构建方法的步骤。
35.此外,为了实现上述目的,本技术还提出一种计算机可读存储介质,所述计算机可读存储介质上存储有指纹谱图数据库的构建程序,所述指纹谱图数据库的构建程序被处理器执行时实现如上任一项所述的指纹谱图数据库的构建方法的步骤。
36.本发明提出的一种指纹谱图数据库的构建方法,该方法在获得复合群中多个不同菌种分别对应的谱图数据后,提取不同菌种在多个谱图数据中的共有峰信息,提取每个菌种在其谱图数据中特有的特征峰信息,将提取得到的共有峰信息和多个特征峰信息合并形成复合群的标准指纹谱图进行入库。基于此,通过将复合群中不同菌种在谱图数据中共有和特有的峰信息进行合并形成标准谱图,有利于避免单独建库时不同菌种的谱图相似性导致菌种的鉴定结果混淆的问题,也有利于避免单一菌种的谱图信息不全导致的鉴定分数过低或与其他菌种的鉴定结果出现混淆,还有利于数据库应用于鉴定复合群内未入库的菌种时避免出现假阳性或鉴定不出的结果,可提高复合群的鉴定分数、鉴定成功率以及未入库的复合群中其他菌种的检出率,从而有效提高复合群鉴定的准确性和鉴定成功率。
附图说明
37.图1为本发明指纹谱图数据库的构建装置一实施例运行涉及的硬件结构示意图;
38.图2为本发明指纹谱图数据库的构建方法一实施例的流程示意图;
39.图3为本发明指纹谱图数据库的构建方法另一实施例的流程示意图;
40.图4为本发明指纹谱图数据库的构建方法又一实施例的流程示意图;
41.图5为本发明指纹谱图数据库的构建方法再一实施例的流程示意图。
42.本发明目的的实现、功能特点及优点将结合实施例,参照附图做进一步说明。
具体实施方式
43.应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
44.本发明实施例的主要解决方案是:获取复合群中多个不同菌种分别对应的谱图数据;提取多个所述谱图数据中的共有峰信息,提取每个所述菌种对应的谱图数据中区别于其他菌种的特征峰信息;合并所述共有峰信息和多个所述菌种对应的多个特征峰信息形成所述复合群的标准指纹谱图,并将所述标准指纹谱图录入所述复合群的指纹谱图数据库。
45.由于现有技术中,复合群在建库的过程中,不同菌种是进行单独建库的,也就是,每个菌种会分别建立一张标准谱图进行入库。然而,单独建库存在复合群中不同菌种的谱图的相似性较高导致出现混淆、建库过程容易出现入库菌株谱图信息不全的结果、数据库仅能包含复合群中部分已知菌种的谱图等问题,这容易导致数据库应用于复合群鉴定时存在鉴定准确性和鉴定成功率不佳的问题。
46.本发明提供上述的解决方案,旨在提高复合群鉴定的准确性和鉴定成功率。
47.本发明实施例提出一种指纹谱图数据库的构建装置,具体用于复合群的指纹谱图数据库构建。
48.在本发明实施例中,参照图1,指纹谱图数据库的构建装置包括:处理器1001(例如cpu),存储器1002等。处理器1001与存储器1002通过通信总线连接。存储器1002可以是高速ram存储器,也可以是稳定的存储器(non-volatile memory),例如磁盘存储器。存储器1002可选的还可以是独立于前述处理器1001的存储装置。
49.本领域技术人员可以理解,图1中示出的装置结构并不构成对装置的限定,可以包括比图示更多或更少的部件,或者组合某些部件,或者不同的部件布置。
50.如图1所示,作为一种计算机可读存储介质的存储器1002中可以包括指纹谱图数据库的构建程序。在图1所示的装置中,处理器1001可以用于调用存储器1002中存储的指纹谱图数据库的构建程序,并执行以下实施例中指纹谱图数据库的构建方法的相关步骤操作。
51.本发明实施例还提供一种指纹谱图数据库的构建方法,应用于上述的指纹谱图数据库的构建装置。
52.参照图2,提出本技术指纹谱图数据库的构建方法一实施例。在本实施例中,所述指纹谱图数据库的构建方法包括:
53.步骤s10,获取复合群中多个不同菌种分别对应的谱图数据;
54.这里的复合群具体为基因高度同源的多个菌种的集合。在本实施例中,复合群指的是非结核分枝杆菌,指的是除结核分枝杆菌复合群和麻风分枝杆菌以外的一大类分枝杆菌的总称,具体包括鸟-胞内分枝杆菌复合群(mac)、脓肿分枝杆菌和偶发分枝杆菌,次要菌种有堪萨斯分枝杆菌、龟分枝杆菌等菌种。在其他实施例中,复合群也可基因高度同源的菌种集合。
55.这里的谱图数据具体为满足预设入库条件的谱图数据。谱图数据具体包括基于各微生物特征性肽指纹图谱形成并满足入库条件的多个谱图及其对应的菌种的属性信息(如菌名、属名、菌株名、中文名等)。
56.步骤s20,提取多个所述谱图数据中的共有峰信息,提取每个所述菌种对应的谱图数据中区别于其他菌种的特征峰信息;
57.这里的共有峰信息具体指的是存在于所有谱图数据中出现概率大于第一设定阈值的信号峰的特征信息(如信号峰的峰强度、峰面积、峰保留时间及其对应的菌种的属性信息等);特征峰信息具体指的是每个菌种的谱图数据出现概率大于第二设定阈值且在复合群中其他菌种的谱图数据中出现概率小于第三设定阈值的信号峰的特征信息(如信号峰的峰强度、峰面积、峰保留时间及其对应的菌种的属性信息等)。
58.具体的,可统计所有谱图数据中出现的不同位置的信号峰在所有菌种对应的所有谱图数据中出现的第一概率,将第一概率大于第一设定阈值的所有信号峰或部分信号峰的特征信息选取作为这里的共有峰信息;在每个菌种对应的谱图数据内出现的不同位置的信号峰内,将第一概率大于第一设定阈值的信号峰以外的其他信号峰的集合作为目标信号峰集合,在每个菌种对应的目标信号峰集合中统计每个目标信号峰在对应菌种的谱图数据内所有谱图中出现的第二概率,将第二概率大于第二设定阈值的部分目标信号峰或全部信号峰的特征信息选取作为这里的特征峰信息。
59.步骤s30,合并所述共有峰信息和多个所述菌种对应的多个特征峰信息形成所述复合群的标准指纹谱图,并将所述标准指纹谱图录入所述复合群的指纹谱图数据库。
60.将共有峰信息和多个菌种的多个特征峰信息进行合并形成复合群峰列表,复合群峰列表可作为这里的复合群的标准指纹谱图,这里的标准指纹谱图同时包含表征复合群内不同菌种共性的信号峰的第一特征信息以及表征复合群内不同菌种特异性的信号峰的第二特征信息。将所获得的标准指纹谱图录入复合群的指纹谱图数据库,便可完成复合群的
指纹谱图数据库的构建。
61.这里的复合群的指纹谱图数据库可用于鉴定待测样本菌群是否为这里的复合群。
62.本发明实施例提出的一种指纹谱图数据库的构建方法,该方法在获得复合群中多个不同菌种分别对应的谱图数据后,提取不同菌种在多个谱图数据中的共有峰信息,提取每个菌种在其谱图数据中特有的特征峰信息,将提取得到的共有峰信息和多个特征峰信息合并形成复合群的标准指纹谱图进行入库。基于此,通过将复合群中不同菌种在谱图数据中共有和特有的峰信息进行合并形成标准谱图,有利于避免单独建库时不同菌种的谱图相似性导致菌种的鉴定结果混淆的问题,也有利于避免单一菌种的谱图信息不全导致的鉴定分数过低或与其他菌种的鉴定结果出现混淆,还有利于数据库应用于鉴定复合群内未入库的菌种时避免出现假阳性或鉴定不出的结果,可提高复合群的鉴定分数、鉴定成功率以及未入库的复合群中其他菌种的检出率,从而有效提高复合群鉴定的准确性和鉴定成功率。
63.进一步的,基于上述实施例,提出本技术指纹谱图数据库的构建方法另一实施例。在本实施例中,参照图3,所述提取多个所述谱图数据中的共有峰信息,提取每个所述菌种对应的谱图数据中区别于其他菌种的特征峰信息的步骤包括:
64.步骤s21,对所述多个不同菌种对应的全部谱图数据进行聚类分析,获得聚类分析结果;
65.具体的,对全部指纹谱图中的所有信号峰进行聚类分析,基于聚类分析可确定复合群中的菌种数量和/或不同菌种对应的聚类距离等信息作为这里的聚类分析结果。
66.步骤s22,根据所述聚类分析结果确定每个所述菌种对应的所述特征峰信息的目标数量;
67.这里的目标数量具体指的是每个菌种对应的区别于其他菌种的所有信号峰的特征信息中参与构建指纹谱图数据库的信号峰所需达到的数量。
68.具体的,不同的菌种数量对应的每个菌种对应的特征峰信息的目标数量可不同,且/或,每个菌种对应的聚类距离不同则其该菌种对应的特征峰信息的目标数量不同。
69.在本实施例中,所述聚类分析结果包括每个所述菌种的聚类距离,所述根据所述聚类分析结果确定每个所述菌种对应的所述特征峰信息的目标数量的步骤包括:根据每个所述菌种的聚类距离对应确定每个菌种对应的所述特征峰信息的目标数量。这里菌种的聚类距离不同则其对应的特征峰信息的目标数量不同。目标数量与聚类距离呈正相关。具体的,可预先建立每个菌种的聚类距离与其对应的目标数量之间的对应关系。该对应关系可以是计算公式、映射关系(如映射表等)等。基于该对应关系可确定每个菌种的聚类距离所对应的特征峰信息的目标数量。
70.步骤s23,根据每个所述菌种对应的目标数量提取每个所述菌种对应的谱图数据中区别于其他菌种的特征峰信息。
71.具体的,可在每个菌种所有区别于其他菌种的信号峰特征信息中,基于峰强度将所有信号峰特征信息按照从大至小依次进行排列,分别将每个菌种排列在前的目标数量个信号峰特征信息作为每个菌种对应的特征峰信息。
72.例如,复合群包括菌种1、菌种2和菌种3,获取的复合群对应的谱图数据包括:菌种1对应的a个谱图,菌种2对应的b个谱图,以及菌种3对应的c个谱图;从a+b+c个谱图中确定共有峰,获得共有峰对应的第一信息;根据聚类分析结果确定每个菌种分别对应的目标数
量,即确定菌种1对应的目标数量为x,确定菌种2对应的目标数量为y,确定菌种3对应的目标数量为z;从菌种1对应的a个谱图中确定a个谱图共有的第一信号峰集合,从第一信号峰集合中提取x个特征峰,从菌种2对应的b个谱图中确定b个谱图共有的第二信号峰集合,从第二信号峰集合中提取y个特征峰,从菌种3对应的c个谱图中确定c个谱图共有的第三信号峰集合,从第三信号峰集合中提取z个特征峰;将x个特征峰的第二信息、y个特征峰的第三信息、z个特征峰的第四信息与上述确定的第一信息合并形成复合群峰列表作为标准指纹图谱。
73.在本实施例中,基于复合群种中不同菌种对应的全部谱图数据的聚类分析结果确定每个菌种用于建库的特征峰信息的目标数量,按照所确定的目标数量提取相应数量的特征峰信息参与建库,有利于进一步提高所获得数据库用于鉴定复合群时的鉴定准确性和鉴定成功率。
74.进一步的,在本实施例中,所述根据每个所述菌种的聚类距离对应确定每个菌种对应的所述特征峰信息的目标数量的步骤包括:
75.根据每个所述菌种的聚类距离与特征距离之间的数量关系,将每个所述菌种的聚类距离转换成每个菌种对应的距离表征值,所述距离表征值为大于1的数值;根据每个菌种对应的距离表征值的对数值确定每个菌种对应的所述特征峰信息的目标数量。
76.这里的特征距离可为预先设置的固定距离,也可从所有菌种对应的多个聚类距离选择其中之一作为特征距离。
77.这里每个菌种对应的数量关系可为每个菌种的聚类距离与特征距离之间的比值、差值或乘积等。
78.按照数量关系将每个菌种的聚类距离转换成大于1的数值作为每个菌种对应的距离表征值。
79.在本实施例中,将所有菌种对应的多个聚类距离中选取数值最小的聚类距离作为特征距离,并设置数值最小的聚类距离所对应的目标菌种的距离表征值为1。基于此,可确定目标菌种以外的每个菌种的聚类距离与特征距离之间的比值,将每个菌种对应获得的比值作为该菌种的距离表征值。
80.具体的,可每个菌种对应的距离表征值的对数值通过计算或查表等方式得到每个菌种所对应的所述特征峰信息的目标数量。
81.在本实施例中,为了进一步提高所确定的目标数量的准确性,步骤s222可包括:按照以下公式计算每个菌种对应的目标数量:
82.mi=a*logdi+b;
83.其中,mi为每个菌种对应的所述特征峰信息的目标数量,a和b均为预设值,di为每个菌种对应的距离表征值,log di为每个菌种对应的距离表征值的对数值。
84.在本实施例中,将每个菌种的聚类距离转换成大于1的距离表征值,再基于每个菌种的距离表征值来确定每个菌种对应的特征峰的目标数量,从而保证聚类距离过大时对应的目标数量过多,有利于保证用于建库的特征峰信息对其对应菌种的特异性表征的准确性,从而进一步提高所获得数据库用于鉴定复合群时的鉴定准确性和鉴定成功率。
85.进一步的,基于上述任一实施例,提出本技术指纹谱图数据库的构建方法又一实施例。在本实施例中,参照图4,所述聚类分析结果还包括所述复合群中的菌种数量,所述步
骤s22包括:
86.步骤s221,当所述菌种数量多于两种时,执行所述根据每个所述菌种的聚类距离对应确定每个菌种对应的所述特征峰信息的目标数量的步骤;
87.例如,菌种数量为3种、4种、5种或更多数量时,可基于每个菌种的聚类距离对应确定每个菌种对应的目标数量。
88.这里基于聚类距离确定对应的菌种的目标数量的具体实现方式可参见上述实施例,在此不作赘述。
89.步骤s222,当所述菌种数量为两种时,确定每个所述菌种对应的谱图数据中区别于其他菌种的信号峰的总数,根据两个所述总数确定每个所述菌种对应的所述特征峰信息的目标数量。
90.具体的,可将上述实施例中提及的每个菌种的目标信号峰集合中信号峰的总数作为每个菌种对应的区别于其他菌种的信号峰的总数。
91.在本实施例中,确定两个所述总数中的最小数值为每个所述菌种对应的目标数量。例如,菌种数量为2,除共有峰以外,菌种1有s根峰,菌种2有t根峰,则每个菌种对应的目标数量相等且均为min(s,t)。
92.在其他实施例中,也可确定两个总数的均值作为每个菌种对应的目标数量。
93.在本实施例中,在菌种数量为两种时每个菌种提取的特征峰信息的目标数量相同且基于各个菌种特有的信号峰的总数确定;在菌种数量多于两种时基于菌种的聚类距离来确定每个菌种所需提取的特征峰信息的目标数量,有利于保证无论复合群内菌种数量有多少所构建的标准数据库可准确反映复合群中每种菌种的特点,从而进一步提高所获得数据库用于鉴定复合群时的鉴定准确性和鉴定成功率。
94.进一步的,基于上述任一实施例,提出本技术指纹谱图数据库的构建方法再一实施例。在本实施例中,参照图5,所述步骤s10包括:
95.步骤s11,按照标准提取法提取所述复合群的样本获得每个所述菌种分别对应的菌株,并采集每个菌种的菌株的多个谱图;
96.在本实施例中,按照标准提取法(例如标准甲酸乙腈提取法等)对复合群的多个菌种的菌株进行提纯采集,获得多个菌株,每个菌株采集多个靶点分别对应的多张谱图,获得每个菌种对应的多个谱图。例如,将样本按照标准甲酸乙腈提取法对复合群种1、复合群种2和复合群种3分别进行采集,每组采集4个靶点*3张,每个菌种对应获得12张谱图。
97.步骤s12,在每个菌种对应的多个谱图中删除满足预设条件的预设个谱图,获得每个菌种对应的多个目标谱图,所述预设条件包括主峰的基线漂移量小于预设漂移量、且谱图内多个特征峰的强度偏差值大于预设差值;
98.这里的预设条件具体为以每个菌种的多个目标谱图中重复性达到要求为目标所设置的条件,满足预设条件的谱图可认为是明显区别于该菌种对应的多个谱图中的其他谱图。
99.例如,在每个菌种的多个谱图中,剔除强度排名前3的主峰质量轴漂移不超过6da、强度差异超过3倍的谱图,获得每个菌种对应的多个目标谱图。
100.步骤s13,将每个菌种的多个目标谱图与对应的菌种的属性信息关联,获得所复合群中每个菌种对应的谱图数据。
101.属性信息具体包括菌名、属名、株名、中文名等。
102.在本实施例中,通过上述方式可使每个菌种中重复性较高的谱图数据才会用于艰苦,从而有利于保证进一步提高后续所构建的数据库的准确性。
103.进一步的,基于上述任一实施例,所述合并所述共有峰信息和多个所述菌种对应的多个特征峰信息形成所述复合群的标准指纹谱图的步骤之后,还包括:根据每个所述谱图数据与所述标准指纹谱图的比对结果,确定每个谱图数据对应的鉴定分值;若每个所述鉴定分值均大于设定分值,则执行所述将所述标准指纹谱图录入所述复合群的指纹谱图数据库的步骤。
104.鉴定分值具体为用于评价鉴定谱图数据对应的菌种属于标准指纹谱图对应的复合群的信度大小的参数。鉴定分值越大则菌种属于标准指纹谱图对应的复合群的可信度越大。
105.在本实施例中,通过所有谱图数据对标准指纹谱图进行反搜,在所有获得的鉴定分值大于设定分值时才将标准指纹谱图录入数据库,从而进一步保证所获得的指纹谱图数据库应用于鉴定复合群时的鉴定准确性和成功率。
106.此外,本发明实施例还提出一种计算机可读存储介质,所述计算机可读存储介质上存储有指纹谱图数据库的构建程序,所述指纹谱图数据库的构建程序被处理器执行时实现如上指纹谱图数据库的构建方法任一实施例的相关步骤。
107.需要说明的是,在本文中,术语“包括”、“包含”或者其任何其他变体意在涵盖非排他性的包含,从而使得包括一系列要素的过程、方法、物品或者系统不仅包括那些要素,而且还包括没有明确列出的其他要素,或者是还包括为这种过程、方法、物品或者系统所固有的要素。在没有更多限制的情况下,由语句“包括一个
……”
限定的要素,并不排除在包括该要素的过程、方法、物品或者系统中还存在另外的相同要素。
108.上述本发明实施例序号仅仅为了描述,不代表实施例的优劣。
109.通过以上的实施方式的描述,本领域的技术人员可以清楚地了解到上述实施例方法可借助软件加必需的通用硬件平台的方式来实现,当然也可以通过硬件,但很多情况下前者是更佳的实施方式。基于这样的理解,本发明的技术方案本质上或者说对现有技术做出贡献的部分可以以软件产品的形式体现出来,该计算机软件产品存储在如上所述的一个存储介质(如rom/ram、磁碟、光盘)中,包括若干指令用以使得一台终端设备(可以是手机,计算机,服务器,指纹谱图数据库的构建装置,或者网络设备等)执行本发明各个实施例所述的方法。
110.以上仅为本发明的优选实施例,并非因此限制本发明的专利范围,凡是利用本发明说明书及附图内容所作的等效结构或等效流程变换,或直接或间接运用在其他相关的技术领域,均同理包括在本发明的专利保护范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1