同步鉴定水生生态系统中浮游藻类和底栖藻类的方法
本发明属于分子生物学的技术领域,具体涉及一种基于高通量测序技术同步鉴定水生生态系统中浮游藻类和底栖藻类的方法。
背景技术:
藻类(algae)是指一群能进行光合作用,营自养生活,无维管束,没有真正根茎叶分化的一类叶状体植物。藻类是海洋、河流、湖泊、水库等水生生态系统中最重要的初级生产者,在碳、氮、磷、硫、硅等元素的生物地球化学循环中发挥重要作用。藻类是生态系统食物链的基础,为生态系统中物种多样性提供了基本保障。藻类分布范围广,生长周期短,对营养盐、光照、温度及扰动等环境变化非常敏感,常作为水质评价中理想的指示生物。生态系统的水体和沉积物中定居的浮游藻类和底栖藻类不断进行交互,塑造了特殊的微生境,为其他动物的生存创造了基本条件,是立体生境的重要建立者和塑造者。
藻类的鉴定及分类是藻类群落组成和功能研究的基础,只有对藻类进行正确的识别和鉴定后,才能充分理解藻类在生态系统中的价值,为生态系统健康发展的宏观调控提供理论依据。
迄今为止,关于真核和原核藻类的物种鉴定方法一直都没有统一的认识。目前,基于藻类细胞形态特征的传统形态学方法可以对藻类物种进行鉴定,应用最为广泛,但存在许多不足。首先,藻类的种类繁多,且许多种属的藻类形态差异细微,只有长期从事该工作的专业人员才能胜任,不同鉴定者的鉴定结果之间的差异可能很大,难以有标准化的检测结果;其次,某些藻类需要经过纯培养后根据其生活史进行鉴定,部分藻类很难分离纯化培养,培养需要大量时间且成功率较低,因此难以快速鉴定;另外,该方法对于尺寸较大且形态特征明显的藻类,较容易鉴定,但对于在显微镜下可识别形态特征较少的藻类,鉴定则非常困难;更重要的是,传统形态学鉴定方法主要偏好于识别环境水体中的浮游藻类,无法对沉积物中的底栖藻类进行准确鉴定。
中国专利申请公开文献cn106048058a(申请号为cn201610647307.x)公开了一种鉴定环氧丙烷皂化废水活性污泥中真核藻类多样性的引物及方法,其通过设计真核藻类特异性引物(上游引物:5’-aaagttaggtgagcg-3’,下游引物:5’-cagtcaatcggtatg-3’),可以从环氧丙烷皂化废水活性污泥宏基因组中特异性扩增获得不同真核藻类,从而鉴定活性污泥中的真核藻类种类。虽然该方法可以对环境样本中的真核藻类进行鉴定,但无法对原核藻类进行特异性扩增和鉴定,影响了对环境系统中所有藻类物种组成及生态功能的探究和评价。
鉴于目前已有鉴定技术的局限性,迫切地需要建立一套可以对水生生态系统中既包含真核藻类又包含原核藻类的全部浮游和底栖藻类进行快速、准确、高效、可标准化鉴定的技术方案。
技术实现要素:
本发明的目的在于提供一种基于高通量测序技术同步鉴定水生生态系统中浮游藻类和底栖藻类的方法,能够对水体和沉积物中的所有原核藻类和真核藻类进行同步鉴定,可以快速、高效地获取可标准化的藻类物种信息。
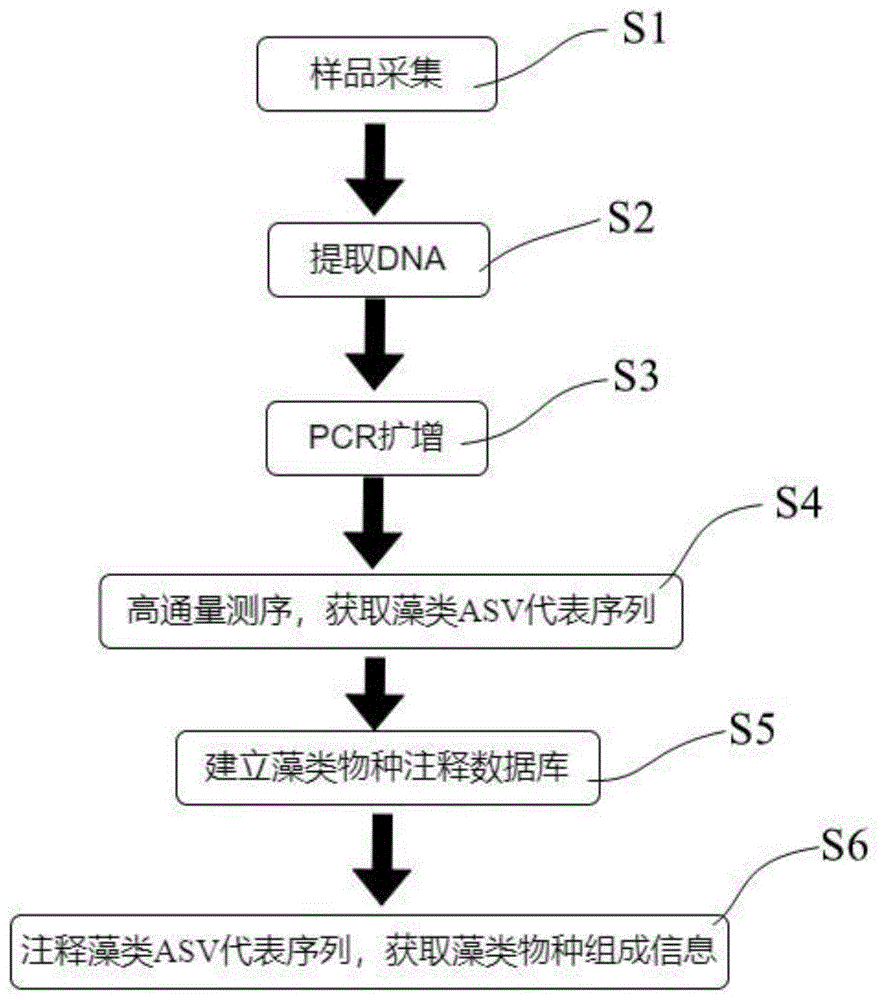
本发明所提供的同步鉴定水生生态系统中浮游藻类和底栖藻类的方法包括以下步骤:步骤s1,从所述水生生态系统中采集水样和沉积物样品;步骤s2,从所述水样和所述沉积物样品中分别提取并纯化dna;步骤s3,以纯化的dna作为模板进行pcr扩增;步骤s4,对pcr扩增的产物进行高通量测序,获取藻类asv代表序列;步骤s5,建立藻类物种注释数据库;步骤s6,将所述藻类asv代表序列与所述藻类物种注释数据库的序列数据进行比对和物种注释,获取藻类物种组成信息。
根据本发明的同步鉴定水生生态系统浮游藻类和底栖藻类的方法,优选地,在所述步骤s1中,从所述水体环境中采集5~10l表层水水样并混合均匀,同时从所述表层水水样对应位置处采集表层沉积物样品。
根据本发明的同步鉴定水生生态系统浮游藻类和底栖藻类的方法,优选地,在所述步骤s3中,利用嵌套引物对进行两轮pcr扩增反应,所述嵌套引物对包括第一引物对和第二引物对。第一轮pcr扩增反应以所述纯化的dna作为模板,以所述第一引物对作为扩增引物,第二轮pcr扩增反应以所述第一轮pcr扩增反应的产物作为模板,以所述第二引物对作为扩增引物。
根据本发明的同步鉴定水生生态系统浮游藻类和底栖藻类的方法,优选地,在所述步骤s3中,所述第一引物对为a23srvf1:5’-ggacaraaagaccctatg-3’和a23srvr1:5’-agatcagcctgttatcc-3’,所述第二引物对为a23srvf2:5’-caraaagaccctatgmagct-3’和a23srvr2:5’-tcagcctgttatccctag-3’。
根据本发明的同步鉴定水生生态系统浮游藻类和底栖藻类的方法,优选地,在所述步骤s5中,建立所述藻类物种注释数据库的过程为:从美国国家生物技术信息中心(ncbi,网址是http://www.ncbi.nlm.nih.gov/)的数据库下载藻类23srdna序列数据,通过去除序列数据中的冗余序列、短于预定长度的序列和/或错误序列的方式优化序列数据后得到所述藻类物种注释数据库。
本发明的方法可以实现同步对水生生态系统的水体和沉积物中的原核藻类物种和真核藻类物种进行快速鉴定,能够为进一步研究水生生态系统中藻类的功能角色奠定了基础。
此外,利用本发明的方法鉴定水生生态系统中的藻类物种时,无需对藻类进行富集、分离和培养,不依赖于操作人员的专业技能,不受藻类细胞形态和细胞尺寸的制约,基于藻类物种间遗传物质的差异,可快速高效地鉴定大批量环境样品中的藻类物种,鉴定结果可重复性高、可标准化。
本发明的技术方案建立了完整的藻类物种注释数据库,与直接注释所有物种的ncbi数据库对比,利用本发明所建立的藻类物种注释数据库可以提高藻类的物种注释效果至30%~60%。
附图说明
为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍。
图1是本发明的同步鉴定水生生态系统中浮游藻类和底栖藻类的方法流程图。
图2是本发明实施例中以从4个监测站点的水样滤出物和沉积物样品中提取的dna为模板分别进行pcr扩增的产物的琼脂糖凝胶电泳结果图。
图3是对本发明实施例中4个监测站点的表层水样中浮游藻类和沉积物中底栖藻类物种组成进行鉴定的结果图。
具体实施方式
下面结合附图并通过具体实施方式来进一步描述本发明的技术方案,本发明的优点和特点将会随着描述而更为清楚。但是应当理解,实施例仅是示例性的,不对本发明的范围构成限制。本领域技术人员应该理解的是,在不偏离本发明的精神和范围下可以对本发明技术方案的细节和形式进行修改或替换,但这些修改和替换均落入本发明的保护范围内。
在下文的描述中,所涉及的方法如无特别说明,则均为本领域的常规方法。所涉及的原料如无特别说明,则均是能从公开商业途径获得的原料。
图1示出了本发明的基于高通量测序技术同步鉴定水生生态系统中浮游藻类和底栖藻类的方法的主要步骤。以下将对各个步骤进行详细的描述。
步骤s1,从待监测的水生生态系统的同一个水体环境中采集水样和沉积物样品。从该水体环境中采集5~10l表层水水样并混合均匀,同时采集表层水水样对应位置处的表层沉积物样品。
步骤s2,从表层水水样和沉积物样品中提取并纯化dna。其中,提取dna之前用0.22μm滤膜对表层水水样进行过滤并收集滤出物,所用的0.22μm滤膜优选是聚碳酸酯膜。可以使用任何合适的市售dna提取试剂盒从滤出物和沉积物样品中提取dna。
步骤s3,以纯化的dna作为模板进行pcr扩增。利用嵌套引物对进行巢式pcr扩增。嵌套引物对包括能通用于水生生态系统中存在的原核藻类和真核藻类的第一引物对和第二引物对。第一轮pcr扩增反应的模板为纯化的dna,第二轮pcr扩增反应以第一轮pcr扩增反应产物作为模板。为了便于描述,将第一轮pcr扩增反应所用的引物对称为第一引物对,第二轮pcr扩增反应所用的引物对称为第二引物对。
本发明的发明人经过大量的研究探索之后,优选地,选择能通用于原核藻类和真核藻类的两个引物对进行pcr扩增,第一引物对是a23srvf1:5’-ggacaraaagaccctatg-3’和a23srvr1:5’-agatcagcctgttatcc-3’,第二引物对是a23srvf2:5’-caraaagaccctatgmagct-3’和a23srvr2:5’-tcagcctgttatccctag-3’。
步骤s4,对pcr扩增得到的产物进行高通量测序,获取藻类的asv(ampliconsequencevariants,扩增子序列变体)代表序列。
步骤s5,建立藻类物种注释数据库。具体地,从ncbi(美国国家生物技术信息中心)数据库(http://www.ncbi.nlm.nih.gov/)下载藻类23srdna序列数据,通过去除冗余序列、短于预定长度的序列和/或错误序列等步骤优化所收集的藻类序列数据,从而建立得到完整的藻类物种注释数据库。
步骤s6,将步骤4获得的所有藻类asv代表序列与藻类物种注释数据库中的序列进行比对和物种注释,获取藻类物种组成信息。
实施例
本实施例以某饮用水水源地的水体为实例,使用本发明的方法对该水体中的浮游藻类和底栖藻类进行监测和鉴定。
1、样品采集和预处理
选取4个监测站点进行采样,将从4个站点所采的水样分别标记为w1~w4,对应的沉积物样品分别标记为s1~s4,对各样品中藻类群落的物种进行监测。
从每个监测站点分别采集表层水,混合得到5l表层水样,于4℃条件下冷藏运回实验室,24小时之内对采集到的所有水样用0.22μm聚碳酸酯膜(millipore,usa)过滤,将承载有滤出物的滤膜贮藏在-80℃冰箱中。
在每个监测站点同步采集表层水对应的水深约0.5m处的表层沉积物样品20ml,置于灭菌离心管中并用干冰保存运回实验室,贮藏在-80℃冰箱中。
2、提取dna
使用
总dna量超过100ng、浓度不低于10ng/ul的dna用于后续的pcr扩增。
3、pcr扩增藻类dna
采用第一引物对(即,上文描述的a23srvf1和a23srvr1)和第二引物对(即,上文描述的a23srvf2和a23srvr2),以上述提取的dna为模板进行巢式pcr扩增反应。
第一轮pcr扩增反应的模板为上述提取得到的dna,所使用的引物对为第一引物对。反应条件为:94℃起始变性温度持续2min;94℃20s,55℃30s,72℃30s,循环反应30次;延伸温度为72℃保持10min。
第二轮pcr扩增反应以第一轮pcr扩增反应的产物作为模板,使用的引物对为第二引物对。反应条件与第一轮pcr反应一致。
通过2%琼脂糖凝胶电泳对pcr产物进行检测,结果如图2所示。
用axyprepdna凝胶回收试剂盒(axygenbiosciences,usa)对pcr产物进行回收、纯化,并用quantifluortm-st荧光定量系统(promega,usa)对pcr产物的浓度进行定量检测,根据不同样本的测序量要求混合样本,以备后续测序使用。
4、藻类dna的高通量测序并生成藻类asv代表序列
将纯化后的pcr扩增产物用于illuminamiseq测序平台进行双端测序(2×300bp),获取所有样品中的dna序列。
测序结束后,使用flash软件对获得的双端序列进行拼接,使用fastp软件按照以下标准对dna序列进行质控优化:(1)以50bp的dna序列片段为移动窗口,当移动窗口内的序列平均序列质量正确率低于99.9%,则截去窗口末端的碱基,当剪切后的dna片段小于50bp时,则剔除该序列片段;(2)根据双端测序的重叠部分将成对的序列进行拼接,其中重合碱基长度要求高于10bp,剔除无法拼接的序列;(3)匹配引物的最大错配为2个核苷酸,任何带有歧义碱基的序列均被剔除。
使用qiime2平台中的dada2方法参照默认参数对优化序列进行序列降噪处理,获得藻类asv代表序列和丰度信息。
5、藻类asv序列的物种注释
采用classify-consensus-blast将上述获得的藻类asv代表序列与藻类物种注释数据库中的序列进行比对和物种注释,确定所有样品中藻类群落的物种组成。
结果如图3所示,表层水水样和沉积物中共检测到藻类12门24纲57目90科139属,其中绿藻门(chlorophyta)、蓝藻门(cyanobacteria)、金藻门(chrysophyta)以及硅藻门(bacillariophyta)的物种最多,分别有45、40、16、12种。在所有检出的藻类物种中,蓝藻的比例最高,其丰度占底栖藻类的75.26%,占浮游藻类的58.16%;其次为金藻,其丰度占底栖藻类的18.74%,占浮游藻类的21.04%。
应该理解,尽管参照上述实施例,已经对本发明进行了具体的描述,但并非对本发明实施方式的限定。对于所属领域的普通技术人员,在不背离权利要求书所定义的范围条件下,可以在上述说明的基础上进行各种形式和细节的变化或变动。这里无需也无法对所有的实施方式予以穷举。因此,落在本发明权利要求的等同要件的含义和范围内的所有变化或变动,均处于本发明创造的保护范围之中。
- 还没有人留言评论。精彩留言会获得点赞!