一种运行于分布式系统的海量测序数据错误修正方法与流程
本发明涉及生物基因技术与计算机科学技术相关的交叉学科领域,尤其涉及一种运行于分布式系统的海量测序数据错误修正方法。
背景技术:
下一代高通量测序(next generation sequencing,NGS,中文名又常称为二代测序或新一代测序)技术让全基因组分析和个性化基因医疗成为可能。下一代测序技术跟传统的 Sanger 测序比起来,有着速度快,费用少的特点,但是他们的缺点是在测序中出现大量的短序列数据及其携带的错误。由于实验技术的局限,这些短序列不可避免会存在一些错误,如果在序列拼接之前没有对这些错误进行修正,算法根据这些错误数据进行拼接,将会降低最终序列的质量。在短序列数据被拼接成为长序列(contig)之前,修复短序列数据是非常重要的一个步骤,是重组可靠长序列的前提和保证。
测序数据中产生的误差一直是一个困扰序列质量以及后续分析的重要问题, 下一代测序中的错误率与碱基质量有关,受测序仪本身、测序试剂、样品等多个因素共同影响。测序误差不但会干扰测序数据正常拼接,而且还无法正确识别样本中存在的遗传信息多态性,难以得到有价值的结果。由于测序实验过程比较复杂,每个过程中存在很多不可控制的随机因素,纯粹依靠实验技术的规范和改进是难以彻底消除测序错误。
下一代测序技术把整条待测序列分解成为短片段(read,被称为读段),对每个短片读段进行多次重复测量。所有的纠错方法都遵循这样一个前提条件:测序出来大多数的读段序列是正确的,只有少数的序列有错误存在。举例来说,在修正错误时,如果有M条相同的序列A,N条相同的序列B,序列A和序列B在规定的Hamming distance的阈值(y)范围内,在这种情况下,一般就认为序列A和序列B是从原始待测序列的同一个区域而来,此时判断数值M和N的大小,数量多的序列就被认为是正确的,数量少的序列则会被修正(依据数量多的序列)。
目前使用的纠错方法主要有以下三种:(1)基于k-spectrum的方法。(2)基于suffix tree/suffix array的方法。(3)基于multiple sequence alignment (MSA)的方法。
现有的错误修正算法计算复杂度高,执行效率低,对计算资源的要求很高,不适合应用在海量测序数据的环境。在处理大量数据时,需要大量内存以及很长的运行时间,特别是在全序列测序产生海量数据的环境下,一般的服务器将无法提供足够的内存和计算能力,需要超级计算机才能够处理。
技术实现要素:
本发明的目的在于克服现有技术的不足,提供一种运行于分布式系统的海量测序数据错误修正方法。
本发明采用的技术方案是:
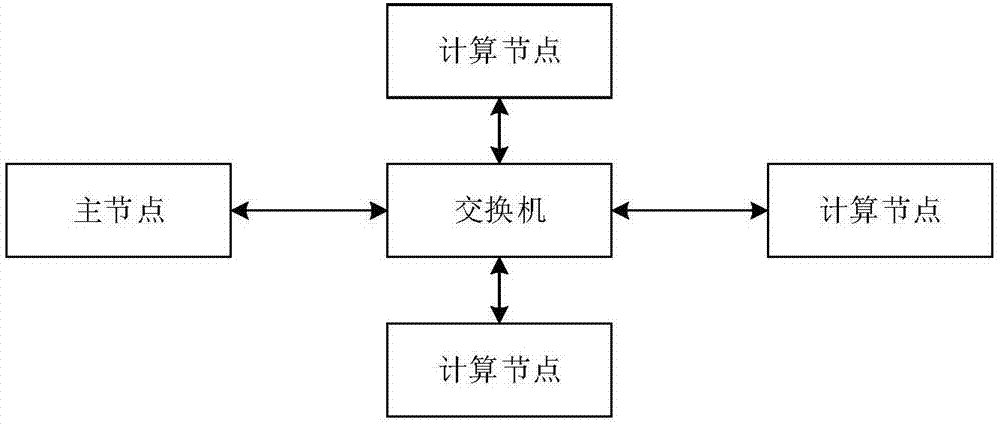
一种运行于分布式系统的海量测序数据错误修正方法,所述分布式系统包括主节点、交换机和若干计算节点,若干计算节点通过交换机连接主节点,所述海量测序数据错误修正方法包括以下步骤:
1)对测序数据进行预处理,确定测序数据的分组标准;
2)对测序数据进行分区处理,平衡分布式系统各个计算节点的负载并传送测序数据到计算节点;
3)对测序数据进行分布式错误修正。
步骤1中确定测序数据的分组标准具体包括以下步骤:
1-1,数据抽样过程:根据要处理的测序数据的特征进行数据抽样,确保抽样测序数据具有一定的代表性;
1-2,抽样测序数据聚类过程:应用序列相似性算法计算抽样测序数据各个短序列之间的相似性,应用统计方法将所述抽样测序数据分别聚为相近的类;
1-3,各类数据特征提取过程:将组成各类的测序数据进行组合和计算,提取特征用于快速判别短序列与该类之间的距离。
步骤2中平衡分布式系统各个计算节点的负载包括以下步骤:
2-1,确定测序数据与每一个样本聚类之间的距离,并根据计算的距离,计算每一个测序数据的归属聚类;
2-2,根据每一个聚类所拥有的测序数据数量,进行平衡负载计算;
2-3,根据平衡负载情况,分配聚类到指定计算节点;
2-4,将测序数据传送到对应的计算节点。
步骤2-2中根据聚类测序数据数量获取聚类标准数据量,聚类标准数据量=min(各类测序数据数量差),每一个聚类对应标准数据量=该类聚类测序数据数量/标准数据量。小数部分向上取整。
步骤2-3中根据聚类标准数据量和计算节点的标准计算能力,进行负载平衡计算,确定聚类数据对应的计算节点,并满足1个计算节点处理1个或多个聚类对应数据,且一个聚类只能分配到一个计算节点进行计算。
步骤2-4中测序数据传送时采用压缩算法将测序数据分别进行压缩,并加入校验码。
步骤2中在数据传送到各个计算节点之前,先对数据进行预先处理,并判别测序数据应传送到哪个节点。
步骤3中对测序数据进行分布式错误修正包括以下步骤:
3-1,各个计算节点中应用错误修正算法对测序数据进行处理,计算评分;
3-2,集成判定计算,将评分数据进行汇总,根据各个计算节点评分确定错误修正方案。
步骤3-1中计算节点接收到测序数据后,运行HiTEC错误修正算法对本节点测序数据进行错误识别处理,计算每一个短序列的错误可能性以及错误位置,并计算错误修正评分。
步骤3-2中各个计算节点将序列编号作为关键值,将评分数据作为计算值,应用哈希函数再次分布到各个计算节点,同一序列的三个错误修正评分都会分布到同一个计算节点,应用选举算法算出该序列的错误修正方案,汇总确定的错误修正方案作为错误修正的结果返回。
本发明采用以上技术方案,充分应用分布式计算平台的性能,提出基于分布式环境的海量生物二代测序错误修正的解决方法。在此方法中,充分考虑测序数据的分布的合理性,以及负载平衡对分布式计算平台性能的影响,通过应用抽样聚类的方式确定测序数据的中心,通过与中心的比较,确定测序数据具体归属。应用单位分割方法,计算单位节点计算能力与单位聚类数据量,以此为负载平衡计算依据,设计负载平衡方法。通过分布测序数据,每个计算节点应用错误修正算法对本节点上数据进行错误以及修正评分,由于每个节点所需要处理的数据量大量减少,显著提高整个系统的错误修正处理效率。最后将评分进行汇总,选举出评分最高的作为错误修正方案。
本发明基于分布式环境的海量测序数据错误修正解决方案,为生物信息学提供切实可用的测序数据错误修正工具,而且为其他海量数据应用解决方案提供新的思路,从而丰富分布式计算的研究内容,推动生物信息学和高性能并行计算的研究和发展。本发明所述方法与集中式系统相比,在处理海量测序数据方面具有速度快,精度高,以及成本低等优势。
附图说明
以下结合附图和具体实施方式对本发明做进一步详细说明;
图1为本发明的分布式系统结构示意图;
图2为本发明一种运行于分布式系统的海量测序数据错误修正方法的流程示意图。
具体实施方式
如图1和图2所示,本发明公开一种运行于分布式系统的海量测序数据错误修正方法,所述分布式系统包括主节点、交换机和若干计算节点,若干计算节点通过交换机连接主节点,每个计算节点可以是PC服务器或者PC台式机,对硬件环境的要求不高,由于可以进行负载平衡,也不要求所有计算节点的配置统一。
所述海量测序数据错误修正方法包括以下步骤:
1)对测序数据进行预处理,确定测序数据的分组标准;
2)对测序数据进行分区处理,平衡分布式系统各个计算节点的负载并传送测序数据到计算节点;
3)对测序数据进行分布式错误修正。
步骤1中确定测序数据的分组标准具体包括以下步骤:
1-1,数据抽样过程:根据要处理的测序数据的特征进行数据抽样,确保抽样测序数据具有一定的代表性;
对要处理的测序数据进行随机抽样,抽样数据量为N/m3,其中N为序列总数,m为计算节点的数量,至少约为1000-3000条。然后采用蒙特卡洛模拟方法对抽样数据进行模拟,最后得到1000-2000条模拟的测序数据集,该数据集能够表示测序数据的整体特征。
1-2,抽样测序数据聚类过程:应用序列相似性算法计算抽样测序数据各个短序列之间的相似性,应用统计方法将所述抽样测序数据分别聚为相近的类;
计算抽样数据集各个短序列之间的汉明距离。然后,应用层次聚类算法,根据汉明距离将这些抽样测序数据分别聚为相近的类。聚类原则有两种,第一种是汉明距离在5以内的聚为同一类,如果同一类的短序列少于3个,说明该类样本过少,取消该类。第二种聚类原则是设定聚类个数n,一般n>6m,应用层次聚类算法将样本数据聚为n类。
1-3,各类数据特征提取过程:将组成各类的测序数据进行组合和计算,提取特征用于快速判别短序列与该类之间的距离。
将各类的样本测序数据分别组成一个连接的长序列,其中类中的短序列按照顺序与下一个序列连接,两个序列之间用$符号分割.应用概率后缀数组算法将每一类组成的长序列构建成一个后缀数组,并计算每一个节点分支概率.该步骤将输出一个数据结构,其中记录该类长序列构成的后缀数组和分支概率,这就是每一类样本测序数据的特征。
对于提取数据特征方法,将类中各个序列进行线性组合,根据出现的频度以及概率确定子序列的权重.
其中,概率后缀数组是基于传统的后缀数组的一种VLMMs实现。与后缀数组一样,PSA可以表示所有N(N + 1)/ 2个从根到叶的子串。基于PSA模型实现的可变长度马尔可夫模型(VLMMs),其每一个节点的字符串的深度表示着子串的长度。通过限制各自的叶节点深度,可以表示相同的字符串的长度,也就可以表示一个给定序列下出现某个状态的条件概率,这就是转移矩阵中的转移概率。转移概率是一个通过给定的路径的符号与所观察到的数据前面的子串路径符号计算而来的相对频率。由子字符串的长度确定的条件概率可以通过PSA模型中的确定的一条路径计算得到。由于后缀数组采用标记起始终止位置的方式来记录每一个节点的字符串,而后缀数组拥有N个节点,因此,采用后缀数组可以在线性的空间中表示所有N(N + 1)/ 2个从根到叶的子串。这样就可以将一个具有N(N + 1)/ 2个转移概率的转移矩阵用一个N个节点的线性数据结构来概率后缀数组表示。
步骤2中平衡分布式系统各个计算节点的负载包括以下步骤:
2-1,确定测序数据与每一个样本聚类之间的距离,并根据计算的距离,计算每一个测序数据的归属聚类;
对于给定一个测序数据s,与样本中每一类中提取的概率后缀数组进行比较计算相似度,具体做法是:从根节点出发,访问PSA中的节点,匹配到相应的节点,并根据该节点的转移概率计算s与该概率后缀数组之间的匹配概率。然后根据匹配概率确定与该测序数据s最相近的3个类为归属聚类。
2-2,根据每一个聚类所拥有的测序数据数量,进行平衡负载计算;根据计算节点的数量和处理能力,计算出标准计算能力,每个节点的计算能力为标准计算能力的整数倍数,至少为1倍。根据聚类测序数据数量,计算出聚类标准数据量,每一个聚类的数量为标准数据量的整数倍数,至少为1倍。
步骤2-2中根据聚类测序数据数量获取聚类标准数据量,聚类标准数据量=min(各类测序数据数量差),每一个聚类对应标准数据量=该类聚类测序数据数量/标准数据量。小数部分向上取整。
2-3,根据平衡负载情况,分配聚类到指定计算节点;
步骤2-3中根据聚类标准数据量和计算节点的标准计算能力,进行负载平衡计算,确定聚类数据对应的计算节点,并满足1个计算节点处理1个或多个聚类对应数据,且一个聚类只能分配到一个计算节点进行计算。其中计算方法为:先计算一个标准计算能力与标准数据量之间的比例,应用背包问题的解决方法计算分配方式。
2-4,将测序数据传送到对应的计算节点。根据上述负载平衡方法,将测序数据传送到对应的计算节点。数据传送到各个计算节点之前,先对数据进行预先处理,并判别测序数据应传送到哪个节点。测序数据传送时采用压缩算法将测序数据分别进行压缩,并加入校验码,以确保压缩包在传输过程中不存在丢失的情况。应用ftp程序将测序数据压缩包传送到对应的计算节点,计算节点接收到传送过来的数据,先进行校验,确认没有传输错误后,将压缩包解压到工作目录,完成接收数据工作。如果发现传输错误,发出重传数据请求,让主节点再次传输该节点的压缩数据包。
步骤3中对测序数据进行分布式错误修正包括以下步骤:
3-1,各个计算节点中应用错误修正算法对测序数据进行处理,计算评分;根据负载平衡方案分布数据后,假设每个计算节点的处理能力相同,每个计算节点要处理的数据量约为3N/m,其中N为测序数据总量,m为节点数量。当结算数量m比较大(如m=50)时,每个节点要处理的数据量显著减少,此时调用常规的测序数据错误修正算法对测序数据进行处理,根据处理结果,返回错误修正评分。
例如,步骤3-1中计算节点接收到测序数据后,运行HiTEC错误修正算法对本节点测序数据进行错误识别处理,计算每一个短序列的错误可能性以及错误位置,并计算错误修正评分。
3-2,集成判定计算,将评分数据进行汇总,根据各个计算节点评分确定错误修正方案。
步骤3-2中各个计算节点将序列编号作为关键值,将评分数据作为计算值,应用哈希函数再次分布到各个计算节点,同一序列的三个错误修正评分都会分布到同一个计算节点,应用选举算法算出该序列的错误修正方案,汇总确定的错误修正方案作为错误修正的结果返回。
本发明采用以上技术方案,充分应用分布式计算平台的性能,提出基于分布式环境的海量生物二代测序错误修正的解决方法。在此方法中,充分考虑测序数据的分布的合理性,以及负载平衡对分布式计算平台性能的影响,通过应用抽样聚类的方式确定测序数据的中心,通过与中心的比较,确定测序数据具体归属。应用单位分割方法,计算单位节点计算能力与单位聚类数据量,以此为负载平衡计算依据,设计负载平衡方法。通过分布测序数据,每个计算节点应用错误修正算法对本节点上数据进行错误以及修正评分,由于每个节点所需要处理的数据量大量减少,显著提高整个系统的错误修正处理效率。最后将评分进行汇总,选举出评分最高的作为错误修正方案。
本发明基于分布式环境的海量测序数据错误修正解决方案,为生物信息学提供切实可用的测序数据错误修正工具,而且为其他海量数据应用解决方案提供新的思路,从而丰富分布式计算的研究内容,推动生物信息学和高性能并行计算的研究和发展。本发明所述方法与集中式系统相比,在处理海量测序数据方面具有速度快,精度高,以及成本低等优势。
- 还没有人留言评论。精彩留言会获得点赞!