用于鉴定单体型的装置和方法与流程
更具体地,本发明涉及用于鉴定多个样本核苷酸序列中的单体型的装置和方法。
背景技术:
在现代生物学和医学中,有许多遗传任务需要完成,例如鉴定遗传性疾病或调查不同物种种群的基因组变异。这些任务需要鉴定单体型,即倾向于一起遗传的等位基因组。尽管单体型分析很重要,但是由于该过程的持续时间很长,而且其计算费用高昂,基本上限制了单体型在医学实践和科学研究中的应用。
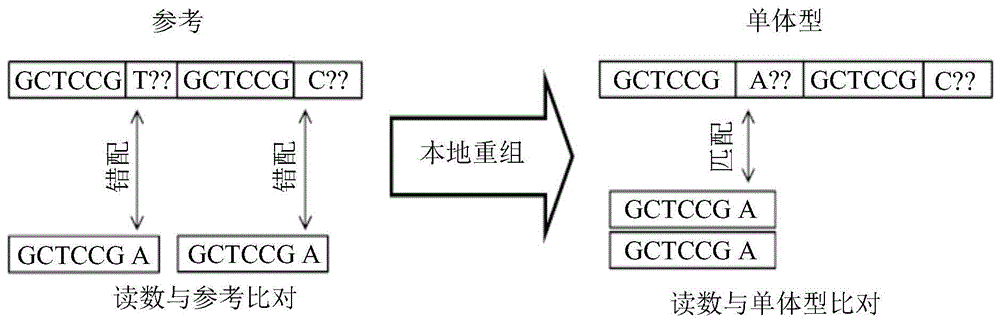
通常,对核苷酸序列进行单体型鉴定,将核苷酸序列映射到参考序列的各区域,其中核苷酸顺应性的概率最大(见图1)。在该映射的基础上,通过从头单体型组装选择这些区域进行单体型分析,所述从头单体型组装不考虑序列的映射并且在所选区域内执行。从头重组方法大大增加了单体型分析的计算复杂性和时间,但是由于基因组中核苷酸序列的重复率高,仍值得采用这种重组方法。从图1中可以看出,如果所述参考具有重复序列,则某一序列可在单体型组装后改变其比对的位置。
因此,如果序列中的大多数核苷酸与所述参考中的重复子序列匹配而其他核苷酸不匹配,则可以进行序列重定位。显然,所述参考中的重复子序列越短,所述序列中其他核苷酸与所述参考不匹配的可能性就越小。
用于单体型分析的区域通常非常短,例如长度为100至500个核苷酸。考虑到该范围的上限,即500个核苷酸,并考虑到由大约3x109个碱基对组成的人类核基因组,令人注意的是,如果我们否认(用于重组)某一序列恰好位于其当前比对中,则所述序列属于当前区域的概率小于10-6(500除以3x109)。从这个角度来看,区域内的重组是没有意义的。
考虑到重组100个核苷酸的序列,其中随机分布着四种不同类型的核苷酸,即使所述序列与参考序列相比包含许多错配(例如,20个),那么为该序列找到具有相同或更好的核苷酸顺应性的另一个比对的概率也小于约10-47(4-(100-20)x500)。这意味着没有重组的单体型分析约每1037基因组(1037≈1/(1037≈1/(3x109x10-47))产生1个错误。随着参考中可重复性的增加,序列重比对的概率上升。因此,必须有一种快速鉴定重复超载区域的方法,其中重组是合理且实用的。
可以理解的是,单体型分析的速度会更高,而且不采用序列重组时的质量不差于采用序列重组。然而,以往的工作并没有提供任何有效的方法用于在不重组的情况下进行多基因组单体型分析。因此,需要一种不重组的单体型分析方法,其中所述方法可以使用序列的当前比对信息,以便快速有效地将这些序列聚合成单体型。同样,还需要一种用于快速鉴定重组有意义的区域的方法。
最新和最有效的单体型分析方法之一是单个个体单体型分析的混合模型(mixturemodelforsingleindividualhaplotyping,简称mixsih),所述模式用二进制表示两种单体型,如matsumotoh.和kiryuh.于2013年bmc基因组学14,s5发表的“mixsih:单个个体单体型分析的混合模型”中所述。基于二进制模型和“最小连接性”得分,所述模式可以准确测量单体型一致性。采用这种方法,mixsih在以下步骤中提取高度准确的单体型片段,如图2所示。
所述mixsih方法首先选择不同的核苷酸,即提取等位基因(步骤1)。为了改善性能,随后将等位基因转变成二进制格式(步骤2)。在步骤3中,使用提议的概率函数选择最可能的等位基因。最后,基于步骤4中的连接性得分选择单体型,所述步骤4中包括图3中所示的子步骤。
然而,现有技术的mixsih方法具有如下若干关键问题:mixsih方法只执行单个个体单体型分析,无法应用于多个基因组;所述mixsih方法专门执行单个个体单体型分析,因此无法产生两个以上的单体型;所述mixsih方法在单体型推断过程中使用复杂的公式,因此无法提供最佳性能;所述mixsih方法不支持从头单体型组装,并且在重复率高的区域中可能丧失单体型的质量;所述mixsih方法未考虑核苷酸鉴定的phred质量,因此无法产生具有最佳精确度的结果。
鉴于上述情况,需要一种改进的装置和方法,提供高效率和高精度的单体型分析,允许对多个基因组采用单体型分析,并且实现产生多于两种单体型的多单体型分析。
技术实现要素:
本发明的目的在于提供一种改进的装置和方法,以保证提供高效率和高精度的单体型分析,允许对多个基因组采用单体型分析,并且实现产生两种以上单体型的多单体型分析。
上述和其它目的通过独立权利要求的主题来实现。据从属权利要求、说明书以及附图,进一步的实现形式是显而易见的。
通常,本发明涉及用于鉴定多个样本核苷酸序列中的单体型的装置和方法。更具体地,提供了一种新颖的装置和方法,用于在核苷酸子序列重复率低的区域中重叠单体型分析,以应对常规单体型分析方法的缺点。与现有技术相比,本发明具有几个显着的优点:首先,本发明提供了一种鉴定含多个基因组的样本中的单体型的方法。与现有方案相比,该方法可以考虑所有可用的等位基因及其可能的组合。其次,本发明开发了一种选择预期的单体型数量的方法。与现有方案相比,该方法可以考虑单体型分析的不同步骤中预期的单体型数量。第三,本发明提供了一种有效聚合单体型的方法,与现有方案相比,通过支持最简单的单体型分析方法可以提高性能。第四,本发明提供了一种通过使用所有可用信息来最精确地产生结果的方法,用于巧妙地组装单体型。最后,本发明提供了一种对具有不同重复率的区域采用适当的组装程序的方法。
更具体地,根据第一方面,提供了一种用于基于参考核苷酸序列鉴定多个样本核苷酸序列中的单体型的装置,其中所述装置包括处理单元,所述处理单元用于:通过基于所述参考核苷酸序列从多个样本核苷酸序列中提取多个等位基因序列来产生初始等位基因序列组,其中所述多个等位基因序列中的每个等位基因序列的每个等位基因(用缺失、插入或单核苷酸多态性表示)与所述参考核苷酸序列中的核苷酸位点相关联;通过将来自所述初始等位基因序列组的在重叠序列部分具有相同等位基因并且属于相同单体型的那些等位基因序列组组合成聚合等位基因序列,基于所述初始等位基因序列组产生第一聚合等位基因序列组,其中第一聚合等位基因序列组包含所述聚合等位基因序列和来自所述初始等位基因序列组的未组合成聚合等位基因序列的等位基因序列;通过连接来自第一聚合等位基因序列组的相邻等位基因序列对,基于第一聚合等位基因序列组产生第二聚合等位基因序列组,其中相邻等位基因序列包含相邻核苷酸位点中的等位基因,但没有重叠等位基因;基于第二聚合等位基因序列组鉴定所述多个样本核苷酸序列中的单体型。
因此,提供了一种用于鉴定单体型的改进装置,允许对多个基因组进行单体型分析,以便高效率和高精度地提供单体型分析的结果,并且能够产生多于两种单体型的多单体型分析。
在第一方面的另一种可能实现方式中,所述处理单元还用于通过从所述初始等位基因序列组中去除偶然变体来过滤所述初始等位基因序列组。
在第一方面的另一种可能实现方式中,所述处理单元用于通过从所述初始等位基因序列组中去除偶然变体来过滤所述初始等位基因序列组,方法是从所述初始等位基因序列组中去除出现频率低于过滤阈值的那些等位基因序列,其中所述出现频率表示等位基因序列在所述初始等位基因序列组中的重复次数。
在第一方面的另一种可能实现方式中,所述处理单元还用于从第一聚合等位基因序列组中去除等位基因序列,所述等位基因序列是第一聚合等位基因序列组中的至少一个其他等位基因序列的各部分。
在第一方面的另一种可能实现方式中,所述处理单元用于通过从所述多个样本核苷酸序列中提取等位基因序列来产生所述初始等位基因序列组,所述等位基因序列具有至少一个核苷酸不匹配相应核苷酸位点处的参考核苷酸序列的相应核苷酸。
在第一方面的另一种可能实现方式中,所述处理单元用于将来自所述初始等位基因序列组的具有重叠等位基因部分的那些等位基因序列组合成聚合序列,其中所述聚合序列包含来自等位基因序列的重叠序列部分和非重叠等位基因,所述等位基因序列按照与等位基因相关的核苷酸位点顺序排序,即根据这些等位基因序列中的每个等位基因序列与参考核苷酸序列的比对排序。
在第一方面的另一种可能实现方式中,如果第二聚合等位基因序列组的等位基因序列数量大于预期值,则所述处理单元还用于通过基于统计学方法计算第二聚合等位基因序列组的每个等位基因序列的概率测量来鉴定所述多个样本核苷酸序列中的单体型,其中所述概率测量通过基于概率测度鉴定第二聚合等位基因序列组中的单体型指示等位基因序列属于单体型的概率。
在第一方面的另一可能实现方式中,所述统计方法包括基于隐马尔可夫模型(hiddenmarkovmodel,简称hmm)的贝叶斯方法。
在第一方面的另一种可能实现方式中,所述处理单元还用于在重复次数低于重复阈值的情况下,确定所述参考核苷酸序列中的重复次数并基于所述参考核苷酸序列鉴定多个样本核苷酸序列中的单体型。
在第一方面的另一种可能实现方式中,所述处理单元还用于:
(i)如果所述参考核苷酸序列具有下一个核苷酸符号,则基于所述下一个核苷酸符号产生哈希码;
(ii)如果所述生成的哈希码已经是一组生成的哈希码的一部分,则增加计数器值,或者,如果所述生成的哈希码不是所述一组生成的哈希码的一部分,则将所述生成的哈希码添加到所述一组生成的哈希码;
(iii)只要所述计数器值小于预定义的阈值计数器值,则重复(i)和(ii);
(iv)如果所述计数器值小于所述预定阈值计数器值,基于所述参考核苷酸序列鉴定所述多个样本核苷酸序列中的单体型。
在第一方面的另一种可能实现方式中,所述处理单元用于基于所述下一个核苷酸符号通过以下方式生成所述哈希码:
将所述核苷酸符号(a、c、g或t)替换为用两位表示的唯一序列;
将所述哈希码的当前值向左移2位;
对所述移位的哈希码和相应用两位表示的唯一序列采用按位or运算;
对所述按位or运算的结果采用二进制掩码,其中所述二进制掩码的前两位为0,所述二进制掩码的其余位为1。
根据第二方面,本发明涉及一种用于基于参考核苷酸序列鉴定多个样本核苷酸序列中的单体型的方法。所述方法包括:通过基于参考核苷酸序列从多个样本核苷酸序列中提取多个等位基因序列来产生初始等位基因序列组,其中所述多个等位基因序列中的每个等位基因序列中的每个等位基因与所述参考核苷酸序列中的核苷酸位点相关联;通过将来自所述初始等位基因序列组的在重叠序列部分具有相同等位基因并且属于相同单体型的那些等位基因序列组组合成聚合等位基因序列,基于所述初始等位基因序列组产生第一聚合等位基因序列组,其中第一聚合等位基因序列组包含所述聚合等位基因序列和来自所述初始等位基因序列组的未组合成聚合等位基因序列的等位基因序列;通过连接来自第一聚合等位基因序列组的相邻等位基因序列对,基于第一聚合等位基因序列组产生第二聚合等位基因序列组,其中相邻等位基因序列包含相邻核苷酸位点中的等位基因,但没有重叠等位基因;基于第二聚合等位基因序列组鉴定所述多个样本核苷酸序列中的单体型。
因此,提供了一种用于鉴定单体型的改进方法,允许对多个基因组进行单体型分析,以便高效率和高精度地提供单体型分析的结果,并且能够产生多于两种单体型的多单体型分析。
在第二方面的另一种可能实现方式中,所述方法还包括通过从所述初始等位基因序列组中去除偶然变体来过滤所述初始等位基因序列组。
在第二方面的另一种可能实现方式中,通过从所述初始等位基因序列组中去除所述偶然变体来过滤所述初始等位基因序列组的步骤包括从所述初始等位基因序列组中去除出现频率低于过滤阈值的那些等位基因序列,其中所述出现频率表示等位基因序列在所述初始的等位基因序列组中的重复次数。
在第二方面的另一种可能实现方式中,所述方法还包括从第一聚合等位基因序列组中去除等位基因序列,所述等位基因序列是第一聚合等位基因序列组中的至少一个其他等位基因序列的各部分。
在第二方面的另一种可能实现方式中,产生所述初始等位基因序列组的步骤包括从所述多个样本核苷酸序列中提取等位基因序列,所述等位基因序列具有至少一个核苷酸不匹配相应核苷酸位点处的参考核苷酸序列的相应核苷酸。
在第二方面的另一种可能实现方式中,组合步骤包括将来自所述初始等位基因序列组的具有重叠等位基因部分的那些等位基因序列组合成聚合序列,其中所述聚合序列包含来自等位基因序列的重叠序列部分和非重叠等位基因,所述等位基因序列按照与等位基因相关的核苷酸位点顺序排序,即根据这些等位基因序列中的每个等位基因序列与参考核苷酸序列的比对排序。
在第二方面的另一种可能实现方式中,如果第二聚合等位基因序列组的等位基因序列数量大于预期值,则鉴定所述多个样本核苷酸序列中的单体型的步骤包括基于统计学方法计算第二聚合等位基因序列组的每个等位基因序列的概率测量,其中所述概率测量通过基于概率测度鉴定第二聚合等位基因序列组中的单体型指示等位基因序列属于单体型的概率。
在第二方面的另一可能实现方式中,所述方法还包括以下步骤:
(i)如果所述参考核苷酸序列具有下一个核苷酸符号,则基于所述下一个核苷酸符号产生哈希码;
(ii)如果所述生成的哈希码已经是一组生成的哈希码的一部分,则增加计数器值,或者,如果所述生成的哈希码不是所述一组生成的哈希码的一部分,则将所述生成的哈希码添加到所述一组生成的哈希码;
(iii)只要所述计数器值小于预定义的阈值计数器值,则重复步骤(i)和(ii);
(iv)如果所述计数器值小于所述预定阈值计数器值,基于所述参考核苷酸序列鉴定所述多个样本核苷酸序列中的单体型。
在第二方面的另一种可能实现方式中,基于所述下一个核苷酸符号产生所述哈希码的步骤包括:将所述核苷酸符号(a、c、g或t)替换为用两位表示的唯一序列;将所述哈希码的当前值向左移2位;对所述移位的哈希码和相应用两位表示的唯一序列采用按位or运算;对所述按位or运算的结果采用二进制掩码,其中所述二进制掩码的前两位为0,所述二进制掩码的其余位为1。
根据第三方面,本发明涉及一种计算机程序,包括:程序代码,用于在计算机或处理器上运行时执行根据第二方面所述的方法。
本发明可以硬件和/或软件的方式来实现。
附图说明
本发明的具体实施例将结合以下附图进行描述,其中:
图1示出了所述参考/单体型序列上核苷酸序列的局部重组的示意图;
图2示出了用于单个个体单体型分析的混合物模型的示意图;
图3示出了用于单个个体单体型分析的混合物模型中的等位基因序列选择的示意图;
图4示出了根据实施例的用于鉴定单体型的装置的示意图;
图5示出了根据实施例鉴定单体型的相应方法的示意图;
图6示出了根据实施例的装置中实现的用于单体型分析的方法的示意图;
图7示出了根据实施例的装置中实现的用于单体型分析的方法的不同阶段的示意图;
图8a-8c示出了鉴定本发明实施例中实现的单体型的示意图;
图9示出了根据实施例的装置中实现的单体型分析的自适应策略的示意图;
图10示出了根据实施例的装置中实现的生成核苷酸序列唯一哈希码的示图;
图11示出了根据实施例的装置中实现的基因组分析工具包中修改后管道的示意图;
图12示出了本发明实施例中实现的debruijn图重组(debruijngraphreassembling,简称dbgr)和重叠组件(overlappingassembly,简称oa)的结果表;
图13示出了本发明实施例中实现的debruijn图重组(debruijngraphreassembling,简称dbgr)和重叠组件(overlappingassembly,简称oa)生成的单体型(染色体4:190610-190645kb)的示意图;
图14a-14b示出了本发明实施例中实现的单体型分析方法与常规debruijn图方法之间的精确度和执行时间的比较的示意图。
在各附图中,相同的或至少功能等同的特征使用相同的标号。
具体实施方式
以下结合附图进行描述,所述附图是本发明的一部分,并通过图解说明的方式示出可以实施本发明的具体方面。可以理解的是,在不脱离本发明范围的情况下,可以利用其它方面,并做出结构或逻辑上的改变。因此,以下详细的描述并不当作限定,本发明的范围由所附权利要求书界定。
例如,可以理解的是,与所描述方法有关的公开对于用于执行所述方法的相应设备或系统也同样适用,反之亦然。例如,如果描述了特定方法步骤,则对应设备可以包括用于执行所描述的方法步骤的单元,即使此类单元没有在图中明确描述或图示。
另外,在以下具体描述以及权利要求中,描述了包含相互连接或进行信号交互的不同功能方框或处理单元的实施例。可以理解的是,本发明也涵盖了包含附加功能方框或处理单元的实施例,这些附加功能方框或处理单元设置在以下所述的实施例中的功能方框或处理单元之间。
最后,可以理解的是,除非另有说明,此处所述的各种示例性方面的特征可以互相结合。
图4示出了用于基于参考核苷酸序列鉴定多个样本核苷酸序列中的单体型的装置400的示意图。如下文的进一步详细描述,装置400包括处理单元401,处理单元401用于:通过基于所述参考核苷酸序列从多个样本核苷酸序列中提取多个等位基因序列来产生初始等位基因序列组,其中所述多个等位基因序列中的每个等位基因序列中的每个等位基因(由缺失、插入或单核苷酸多态性表示)与所述参考核苷酸序列中的核苷酸位点相关联;通过将来自所述初始等位基因序列组的在重叠序列部分具有相同等位基因并且属于相同单体型的那些等位基因序列组组合成聚合等位基因序列,基于所述初始等位基因序列组产生第一聚合等位基因序列组,其中第一聚合等位基因序列组包含所述聚合等位基因序列和来自所述初始等位基因序列组的未组合成聚合等位基因序列的等位基因序列;通过连接来自第一聚合等位基因序列组的相邻等位基因序列对,基于第一聚合等位基因序列组产生第二聚合等位基因序列组,其中相邻等位基因序列包含相邻核苷酸位点中的等位基因,但没有重叠等位基因;基于第二聚合等位基因序列组鉴定所述多个样本核苷酸序列中的单体型。
图5示出了用于基于参考核苷酸序列鉴定多个样本核苷酸序列中的单体型的相应方法500的步骤。方法500包括以下步骤:用于通过基于所述参考核苷酸序列从多个样本核苷酸序列中提取多个等位基因序列来产生(501)初始等位基因序列组,其中所述多个等位基因序列中的每个等位基因序列中的每个等位基因(由缺失、插入或单核苷酸多态性表示)与所述参考核苷酸序列中的核苷酸位点相关联;通过将来自所述初始等位基因序列组的在重叠序列部分具有相同等位基因并且属于相同单体型的那些等位基因序列组组合成聚合等位基因序列,基于所述初始等位基因序列组产生(503)第一聚合等位基因序列组,其中第一聚合等位基因序列组包含所述聚合等位基因序列和来自所述初始等位基因序列组的未组合成聚合等位基因序列的等位基因序列;通过连接来自第一聚合等位基因序列组的相邻等位基因序列对,基于第一聚合等位基因序列组产生(505)第二聚合等位基因序列组,其中相邻等位基因序列包含相邻核苷酸位点中的等位基因,但没有重叠等位基因;基于第二聚合等位基因序列组鉴定(507)所述多个样本核苷酸序列中的单体型。
下面将描述图4中示出的装置400和图5中示出的方法500的其他实施例、实现方式和细节,其中方法500也将被称为重叠单体型分析。
方法500(以及相应的装置400)的另一实施例在图6中示为重叠单体型分析方法600。重叠单体型分析方法600包括以下主要步骤:601从核苷酸符号序列中提取等位基因序列;603使用预定义的过滤阈值从所述等位基因序列中滤去稀有等位基因;605在重叠中聚合具有相同等位基因的等位基因序列;607去除属于其他等位基因序列片段的等位基因序列;609聚合等位基因序列,但没有重叠等位基因;如果组装的单体型数量大于预期(即,大于预定阈值),则611选择所述等位基因中概率最大的等位基因。
图7示出了装置400和方法500中实现的不同重叠单体型分析阶段的示意图。
基于多个样本核苷酸序列与所述参考核苷酸序列的比对,第一阶段是比较它们和选择等位基因,例如,核苷酸错配、缺失或插入。每个等位基因均包含有关其位置(即,所述参考序列内的绝对位置)、符号和/或变化类型的信息,例如单核苷酸多态性、缺失和插入。选定的等位基因序列包括有关其在所述参考中的起始和结束的信息以及一组有界等位基因。提取的等位基因序列可用于以下步骤中的单体型聚合。
因此,如上所述,装置400的处理单元401用于通过基于所述参考核苷酸序列从所述多个样本核苷酸序列中提取多个等位基因序列产生初始等位基因序列组,其中所述多个等位基因序列中的每个等位基因序列中的每个等位基因与所述参考核苷酸序列中的核苷酸位点相关联。
此外,如上所述,装置400的处理单元401用于通过从所述多个样本核苷酸序列中提取等位基因序列来产生所述初始等位基因序列组,所述等位基因序列具有至少一个核苷酸不匹配相应核苷酸位点处的参考核苷酸序列的相应核苷酸。
第二阶段是基于所述等位基因序列、每个单体型的过滤阈值的输入值和单体型的预期数量滤去所有偶然和稀有等位基因。为了应用过滤,可以首先按照公式
在过滤后,装置400的处理单元401可以开始在图7的阶段3中将等位基因序列聚合成单体型。如上所述,为此目的,处理单元401用于通过将来自所述初始等位基因序列组的在重叠序列部分具有相同等位基因并且属于相同单体型的那些等位基因序列组组合成聚合等位基因序列,基于所述初始等位基因序列组产生第一聚合等位基因序列组,其中第一聚合等位基因序列组包含所述聚合等位基因序列和来自所述初始等位基因序列组的未组合成聚合等位基因序列的等位基因序列。
根据一实施例,装置400的处理单元401用于将来自所述初始等位基因序列组的具有重叠等位基因部分的那些等位基因序列组合成聚合序列,其中所述聚合序列包含来自等位基因序列的重叠序列部分和非重叠等位基因,所述等位基因序列按照与等位基因相关的核苷酸位点顺序排序,即根据这些等位基因序列中的每个等位基因序列与参考核苷酸序列的比对排序。这方面在图8a-c中进一步说明。举例来说,在不同序列中存在两个等位基因gcc(在位点1-3处)和ta(在位点6-7处),如图8a中所示。所述两个等位基因是来自相同的单体型还是来自不同的单体型,还不确定。将等位基因移动到相同单体型中的原因可能是等位基因处于相同的序列中或者是不同序列具有重叠序列部分。根据上述原因,如图8b所示,等位基因gcccc(位点1-5)和ctta(位点4-7)属于不同的单体型,因为所述等位基因处于不同的序列中并且具有彼此在位点5重叠的不同等位基因c和t。另一方面,如图8c中所示,等位基因gcccc(位点1-5)和ccat(位点4-7)位于相同的单体型中,因为所述等位基因的重叠序列部分在位点4和5处包含相同的等位基因cc。因此,可以基于鉴定彼此重叠的不同等位基因发现不同的单体型,而具有相同重叠等位基因的序列可以合并成一个单体型。因此,为找到等位基因聚合的所有可能变体,根据一实施例,所述变体可以在各种循环中合并,直到新的等位基因聚合无法与其他等位基因聚合为止。
在图7的阶段4中,根据一实施例,装置400的处理单元401还用于从第一聚合等位基因序列组中去除等位基因序列,所述等位基因序列是第一聚合等位基因序列组中的至少一个其他等位基因序列的各部分,即所述至少一个其他等位基因序列的各片段。
当所述聚合等位基因序列彼此之间没有重叠的等位基因时,它们可能处于相同或不同的单体型中。因此,如上所述,在图7的阶段5中,装置400的处理单元401用于通过连接来自第一聚合等位基因序列组的相邻等位基因序列对,基于第一聚合等位基因序列组产生第二聚合等位基因序列组,其中相邻等位基因序列包含相邻核苷酸位点中的等位基因,但没有重叠等位基因。
在图7的阶段6中,如果第二聚合等位基因序列组的等位基因序列数量大于预期值(例如,预定阈值),装置400的处理单元401还用于通过基于统计学方法计算第二聚合等位基因序列组的每个等位基因序列的概率测量来鉴定所述多个样本核苷酸序列中的单体型,其中所述概率测量通过基于概率测度鉴定第二聚合等位基因序列组中的单体型指示等位基因序列属于单体型的概率。
所述统计方法包括基于隐马尔可夫模型(hiddenmarkovmodel,简称hmm)的贝叶斯方法,即shuyings.于2007年多伦多发表的哲学博士学位论文“使用马尔可夫链采样高效的隐马尔可夫模型的单体型推理”中所述的hmm对方法。例如,在基因组分析工具包中实现该方法。
如上所述,在最后阶段,装置400的处理单元401用于基于第二聚合等位基因序列组鉴定所述多个样本核苷酸序列中的单体型,并且这些单体型是所述重叠方法的输出。
图9示出了根据实施例的装置400和方法500中实现的单体型分析的自适应策略的示意图。在一实施例中,装置400用于确定所述参考序列是否具有任何重复。如果重复次数(也称为频率)大于预定阈值,则装置400可以用于使用传统的从头组装,特别是debruijn图重组。否则,也就是说,如果重复次数小于所述预定阈值,则装置400可以用于使用由本发明实施例实现的重叠单体型分析。
在一实施例中,所述自适应单体型分析方法包括一种新颖的哈希码生成方法,如图10所示。根据实施例的装置400中实现的哈希码生成包括以下主要步骤:首先,用0初始化整数count和哈希码,然后初始化空set;第二,如果核苷酸符号的参考序列具有下一个核苷酸符号,则选择所述下一个核苷酸符号;基于所选择的核苷酸符号产生唯一哈希码;如果所述set包含所述哈希码,则递增所述count的值,或者将所述哈希码添加到所述set中;如果所述count值等于预定阈值,则完成所述循环并使用所述从头组件(例如,所述debruijn图方法);第三,如果在计算区域中所有相同的哈希码之后,所述count值仍然低于所述预定阈值,则通过重叠单体型分析方法来生成单体型。
自适应单体型分析方法包括三个主要阶段。在阶段1中,首先初始化整数count和具有0值的哈希码,并创建带有整数的空set,这些整数将在下一步中使用。
在阶段2中,如果当前区域内的参考序列具有下一个核苷酸符号,则选择该符号用于生成唯一哈希码,这将在下面进一步描述。如果set包含所述生成的哈希码,则所述count值会递增,否则所述哈希码会添加到所述set中。当所述count递增时,将检查其是否等于预定阈值;如果为真,则所述循环结束并且所述头组装将用于当前区域,其中从所述头组装方法可以包括所述debruijn图的已知重组,例如,在所述开源软件基因组分析工具包中实现所述debruijn图的已知重组。
在阶段3中,如果所述循环结束并且所述count值仍然低于所述预定阈值,则可以采用所述重叠单体型分析方法来生成单体型。
根据一实施例,所述自适应单体型分析的效率主要由所述唯一哈希码的生成决定。该哈希码方法应用于预定长度的核苷酸子序列,并包括图10中所示的以下步骤:第一步,用0至3(即,a:0;c:1、g:2、t:3)的相应值替换核苷酸符号;第二步,将哈希码的当前值左移2位;第三步,对所述前一步骤的结果和所述核苷酸值(来自第一步)采用所述按位or;第四步,对所述前一步骤的结果采用二进制掩码,其中最后2*(预定义的子序列长度)位用1填充,其他用0填充,并将所述结果作为所述哈希码的新值返回。
因此,根据一实施例,自适应单体型分析的方法可以通过生成唯一哈希码来有效地对具有不同重复序列的核苷酸序列的基因组区域进行单体型分析,以快速鉴定预定长度的重复子序列,因此可以确定采用所述新颖方法,即重叠单体型,所述重叠单体型适用于具有低重复率的区域,也可以采用所述从头组装方法,该方法适用于具有高重复率的区域。
根据一实施例,重叠单体型分析500的方法用于具有高等位基因频率的食道鳞状细胞癌的基因组。使用由broad研究所提供的开源软件基因组分析工具包的修改版本。图11中示出了所述基因组分析工具包的修改管道的示意图。利用基因组分析工具包可以实现鉴定活性区域进行单体型分析1101,使用debruijn图1102从头组装合理的单体型,以及通过hmm对1103选择单体型。在该实施例中,所述debruijn图组装被替换为所述重叠单体型分析组装(上述步骤1至5)。单体型分析的步骤实施、输入参数和结果的特性将在下面进一步描述。
为了比较所述原始实施例和修改后实施例的结果,根据实施例提供单体型预期数量2和每个单体型的过滤阈值3%作为重叠单体型分析的输入。对具有高突变频率的食管鳞状细胞癌基因组的四个区间进行了分析。
图12所示表格列出了使用debruijn图重组(debruijngraphreassembling,简称dbgr)和重叠组装(overlappingassembly,简称oa)对基因组的不同区间进行单体型分析的结果和执行时间。从图12的表格中可以看出,根据本发明实施例通过所述重叠方法鉴定的等位基因的质量和数量通常优于通过所述常规算法鉴定的等位基因的质量和数量;尤其是,所述重叠单体型分析方法的执行时间提高了3到4倍。
图13对所述单体型分析结果进行了可视化,其中示出了本发明实施例中实现的debruijn图重组和重叠组装产生的区间190610至190645kb中的染色体4的单体型。图13示出了通过所述两种方法鉴定的等位基因几乎相同,并且所述鉴定的等位基因的数量也非常相似,这证实了由本发明实施例实现的重叠组装(overlappingassembly,简称oa)可以至少与所述传统debruijn图重组(debruijngraphreassembling,简称dbgr)一样好。
根据一实施例,所提出的自适应单体型分析的方法可以有效地鉴定人类基因组中的单体型,其中可以使用所述基因组分析工具包的修改版本来执行所述自适应单体型分析。同样,所述输入的参数包括单体型预期数量2和每个单体型的过滤阈值3%。在所述人类基因组na12878的第20号染色体中鉴定的单体型构成了由加州大学伯克利分校提供的数据集。对所述单体型分析质量的评估由加州大学伯克利分校提供的开源软件smash工具进行,并在下面的图14a-14b中示出。
图14a所示的示意图说明了本发明实施例中实现的自适应单体型分析与所述debruijn图之间作为重复值(r)函数的精确度的比较,其中y轴表示所述自适应单体型分析的精确度与debruijn图的精确度之比,x轴表示重复水平(r)。
类似地,图14b所示的示意图说明了本发明实施例中实现的自适应单体型分析与debruijn图之间作为重复值(r)函数的执行时间的比较,其中y轴表示所述自适应单体型分析的执行时间与所述debruijn图的执行时间之比,x轴表示重复水平(r)。
在所述重叠单体型分析方法的以下步骤中,可以对重复子序列的长度和数量使用预定义的重复水平(r)。在生成用于创建二进制掩码的唯一哈希码期间:最后的2xr位用1填充。在实施所述自适应策略期间,如果所鉴定的重复子序列的数量高于r,则可以使用所述debruijn图方法的从头重组进行单体型分析,或者如果所述鉴定的重复子序列的数量低于r,则可以使用本发明实施例中实现的重叠组件。
根据图14a-14b,在重复阈值r=9时,在本发明实施例中实现的自适应单体型的执行时间比所述debruijn图重组的执行时间短2倍,且精度没有降低。最佳精度的结果显示为r=8,执行时间提高了1.7倍。根据这些结果,当对所述人类基因组采用本发明实施例中实现的自适应单体型分析时,可以推荐使用r=8值作为所述长度和重复子序列数量的阈值。
尽管本发明的特定特征或方面可能已经仅结合几种实施方式或实施例中的一种进行公开,但此类特征或方面可以和其它实施方式或实施例中的一个或多个特征或方面相结合,只要对任何给定或特定的应用有需要或有利即可。而且,在一定程度上,术语“包括”、“有”、“具有”或这些词的其它变形在详细的说明书或权利要求书中使用,这类术语和所述术语“包括”是类似的,都是表示包括的含义。同样,术语“示例性地”、“例如”和“如”仅表示为示例,而不是最好或最佳的。可以使用术语“耦合”和“连接”及其派生词。应当理解,这些术语可以用于指示两个元件彼此协作或交互,而不管它们是直接物理接触还是电接触,或者它们彼此不直接接触。
尽管本文中已说明和描述特定方面,但本领域普通技术人员应了解,多种替代和/或等效实现形式可在不脱离本发明的范围的情况下替代所示和描述的特定方面。本申请旨在覆盖本文论述的特定方面的任何修改或变更。
尽管以上权利要求书中的元件是利用对应的标签按照特定顺序列举的,但是除非对权利要求的阐述另有暗示用于实施部分或所有这些元件的特定顺序,否则这些元件不必限于以所述特定顺序来实施。
通过以上启示,对于本领域技术人员来说,许多替代、修改和变化是显而易见的。当然,本领域技术人员容易认识到除本文所述的应用之外,还存在本发明的众多其它应用。虽然已参考一个或多个特定实施例描述了本发明,但本领域技术人员将认识到在不偏离本发明的范围的前提下,仍可对本发明作出许多改变。因此,应理解,只要是在所附权利要求书及其等效物的范围内,可以用不同于本文具体描述的方式来实施本发明。
- 还没有人留言评论。精彩留言会获得点赞!