一种基于相似性网络融合算法的生物数据网络处理方法与流程
本发明涉及一种基于相似性网络融合算法的生物数据网络处理方法,属于生物信息分析技术领域。
背景技术:
伴随着人类基因组计划的发展,生物信息学也在迅速完善和发展。高通量测序技术的发展促进了更全面更深入地对基因组进行分析。随着测序成本的不断降低,包括基因组学、转录组学等在内的多组学数据不断积累,海量生物学数据有助于全面地有效地挖掘其中所蕴含的生物学知识,为生物信息分析提供了丰富的数据资源,同时也带来了新的挑战。随着具备大数据特征的生物学数据的不断积累及精准医疗战略计划的开启,生物信息分析的重要性也日益增长,对当前相关领域的发展具有重大的推动意义。而如何通过生物实验数据挖掘出生物网络的潜在变化,一直是以系统的方法研究生命现象的热点和难点。常规的方法往往只能同时针对某一生物类型数据进行分析,而无法同时对多种生物类型数据进行分析,无法利用不同类型数据蕴含的不同特征。
肖斌(《luminal亚型乳腺癌细胞与正常乳腺细胞的circrna表达谱差异分析》,南方医科大学学报,2018,38(8),1014-1019.)等报道的是一种单因素的生物信息分析。通过两种细胞的circrna表达谱,提取数据后,对采集到的阵列图像进行分位数归一化和后续数据处理,进行火山图和聚类热图分析,得到luminal亚型乳腺癌细胞与正常乳腺细胞的circrna表达差异较大,其中表达上调或下调的circrna有望成为luminal亚型乳腺癌诊断的新靶标的结论。然而在实际的疾病基因关系中,通常是由多种类型基因相互影响共同作用于细胞进而产生疾病,单一数据的分析存在一定的局限性。
刘玉智(《表达谱芯片与dna甲基化芯片综合分析探索鼻咽癌发生、发展的分子靶标》,临床检验杂志,2018,(8),574-578.)分别分析基因表达芯片数据和dna甲基化芯片数据,利用r语言的相关工具包对表达谱芯片进行差异表达分析,对dna甲基化芯片进行差异甲基化位点分析,利用david数据库对筛选出的差异表达基因进行基因功能分析和信号通路分析,最后筛选出4个与鼻咽癌发生、发展相关的分子靶标和潜在的治疗靶点。虽然这篇文章已经运用到多种类型数据,获得了鼻咽癌相关治疗靶点,但是其实质仍是对单一类型数据进行数据处理,并没有在同一时间融合多种数据的特征进行数据分析。
技术实现要素:
为了解决目前存在的只能从单一类型数据进行分析而没有在同一时间融合多种类型的数据的特征进行数据分析从而确定疾病亚型的问题,本发明提供了一种基于相似性网络融合算法的生物数据网络处理方法。
一种生物数据网络处理方法,所述方法包括:
s1:根据不同生物数据类型的样本数据集分别构建各个类型对应的样本相似性矩阵;
s2:根据s1构建的各个类型对应的样本相似性矩阵,采用snf算法构建多种类型的样本数据的融合相似性矩阵;
s3:采用谱聚类方法将s2得到的多种类型的样本数据对应的融合相似性矩阵进行聚类确定样本数据所属子类。
可选的,所述s1包括:
对包含有不同生物数据类型的样本数据集中的每一类型数据进行归一化处理;
归一化后计算同一类型的样本间的欧式距离,构建距离矩阵;
采用高斯热核函数构建各个类型的样本数据的样本相似性矩阵。
可选的,所述欧式距离dij计算公式为:
其中,样本数据集共包含m个类型的样本数据,样本个数为n,mv为每个类型的样本数据所包含的基因数量,v=1…m,xik代表样本i的第k个基因,i、j取值范围为[1,n],k取值范围为[1,mv];xjk代表样本j的第k个基因。
可选的,所述采用高斯热核函数构建各个类型的样本数据的样本相似性矩阵,包括:
每个类型的样本数据的样本相似性矩阵记为wv,则各个类型的样本数据的样本相似性矩阵为:
其中,μ为一个超参数,取值范围为[0.3,0.8];εij为用于消除缩放比例问题的参数。
可选的,εij定义为:
其中,ni代表除样本i之外的样本,mean(d(i,ni))为样本xi到其他样本ni的距离均值。
可选的,所述s2包括:
在得到s1构建的各个类型对应的样本相似性矩阵wv后,根据下述公式得到各个类型的样本数据对应的归一化权值矩阵p(v):
∑f≠iwif表示样本i与同一类型的样本数据中其他所有样本的相似性之和,f取值范围为[1,n];
定义用来测量局部亲和力的核矩阵s,记各个类型的样本数据对应的核矩阵为s(v):
采用snf算法更新每个数据类型对应的样本相似性矩阵wv,迭代预定次数后,得到更新后的p(v)′:
其中,∑k≠vp(k)表示除了当前数据类型v之外的所有数据类型对应的归一化矩阵p(v)之和;
融合所有数据类型的相似性矩阵得到融合相似性矩阵p:
可选的,所述迭代预定次数为迭代10-20次。
可选的,所述方法还包括:根据样本相似性矩阵得到样本相似性网络。
本发明的第二个目的在于提供上述方法在分析疾病亚型鉴定方面的应用。
本发明的第三个目的在于提供上述方法在生物信息分析技术领域内的应用。
本发明有益效果是:
通过采用snf算法,通过首先对生物多种遗传信息如mrna,mirna,lncrna等构建相似性网络,再使用snf算法对相似矩阵进行融合,创建可用的样本网络,利用谱聚类进行聚类,分析网络之间的关系,从而用于疾病致病机理的发现,早期诊断和后期治疗等领域。本发明方法能够利用不同类型的数据的互补性得到更为综合的结果,大大优于单一数据的分析和建立,为后续的综合分析建立基础。
附图说明
为了更清楚地说明本发明实施例中的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1是本发明通过snf算法进行融合得到的融合相似性矩阵对应的融合相似性网络示意图。
图2是本发明构建的样本相似性网络图。
图3是本发明使用谱聚类对融合相似性矩阵进行聚类分析结果图。
图4是对图3所示的聚类分析结果标出明显方块后的示意图。
具体实施方式
为使本发明的目的、技术方案和优点更加清楚,下面将结合附图对本发明实施方式作进一步地详细描述。
实施例一:
本实施例提供一种基于相似性网络融合算法的生物数据网络处理方法,为详细进行说明,本实施例以采用mrna,mirna,lncrna三种类型数据作为数据集为例,分别以这些数据构建相似性矩阵进行融合,再构建样本网络进行分析。下述过程中输入的是胰腺癌患者共177个样本的mrna,mirna,lncrna三种类型的数据,该样本数据来源于tcga数据库(https://www.cancer.gov/tcga)。
本实施例提供的基于相似性网络融合算法的生物数据网络处理方法,包括:
(1)分别构建各个类型样本数据对应的样本相似性矩阵以及样本相似性网络;
假设样本数据集为{x1,x2,...,xn},该样本数据集共包含m个类型的样本数据,样本个数为n,每个类型的样本数据所包含的基因数量分别为mv(v=1…m);本实施例中n=177,m=3,mrna,mirna,lncrna三种类型的样本数据中分别包含的基因数量分别为m1=8073,m2=557,m3=17914。
首先,对上述177个样本的mrna,mirna,lncrna不同类型的生物数据集中的每一类型数据进行归一化处理,归一化后计算样本间的欧式距离,构建距离矩阵,再由高斯热核函数构建各个类型的样本数据的样本相似性矩阵wv,其中v=1…m。则三个类型的样本数据分别对应的样本相似性矩阵分别为w1,w2,w3;
为描述简便,下述以构建一个类型的样本数据对应的样本相似性矩阵以及样本相似性网络个过程为例进行说明,多种类型则需要分别对每个类型的样本数据进行下述过程。
归一化公式为:
u为均值,σ为标准差,x为样本数据。
欧式距离计算公式为:
i、j取值范围为[1,n],本实施例中i,j∈(1,177),xik代表样本i的第k个基因,k取值范围为[1,mv]。
由高斯热核函数构建每个类型的样本数据的样本相似性矩阵wv为:
其中,wij表示样本i与样本j的相似性,μ是一个超参数,取值范围为[0.3,0.8];dij表示样本i与样本j的欧几里得距离;εij是用于消除缩放比例问题的参数,εij定义为
其中,ni代表除样本i之外的样本,mean(d(i,ni))为样本xi到其他样本ni的距离均值;
在得到每个类型的样本数据的样本相似性矩阵wv后,以图的形式进行表示,得出每个类型的样本数据对应的样本相似性网络。
(2)相似网络融合
在得到以不同生物数据类型构建的样本相似性矩阵w后,利用相似网络融合(similaritynetworkfusion,snf)算法,迭代更新状态矩阵,状态矩阵即每次迭代时输入的样本相似性矩阵,最终得到多种类型的样本数据的融合相似性矩阵,构建融合样本网络,从而进行下一步分析。
snf算法是使用样本网络作为整合基础,为每个数据类型构建样本相似性网络,并使用非线性组合方法将这些网络集成到单个相似性的网络中的方法。snf算法超越了目前捕获连续的表型的分型策略,大大优于单一数据的分析和建立,在识别肿瘤亚型,预测生存时是十分有效的。
基于snf算法的相似网络迭代融合,使上述各个数据类型很好地整合在一起,从而从一个全面的角度进一步挖掘生物信息。
在得到各个数据类型的样本相似性矩阵wv后,根据下述公式得到各个类型的样本数据对应的归一化权值矩阵p(v):
∑f≠iwif表示样本i与同一类型的样本数据中其他所有样本的相似性之和,f取值范围为[1,n];
该归一化矩阵p(v)不受对角线自相似性的影响,避免了数值的不稳定性。
定义用来测量局部亲和力的核矩阵s,记各个类型的样本数据对应的核矩阵为s(v):
采用snf算法更新每个数据类型对应的样本相似性矩阵wv,迭代预定次数后,预定次数取10-20次,得到更新后的p(v)′:
其中,∑k≠vp(k)表示除了当前数据类型v之外的所有数据类型对应的归一化矩阵p(v)之和;
本实施例中3个类型对应的样本相似性矩阵
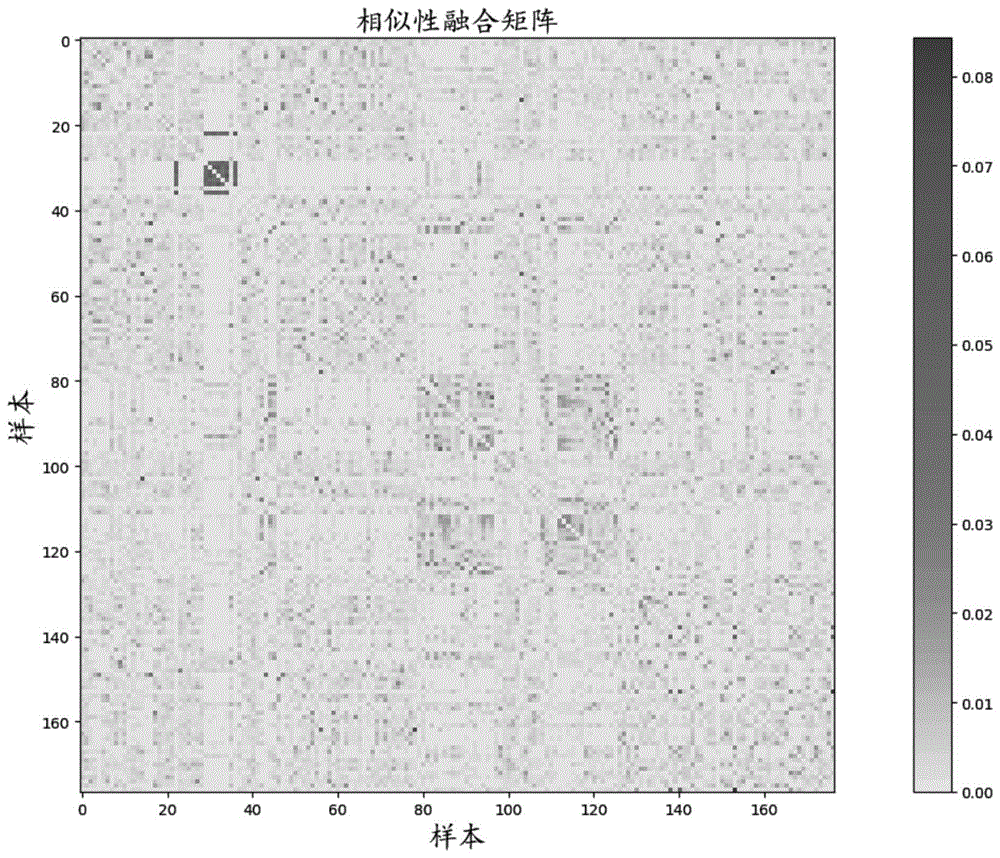
在m个样本相似性网络上进行数据的特征融合过程中,如果两个样本i和j在所有数据类型中都是相似的,那么它们的相似性将通过融合过程得到增强,反之亦然。融合所有数据类型的相似性矩阵得到融合相似性矩阵p:
融合相似性矩阵p对应的融合相似性网络如图1所示,由它构建样本相似性网络,如图2所示。
(3)谱聚类
用谱聚类方法将上述得到的融合相似性矩阵p聚类而得到子类。
假设聚类总数为c,每一个样本xi拥有一个标签指示向量yi∈{0,1}c,当xi属于第c个聚类时,c的取值范围为[1,c],
yi(k)=1
否则,
yi(k)=0
用划分矩阵
s.t.qtq=i
其中q=y(yty)-1/2为规模划分矩阵;l+=i-d-1/2pd-1/2表示融合相似性矩阵p的标准化拉普拉斯矩阵;矩阵d是融合相似性矩阵p对应的相似性网络的度的矩阵,对角线元素为对应位置节点的度,非对角线元素设置为0。目标函数可以用特征向量分解问题来表征。通过计算最小的k特征向量,并对约简后的数据应用k-均值算法,得到样本的聚类,分析结果如图3所示,样本已被聚类为三个子类,对比图1,图3中可以看出三个明显的大小不一的方块,每个方块代表一个子类,将这三个明显方块标出示意图如图4所示。
本发明通过采用snf算法,通过计算样本相似性、进行相似性网络融合来创建一个生物信息的全面视图的计算模型。snf算法可以保持较高的信噪比,使各个数据类型可以很好地整合在一起。而谱聚类算法可分析网络节点间的关系。本发明方法能够集中多种数据类型的特征,解决了单数据分析的局限性,为后续的综合分析诸如疾病亚型鉴定等建立了基础。
本发明实施例中的部分步骤,可以利用软件实现,相应的软件程序可以存储在可读取的存储介质中,如光盘或硬盘等。
以上所述仅为本发明的较佳实施例,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!