一种构建铬还原酶蛋白数据库的方法与流程
本发明属于生物技术领域,涉及一种构建铬还原酶蛋白数据库的方法,具体涉及一种基于数据库关键词搜索及比对后筛选建立铬还原酶蛋白数据库的生物信息方法。
背景技术:
重金属污染是自然界普遍存在的一种工业污染。铬(cr)作为重金属中毒性极强的一种污染物,其处理和回收更是成为世界各国科学家研究重金属污染的重中之重。cr污染不仅已经成为危害环境,影响人类健康的元凶之一,同时也严重损伤农作物机体。cr污染的来源主要来源于皮革、电镀、颜料及木材防腐等行业。cr虽然是人和动物所必需的微量元素,但是过量的cr有致癌作用,尤其是六价铬cr(vi)是一种危害极大的致癌物质,如今cr渣已成为铬污染的重要环境问题。
六价的氧化形式cr(vi)由于具有强烈的生物毒性和极强的水相迁移性,而cr(iii)在环境中易形成非晶型的氢氧化物沉淀,且生物毒性较小,因此将cr(vi)还原成cr(iii)已成为一种公认的治理cr(vi)污染的有效策略。自从20世纪70年代romanenko等首次在厌氧环境中发现了具有六价铬还原能力的细菌以来,细菌介导的cr(vi)还原由于具有反应温和、能耗低且无二次污染的特点,被认为是最具潜力的cr(vi)环境修复方法之一。具有cr(vi)还原能力的菌株分别来自无色杆菌、土壤细菌、芽饱杆菌、脱硫弧菌、肠杆菌、大肠杆菌、微球菌、硫杆菌以及假单胞菌等多个不同的种属,种类十分繁多,其中除了大肠杆菌、芽抱杆菌、硫杆菌以及假单胞菌等种属的菌株能在好氧条件下将cr(vi)还原之外,绝大多数种类的菌株都只能在厌氧的情况下还原cr(vi)。
微生物对cr(vi)还原主要是酶的作用,目前已经发现了不同物种来源的cr(vi)还原酶,并公布了其蛋白序列,但是目前缺少cr(vi)还原酶蛋白数据库,对cr(vi)还原酶多样性及物种来源缺乏系统的了解。
技术实现要素:
基于目前缺少cr(vi)还原酶蛋白数据库的现实,本发明的首要目的是提供一种cr(vi)还原酶蛋白数据库的构建方法。本发明首次提出以下载生物信息数据库(如ncbi、uniprot)中含有cr(vi)还原酶关键词的蛋白序列,应用生物信息学方法对序列进行长度处理及物种分析,序列比对后筛选确定cr(vi)还原酶蛋白的关键技术。该方法的关键步骤为cr(vi)还原酶蛋白的生物信息学鉴定,以及根据序列注册号获取其详细物种信息。
本发明的另一目的在于提供一种铬还原酶蛋白数据库。
本发明的目的通过下述技术方案实现:
一种构建铬还原酶蛋白数据库的方法,包括如下步骤:
(1)数据获取:从现有的蛋白数据库中批量下载相关cr(vi)还原酶蛋白序列;
(2)数据过滤:按如下规则对步骤(1)得到的cr(vi)还原酶蛋白序列进行过滤:
①删除和cr(vi)还原酶不相关的序列;
②删除物种名重复序列(菌株名不同视为不同的物种),物种名重复的序列只保留蛋白氨基酸序列长度最长的一条,去除来源于同一物种并且长度相同相似性为100%的序列;
③删除含有序列名字含有partical、uncultured和unclassifed中的一个词或至少两个词的序列;
(3)长度处理:筛选保留长度分布于178~218个氨基酸之间cr(vi)还原酶蛋白序列;
(4)序列比对:clustalw将筛选后的cr(vi)还原酶蛋白序列进行聚类分析,构建每条蛋白氨基酸序列的相似性矩阵,删除氨基酸序列与其它任何一条序列相似性低于30%的序列;
(5)物种分析:以ncbi的prot.accession2taxid为基础,寻找每条cr(vi)还原酶蛋白对应的物种来源信息;
(6)构建数据库:整合所有蛋白序列,完成cr(vi)还原酶蛋白数据库的构建。
步骤(1)中所述的蛋白数据库优选为ncbi数据库(https://www.ncbi.nlm.nih.gov/)和/或uniprot(https://www.uniprot.org/)数据库中的至少一种。
步骤(1)中所述的相关cr(vi)还原酶蛋白序列优选为具有关键词chromate、chromium、cr、crreductase中的一个或至少两个关键词的蛋白序列。
步骤(2)中所述的和cr(vi)还原酶蛋白不相关的序列优选序列名字中未含有关键词cr(vi)、chromium、chromate的序列。
步骤(5)中所述的prot.accession2taxid可从ftp.ncbi.nih.gov/pub/taxonomy/accession2taxid下载获得。
在本发明中,筛选得到的蛋白序列同时包含目标铬还原酶蛋白的关键词注释信息及其对应的物种信息,序列长度等信息。
本发明在关键词筛选以后用clustalw将蛋白进行全局比对后,构建每条蛋白氨基酸序列的相似性矩阵,删除氨基酸序列与其它任何一条序列相似性低于30%的序列,保留具有一定序列保守性的cr(vi)还原酶蛋白。
本专利利用ncbi的prot.accession2taxid文件,能快速准确的获得cr(vi)还原酶蛋白的物种来源及其系统进化地位。
一种铬还原酶蛋白数据库,通过所述的构建铬还原酶蛋白数据库的方法得到。
本发明相对于现有技术具有如下的优点及效果:
本发明是首个建立铬还原酶蛋白数据库的方法,利用本发明提供的方法,可以完成铬还原酶蛋白序列数据库的构建,不仅为了解自然铬还原细菌的微生物多样性及主要功能类群提供基础数据,同时可为铬还原酶蛋白基因的引物设计、数据分析和序列的物种注释提供数据支撑。
附图说明
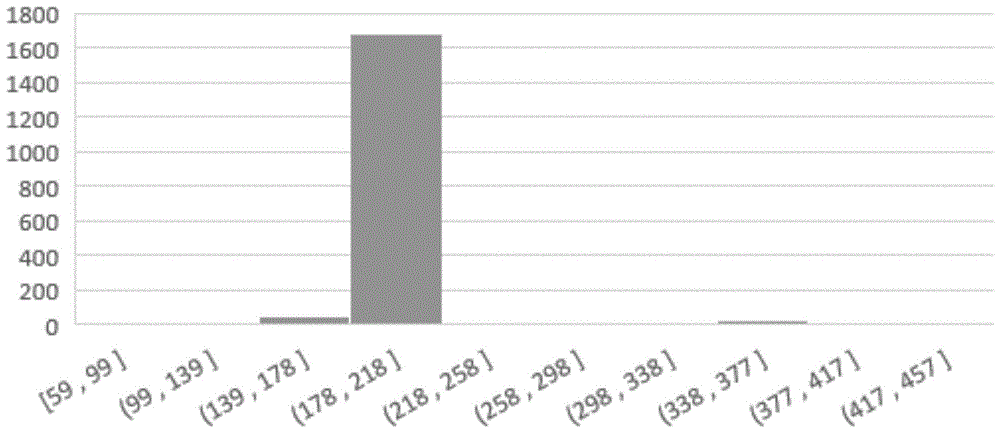
图1是cr(vi)还原酶蛋白长度分布特征分析图。
图2是cr(vi)还原酶蛋白序列的全局比对部分结果图。
图3是cr(vi)还原酶蛋白在门(phylum)水平的物种组成结果分析图。
图4是cr(vi)还原酶蛋白在科(family)水平的物种组成结果分析图。
具体实施方式
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
实施例
(1)在ncbi(https://www.ncbi.nlm.nih.gov/)及uniprot蛋白数据库(https://www.uniprot.org/)搜索含有关键词chromate和/或chromium和/或crreductase,得到对应的蛋白序列。
部分结果示例如下:
>tr|a0a037yn28|a0a037yn28_ecolxchromatereductaseos=escherichiacoliox=562gn=azrpe=4sv=1
mseklqvvtllgslrkgsfngmvartlpkiapasmevnalpsiadiplydadvqqedgfpatvealaeqirqadgvvivtpeynysvpgglknaidwlsrlpdqplagkpvliqtssmgviggarcqyhlrqilvfldamvmnkpefmggviqnkvdpqtgevidqgtldhltgqltafgefiqrvki
>tr|a0a060|a0a060_ecolxchromatereductase(fragment)os=escherichiacoliox=562gn=chrrpe=4sv=1
mseklqvvtllgslrkgsfngmvartlpkiapasmevnalpsiadiplydadvqqeegfpatvealaeqirqadgvvivtpeynysvpgglknaidwlsrlpdqplagkpvliqtssmgviggarcqyhlrqilvfldamvmnkpefmggviqnkvdpqtgevidqgtldhltgqltafgefiqrvki
>tr|a0a0e8fvu2|a0a0e8fvu2_borptchromatereductaseos=bordetellapertussisox=520gn=azrpe=4sv=1
mstykiavfvdslraasfnlrlaraleklapanfefehvslgdvplynqdhendmaapaaklkqqiadahgilfvtpehnrsipaalknaidwgsrpwghnswigktagivgtspsaagtamaqqhlrnilaaegvnvlttpevflqfkeglideqfnitnedtrkflqgwldryvawvtrlag
>tr|d3r778|d3r778_bifabchromatereductaseos=bifidobacteriumanimalissubsp.lactis(strainbb-12)ox=552531gn=bif_01684pe=3sv=1
makiihndtvdvllervsirafkdeaigldvlntieqaaqntassqylndwsairikdpavkteiaricnqdyianapvlyifvldqyrnsliaqsegvavdsdeftlknsyrfsqaqndavlalhametaaesfelgtvilgsilgnipelvkllalpkytypvlglalgkpdqhpdpkprlprdiqffddsynaepnrildgireydrevqayydtrgkdlseraysdmttkkatstaalemgfeharaqgfdlek
>tr|a0a087b2b5|a0a087b2b5_9bifichromatereductase/nadph-dependentfmnreductase/oxygen-insensitivenadphnitroreductaseos=bifidobacteriumkashiwanohensejcm15439=dsm21854ox=1150460gn=bkas_1610pe=3sv=1
msnilhnatvdtllerrsirkfkpkplsndivetletvaqhaassqflndwsairitdpatkkrlaeiggqpyiatapllyvfvldehrnaaiaaskgvetasdeftlngsyrytqaqndavlalhametaayslglgcvilgsllndvsalidllnlpeytypvlglaigkpdqdpalkprmprsmqffeneypsnnetllaglrdfdeevhqyydlrnterpvdtfsdqiasnavdsgvtsktvapnakrqgfrler
……
(2)删除序列名字中未含有关键词cr(vi)、chromium、chromate的序列,删除物种名重复序列(菌株名不同视为不同的物种),物种名重复的序列只保留蛋白氨基酸序列长度最长的一条;去除来源于同一物种并且长度相同相似性为100%的序列;删除序列名字含有partical/uncultured/unclassifed标注的序列;
(3)分析序列的长度分布特征:如图1所示,cr(vi)还原酶蛋白长度主要分布于178~218个氨基酸之间;从而筛选出完整性较高的序列;例如,如果序列太短可能来源于功能未确定的克隆文库序列。
(4)采用clustalw方法对cr(vi)还原酶蛋白序列进行对序列全局比对(图2),构建每条蛋白氨基酸序列的相似性矩阵,删除氨基酸序列与其它任何一条序列相似性低于30%的序列;
(5)从ncbi的prot.accession2taxid寻找蛋白gi号对应的物种信息。如gi|1214173918蛋白搜索出对应的物种信息为candidatusmancarchaeumacidiphilum,所述的prot.accession2taxid从ftp.ncbi.nih.gov/pub/taxonomy/accession2taxid下载;
(6)整合cr(vi)还原酶蛋白的物种信息,获得以下序列信息(部分结果示例);
>gi|1214173918|gb|asi14193.1|chromatereductase,classi,flavoprotein[candidatusmancarchaeumacidiphilum]
mensdivilgicgslrkdsynrvilnaaaesgvegvkfkfadigdlplynqdldnnlpepvkrlkhdiesadallivtpeynrsvpgplknaidwasrpygknsfdekpvavlgatvgrfvgtalvqyhlrgilafmnahlmenpqmfitevgekvkdgklvdeetkegikkvvdslkkwairiaieeddasefgl
>gi|1139286884|emb|sjk85212.1|chromatereductase,classi,flavoprotein[cuniculiplasmadivulgatum]
mvtgskkvkitafggsfrkdsynmkllkecqrlmpegseleivsikeiplynqdldenqpepvrifrnqikkadgfliatpeydfsipgflkntldyisrppmenpftgkigaimsaspsmlggsraqyhlrqvmgfmdarvinrpevfisfahtkfdenghltdemaidlikkllerlvdeirkqknivn
>gi|14209680|gb|aak56852.1|af375641_1chromatereductase[pseudomonasputida]
msqvysvavvvgslrkesynrkvaralselapsslalkiveigdlplynedveaeappeawkrfreeirrsdavlfvtpehnrsvpgclknaidvgsrpygqsawsgkptavvsvspgaiggfganhavrqslvfldmpcmqmpeayiggaaslfddsgklndktrpflqafvdkfaswvklnrav
>gi|359286686|gb|aev18370.1|chromatereductase[geobacillusthermoleovoransccb_us3_uf5]
mmnivalvgslrkdsynrklaefmrerykdkfqldiadigalphynqddeldppevvaafkrqvadadgvlivtpeynwsvpgvlknaldwlsrvdkvlvnkpvmivgvstgmmgtiraqlhlrqilqspglaakvlppagnevlvnvasqkfdeagnlvdaatvefldqvigrfiewvgr
>gi|504403203|ref|wp_014590305.1|multispecies:chromatereductase[pseudomonas]
msqvysvavvvgslrkesynrkvaralselapsslalkiveigdlplynedieaeappetwkrfrdeirrsdavlfvtpeynrsvpgclknaidvgsrpygqsawsgkptavvsvspgaiggfganhavrqslvfldmpcmqmpeaylggaaslfedsgklndktrpflqafvdkfaswvklnrav
(7)整合所有蛋白序列,完成cr(vi)还原酶蛋白数据库的构建,一共含1770条序列,来源于10个门(phylum),23个纲(class),56个目(order),115个科(family)及316个属(genus)。其中在门水平以变形菌proteobacteria(占到85.6%)、放线菌actinobacteria(6.2%)以及厚壁菌firmicutes(5.0%)为优势(图3)。而在科水平占优势的包括肠杆菌enterobacteriaceae(46.7%)和假单胞菌pseudomonadaceae(7.9%)(图4)。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
序列表
<110>广东省生态环境技术研究所
<120>一种构建铬还原酶蛋白数据库的方法
<160>10
<170>siposequencelisting1.0
<210>1
<211>188
<212>prt
<213>大肠杆菌(escherichiacoli)
<220>
<223>铬还原酶蛋白氨基酸序列
<400>1
metserglulysleuglnvalvalthrleuleuglyserleuarglys
151015
glyserpheasnglymetvalalaargthrleuprolysilealapro
202530
alasermetgluvalasnalaleuproserilealaaspileproleu
354045
tyraspalaaspvalglnglngluaspglypheproalathrvalglu
505560
alaleualagluglnileargglnalaaspglyvalvalilevalthr
65707580
proglutyrasntyrservalproglyglyleulysasnalaileasp
859095
trpleuserargleuproaspglnproleualaglylysprovalleu
100105110
ileglnthrsersermetglyvalileglyglyalaargcysglntyr
115120125
hisleuargglnileleuvalpheleuaspalametvalmetasnlys
130135140
progluphemetglyglyvalileglnasnlysvalaspproglnthr
145150155160
glygluvalileaspglnglythrleuasphisleuthrglyglnleu
165170175
thralapheglyglupheileglnargvallysile
180185
<210>2
<211>188
<212>prt
<213>大肠杆菌(escherichiacoli)
<220>
<223>铬还原酶蛋白氨基酸序列
<400>2
metserglulysleuglnvalvalthrleuleuglyserleuarglys
151015
glyserpheasnglymetvalalaargthrleuprolysilealapro
202530
alasermetgluvalasnalaleuproserilealaaspileproleu
354045
tyraspalaaspvalglnglnglugluglypheproalathrvalglu
505560
alaleualagluglnileargglnalaaspglyvalvalilevalthr
65707580
proglutyrasntyrservalproglyglyleulysasnalaileasp
859095
trpleuserargleuproaspglnproleualaglylysprovalleu
100105110
ileglnthrsersermetglyvalileglyglyalaargcysglntyr
115120125
hisleuargglnileleuvalpheleuaspalametvalmetasnlys
130135140
progluphemetglyglyvalileglnasnlysvalaspproglnthr
145150155160
glygluvalileaspglnglythrleuasphisleuthrglyglnleu
165170175
thralapheglyglupheileglnargvallysile
180185
<210>3
<211>184
<212>prt
<213>百日咳杆菌(bordetellapertussis)
<220>
<223>铬还原酶蛋白氨基酸序列
<400>3
metserthrtyrlysilealavalphevalaspserleuargalaala
151015
serpheasnleuargleualaargalaleuglulysleualaproala
202530
asnphegluphegluhisvalserleuglyaspvalproleutyrasn
354045
glnasphisgluasnaspmetalaalaproalaalalysleulysgln
505560
glnilealaaspalahisglyileleuphevalthrprogluhisasn
65707580
argserileproalaalaleulysasnalaileasptrpglyserarg
859095
protrpglyhisasnsertrpileglylysthralaglyilevalgly
100105110
thrserproseralaalaglythralametalaglnglnhisleuarg
115120125
asnileleualaalagluglyvalasnvalleuthrthrprogluval
130135140
pheleuglnphelysgluglyleuileaspgluglnpheasnilethr
145150155160
asngluaspthrarglyspheleuglnglytrpleuaspargtyrval
165170175
alatrpvalthrargleualagly
180
<210>4
<211>258
<212>prt
<213>bifidobacteriumanimalissubsp.lactis
<220>
<223>铬还原酶蛋白氨基酸序列
<400>4
metalalysileilehisasnaspthrvalaspvalleuleugluarg
151015
valserileargalaphelysaspglualaileglyleuaspvalleu
202530
asnthrilegluglnalaalaglnasnthralaserserglntyrleu
354045
asnasptrpseralaileargilelysaspproalavallysthrglu
505560
ilealaargilecysasnglnasptyrilealaasnalaprovalleu
65707580
tyrilephevalleuaspglntyrargasnserleuilealaglnser
859095
gluglyvalalavalaspseraspgluphethrleulysasnsertyr
100105110
argpheserglnalaglnasnaspalavalleualaleuhisalamet
115120125
gluthralaalagluserphegluleuglythrvalileleuglyser
130135140
ileleuglyasnileprogluleuvallysleuleualaleuprolys
145150155160
tyrthrtyrprovalleuglyleualaleuglylysproaspglnhis
165170175
proaspprolysproargleuproargaspileglnphepheaspasp
180185190
sertyrasnalagluproasnargileleuaspglyileargglutyr
195200205
asparggluvalglnalatyrtyraspthrargglylysaspleuser
210215220
gluargalatyrseraspmetthrthrlyslysalathrserthrala
225230235240
alaleuglumetglyphegluhisalaargalaglnglypheaspleu
245250255
glulys
<210>5
<211>259
<212>prt
<213>bifidobacteriumkashiwanohensejcm15439
<220>
<223>铬还原酶蛋白氨基酸序列
<400>5
metserasnileleuhisasnalathrvalaspthrleuleugluarg
151015
argserilearglysphelysprolysproleuserasnaspileval
202530
gluthrleugluthrvalalaglnhisalaalaserserglnpheleu
354045
asnasptrpseralaileargilethraspproalathrlyslysarg
505560
leualagluileglyglyglnprotyrilealathralaproleuleu
65707580
tyrvalphevalleuaspgluhisargasnalaalailealaalaser
859095
lysglyvalgluthralaseraspgluphethrleuasnglysertyr
100105110
argtyrthrglnalaglnasnaspalavalleualaleuhisalamet
115120125
gluthralaalatyrserleuglyleuglycysvalileleuglyser
130135140
leuleuasnaspvalseralaleuileaspleuleuasnleuproglu
145150155160
tyrthrtyrprovalleuglyleualaileglylysproaspglnasp
165170175
proalaleulysproargmetproargsermetglnphephegluasn
180185190
glutyrproserasnasngluthrleuleualaglyleuargaspphe
195200205
aspglugluvalhisglntyrtyraspleuargasnthrgluargpro
210215220
valaspthrpheseraspglnilealaserasnalavalaspsergly
225230235240
valthrserlysthrvalalaproasnalalysargglnglyphearg
245250255
leugluarg
<210>6
<211>196
<212>prt
<213>candidatusmancarchaeumacidiphilum
<220>
<223>铬还原酶蛋白氨基酸序列
<400>6
metgluasnseraspilevalileleuglyilecysglyserleuarg
151015
lysaspsertyrasnargvalileleuasnalaalaalaglusergly
202530
valgluglyvallysphelysphealaaspileglyaspleuproleu
354045
tyrasnglnaspleuaspasnasnleuprogluprovallysargleu
505560
lyshisaspilegluseralaaspalaleuleuilevalthrproglu
65707580
tyrasnargservalproglyproleulysasnalaileasptrpala
859095
serargprotyrglylysasnserpheaspglulysprovalalaval
100105110
leuglyalathrvalglyargphevalglythralaleuvalglntyr
115120125
hisleuargglyileleualaphemetasnalahisleumetgluasn
130135140
proglnmetpheilethrgluvalglyglulysvallysaspglylys
145150155160
leuvalaspglugluthrlysgluglyilelyslysvalvalaspser
165170175
leulyslystrpalaileargilealaileglugluaspaspalaser
180185190
glupheglyleu
195
<210>7
<211>191
<212>prt
<213>cuniculiplasmadivulgatum
<220>
<223>铬还原酶蛋白氨基酸序列
<400>7
metvalthrglyserlyslysvallysilethralapheglyglyser
151015
phearglysaspsertyrasnmetlysleuleulysglucysglnarg
202530
leumetprogluglysergluleugluilevalserilelysgluile
354045
proleutyrasnglnaspleuaspgluasnglnprogluprovalarg
505560
ilepheargasnglnilelyslysalaaspglypheleuilealathr
65707580
proglutyrasppheserileproglypheleulysasnthrleuasp
859095
tyrileserargproprometgluasnprophethrglylysilegly
100105110
alailemetseralaserprosermetleuglyglyserargalagln
115120125
tyrhisleuargglnvalmetglyphemetaspalaargvalileasn
130135140
argprogluvalpheileserphealahisthrlyspheaspgluasn
145150155160
glyhisleuthraspglumetalaileaspleuilelyslysleuleu
165170175
gluargleuvalaspgluilearglysglnlysasnilevalasn
180185190
<210>8
<211>186
<212>prt
<213>pseudomonasputida
<220>
<223>铬还原酶蛋白氨基酸序列
<400>8
metserglnvaltyrservalalavalvalvalglyserleuarglys
151015
glusertyrasnarglysvalalaargalaleusergluleualapro
202530
serserleualaleulysilevalgluileglyaspleuproleutyr
354045
asngluaspvalglualaglualaproproglualatrplysargphe
505560
argglugluileargargseraspalavalleuphevalthrproglu
65707580
hisasnargservalproglycysleulysasnalaileaspvalgly
859095
serargprotyrglyglnseralatrpserglylysprothralaval
100105110
valservalserproglyalaileglyglypheglyalaasnhisala
115120125
valargglnserleuvalpheleuaspmetprocysmetglnmetpro
130135140
glualatyrileglyglyalaalaserleupheaspaspserglylys
145150155160
leuasnasplysthrargpropheleuglnalaphevalasplysphe
165170175
alasertrpvallysleuasnargalaval
180185
<210>9
<211>181
<212>prt
<213>geobacillusthermoleovoransccb_us3_uf5
<220>
<223>铬还原酶蛋白氨基酸序列
<400>9
metmetasnilevalalaleuvalglyserleuarglysaspsertyr
151015
asnarglysleualagluphemetarggluargtyrlysasplysphe
202530
glnleuaspilealaaspileglyalaleuprohistyrasnglnasp
354045
aspgluleuaspproprogluvalvalalaalaphelysargglnval
505560
alaaspalaaspglyvalleuilevalthrproglutyrasntrpser
65707580
valproglyvalleulysasnalaleuasptrpleuserargvalasp
859095
lysvalleuvalasnlysprovalmetilevalglyvalserthrgly
100105110
metmetglythrileargalaglnleuhisleuargglnileleugln
115120125
serproglyleualaalalysvalleuproproalaglyasngluval
130135140
leuvalasnvalalaserglnlyspheaspglualaglyasnleuval
145150155160
aspalaalathrvalglupheleuaspglnvalileglyargpheile
165170175
glutrpvalglyarg
180
<210>10
<211>186
<212>prt
<213>pseudomonas
<220>
<223>铬还原酶蛋白氨基酸序列
<400>10
metserglnvaltyrservalalavalvalvalglyserleuarglys
151015
glusertyrasnarglysvalalaargalaleusergluleualapro
202530
serserleualaleulysilevalgluileglyaspleuproleutyr
354045
asngluaspileglualaglualaproprogluthrtrplysargphe
505560
argaspgluileargargseraspalavalleuphevalthrproglu
65707580
tyrasnargservalproglycysleulysasnalaileaspvalgly
859095
serargprotyrglyglnseralatrpserglylysprothralaval
100105110
valservalserproglyalaileglyglypheglyalaasnhisala
115120125
valargglnserleuvalpheleuaspmetprocysmetglnmetpro
130135140
glualatyrleuglyglyalaalaserleuphegluaspserglylys
145150155160
leuasnasplysthrargpropheleuglnalaphevalasplysphe
165170175
alasertrpvallysleuasnargalaval
180185
- 还没有人留言评论。精彩留言会获得点赞!