基于人工智能的蛋白质配体结合原子的识别方法、装置与流程
1.本技术涉及人工智能技术,尤其涉及一种基于人工智能的蛋白质配体结合原子的识别方法、装置、电子设备及计算机可读存储介质。
背景技术:
2.人工智能(ai,artificial intelligence)是计算机科学的一个综合技术,通过研究各种智能机器的设计原理与实现方法,使机器具有感知、推理与决策的功能。人工智能技术是一门综合学科,涉及领域广泛,例如自然语言处理技术以及机器学习/深度学习等几大方向,随着技术的发展,人工智能技术将在更多的领域得到应用,并发挥越来越重要的价值。
3.基于人工智能发掘蛋白质的结构的技术,对于辅助药物设计有重要价值,例如,通过分析有利和不利的蛋白-配体相互作用,构建结构活性之间的构效关系来指导药物的发现,其中,准确识别蛋白质结构中的蛋白质配体结合原子是关键关节。
4.相关技术中缺乏基于人工智能来识别蛋白质的配体结合原子的有效方案,主要依赖于蛋白质结构外部的虚拟结合位点来确定配体结合原子,导致配体结合原子的识别准确率不高。
技术实现要素:
5.本技术实施例提供一种基于人工智能的蛋白质配体结合原子的识别方法、装置、电子设备及计算机可读存储介质,能够提高配体结合原子的识别准确率。
6.本技术实施例的技术方案是这样实现的:
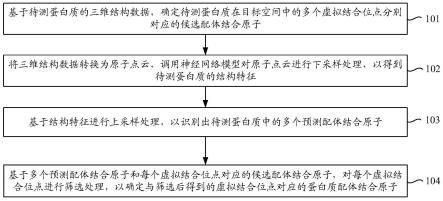
7.本技术实施例提供一种基于人工智能的蛋白质配体结合原子的识别方法,包括:
8.基于待测蛋白质的三维结构数据,确定所述待测蛋白质在目标空间中的多个虚拟结合位点分别对应的候选配体结合原子;
9.将所述三维结构数据转换为原子点云,调用神经网络模型对所述原子点云进行下采样处理,以得到所述待测蛋白质的结构特征,以及
10.基于所述结构特征进行上采样处理,以识别出所述待测蛋白质中的多个预测配体结合原子;
11.基于多个所述预测配体结合原子和每个所述虚拟结合位点对应的候选配体结合原子,对每个所述虚拟结合位点进行筛选处理,以确定与筛选后得到的虚拟结合位点对应的蛋白质配体结合原子。
12.本技术实施例提供一种基于人工智能的蛋白质配体结合原子的识别装置,包括:
13.确定模块,用于基于待测蛋白质的三维结构数据,确定所述待测蛋白质在目标空间中的多个虚拟结合位点分别对应的候选配体结合原子;
14.转换模块,用于将所述三维结构数据转换为原子点云;
15.预测模块,用于调用神经网络模型对所述原子点云进行下采样处理,以得到所述
待测蛋白质的结构特征,以及用于基于所述结构特征进行上采样处理,以识别出所述待测蛋白质中的多个预测配体结合原子;
16.筛选模块,用于基于多个所述预测配体结合原子和每个所述虚拟结合位点对应的候选配体结合原子,对每个所述虚拟结合位点进行筛选处理,以确定与筛选后得到的虚拟结合位点对应的蛋白质配体结合原子。
17.上述方案中,所述确定模块,还用于:
18.分别以配体中各个原子为球心,并以识别半径构建球体,确定由多个所述球体构成的目标空间;
19.基于所述待测蛋白质的三维结构数据,确定所述待测蛋白质的空间结构信息;
20.基于所述待测蛋白质的空间结构信息,确定所述待测蛋白质在所述目标空间中的多个虚拟结合位点;
21.确定每个所述虚拟结合位点分别对应的候选配体结合原子。
22.上述方案中,所述确定模块,还用于:
23.以配体中各个原子为球心,依次基于多个候选识别半径构建球体,得到由同一候选识别半径构建的多个球体所构成的多个目标空间,其中,每个所述目标空间中的球体的候选识别半径不同;
24.确定蛋白质样本在每个所述目标空间中的样本原子集合;
25.确定所述样本原子集合中的实际配体结合原子与非配体结合原子的样本比例,确定符合预设样本比例的样本比例对应的样本原子集合;
26.将确定的样本原子集合所处的目标空间对应的候选识别半径,作为所述识别半径。
27.上述方案中,所述神经网络模型包括j个下采样模块,j为大于或等于2的正整数;所述预测模块,还用于:
28.基于所述待测蛋白质的原子点云确定所述神经网络模型的输入特征,并作为第1个下采样模块的输入特征;
29.通过第j个下采样模块对所述第j个下采样模块的输入特征进行卷积操作,得到所述第j个下采样模块对应的结构特征,对所述第j个下采样模块对应的结构特征进行池化操作,并将所述池化操作的结果作为第j+1个下采样模块的输入特征;
30.其中,j为取值从1开始递增的整数变量,且取值满足1≤j≤j-1;
31.通过第j个下采样模块对所述第j个下采样模块的输入特征进行卷积操作,得到所述第j个下采样模块对应的结构特征,并作为所述待测蛋白质的结构特征。
32.上述方案中,所述预测模块,还用于:
33.基于所述待测蛋白质的原子点云确定所述待测蛋白质的残基类型、原子类型和原子的空间坐标;
34.基于所述待测蛋白质的残基类型、原子类型和原子的空间坐标确定所述神经网络模型的输入特征。
35.上述方案中,所述神经网络模型还包括j个上采样模块和输出模块,所述预测模块,还用于:
36.通过所述第j个下采样模块对所述待测蛋白质的结构特征进行池化操作,并将所
述池化操作的结果作为第1个上采样模块的输入特征;
37.通过第i个上采样模块对所述第i个上采样模块的输入特征进行卷积操作和上采样操作,得到所述第i个上采样模块的输出特征;
38.将所述第i个上采样模块的输出特征与第j+1-i个下采样模块对应的结构特征串联,并将串联结果作为第i+1个上采样模块的输入特征;
39.其中,i为取值从1开始递增的整数变量,且取值满足1≤i≤j-1;
40.通过第j个上采样模块对所述第j个上采样模块的输入特征进行卷积操作和上采样操作,得到所述第j个上采样模块的输出特征;
41.将所述第j个上采样模块的输出特征与第1个下采样模块对应的结构特征串联,并将串联结果作为所述输出模块的输入特征;
42.通过所述输出模块对所述输出模块的输入特征进行线性组合和分类处理,以识别出所述待测蛋白质中的多个预测配体结合原子。
43.上述方案中,所述筛选模块,还用于:
44.确定每个所述虚拟结合位点对应的多个候选配体结合原子与所述多个预测配体结合原子之间的公共原子;
45.确定所述公共原子在所述多个候选配体结合原子中所占的比例,以筛选得到大于比例阈值的比例对应的多个虚拟结合位点;
46.将筛选得到的所述多个虚拟结合位点对应的多个候选配体结合原子作为所述蛋白质配体结合原子。
47.上述方案中,所述筛选模块,还用于:
48.对筛选得到的所述多个虚拟结合位点进行排序;
49.确定排序后的多个虚拟结合位点的几何中心。
50.上述方案中,所述筛选模块,还用于:
51.基于所述比例对筛选得到的所述多个虚拟结合位点进行降序排序,或
52.基于对应的公共原子的数量对筛选得到的所述多个虚拟结合位点进行降序排序。
53.上述方案中,所述筛选模块,还用于:
54.以每个所述蛋白质配体结合原子为球心、以识别半径构建球体;
55.当所述球体与配体相交时,确定所述球体对应的蛋白质配体结合原子识别正确。
56.上述方案中,当所述待测蛋白质的实际配体结合原子为已知信息时,所述筛选模块,还用于:
57.确定筛选后得到的每个虚拟结合位点对应的蛋白质配体结合原子与所述实际配体结合原子的交并比;
58.基于所述交并比对筛选后得到的每个虚拟结合位点再次进行筛选,得到最终的虚拟结合位点以及对应的蛋白质配体结合原子。
59.上述方案中,所述基于人工智能的蛋白质配体结合原子的识别装置还包括训练模块,用于:
60.基于蛋白质样本中各原子与配体的距离为所述各原子添加标签,所述标签的类型包括实际配体结合原子和非配体结合原子;
61.调用所述神经网络模型对添加标签后的蛋白质样本的原子点云进行预测处理,得
到所述原子点云中每个原子的识别结果,所述识别结果表征相应的原子是否是配体结合原子;
62.基于所述原子点云中每个原子的标签与对应的识别结果的误差,在所述神经网络模型中反向传播,以更新所述神经网络模型的参数。
63.本技术实施例提供一种电子设备,包括:
64.存储器,用于存储可执行指令;
65.处理器,用于执行所述存储器中存储的可执行指令时,实现本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法。
66.本技术实施例提供一种计算机可读存储介质,存储有可执行指令,用于引起处理器执行时,实现本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法。
67.本技术实施例具有以下有益效果:
68.基于待测蛋白质的多个虚拟结合位点确定候选配体结合原子,然后调用神经网络模型对待测蛋白质的原子点云进行下采样处理和上采样处理,以识别出预测配体结合原子,基于预测配体结合原子对多个虚拟结合位点进行筛选,从而可以过滤掉识别错误的虚拟结合位点对应的候选配体结合原子,以准确识别蛋白质配体结合原子。
附图说明
69.图1a是本技术实施例提供的识别系统100的架构示意图;
70.图1b是本技术实施例提供的识别系统100的架构示意图;
71.图2是本技术实施例提供的服务器200的结构示意图;
72.图3a是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图;
73.图3b是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图;
74.图3c是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图;
75.图3d是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图;
76.图3e是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图;
77.图4是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别示意图;
78.图5是本技术实施例提供的确定候选配体结合原子的示意图;
79.图6是本技术实施例提供的基于ssc的u-net的结构示意图;
80.图7是本技术实施例提供的pointsite模型的结构示意图;
81.图8是本技术实施例提供的基于pdb生成的原子级别标注的示意图。
具体实施方式
82.为了使本技术的目的、技术方案和优点更加清楚,下面将结合附图对本技术作进一步地详细描述,所描述的实施例不应视为对本技术的限制,本领域普通技术人员在没有
做出创造性劳动前提下所获得的所有其它实施例,都属于本技术保护的范围。
83.在以下的描述中,涉及到“一些实施例”,其描述了所有可能实施例的子集,但是可以理解,“一些实施例”可以是所有可能实施例的相同子集或不同子集,并且可以在不冲突的情况下相互结合。
84.除非另有定义,本文所使用的所有的技术和科学术语与属于本技术的技术领域的技术人员通常理解的含义相同。本文中所使用的术语只是为了描述本技术实施例的目的,不是旨在限制本技术。
85.对本技术实施例进行进一步详细说明之前,对本技术实施例中涉及的名词和术语进行说明,本技术实施例中涉及的名词和术语适用于如下的解释。
86.1)残基:在蛋白质的序列中,氨基酸之间的氨基和羧基脱水成键,剩余的结构部分称为氨基酸残基。
87.2)配体:与蛋白质可逆结合的化学物质称为配体,配体包括金属离子、有机或无机分子、多糖和短肽等聚合物。多数蛋白质通过与其他配体相互作用来执行生物功能。配体在蛋白质上的配体结合位点与配体在大小、形状、电荷、亲水性和疏水性上是互补的。
88.3)蛋白质结合口袋(以下简称口袋):指蛋白质表面或内部具有适合与配体结合的空腔,口袋周围的氨基酸残基决定了它的形状、位置、物化特性以及功能。
89.4)配体结合位点:配体与蛋白质的结合位置,一般位于蛋白质的疏水口袋上。
90.5)蛋白质三维结构数据文件(pdb,protein data bank):通过记录蛋白质中每一个氨基酸上的每一个原子的三维坐标来存储蛋白质的三维结构数据。pdb还包括结构说明,如二硫键、螺旋、片层、活性位点等信息。
91.6)cameo:是一个公开的蛋白结构的预测的基准,分为困难示例和简单示例。
92.7)交并比:是集合a与集合b的交叠率,即它们的交集与并集的比值(a∩b)/(a∪b),最理想情况是完全重叠,即比值为1。交并比作为检测评价函数,用于评价识别的配体结合位点的准确性。
93.8)体素化:是将物体的几何形式表示转换成最接近该物体的体素表示形式,产生体数据集,其不仅包含物体的表面信息,而且能描述物体的内部属性。
94.9)点云数据:指在一个三维坐标系统中的一组向量的集合。这些向量通常以三维坐标的形式表示,用来代表一个物体的外表面形状。点云数据还可以表示一个点的颜色,灰度值、深度、分割结果等。例如,pi={xi,yi,zi,
…
}表示空间中的一个点,则point cloud(点云)={p1,p2,p3,
…
pn}表示一组点云数据。
95.10)感受野:是神经网络模型中每一层输出的特征图上每个像素点在神经网络模型的原始输入图像上映射的区域大小。
96.在计算生物学中存在一个基本的问题,即基于给定的蛋白质结构,准确地识别配体结合位点以及形成配体结合位点的原子,即配体结合原子。
97.对于理解蛋白质功能和设计基于蛋白质结构的药物而言,准确识别蛋白质结构上的配体结合位点非常重要。在细胞环境中,大多数蛋白质通过与其他配体相互作用来执行生物功能,因此,蛋白质外部配体结合位点的准确识别对于确定蛋白质的生物功能至关重要。在基于结构的药物设计(sbdd,structure-based drug design)中,需要确定出配体结合位点,并筛选出于配体结合位点对应的药物小分子,因此,在sbdd中,配体结合位点的准
确识别也是不可或缺的一步。然而,使用实验技术检测蛋白质的配体结合位点昂贵又耗时,通常还需要测量出蛋白质-配体复合物的三维结构。已知三维结构的蛋白质在pdb中只有10万多个。然而,蛋白质序列数据库中却有几百万条蛋白质序列。也就是说,绝大多数蛋白质以及蛋白质-配体复合物的三维结构是未知的。
98.相关技术中用于确定蛋白质配体结合位点的方法主要包括基于模板的方法和无模板的方法。形式上,蛋白质的配体结合位点与对应的配体原子在一定距离内,距离可为其中,为长度单位,表示10-10
米。无模板方法包括以口袋为中心的方法,它包括以下两个步骤:(i)以配体(如药物小分子)中任一原子为球心,以特定半径(即识别半径)构建球体,识别在球体范围内的蛋白质上的虚拟结合位点,可将虚拟结合位点看作可能的配体结合位点或口袋的一部分;(ii)将虚拟结合位点对应的原子作为配体结合原子。
99.以口袋为中心的方法包括几何策略、能量策略和机器学习。几何策略指利用蛋白质的三维结构来搜索蛋白质的空腔和口袋,进而识别配体结合位点的方法,它可以进一步分为网格扫描、ligsiet、ligsite_csc、probe sphere、α形状和alpha sphere(如fpocket)。能量策略通过计算蛋白质各个位置的原子与配体的结合能量来确定配体结合位点,它可以进一步分为sitehound,q-sitefinder,pocketfinder。机器学习利用影响配体结合位点的残基和/或原子水平的蛋白质特征,通过机器学习算法(如基于三维卷积神经网络模型的deepsite、基于随机森林的p2rank)识别配体结合位点。在确定配体结合位点之后,可确定配体结合位点对应的配体结合原子。
100.可见,相关技术中,以口袋为中心的方法需要先确定虚拟结合位点,然后基于虚拟结合位点确定配体结合原子。然而,虚拟结合位点不是实际的原子,不能体现蛋白质结构的局部连通性和全局三维信息,也不能体现蛋白质的原子和/或残基的物理化学特征,因此,通过虚拟结合位点确定配体结合原子的准确率不高。
101.本技术实施例提供一种基于人工智能的蛋白质配体结合原子的识别方法,能有效地提高配体结合原子的识别准确率。
102.本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法可以由各种电子设备实施,例如,可以由终端或服务器单独实施,也可以由服务器和终端协同实施。例如终端独自承担下文所述的基于人工智能的蛋白质配体结合原子的识别方法,或者,终端向服务器发送针对待测蛋白质的配体结合原子识别请求,服务器根据接收的识别请求执行基于人工智能的蛋白质配体结合原子的识别方法。
103.本技术实施例提供的用于识别蛋白质配体结合原子的电子设备可以是各种类型的终端设备或服务器,其中,服务器可以是独立的物理服务器,也可以是多个物理服务器构成的服务器集群或者分布式系统,还可以是提供云服务、云数据库、云计算、云函数、云存储、网络服务、云通信、中间件服务、域名服务、安全服务、cdn、以及大数据和人工智能平台等基础云计算服务的云服务器;终端可以是平板电脑、笔记本电脑、台式计算机等,但并不局限于此。终端以及服务器可以通过有线或无线通信方式进行直接或间接地连接,本技术对此不做限制。
104.以服务器为例,例如可以是部署在云端的服务器集群,向用户开放人工智能云服务(aiaas,ai as a service),aiaas平台会把几类常见的ai服务进行拆分,并在云端提供独立或者打包的服务,这种服务模式类似于一个ai主题商城,所有的用户都可以通过应用
程序编程接口的方式来接入使用aiaas平台提供的一种或者多种人工智能服务。
105.例如,其中的一种人工智能云服务可以为配体结合原子识别服务,即云端的服务器封装有本技术实施例提供的配体结合原子识别的程序。云端的服务器基于蛋白质样本进行有监督的训练,得到可用于识别蛋白质的配体结合原子的神经网络模型。此后,终端通过调用云服务中的配体结合原子识别服务,以使部署在云端的服务器通过上述神经网络模型对待测蛋白质进行预测处理,识别出待测蛋白质中的预测配体结合原子,将预测配体结合原子作为蛋白质配体结合原子返回给终端。
106.在一些实施例中,以服务器和终端协同实施本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法为例进行说明一个示例性的识别系统。参见图1a,图1a是本技术实施例提供的识别系统100的架构示意图。终端400通过网络300连接服务器200,网络300可以是广域网或者局域网,又或者是二者的组合。终端向服务器发送针对待测蛋白质的配体结合原子识别请求,服务器根据接收的识别请求执行基于人工智能的蛋白质配体结合原子的识别方法。
107.在一些实施例中,终端400响应于用户的识别触发操作,生成针对待测蛋白质的识别请求,并向服务器200发送识别请求。服务器200接收来自终端400的识别请求后,获取待测蛋白质对应的pdb,基于pdb确定待测蛋白质的三维结构数据,并基于待测蛋白质的三维结构数据确定候选配体结合原子。通过神经网络模型识别出待测蛋白质的预测配体结合原子,基于预测配体结合原子从候选配体结合原子中筛选出蛋白质配体结合原子,将蛋白质配体结合原子发送给终端400。
108.本技术实施例还可以通过区块链技术来实现,参见图1b,图1b是本技术实施例提供的识别系统100的架构示意图。区块链网络500(示例性示出了区块链网络500包括的节点510-1、节点510-2和节点510-3)中存储有pdb,服务器从区块链网络500获取pdb,基于pdb中的蛋白质样本训练得到神经网络模型,并将训练好的神经网络模型部署到线上。当服务器200接收到针对待测蛋白质的识别请求时,从区块链网络500获取待测蛋白质对应的pdb,基于pdb确定待测蛋白质的三维结构数据,并基于待测蛋白质的三维结构数据确定候选配体结合原子。之后,通过训练好的神经网络模型识别出待测蛋白质的预测配体结合原子,基于预测配体结合原子从候选配体结合原子中筛选出蛋白质配体结合原子,将蛋白质配体结合原子发送给终端400。
109.以实施本技术实施例的电子设备为图1a示出的服务器200为例,说明本技术实施例提供的电子设备的结构。参见图2,图2是本技术实施例提供的服务器200的结构示意图,图2所示的服务器200包括:至少一个处理器410、存储器440、至少一个网络接口420。服务器200中的各个组件通过总线系统430耦合在一起。可理解,总线系统430用于实现这些组件之间的连接通信。总线系统430除包括数据总线之外,还包括电源总线、控制总线和状态信号总线。但是为了清楚说明起见,在图2中将各种总线都标为总线系统430。
110.处理器410可以是一种集成电路芯片,具有信号的处理能力,例如通用处理器、数字信号处理器(dsp,digital signal processor),或者其他可编程逻辑器件、分立门或者晶体管逻辑器件、分立硬件组件等,其中,通用处理器可以是微处理器或者任何常规的处理器等。
111.存储器440可以是可移除的,不可移除的或其组合。示例性的硬件设备包括固态存
储器,硬盘驱动器,光盘驱动器等。存储器440可选地包括在物理位置上远离处理器410的一个或多个存储设备。
112.存储器440包括易失性存储器或非易失性存储器,也可包括易失性和非易失性存储器两者。非易失性存储器可以是只读存储器(rom,read only memory),易失性存储器可以是随机存取存储器(ram,random access memory)。本技术实施例描述的存储器440旨在包括任意适合类型的存储器。
113.在一些实施例中,存储器440能够存储数据以支持各种操作,这些数据的示例包括程序、模块和数据结构或者其子集或超集,下面示例性说明。
114.操作系统441,包括用于处理各种基本系统服务和执行硬件相关任务的系统程序,例如框架层、核心库层、驱动层等,用于实现各种基础业务以及处理基于硬件的任务;
115.网络通信模块442,用于经由一个或多个(有线或无线)网络接口420到达其他计算设备,示例性的网络接口420包括:蓝牙、无线相容性认证(wifi)、和通用串行总线(usb,universal serial bus)等;
116.在一些实施例中,本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别装置可以采用软件方式实现,图2示出了存储在存储器440中的基于人工智能的蛋白质配体结合原子的识别装置443,其可以是程序和插件等形式的软件,包括以下软件模块:确定模块4431、转换模块4432、预测模块4433、筛选模块4434和训练模块4435,这些模块是逻辑上的,因此根据所实现的功能可以进行任意的组合或进一步拆分。将在下文中说明各个模块的功能。
117.下面将结合附图对本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法进行说明。以下是以服务器为基于人工智能的蛋白质配体结合原子的识别的执行主体进行说明,具体可由服务器通过运行上文的各种计算机程序来实现的;当然,根据对下文的理解,不难看出也可以由终端和服务器协同实施本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法。
118.参见图3a,图3a是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图,将结合图3a示出的步骤进行说明。
119.在步骤101中,基于待测蛋白质的三维结构数据,确定待测蛋白质在目标空间中的多个虚拟结合位点分别对应的候选配体结合原子。
120.在一些实施例中,待测蛋白质的三维数据结构是根据pdb得到的。虚拟结合位点是配体与待测蛋白质的可能的配体结合位点。参见图4,图4是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别示意图。在图4中,存在配体401和配体402,并在图4中以虚线标出了配体401识别半径范围内的配体结合原子和配体402识别半径范围内的配体结合原子。通过以口袋为中心的方法识别出虚拟结合位点403和虚拟结合位点404。在图4中以虚线标出了虚拟结合位点403对应的多个候选配体结合原子,以及虚拟结合位点404对应的多个候选配体结合原子。
121.在一些实施例中,基于待测蛋白质的三维结构数据,确定待测蛋白质在目标空间中的多个虚拟结合位点分别对应的候选配体结合原子,可采用如下方式实现:分别以配体中各个原子为球心,并以识别半径构建球体,确定由多个球体构成的目标空间;基于待测蛋白质的三维结构数据,确定待测蛋白质的空间结构信息;基于待测蛋白质的空间结构信息,
确定待测蛋白质在目标空间中的多个虚拟结合位点;确定每个虚拟结合位点分别对应的候选配体结合原子。
122.参见图5,图5是本技术实施例提供的确定候选配体结合原子的示意图。因为配体和配体结合位点在一定距离(识别半径)内,所以,当配体为小分子或聚合物时,以配体中各个原子为球心,以识别半径构建球体,可以确定出可能的配体结合原子。确定待测蛋白质的空间结构信息,即确定待测蛋白质中各个原子的空间位置。多个球体构成的目标空间501与待测蛋白质502相交的位置即为虚拟结合位点,图5中有虚拟结合位点503和虚拟结合位点504,每个虚拟结合位点都对应多个候选配体结合原子。
123.在一些可能的示例中,可通过图3b所示的步骤1011至步骤1014确定识别半径,图3b是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图。
124.在步骤1011中,以配体中各个原子为球心,依次基于多个候选识别半径构建球体,得到由同一候选识别半径构建的多个球体所构成的多个目标空间,其中,每个目标空间中的球体的候选识别半径不同。
125.例如,多个候选识别半径分别为和配体包括原子1、原子2和原子3。分别以原子1、原子2和原子3为球心,以为半径构建球体,可以得到目标空间1;分别以原子1、原子2和原子3为球心,以为半径构建球体,可以得到目标空间2;分别以原子1、原子2和原子3为球心,以为半径构建球体,可以得到目标空间3。
126.在步骤1012中,确定蛋白质样本在每个目标空间中的样本原子集合。
127.确定pdb中蛋白质样本的空间结构信息,并基于蛋白质样本的空间结构信息确定蛋白质样本与目标空间1相交的位置(即虚拟结合位点)所对应的多个样本原子,记为样本原子集合1;同样地,基于蛋白质样本与目标空间2确定样本原子集合2,基于蛋白质样本与目标空间3确定样本原子集合3。
128.在步骤1013中,确定样本原子集合中的实际配体结合原子与非配体结合原子的样本比例,确定符合预设样本比例的样本比例对应的样本原子集合。
129.因为pdb中的样本蛋白质的实际配体结合原子与非配体结合原子是已知的,所以,可以分别确定样本原子集合1、样本原子集合2和样本原子集合3中实际配体结合原子个数与非配体结合原子个数之比,即样本比例。之后,确定与预设样本比例相同或最接近的样本比例对应的样本原子集合。例如,样本原子集合1、样本原子集合2和样本原子集合3分别对应的样本比例为0.2、0.3和0.45,而预设样本比例为0.5,则将样本原子集合3作为需要的样本原子集合。
130.在步骤1014中,将确定的样本原子集合所处的目标空间对应的候选识别半径,作为识别半径。
131.根据步骤1011至步骤1013的示例,将样本原子集合3所处的目标空间3对应的候选识别半径作为识别半径。
132.在一些可能的示例中,识别半径可以是中的任意一个值。
133.在步骤102中,将三维结构数据转换为原子点云,调用神经网络模型对原子点云进行下采样处理,以得到待测蛋白质的结构特征。
134.如图4所示,图4示出了具有配体的蛋白质三维结构41、原子级别的点云42和点云分割结果43,其中,点云分割结果43是通过点云分割的以蛋白质为中心的方法得到的。从点云分割结果中可以确定预测配体结合原子44,即图4所示蛋白质中由虚线圈出的部分44。
135.在一些实施例中,因为三维结构数据包括原子的三维坐标、深度、灰度值等信息,所以可以根据三维结构数据确定待测蛋白质对应的原子点云,如图4中由蛋白质三维结构转换得到原子级别的点云。
136.在一些实施例中,神经网络模型包括j个下采样模块,j为大于或等于2的正整数;调用神经网络模型对原子点云进行下采样处理,以得到待测蛋白质的结构特征,可通过图3c所示的步骤1021至步骤1023实现,图3c是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图。
137.在步骤1021中,基于待测蛋白质的原子点云确定神经网络模型的输入特征,并作为第1个下采样模块的输入特征。
138.在一些可能的示例中,神经网络模型可以是基于子流体稀疏卷积(ssc,submanifold sparse convolutional)的u-net。首先,基于待测蛋白质的原子点云确定待测蛋白质的残基类型、原子类型和原子的空间坐标。然后,基于待测蛋白质的残基类型、原子类型和原子的空间坐标确定神经网络模型(基于ssc的u-net)的输入特征,即第1个下采样模块的输入特征。
139.在步骤1022中,通过第j个下采样模块对第j个下采样模块的输入特征进行卷积操作,得到第j个下采样模块对应的结构特征,对第j个下采样模块对应的结构特征进行池化操作,并将池化操作的结果作为第j+1个下采样模块的输入特征;其中,j为取值从1开始递增的整数变量,且取值满足1≤j≤j-1。
140.如图6所示,图6是本技术实施例提供的基于ssc的u-net的结构示意图。基于ssc的u-net包括编码器和解码器两部分,其中,编码器包括3个下采样模块,即j=3。第1个下采样模块601由输入层、卷积层和卷积块构成,第2个下采样模块602和第3个下采样模块603由卷积层和卷积块构成。其中,卷积块表示多个卷积层。
141.在第1个下采样模块601中对输入特征执行卷积操作,得到对应的结构特征,结构特征可以包括位置、颜色、深度等信息。对结构特征执行池化操作,将池化操作的结果作为输入特征输入第2个下采样模块602。通过第2个下采样模块602对池化操作的结果继续执行卷积操作和池化操作,并将池化操作的结果作为第3个下采样模块603的输入特征。
142.其中,池化操作用于提取结构特征,池化操作用于降维,从而得到逐渐减小的分辨率。
143.在图6中,对于输入层,激活点是点云中的点,对于非输入层,如果一个点的感受野中存在激活点,则该点也为激活点。非激活点是除激活点之外的点。基于ssc的u-net在执行卷积操作时,只对激活点执行卷积操作,对非激活点不执行卷积操作。如此,可以保持基于ssc的u-net中每一层的稀疏性,避免子流形扩张,并节约计算资源和内存消耗。其中,子流形表示输入特征是稀疏的,因为它的有效维数低于它所在的空间,例如二维空间中的一维曲线,或三维空间中的二维曲面。当输入特征包括具有两个或更多维度的空间中的一维曲线或者三个或更多维度中的二维表面时,随着层数的加深,激活点的数量快速增长,输入特征不再具备稀疏性,即带来子流形扩张问题。
144.由图6可以看出,随着层数的加深,激活点对应的感受野在逐渐增大,从而基于ssc的u-net可以更好地学习待测蛋白质的全局信息和几何信息。
145.在步骤1023中,通过第j个下采样模块对第j个下采样模块的输入特征进行卷积操作,得到第j个下采样模块对应的结构特征,并作为待测蛋白质的结构特征。
146.在第3个下采样模块603中,对输入特征,即第2个下采样模块602输出的池化结果进行卷积操作,得到对应的结构特征,将第3个下采样模块603对应的结构特征作为待测蛋白质的结构特征。
147.在步骤103中,基于结构特征进行上采样处理,以识别出待测蛋白质中的多个预测配体结合原子。
148.在一些实施例中,神经网络模型还包括j个上采样模块和输出模块;基于结构特征进行上采样处理,以识别出待测蛋白质中的多个预测配体结合原子,可通过图3d所示的步骤1031至步骤1036实现,图3d是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图。
149.在步骤1031中,通过第j个下采样模块对待测蛋白质的结构特征进行池化操作,并将池化操作的结果作为第1个上采样模块的输入特征。
150.在一些可能的示例中,基于ssc的u-net中的解码器还包括3个上采样模块和1个输出模块。第1个上采样模块604的输入特征是第3个下采样模块603输出的池化操作的结果。
151.在步骤1032中,通过第i个上采样模块对第i个上采样模块的输入特征进行卷积操作和上采样操作,得到第i个上采样模块的输出特征。
152.如图6所示,图6中第1个上采样模块604由卷积层、卷积块和反卷积层构成,第2个上采样模块605和第3个上采样模块606均由卷积块和反卷积层构成。以i=2为例进行说明,在第2个上采样模块605中对其输入特征进行卷积操作,从而进一步提取结构特征。之后,进行上采样操作,以将抽象的结构特征的尺寸还原解码到与第2个下采样模块602一致的尺寸。其中,上采样操作可使上采样模块对应的分辨率增加。经过上采样操作后,得到第2个上采样模块605的输出特征。
153.在步骤1033中,将第i个上采样模块的输出特征与第j+1-i个下采样模块对应的结构特征串联,并将串联结果作为第i+1个上采样模块的输入特征;其中,i为取值从1开始递增的整数变量,且取值满足1≤i≤j-1。
154.在一些可能的示例中,仍以i=2为例进行说明,将第2个上采样模块605的输出特征a与第2个下采样模块602对应的结构特征b串联,将串联结果[a,b]作为第3个上采样模块606的输入特征。
[0155]
在一些可能的示例中,步骤1033说明了基于ssc的u-net中的跳跃连接的过程。跳跃连接可将下采样模块对应的结构特征叠加到上采样模块的输入特征中,从而有助于还原下采样处理所带来的信息损失。同时,跳跃连接还可以解决网络层数较深时梯度消失的问题,有助于梯度的反向传播,加快训练过程。
[0156]
在步骤1034中,通过第j个上采样模块对第j个上采样模块的输入特征进行卷积操作和上采样操作,得到第j个上采样模块的输出特征。
[0157]
在步骤1035中,将第j个上采样模块的输出特征与第1个下采样模块对应的结构特征串联,并将串联结果作为输出模块的输入特征。
[0158]
在第3个上采样模块606中,对输入特征执行卷积操作和上采样操作,得到输出特征,将输出特征与第1个下采样模块601对应的结构特征串联,并将串联结果作为输出模块607的输入特征。
[0159]
在步骤1036中,通过输出模块对输出模块的输入特征进行线性组合和分类处理,以识别出待测蛋白质中的多个预测配体结合原子。
[0160]
在一些可能的示例中,输出模块607由线性层和分类层构成。输出模块607的线性层对输入特征进行线性组合,并通过分类层对线性组合后的特征进行分类处理,得到识别结果,识别结果包括预测配体结合原子和非预测配体结合原子。其中,分类处理可以通过softmax函数实现。
[0161]
在一些实施例中,基于ssc的u-net的训练过程如下。首先,基于蛋白质样本中各原子与配体的距离为各原子添加标签,标签的类型包括实际配体结合原子和非配体结合原子。例如,为距离小于识别半径的原子添加标签1,表示实际配体结合原子;为距离大于等于识别半径的原子添加标签0,表示非配体结合原子。
[0162]
然后,调用基于ssc的u-net对添加标签后的蛋白质样本的原子点云进行预测处理,即进行上采样处理和下采样处理,得到原子点云中每个原子的识别结果,识别结果表征相应的原子是否是配体结合原子。
[0163]
最后,基于原子点云中每个原子的标签与对应的识别结果的误差,在基于ssc的u-net中反向传播误差,以更新基于ssc的u-net的参数,从而得到训练好的基于ssc的u-net,用于蛋白质中预测配体结合原子的识别。
[0164]
在步骤104中,基于多个预测配体结合原子和每个虚拟结合位点对应的候选配体结合原子,对每个虚拟结合位点进行筛选处理,以确定与筛选后得到的虚拟结合位点对应的蛋白质配体结合原子。
[0165]
在图4中,经过筛选处理后,只有虚拟结合位点404得以保留,因为虚拟结合位点404和配体402所处位置相同,因此虚拟结合位点404识别正确。
[0166]
在一些实施例中,如图3e所示,图3e是本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别方法的流程示意图。图3e中,步骤104可以通过如下步骤1041至步骤1043实现。
[0167]
在步骤1041中,确定每个虚拟结合位点对应的多个候选配体结合原子与多个预测配体结合原子之间的公共原子。
[0168]
例如,预测配体结合原子一共有10个,虚拟结合位点e对应10个候选配体结合原子,这10个候选配体结合原子与预测配体结合原子存在2个公共原子。虚拟结合位点f对应12个候选配体结合原子,这12个候选配体结合原子与预测配体结合原子存在3个公共原子。
[0169]
在步骤1042中,确定公共原子在多个候选配体结合原子中所占的比例,以筛选得到大于比例阈值的比例对应的多个虚拟结合位点。
[0170]
其中,确定公共原子在多个候选配体结合原子中所占的比例,即确定二者的数量之间的比例。筛选得到大于比例阈值的比例对应的多个虚拟结合位点,即确定在多个候选配体结合原子中所占的比例大于比例阈值的公共原子,并确定该公共原子所属的虚拟结合位点。
[0171]
例如,对于虚拟结合位点e,2个公共原子在10个候选配体结合原子中所占的比例
为0.2;对于虚拟结合位点f,3个公共原子在12个候选配体结合原子中所占的比例为0.25。比例阈值可以是0.05、0.1、0.2等。当比例阈值为0.1时,因为0.2和0.25均大于0.1,所以虚拟结合位点e和虚拟结合位点f均未被筛选掉。
[0172]
在步骤1043中,将筛选得到的多个虚拟结合位点对应的多个候选配体结合原子作为蛋白质配体结合原子。
[0173]
例如,将虚拟结合位点e对应的10个候选配体结合原子和虚拟结合位点f对应的12个候选配体结合原子作为蛋白质配体结合原子。
[0174]
在一些实施例中,将筛选得到的多个虚拟结合位点作为蛋白质的配体结合位点。在确定蛋白质配体结合原子之后,还可以对筛选得到的多个虚拟结合位点(即蛋白质的配体结合位点)进行排序,例如,基于比例对筛选得到的多个虚拟结合位点进行降序排序,此时,虚拟结合位点f的排序在虚拟结合位点e之前。也可以基于对应的公共原子的数量对筛选得到的多个虚拟结合位点进行降序排序,此时,因为虚拟结合位点f对应的公共原子的数量(3个)比虚拟结合位点e对应的公共原子的数量(2个)更多,所以,虚拟结合位点f的排序仍在虚拟结合位点e之前。在确定虚拟结合位点的排序后,确定各个虚拟结合位点的几何中心,并输出蛋白质的配体结合位点(即上述排序后的虚拟结合位点)、对应的配体结合原子以及配体结合位点的几何中心。
[0175]
在一些实施例中,为了验证上文所确定的蛋白质配体结合原子是否识别正确,可以每个蛋白质配体结合原子为球心、以识别半径构建球体;当球体与配体相交时,确定该球体对应的蛋白质配体结合原子识别正确。
[0176]
在一些实施例中,当待测蛋白质的实际配体结合原子为已知信息时,确定筛选后得到的每个虚拟结合位点对应的蛋白质配体结合原子与实际配体结合原子的交并比。仍以虚拟结合位点e和虚拟结合位点f为例进行说明,例如,存在5个实际配体结合原子,虚拟结合位点e对应的10个蛋白质配体结合原子和5个实际配体结合原子的公共原子为4个,则对应的交并比为0.36。虚拟结合位点f对应的12个蛋白质配体结合原子和5个实际配体结合原子的公共原子为5个,则对应的交并比为0.42。
[0177]
在确定交并比之后,可基于交并比对筛选后得到的每个虚拟结合位点再次进行筛选,得到最终的虚拟结合位点(即蛋白质的配体结合位点)以及对应的蛋白质配体结合原子。例如,若设置了将交并比低于0.4的虚拟结合位点过滤掉,则虚拟结合位点e将被过滤掉,而虚拟结合位点f得以保留,虚拟结合位点f为最终的虚拟结合位点,可确定与虚拟结合位点f对应的蛋白质配体结合原子。如此,可对虚拟结合位点进一步过滤,提高所识别的配体结合位点的准确性。
[0178]
在一些实施例中,当只需要确定蛋白质配体结合原子,不需要确定配体结合位点时,也可以在将待测蛋白质转换为原子点云后,单独使用基于ssc的u-net对原子点云进行点云分割,识别出预测配体结合原子并输出。
[0179]
可以看出,本技术实施例基于待测蛋白质的多个虚拟结合位点确定候选配体结合原子,然后调用神经网络模型对待测蛋白质的原子点云进行下采样处理和上采样处理,以识别出预测配体结合原子,基于预测配体结合原子对多个虚拟结合位点进行筛选,从而可以过滤掉识别错误的虚拟结合位点对应的候选配体结合原子,识别出蛋白质配体结合原子,有效地提高了配体结合原子的识别准确率。
[0180]
下面,将说明本技术实施例在一个辅助药物设计的应用场景中识别蛋白质配体结合原子的示例性应用。
[0181]
对于给定待测蛋白质对应的pdb,需要识别所有可能的配体结合位点和配体结合原子,并以排序的方式输出每个配体结合位点的几何中心。
[0182]
存在两种用于识别蛋白质的配体结合位点的无模板方法:以口袋为中心的方法和以蛋白质为中心的方法。以口袋为中心的方法需要先确定虚拟结合位点,然后基于虚拟结合位点确定配体结合原子。以蛋白质为中心的方法直接确定蛋白质结构上的配体结合原子。
[0183]
本技术实施例提出一种点云分割方法—pointsite,并提出对应的pointsite模型,可在原子水平上进行蛋白质的配体结合位点和配体结合原子的识别,pointsite的本质是以蛋白质为中心的方法。在通过pointsite进行识别之前,需要先通过以口袋为中心的方法确定出虚拟结合位点。
[0184]
参见图7,图7是本技术实施例提供的pointsite模型的结构示意图,pointsite模型包括转换模块、预测模块和筛选模块。在转换模块中,基于待测蛋白质对应的pdb确定其三维结构数据,并将三维结构数据转换为原子点云,原子点云中每个点代表一个原子。在预测模块中,通过神经网络模型,如基于ssc的u-net对待测蛋白质的原子点云进行分割,识别出预测配体结合原子。其中,u-net的输入特征(包括待测蛋白质的残基类型、原子类型和原子的空间坐标)是基于原子点云确定的。在筛选模块中,基于预测配体结合原子对以口袋为中心的方法确定出的虚拟结合位点进行过滤和重排,从而实现精确的配体结合位点和对应的配体结合原子的识别。
[0185]
以下对pointsite模型的各个模块进行介绍。
[0186]
转换模块:给定配体数据集已知的蛋白质,配体数据集如scpdb,它是一个来自pdb的药物结合位点的注释数据库。蛋白质的所有信息都在pdb中呈现,包括蛋白质中各原子及对应的空间坐标、配体中各原子及对应的坐标、蛋白质残基等。通过转换模块将pdb转换为原子点云。转换模块的输入为pdb,输出为蛋白质中各原子的特征,包括21个残基类型(a、r、n、d、c、q、e、g、h、i、l、k、m、f、p、s、t、w、y、v、unk)、5个原子类型(c、n、o、s、unk)和原子的3个空间坐标,它们构成了一个29维向量。
[0187]
如果蛋白质的原子位于以配体的重原子为球心,以识别半径为半径的球体中,将该原子标记为实际配体结合原子(标签为1),否则标记为非配体结合原子(标签为0)。通过这种设置,蛋白质中每个原子的基本事实标签为0/1。
[0188]
识别半径的取值范围为考虑到以下三个因素,选取为识别半径。(a)适用于scpdb;(b)当识别半径为时,会考虑那些在与配体的隐含相互作用中与配体结合原子接触的原子;(c)当识别半径为时,可以缓解标签为0的原子与标签为1的原子数量不平衡的问题。
[0189]
本技术实施例还提供一种软件包lig_tool,用于生成原子级别标注,以便后续预测配体结合原子的识别。如图8所示,图8是本技术实施例提供的基于pdb生成的原子级别标注的示意图。图8中,第一列atom(原子)是标准残基的原子,后面的列依次为标准残基中各原子的原子编号、原子名称、残基名称(链标识符a)、残基编号、空间坐标(xyz坐标)、占有
率、温度因子,链标识符a、原子类型的元素符号。
[0190]
预测模块:考虑到每个蛋白质的原子数目通常超过10k,如果在体素化表示上利用传统的三维卷积神经网络模型,会引发子流形扩张。这将大大减少前一个卷积层输入特征的稀疏性和下一个卷积层的几何信息。
[0191]
更严重的是,蛋白质原子在体素化表示的整个空间中只占相对较小的比例,而空体素占较大的比例。若使用传统的三维卷积神经网络模型将会浪费大量的计算资源,并且由于内存限制不能将整个点云作为输入,这对于配体结合位点的上下文学习(获取蛋白质的结构信息)和识别极为不利。
[0192]
若使用deepsite,通过滑动窗口和下采样操作来预测蛋白质配体结合位点,因为这些操作不会将蛋白质所有的原子点云都作为输入,因此也不利于配体结合位点的上下文学习。
[0193]
相对地,ssc网络模型可以保持每个卷积层的输入特征的稀疏性,并将各点标记为激活点/非激活点(如图6所示)。因此,可以在ssc网络模型中输入完整的原子点云,同时可以考虑原子之间的连通性以及蛋白质原子和残基的物理化学特征,以便更好的学习全局上下文信息。
[0194]
受ssc和输入特征的稀疏性的启发,利用基于ssc的u-net进行配体结合原子预测。需要说明的是,ssc仅通过哈希表在三维特征映射中存储非空组件(即激活点),因此基于ssc的u-net中的卷积运算仅考虑非空组件。
[0195]
图6中,激活点以白色方块显示,而非激活点以黑色方块显示。可见,仅当内核的中心节点是激活的时,sscs输出节点才是激活的,即卷积运算仅考虑非空组件,因此不会出现子流形扩张问题,保证了后续特征映射的稀疏性。
[0196]
此外,通过基于ssc的u-net中的多尺度(多个不同的卷积核)从不同分辨率的点云中获取蛋白质的结构特征。图6中,一共有4个尺度,对应4种不同的分辨率。图6中基于ssc的u-net可以分为编码器和解码器两部分,其中,编码器包括3个下采样模块,解码器包括3个上采样模块和1个输出模块。每个下采样模块都包含一个步长为2的ssc,用于对每个下采样模块的输入特征进行卷积操作和池化操作。在解码器中使用跳跃连接,将上采样模块对其输入特征进行卷积操作和上采样操作后得到的输出特征与编码器中具有相同分辨率的下采样模块输出的结构特征进行连接,作为解码器中下一个模块的输入特征。最后,解码器输出预测配体结合原子。可见,基于ssc的u-net的这种结构不仅可以将所有的原子点云作为有效输入,而且可以捕捉到更好的全局上下文和蛋白质结构的几何信息。
[0197]
可以看出,图6中,随着层数的加深,感受野也在逐渐扩大,基于ssc的u-net可以通过逐渐扩大的感受野和跳跃连接来学习蛋白质的全局和几何信息,这反过来可以帮助捕获蛋白质的三维结构和配体结合原子彼此间的复杂关系。
[0198]
此外,本技术实施例中的pointsite是一种数据驱动方法,它可以有效地学习整个数据库的特征。一旦pointsite模型被训练出来了,蛋白质的配体结合原子的识别时间将很短,实际中不到1秒,并且整个包是轻量级的。
[0199]
筛选模块:以口袋为中心的方法通常需要额外的信息(例如,蛋白质结构外部的口袋的几何信息和/或能量特征)为识别下游的配体结合位点定义虚拟结合位点,而pointsite利用的是蛋白质结构中原子的固有信息。因此,可以结合以口袋为中心的方法和
以蛋白质为中心的pointsite,来识别出蛋白质的配体结合位点,并对配体结合位点排序。
[0200]
以口袋为中心的方法,如fpocket或sitebound等,其输出是虚拟结合位点的排序列表,排序列表中的虚拟结合位点需要满足相应标准,如需要具有高相互作用能量。在通过以口袋为中心的方法得到包括k个虚拟结合位点的排序列表后,需要基于预测配体结合原子对k个虚拟结合位点进行过滤筛选,并确定与筛选得到的虚拟结合位点的距离在识别半径内的配体结合原子。对于第m个候选虚拟结合位点,通过如下公式(1)进行过滤:
[0201]
ratiom=|sba∩ibam|1/|ibam|1ꢀꢀꢀ
(1)
[0202]
其中,m为大于等于1且小于等于k的正整数,k为大于1的正整数,||1表示计数,sba代表预测配体结合原子,ibam代表第m个虚拟结合位点对应的配体结合原子,|sba∩ibam|1代表第m个虚拟结合位点对应的配体结合原子与预测配体结合原子公共的原子的个数。ratiom代表公共原子在第m个虚拟结合位点对应的配体结合原子中所占的比例。如果ratiom小于比例阈值(如0.1),则第m个虚拟结合位点将被过滤掉。过滤后,k个虚拟结合位点将减少为k1个虚拟结合位点。将k1个虚拟结合位点作为蛋白质的配体结合位点。基于k1个虚拟结合位点可确定与之对应的配体结合原子。对k1个虚拟结合位点根据对应的ratiom的大小进行排序。
[0203]
在基于上述筛选得到k1个虚拟结合位点的基础上,还可基于公共的原子的个数再次进行排序,即根据第n个虚拟结合位点对应的|sba∩iban|1的大小对k1个虚拟结合位点进行排序,其中,n为大于等于1且小于等于k1的正整数。确定排序后的k1个虚拟结合位点各自的几何中心,输出每个虚拟结合位点的几何中心。
[0204]
这种对虚拟结合位点过滤排序的方法不仅满足dca标准,即保证配体结合位点和配体的距离在一定的范围内,还能提高识别出的配体结合位点对应的交并比。
[0205]
参见表1,表1显示了fpocket、sitehound、metapocket2、deepsite、p2rank和本技术实施例提供的pointsite分别在b277数据集、dt198数据集、astex85数据集、chen251数据集、coach420数据集和holo4k数据集上的交并比数据。
[0206][0207][0208]
表1根据不同算法得到的配体结合位点对应的交并比
[0209]
其中,b277是包含277种处于结合状态的蛋白质的数据集,dt198是包含198种药物-靶标复合物的数据集,astex85是包括85个条目的数据集,chen251是包含251个条目的数据集,coach420是包含420种蛋白质的数据集,holo4k是包含4543个蛋白质-配体复合物的大型数据集。
[0210]
从表1中可以看出,本技术实施例提供的pointsite在表1中除chen251数据集以外的数据集中的交并比均高于其他数据集,且在chen251数据集中的交并比仅次于p2rank,表
明本技术实施例提供的pointsite可以大大提高配体结合位点和配体结合原子识别的准确性。
[0211]
在配体结合位点识别方面,真正的挑战是在没有结合配体的蛋白质结构中识别出配体结合位点。因此,本技术实施例通过对具有配体的蛋白质进行训练,得到基于ssc的u-net,通过基于ssc的u-net对蛋白质的原子点云进行分割,识别出预测配体结合原子,基于预测配体结合原子可对未结合配体的蛋白质中的配体结合位点进行精确的识别。
[0212]
此外,不管是对于具有原始结构的蛋白质三维结构(通过实验测量得到蛋白质三维结构),还是对于从头预测的蛋白质三维结构(蛋白质三维结构未知,需要通过计算生物学计算得到蛋白质三维结构),本技术实施例都可以准确地识别出配体结合原子,这对于结合原子辅助的小分子(配体)的设计提供了必要的帮助。对于cameo中的困难目标,如折叠的蛋白质,pointsite同样可以保持稳定的性能,可见,pointsite具有强大的泛化能力。
[0213]
在一些可能的示例中,pointsite还可以检测出蛋白质中与抗病毒药物(配体)结合的配体结合位点,从而帮助实现抗病毒药物的提前“占位”,避免病毒与蛋白质的结合。
[0214]
可以看出,本技术实施例通过细粒度的原子级别的配体结合原子表示和通过稀疏卷积得到的增强的特征,可使得最终得到的配体结合位点对应的交并比远远高于相关技术中的交并比,证明了pointsite可实现对配体结合位点和配体结合原子精确的识别。对于相关技术中以口袋为中心的方法得到的虚拟结合位点,本技术实施例提出的pointsite可以作为筛选工具,通过过滤掉与预测配体结合原子缺乏公共原子的虚拟结合位点,以此显著降低配体结合位点的假阳性(将非配体结合位点检测为配体结合位点)。
[0215]
通过以口袋为中心的方法确定出虚拟结合位点,通过pointsite对虚拟结合位点进行过滤,还可基于交并比进一步对虚拟结合位点进行过滤,得到最终的配体结合位点,如此,pointsite在各种标准基准(如dca标准)、cameo中困难示例以及未结合配体的蛋白质上均实现了最佳结果。
[0216]
下面继续说明本技术实施例提供的基于人工智能的蛋白质配体结合原子的识别装置443实施为软件模块的示例性结构。在一些实施例中,如图2所示,存储在存储器440的基于人工智能的蛋白质配体结合原子的识别装置443中的软件模块可以包括:确定模块4431、转换模块4432、预测模块4433、筛选模块4434和训练模块4435。
[0217]
确定模块4431,用于基于待测蛋白质的三维结构数据,确定待测蛋白质在目标空间中的多个虚拟结合位点分别对应的候选配体结合原子。转换模块4432,用于将三维结构数据转换为原子点云。预测模块4433,用于调用神经网络模型对原子点云进行下采样处理,以得到待测蛋白质的结构特征,以及用于基于结构特征进行上采样处理,以识别出待测蛋白质中的多个预测配体结合原子。筛选模块4434,用于基于多个预测配体结合原子和每个虚拟结合位点对应的候选配体结合原子,对每个虚拟结合位点进行筛选处理,以确定与筛选后得到的虚拟结合位点对应的蛋白质配体结合原子。
[0218]
在一些实施例中,确定模块4431,还用于分别以配体中各个原子为球心,并以识别半径构建球体,确定由多个球体构成的目标空间;基于待测蛋白质的三维结构数据,确定待测蛋白质的空间结构信息;基于待测蛋白质的空间结构信息,确定待测蛋白质在目标空间中的多个虚拟结合位点;确定每个虚拟结合位点分别对应的候选配体结合原子。
[0219]
在一些实施例中,确定模块4431,还用于以配体中各个原子为球心,依次基于多个
候选识别半径构建球体,得到由同一候选识别半径构建的多个球体所构成的多个目标空间,其中,每个目标空间中的球体的候选识别半径不同;确定蛋白质样本在每个目标空间中的样本原子集合;确定样本原子集合中的实际配体结合原子与非配体结合原子的样本比例,确定符合预设样本比例的样本比例对应的样本原子集合;将确定的样本原子集合所处的目标空间对应的候选识别半径,作为识别半径。
[0220]
在一些实施例中,神经网络模型包括j个下采样模块,j为大于或等于2的正整数;预测模块4433,还用于基于待测蛋白质的原子点云确定神经网络模型的输入特征,并作为第1个下采样模块的输入特征;通过第j个下采样模块对第j个下采样模块的输入特征进行卷积操作,得到第j个下采样模块对应的结构特征,对第j个下采样模块对应的结构特征进行池化操作,并将池化操作的结果作为第j+1个下采样模块的输入特征;其中,j为取值从1开始递增的整数变量,且取值满足1≤j≤j-1;通过第j个下采样模块对第j个下采样模块的输入特征进行卷积操作,得到第j个下采样模块对应的结构特征,并作为待测蛋白质的结构特征。
[0221]
在一些实施例中,预测模块4433,还用于基于待测蛋白质的原子点云确定待测蛋白质的残基类型、原子类型和原子的空间坐标;基于待测蛋白质的残基类型、原子类型和原子的空间坐标确定神经网络模型的输入特征。
[0222]
在一些实施例中,神经网络模型还包括j个上采样模块和输出模块,预测模块4433,还用于通过第j个下采样模块对待测蛋白质的结构特征进行池化操作,并将池化操作的结果作为第1个上采样模块的输入特征;通过第i个上采样模块对第i个上采样模块的输入特征进行卷积操作和上采样操作,得到第i个上采样模块的输出特征;将第i个上采样模块的输出特征与第j+1-i个下采样模块对应的结构特征串联,并将串联结果作为第i+1个上采样模块的输入特征;其中,i为取值从1开始递增的整数变量,且取值满足1≤i≤j-1;通过第j个上采样模块对第j个上采样模块的输入特征进行卷积操作和上采样操作,得到第j个上采样模块的输出特征;将第j个上采样模块的输出特征与第1个下采样模块对应的结构特征串联,并将串联结果作为输出模块的输入特征;通过输出模块对输出模块的输入特征进行线性组合和分类处理,以识别出待测蛋白质中的多个预测配体结合原子。
[0223]
在一些实施例中,筛选模块4434,还用于确定每个虚拟结合位点对应的多个候选配体结合原子与多个预测配体结合原子之间的公共原子;确定公共原子在多个候选配体结合原子中所占的比例,以筛选得到大于比例阈值的比例对应的多个虚拟结合位点;将筛选得到的多个虚拟结合位点对应的多个候选配体结合原子作为蛋白质配体结合原子。
[0224]
在一些实施例中,筛选模块4434,还用于对筛选得到的多个虚拟结合位点进行排序;确定排序后的多个虚拟结合位点的几何中心。
[0225]
在一些实施例中,筛选模块4434,还用于基于比例对筛选得到的多个虚拟结合位点进行降序排序,或基于对应的公共原子的数量对筛选得到的多个虚拟结合位点进行降序排序。
[0226]
在一些实施例中,筛选模块4434,还用于以每个蛋白质配体结合原子为球心、以识别半径构建球体;当球体与配体相交时,确定球体对应的蛋白质配体结合原子识别正确。
[0227]
在一些实施例中,当待测蛋白质的实际配体结合原子为已知信息时,筛选模块4434,还用于确定筛选后得到的每个虚拟结合位点对应的蛋白质配体结合原子与实际配体
结合原子的交并比;基于交并比对筛选后得到的每个虚拟结合位点再次进行筛选,得到最终的虚拟结合位点以及对应的蛋白质配体结合原子。
[0228]
在一些实施例中,基于人工智能的蛋白质配体结合原子的识别装置443还包括训练模块4435,用于基于蛋白质样本中各原子与配体的距离为各原子添加标签,标签的类型包括实际配体结合原子和非配体结合原子;调用神经网络模型对添加标签后的蛋白质样本的原子点云进行预测处理,得到原子点云中每个原子的识别结果,识别结果表征相应的原子是否是配体结合原子;基于原子点云中每个原子的标签与对应的识别结果的误差,在神经网络模型中反向传播,以更新神经网络模型的参数。
[0229]
本技术实施例提供一种存储有可执行指令的计算机可读存储介质,其中存储有可执行指令,当可执行指令被处理器执行时,将引起处理器执行本技术实施例提供的方法,例如,如图3a示出的基于人工智能的蛋白质配体结合原子的识别方法。
[0230]
在一些实施例中,计算机可读存储介质可以是fram、rom、prom、eprom、eeprom、闪存、磁表面存储器、光盘、或cd-rom等存储器;也可以是包括上述存储器之一或任意组合的各种设备。
[0231]
在一些实施例中,可执行指令可以采用程序、软件、软件模块、脚本或代码的形式,按任意形式的编程语言(包括编译或解释语言,或者声明性或过程性语言)来编写,并且其可按任意形式部署,包括被部署为独立的程序或者被部署为模块、组件、子例程或者适合在计算环境中使用的其它单元。
[0232]
作为示例,可执行指令可以但不一定对应于文件系统中的文件,可以可被存储在保存其它程序或数据的文件的一部分,例如,存储在超文本标记语言(html,hyper text markup language)文档中的一个或多个脚本中,存储在专用于所讨论的程序的单个文件中,或者,存储在多个协同文件(例如,存储一个或多个模块、子程序或代码部分的文件)中。
[0233]
作为示例,可执行指令可被部署为在一个计算设备上执行,或者在位于一个地点的多个计算设备上执行,又或者,在分布在多个地点且通过通信网络互连的多个计算设备上执行。
[0234]
综上所述,本技术实施例基于待测蛋白质的多个虚拟结合位点确定候选配体结合原子,然后调用神经网络模型对待测蛋白质的原子点云进行下采样处理和上采样处理,以识别出预测配体结合原子,基于预测配体结合原子对多个虚拟结合位点进行筛选,从而可以过滤掉识别错误的虚拟结合位点对应的候选配体结合原子,识别出蛋白质配体结合原子。
[0235]
以上所述,仅为本技术的实施例而已,并非用于限定本技术的保护范围。凡在本技术的精神和范围之内所作的任何修改、等同替换和改进等,均包含在本技术的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1