蛋白质结构预测模型的训练方法和蛋白质结构预测方法与流程
1.本技术实施例涉及计算机技术领域,尤其涉及一种蛋白质结构预测模型的训练方法和蛋白质结构预测方法。
背景技术:
2.对蛋白质结构进行预测可以有助于了解蛋白质的作用,了解蛋白质如何行使其生物功能,以及认识蛋白质与蛋白质或其它分子之间的相互作用,这无论是对于生物学还是对于医学和药学,都是非常重要的。而蛋白质结构预测通常需要先对蛋白质二级结构进行预测,进而根据确定出的蛋白质二级结构得到完整的蛋白质结构。
3.蛋白质结构预测的预测精度,尤其是蛋白质二级结构预测的预测精度,取决于特征表征矩阵的质量,特征表征矩阵为用来表征蛋白质的氨基酸序列的特定位置的评分矩阵。特征表征矩阵的质量越高,蛋白质结构预测的预测精度就越高。然而,一些蛋白质不具有高质量的特征表征矩阵,其特征表征矩阵的质量较低,而目前利用低质量的特征表征矩阵进行蛋白质结构预测,预测精度较低。
技术实现要素:
4.为解决相关技术中存在的技术问题,本技术实施例提供一种蛋白质结构预测模型的训练方法和蛋白质结构预测方法,可以提高蛋白质结构预测的预测精度。
5.为达到上述目的,本技术实施例的技术方案是这样实现的:
6.第一方面,本技术实施例提供了一种蛋白质结构预测模型的训练方法,所述方法包括:
7.获取包含多个氨基酸序列样本的训练数据集;每个氨基酸序列样本的第一表征矩阵是根据关联的同源序列集合确定的,且所述同源序列集合中的同源序列的数量不小于设定阈值;每个氨基酸序列样本的第二表征矩阵是根据关联的同源序列集合的下采样结果确定的;
8.基于所述训练数据集,对所述蛋白质结构预测模型包含的特征增强网络和结构预测网络进行迭代训练,直到满足设定的收敛条件为止,其中,一次迭代训练过程包括:
9.基于从所述训练数据集抽取的氨基酸序列样本以及对应的第二表征矩阵,通过所述特征增强网络,得到相应的增强样本表征矩阵,并根据所述增强样本表征矩阵与所述氨基酸序列样本的第一表征矩阵,确定第一损失值;
10.将所述增强样本表征矩阵和所述氨基酸序列样本进行拼接后分别输入所述结构预测网络和用于辅助训练的分类网络,并基于获得的输出结果,确定第二损失值;
11.根据所述第一损失值和所述第二损失值,分别对所述特征增强网络和所述结构预测网络进行参数调整。
12.第二方面,本技术实施例还提供了一种蛋白质结构预测方法,所述方法包括:
13.将待处理蛋白质对应的蛋白质信息矩阵输入已训练的蛋白质结构预测模型,得到
所述待处理蛋白质中的各个氨基酸所属的设定结构类别;所述蛋白质结构预测模型是通过第一方面中任一项所述的训练方法得到的;所述蛋白质信息矩阵是基于所述待处理蛋白质的氨基酸序列获得的;
14.根据所述各个氨基酸所属的设定结构类别,确定所述待处理蛋白质的结构。
15.第三方面,本技术实施例还提供了一种蛋白质结构预测模型的训练装置,所述装置包括:
16.数据获取单元,用于获取包含多个氨基酸序列样本的训练数据集;每个氨基酸序列样本的第一表征矩阵是根据关联的同源序列集合确定的,且所述同源序列集合中的同源序列的数量不小于设定阈值;每个氨基酸序列样本的第二表征矩阵是根据关联的同源序列集合的下采样结果确定的;
17.模型训练单元,用于基于所述训练数据集,对所述蛋白质结构预测模型包含的特征增强网络和结构预测网络进行迭代训练,直到满足设定的收敛条件为止,其中,一次迭代训练过程包括:基于从所述训练数据集抽取的氨基酸序列样本以及对应的第二表征矩阵,通过所述特征增强网络,得到相应的增强样本表征矩阵,并根据所述增强样本表征矩阵与所述氨基酸序列样本的第一表征矩阵,确定第一损失值;将所述增强样本表征矩阵和所述氨基酸序列样本进行拼接后分别输入所述结构预测网络和用于辅助训练的分类网络,并基于获得的输出结果,确定第二损失值;根据所述第一损失值和所述第二损失值,分别对所述特征增强网络和所述结构预测网络进行参数调整。
18.在一种可选的实施例中,所述数据获取单元,具体用于:
19.基于所述氨基酸序列样本,从氨基酸序列数据库中查找所述氨基酸序列样本对应的多个同源序列,并将所述多个同源序列组成所述氨基酸序列样本关联的同源序列集合;
20.对所述氨基酸序列样本关联的同源序列集合进行下采样,得到同源序列集合的下采样结果;
21.根据所述同源序列集合的下采样结果,确定所述氨基酸序列样本的第二表征矩阵。
22.在一种可选的实施例中,所述模型训练单元,具体用于:
23.将所述氨基酸序列样本输入矩阵生成网络,得到所述氨基酸序列样本的初始样本表征矩阵;所述矩阵生成网络是基于自然语言处理模型训练得到的;
24.将所述氨基酸序列样本、所述氨基酸序列样本的第二表征矩阵和所述初始样本表征矩阵进行拼接,得到第一蛋白质信息样本矩阵;
25.将所述第一蛋白质信息样本矩阵输入所述特征增强网络,得到所述氨基酸序列样本对应的增强样本表征矩阵。
26.在一种可选的实施例中,所述模型训练单元,还用于:
27.将所述增强样本表征矩阵和所述氨基酸序列样本进行拼接,得到第二蛋白质信息样本矩阵;
28.将所述第二蛋白质信息样本矩阵输入所述结构预测网络,得到所述氨基酸序列样本对应的结构预测数据;
29.将所述第二蛋白质信息样本矩阵输入所述分类网络,得到所述氨基酸序列样本对应的结构预测参考数据;
30.将所述结构预测数据与所述结构预测参考数据进行比对,确定所述第二损失值。
31.在一种可选的实施例中,所述模型训练单元,还用于:
32.将所述氨基酸序列样本和所述氨基酸序列样本的第一表征矩阵进行拼接,得到第一辅助训练信息矩阵,并将所述第一辅助训练信息矩阵输入所述分类网络,得到所述氨基酸序列样本对应的第一辅助结构预测数据;
33.将所述氨基酸序列样本和所述氨基酸序列样本的第二表征矩阵进行拼接,得到第二辅助训练信息矩阵,并将所述第二辅助训练信息矩阵输入所述分类网络,得到所述氨基酸序列样本对应的第二辅助结构预测数据;
34.将所述氨基酸序列样本和所述氨基酸序列样本的初始样本表征矩阵进行拼接,得到第三辅助训练信息矩阵,并将所述第三辅助训练信息矩阵输入所述分类网络,得到所述氨基酸序列样本对应的第三辅助结构预测数据;
35.基于所述结构预测参考数据、所述第一辅助结构预测数据、所述第二辅助结构预测数据和所述第三辅助结构预测数据,确定第三损失值;
36.根据所述第一损失值、所述第二损失值和所述第三损失值,对所述特征增强网络和所述结构预测网络进行参数调整。
37.在一种可选的实施例中,所述模型训练单元,还用于:
38.根据所述第一损失值、所述第二损失值和所述第三损失值的加权和,确定联合损失值;
39.根据所述联合损失值,对所述特征增强网络和所述结构预测网络进行参数调整。
40.第四方面,本技术实施例还提供了一种蛋白质结构预测装置,所述装置包括:
41.类别确定单元,用于将待处理蛋白质对应的蛋白质信息矩阵输入已训练的蛋白质结构预测模型,得到所述待处理蛋白质中的各个氨基酸所属的设定结构类别;所述蛋白质结构预测模型是通过第一方面中任一项所述的训练方法得到的;所述蛋白质信息矩阵是基于所述待处理蛋白质的氨基酸序列获得的;
42.结构确定单元,用于根据所述各个氨基酸所属的设定结构类别,确定所述待处理蛋白质的结构。
43.在一种可选的实施例中,所述装置还包括矩阵生成单元,用于:
44.将所述氨基酸序列输入矩阵生成网络,基于所述矩阵生成网络对所述氨基酸序列进行同源信息表达,得到所述待处理蛋白质的初始同源信息表征矩阵;所述矩阵生成网络是基于自然语言处理模型训练得到的;
45.将所述氨基酸序列和所述初始同源信息表征矩阵进行拼接,得到所述待处理蛋白质对应的蛋白质信息矩阵。
46.在一种可选的实施例中,所述类别确定单元,具体用于:
47.将待处理蛋白质对应的蛋白质信息矩阵输入所述蛋白质结构预测模型的特征增强网络,基于所述特征增强网络确定所述待处理蛋白质对应的增强同源信息表征矩阵;
48.将所述氨基酸序列和所述增强同源信息表征矩阵进行拼接,得到增强蛋白质信息矩阵;
49.将所述增强蛋白质信息矩阵输入所述蛋白质结构预测模型的结构预测网络,基于所述结构预测网络确定所述待处理蛋白质中的各个氨基酸所属的设定结构类别。
50.在一种可选的实施例中,所述特征增强网络包括卷积子网络、长短期记忆子网络、拼接层和至少一个全连接层;所述类别确定单元,还用于:
51.将所述蛋白质信息矩阵输入所述卷积子网络,基于所述卷积子网络获得所述蛋白质信息矩阵的基础特征;
52.将所述蛋白质信息矩阵输入所述长短期记忆子网络,基于所述长短期记忆子网络获得所述蛋白质信息矩阵的时序特征;
53.将所述基础特征和所述时序特征输入所述拼接层,基于所述拼接层对所述基础特征和所述时序特征进行拼接,得到合成特征;
54.将所述合成特征输入所述全连接层,基于所述全连接层得到所述待处理蛋白质对应的增强同源信息表征矩阵。
55.在一种可选的实施例中,所述结构预测网络包括卷积子网络、长短期记忆子网络、拼接层和多分类层;所述类别确定单元,还用于:
56.将所述增强蛋白质信息矩阵输入所述卷积子网络,基于所述卷积子网络获得所述增强蛋白质信息矩阵的基础特征;
57.将所述增强蛋白质信息矩阵输入所述长短期记忆子网络,基于所述长短期记忆子网络获得所述增强蛋白质信息矩阵的时序特征;
58.将所述基础特征和所述时序特征输入所述拼接层,基于所述拼接层对所述基础特征和所述时序特征进行拼接,得到合成特征;
59.将所述合成特征输入所述多分类层,基于所述多分类层得到所述待处理蛋白质中的各个氨基酸所属的设定结构类别。
60.第五方面,本技术实施例还提供了一种计算机可读存储介质,所述计算机可读存储介质内存储有计算机程序,所述计算机程序被处理器执行时,实现第一方面的蛋白质结构预测模型的训练方法。
61.第六方面,本技术实施例还提供了一种计算机可读存储介质,所述计算机可读存储介质内存储有计算机程序,所述计算机程序被处理器执行时,实现第二方面的蛋白质结构预测方法。
62.第七方面,本技术实施例还提供了一种电子设备,包括存储器和处理器,所述存储器上存储有可在所述处理器上运行的计算机程序,当所述计算机程序被所述处理器执行时,使得所述处理器实现第一方面的蛋白质结构预测模型的训练方法。
63.第八方面,本技术实施例还提供了一种电子设备,包括存储器和处理器,所述存储器上存储有可在所述处理器上运行的计算机程序,当所述计算机程序被所述处理器执行时,使得所述处理器实现第二方面的蛋白质结构预测方法。
64.本技术实施例提供的蛋白质结构预测模型的训练方法和蛋白质结构预测方法,训练数据集中的每个氨基酸序列样本均具有第一表征矩阵和第二表征矩阵,第一表征矩阵是根据氨基酸序列样本关联的同源序列集合确定的高质量的特征表征矩阵,第二表征矩阵是根据该同源序列集合的下采样结果确定的低质量的特征表征矩阵。本技术实施例基于各个氨基酸序列样本的第一表征矩阵和第二表征矩阵,并采用知识蒸馏的方法,借助辅助训练的分类网络对蛋白质结构预测模型包含的特征增强网络和结构预测网络进行联合训练,得到的蛋白质结构预测模型,可以基于待处理蛋白质的低质量的特征表征矩阵,对待处理蛋
白质的结构进行预测,获得精度较高的蛋白质结果预测结果,提高蛋白质结构预测的预测精度。
附图说明
65.为了更清楚地说明本技术实施例中的技术方案,下面将对实施例描述中所需要使用的附图作简要介绍,显而易见地,下面描述中的附图仅仅是本技术的一些实施例,对于本领域的普通技术人员来讲,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。
66.图1为本技术实施例提供的一种蛋白质结构预测模型的训练方法的应用场景图;
67.图2为本技术实施例提供的一种蛋白质结构预测模型的训练方法的流程示意图;
68.图3为本技术实施例提供的另一种蛋白质结构预测模型的训练方法的流程示意图;
69.图4为本技术实施例提供的另一种蛋白质结构预测模型的训练方法的流程示意图;
70.图5为本技术实施例提供的一种蛋白质结构预测方法的流程示意图;
71.图6为本技术实施例提供的另一种蛋白质结构预测方法的流程示意图;
72.图7为本技术实施例提供的一种特征增强网络的结构示意图;
73.图8为本技术实施例提供的一种结构预测网络的结构示意图;
74.图9为本技术实施例提供的一种蛋白质结构预测方法与相关方法的性能效果对比柱状图;
75.图10为本技术实施例提供的一种蛋白质结构预测模型的训练装置的结构框图;
76.图11为本技术实施例提供的一种蛋白质结构预测装置的结构框图;
77.图12为本技术实施例提供的另一种蛋白质结构预测装置的结构框图;
78.图13为本技术实施例提供的一种电子设备的结构示意图。
具体实施方式
79.为了使本技术的目的、技术方案和优点更加清楚,下面将结合附图对本技术作进一步地详细描述,显然,所描述的实施例仅仅是本技术一部分实施例,而不是全部的实施例。基于本技术中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其它实施例,都属于本技术保护的范围。
80.需要说明的是,本技术的文件中涉及的术语“包括”和“具有”以及它们的变形,意图在于覆盖不排他的包含,例如,包含了一系列步骤或单元的过程、方法、系统、产品或设备不必限于清楚地列出的那些步骤或单元,而是可包括没有清楚地列出的或对于这些过程、方法、产品或设备固有的其它步骤或单元。
81.以下对本技术实施例中的部分用语进行解释说明,以便于本领域技术人员理解。
82.(1)蛋白质二级结构预测(protein secondary structure prediction,pssp):通常被认为是蛋白结构预测的第一步,可以是一个序列标注问题。一条蛋白质序列由氨基酸链组成,每个位置可以是20个基础氨基酸中的任意一种,pssp的作用是将每个位置的标签预测出来。
83.(2)多序列对准(multiple sequence alignment,msa):把两个以上的氨基酸序列进行对齐,逐列比较每个序列所包含的氨基酸的异同,使得参与比对的氨基酸序列有尽可能多的列具有相同的氨基酸,并且相同残基的位点位于同一列,以便发现不同序列之间的相似部分,从而推测这些氨基酸序列在结构和功能上面的相似关系。
84.(3)特定位置的评分矩阵(position-specific scoring matrix,pssm):可以通过多序列对准msa计算得到,并且pssm的质量可以决定pssp的精度,当pssm的质量越高时,pssp的精度就越高,当pssm的质量越低时,pssp的精度也就越低。
85.(4)知识蒸馏(knowledge distillation,kd):通过将老师模型学习到的知识转移给学生网络的方式来提升学生网络的性能。
86.(5)对比学习(contrastive learning,cl):通过对正负样本的学习来增加不同类别样本的类间距离减少同类样本之间的类内距离。
87.下文中所用的词语“示例性”的意思为“用作例子、实施例或说明性”。作为“示例性”所说明的任何实施例不必解释为优于或好于其它实施例。
88.文中的术语“第一”、“第二”仅用于描述目的,而不能理解为明示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者隐含地包括一个或者更多个该特征,在本技术实施例的描述中,除非另有说明,“多个”的含义是两个或两个以上。
89.本技术实施例涉及人工智能(artificial intelligence,ai)和机器学习技术,基于人工智能中的计算机视觉(computer vision,cv)技术、语音处理技术(speech technology)和机器学习(machine learning,ml)而设计。
90.人工智能是利用数字计算机或者数字计算机控制的机器模拟、延伸和扩展人的智能,感知环境、获取知识并使用知识获得最佳结果的理论、方法、技术及应用系统。换句话说,人工智能是计算机科学的一个综合技术,它企图了解智能的实质,并生产出一种新的能以人类智能相似的方式做出反应的智能机器。人工智能也就是研究各种智能机器的设计原理与实现方法,使机器具有感知、推理与决策的功能。人工智能技术主要包括计算机视觉技术、语音处理技术、以及机器学习/深度学习等几大方向。
91.随着人工智能技术研究和进步,人工智能在多个领域展开研究和应用,例如常见的智能家居、图像检索、视频监控、智能音箱、智能营销、无人驾驶、自动驾驶、无人机、机器人、智能医疗等,相信随着技术的发展,人工智能将在更多的领域得到应用,并发挥越来越重要的价值。
92.机器学习是一门多领域交叉学科,涉及概率论、统计学、逼近论、凸分析、算法复杂度理论等多门学科。专门研究计算机怎样模拟或实现人类的学习行为,以获取新的知识或技能,重新组织已有的知识结构使之不断改善自身的性能。机器学习是人工智能的核心,是使计算机具有智能的根本途径,其应用遍及人工智能的各个领域。机器学习和深度学习通常包括人工神经网络、置信网络、强化学习、迁移学习、归纳学习等技术。本技术实施例通过对基于机器学习或深度学习的蛋白质结构预测模型进行训练,得到已训练的蛋白质结构预测模型,并在蛋白质结构预测过程中,通过基于机器学习或深度学习的蛋白质结构预测模型确定待处理蛋白质中的各个氨基酸所属的设定结构类别。
93.为更好地理解本技术实施例提供的技术方案,下面对本技术实施例提供的技术方
案适用的应用场景做一些简单介绍,需要说明的是,以下介绍的应用场景仅用于说明本技术实施例而非限定。在具体实施时,可以根据实际需要灵活地应用本技术实施例提供的技术方案。
94.本技术实施例所提供的蛋白质结构预测方法可以应用于图1示出的应用场景中。参见图1所示,该应用场景中包括多个终端设备11和服务器12。终端设备11与服务器12之间可以通过有线连接方式或无线连接方式进行连接并传输数据。例如,终端设备11与服务器12可以通过数据线连接或者通过有线网络连接;终端设备11与服务器12也可以通过射频模块、wifi模块或者无线网络连接。
95.其中,终端设备11可以是计算机、笔记本、掌上电脑(personal digital assistant,pda)、平板电脑等。服务器12可以是一台服务器或由若干台服务器组成的服务器集群或云计算中心,或者是一个虚拟化平台,也可以是个人计算机、大中型计算机或计算机集群等。根据实现需要,本技术实施例中的应用场景中可以具有任意数目的终端设备和服务器。本技术对此不做特殊限定。本技术实施例提供的蛋白质结构预测方法和蛋白质结构预测模型的训练方法可以由服务器12执行,也可以由终端设备11和服务器12协作执行。
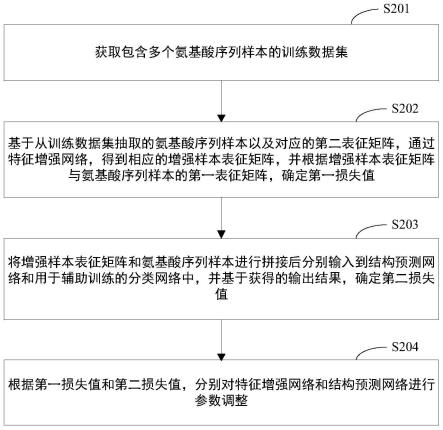
96.例如,某个医疗研究机构设置有一台服务器12,用于对蛋白质结构预测模型进行训练和预测蛋白质结构。在该医疗研究机构内部的各个实验室均设置有终端设备11。研究人员可以先采用包含多个氨基酸序列样本的训练数据集对服务器12上的蛋白质结构预测模型进行训练,得到已训练的蛋白质结构预测模型。在得到已训练的蛋白质结构预测模型后,可以通过自己实验室的终端设备11将获取到的待处理蛋白质的氨基酸序列传输至服务器12,服务器12在接收到氨基酸序列后,可以先根据该氨基酸序列确定出待处理蛋白质对应的蛋白质信息矩阵,再将蛋白质信息矩阵输入已训练的蛋白质结构预测模型,得到待处理蛋白质中的各个氨基酸所属的设定结构类别,进而可以根据各个氨基酸所属的设定结构类别,确定出待处理蛋白质的结构。
97.为进一步说明本技术实施例提供的技术方案,下面结合附图以及具体实施方式对此进行详细的说明。虽然本技术实施例提供了如下述实施例或附图所示的方法操作步骤,但基于常规或者无需创造性的劳动在所述方法中可以包括更多或者更少的操作步骤。在逻辑上不存在必要因果关系的步骤中,这些步骤的执行顺序不限于本技术实施例提供的执行顺序。所述方法在实际的处理过程中或者装置执行时,可按照实施例或者附图所示的方法顺序执行或者并行执行。
98.图2示出了本技术实施例提供的一种蛋白质结构预测模型的训练方法的流程图,该方法可以由图1中的服务器12执行,也可以由终端设备11或其它电子设备执行。示例性地,下文以用于训练蛋白质结构预测模型的计算机作为执行主体,说明本技术实施例的蛋白质结构预测模型的训练方法的具体实现过程。如图2所示,该蛋白质结构预测模型的训练方法包括如下步骤:
99.步骤s201,获取包含多个氨基酸序列样本的训练数据集。
100.获取到的训练数据集中可以包括多个氨基酸序列样本,每个氨基酸序列样本的第一表征矩阵是根据关联的同源序列集合确定的,并且同源序列集合中的同源序列的数量不小于设定阈值。此外,每个氨基酸序列样本的第二表征矩阵是根据关联的同源序列集合的下采样结果确定的。
101.基于该训练数据集,可以对蛋白质结构预测模型包含的特征增强网络和结构预测网络进行迭代训练,直到满足设定的收敛条件为止,其中,一次迭代训练过程可以包括步骤s202至步骤s204。
102.在一种实施例中,可以先基于氨基酸序列样本,从氨基酸序列数据库中查找氨基酸序列样本对应的多个同源序列,并将多个同源序列组成氨基酸序列样本关联的同源序列集合。再对氨基酸序列样本关联的同源序列集合进行下采样,得到同源序列集合的下采样结果。最后根据同源序列集合的下采样结果,确定出氨基酸序列样本的第二表征矩阵。例如,基于氨基酸序列样本d0,可以从氨基酸序列数据库中查找到与氨基酸序列样本d0对应的200个同源序列,然后从200个同源序列中随机抽取出50个同源序列,并根据抽取出的50个同源序列确定出氨基酸序列样本d0的第二表征矩阵。
103.步骤s202,基于从训练数据集抽取的氨基酸序列样本以及对应的第二表征矩阵,通过特征增强网络,得到相应的增强样本表征矩阵,并根据增强样本表征矩阵与氨基酸序列样本的第一表征矩阵,确定第一损失值。
104.可以先将氨基酸序列样本输入到矩阵生成网络中,得到氨基酸序列样本的初始样本表征矩阵。该矩阵生成网络可以是基于双向注意力机制的预训练语言模型(bidirectional encoder representations from transformers,bert)训练得到的。矩阵生成网络也可以是基于其他自然语言处理模型训练得到的,本技术在此不作限定。
105.然后将氨基酸序列样本、氨基酸序列样本的第二表征矩阵和初始样本表征矩阵进行拼接,得到第一蛋白质信息样本矩阵,并将第一蛋白质信息样本矩阵输入到特征增强网络中,可以得到氨基酸序列样本对应的增强样本表征矩阵。
106.根据增强样本表征矩阵与氨基酸序列样本的第一表征矩阵,可以确定出第一损失值。在计算第一损失值时,可以采用均方误差(mean square error,mse)损失函数来计算第一损失值。第一损失值是判定实际的输出第一表征矩阵与期望的输出增强样本表征矩阵的接近程度。第一损失值越小,说明实际的输出越接近期望的输出。第一损失值也可以采用其他损失函数来计算得到,本技术实施例在此不作限定。
107.步骤s203,将增强样本表征矩阵和氨基酸序列样本进行拼接后分别输入到结构预测网络和用于辅助训练的分类网络中,并基于获得的输出结果,确定第二损失值。
108.将增强样本表征矩阵和氨基酸序列样本进行拼接,可以得到第二蛋白质信息样本矩阵。将第二蛋白质信息样本矩阵输入到结构预测网络中,可以得到氨基酸序列样本对应的结构预测数据。将第二蛋白质信息样本矩阵输入到分类网络中,可以得到氨基酸序列样本对应的结构预测参考数据。将结构预测数据与结构预测参考数据进行比对,可以确定第二损失值。
109.在计算第二损失值时,可以采用知识蒸馏(knowledge distillation,kd)损失函数计算第二损失值。第二损失值是判定实际的输出结构预测数据与期望的输出结构预测参考数据的接近程度。第二损失值越小,说明实际的输出越接近期望的输出。第二损失值也可以采用其他损失函数来计算得到,本技术实施例在此不作限定。
110.步骤s204,根据第一损失值和第二损失值,分别对特征增强网络和结构预测网络进行参数调整。
111.在一种实施例中,可以仅根据第一损失值和第二损失值的加权和,确定联合损失
值,并判断联合损失值是否收敛至预设的目标值。如果联合损失值小于或等于预设的目标值,或者,连续n次训练得到的联合损失值的变化幅度小于或等于预设的目标值时,认为联合损失值已收敛至预设的目标值,说明联合损失值收敛;否则,说明联合损失值尚未收敛。如果联合损失值未收敛,则分别对特征增强网络和结构预测网络的参数进行调整,调整参数后,返回执行从训练数据集中抽取基酸序列样本步骤,继续下一轮的训练过程。如果联合损失值收敛,则将当前得到的特征增强网络和结构预测网络作为已训练的蛋白质结构预测模型。采用根据第一损失值和第二损失值的加权和确定的联合损失值对蛋白质结构预测模型进行训练,得到的蛋白质结构预测模型可以对组成蛋白质结构的氨基酸所属的设定结构类别进行准确预测。
112.在另一种实施例中,将氨基酸序列样本和氨基酸序列样本的第一表征矩阵进行拼接,得到第一辅助训练信息矩阵,并将第一辅助训练信息矩阵输入到分类网络中,可以得到氨基酸序列样本对应的第一辅助结构预测数据。将氨基酸序列样本和氨基酸序列样本的第二表征矩阵进行拼接,得到第二辅助训练信息矩阵,并将第二辅助训练信息矩阵输入到分类网络中,可以得到氨基酸序列样本对应的第二辅助结构预测数据。将氨基酸序列样本和氨基酸序列样本的初始样本表征矩阵进行拼接,得到第三辅助训练信息矩阵,并将第三辅助训练信息矩阵输入分类网络,得到氨基酸序列样本对应的第三辅助结构预测数据。基于结构预测参考数据、第一辅助结构预测数据、第二辅助结构预测数据和第三辅助结构预测数据,确定第三损失值。并根据第一损失值、第二损失值和第三损失值的加权和,确定出联合损失值,并根据该联合损失值,对特征增强网络和结构预测网络进行参数调整。根据第一损失值、第二损失值和第三损失值来确定联合损失值,可以进一步提高对蛋白质结构预测模型进行训练的训练效果,并且也可以进一步提高得到的蛋白质结构预测模型对组成蛋白质结构的氨基酸所属的设定结构类别进行预测的预测精度。
113.本技术实施例提供的蛋白质结构预测模型的训练方法,训练数据集中的每个氨基酸序列样本均具有第一表征矩阵和第二表征矩阵,第一表征矩阵是根据氨基酸序列样本关联的同源序列集合确定的高质量的特征表征矩阵,第二表征矩阵是根据该同源序列集合的下采样结果确定的低质量的特征表征矩阵。本技术实施例基于各个氨基酸序列样本的第一表征矩阵和第二表征矩阵,并采用知识蒸馏的方法,借助辅助训练的分类网络对蛋白质结构预测模型包含的特征增强网络和结构预测网络进行联合训练,可以得到蛋白质结构预测模型。从而可以利用已训练的蛋白质结构预测模型,基于待处理蛋白质的低质量的特征表征矩阵,对待处理蛋白质的结构进行预测,获得精度较高的蛋白质结果预测结果,提高蛋白质结构预测的预测精度。
114.图3示出了本技术提出的蛋白质结构预测模型的训练方法的详细实现过程,如图3所示,可以包括如下步骤:
115.步骤s301,获取训练数据集。
116.获取到的训练数据集中可以包括多个氨基酸序列样本,并且每个氨基酸序列样本都对应有第一表征矩阵、第二表征矩阵和初始样本表征矩阵。其中,氨基酸序列样本对应的第一表征矩阵是根据氨基酸序列样本关联的同源序列集合确定的,氨基酸序列样本对应的第二表征矩阵是根据氨基酸序列样本关联的同源序列集合的下采样结果确定的,氨基酸序列样本对应的初始样本表征矩阵是将氨基酸序列样本输入到已训练的矩阵生成网络中得
到的。
117.步骤s302,从训练数据集中抽取氨基酸序列样本。
118.可以预先获取训练数据集,对蛋白质结构预测模型进行训练时,从训练数据集中抽取氨基酸序列样本作为训练样本数据。
119.步骤s303,将抽取的氨基酸序列样本以及对应的第二表征矩阵和初始样本表征矩阵输入到待训练的特征增强网络中,得到相应的增强样本表征矩阵。
120.在采用训练数据集对蛋白质结构预测模型进行训练时,可以抽取训练数据集中的氨基酸序列样本,并将抽取的氨基酸序列样本以及对应的第二表征矩阵和初始样本表征矩阵输入到待训练的蛋白质结构预测模型所包含的特征增强网络中。基于特征增强网络,可以得到氨基酸序列样本对应的增强样本表征矩阵。
121.步骤s304,根据增强样本表征矩阵与氨基酸序列样本的第一表征矩阵,确定第一损失值。
122.将增强样本表征矩阵与氨基酸序列样本的第一表征矩阵进行比对,可以确定出第一损失值。在计算第一损失值时,可以采用mse损失函数来计算第一损失值。第一损失值是判定实际的输出第一表征矩阵与期望的输出增强样本表征矩阵的接近程度。第一损失值越小,说明实际的输出越接近期望的输出。
123.步骤s305,将增强样本表征矩阵和氨基酸序列样本进行拼接后输入到待训练的结构预测网络中,得到氨基酸序列样本对应的结构预测数据。
124.可以将增强样本表征矩阵和氨基酸序列样本进行拼接,得到第二蛋白质信息样本矩阵,并将第二蛋白质信息样本矩阵输入到待训练的蛋白质结构预测模型所包含的结构预测网络中。基于结构预测网络,可以得到氨基酸序列样本对应的结构预测数据。
125.步骤s306,将增强样本表征矩阵和氨基酸序列样本进行拼接后输入到用于辅助训练的分类网络中,得到氨基酸序列样本对应的结构预测参考数据。
126.可以将增强样本表征矩阵和氨基酸序列样本进行拼接,得到第二蛋白质信息样本矩阵,并将第二蛋白质信息样本矩阵输入到用于辅助训练的分类网络中,并且该分类网络是已训练的分类网络。基于分类网络,可以得到氨基酸序列样本对应的结构预测参考数据。
127.步骤s307,根据氨基酸序列样本对应的结构预测数据和结构预测参考数据,确定第二损失值。
128.将氨基酸序列样本对应的结构预测数据与氨基酸序列样本对应的结构预测参考数据进行比对,可以确定出第二损失值。在计算第二损失值时,可以采用kd损失函数计算第二损失值。第二损失值是判定实际的输出结构预测数据与期望的输出结构预测参考数据的接近程度。第二损失值越小,说明实际的输出越接近期望的输出。
129.步骤s308,将氨基酸序列样本和氨基酸序列样本的第一表征矩阵进行拼接后输入到分类网络中,得到氨基酸序列样本对应的第一辅助结构预测数据。
130.可以将氨基酸序列样本和氨基酸序列样本的第一表征矩阵进行拼接,得到第一辅助训练信息矩阵,并将第一辅助训练信息矩阵输入到已训练的分类网络中。基于分类网络可以得到氨基酸序列样本对应的第一辅助结构预测数据。
131.步骤s309,将氨基酸序列样本和氨基酸序列样本的第二表征矩阵进行拼接后到输入分类网络中,得到氨基酸序列样本对应的第二辅助结构预测数据。
132.可以将氨基酸序列样本和氨基酸序列样本的第二表征矩阵进行拼接,得到第二辅助训练信息矩阵,并将第二辅助训练信息矩阵输入到已训练的分类网络中。基于分类网络可以得到氨基酸序列样本对应的第二辅助结构预测数据。
133.步骤s310,将氨基酸序列样本和氨基酸序列样本的初始样本表征矩阵进行拼接后输入到分类网络中,得到氨基酸序列样本对应的第三辅助结构预测数据。
134.将氨基酸序列样本和氨基酸序列样本的初始样本表征矩阵进行拼接,得到第三辅助训练信息矩阵,并将第三辅助训练信息矩阵输入到已训练的分类网络中。基于分类网络可以得到氨基酸序列样本对应的第三辅助结构预测数据。
135.步骤s311,根据结构预测参考数据、第一辅助结构预测数据、第二辅助结构预测数据和第三辅助结构预测数据,确定第三损失值。
136.将结构预测参考数据、第一辅助结构预测数据、第二辅助结构预测数据和第三辅助结构预测数据进行比对,可以确定出第三损失值。在计算第三损失值时,可以采用对比学习(contrastive learning,cl)损失函数计算第三损失值。第三损失值是判定实际的输出结构预测参考数据与期望的输出第一辅助结构预测数据、第二辅助结构预测数据和第三辅助结构预测数据的接近程度。第三损失值越小,说明实际的输出越接近期望的输出。第三损失值也可以采用其他损失函数来计算得到,本技术实施例在此不作限定。
137.在一种实施例中,当对组成蛋白质结构的氨基酸的设定结构类别的预测精度要求不高时,也可以不用确定第三损失值,仅根据确定出的第一损失值和第二损失值对蛋白质结构预测模型进行训练。
138.步骤s312,根据第一损失值、第二损失值和第三损失值的加权和,确定联合损失值。
139.步骤s313,确定联合损失值是否收敛至预设的目标值;如果否,执行步骤s314;如果是,执行步骤s315。
140.可以将第一损失值、第二损失值和第三损失值进行加权和操作,得到联合损失值。判断联合损失值是否收敛至预设的目标值,如果联合损失值小于或等于预设的目标值,或者,连续n次训练得到的联合损失值的变化幅度小于或等于预设的目标值时,认为联合损失值已收敛至预设的目标值,说明联合损失值收敛;否则,说明联合损失值尚未收敛。
141.步骤s314,根据确定的联合损失值分别对待训练的特征增强网络和结构预测网络的参数进行调整。
142.如果联合损失值未收敛,则分别对蛋白质结构预测模型所包含的特征增强网络和结构预测网络的参数进行调整,调整模型参数后,返回执行步骤s302,继续下一轮的训练过程。
143.步骤s315,结束训练得到已训练的蛋白质结构预测模型。
144.如果联合损失值收敛,则将当前得到的特征增强网络和结构预测网络作为已训练的蛋白质结构预测模型。
145.参阅图4所示,下面采用一个具体的应用场景,对以上实施例做出进一步详细说明:
146.假设获取到的训练数据集中包含有氨基酸序列样本s,则可以从氨基酸序列数据库中查找氨基酸序列样本s对应的多个同源序列,并将多个同源序列组成氨基酸序列样本s
关联的同源序列集合。根据氨基酸序列样本s关联的同源序列集合可以确定出氨基酸序列样本s对应的第一表征矩阵xh。对氨基酸序列样本s关联的同源序列集合进行下采样,可以得到同源序列集合的下采样结果,根据同源序列集合的下采样结果,可以确定氨基酸序列样本s对应的第二表征矩阵x
l
。
147.基于训练数据集中的氨基酸序列样本s,可以对蛋白质结构预测模型包含的特征增强网络和结构预测网络进行迭代训练,直到满足设定的收敛条件为止,其中,一次迭代训练过程可以包括:
148.将氨基酸序列样本s输入到已训练的矩阵生成网络中,基于矩阵生成网络可以得到氨基酸序列样本s对应的初始样本表征矩阵xb。将氨基酸序列样本s、第二表征矩阵x
l
和初始样本表征矩阵xb进行拼接,得到第一蛋白质信息样本矩阵(s,xb,x
l
),并将第一蛋白质信息样本矩阵(s,xb,x
l
)输入到待训练的特征增强网络fe中,基于特征增强网络fe可以得到氨基酸序列样本s对应的增强样本表征矩阵xe。根据氨基酸序列样本s对应的第一表征矩阵xh和氨基酸序列样本s对应的增强样本表征矩阵xe,可以计算出第一表征矩阵xh与增强样本表征矩阵xe之间的mse损失值lm。
149.将氨基酸序列样本s与增强样本表征矩阵xe进行拼接,可以得到第二蛋白质信息样本矩阵(s,xe),并将第二蛋白质信息样本矩阵(s,xe)输入到待训练的结构预测网络fs中,基于结构预测网络fs可以得到氨基酸序列样本s对应的结构预测数据fs(s,xe)。将第二蛋白质信息样本矩阵(s,xe)输入到已训练的分类网络f
t
中,基于分类网络f
t
可以得到氨基酸序列样本s对应的结构预测参考数据f
t
(s,xe)。根据氨基酸序列样本s对应的结构预测数据fs(s,xe)和氨基酸序列样本s对应的结构预测参考数据f
t
(s,xe),可以计算出结构预测数据fs(s,xe)与结构预测参考数据f
t
(s,xe)之间的kd损失值ld。
150.将氨基酸序列样本s与第一表征矩阵xh进行拼接,可以得到第一辅助训练信息矩阵(s,xh),并将第一辅助训练信息矩阵(s,xh)输入到已训练的分类网络f
t
中,基于分类网络f
t
可以得到氨基酸序列样本s对应的第一辅助结构预测数据f
t
(s,xh)。将氨基酸序列样本s与第二表征矩阵x
l
进行拼接,可以得到第二辅助训练信息矩阵(s,x
l
),并将第二辅助训练信息矩阵(s,x
l
)输入到已训练的分类网络f
t
中,基于分类网络f
t
可以得到氨基酸序列样本s对应的第二辅助结构预测数据f
t
(s,x
l
)。将氨基酸序列样本s与初始样本表征矩阵xb进行拼接,可以得到第三辅助训练信息矩阵(s,xb),并将第二辅助训练信息矩阵(s,xb)输入到已训练的分类网络f
t
中,基于分类网络f
t
可以得到氨基酸序列样本s对应的第三辅助结构预测数据f
t
(s,xb)。根据结构预测参考数据f
t
(s,xe)、第一辅助结构预测数据f
t
(s,xh)、第二辅助结构预测数据f
t
(s,x
l
)和第三辅助结构预测数据f
t
(s,xb),可以计算出结构预测参考数据f
t
(s,xe)、第一辅助结构预测数据f
t
(s,xh)、第二辅助结构预测数据f
t
(s,x
l
)和第三辅助结构预测数据f
t
(s,xb)之间的cl损失值l
l
。
151.根据mse损失值lm、kd损失值ld和cl损失值l
l
的加权和,确定出联合损失值,再根据联合损失值分别对特征增强网络和结构预测网络进行参数调整。
152.图5示出了本技术实施例提供的一种蛋白质结构预测方法的流程图,该方法可以由图1中的服务器12执行,也可以由终端设备11或其它电子设备执行。示例性地,下文以用于预测蛋白质结构的计算机作为执行主体,说明本技术实施例的蛋白质结构预测方法的具体实现过程。如图5所示,该蛋白质结构预测方法包括如下步骤:
153.步骤s501,将待处理蛋白质对应的蛋白质信息矩阵输入已训练的蛋白质结构预测模型,得到待处理蛋白质中的各个氨基酸所属的设定结构类别。
154.可以将氨基酸序列输入到矩阵生成网络中,基于矩阵生成网络对氨基酸序列进行同源信息表达,得到待处理蛋白质的初始同源信息表征矩阵。其中,矩阵生成网络是基于bert模型训练得到的。将氨基酸序列和初始同源信息表征矩阵进行拼接,可以得到待处理蛋白质对应的蛋白质信息矩阵。
155.在得到已训练的蛋白质结构预测模型后,可以将待处理蛋白质对应的蛋白质信息矩阵输入到已训练的蛋白质结构预测模型所包含的特征增强网络中,基于特征增强网络可以确定待处理蛋白质对应的增强同源信息表征矩阵。将氨基酸序列和增强同源信息表征矩阵进行拼接,可以得到增强蛋白质信息矩阵,并将增强蛋白质信息矩阵输入到已训练的蛋白质结构预测模型所包含的结构预测网络中,基于结构预测网络可以确定待处理蛋白质中的各个氨基酸所属的设定结构类别。
156.步骤s502,根据各个氨基酸所属的设定结构类别,确定待处理蛋白质的结构。
157.根据得到的待处理蛋白质中的各个氨基酸所属的设定结构类别,可以确定出待处理蛋白质的结构。
158.蛋白质二级结构是指多肽主链骨架原子沿一定的轴盘旋或折叠而形成的特定的构象,即肽链主链骨架原子的空间位置排布,不涉及氨基酸残基侧链。并且,蛋白质二级结构的主要形式包括α-螺旋、β-折叠、β-转角和无规卷曲。可以根据待处理蛋白质中的各个氨基酸所属的设定结构类别,确定出待处理蛋白质的二级结构,即可以先确定出待处理蛋白质中的各个氨基酸属于α-螺旋、β-折叠、β-转角和无规卷曲这四种主要形式中的哪一种形式,进而根据各个氨基酸所属的形式,确定出蛋白质二级结构。
159.具体地,如图6所示,可以采用一个具体的应用场景,对以上实施例做出进一步详细说明:
160.假设待处理蛋白质中包含有氨基酸d0、氨基酸d1和氨基酸d2共3个氨基酸组成的氨基酸序列s。
161.步骤s601,将氨基酸序列s输入矩阵生成网络,得到待处理蛋白质的初始同源信息表征矩阵。
162.可以将氨基酸序列s输入到矩阵生成网络中,基于矩阵生成网络对氨基酸序列进行同源信息表达,得到待处理蛋白质的初始同源信息表征矩阵。
163.步骤s602,将氨基酸序列s和初始同源信息表征矩阵进行拼接,得到待处理蛋白质对应的蛋白质信息矩阵。
164.步骤s603,将待处理蛋白质对应的蛋白质信息矩阵输入已训练的蛋白质结构预测模型,得到待处理蛋白质中的氨基酸d0属于α-螺旋结构类别,氨基酸d1属于β-折叠结构类别,氨基酸d2属于β-转角结构类别。
165.将待处理蛋白质对应的蛋白质信息矩阵输入到已训练的蛋白质结构预测模型中,基于蛋白质结构预测模型可以确定出待处理蛋白质中的氨基酸d0属于α-螺旋结构类别,氨基酸d1属于β-折叠结构类别,氨基酸d2属于β-转角结构类别。
166.步骤s604,根据氨基酸d0、氨基酸d1和氨基酸d2所属的结构类别,确定待处理蛋白质的结构。
167.根据氨基酸d0属于α-螺旋结构类别,氨基酸d1属于β-折叠结构类别,氨基酸d2属于β-转角结构类别,可以确定出待处理蛋白质的二级结构。
168.在一种实施例中,如图7所示,特征增强网络可以包括卷积子网络、长短期记忆子网络、拼接层和至少一个全连接层。可以将蛋白质信息矩阵输入到卷积子网络中,基于卷积子网络获得蛋白质信息矩阵的基础特征。将蛋白质信息矩阵输入到长短期记忆子网络中,基于长短期记忆子网络获得蛋白质信息矩阵的时序特征。将基础特征和时序特征输入到拼接层中,基于拼接层对基础特征和时序特征进行拼接,得到合成特征。将合成特征输入到全连接层中,基于全连接层得到待处理蛋白质对应的增强同源信息表征矩阵。
169.特征增强网络中的卷积子网络和长短期记忆子网络也可以由其他网络来替代,本技术实施例在此不作限定。例如,可以采用transformer网络来替代卷积子网络和长短期记忆子网络。
170.在另一种实施例中,如图8所示,结构预测网络包括卷积子网络、长短期记忆子网络、拼接层和多分类层。可以将增强蛋白质信息矩阵输入到卷积子网络,基于卷积子网络获得增强蛋白质信息矩阵的基础特征。将增强蛋白质信息矩阵输入到长短期记忆子网络中,基于长短期记忆子网络获得增强蛋白质信息矩阵的时序特征。将基础特征和时序特征输入到拼接层中,基于拼接层对基础特征和时序特征进行拼接,得到合成特征。将合成特征输入到多分类层中,基于多分类层得到待处理蛋白质中的各个氨基酸所属的设定结构类别。
171.在一些实施例中,可以在所有关键数据上将本技术提出的蛋白质结构预测方法与real方法和bagging方法进行比较,当设定结构类别为三分类时,比较的结果可以如表1所示:
172.表1
[0173][0174]
如表1所示,msa counts为多序列对准的数量,bc40、cullpdb和cb513为关键数据集,number为从关键数据集中查找出的具有msa counts的氨基酸序列,real和bagging为与本技术提出的蛋白质结构预测方法进行比较的相关方法。从表1中可以看出,当设定结构类别为三分类时,本技术提出的蛋白质结构预测方法的性能高于real方法和bagging方法。
[0175]
当设定结构类别为八分类时,比较的结果可以如表2所示:
[0176]
表2
[0177][0178]
如表2所示,当设定结构类别为八分类时,本技术提出的蛋白质结构预测方法的性能同样高于real方法和bagging方法。
[0179]
图8为在所有关键数据上将本技术提出的蛋白质结构预测方法与bagging方法进行比较的柱状图。如图8所示,pssm-distill为本技术提出的蛋白质结构预测方法,可以分别在包含有不同的msa count和msa meff区间内比较pssm-distill方法和bagging方法的预测精度。从图9中可以看出,pssm-distill方法的预测精度远高于bagging方法的预测精度。
[0180]
在另一些实施例中,可以将本技术提出的蛋白质结构预测模型的训练方法中的bert、mse和cl分别剥离后,比较本技术提出的蛋白质结构预测方法与分别剥离bert、mse和cl后得到的蛋白质结构预测方法的性能,比较的结果可以如表3所示:
[0181]
表3
[0182][0183]
如表3所示,our为本技术提出的蛋白质结构预测方法,w/o bert为剥离bert部分后得到的蛋白质预测方法,w/o cl为剥离cl部分后得到的蛋白质预测方法,w/o mse为剥离mse部分后得到的蛋白质预测方法。从表3中可以看出,本技术提出的蛋白质结构预测方法的性能分别高于剥离bert、cl和mse部分的蛋白质结构预测方法的性能。
[0184]
与图2所示的蛋白质结构预测模型的训练方法基于同一发明构思,本技术实施例中还提供了一种蛋白质结构预测模型的训练装置,该蛋白质结构预测模型的训练装置可以布设在服务器或终端设备中。由于该装置是本技术蛋白质结构预测模型的训练方法对应的装置,并且该装置解决问题的原理与该方法相似,因此该装置的实施可以参见上述方法的实施,重复之处不再赘述。
[0185]
图10示出了本技术实施例提供的一种蛋白质结构预测模型的训练装置的结构示意图,如图10所示,该蛋白质结构预测模型的训练装置包括数据获取单元1001和模型训练单元1002。
[0186]
其中,数据获取单元1001,用于获取包含多个氨基酸序列样本的训练数据集;每个氨基酸序列样本的第一表征矩阵是根据关联的同源序列集合确定的,且同源序列集合中的同源序列的数量不小于设定阈值;每个氨基酸序列样本的第二表征矩阵是根据关联的同源序列集合的下采样结果确定的;
[0187]
模型训练单元1002,用于基于训练数据集,对蛋白质结构预测模型包含的特征增强网络和结构预测网络进行迭代训练,直到满足设定的收敛条件为止,其中,一次迭代训练
过程包括:基于从训练数据集抽取的氨基酸序列样本以及对应的第二表征矩阵,通过特征增强网络,得到相应的增强样本表征矩阵,并根据增强样本表征矩阵与氨基酸序列样本的第一表征矩阵,确定第一损失值;将增强样本表征矩阵和氨基酸序列样本进行拼接后分别输入结构预测网络和用于辅助训练的分类网络,并基于获得的输出结果,确定第二损失值;根据第一损失值和第二损失值,分别对特征增强网络和结构预测网络进行参数调整。
[0188]
在一种可选的实施例中,数据获取单元1001,具体用于:
[0189]
基于氨基酸序列样本,从氨基酸序列数据库中查找氨基酸序列样本对应的多个同源序列,并将多个同源序列组成氨基酸序列样本关联的同源序列集合;
[0190]
对氨基酸序列样本关联的同源序列集合进行下采样,得到同源序列集合的下采样结果;
[0191]
根据同源序列集合的下采样结果,确定氨基酸序列样本的第二表征矩阵。
[0192]
在一种可选的实施例中,模型训练单元1002,具体用于:
[0193]
将氨基酸序列样本输入矩阵生成网络,得到氨基酸序列样本的初始样本表征矩阵;矩阵生成网络是基于自然语言处理模型训练得到的;
[0194]
将氨基酸序列样本、氨基酸序列样本的第二表征矩阵和初始样本表征矩阵进行拼接,得到第一蛋白质信息样本矩阵;
[0195]
将第一蛋白质信息样本矩阵输入特征增强网络,得到氨基酸序列样本对应的增强样本表征矩阵。
[0196]
在一种可选的实施例中,模型训练单元1002,还用于:
[0197]
将增强样本表征矩阵和氨基酸序列样本进行拼接,得到第二蛋白质信息样本矩阵;
[0198]
将第二蛋白质信息样本矩阵输入结构预测网络,得到氨基酸序列样本对应的结构预测数据;
[0199]
将第二蛋白质信息样本矩阵输入分类网络,得到氨基酸序列样本对应的结构预测参考数据;
[0200]
将结构预测数据与结构预测参考数据进行比对,确定第二损失值。
[0201]
在一种可选的实施例中,模型训练单元1002,还用于:
[0202]
将氨基酸序列样本和氨基酸序列样本的第一表征矩阵进行拼接,得到第一辅助训练信息矩阵,并将第一辅助训练信息矩阵输入分类网络,得到氨基酸序列样本对应的第一辅助结构预测数据;
[0203]
将氨基酸序列样本和氨基酸序列样本的第二表征矩阵进行拼接,得到第二辅助训练信息矩阵,并将第二辅助训练信息矩阵输入分类网络,得到氨基酸序列样本对应的第二辅助结构预测数据;
[0204]
将氨基酸序列样本和氨基酸序列样本的初始样本表征矩阵进行拼接,得到第三辅助训练信息矩阵,并将第三辅助训练信息矩阵输入分类网络,得到氨基酸序列样本对应的第三辅助结构预测数据;
[0205]
基于结构预测参考数据、第一辅助结构预测数据、第二辅助结构预测数据和第三辅助结构预测数据,确定第三损失值;
[0206]
根据第一损失值、第二损失值和第三损失值,对特征增强网络和结构预测网络进
行参数调整。
[0207]
在一种可选的实施例中,模型训练单元1002,还用于:
[0208]
根据第一损失值、第二损失值和第三损失值的加权和,确定联合损失值;
[0209]
根据联合损失值,对特征增强网络和结构预测网络进行参数调整。
[0210]
与图5所示的蛋白质结构预测方法基于同一发明构思,本技术实施例中还提供了一种蛋白质结构预测装置,该蛋白质结构预测装置可以布设在服务器或终端设备中。由于该装置是本技术蛋白质结构预测方法对应的装置,并且该装置解决问题的原理与该方法相似,因此该装置的实施可以参见上述方法的实施,重复之处不再赘述。
[0211]
图11示出了本技术实施例提供的一种蛋白质结构预测装置的结构示意图,如图11所示,该蛋白质结构预测装置包括类别确定单元1101和结构确定单元1102。
[0212]
其中,类别确定单元1101,用于将待处理蛋白质对应的蛋白质信息矩阵输入已训练的蛋白质结构预测模型,得到待处理蛋白质中的各个氨基酸所属的设定结构类别;蛋白质结构预测模型是通过蛋白质结构预测模型的训练方法中任一项的训练方法得到的;蛋白质信息矩阵是基于待处理蛋白质的氨基酸序列获得的;
[0213]
结构确定单元,用于根据各个氨基酸所属的设定结构类别,确定待处理蛋白质的结构。
[0214]
在一种可选的实施例中,如图12所示,上述蛋白质结构预测装置还可以包括矩阵生成单元1201,用于:
[0215]
将氨基酸序列输入矩阵生成网络,基于矩阵生成网络对氨基酸序列进行同源信息表达,得到待处理蛋白质的初始同源信息表征矩阵;矩阵生成网络是基于自然语言处理模型训练得到的;
[0216]
将氨基酸序列和初始同源信息表征矩阵进行拼接,得到待处理蛋白质对应的蛋白质信息矩阵。
[0217]
在一种可选的实施例中,类别确定单元1101,具体用于:
[0218]
将待处理蛋白质对应的蛋白质信息矩阵输入蛋白质结构预测模型的特征增强网络,基于特征增强网络确定待处理蛋白质对应的增强同源信息表征矩阵;
[0219]
将氨基酸序列和增强同源信息表征矩阵进行拼接,得到增强蛋白质信息矩阵;
[0220]
将增强蛋白质信息矩阵输入蛋白质结构预测模型的结构预测网络,基于结构预测网络确定待处理蛋白质中的各个氨基酸所属的设定结构类别。
[0221]
在一种可选的实施例中,特征增强网络包括卷积子网络、长短期记忆子网络、拼接层和至少一个全连接层;类别确定单元1101,还用于:
[0222]
将蛋白质信息矩阵输入卷积子网络,基于卷积子网络获得蛋白质信息矩阵的基础特征;
[0223]
将蛋白质信息矩阵输入长短期记忆子网络,基于长短期记忆子网络获得蛋白质信息矩阵的时序特征;
[0224]
将基础特征和时序特征输入拼接层,基于拼接层对基础特征和时序特征进行拼接,得到合成特征;
[0225]
将合成特征输入全连接层,基于全连接层得到待处理蛋白质对应的增强同源信息表征矩阵。
[0226]
在一种可选的实施例中,结构预测网络包括卷积子网络、长短期记忆子网络、拼接层和多分类层;类别确定单元1101,还用于:
[0227]
将增强蛋白质信息矩阵输入卷积子网络,基于卷积子网络获得增强蛋白质信息矩阵的基础特征;
[0228]
将增强蛋白质信息矩阵输入长短期记忆子网络,基于长短期记忆子网络获得增强蛋白质信息矩阵的时序特征;
[0229]
将基础特征和时序特征输入拼接层,基于拼接层对基础特征和时序特征进行拼接,得到合成特征;
[0230]
将合成特征输入多分类层,基于多分类层得到待处理蛋白质中的各个氨基酸所属的设定结构类别。
[0231]
与上述方法实施例和装置实施例基于同一发明构思,本技术实施例中还提供了一种电子设备。该电子设备可以是服务器,如图1所示的服务器100。在该实施例中,电子设备的结构可以如图13所示,包括存储器1301,通讯模块1303以及一个或多个处理器1302。
[0232]
存储器1301,用于存储处理器1302执行的计算机程序。存储器1301可主要包括存储程序区和存储数据区,其中,存储程序区可存储操作系统,以及运行即时通讯功能所需的程序等;存储数据区可存储各种即时通讯信息和操作指令集等。
[0233]
存储器1301可以是易失性存储器(volatile memory),例如随机存取存储器(random-access memory,ram);存储器1301也可以是非易失性存储器(non-volatile memory),例如只读存储器,快闪存储器(flash memory),硬盘(hard disk drive,hdd)或固态硬盘(solid-state drive,ssd)、或者存储器1301是能够用于携带或存储具有指令或数据结构形式的期望的程序代码并能够由计算机存取的任何其他介质,但不限于此。存储器1301可以是上述存储器的组合。
[0234]
处理器1302,可以包括一个或多个中央处理单元(central processing unit,cpu)或者为数字处理单元等等。处理器1302,用于调用存储器1301中存储的计算机程序时实现上述蛋白质结构预测模型的训练方法和蛋白质结构预测方法。
[0235]
通讯模块1303用于与终端设备和其他电子设备进行通信。如果电子设备为服务器,则服务器可以通过通讯模块1303接收终端设备发送的氨基酸序列。
[0236]
本技术实施例中不限定上述存储器1301、通讯模块1303和处理器1302之间的具体连接介质。本公开实施例在图13中以存储器1301和处理器1302之间通过总线1304连接,总线1304在图13中以粗线表示,其它部件之间的连接方式,仅是进行示意性说明,并不引以为限。总线1304可以分为地址总线、数据总线、控制总线等。为便于表示,图13中仅用一条粗线表示,但并不表示仅有一根总线或一种类型的总线。
[0237]
在另一种实施例中,电子设备也可以是平板电脑、台式机、笔记本电脑等。
[0238]
根据本技术的一个方面,提供了一种计算机程序产品或计算机程序,该计算机程序产品或计算机程序包括计算机指令,该计算机指令存储在计算机可读存储介质中。计算机设备的处理器从计算机可读存储介质读取该计算机指令,处理器执行该计算机指令,使得该计算机设备执行上述实施例中的蛋白质结构预测模型的训练方法和蛋白质结构预测方法。所述程序产品可以采用一个或多个可读介质的任意组合。可读介质可以是可读信号介质或者可读存储介质。可读存储介质例如可以是——但不限于——电、磁、光、电磁、红外
线、或半导体的系统、装置或器件,或者任意以上的组合。可读存储介质的更具体的例子(非穷举的列表)包括:具有一个或多个导线的电连接、便携式盘、硬盘、随机存取存储器(ram)、只读存储器(rom)、可擦式可编程只读存储器(eprom或闪存)、光纤、便携式紧凑盘只读存储器(cd-rom)、光存储器件、磁存储器件、或者上述的任意合适的组合。
[0239]
以上所述,仅为本技术的具体实施方式,但本技术的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本技术揭露的技术范围内,可轻易想到变化或替换,都应涵盖在本技术的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1