抗原呈递预测模型的训练方法及其预测方法、设备和介质与流程
1.本公开涉及人工智能技术领域,特别涉及一种抗原呈递预测模型的训练方法及其预测方法、设备和介质。
背景技术:
2.最近免疫治疗取得的成功证明了一个长期的假设:肿瘤是具有免疫原性的并且能够引发获得性免疫反应。肿瘤细胞中的主要组织相容性复合物(major histocompatibility complex,mhc)或者人类白细胞抗原(human leukocyte antigen,hla)能够呈递肿瘤特异性抗原,这些抗原能够被cd8+(一种白细胞分化抗原)的杀伤性t细胞所识别。多年来研究人员一直在探究能够刺激t细胞产生对肿瘤有效反应的抗原的性质。
3.肿瘤抗原能够分为两大类:自身和非自身抗原。肿瘤相关的自身抗原包括癌症-睾丸(种系)抗原,分化抗原和癌症细胞过度表达的抗原。自身抗原的优点是在不同的肿瘤病人和肿瘤中共享。但是,这些自身抗原受到免疫耐受的限制,因此只能引起很弱的或者不能引起t细胞应答。肿瘤所积累的非同义体突变很久以前就被假设为能够被获得性免疫系统所识别的非自身抗原(也称肿瘤新生抗原),因此能够引起更加强烈的免疫反应。对肿瘤新生抗原的最初报道已经有二十年之久,但是对新生抗原的综合鉴定一直以来都是一件非常困难的事情,直到下一代测序技术的出现才得到提高。随着下一代测序的广泛使用,数项研究已经展示了非同义突变与免疫治疗高度相关。肿瘤中的突变负荷能够预测病人对免疫检查位点抑制疗法的响应,新抗原特异性的t细胞在越来越多的肿瘤病人中检测到。其中,新生抗原在肿瘤作用的直接证据来自于靶向新生抗原的肿瘤浸润性淋巴细胞(tumor infiltrating lymphocyte,til)疗法在病人中引起转移性肿瘤的消退。另外,在前临床研究中,靶向新抗原的治疗疫苗引起了肿瘤的消退。除了非同义突变外,还有其他种类的新抗原来源,包括因肿瘤特异性剪接或肿瘤细胞中mhci呈递机制发生变化导致的多肽转录后修饰,内源性逆转录病毒和在癌症中找到的其他病毒感染等。
4.肿瘤新生抗原作为抗肿瘤免疫力的重要驱动因素,其鉴定具有重要的临床应用,包括作为预测和药效生物标记,新型个体化肿瘤免疫疗法的设计,包括til疗法和疫苗。但是,一个主要的挑战是只有一小部分的突变引入的抗原能够被mhc呈递,被呈递的抗原中只有更小的一部分具有免疫原性。因为抗原的免疫原性验证需要花费巨大人力物力,通量很小,导致目前所积累的数据较少,进一步导致难以建立免疫原性预测模型。而抗原能否与hla结合和呈递到细胞表面已经可以高通量验证从而积累了大量的数据,因此更多的实验室希望通过预测抗原呈递或与hla结合来协助选择免疫原性抗原。肿瘤新生抗原筛选联盟(tumor neoantigen selection alliance,tesla)的研究表明抗原呈递主要包含了抗原与hla的亲和力和表达量等特征,虽然hla亲和力软件众多且准确性已经达到一定水平,但整合多个特征的抗原呈递预测软件较少,准确率也有待更多数据的评估确定。
5.但是,目前的抗原呈递预测算法对于抗原呈递预测表现均不是很好,在外部的质谱数据独立评估上均较差。
技术实现要素:
6.本公开的主要目的在于,提供一种抗原呈递预测模型的训练方法及其预测方法、设备和介质,以改善现有技术中存在的上述缺陷。
7.本公开是通过下述技术方案来解决上述技术问题:
8.作为本公开的一方面,提供一种抗原呈递预测模型的训练方法,包括:
9.获取抗原序列训练数据及用于表征抗原序列呈递概率的呈递概率训练数据;
10.将所述抗原序列训练数据输入至待训练的抗原呈递预测模型;
11.通过所述抗原呈递预测模型对所述抗原序列训练数据进行呈递预测处理以预测出所述抗原序列训练数据对应的呈递概率,其中,通过所述抗原呈递预测模型的特征编码模块对所述抗原序列训练数据进行氨基酸编码及向量映射处理;以及,
12.根据预测出的所述呈递概率及所述呈递概率训练数据训练所述抗原呈递预测模型。
13.作为可选实施方式,所述抗原序列训练数据包括主要由氨基酸组成的多肽序列、短肽序列以及短肽对应的pseudo序列(在编码mhc序列时,只考虑那些与肽紧密结合的残基,这些残基形成一个短序列,称为mhc伪序列(mhc pseudo sequence));和/或,
14.所述呈递概率训练数据包括label值(0或1,0表示呈递性差,1表示呈递性强)。
15.作为可选实施方式,所述特征编码模块包括embedding模块(一种数据处理模块)。
16.作为可选实施方式,所述通过所述抗原呈递预测模型对所述抗原序列训练数据进行呈递预测处理以预测出所述抗原序列训练数据对应的呈递概率的步骤,还包括:
17.通过所述抗原呈递预测模型的第一特征提取模块对进行氨基酸编码及向量映射处理后的抗原序列训练数据进行多肽序列一维卷积特征提取处理。
18.作为可选实施方式,所述第一特征提取模块包括一维cnn(卷积神经网络)模块。
19.作为可选实施方式,所述一维cnn模块包括三个并联的卷积结构。
20.作为可选实施方式,所述三个并联的卷积结构包括卷积核尺寸分别为1*1、1*5、1*9的一层卷积层。
21.作为可选实施方式,所述通过所述抗原呈递预测模型对所述抗原序列训练数据进行呈递预测处理以预测出所述抗原序列训练数据对应的呈递概率的步骤,还包括:
22.通过所述抗原呈递预测模型的第二特征提取模块对进行多肽序列一维卷积特征提取处理后的抗原序列训练数据进行多肽序列有序特征提取处理。
23.作为可选实施方式,所述第二特征提取模块包括rnn(循环神经网络)模块。
24.作为可选实施方式,所述rnn模块包括至少两层bigru(神经网络结构的一种)。
25.作为可选实施方式,所述通过所述抗原呈递预测模型对所述抗原序列训练数据进行呈递预测处理以预测出所述抗原序列训练数据对应的呈递概率的步骤,还包括:
26.通过所述抗原呈递预测模型的特征融合模块对进行多肽序列有序特征提取处理后的抗原序列训练数据进行特征融合处理。
27.作为可选实施方式,所述特征融合模块包括全连接模块。
28.作为可选实施方式,所述将所述抗原序列训练数据输入至待训练的抗原呈递预测模型的步骤之前,所述训练方法还包括:
29.对获取到的所述抗原序列训练数据进行预处理。
30.作为可选实施方式,所述预处理包括无效数据过滤处理、字符串序列数字化处理及归一化处理中的至少一种。
31.作为可选实施方式,还包括:
32.获取抗原序列测试数据;
33.将所述抗原序列测试数据输入至训练出的所述抗原呈递预测模型,以对训练出的所述抗原呈递预测模型进行测试,以获取训练并测试后的所述抗原呈递预测模型。
34.作为本公开的另一方面,提供一种抗原呈递预测方法,包括:
35.获取抗原序列数据;
36.将所述抗原序列数据输入至通过如上述的抗原呈递预测模型的训练方法训练出的抗原呈递预测模型;
37.通过训练出的抗原呈递预测模型对所述抗原序列数据进行呈递预测处理;以及,
38.输出预测出的所述抗原序列数据对应的呈递概率。
39.作为本公开的另一方面,提供一种抗原呈递预测方法,包括:
40.获取抗原序列数据;
41.将所述抗原序列数据输入至抗原呈递预测模型;
42.通过所述抗原呈递预测模型对所述抗原序列训练数据进行呈递预测处理以预测出所述抗原序列数据对应的呈递概率,其中,通过所述抗原呈递预测模型的特征编码模块对所述抗原序列数据进行氨基酸编码及向量映射处理;以及,
43.输出预测出的所述抗原序列数据对应的呈递概率。
44.作为可选实施方式,所述抗原序列数据包括主要由氨基酸组成的多肽序列、短肽序列以及短肽对应的pseudo序列。
45.作为可选实施方式,所述通过所述抗原呈递预测模型对所述抗原序列数据进行呈递预测处理以预测出所述抗原序列数据对应的呈递概率的步骤,还包括:
46.通过所述抗原呈递预测模型的第一特征提取模块对进行氨基酸编码及向量映射处理后的抗原序列训练数据进行多肽序列一维卷积特征提取处理;
47.通过所述抗原呈递预测模型的第二特征提取模块对进行多肽序列一维卷积特征提取处理后的抗原序列训练数据进行多肽序列有序特征提取处理。
48.作为可选实施方式,所述通过所述抗原呈递预测模型对所述抗原序列数据进行呈递预测处理以预测出所述抗原序列数据对应的呈递概率的步骤,还包括:
49.通过所述抗原呈递预测模型的特征融合模块对进行多肽序列有序特征提取处理后的抗原序列训练数据进行特征融合处理。
50.作为本公开的另一方面,提供一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行计算机程序时实现如上述的抗原呈递预测模型的训练方法或如上述的抗原呈递预测方法。
51.作为本公开的另一方面,提供一种计算机可读介质,其上存储有计算机指令,所述计算机指令在由处理器执行时实现如上述的抗原呈递预测模型的训练方法或如上述的抗原呈递预测方法。
52.根据本公开内容,本领域技术人员可以理解本公开内容的其它方面。
53.本公开的积极进步效果在于:
54.本公开利用深度学习技术有效地建立了基于抗原序列的神经网络算法模型,在大量的抗原序列训练数据以及对应表征呈递能力的呈递概率数据(label数据)的训练下,模型能够挖掘输入抗原序列的深层特征,并且学习到这些深层特征与label之间的一一对应关系,在模型完成参数自动优化后,模型便具有对输入抗原序列数据的预测判决能力,从而有效地解决了预测新抗原呈递能力较差的问题,提升了预测抗原呈递能力的准确性和效率。
55.本公开针对输入序列编码问题,提出用nlp(自然语言处理)中常用的word embedding(将序列所属x空间的位点对应氨基酸映射为到y空间的多维向量)方法来对氨基酸位点进行向量初始化,这种编码方式在数据对应的场景下适用性强,这样能够让氨基酸初始化向量表达更加有意义。
56.本公开针对序列位点深层特征提取问题,考虑了利用特征提取能力更强的一维cnn神经网络结构来提取序列的特征。
57.本公开为了考虑序列中位点的相关性以及序列整体性,模型搭建用到rnn模块这种具有记忆功能的针对序列建模的神经网络技术。
附图说明
58.在结合以下附图阅读本公开的实施例的详细描述之后,能够更好地理解本公开的所述特征和优点。在附图中,各组件不一定是按比例绘制,并且具有类似的相关特性或特征的组件可能具有相同或相近的附图标记。
59.图1为根据本公开内容的一实施例的抗原呈递预测模型的训练方法的流程示意图。
60.图2为根据本公开内容的一可选实施方式的训练模型场景的流程示意图。
61.图3为根据本公开内容的一可选实施方式的抗原呈递预测模型的结构示意图。
62.图4为根据本公开内容的另一实施例的抗原呈递预测方法的流程示意图。
63.图5为根据本公开的另一实施例的实现抗原呈递预测模型的训练方法或抗原呈递预测方法的电子设备的结构示意图。
具体实施方式
64.下面通过实施例的方式进一步说明本公开,但并不因此将本公开限制在所述的实施例范围之中。
65.应当注意,在说明书中对“一实施例”、“可选实施例”、“另一实施例”等的引用指示所描述的实施例可以包括特定的特征、结构或特性,但是每个实施例可能不一定包括该特定的特征、结构或特性。而且,这样的短语不一定指代相同的实施例。此外,当结合实施例描述特定特征、结构或特性时,无论是否被明确描述,结合其它实施例来实现这样的特征、结构或特性都在相关领域的技术人员的知识范围内。
66.在本公开内容的描述中,需要理解的是,术语“中心”、“横向”、“上”、“下”、“左”、“右”、“竖直”、“水平”、“顶”、“底”、“内”、“外”等指示的方位或位置关系为基于附图所示的方位或位置关系,仅是为了便于描述本公开内容和简化描述,而不是指示或暗示所指的装置或元件必须具有特定的方位、以特定的方位构造和操作,因此不能理解为对本公开内容
的限制。此外,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者隐含地包括一个或者更多个该特征。在本公开内容的描述中,除非另有说明,“多个”的含义是两个或两个以上。另外,术语“包括”及其任何变形,意图在于覆盖不排他的包含。
67.在本公开内容的描述中,需要说明的是,除非另有明确的规定和限定,术语“安装”、“相连”、“连接”应做广义理解,例如,可以是固定连接,也可以是可拆卸连接,或一体地连接;可以是机械连接,也可以是电连接;可以是直接相连,也可以通过中间媒介间接相连,可以是两个元件内部的连通。对于本领域的普通技术人员而言,可以具体情况理解上述术语在本公开内容中的具体含义。
68.这里所使用的术语仅仅是为了描述具体实施例而不意图限制示例性实施例。除非上下文明确地另有所指,否则这里所使用的单数形式“一个”、“一项”还意图包括复数。还应当理解的是,这里所使用的术语“包括”和/或“包含”规定所陈述的特征、整数、步骤、操作、单元和/或组件的存在,而不排除存在或添加一个或更多其他特征、整数、步骤、操作、单元、组件和/或其组合。
69.目前,一般采用的抗原呈递预测算法主要具有以下缺陷:
70.1、在数据处理上,目前的抗原呈递预测算法使用通用的传统编码方式,导致缺乏一定的适应性,不一定在呈递数据场景下适用;
71.2、在算法建模上,通过简单的线性网络结构来建立模型,忽略了挖掘输入数据的深层特征以及序列的位点相互关系,这样的算法模型表达能力显然不足。
72.综上所述,目前的抗原呈递预测算法对于抗原呈递预测表现均不是很好,在外部的质谱数据独立评估上均较差。
73.为了克服目前存在的上述缺陷,本实施例提供一种抗原呈递预测模型的训练方法,包括:获取抗原序列训练数据及用于表征抗原序列呈递概率的呈递概率训练数据;将抗原序列训练数据输入至待训练的抗原呈递预测模型;通过抗原呈递预测模型对抗原序列训练数据进行呈递预测处理以预测出抗原序列训练数据对应的呈递概率,其中,通过抗原呈递预测模型的特征编码模块对抗原序列训练数据进行氨基酸编码及向量映射处理;以及,根据预测出的呈递概率及呈递概率训练数据训练抗原呈递预测模型。
74.在本实施例中,抗原呈递预测模型的训练方法优选适用于hla抗原呈递预测模型的训练方法,当然本实施例可适用于所有多肽序列的亲和力,抗原呈递和免疫原性模型,包括但不限于i类和ii类人类白细胞抗原的相关预测,因此本实施例并不限定应用场景,可根据实际需求或可能出现的需求进行相应的选择及调整。
75.在本实施例中,特征编码模块优选为embedding模块,但并不仅限于此,可根据实际需求或可能出现的需求进行相应的选择及调整。
76.在本实施例中,利用深度学习技术有效地建立了基于抗原序列的神经网络算法模型,在大量的抗原序列训练数据以及对应表征呈递能力的呈递概率数据(label数据)的训练下,模型能够挖掘输入抗原序列的深层特征,并且学习到这些深层特征与label之间的一一对应关系,在模型完成参数自动优化后,模型便具有对输入抗原序列数据的预测判决能力,从而有效地解决了预测新抗原呈递能力较差的问题,提升了预测抗原呈递能力的准确性和效率。
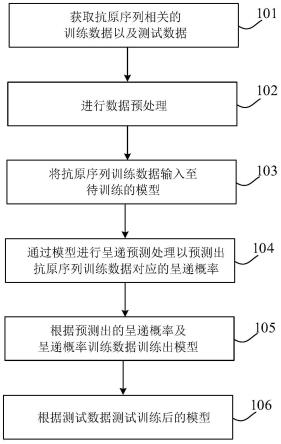
77.在本实施例中,针对输入序列编码问题,可利用nlp(自然语言处理)中常用的word embedding(将序列所属x空间的位点对应氨基酸映射为到y空间的多维向量)方法来对氨基酸位点进行向量初始化,这种编码方式在数据对应的场景下适用性强,这样能够让氨基酸初始化向量表达更加有意义。
78.为了克服目前存在的上述缺陷,首先,本实施例的抗原呈递预测模型考虑了输入序列数字化的编码方式,普适性的编码方式难以让编码后的数据适用于抗原呈递场景,需要用适应性强的编码方式来进行抗原序列编码。因此,输入序列采用什么类型的编码模型,能够让氨基酸初始化向量表达更加有意义,是本实施例解决的技术问题之一。
79.其次,本实施例的抗原呈递预测模型还考虑了挖掘输入序列编码向量的深层特征,即让其非线性映射后的特征表达意义更加丰富,然后才能进行各种非线性组合,从而体现出更加强大的表达能力。另外,还需要结合序列位点的相互关系,考虑序列的局部性和整体性,这样才能更加全面的进行序列特征表达,因此,采用什么类型的算法结构进行建模,也是本实施例解决的技术问题之一。
80.基于此,参考图2所示,在本实施例中,首先,对训练数据集进行数据化预处理,并输入已初始化的算法模型进行训练,得到训练好具备预测能力的抗原呈递预测模型;接着,测试输入数据同样经过数字化预处理,输入已训练好的抗原呈递预测模型;最终得到每条输入数据的呈递score(分值),其值位于0~1,表示其呈递概率,越接近1,其呈递能力越强。
81.具体地,作为一实施例,如图1所示,本实施例提供的抗原呈递预测模型的训练方法,主要包括以下步骤:
82.步骤101、获取抗原序列相关的训练数据以及测试数据。
83.在本步骤中,获取抗原序列训练数据、用于表征抗原序列呈递概率的呈递概率训练数据和抗原序列测试数据。
84.具体地,所有抗原序列相关的数据分为训练数据和测试数据,这两者唯一的不同点在于训练数据除了输入数据(抗原序列训练数据)外还包含输入数据一一对应的输出数据,即表征输入的抗原序列呈递性的label值(0或1,0表示呈递性差,1表示呈递性强)。
85.步骤102、进行数据预处理。
86.在本步骤中,对获取到的抗原序列训练数据和抗原序列测试数据进行预处理。
87.作为一可选实施方式,在本步骤中,预处理包括无效数据过滤处理、字符串序列数字化处理及归一化处理。
88.具体地,对于输出数据不作任何处理,而对于输入数据,不管是训练数据中的输入数据还是测试数据中的输入数据都需要经过相同方式的预处理,包括无效数据过滤处理、字符串序列数字化处理。
89.其中,无效数据过滤主要是过滤掉一些不符合要求的数据,比如有位点字符错误的字符串数据、字符串长度、短肽长度《8的一些数据等(本实施例中输入的多肽的长度不固定),这些数据在本实施例的数据集中被认为是极小概率样本数据或错误数据,不参与模型训练,需要在训练集中进行过滤。而对于测试集,这类数据不直接过滤处理,但也不能输入模型,其表征呈递性的score数值不通过模型预测来得到,而是直接赋予数值0,表示呈递性低。
90.字符串序列数字化处理主要是针对输入中的短肽序列以及短肽对应的pseudo序
列(在编码mhc序列时,只考虑那些与肽紧密结合的残基,这些残基形成一个短序列,称为mhc伪序列(mhc pseudo sequence)),这些序列都是主要由氨基酸组成。而氨基酸一共有20种,可分别编码为0-19,考虑到序列中可能存在一些错误或者未知位点,可将这些位点设置为字符“unk”,对应编码为20。另外设置一个换行符“eos”,编码为21,便于训练模型结构中的embedding模块,详细的氨基酸及其对应编码方式如下所示:
91.alphabet_dict={'a':0,'c':1,'d':2,'e':3,'f':4,'g':5,'h':6,'i':7,'k':8,'l':9,'m':10,'n':11,'p':12,'q':13,'r':14,'s':15,'t':16,'v':17,'w':18,'y':19,'《unk》':20,'《eos》':21}。
92.alphabet_dict为编码字典,根据字典可查询到每个氨基酸所对应的编码数字,例如氨基酸'e'和'h'分别编码为3和6。
93.另外,需要进行的预处理操作便是归一化操作,主要是针对输入数据中的表达量特征。在本实施例中,其数值范围在0-170之间(大于170的表达量直接设置为170),为了便于模型训练收敛,需要将输入数据中的表达量特征数值范围归一化到0-1。
94.步骤103、将抗原序列训练数据输入至待训练的模型。
95.在本步骤中,将所述抗原序列训练数据输入至待训练的已初始化的抗原呈递预测模型。
96.作为一可选实施方式,抗原呈递预测模型主要包括特征编码模块、第一特征提取模块、第二特征提取模块及特征融合模块,当然本实施例并不具体限定抗原呈递预测模型的主要结构,可根据实际需求或可能出现的需求进行相应的选择及调整。
97.作为一优选实施方式,第一特征提取模块主要包括一维cnn模块,优选地,一维cnn模块包括三个并联的卷积结构,更优选地,参考图3所示,三个并联的卷积结构包括卷积核尺寸分别为1*1(conv 1d 1)、1*5(conv 1d 5)、1*9(conv 1d 9)的一层卷积层,当然本实施例并不具体限定第一特征提取模块的主要结构,可根据实际需求或可能出现的需求进行相应的选择及调整。
98.作为一优选实施方式,第二特征提取模块主要包括rnn模块,优选地,参考图3所示,rnn模块包括至少两层bigru(bigru_1、bigru_2),当然本实施例并不具体限定第二特征提取模块的主要结构,可根据实际需求或可能出现的需求进行相应的选择及调整。
99.作为一优选实施方式,参考图3所示,特征融合模块主要包括全连接(fc)模块(fc_linear),当然本实施例并不具体限定特征融合模块的主要结构,可根据实际需求或可能出现的需求进行相应的选择及调整。
100.具体地,参考图3所示,输入数据peptide_input、pseudo_input以及expression_input分别对应抗原序列预处理后的数字化输入、pseudo序列数字化后的数据以及归一化后的表达量数据。
101.对于输入peptide_input、pseudo_input每个位点只进行了编码,并没有进行特征化处理,假设预处理后的每个输入peptide_input和pseudo_input分别为和其中ai和bi均为整数,分别对应peptide序列和pseudo序列通过编码字典alphabet_dict编码得到的数字。将每个位点数字变成(1,1)的向量,则输入peptide_input和pseudo_input分别是维度为(n
p
,1)和(ns,1)的向量。
102.在本实施例中,使用word embedding方法来对氨基酸位点进行向量初始化。具体地,将序列空间中的某个氨基酸,通过一定的方法,映射或者说嵌入(embedding)到另一个数值向量空间,这样每个氨基酸同样可变成多维向量。在本发明中,主要是通过gensim工具包中的word2vec模块将每个氨基酸映射成一维向量,其中word2vec模块就是一个简单的神经网络模块,也需要数据训练,输入的是(ns,1)格式数据,输出(ns,n
emb
)格式数据,其中n
emb
便是1维扩展到高维后每个氨基酸特征向量长度,即每个位点的向量为(1,n
emb
)。
103.为使word embedding出来的向量具有较强的场景意义,需要提前使用抗原呈递相关训练数据集来训练word2vec模型(word2vec模型在整个算法模型中是一个模块,其中参数会参与训练,进行参数优化),让该模型学习序列表达意义,完成word2vec模型训练后,输入序列通过word2vec模型得到的embedding向量表达意义更加丰富,不同氨基酸之间的特征向量也不是完全的相互独立,存在一定的相关性,具体相关程度依赖于场景数据。
104.在模型结构上,参考图3所示,两个embedding通道出来的特征数据和expression_input通过concat操作级联到一起,构成单个输入特征数据。假设两个embedding通道出来的数据维度分别是(n
p
,n
emb
)和(ns,n
emb
),而expression_input维度为(1,1),第二个维度不为n
emb
,不能通过concat操作级联到一起,本发明所采取的措施是将expression_input复制n
emb
份,得到维度为(1,n
emb
)的数据,然后通过concat操作级联后得到的数据维度为(n
p
+ns+1,n
emb
),即在第一个维度上,将三个输入通过concat操作首尾相连得到n
p
+ns+1维度级联输入数据。
105.在本实施例中,利用特征提取能力更强的一维cnn神经网络结构来提取序列的特征,参考图3所示,图3中conv 1d为1维的卷积操作。为了提取更丰富的特征,本实施例选择三种尺度的卷积核来进行卷积操作,其卷积尺寸分别为1、5、9,对应的padding(填充)分别是0、2、4,即保证卷积前后序列长度不发生变化。三个通道的卷积操作完成后,分别得到维度为(n
p
+ns+1,cn1)、(n
p
+ns+1,cn5)和(n
p
+ns+1,cn9)的输出特征维度数据,其中cn1、cn5以及cn9分别对应1、5、9卷积核的个数,即扩展后的特征向量长度。得到三种输出后,再次通过concat操作,在第二个维度上进行级联操作,得到(n
p
+ns+1,cn)维度数据,其中cn等于cn1+cn5+cn9。
106.通过卷及操作提取到深层特征后,需要考虑序列的局部以及整体,本实施例设计的网络结构中,将完成卷积操作后得到的特征数据经过rnn网络层来进行前后特征向量的有序融合,可选用的rnn类型网络结构主要有一般的rnn、lstm、bilstm、gru以及bigru等,本实施例选择bigru这种考虑正向和反向且运行效率较高的rnn类型网络结构来提取特征。相对于其他类型的rnn结构,bigru能保证效率的同时,算法效果最佳。
107.为了让特征提取和融合更加充分,在模型中,本实施例设计了两层bigru结构。通过两层bigru结构后,得到的融合特征维度为(n
p
+ns+1,rn),其中rn便是bigru结构中的隐藏层单元数,也是输出特征第二个维度大小。
108.完成以上的卷积特征提取和有序特征融合后,接下来便是利用全连接网络层fc来进行维度映射,即所有特征按一定规则融合输出二维特征。不过,考虑到bigru操作可能会丢失一些重要的卷积特征,本实施例利用concat操作将卷积特征和bigru特征进行了级联,得到输出为(n
p
+ns+1,rn+cn)的特征数据。最后经过fc层的特征融合和降维操作得到(1,2)的输出数据,而本实施例的算法模型是预测输入序列的呈递概率,即输出一个0-1之间的分
数值,因此,首先需要将维度为(1,2)的输出结果进行softmax计算,其中softmax函数如下:
[0109][0110]
其中i为输出结果(1,2)中每个元素,通过该函数的变换后得到输出结果位于0-1之间的概率值,然后取第二个维度的数值,便得到一个数值位于0-1之间的score值。
[0111]
步骤104、通过模型进行呈递预测处理以预测出抗原序列训练数据对应的呈递概率。
[0112]
在本步骤中,通过抗原呈递预测模型对抗原序列训练数据进行呈递预测处理以预测出抗原序列训练数据对应的呈递概率。
[0113]
作为一优选实施方式,在本步骤中,通过抗原呈递预测模型的特征编码模块对抗原序列训练数据进行氨基酸编码及向量映射处理;还通过抗原呈递预测模型的第一特征提取模块对进行氨基酸编码及向量映射处理后的抗原序列训练数据进行多肽序列一维卷积特征提取处理;还通过抗原呈递预测模型的第二特征提取模块对进行多肽序列一维卷积特征提取处理后的抗原序列训练数据进行多肽序列有序特征提取处理;还通过抗原呈递预测模型的特征融合模块对进行多肽序列有序特征提取处理后的抗原序列训练数据进行特征融合处理。
[0114]
具体地,模型在刚开始创建并完成初始化后,模型涉及每一个参数都是随机化参数,模型不具备任何的表达能力,模型接受输入数据后的输出结果没有任何实际意义。所以,模型需要经过训练,让其输出结果具有一定的意义。
[0115]
模型可以理解为函数,所谓模型训练便是用预处理好的训练数据,通过一定的方法确定模型的参数,一旦参数数值确定即训练完成后,输入代入模型,输出有意义的数值。在本发明中,训练数据中的输出label表示抗原数据的呈递能力,所以通过大量抗原输入和表征其呈递能力的输出label一一对应的数据训练后,模型便通过一定的方法学习到了抗原输入数据和输出label之间的映射关系。一旦训练完成,即确定模型参数后,模型便具有预测输入抗原数据呈递性能力。
[0116]
步骤105、根据预测出的呈递概率及呈递概率训练数据训练出模型。
[0117]
在本步骤中,根据预测出的呈递概率及呈递概率训练数据训练出抗原呈递预测模型。
[0118]
步骤106、根据测试数据测试训练后的模型。
[0119]
在本步骤中,将抗原序列测试数据输入至训练出的抗原呈递预测模型,以对训练出的抗原呈递预测模型进行测试,以获取训练并测试后的抗原呈递预测模型。
[0120]
具体地,模型测试即实现对输入抗原数据呈递性的预测能力,可以直接用本实施例所描述的预处理后的测试数据进行模型测试。
[0121]
下面以具体的实验数据来进行算法模型的实施操作,更详细的描述算法的子模块结构以及相应的输入和输出。
[0122]
假设实验输入数据由两组数据构成批次处理,数据为:
[0123]
peptide_input(2,12)—》[[
‘
l’,
‘
d’,
‘
e’,
‘
a’,
‘
l’,
‘
q’,
‘
w’,
‘
h’,
‘
l’,
‘
l’,
‘
l’,
‘
l’],
[0124]
[
‘
d’,
‘
e’,
‘
a’,
‘
l’,
‘
q’,
‘
w’,
‘
h’,
‘
d’,
‘
l’,
‘
l’,
‘
l’,
‘
l’]]
[0125]
pseudo_input(2,35)—》[['y','y','s','e','y','r','n','i','y','a','q','t','d','e','s','n','l','y',
[0126]
'l','r','y','n','f','y','t','w','a','v','l','t','y','t','w','y','a'],
[0127]
['y','y','s','e','y','r','n','i','y','a','q','t','d','e','s','n','l','y',
[0128]
'l','r','y','n','f','y','t','w','a','v','l','t','y','t','w','y','a']]
[0129]
expression_input(2,1)—》[[[3.6525]],[[3.6525]]]
[0130]
1:实验输入字符矩阵数据进入到的第一个网络层便是embedding层,该层主要由word2vec模型组成,其作用是将输入的peptide_input和pseudo_input的字符矩阵将全部编码成数字矩阵x_pep和x_pse:
[0131]
peptide_input(2,12)—》[[9,2,3,0,9,13,18,6,0,0,0,0],[2,3,0,9,13,18,6,2,0,0,0,0]]
[0132]
pseudo_input(2,35)—》[[19,19,15,3,19,14,11,7,19,0,13,16,2,3,15,11,9,19,9,14,19,11,4,19,16,18,0,17,9,16,19,16,18,19,0],[19,19,15,3,19,14,11,7,19,0,13,16,2,3,15,11,9,19,9,14,19,11,4,19,16,18,0,17,9,16,19,16,18,19,0]]
[0133]
经过编码后的数字矩阵数据x_pep和x_pse以及另外一个输入接着通过concat操作进行级联操作变成一个级联矩阵数据x,其维度为(2,48,25),其中25便是上节描述的特征向量维度n
emb
,处理后的实验数据为:
[0134][0135]
2:经过concat后的数据接下来便是经过三个并联的卷积结构进行第一步的特征提取,三个卷积结构分别由卷积核大小为1*3、1*5、1*9的一层卷积层构成。卷积操作的输入特征通道数均为n
emb
,而输出通道均为64,即上节中的cn1、cn5以及cn9均为64,级联矩阵数据x经过分别经过三个并联的卷积结构操作后,得到维度均为(2,48,64),接着进行concat级联操作得到输出特征矩阵x_conved,其维度为(2,48,192),实验数据如下所示:
[0136][0137]
3:经过初步特征提取后得到的卷积特征矩阵x_conved接着进行经过由两层bigru组成的网络层进行第二步的特征提取,输入实验数据x_conved经过两层bigru处理后便得到维度为(2,48,192)的输出特征矩阵x_bigrued,其中192便是上节中描述的
rn
,即bigru并没有让特征维度大小进行改变,随后x_bigrued便和x_conved进行concat操作,得到最后提取到特征矩阵数据x_fea,其维度为(2,48,384),实验数据x_fea结果如下所示:
[0138][0139]
4:经过特征提取后得到的特征矩阵数据x_fea接下来便经过最后的全连接层来进行最后的特征融合,得到融合后的输出矩阵x_out,其维度为(2,2),实验数据x_out结果如下所示:
[0140]
[[3.4756,-3.5319],
[0141]
[[4.6604,-4
·
6849]]
[0142]
最后经过上节中softmax函数处理,便得到最后的预测分数y=[[9.99098e-01,9.04278e-04],[9.99912e-01,8.73592e-05]]。其中两组数据,每组数据是一个二维的向量,向量第一个维度表示不呈递的概率,第二个维度表示呈递的概率。为了得到呈递概率,选择每组数据的第二个维度,得到最终两组数据的呈递score为[9.04278e-04,8.73592e-05]。
[0143]
作为另一实施例,如图4所示,本实施例还提供一种抗原呈递预测方法,主要包括以下步骤:
[0144]
步骤201、获取抗原序列数据;
[0145]
步骤202、将抗原序列数据输入至训练出的抗原呈递预测模型,具体将抗原序列数据输入至通过如上述的抗原呈递预测模型的训练方法训练出的抗原呈递预测模型;
[0146]
步骤203、通过训练出的抗原呈递预测模型对抗原序列数据进行呈递预测处理;
[0147]
步骤204、输出预测出的抗原序列数据对应的呈递概率。
[0148]
以下具体说明本实施例提供的抗原呈递预测模型的训练方法及其预测方法的具体操作环境及使用场景。
[0149]
可选环境的说明:
[0150]
在本实施例中,实施所需计算资源可选择gpu(图形处理器),也可选择cpu(中央处理器),由于使用gpu相对效率更高,因此优选gpu。本实施例基于ubuntu20.04环境下的python(计算机编程语言)开发,因此需要配置相应的运行环境,具体的可选环境配置要求如下所示:
[0151][0152][0153]
环境安装完成后,实验人员可直接在工程目录下执行以下代码运行命令便可测试自己数据:
[0154]
python predict.py
‑‑
test_csv_path test_input.csv
‑‑
pred_save_path predict_result.csv
[0155]
其中,predict.py是代码python代码文件,命令中需传入两个命令参数test_csv_path和pred_save_path,分别代表待测试的csv格式多肽抗原数据文件路径以及预测结果保存路径。
[0156]
其中输入数据样例如下所示:
[0157]
peptide expmhc
[0158]
liedhfdvt 14.5525 b4002
[0159]
iqvvgvetd 0.00125 b4002
[0160]
tktqkirll 0.02625 b4002
[0161]
gislqkklq 41.2275 b4002
[0162]
lkakiinic 0.005 b4002
[0163]
spiiilnlv 10.195 b4002
[0164]
tpserptaq 7.33625 b4002
[0165]
如上所示,输入文件中包含多肽抗原数据peptide、对应的表达量exp以及对应分型mhc,第三列mhc表示该多肽属于哪种分型,并不是算法输入必须的数据。
[0166]
算法模型输入还需要peptide对应假序列,这个数据不需要用户输入,数据预处理流程中程序会根据输入peptide序列对应mhc来查询对应的pseudo序列(每条peptide序列都有对应的mhc,而mhc可以查询到对应得pseudo序列,不同mhc也可能具有相同的pseudo序列;输入mhc只是便于数据分析,例如保证训练集中各种mhc数据平衡,测试查看各种mhc效果,实际上mhc不属于模型输入),而查询字典则保存在本地。
[0167]
如果出现查询不到pseudo序列情况,则将输入序列判定为无效序列数据。即当实验数据集中的mhc种类有限,pseudo也有限时,训练好的模型在后期使用时候可能出现新的分型查询不到pseudo序列情况,这时候将pseudo序列对应的peptide序列判定为无效序列。
[0168]
准备好测试数据后,执行命令便可以得到每条多肽序列的呈递分数,其结果保存在predict_result.csv文件中,结果样例如下所示:
[0169]
peptide score mhc predict
[0170]
liedhfdvt 0.0212 b4002 0
[0171]
iqvvgvetd 0.013 b4002 0
[0172]
tktqkirll 0.0426 b4002 0
[0173]
gislqkklq 0.0082 b4002 0
[0174]
lkakiinic 0.0255 b4002 0
[0175]
spiiilnlv 0.0058 b4002 0
[0176]
tpserptaq 0.0027 b4002 0
[0177]
rallnlhtd 0.0123 b4002 0
[0178]
其中第二列为每条多肽的呈递概率,第四列为0或者1,即概率大于0.5则为1,否则为0,代表该条抗原多肽是否可以被呈递。
[0179]
本实施例提供的抗原呈递预测模型的训练方法及其预测方法,利用深度学习技术有效地建立了基于抗原序列的神经网络算法模型,在大量的抗原序列训练数据以及对应表征呈递能力的呈递概率数据的训练下,模型能够挖掘输入抗原序列的深层特征,并且学习到这些深层特征与label之间的一一对应关系,在模型完成参数自动优化后,模型便具有对输入抗原序列数据的预测判决能力,从而有效地解决了预测新抗原呈递能力较差的问题,提升了预测抗原呈递能力的准确性和效率。
[0180]
本实施例提供的抗原呈递预测模型的训练方法及其预测方法,针对输入序列编码问题,提出用nlp中常用的word embedding方法来对氨基酸位点进行向量初始化,这种编码方式在数据对应的场景下适用性强,这样能够让氨基酸初始化向量表达更加有意义。
[0181]
本实施例提供的抗原呈递预测模型的训练方法及其预测方法,针对序列位点深层特征提取问题,考虑了利用特征提取能力更强的一维cnn神经网络结构来提取序列的特征。
[0182]
本实施例提供的抗原呈递预测模型的训练方法及其预测方法,为了考虑序列中位点的相关性以及序列整体性,模型搭建用到rnn模块这种具有记忆功能的针对序列建模的神经网络技术。
[0183]
本实施例提供的抗原呈递预测模型的训练方法及其预测方法,在保证准确的预测抗原呈递能力的同时,还能够保持高效性。本实施例利用gpu加速计算每秒能够处理10万个
样本,非常高效,而且本实施例涉及的模型占用大小只有3mb左右,极大地降低了占用空间。
[0184]
图5为根据本实施例提供的一种电子设备的结构示意图。电子设备包括存储器、处理器及存储在存储器上并可在处理器上执行的计算机程序,处理器执行程序时实现如上实施例中的抗原呈递预测模型的训练方法或抗原呈递预测方法。图5显示的电子设备30仅仅是一个示例,不应对本公开实施例的功能和使用范围带来任何限制。
[0185]
如图5所示,电子设备30可以以通用计算设备的形式表现,例如其可以为服务器设备。电子设备30的组件可以包括但不限于:上述至少一个处理器31、上述至少一个存储器32、连接不同系统组件(包括存储器32和处理器31)的总线33。
[0186]
总线33包括数据总线、地址总线和控制总线。
[0187]
存储器32可以包括易失性存储器,例如随机存取存储器(ram)321和/或高速缓存存储器322,还可以进一步包括只读存储器(rom)323。
[0188]
存储器32还可以包括具有一组(至少一个)程序模块324的程序/实用工具325,这样的程序模块324包括但不限于:操作系统、一个或者多个应用程序、其它程序模块以及程序数据,这些示例中的每一个或某种组合中可能包括网络环境的实现。
[0189]
处理器31通过执行存储在存储器32中的计算机程序,从而执行各种功能应用以及数据处理,例如本公开如上实施例中的抗原呈递预测模型的训练方法或抗原呈递预测方法。
[0190]
电子设备30也可以与一个或多个外部设备34(例如键盘、指向设备等)通信。这种通信可以通过输入/输出(i/o)接口35进行。并且,模型生成的设备30还可以通过网络适配器36与一个或者多个网络(例如局域网(lan),广域网(wan)和/或公共网络,例如因特网)通信。如图5所示,网络适配器36通过总线33与模型生成的设备30的其它模块通信。应当明白,尽管图中未示出,可以结合模型生成的设备30使用其它硬件和/或软件模块,包括但不限于:微代码、设备驱动器、冗余处理器、外部磁盘驱动阵列、raid(磁盘阵列)系统、磁带驱动器以及数据备份存储系统等。
[0191]
应当注意,尽管在上文详细描述中提及了电子设备的若干单元/模块或子单元/模块,但是这种划分仅仅是示例性的并非强制性的。实际上,根据本公开的实施方式,上文描述的两个或更多单元/模块的特征和功能可以在一个单元/模块中具体化。反之,上文描述的一个单元/模块的特征和功能可以进一步划分为由多个单元/模块来具体化。
[0192]
本实施例还提供了一种计算机可读存储介质,其上存储有计算机程序,程序被处理器执行时实现如上实施例中的抗原呈递预测模型的训练方法或抗原呈递预测方法中的步骤。
[0193]
其中,可读存储介质可以采用的更具体可以包括但不限于:便携式盘、硬盘、随机存取存储器、只读存储器、可擦拭可编程只读存储器、光存储器件、磁存储器件或上述的任意合适的组合。
[0194]
在可能的实施方式中,本公开还可以实现为一种程序产品的形式,其包括程序代码,当程序产品在终端设备上执行时,程序代码用于使终端设备执行实现如上实施例中的抗原呈递预测模型的训练方法或抗原呈递预测方法中的步骤。
[0195]
其中,可以以一种或多种程序设计语言的任意组合来编写用于执行本公开的程序代码,程序代码可以完全地在用户设备上执行、部分地在用户设备上执行、作为一个独立的
软件包执行、部分在用户设备上部分在远程设备上执行或完全在远程设备上执行。
[0196]
虽然以上描述了本公开的具体实施方式,但是本领域的技术人员应当理解,这仅是举例说明,本公开的保护范围是由所附权利要求书限定的。本领域的技术人员在不背离本公开的原理和实质的前提下,可以对这些实施方式做出多种变更或修改,但这些变更和修改均落入本公开的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1