基于组织病理学图像和基因表达数据的胃癌患者生存风险预测方法
1.本发明涉及癌症风险预测模型构建技术领域,尤其是涉及一种基于组织病理学图像和基因表达数据的胃癌患者生存风险预测方法。
背景技术:
2.组织病理学图像作为肿瘤诊断和预后的临床金标准,指导临床医生做出更精确的治疗决策。病理学家通过在显微镜下评估肿瘤细胞的形态等特征进行分级。然而,手动评估组织病理学图像是非常耗时、主观且不可重复的,特别是对于在偏远地区工作的病理学家。因此,直接从组织病理学图像预测肿瘤患者生存风险的全自动模型引起了极大的关注。这种计算机辅助工具可以用来提高病理学家的效率和准确性,并最终为患者提供更好的治疗。
3.准确预测患者的生存时间或进展将有助于临床医生做出早期治疗决定,这对患者的健康至关重要。例如,可以建议低风险患者选择相对保守的治疗策略。预测患者生存风险的方法主要分为三类:基于图像结构化特征的模型、基于深度卷积网络的模型和多模态融合模型。
4.基于图像结构化特征的模型通常从非结构化的组织病理学图像中提取结构化的图像特征,包括细胞形状、大小和纹理。然而,结构化图像特征表示图像信息的能力有限。近年来,研究人员试图利用深度学习直接从组织病理学图像中学习生存相关信息。
5.基于卷积神经网络(cnn)的模型通常使用cnn进行生存分析,用非线性深度全连接网络代替传统cox比例风险模型的线性风险函数。根据是否需要人工标记感兴趣的区域(region of interests,rois),基于cnn的生存模型可以分为两类:基于rois的方法和基于整张组织病理学图像(whole slide image,wsi)的方法。基于rois的方法需要病理学医师在wsi上标注rois,不是端到端的全自动分析。基于wsi的方法直接使用wsi来预测患者的预后。据调查所知,目前还没有一种利用胃癌组织病理图像预测胃癌患者生存风险的模型。
6.多模态融合模型整合了组织病理学图像和其他组学数据,以提高预后的准确性。例如gscnn方法将胶质母细胞瘤患者的基因组生物标志物和病理学图像整合到一个统一的预测框架中,预测肿瘤患者的生存。然而gscnn忽略了患者的临床信息。据调查所知,目前还没有一个整合组织病理图像、临床数据和基因表达数据的胃癌生存预测模型。
技术实现要素:
7.本发明的目的就是为了克服上述现有技术存在的缺陷而提供一种基于组织病理学图像和基因表达数据的胃癌患者生存风险预测方法。
8.本发明的目的可以通过以下技术方案来实现:
9.基于组织病理学图像和基因表达数据的胃癌患者生存风险预测方法,包括如下步骤:
10.步骤1:数据收集。获得胃癌患者的组织病理学图像、临床数据和基因表达数据。
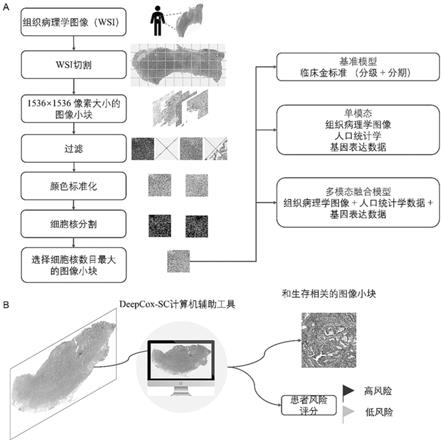
11.步骤2:基于组织病理学图像建立胃癌患者生存风险预测模型。对组织病理学图像进行预处理(切割成图像小块、颜色标准化以及细胞核分割处理),选取组织病理学图像中细胞核数目最大的图像小块,基于卷积神经网络构建生存预测模型,模型输出为deepcox-sc风险评分,表示估计的患者死亡风险。
12.步骤3:建立多模态融合预测模型。基于deepcox-sc风险评分,人口统计学和基因表达数据构建deepcox-sc多模态融合模型。
13.所述步骤2基于组织病理学图像建立胃癌患者生存风险预测模型具体包括:
14.步骤2.1:整张组织病理学图像(whole slide image,wsi)裁剪成图像小块。将20倍放大的wsi裁剪成1536
×
1536像素的图像小块,空白背景超过30%的图像小块被过滤掉。
15.步骤2.2:颜色标准化。采用现有macenko方法将图像颜色归一化到标准染色的组织病理学图像。
16.步骤2.3:细胞核分割。采用分层多级阈值方法(hierarchical multilevel thresholding)对每个图像小块的细胞核进行分割,然后选择细胞核数量最大的图像小块。
17.步骤2.4:将细胞核数目最大的图像小块作为神经网络的输入,最终输出患者死亡风险的预测值(deepcox-sc风险评分)。神经网络通过卷积层和池化层提取组织病理学图像的空间特征,然后将这些特征连接到全连接层上。该卷积神经网络的输出为单节点,代表估计的患者死亡风险,即deepcox-sc风险评分。
18.所述步骤3建立多模态融合预测模型具体包括:
19.步骤3.1:高维基因数据变量选择。高维基因表达数据经过确定独立筛选方法和带lasso惩罚项的cox模型进行变量选择,并获得十个基因,包括chaf1a、repin1、serpine1、htra3、pwp2、gpr173、ncln、nt5e、myl4和ywhabp2。
20.步骤3.2:基于deepcox-sc风险评分、人口统计学(年龄)和步骤3.1筛选到的10个基因构建deepcox-sc多模态融合模型,预测胃癌患者生存风险。
21.本发明提供的基于组织病理学图像和基因表达数据的胃癌患者生存风险预测方法,相较于现有技术至少包括如下有益效果:
22.1)相比于随机选择图像小块,本发明经过图像预处理(切割成图像小块、颜色标准化以及细胞核分割处理),选取细胞核数目最大的图像小块,证明了这些图像小块包含更多和生存相关的信息,提高对胃癌患者生存风险预测能力。
23.2)传统方法提取组织病理学图像的特征(将非结构化的图像转化为结构化的图像特征),该结构化特征表示图像的能力很有限。本发明直接采用组织病理学图像作为深度卷积神经网络的输入构建胃癌患者生存风险预测模型。
24.3)相比仅基于单个模型数据建模,本发明利用组织病理学图像,临床信息和基因表达数据构建多模态融合模型预测胃癌患者生存风险。相比于单模态模型,多模态融合模型提高了模型预测准确度。
25.4)本发明方法整合组织病理学图像、临床数据和基因表达数据,采用深度卷积神经网络构建多模态融合预测模型,相比仅基于单模态数据的预测模型,预测准确度得到了显著的提升。本发明选取细胞核数目最大的图像小块构建预测模型,相比于随机选择图像小块的方法,提高了模型预测效果和稳健性。模型准确性超过了病理学家手动标注的准确
性,可作为辅助工具来提高病理学家的效率和准确性。
附图说明
26.图1为实施例中deepcox-sc模型架构示意图,其中,部分a为deepcoxsc预处理流程示意图,部分b为deepcox-sc模型以wsi作为输入,输出患者细胞核数目最大的图像小块和预测的风险评分的原理示意图。
27.图2为实施例中森林图展示单因素和多因素cox回归分析结果,其中,部分a为单因素cox回归分析,变量采用分类编码方式;部分b为单因素cox回归分析,变量采用连续性编码方式;部分c为多因素cox回归分析。
28.图3为实施例中不同模型的c-index和时间依赖性auc值,其中,部分a为采用十折交叉验证比较不同模型的c-index,实线灰色小提琴图表示仅基于图像的模型:deepcox-sc模型,随机选择图像小块模型,提取wsi结构化特征的模型;虚线灰色小提琴图表示整合组织病理学图像和人口统计数据(年龄)的模型;实线白色小提琴图为整合组织病理学图像、人口统计数据(年龄)和基因表达数据的多模态融合模型;deepcox-sc多模态融合模型与多模态融合基准模型(包括等级、阶段、年龄和基因)c-index基本相同;部分b为不同模型的1年auc值。
29.图4为实施例中高风险组和低风险组的生存差异,其中,部分a为分级ii级患者deepcox-sc风险评分的kaplan-meier图,部分b为分级iii级患者kaplan-meier图,部分c为分期ii期患者kaplan-meier图,部分d为分期iii期患者kaplan-meier图。
具体实施方式
30.下面结合附图和具体实施例对本发明进行详细说明。显然,所描述的实施例是本发明的一部分实施例,而不是全部实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动的前提下所获得的所有其他实施例,都应属于本发明保护的范围。
31.实施例
32.本发明涉及一种基于组织病理学图像和基因表达数据的胃癌患者生存风险预测方法,该方法旨在提供一个完全自动化的深度卷积神经网络模型,直接从胃癌组织病理学图像中预测患者生存风险(deepcox-sc模型)。利用tcga(the cancer genome atlas,癌症基因组数据集)中的胃癌数据,发现deepcox-sc的预后准确性超过了病理学家手动标注的准确性。通过进一步整合组织病理学图像、临床数据和高维基因表达数据构建deepcox-sc多模态融合模型,进一步提高预测精度。本发明方法的实现方案具体包括以下内容:
33.1、数据集准备
34.收集胃癌患者的组织病理图像、临床数据和基因表达数据。组织病理图像为福尔马林固定石蜡包埋(formalin-fixed paraffin-embedded,ffpe)的组织,ffpe切片是医学诊断的金标准。
35.2、wsi预处理
36.步骤1:将wsi裁剪成图像小块。wsi通常超过100000
×
100000像素大小。直接使用wsi作为cnn的输入是不现实的,输入图像太大会增加需要估计的参数量和计算能力。考虑到这些因素,本发明wsi被裁剪成1536
×
1536像素的图像小块。由于wsi的边缘包含白色背
景,当将wsi处理成图像小块时,有些图像小块包含白色背景。空白背景超过30%的图像小块被过滤掉。
37.步骤2:颜色标准化。许多因素(如病理学图像染色和wsi数字化过程)都会导致两张组织病理学图像的外观存在差异。这种差异显著影响了cnn模型的性能。因此,使用现有macenko方法将图像颜色归一化到标准染色的组织病理学图像。标准染色的组织病理学图像从histomicstk中下载获得。
38.步骤3:细胞核分割。经过图像小块过滤后,每个样本平均剩余2937个图像小块(1536
×
1536像素)。每个样本的所有图像小块不能直接用于cnn模型。细胞核的数量、大小、结构等细胞特征在肿瘤的诊断和肿瘤分级(grading)中起着重要作用。采用分层多级阈值方法(hierarchical multilevel thresholding)对每个图像小块的细胞核进行分割,然后选择细胞核数量最大的图像小块。选取的图像小块(1536
×
1536像素)由9个512
×
512像素大小的小块构成,本发明使用cellprofiler公共软件提取512
×
512像素大小的小块的1100个特征。单因素cox回归结果显示,cellprofiler公共软件提取的1100个特征中,基于zernike矩提取细胞核面积特征(median_primaryobject_areashape_zernike_7_7)的p值最小,系数绝对值最大。本发明在9个512
×
512像素大小的小块中选择特征(median_primaryobject_areashape_zernike_7_7)最小的图像小块,因为该特征的系数为负。用选取的含有更多和生存信息相关的图像小块(512
×
512像素)来训练cnn模型。
39.3、高维基因数据预处理
40.首先使用确定独立筛选(sure independent screening,sis)变量选择方法将60483个基因减少到中等维度(n/log(n),其中n为患者数量)。然后,通过在cox比例风险模型中加入lasso惩罚项进行变量选择。最后将十折交叉验证中系数不为零的基因取交集。共获得10个基因:chaf1a、repin1、serpine1、htra3、pwp2、gpr173、ncln、nt5e、myl4、ywhabp2。
41.4、deepcox-sc模型架构
42.图1为deepcox-sc模型架构。wsi预处理(将wsi裁剪成小块、过滤和颜色归一化)后,每个患者大约有数千个图像小块。以往的许多研究随机选择一个或多个小块作为cnn模型的输入。本发明推测细胞核数目最多的小块包含更多和生存相关的信息。因此,本发明采用分层多级阈值法分割细胞核,选择细胞核数目最大的小块。deepcox-sc模型的输入为细胞核数目最大的图像小块,最终输出对患者死亡风险的预测(deepcox-sc风险评分)。deepcox-sc模型利用在imagenet上预训练的inceptionresnetv2卷积层提取组织病理学图像的空间特征,然后将这些特征连接到全连接层上。最后一层为单个节点,代表估计的风险函数利用患者生存时间和事件(死亡)优化cox模型似然函数(公式(1))。
[0043][0044]
式中,θ表示卷积神经网络参数;共有n个样本,表示生存时间大于第i个样本的所有个体集合;δi为第i个样本是否删失的示性函数,1表示第i样本死亡,0表示第i个样本删失。
[0045]
deepcox-sc多模态融合模型整合基于组织病理学图像获得的deepcox-sc风险评分,临床资料(年龄)和经变量筛选后获得的10个基因表达数据,提高胃癌患者生存预测。
[0046]
5、deepcox-sc模型训练
[0047]
十折交叉验证:使用十倍交叉验证对模型进行训练。采用分层随机抽样将数据划分为十折,每折中的删失率和时间分布保持一致。36个患者含有两张wsi,如果一个患者被分配到训练集,那么该患者的所有的wsi均被分配到训练集。该方法保证了一个样本的组织病理学图像同时存在于训练集和验证集中,有效避免了信息泄露。
[0048]
数据增强:通过数据增强,防止训练数据不足导致过拟合,提高泛化性能。本发明对训练数据进行了40度的随机旋转,水平和垂直随机翻转。将训练集和验证集图像的像素值归一化到0-1。
[0049]
超参数:采用adam优化器最小化损失函数(式(1)),初始学习率(learning rate)为0.002,衰减(decay)为0.05。模型训练时的批量大小(mini-batch size)为32,训练周期(epoch)为100次。为了避免过拟合,根据验证集的性能让训练过程早停。当验证集上的损失函数(loss)在10个epoch没有减少时,训练过程将中断。
[0050]
硬件和软件:使用tensorflow-v2.2.0框架在上海交通大学pi 2.0高性能超算上进行训练,使用nvidia tesla v100,每个gpu 32gb内存。使用histomicstk(https://digitalslidearchive.github.io/histomicstk/)进行基础组织病理学图像分析。
[0051]
6、模型评估
[0052]
采用验证集上的harrell’s c-index和时间依赖性auc值评估预测精度。c-index用于衡量验证集的预测风险和实际生存之间的一致性。计算10个验证集上c-index和auc值的平均值和标准差(sd)。采用wilcoxon符号秩检验比较不同模型之间的性能差异。进行单因素和多因素cox回归分析,采用似然比检验计算多因素cox模型的p值。绘制kaplan-meier生存曲线,采用log-rank检验比较各组之间的生存差异。所有的统计分析都在r版本3.6.1中执行。
[0053]
为验证本发明的准确性,本实施例的实例验证内容如下:
[0054]
1)数据集
[0055]
胃癌的组织病理图像、临床数据和基因表达数据来源于the cancer genome atlas(https://portal.gdc.cancer.gov/projects/tcga-stad)数据集(tcga)。首先,本实施例下载了416名患者的442张组织病理学切片(whole-slide image,wsi),其中36名患者含有两张wsi。进一步排除41例没有20倍放大的wsi的患者和18例缺失生存数据的患者。最后,对357例患者的382张wsi进行分析。表1为357例胃癌患者(男性234例,女性123例)的临床特征。3例患者的年龄缺失,354例患者诊断时的中位数年龄为66.5岁。删失率(censoring rate)为0.6(142例死亡)。存活患者的中位生存时间为357天,死亡患者的中位生存时间为616天。
[0056]
临床分期(stage)以美国癌症联合委员会(ajcc)胃癌tnm分期系统为基础。临床分期表示原发肿瘤的大小和癌细胞在体内扩散的程度。临床分期与癌症预后密切相关,通常情况下低分期预后较好。病理学分级(grade)取决于癌细胞在显微镜下的形态,根据who标准进行分类。
[0057]
表1胃癌患者的临床特征
[0058][0059]
2)单因素和多因素cox回归
[0060]
本实施例使用10折交叉验证来训练和验证预后模型(deepcox-sc模型)。模型输出为deepcox-sc模型预测的风险评分,即deepcox-sc风险评分,表示患者的死亡风险。整合验证集中患者的deepcox-sc风险评分。根据deepcox-sc风险评分中位数,将患者分为高风险组和低风险组。
[0061]
本实施例采用单因素和多因素cox回归模型评估deepcox-sc风险评分的预后能力。在单因素分析中,将deepcox-sc风险评分和临床资料(年龄、性别、种族、分期和分级)编码为连续和分类变量(图2中a部分和图2中b部分)。临床分期和分级是影响预后的重要因素,分期和分级越高,死亡风险越高。这也可以从分期和分级的风险比(hazard ratio,hr)
中看出,分期和分级越高,风险比越高(图2中a部分)。ii期、iii期、iv期的风险比分别为1.936、3.015、5.856(i期为参照)。g2级、g3级、gx级的风险比分别为1.254、1.719、2.287(g1为参考)。仅当临床分级编码为连续变量时,分级与生存风险显著相关。无论分期为分类变量还是连续变量,均能显著预测生存。然而,连续性变量的p值更显著(似然比检验,2e-07vs 5e-06)。为了考虑这种相关性,本实施例在多因素分析中将分期和分级编码为连续变量(图2中c部分)。
[0062]
为了评估deepcox-sc风险评分的独立预后能力,本实施例进行了多因素分析。多因素分析包括deepcox-sc风险评分、年龄、性别、分期和分级。在调整年龄、性别、分期和分级后,deepcox-sc风险评分仍是生存的一个重要预测变量(hr:1.528,95%ci:1.305-1.78,p值=1.445e-7)。年龄(hr:1.026,95%ci:1.009-1.044,p值=2.18e-03),分期(hr:1.784,95%ci:1.429-2.227,p值=3.26e-07)和分级(hr:1.388,95%ci:1.021-1.888,p值=3.65e-02)在多因素cox回归模型中也具有显著性。
[0063]
3)评价deepcox-sc模型的预测精度
[0064]
本实施例采用十折交叉验证来评估预测的准确性。预测的准确性通过c-index和时间依赖性auc值来衡量。c-index表示预测风险与实际生存之间的一致性。c-index范围在0-1之间,c-index得分越高,预测越准确。对仅基于图像的方法,deepcox-sc与另外两种模型进行比较:随机选择图像小块的模型和基于结构化特征的模型(图3中的实线灰色小提琴图)。
[0065]
随机选择图像小块的模型:该模型随机选择图像小块作为cnn模型的输入。deepcox-sc模型选择细胞核数目最多的图像小块,进一步说明该图像小块含有更多和生存相关的信息。
[0066]
基于结构化特征的模型:该模型提取图像小块的结构化特征。本实施例使用cellprofiler公共软件从每个图像小块中提取特征。采用cellprofiler公共软件中的染色分离模块(unmixcolour)将组织病理图像分离为苏木精染色灰度图像和伊红染色灰度图像。对于每个图像小块,使用cellprofiler公共软件中的“纹理测量”、“强度测量”、“大小形状测量”、“粒度测量”和“对象强度分布测量”模块获得1100个特征。计算每个图像小块各特征的均值和标准差。首先使用确定独立筛选(sure independent screening,sis)变量选择方法将1100个特征减少到中等维度(n/log(n),其中n为患者数量)。然后,通过在cox比例风险模型中加入lasso惩罚项进行变量选择。最后将十折中系数不为零的特征取并集(35个特征)进行cox回归分析。由于特征的交集为零,所以计算并集。
[0067]
对于胃癌患者,deepcox-sc模型预测生存的c-index和1年的auc分别为0.660
±
0.057和0.701
±
0.146(均值
±
标准差)(图3中a部分、图3中b部分)。deepcox-sc模型超过了随机选择图像小块的模型(c-index 0.609
±
0.078)。这表明细胞核数目最大的图像小块能更好的预测生存。细胞核数目最大的图像小块提高了8.37%的性能。基于结构化特征的模型性能较差(c-index 0.601
±
0.065)。
[0068]
单因素和多因素cox回归模型结果显示,年龄是影响生存的重要因素。如图3虚线灰色小提琴图所示,本实施例进一步整合了deepcox-sc风险评分和年龄(基于图像和年龄的模型)。为了比较,本实施例建立了基于临床分级、分期和年龄的基准模型。如前所述,考虑到临床分级、分期与患者生存风险的相关性,将分级和分期编码为连续变量。
[0069]
基准模型包括临床分级、分期和年龄,该基准模型的c-index为0.649
±
0.099(图3中a部分、图3中b部分的虚线灰色小提琴图)。整合deepcox-sc风险评分和人口统计学(年龄)的结果略好于基准模型(c-index为0.667
±
0.050)。综上所述,全自动化deepcox-sc模型可以直接用病理图像和临床数据预测患者生存风险,预测的准确性与病理学家的准确性相当。
[0070]
4)整合基因表达数据,提高预后准确性
[0071]
多模态融合模型整合组织病理学图像、临床资料(年龄)和基因表达数据(图3中实线白色小提琴图)。高维基因表达数据的预处理类似于组织病理学图像特征的预处理,即使用sis进行初步筛选,然后再基于lasso惩罚项进行变量选择。最后获得十折中系数不为零的基因交集。共获得10个基因:chaf1a、repin1、serpine1、htra3、pwp2、gpr173、ncln、nt5e、myl4、ywhabp2。
[0072]
整合基因表达数据后,deepcox-sc的c-index从0.660
±
0.057提高到0.744
±
0.070(平均提高13%)。deepcox-sc多模态融合模型(包括组织病理学图像、年龄和基因表达)的表现显著优于deepcox-sc模型(wilcoxon符号秩检验的p值为0.053)。多模态融合基准模型(临床分级、分期、年龄、基因表达)的c-index为0.754
±
0.065,略高于deepcox-sc多模态融合模型(0.744
±
0.070)。deepcox-sc多模态融合模型的1年和2年auc(0.800
±
0.091,0.833
±
0.055)均大于多模态融合基准模型(0.781
±
0.096,0.820
±
0.084)。deepcox-sc多模态融合模型也优于之前使用ct图像预测胃癌生存率的研究(c-index0.724)。综上所述,deepcox-sc多模态融合模型能够准确预测胃癌患者的生存。
[0073]
在多模态融合模型中使用的10个基因中有6个基因与生存相关。组蛋白伴侣chaf1a在许多癌症中促进细胞侵袭并抑制细胞凋亡。复制启动因子repin1与生存结局密切相关。serpine1在胃癌中是一种促癌因子。htra3促进肿瘤转移。pwp2为胃癌预后相关基因。nt5e和多种癌症患者的生存相关。其他三个基因(ncln、myl4和gpr173)在肿瘤中表达异常。ywhabp2是一个酪氨酸羟化酶假基因,酪氨酸羟化酶对动物的发育和生存至关重要。
[0074]
5)亚群分析
[0075]
ii期、iii期、g2期、g3期患者分别为113、163、125、216例。以113例ii期患者为例,尽管同属相同的临床分期,但这些患者的生存风险却不同。将这些患者分为有显著生存差异的亚组是有意义的。根据deepcox-sc风险评分中位数,将113例ii期患者分为高风险和低风险两个亚组,两组之间具有统计学显著的生存差异(图4中a部分)。g2、g3、ii期和iii期两组间生存差异的log-rank p值分别为5.66e-4、1.97e-4、1.29e-3和9.22e-3(图4中a部分、图4中b部分、图4中c部分、图4中d部分)。
[0076]
以上所述,仅为本发明的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的工作人员在本发明揭露的技术范围内,可轻易想到各种等效的修改或替换,这些修改或替换都应涵盖在本发明的保护范围之内。因此,本发明的保护范围应以权利要求的保护范围为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1