一种同源重组修复基因变异的解读方法与流程
1.本技术属于医学信息技术领域,更具体地说,它涉及一种同源重组修复基因变异的解读方法。
背景技术:
2.dna双链断裂(double strand breaks)是一种dna损伤类型,严重时还会导致染色体的断裂和重排等,由于没有互补链进行修复,所以dna序列难以恢复,造成遗传信息丢失,这种dna双链断裂需要同源重组修复。若同源重组修复能力缺失即发生hrd,则会导致基因组丧失稳定性,在基因组不稳定的情况下容易积累dna损伤,以此恶性循环,导致癌症发生。hrd对铂类或者parp抑制剂的使用具有很重要的指导意义。parp是一种重要的dna修复酶,具有应答dna损伤、维持基因组稳定等作用。parp抑制剂会影响parp功能从而阻止dna修复功能,对于存在hrd的癌细胞,parp抑制剂会进一步增加基因组的不稳定性,使癌细胞死亡。
3.hrd一般由同源重组修复通路中基因变异或者表观变异引起,同源重组修复通路中包含brca1/2、rad52/rad22、palb2、rad51家族、brip1/bach1、atm和chek2等基因。研究表明存在brca1突变的女性,会存在50
‑
85%和15
‑
45%的概率罹患乳腺癌和卵巢癌,同时对于铂类药物,parp抑制剂存在敏感。hrr基因突变在临床上均具有重要的诊断和预后治疗价值。目前针对肿瘤同源重组修复状态检测的方式多种多样,包括brca1/2双基因检测,hrr基因panel检测,肿瘤hrd状态检测等,但由于hrr家族基因发挥抑癌基因功能,无明确热点变异区域,因此对于检测到的hrr基因突变进行准确解读注释是临床应用的重要前提。
4.目前国际临床应用中公认的变异解读分级体系为acmg/amp等组织2015年发表的的遗传性变异解读共识以及2017年发表的肿瘤体细胞变异解读共识,其中acmg/amp等组织2015年发表的遗传性变异分级为五级分类(致病
‑
pathogenic;疑似致病
‑
likely pathogenic;意义不明
‑
vus;疑似良性
‑
likely benign;良性
‑
benign);acmg/amp等组织2017年发表的肿瘤体细胞变异分级为四级分类(i级,ii级,iii级,iv级)。由于两种体系之间使用的分类标准存在差异,国际上多个公共数据库收集的变异位点包括clinvar、brcaexchange、clvic、oncokb等使用不同的变异分类标准,解读规则不统一导致hrr基因变异使用不同体系的解读注释结果存在差异,潜在增加了临床应用的困难进而会对临床诊疗判断产生影响。
技术实现要素:
5.为了统一评级标准,本技术提供一种同源重组修复(hrr)基因变异的解读方法,该解读方法是基于生物信息学注释流程以及标准化受检者临床数据得到的标准化数据而进行快速位点致病性的判读。该解读方法同时适用于肿瘤及其配对(或无配对),充分结合最新分析技术,包括hrd,mutational signature分析在内的基因测序结果证据与样本自身临床遗传学证据对同源重组修复基因变异情况进行解读,该解读方法更加匹配临床需求。
6.本技术是通过以下方案实现的:
本技术提供一种同源重组修复基因变异的解读方法,所述方法包括生物信息学解读方法和临床数据的解读方法。
7.现有技术中,较多的是对受检者生物信息学的解读,而没有将受检者的生物信息学与受检者的临床相关数据相结合,因此,本技术提供同源重组修复基因变异的解读方法,不仅包括受检者生物信息学的解读,还包括受检者的临床数据的解读,将生物信息学数据和临床数据相结合,从而使对受试者的基因组变异解读更加准确,更准确地辅助诊疗。
8.在本技术的一个具体实施方式中,所述生物信息学解读方法包括:创建同源重组修复基因变异信息的注释文件;确定同源重组修复基因致病等级分类、变异分类及致病概率;对变异证据进行分级,优化证据模块内容,建立变异解读规则;获得判读结果。
9.本技术对现有的生物信息学解读方法进行了优化,充分结合最新分析技术,包括hrd,mutational signature分析在内的基因测序结果证据,为准确分析受检者的生物信息学提供了更多的证据。
10.在本技术的一个具体实施方式中,所述注释文件的信息包括:基因名称、基因组位置、变异类型、染色体编号、hgvs格式碱基变化、转录本信息、hgvs格式氨基酸变化、人群频率、软件预测和参考数据库的变异注释信息。
11.在本技术的一个具体实施方式中,确定同源重组修复基因致病等级分类、变异分类及致病概率包括:同源重组修复基因变异致病等级分类、变异分类及致病概率由强到弱分为5类包括:致病的,5类,致病概率为>99%;可能致病的,4类,致病概率为90%~99%;意义不明确的,3类,致病概率为10%~90%;可能良性,2类,致病概率1%~10%;良性,1类,致病概率为<1%。
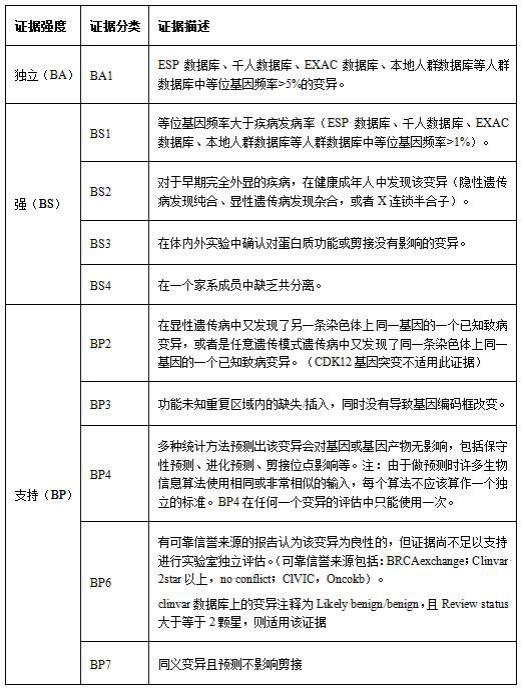
12.在本技术的一个具体实施方式中,所述变异证据包括良性证据和致病性证据,所述良性证据包括独立证据ba、强证据bs、支持证据bp;所述致病性证据包括非常强证据pvs、强证据ps、中等证据pm和支持证据pp。
13.在本技术的一个具体实施方式中,所述优化良性证据,包括删除证据bp1:已知一个疾病的致病原因是由于某基因的截断变异,在此基因中所发现的错义变异。
14.在本技术的一个具体实施方式中,所述优化良性证据,包括删除证据bp5:在已经有另一分子致病基因的病例中发现的变异。
15.在本技术的一个具体实施方式中,所述优化良性证据,包括修正证据bp2:当突变基因为cdk12时,不适用此证据。
16.在本技术的一个具体实施方式中,所述优化致病性证据,包括方式一、修正ps2证据为:如果文献查询到位点曾经以经过家系验证的新发突变出现,也可以适用该证据。
17.在本技术的一个具体实施方式中,所述优化致病性证据,包括方式二、修正pm1证据为:位于突变热点区域或者非常明确的功能结构域,且该区域无任何良性突变;体细胞数据库或算法显示相同位置变异为热点变异,q
‑
value < 0.1或者复现>3次。
18.在本技术的一个具体实施方式中,所述优化致病性证据,包括方式三、修正pm2证据为:esp 数据库、千人数据库、exac 数据库、本地人群数据库中正常对照人群中,针对brca1和brca2基因未发现变异或频率<0.0003;针对非brca1和brca2的其他基因未发现变异或频率<0.0001。
19.在本技术的一个具体实施方式中,所述优化致病性证据,包括方式四、删除证据
pm3:隐性遗传模式的致病基因下,检测到于另一个已知致病/可能致病变异互为反式存在。
20.在本技术的一个具体实施方式中,所述优化致病性证据,包括方式五、修正证据pm6为:如果文献查询到位点曾经以未经家系验证的新发突变出现,也可以适用该证据。
21.在本技术的一个具体实施方式中,所述优化致病性证据,包括方式六、删除证据pp2:对某个基因来说,如果这个基因的错义变异是造成某种疾病的原因,并且这个基因中良性变异所占的比例很小,在这样的基因中所发现的新的错义变异。
22.在本技术的一个具体实施方式中,所述优化致病性证据,包括方式七、修正pp4证据注释如下:(1)受检者临床数据≥20分;(2)brca1和/或brca2基因变异时,受检者hrd为阳性;(3)brca1和/或brca2基因变异时,且mutational signature符合突变特征3;(4)对于乳腺癌、卵巢癌患者,表型需满足nccn 指南中的至少 2 条;受检者至少满足上述四种情形之一则适用pp4证据。
23.在本技术一个具体实施方式中,所述优化致病性证据,包括方式八、增加pp5证据注释:clinvar数据库上的变异注释为pathogenic/likely pathogenic,且review status大于等于2颗星,则适用pp5证据。
24.在本技术一个具体实施方式中,所述优化致病性证据,包括增加bp6证据注释:clinvar数据库上的变异注释为likely benign/benign,且review status大于等于2颗星,则适用bp6证据。
25.在本技术的一个具体实施方式中,所述优化致病性证据,包括确定证据pp5和bp6中,可靠信誉来源的支持性数据库包括:brcaexchange、clinvar 2star、no conflict、clvic和oncokb,其中2star为位点在clinvar数据库中的“review status”状态。
26.在本技术一个具体实施方式中,所述受检者临床数据通过以下方法获得:当所述年龄≤35时,赋予第一分值5分;当35<年龄≤50时,赋予第一分值4分;当50<年龄≤70时,赋予第一分值3分;当年龄>70时,赋予第一分值1分;当肿瘤类型为卵巢癌时:所述分型为浆液性卵巢癌,则赋予第二分值4分;所属分型为滤泡性卵巢癌,则赋予第二分值1分;所述分型为子宫内膜样癌,则赋予第二分值1分;当卵巢癌为其他分型时,则赋予第二分值0分;当肿瘤类型不是卵巢癌时,则赋予第二分值0分;当肿瘤类型为乳腺癌时,所述分型为三阴性,则赋予第三分值4分;所述分型为激素阳性,则赋予第三分值1分;所述分型为her2阳性,则赋予第三分值1分;当肿瘤类型不是乳腺癌时,则赋予第三分值0分;当肿瘤为前列腺癌时:gs评分>7分时,则赋予第四分值4分;gs评分<7分时,则赋予第四分值1分;当肿瘤类型不是前列腺癌时,则赋予第四分值0分;当肿瘤类型为其他类型时,则赋予第五分值1分,当肿瘤类型为卵巢癌、乳腺癌或前列腺癌时,则赋予第五分值0分;当肿瘤是多原发时,则赋予第六分值5分;当肿瘤不是多原发时,则赋予第六分值1分;当肿瘤对铂类药物敏感时,则赋予第七分值4分;当肿瘤耐铂类药物时,则赋予第七分值1分;当肿瘤对铂类药物是否敏感为未知时,则赋予第七分值0分;当肿瘤对parp抑制剂敏感时,则赋予第八分值5分;当肿瘤对parp抑制剂不敏感时,则赋予第八分值1分;当肿瘤对parp抑制剂是否敏感为未知时,则赋予第八分值0分;当一级亲属患癌人数为n时,则赋予第九分值2n分,其中,n为0,1,2,3...;当二级亲属患癌人数为0时,则赋予第十分值0分,当二级亲属患癌人数为m时,则赋予第十分值m
‑
1分,其中m为1,2,3...;当三级亲属患癌人数为q时,当q为0时,则赋予第十一分值0分;当q为基数时,则赋予第十一分值(q
‑
1)/2分,当q为偶数时,则赋予第十一分值q/2分;所
述受检者临床数据的分数是第一分值、第二分值、第三分值、第四分值、第五分值、第六分值、第七分值、第八分值、第九分值、第十分值和第十一分值的总和。
27.在本技术的一个具体实施方式中,所述同源重组修复基因选自brca1,brca2,atm,cdk12,bard1,brip1,chek1,chek2,fanca,fancl,rad51b,rad51c,rad51d和rad54l中的一种或多种。
28.本技术提供的参比电极至少具有以下有益效果之一:本技术提供的同源重组修复基因变异的解读方法,其通过将受检者的生物信息学解读方法和临床数据相结合,从而使对受试者的基因组变异解读更加准确,更贴近临床需求。
附图说明
29.图1为本技术实施例中提供的与f1cdx检测结果的对比图。
具体实施方式
30.除非另有定义,本技术中使用的所有技术和科学术语具有与本技术所述技术领域的普通技术人员通常理解的相同含义。
31.下面将结合本技术实施例,对本技术的技术方案进行清楚、完整地描述,显然,所描述的实施例仅是本技术一部分实施例,而不是全部的实施例。基于本技术中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本技术保护的范围。实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。
32.术语解释:hrr:homologous recombination repair,同源重组修复hrd:homologous recombination deficiency,同源重组修复缺陷mutational signature:突变信号指纹parp:poly adp
‑
ribose polymerase,多聚adp核糖聚合酶hgvs:人类基因组变异协会(hgvs:human genome variation society),其建立的命名规则目的在于对基因信息、转录信息、蛋白信息的变异情况给予标准化的命名方式,形成国际学术界通用的规范。
33.acmg/amp指南:美国遗传协会(acmg,american college of medical genetics)和美国分子病理协会(amp,association for molecular pathology),共同颁布和制定了人类基因变异致病性解读指南。
34.clinvar:是ncbi主办的与疾病相关的人类基因组变异数据库。它是一个开放的数据库,整合了dbsnp、dbvar、pubmed、omim等多个数据库在遗传变异和临床表型方面的数据信息,提交结果由专家进行审核并形成标准。
35.brcaexchange:是专门针对brca1/2基因变异收录及解读的开放、开源的数据库,按照enigma标准进行解读,并将结果提交联盟专家进行审核。
36.oncokb:oncokb是由memorial sloan kettering cancer center(msk)维护的全面的精准肿瘤学知识库,包含来自fda、nccn、asco、clinicaltrials和科学文献的专业指导
方针和建议,治疗策略和肿瘤专家或肿瘤协会共识。数据库将变异位点进行了1~4级的临床证据和生物学证据分级。
37.clvic:clinical interpretation of variants in cancer,civic是一个开放访问,开源,社区驱动的网络资源,用于临床解释癌症变异数据平台。
38.varsome:the human genomic variant search engine,varsome是一个用于人类遗传变异的搜索引擎、聚合器和影响分析工具。在多项变异注释分类中,varsome作为重要的在线变异分类平台被提及。
39.f1cdx:foundationone
®
cdx,是首款突破性的基于ngs的体外诊断产品,能对任何实体肿瘤进行诊断,在体外诊断领域具有里程碑式的意义,是首个获得 fda 批准的针对泛肿瘤的全面的伴随诊断检测。foundationone
®
cdx在2019年获批作为parp抑制剂奥拉帕利在前列腺癌中的伴随诊断,对14个hrr基因进行有害突变检测和筛选。
40.intervar:intervar是一种生物信息学软件工具,用于基于acmg/amp 2015 指南对遗传变异的临床分类解释。intervar的输入源支持由annovar生成的带注释的文件,intervar的输出可将变异分类为“良性”、“可能良性”、“不确定性”、“可能致病”和“致病”。
41.实施例1本实施例提供的同源重组修复基因变异的解读方法,具体包括如下步骤:1. 受检者数据收集整理(1)获取受检者生物信息学数据按照hgvs格式要求输出生物信息学数据的注释文件,包括变异所属基因信息,基因名称、转录本信息、染色体编号、基因组位置、hgvs格式碱基变化、hgvs格式氨基酸变化、变异类型、人群频率、软件预测、参考数据库的变异注释信息,家系/亲属测序变异结果,以及样本hrd检测结果和mutational signature信息。
42.(2)通过以下方法获取受检者临床数据当所述年龄≤35时,赋予第一分值5分;当35<年龄≤50时,赋予第一分值4分;当50<年龄≤70时,赋予第一分值3分;当年龄>70时,赋予第一分值1分;当肿瘤类型为卵巢癌时:所述分型为浆液性卵巢癌,则赋予第二分值4分;所属分型为滤泡性卵巢癌,则赋予第二分值1分;所述分型为子宫内膜样癌,则赋予第二分值1分;当卵巢癌为其他分型时,则赋予第二分值0分;当肿瘤类型不是卵巢癌时,则赋予第二分值0分;当肿瘤类型为乳腺癌时,所述分型为三阴性,则赋予第三分值4分;所述分型为激素阳性,则赋予第三分值1分;所述分型为her2阳性,则赋予第三分值1分;当肿瘤类型不是乳腺癌时,则赋予第三分值0分;当肿瘤为前列腺癌时:gs评分>7分时,则赋予第四分值4分;gs评分<7分时,则赋予第四分值1分;当肿瘤类型不是前列腺癌时,则赋予第四分值0分;当肿瘤类型为其他类型时,则赋予第五分值1分,当肿瘤类型为卵巢癌、乳腺癌或前列腺癌时,则赋予第五分值0分;当肿瘤是多原发时,则赋予第六分值5分;当肿瘤不是多原发时,则赋予第六分值1分;当肿瘤对铂类药物敏感时,则赋予第七分值4分;当肿瘤耐铂类药物时,则赋予第
七分值1分;当肿瘤对铂类药物是否敏感为未知时,则赋予第七分值0分;当肿瘤对parp抑制剂敏感时,则赋予第八分值5分;当肿瘤对parp抑制剂不敏感时,则赋予第八分值1分;当肿瘤对parp抑制剂是否敏感为未知时,则赋予第八分值0分;当一级亲属患癌人数为n时,则赋予第九分值2n分,其中,n为0,1,2,3...;当二级亲属患癌人数为0时,则赋予第十分值0分,当二级亲属患癌人数为m时,则赋予第十分值m
‑
1分,其中m为1,2,3...;当三级亲属患癌人数为q时,当q为0时,则赋予第十一分值0分;当q为基数时,则赋予第十一分值(q
‑
1)/2分,当q为偶数时,则赋予第十一分值q/2分;所述受检者临床数据的分数是第一分值、第二分值、第三分值、第四分值、第五分值、第六分值、第七分值、第八分值、第九分值、第十分值和第十一分值的总和。
43.为评定变异结果,本技术变异证据包括良性证据和致病性证据。良性证据按照由强到弱氛围3个等级:独立证据ba、强证据bs和支持证据bp;致病性证据按照由强到弱分为4个等级:非常强证据pvs、强证据ps、中等证据pm和支持证据pp,证据分类描述见表1和表2。
44.表1 良性证据等级分类描述
表2 致病性证据等级分类描述
本技术中同源重组修复基因致病等级分类、变异分类及致病概率包括:同源重组修复基因变异致病等级分类、变异分类及致病概率由强到弱分为5类包括:致病的(pathogenic,简写p),5类,致病概率为>99%;可能致病的(likely pathogenic,简写lp),4
类,致病概率为90%~99%;意义不明确的(uncertain significance,简写us),3类,致病概率为10%~90%;可能良性(likely benign,简写lb),2类,致病概率1%~10%;良性(benign,简写b),1类,致病概率为<1%。变异判读按照表3的判读标准,进行最终的打分判定。
45.表3 变异分类判读规则为验证本技术构建的解读方法对变异解读的准确性,下面将结合更具体的实施案例来进一步说明本技术的有益效果。
46.实施例2
为更好地进行案例说明,本实施例利用实施例1中的解读方法解读1例中国人的检测结果,具体如下。
47.患者信息:患者李某,68岁,确诊为浆液性卵巢癌,50岁诊断为左侧浸润性乳腺癌,无家族史,患者对一线铂类药物存在敏感。
48.患者的hrr基因检测突变结果、hrd检测结果、主要mutation signature结果以及患者临床数据结果如下:患者的hrd
‑
score检测状态为high。
49.患者的主要mutation signature为特征3。
50.患者的临床数据为18。
51.患者hrr基因检测结果显示存在三个变异基因,分别为:1)brca2 c.6322c>t p.r2108c2)brca1 c.2717dela p.k906fs3)atm c.5793t>c p.a1931a对上述三个变异基因分别进行变异解读过程,获得判读结果,以下以1)brca2 c.6322c>t p.r2108c为例进行说明。
52.1)brca2 c.6322c>t p.r2108c该变异在db
‑
patient中出现342/33617次,在db
‑
normal中出现4319/238891次,计算or值为or=0.56<1且95%ci不包含1(bs2)。
53.该变异为胚系变异,在clinvar上收录为有害变异(bp2)。
54.该变异收录为benign且reveiw status(clndsdb)为rev iewed_by_expert_panel/no conflict,即等于2颗星(bp6)。
55.判读结果:依据本技术的变异解读方法,判定该变异为2
‑
可能良性的突变(likely benign:bs2+ bp2+ bp6+pp4)。
56.按照同样的方法判断:2)brca1 c.2717dela p.k906fs该变异符合pm2、ps4、pvs1和pp5证据,依据本技术的变异解读方法,判定该变异为5
‑
致病的突变。
57.3)atm c.5793t>c p.a1931a该变异符合bs1、bp7、bp2和bp6证据,依据本技术的变异解读方法,判定该变异为2
‑
可能良性的突变。
58.最终分析该患者存在brca1 p.k906fs有害突变。
59.实施例3按照实施例1中的解读方法,本实施例中对来源于中国人群肿瘤样本的191个hrr基因变异进行解读,并与f1cdx解读结果进行比较,其实验结果如图1,表4、表5
‑
1、表5
‑
2和表5
‑
3所示。
60.图1中,“182”表示fmi分级为182个hrr基因变异为致病性;“9”表示fmi分级为9个hrr基因变异为意义不明,而通过本技术提供的同源重组修复基因变异的解读方法判读的结果为191个hrr基因变异均为可能致病。
61.表4与fmi分级的结果一致的部分hrr基因变异
表5
‑
1至表5
‑
3示出了利用实施例1中的解读方法解读结果与fmi分级的结果不一致的9个hrr基因变异。
63.从图1,表4中可以看出,利用本技术提供的同源重组修复基因变异的解读方法所判读的结果包括未分类的样本、分类为致病的样本与f1cdx检测结果的完全一致。
64.从图1,表5
‑
1至5
‑
3中可以看出,对于f1cdx检测结果为意义不明的9个位点,根据本技术提供的解读方法判读结果为“可能致病”。而该9个位点通过clinvar突变数据有害性均确认为“可能致病或致病性”,其与本技术实施例1中提供的解读方法判读的结果一致,可见,本技术提供的解读方法更加准确,更贴近于临床需求。
65.本具体实施例仅仅是对本技术的解释,其并不是对本技术的限制,本领域技术人员在阅读完本说明书后可以根据需要对本实施例做出没有创造性贡献的修改,但只要在本技术的权利要求范围内都受到专利法的保护。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1