基于全基因组测序完成待测样本的核型分析的方法、系统和计算机可读介质与流程
1.本发明涉及生物信息领域,具体地,本发明涉及确定待测样本预定大片段拷贝数(cna)变异的方法、系统和计算机可读介质。
背景技术:
2.急性髓系白血病(aml)也称急性非淋巴细胞白血病(急非淋),是骨髓祖细胞不受控的增殖引起的血液系统恶性肿瘤,其临床发病率较高,复发率高,治愈率低。aml是由获得性髓系定向祖细胞变异引起,以造血干细胞分化受阻、细胞克隆性增值异常以及凋亡抵抗为主要特征。近年来其发病率呈逐年上升趋势,严重威胁着人类的健康,我国已将其列入十大恶性肿瘤之一、由于aml起病急,发病重,病情进展迅速,若延误治疗病人常在半年内死亡,因此对aml的早期诊断和预后监测是提高aml患者生存率的最佳途径。
3.骨髓增生异常综合症(myelodysplastic syndrome,mds)是一组起源于造血髓系定向干细胞或多能干细胞的异质性克隆性疾患,主要特征是无效造血和高危演变为急性髓系白血病,临床表现为贫血,可伴有感染或出血,部分病人可无症状。血象可呈全血细胞减少,或任何一系及二系血细胞减少。中国骨髓增生异常综合征发病率趋高已达十万分之三,多累及中老年人,50岁以上的病例占50%~70%,且患者发病年龄比西方国家年轻十岁左右。
4.现有的与染色体拷贝数变异(copy number variation)相关的疾病(如aml和mds)的检测和预后方法,存在检测不准确,灵敏度不高,且耗时较长的问题,不能满足科研和临床检测需求。因此亟需开发快速检测、检测更准确且具有较高灵敏度的cnv检测方法。
技术实现要素:
5.本发明旨在至少在一定程度上解决相关技术中的技术问题之一。为此,本发明的一个目的在于提供一种确定待测样本预定染色体变异的方法,采用二代测序和动态cutoff相结合,能够快速、准确地检测cnv大片段,同时根据相关疾病的临床指南(如急性髓系白血病、骨髓增生异常综合症的nccn临床指南),根据检测出染色体拷贝数变异的位置和突变类型(比如5q,7q的缺失),从而指导临床治疗或预后(具有5q,7q缺失,预后不好)。本发明提供的检测方法灵敏度高,分辨率低,检测结果更准确,通过与预后相关的基因的位置和变异在染色体上的位置注释,能够用于与染色体拷贝数变异相关的疾病的预后。
6.为此本发明第一方面提供一种确定待测样本预定染色体变异的方法。根据本发明的实施方案,所述方法包括:
7.(1)将所述预定染色体的参考序列进行划分,以便获得窗口序列bins;
8.(2)将来自所述待测样本的测序数据与所述参考序列进行比对,所述测序数据由多个测序读段reads构成,以便确定每个测序读段的位置;
9.(3)基于步骤(2)的比对结果和所述测序读段的位置,分别统计所述多个窗口序列
中的每一个窗口中匹配上的读段数目;
10.(4)基于步骤(3)的统计结果,对每个窗口的所述匹配测序读段数目进行过滤和校正,以便获得待测样本的过滤和校正后的测序读段;
11.(5)对步骤(4)所获得的待测样本的过滤和校正后的每个窗口中的测序读段数目进行取对数处理,并减去多个正常样本在同一bins下的测序读段数目的平均值的对数值,以便获得所述窗口的测序读段数目的对数值log r ratio;
12.(6)基于所述待测样本的每个bins的log r ratio,将相邻的且log r ratio值接近或相等的bins连接起来,以便获得多个基因组片段,基于每一个所述基因组片段,获取所述待测样本中每个基因组片段的平均log r ratio值和对应的bins数目;
13.(7)利用所述待测样本中基因组片段的平均log r ratio值和对应的bins数目,获取所述待测样本中所述基因组片段的对应cutoff值;
14.(8)针对所述基因组片段中的每一个,获取所述平均log r ratio值的绝对值与所述cutoff值的绝对值的差值,以便获取待测样本中每一个基因组片段的substract值,其中,所述substract值大于0,是所述待测样本预定染色体具有cnv变异的指示。
15.本发明提供的确定待测样本预定染色体变异的方法,采用二代测序和动态cutoff相结合,能够快速、准确地检测cnv大片段,同时根据结果基于染色体核型分析,能够检测与染色体拷贝数变异相关的疾病。本发明提供的检测方法灵敏度高,分辨率低,检测结果更准确,通过与预后相关的基因的位置和变异在染色体上的位置注释,能够用于与染色体拷贝数变异相关的疾病的预后。本发明提供的确定待测样本预定染色体变异的方法中,通过减去多个正常样本在同一bins下的测序读段数目的平均值的对数值来去除系统误差和背景噪音,从而更准确地检测预定染色体的cnv变异。
16.发明人将待测样本的过滤和校正后的每个窗口中的测序读段数目进行取对数处理,并减去多个正常样本在同一bins下的测序读段数目的平均值的对数值,获得所述窗口的测序读段数目的对数值log r ratio,该方法能够去除系统误差和背景噪音,保证结果的准确度。
17.根据本发明的实施方案,步骤(1)进一步包括:
18.将所述预定染色体的参考序列划分为多个相同长度的窗口序列,所述窗口的长度根据测序深度确定。
19.根据本发明的实施方案,在步骤(4)中,对每个窗口的所述匹配测序读段数目进行过滤包括保留1%-99%分位数的bins。
20.在本发明中,对测序读段进行过滤时,根据测序深度来确定保留多少百分比分位数的的bins。
21.根据本发明的实施方案,所述过滤和校正包括:过滤低质量测序读段和校正测序深度、gc含量、比对率、n比例校正。
22.根据本发明的实施方案,在步骤(4)中,对每个窗口的所述匹配测序读段数目进行校正进一步包括构建正常对照参考集,所述正常对照参考集通过以下方法获得:
23.采用(1)-(4)相同步骤,获得多个正常样本过滤和校正后的测序读段,以便获得多个正常样本的测序读段数目的平均值的对数值。
24.根据本发明的实施方案,所述正常样本的数目不少于20个,且各个正常样本来自
于不同的个体。
25.根据本发明的实施方案,所述正常样本为没有cnv变异的样本。
26.根据本发明的实施方案,所述cutoff值为动态cutoff值。
27.根据本发明的实施方案,在步骤(7)中,利用所述待测样本中基因组片段的平均log r ratio值和对应的bins数目,基于最小二乘法进行曲线拟合,以便获取所述待测样本中所述基因组片段的平均log r ratio值的cutoff值。根据本发明的实施方案,选取每个基因组片段中深度》4x的已知snp位点,计算纯合与杂合的数目和比例;并与所述正常对照参考集对比,计算z-score,
28.其中,杂合位点数目的zscore《-3和杂合/纯合的占比zscore《-3,则该样本存在获得性杂合性缺失(loh)。
29.基于检测到的存在变异的片段的位置以及人的所有基因在染色体上的位置,进行注释。注释出位于变异片段的所有基因,并挑选出跟疾病治疗和预后相关的突变。
30.根据本发明的实施方案,所述待测样本来源于疑似癌症患者。
31.根据本发明的实施方案,所述待测样本为血液、脑脊液、体液、尿液、唾液或皮肤。
32.根据本发明的实施方案,测序数据是通过对血浆游离的dna进行全基因组建库后,使用二代测序仪测序获得的,平均测序深度为小于0.1x、0.3x、0.5x、1x、2x、3x、4x或5x。
33.本发明第二方面提供一种确定待测样本预定染色体变异的系统。根据本发明的实施方案,所述系统包括:
34.窗口划分装置,所述窗口划分装置被配置为适于将所述预定染色体的参考序列进行划分,以便获得窗口序列bins;
35.比对装置,所述比对装置与所述窗口划分装置相连,所述比对装置被配置为适于将将来自所述待测样本的测序数据与所述参考序列进行比对,所述测序数据由多个测序读段reads构成,以便确定每个测序读段的位置;统计装置,所述统计装置与所述比对装置相连,所述统计装置被配置为适于基于所述比对装置的比对结果和所述测序读段的位置,分别统计所述多个窗口序列中的每一个窗口中匹配上的读段数目;
36.校正装置,所述校正装置与所述统计装置相连,所述校正装置被配置为适于对每个窗口的所述匹配测序读段数目进行过滤和校正,以便获得待测样本的过滤和校正后的测序读段;
37.取对数装置,所述取对数装置与所述校正装置相连,所述取对数装置被配置为适于对所述校正装置所获得的待测样本的过滤和校正后的每个窗口中的测序读段数目进行取对数处理,并减去多个正常样本在同一bins下的测序读段数目的平均值的对数值,以便获得所述窗口的测序读段数目的对数值log r ratio;
38.片段化获取装置,所述片段化获取装置与所述取对数装置相连,所述片段化获取装置被配置为适于基于所述待测样本的每个bins的log r ratio,将相邻的且log r ratio值接近或相等的bins连接起来,以便获得多个基因组片段,基于每一个所述基因组片段,获取所述待测样本中每个基因组片段的平均log r ratio值和对应的bins数目;
39.cutoff值获取装置,所述cutoff值获取装置与所述片段化获取装置相连,所述cutoff值获取装置被配置为适于利用所述待测样本中基因组片段的平均log r ratio值和对应的bins数目,获取所述待测样本中所述基因组片段的对应cutoff值;
40.确定染色体变异装置,所述确定染色体变异装置与所述cutoff值获取装置相连,所述确定染色体变异装置被配置为适于针对所述基因组片段中的每一个,获取所述平均log r ratio值的绝对值与所述cutoff值的绝对值的差值,以便获取待测样本中每一个基因组片段的substract值,其中,所述substract值大于0,是所述待测样本预定染色体具有cnv变异的指示。根据本发明的实施方案,所述窗口划分装置被配置为适于将所述预定染色体的参考序列划分为多个相同长度的窗口序列。
41.根据本发明的实施方案,所述校正装置适于对每个窗口的所述匹配测序读段数目进行过滤包括保留1%-99%分位数的bins。
42.根据本发明的实施方案,所述过滤和校正包括:过滤低质量测序读段和校正测序深度、gc含量、比对率、n比例校正。
43.根据本发明的实施方案,所述校正装置适于对每个窗口的所述匹配测序读段数目进行校正进一步包括构建正常对照参考集,所述正常对照参考集通过以下方法获得:
44.采用窗口划分装置、比对装置、统计装置获得多个正常样本过滤和校正后的测序读段,以便获得多个正常样本的测序读段数目的平均值的对数值。
45.根据本发明的实施方案,所述正常样本的数目不少于20个,且各个正常样本来自于不同的个体。
46.根据本发明的实施方案,所述正常样本为没有cnv变异的样本。
47.根据本发明的实施方案,所述cutoff值为动态cutoff值。
48.根据本发明的实施方案,所述cutoff值获取装置适于利用所述待测样本中基因组片段的平均log r ratio值和对应的bins数目,基于最小二乘法进行曲线拟合,以便获取所述待测样本中所述基因组片段的平均log r ratio值的cutoff值。
49.根据本发明的实施方案,选取每个基因组片段中深度》4x的已知snp位点,计算纯合与杂合的数目和比例;并与所述正常对照参考集对比,计算z-score。
50.其中,杂合位点数目的zscore《-3和杂合/纯合的占比zscore《-3,则该样本存在获得性杂合性缺失(loh)。
51.根据本发明的实施方案,所述待测样本来源于疑似癌症患者。
52.根据本发明的实施方案,所述待测样本为血液、脑脊液、体液、尿液、唾液或皮肤。
53.根据本发明的实施方案,测序数据是通过对血浆游离的dna进行全基因组建库后,使用二代测序仪测序获得的,平均测序深度为小于0.1x、0.3x、0.5x、1x、2x、3x、4x或5x。
54.本发明第三方面提供第二方面所述的确定待测样本预定染色体变异的系统在疾病预后中的用途。根据本发明的实施方案,所述疾病为与染色体拷贝数变异相关的疾病。
55.根据本发明的实施方案,所述与染色体拷贝数变异相关的疾病包括血液肿瘤疾病。
56.根据本发明的实施方案,所述血液肿瘤疾病选自急性髓系白血病、骨髓增生异常综合症。
57.本发明第四方面提供一种与染色体拷贝数变异相关的疾病的预后方法。根据本发明的实施方案,所述方法包括:
58.基于第一方面所述的方法确定待测样本中预定染色体是否具有cnv变异,根据基因的位置和变异在染色体上的位置注释,以便对与染色体拷贝数变异相关的疾病进行预
后,其中,所述基因为与预后相关的基因。
59.本发明第五方面提供一种计算机可读存储介质,其上存储有计算机程序。根据本发明的实施方案,该程序被处理器执行时实现第一方面所述方法的步骤。
60.本发明第六方面提供一种电子设备。根据本发明的实施方案,所述电子设备包括:
61.第五方面所述的计算机可读存储介质;以及
62.一个或者多个处理器,用于执行所述计算机可读存储介质中的程序。
63.本发明的附加方面和优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。
附图说明
64.本发明的上述和/或附加的方面和优点从结合下面附图对实施例的描述中将变得明显和容易理解,其中:
65.图1显示了根据本发明实施例的确定待测样本预定染色体变异的系统的结构示意图;、
66.图2显示了实施例2中窗口大小分别与变异系数的对数似然值和赤池信息量准则之间的关系;
67.图3显示了实施例2中待测样本测序读段数据的gc分布图;
68.图4显示了不同染色体的在正常人群例的分布,表示其波动范围的大小;
69.图5显示了两个样本中不同片段的bins数目,以及对应的cutoff值;
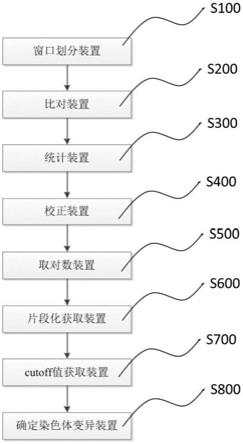
70.图6显示了实施例4中mds样本临床(fish)检测结果和cnv分析结果;
71.图7显示了实施例5中aml样本cnv分析结果;以及该区域包含的部分基因;
72.图8显示了基于本发明的方法相比传统的方法,多检出cnv变异占比,根据aml/mds的相关指南,重新定义这些样本预后;
73.图9显示了在癌症基因pik3ca区域内,检测出来的loh阳性和阴性示例。
具体实施方式
74.下面详细描述本发明的实施例。下面描述的实施例是示例性的,仅用于解释本发明,而不能理解为对本发明的限制。实施例中未注明具体技术或条件的,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的常规产品。
75.为了描述方便,本发明所提出的确定待测样本预定染色体变异的系统的结构示意图可参考图1。根据本发明的实施例,所述系统包括:
76.窗口划分装置s100,所述窗口划分装置s100被配置为适于将所述预定染色体的参考序列进行划分,以便获得窗口序列bins;
77.比对装置s200,所述比对装置s200与所述窗口划分装置s100相连,所述比对装置s200被配置为适于将来自所述待测样本的测序数据与所述参考序列进行比对,所述测序数据由多个测序读段reads构成,以便确定每个测序读段的位置;;
78.统计装置s300,所述统计装置s300与所述比对装置s200相连,所述统计装置s300被配置为适于基于所述比对装置s200的比对结果和所述测序读段的位置,分别统计所述多
个窗口序列中的每一个窗口中匹配上的读段数目;
79.校正装置s400,所述校正装置s400与所述统计装置s300相连,所述校正装置s400被配置为适于对每个窗口的所述匹配测序读段数目进行过滤和校正,以便获得待测样本的过滤和校正后的测序读段;
80.取对数装置s500,所述取对数装置s500与所述校正装置s400相连,所述取对数装置s500被配置为适于对所述校正装置s400所获得的待测样本的过滤和校正后的每个窗口中的测序读段数目进行取对数处理,并减去多个正常样本在同一bins下的测序读段数目的平均值的对数值,以便获得所述窗口的测序读段数目的对数值log r ratio;
81.片段化获取装置s600,所述片段化获取装置s600与所述取对数装置s500相连,所述片段化获取装置s600被配置为适于基于所述待测样本的每个bins的log r ratio,将相邻的且log r ratio值接近或相等的bins连接起来,以便获得多个基因组片段,基于每一个所述基因组片段,获取所述待测样本中每个基因组片段的平均log r ratio值和对应的bins数目;
82.cutoff值获取装置s700,所述cutoff值获取装置s700与所述片段化获取装置s600相连,所述cutoff值获取装置s700被配置为适于利用所述待测样本中基因组片段的平均log r ratio值和对应的bins数目,获取所述待测样本中所述基因组片段的对应cutoff值;
83.确定染色体变异装置s800,所述确定染色体变异装置s800与所述cutoff值获取装置s700相连,所述确定染色体变异装置s800被配置为适于针对所述基因组片段中的每一个,获取所述平均log r ratio值的绝对值与所述cutoff值的绝对值的差值,以便获取待测样本中每一个基因组片段的substract值,其中,所述substract值大于0,是所述待测样本预定染色体具有cnv变异的指示。
84.在本发明中,“log r ratio值接近或相等”是利用现有的模型进行确定的,例如通过隐马尔可夫(hmm)模型来确定log r ratio值是否接近或相等。
85.根据本发明的实施例,所述窗口划分装置s100被配置为适于将所述预定染色体的参考序列划分为多个相同长度的窗口序列。根据本发明的实施例,所述校正装置s400适于对每个窗口的所述匹配测序读段数目进行过滤包括保留1%-99%分位数的bins,所述过滤和校正包括:过滤低质量测序读段和校正测序深度、gc含量、比对率、n比例校正。
86.根据本发明的实施例,所述校正装置适于对每个窗口的所述匹配测序读段数目进行校正进一步包括构建正常对照参考集,所述正常对照参考集通过以下方法获得:
87.采用窗口划分装置、比对装置、统计装置获得多个正常样本过滤和校正后的测序读段,以便获得多个正常样本的测序读段数目的平均值的对数值;
88.根据本发明的实施例,所述正常样本的数目不少于20个,且各个正常样本来自于不同的个体;
89.根据本发明的实施例,所述正常样本为没有cnv变异的样本。
90.根据本发明的实施例,所述cutoff值为动态cutoff值。
91.下面详细描述本发明的实施例。下面描述的实施例是示例性的,仅用于解释本发明,而不能理解为对本发明的限制。
92.实施例中未注明具体技术或条件的,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的常
规产品。
93.实施例1:建库和测序
94.抽血aml或者mds患者的骨髓,对骨髓样本(新鲜的细胞或者组织)进行细胞破碎,基因组dna的提取,提取后按照下面的方法进行打断建库。本实施例适用于所有的人的新鲜组织和细胞样本。
95.1、基因组dna打断
96.1)采用covaris打断仪,将基因组dna打断至所需主带范围:pe50推荐主带约180bp,pe100推荐主带约280bp,pe150推荐主带约420bp。
97.检查covaris水槽中注入的去离子水液面到达或超过运行标记,确保水面没过打断管玻璃部分;参考covaris仪器使用说明进行实验操作;
98.表1
99.组分用量基因组dnaxμl/(总量1ug)1x low te buffer(50-x)μl总体积50μl
100.2)在1.5ml的pcr管中将样品使用1x low te buffer稀释为50μl(1ug)。
101.3)将稀释好的样品小心地加入打断管中,避免过程中出现气泡,将打断管放入打断仪中,参考下述打断程序运行打断仪。
102.打断仪参数设定如下表2所示(可根据仪器型号调整):
103.表2
[0104][0105][0106]
2、片段筛选
[0107]
打断后dna分布范围较宽,通常需要进行片段筛选以控制最终文库片段集中度。
[0108]
磁珠片段筛选提供了500ng gdna打断后(体积为80μl),在末端修复之前进行dna片段双筛步骤,第一步中加入64μl磁珠,第二步中加入16μl磁珠进行片段筛选,最终得到主带280bp的目的片段。
[0109]
片段筛选实验步骤如下:
[0110]
1)提前30min取出dna clean beads置于室温,使用前充分震荡混匀;
[0111]
2)吸取所有打断产物至新的1.5ml ep管中,若体积不足80μl,用te buffer补足;
[0112]
3)吸取64μl dna clean beads至含有80μl打断产物的离心管中,用移液器轻轻吸打至少10次至完全混匀,最后一次应确保将吸头中所有液体及磁珠都打入ep管中;
[0113]
4)室温孵育5min。瞬时离心,将ep管置于磁力架上,静置2-5min至液体澄清,吸取
上清至新的1.5ml ep管中。注意:此步保留上清,丢弃磁珠;
[0114]
5)吸取16μl dna clean beads至140上清管中,用移液器轻轻吸打至少10次至完全混匀;
[0115]
6)室温孵育5min;
[0116]
7)瞬时离心,将ep管置于磁力架,静置2-5min至液体澄清,用移液器小心吸取并丢弃上清;
[0117]
8)保持ep管置于磁力架上,加入200μl新鲜配制的80%乙醇漂洗磁珠及管壁,小心吸取并丢弃上清;
[0118]
9)重复步骤8,尽量吸干管内液体;
[0119]
10)保持ep管固定于磁力架上,打开ep管管盖,室温干燥,直至磁珠表面无反光、无开裂;
[0120]
11)将ep管从磁力架上取下,加入32μl te buffer进行dna洗脱,用移液器轻轻吸打至少10次至完全混匀;
[0121]
12)室温孵育5min,瞬时离心,将ep管置于磁力架上,静置2-5min至液体澄清,将30μl上清液转移到新的1.5ml ep管中;
[0122]
13)样本dna的定量和质控
[0123]
取1μl样本使用qubit4.0 fluorometer(qubit dsdna hs assay kit)进行文库浓度测定,记录文库浓度。
[0124]
取1μl样本使用agilent 2100(agilent dna 1000assay kit)进行片段大小检测,正常打断后样品主峰约在300bp。
[0125]
3.末端修复及加a
[0126]
1)根据样本浓度,取适量样本(50ng)至新的0.2ml pcr管,用te buffer补充至总体积40μl。
[0127]
2)在冰上配制末端修复反应液(见下表3):
[0128]
表3
[0129]
组分用量erat buffer7.1μlerat enzyme mix2.9μl总体积10μl
[0130]
3)用移液器吸取10μl配制好的末端修复反应液加入步骤1)的pcr管中,涡旋震荡3次,每次3s,瞬时离心将反应液收集至管底。
[0131]
4)将上述pcr管置于pcr仪上,运行pcr仪程序,设置pcr仪参数。热盖温度设置85℃;加热模块设置:37℃,30min;65℃,15min;4℃,∞。
[0132]
5)程序运行完成后瞬时离心将反应液收集至管底,立即进行下步连接反应。
[0133]
注意:不建议在此处停止,请继续做步骤3。如果必须停止,末端修复产物可以放在-20℃冰箱过夜,但产量可能会下降20%左右。
[0134]
4、接头连接
[0135]
1)在上述步骤3反应的pcr管中加入5μl对应的mgieasy dna adapters,涡旋震荡3次,每次3s,瞬时离心将反应液收集至管底。
[0136]
2)在冰上配制接头连接反应液(见下表4):
[0137]
表4
[0138]
组分用量ligation buffer23.4μldna ligase1.6μl总体积25μl
[0139]
3)用移液器缓慢吸取25μl配制好的接头连接反应液加入步骤1)的pcr管中,涡旋震荡6次,每次3s,瞬时离心将反应液收集至管底。
[0140]
4)运行pcr仪程序(要求无热盖),设置pcr仪参数,加热模块设置:23℃,30min,4℃,∞。
[0141]
5)程序结束后,瞬时离心将反应液收集至管底,加入20μl te buffer至总体系100μl,全部转移到新的1.5ml离心管中。立即进行下步实验。
[0142]
5、连接产物纯化
[0143]
1)将dna clean beads磁珠提前30min放至室温,并震荡混匀备用;
[0144]
2)用移液器吸取50μl dna clean beads至上步反应结束后的接头连接产物中,并轻轻吸打至少10次至完全混匀,最后一次应确保将吸头中所有液体及磁珠都打入离心管中。
[0145]
3)轻轻吸打混匀6次;
[0146]
4)室温静置孵育5min,将pcr管置于磁力架上5min使溶液澄清;
[0147]
5)移除上清,pcr管继续放置在磁力架上,向pcr管内加入200μl 85%乙醇溶液,静置30s;
[0148]
6)移除上清,再向pcr管内加入200μl 85%乙醇溶液,静置30s后彻底移除上清(建议使用10μl移液器移除底部残留乙醇溶液);
[0149]
7)室温静置至磁珠干燥,使残留乙醇彻底挥发;
[0150]
8)加入40μl的te buffer,把离心管从磁力架取下,轻轻吸打重悬磁珠,避免产生气泡,室温静置5min;
[0151]
注意:当样本dna=50ng,连接产物纯化时洗脱40μl te buffer,取19μl进行pcr。
[0152]
当样本dna《50ng,推荐连接产物纯化时洗脱21μl te buffer,取19μl进行pcr。
[0153]
9)将离心管置于磁力架上5min,使溶液澄清;
[0154]
10)用移液器吸取38μl上清液,转移到新的1.5ml离心管中(置于冰盒上),在反应管上标记样本编号,准备下一步反应。
[0155]
6、pre-pcr反应
[0156]
1)取19μl步骤4的连接纯化后产物于新的0.2mlpcr管中。
[0157]
2)在冰上按下列表格配制pcr反应体系,如下表5所示:
[0158]
表5
[0159]
组分用量pcr enzyme mix25μlpcr primer mix6μl总体积31μl
[0160]
3)用移液器吸取31μl配制好的pcr反应液加入步骤1的pcr管中,涡旋震荡3次,每次3s,瞬时离心将反应液收集至管底。
[0161]
4)将样品置于pcr仪上,运行pcr程序,如下表6所示:
[0162]
表6
[0163][0164]
5)程序运行完成后,瞬时离心将反应液收集至管底,立即进行双轮磁珠的分选纯化。
[0165]
7、扩增后纯化
[0166]
1)将dna clean beads磁珠提前30min放至室温,并震荡混匀备用;
[0167]
2)用移液器吸取50μl dna clean beads至上步反应结束后的扩增产物中,并轻轻吸打至少10次至完全混匀,最后一次应确保将吸头中所有液体及磁珠都打入离心管中。
[0168]
3)轻轻吸打混匀6次;
[0169]
4)室温静置孵育5min,将pcr管置于磁力架上5min使溶液澄清;
[0170]
5)移除上清,pcr管继续放置在磁力架上,向pcr管内加入200μl 85%乙醇溶液,静置30s;
[0171]
6)移除上清,再向pcr管内加入200μl 85%乙醇溶液,静置30s后彻底移除上清(建议使用10μl移液器移除底部残留乙醇溶液);
[0172]
7)室温静置至磁珠干燥,使残留乙醇彻底挥发;
[0173]
8)加入32μl的te buffer,把离心管从磁力架取下,轻轻吸打重悬磁珠,避免产生气泡,室温静置5min;
[0174]
9)将离心管置于磁力架上5min,使溶液澄清;
[0175]
10)用移液器吸取30μl上清液,转移到新的1.5ml离心管中(置于冰盒上),在反应管上标记样本编号,准备下一步反应。
[0176]
11)取1μl样本使用qubit4.0 fluorometer(qubit dsdna hs assay kit)进行文库浓度测定,记录文库浓度。要求最终pcr产物的摩尔产量≥1pmol。
[0177]
12)取1μl样本使用agilent2100(agilent dna 1000assay kit)进行片段检测。
[0178]
8、pooling与测序
[0179]
将不同文库pooling在一起,加载到华大基因测序平台(mgi-2000)进行测序。
[0180]
实施例2cnv检测
[0181]
(1)按照实施例1的方法,完成对样本的建库测序,获得下机数据,过滤掉低质量等reads后,使用比对软件(bwa)将这些测序reads比对到人的参考基因组上。
[0182]
(2)过滤比对结果,要求比对质量值》30,去除重复的reads,不正常配对的reads等。使用bedtools里面的工具获得reads1的比对起始位置。
[0183]
(3)根据比对起始位置,发明人通过已经发布的方法(gusnanto et al.(2014)),计算出不同区间对应的赤池信息量准则(akaike’s information criterion)和交叉验证对数似然估计值(cross validation log-likelihood)。如图2所示,选取aic最小值(或者对数似然值最大)对应的区间大小,最终选取100,000bp作为区间大小。
[0184]
(4)将人的参考基于组,每个100,000bp,划分为一个区间(bin),统计每个区间的比对reads;
[0185]
(5)bins的过滤包括:1)mappability》0.5;2)n的比例《0.5;3)不在从ucsc上下载的region文件wgencodedacmapabilityconsensusexcludable.bed和wgencodedukemapabilityregionsexcludable.bed;
[0186]
(6)每个样本的reads数,相对于bins的长度校正(除以该bin非n的比例)。
[0187]
(7)根据每个bin的gc值:统计每个窗口(bin)内a、t、c、g碱基的数量;以及g和c的数量。gc所占的比值,为该窗口的gc含量,图3为待测样本测序读段数据的gc分布图。
[0188]
(8)mappability计算:根据从ucsc下载的encode’s mappability bigwig文件,将文件中的每个region的mappability与bin比较,计算出每个bin里面所有region的mappability的平均值,作为该bin的mappability值。
[0189]
(9)过滤掉reads数目异常的bins:保留1%-99%分位数的bins;
[0190]
(10)将每个bin的gc和mappability组合,并按照它们的组合进行分组,同时计算每个gc和mappability组合对应所有bins的reads数目中位数。使用loess方法计算拟合曲线,对每个bins的reads进行校正。
[0191]
(11)将每个bins除以样本bins的中位值,校正测序数据量。
[0192]
(12)基于对应的同类型的正常样本测序数据,按照上面的处理,得到每个bins的在所有样本中的中位值作为基线。计算待测样本的log r ratio=log(rc)-log(rcnor);其中rcnor为normal参考集在同一个bins的平均值。
[0193]
(13)使用已经发表的r软件包dnacopy(https://bioconductor.org/packages/release/bioc/html/dnacopy.html)对bins的值进行平滑,校正异常值(smooth);
[0194]
(14)使用已经发表的算法(比如:循环二元分割法(cbs)将bins合并成片段(dnacopy),隐马尔可夫模型(hmmcopy:lai d,ha g,shah s(2019))最终得到该样本的cnv结果。
[0195]
表7:hz042的基于隐马尔可夫模型得到的cnv结果
[0196]
[0197]
[0198][0199]
实施例3cnv大片段变异类型检测
[0200]
(1)基于实施例2中分析方法得到cnv的片段大小(片段包含的bins数目),以及片段对应的log r ratio。该样本来自一个正常的人,根据上面的结果可以看出,长度越小,
logr的值越大(负数变的越小)。因此,根据500例左右的正常人(无cnv变异)检测出来的不同大小的基因组片段和对应的logr构建动态cutoff方法。
[0201]
(2)根据正常人群的测序分析结果,分析片段的大小和对应的log r ratio。发现随着片段所含biomarker个数的增加(即片段的长度变大),log r ratio的波动范围明显缩小。因此,可以认为片段越小,log r ratio波动大,背景噪音明显,cutoff要偏大。片段越长,log r ratio波动小,背景噪音偏小,cutoff可以适当减小。
[0202]
(3)同时通过对正常人群的研究发现,不同基因组区域(染色体)由于本身的序列特性,其背景噪音的波动范围是不一样的(如图4所示);在正常人群的血液样本中,chr19的标准偏差(sd,代表波动范围)显著大于其他染色体的。同时,也发现不同正常样本的测序均匀性不一,也就是不同人的样本,可能由于样本质量的问题,测序均一性有不同波动。
[0203]
(4)基于上面的发现,我们根据背景噪音将样本分组,并对不同染色体区域,选取每个正常样本参考组中的边界数据点,基于最小二乘法进行曲线拟合(nls),得到每个样本、每个片段,在其相应背景噪声以及biomarker个数的条件下,平均log r ratio的cutoff值(如图5所示),并将每个片段的平均log r ratio绝对值与相应cutoff值的差值,作为该片段substract。、
[0204]
(5)挑选substract》0的片段,作为最终具有cnv变异的标签。并且可以根据偏离cutoff的大小,判断片段变异的程度。图5显示了两个样本中每个片段的cutoff值。
[0205]
实施例4对mds样本进行cnv检测
[0206]
按照实施例2和3的方法,对待检测mds(骨髓增生异常综合征)样本进行cnv检测。患者为68岁,临床确诊为骨髓增生异常综合征。如附图6所示,患者在2020年7月2日接受fish检测发现异常核型-7/7q,采用上述cnv检测方法发现样本存在染色体7号的缺失,与临床上的fish结果一致。根据nccn指南提示该样本预后不良。
[0207]
实施例5对样本进行cnv检测
[0208]
使用实施例2和3中同样方法,对另外一例aml(急性髓系白血病)样本进行cnv分析,检测结果如下:
[0209]
对应异常片段的检测结果如图7所示,对图7中检测到的异常片段进行注释。
[0210]
根据基因的位置和变异在染色体上的位置注释,结果如图8所示。表明该样本的cnv突变包含基于kmt2anm_001197104。kmt2a明显扩增,该基因编码一个转录共激活因子,它在早期发育和造血过程中在调节基因表达中起着至关重要的作用。根据aml的相关指南:该基因拷贝数的变化与病人的治疗和预后有关系。
[0211]
图8中结果表明,采用本发明的确定待测样本预定大片段拷贝数(cna)变异的方法,对已知病例的分析结果与细胞遗传学结果一致性能够达到91%,说明本发明的方法能够快速、准确地检测cnv大片段。
[0212]
实施例6
[0213]
基于上述方法,对20例aml和mds进行同样的cnv变异检测,并与临床常规的检测结果对比。并按照指南中进行分组比较一致性,以及整体的一致性。具体分组为chr5/chr5q,chr7/chr7q是否存在缺失,复杂核型:改样本存在3条及以上的染色体变异;非复杂核型:改样本包含1条或2条染色体变异。正常核型:该样本核型正常,不具有染色体变异。根据nccn指南或者相关的aml/mds国际共识:5q和7q是否缺失,以及染色体变异的数目(正常核型,非
复杂核型,复杂核型)对aml和mds的预后是不一样的。医生根据不同的预后,修订治疗
[0214]
实施例7
[0215]
根据正常样本,选取人群中多态性(snp)位点频率在5%~95%之间的已知snp位点,并挑选出目标区域(如图9)深度大于4x以上的位点。统计这些挑选出来位点的reads支持数,计算基因型。从而获得纯合的位点数目和杂合位点数目。通过正常参照样本在同一个区域的统计结果,计算出该区域的杂合位点数目和杂合/纯合比例,同时计算出这两个统计量的均值和标准差,对待测样本计算出其zscore。图9显示待测2个样本(正常和发生loh)的杂合位点数目和对应的突变频率。其中n2几乎都是纯合位点(突变频率在0%和100%),在该区域杂合位点数目的z-score和杂合/纯合的占比计算出的zscore都是显著小于-3的。
[0216]
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必须针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。此外,在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。
[0217]
尽管上面已经示出和描述了本发明的实施例,可以理解的是,上述实施例是示例性的,不能理解为对本发明的限制,本领域的普通技术人员在本发明的范围内可以对上述实施例进行变化、修改、替换和变型。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1