一种基于生成对抗网络的单细胞RNA序列缺失值填补方法
一种基于生成对抗网络的单细胞rna序列缺失值填补方法
技术领域
1.本发明属于生物信息领域,具体涉及一种基于生成对抗网络的单细胞rna序列缺失值填补方法。
背景技术:
2.随着高通量测序技术的发展,基因组学测序中单细胞rna测序(scrna-seq)技术成为近年来的一个热门话题。相比于之前的批量rna测序(bulk rna-seq)技术而言,scrna-seq具有相对较高的噪声水平,特别是由于所谓的辍学事件(dropout event)。在scrna-seq数据集的基因细胞表达矩阵中观察到的零由真零和缺失值零组成。真零是基因不表达导致的,而缺失值零是由所谓的辍学事件引起的。辍学事件是一种特殊类型的缺失值,这是由于测序实验中的低rna输入和单细胞水平上基因表达模式的随机性造成的。然而,大多数为scrna序列分析开发的统计工具并没有明确地解决这些辍学事件。
3.利用缺失值填补方法对缺失值进行估计可以保留原始数据中的重要信息,从而避免删除包含缺失值而信息量大的有效基因。又因为缺失值填补方法将不完整数据集转换成完整数据集,从而在数据挖掘的过程当中能够在完整的数据集上,而不是在数据集的子集上进行分析,从而避免分析出的结果产生局部规律的错误,因此,对缺失值填补方法进行研究是十分必要的。
4.深度学习发展迅速,在图像分类、语音识别等领域取得了较好的成绩。一些研究者已经将最广泛使用的卷积神经网络(convolutional neural networks,cnn)和循环神经网络(recurrent neural network,rnn)框架转移到生物医学领域。但总体而言,深度学习在生物医学领域的应用,尤其是在基因组领域的应用还非常有限。造成这种现象的原因主要有以下两点:首先,常用的rnn/cnn不适用于基因组数据建模;其次,对于深度学习的非专业人士而言,没有一个友好的自动化机器学习框架。
5.目前基于机器学习方法的缺失值填补方法的表现也并不理想。其原因在于:其一是原始数据中大量的零,使得机器学习方法很难提取数据深层的信息,反而将大部分的零视为真零,即不进行填补,因此机器学习方法填补后的数据较为离散。其二是在使用某些基于机器学习的缺失值填补方法进行填补后,输出的数据中含有负值,但现实的基因表达值应该均为非负值。
6.综上所述,基于现有的机器学习方法进行缺失值填补时很难提取到数据的深层信息,导致对缺失数据填补的准确率较低。
技术实现要素:
7.本发明的目的是为解决采用现有方法对缺失数据填补的准确率低的问题,而提出了一种基于生成对抗网络的单细胞rna序列缺失值填补方法。
8.本发明为解决上述技术问题所采取的技术方案是:
9.一种基于生成对抗网络的单细胞rna序列缺失值填补方法,所述方法具体包括以
下步骤:
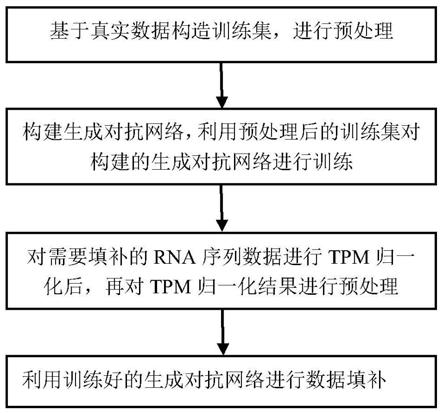
10.步骤一、基于rna序列真实数据构造训练集;
11.步骤二、构建生成对抗网络,所述生成对抗网络中包括生成器和判别器,其中,生成器是由编码模块和解码模块组成的自编码器;
12.利用训练集对构建的生成对抗网络进行训练;
13.步骤三、对需要填补的rna序列数据进行tpm归一化后,再对tpm归一化结果进行预处理,训练好的生成对抗网络根据预处理结果生成填补后的rna序列数据。
14.进一步地,所述步骤一的具体过程为:
15.步骤一一、从数据集usoskin中获取rna序列数据,作为训练集;
16.步骤一二、获取的每条rna序列数据均根据设置的缺失参数,来生成带有缺失值的rna序列数据,并给生成的带有缺失值的rna序列数据打上标签;
17.并分别对生成的每条带有缺失值的rna序列数据进行tpm归一化,分别获得各条带有缺失值的rna序列数据所对应的tpm归一化后数据;
18.步骤一三、对tpm归一化后的数据进行预处理,分别获得每条rna序列数据所对应的预处理后矩阵;
19.将预处理后的矩阵作为训练生成对抗网络的输入。
20.进一步地,所述预处理的方式为基因选择和对数转换。
21.进一步地,所述步骤二中,生成器的损失函数为:
22.利用drimpute对预处理后的矩阵进行填补,将填补结果作为方向约束来建立生成器的损失函数:
[0023][0024]
其中,e是编码器矩阵、d是解码器矩阵,r为中间变量,σ是神经网络中应用于编码器层的sigmoid激活函数,||
·
||o表示针对存在的非零计数计算损耗,δ是方向约束权重,用于控制方向约束项对成本函数的贡献,λ是正则化系数,f
′
是方向约束,||
·
||f是f范数。
[0025]
进一步地,所述中间变量r为:
[0026]
r=mox
ꢀꢀꢀ
(2)
[0027]
其中,o是hadamard乘积,m是二进制掩码,x代表预处理后的矩阵。
[0028]
进一步地,所述步骤二中,判别器的损失函数为:
[0029][0030]
其中,ak是第k个缺失数据的真实值通过判别器输出的标签值,是生成器输出的第k个缺失数据的预测值通过判别器输出的标签值,k=1,2,
…
,k,k是缺失数据的总个数,w是判别器的参数集。
[0031]
进一步地,所述自编码器的正则化系数λ的取值在0~12000之间。
[0032]
进一步地,所述自编码器的隐藏层层数在1500~8000之间。
[0033]
更进一步地,所述生成对抗网络的学习率初始值在10-5
~10-3
之间。
[0034]
本发明的有益效果是:
[0035]
本发明通过生成对抗网络对rna序列缺失值进行填补,在生成器部分,引入由drimpute填补完成的数据作为方向约束,在使用自编码器生成缺失值的同时,在损失函数中加入方向约束项,并对解码层给予了一个relu激活函数来解决填补后的数据出现负值的情况。实验证明,采用本发明的方法可以显著提高数据填补的准确率。
附图说明
[0036]
图1是本发明方法的流程图。
[0037]
图2(a)是原始模拟数据当v=8时,直接聚类的结果图;
[0038]
rtsne_1和rtsne_2是对数据使用t_sne进行降维后得到的两列值;
[0039]
图2(b)是原始模拟数据当v=8时,采用scimpute方法进行填补后再聚类的结果图;
[0040]
图2(c)是原始模拟数据当v=8时,采用autoimpute方法进行填补后再聚类的结果图;
[0041]
图2(d)是原始模拟数据当v=8时,采用本发明方法进行填补后再聚类的结果图。
具体实施方式
[0042]
具体实施方式一、结合图1说明本实施方式。本实施方式所述的一种基于生成对抗网络的单细胞rna序列缺失值填补方法,所述方法具体包括以下步骤:
[0043]
步骤一、基于rna序列真实数据构造训练集;
[0044]
步骤二、构建生成对抗网络,所述生成对抗网络中包括生成器和判别器,其中,生成器是由编码模块和解码模块组成的自编码器;
[0045]
利用训练集对构建的生成对抗网络进行训练;
[0046]
步骤三、对需要填补的rna序列数据进行tpm归一化后,再对tpm归一化结果进行预处理(即基因选择和对数转换),训练好的生成对抗网络根据预处理结果生成填补后的rna序列数据。
[0047]
具体实施方式二:本实施方式与具体实施方式一不同的是,所述步骤一的具体过程为:
[0048]
步骤一一、从数据集usoskin中获取rna序列数据,作为训练集;
[0049]
数据集usoskin由文献1(usoskin,d.et al.unbiased classifcation of sensory neuron types by large-scale single-cell rna sequencing.nat.neuroscience 18,145(2015))发表共享。
[0050]
步骤一二、获取的每条rna序列数据均根据设置的缺失参数,来生成带有缺失值的rna序列数据,并给生成的带有缺失值的rna序列数据打上标签;
[0051]
标签代表的是每个基因位点是否为缺失值,如果是缺失值,则自编码器向着目标数据学习,使对缺失数据的预测结果尽量接近于目标数据,以得到训练好的自编码器参数;
[0052]
并分别对生成的每条带有缺失值的rna序列数据进行tpm归一化,分别获得各条带有缺失值的rna序列数据所对应的tpm归一化后数据;
[0053]
步骤一三、对tpm归一化后的数据进行预处理,分别获得每条rna序列数据所对应
的预处理后矩阵;
[0054]
将预处理后的矩阵作为训练生成对抗网络的输入。
[0055]
其它步骤及参数与具体实施方式一相同。
[0056]
具体实施方式三:本实施方式与具体实施方式一或二不同的是,所述预处理的方式为基因选择和对数转换。
[0057]
其它步骤及参数与具体实施方式一或二相同。
[0058]
具体实施方式四:本实施方式与具体实施方式一至三之一不同的是,所述步骤二中,生成器的损失函数为:
[0059]
利用drimpute对预处理后的矩阵进行填补,将填补结果作为方向约束来建立生成器的损失函数:
[0060][0061]
其中,e是编码器矩阵、d是解码器矩阵,r为中间变量,σ是神经网络中应用于编码器层的sigmoid激活函数,||
·
||o表示针对存在的非零计数计算损耗,δ是方向约束权重,用于控制方向约束项对成本函数的贡献,λ是正则化系数,f
′
是方向约束,||
·
||f是f范数。
[0062]
其它步骤及参数与具体实施方式一至三之一相同。
[0063]
具体实施方式五:本实施方式与具体实施方式一至四之一不同的是,所述中间变量r为:
[0064]
r=mox
ꢀꢀꢀ
(2)
[0065]
其中,o是hadamard乘积,m是含有1的二进制掩码,r包含一个非零项,其他地方是0,x代表预处理后的矩阵。
[0066]
预处理后的矩阵x经过生成器的输出为:ψ是神经网络中应用于解码器层的relu激活函数。
[0067]
其它步骤及参数与具体实施方式一至四之一相同。
[0068]
具体实施方式六:本实施方式与具体实施方式一至五之一不同的是,所述步骤二中,判别器的损失函数为:
[0069][0070]
其中,ak是第k个缺失数据的真实值通过判别器输出的标签值,是生成器输出的第k个缺失数据的预测值通过判别器输出的标签值,k=1,2,
…
,k,k是缺失数据的总个数,w是判别器的参数集。
[0071]
本实施方式中,在缺失数据的位置上,将生成器生成的填补数据标注为0,将真实数据标注为1,将生成的填补数据和真实数据共同输入判别器进行真伪判别,判别器输出填补数据和真实数据对应的真伪标签。
[0072]
其它步骤及参数与具体实施方式一至五之一相同。
[0073]
具体实施方式七:本实施方式与具体实施方式一至六之一不同的是,所述自编码
器的正则化系数λ的取值在0~12000之间。
[0074]
正则化系数用于防止非零值过度拟合,在本发明中正则化系数也用于控制正则项对成本函数的贡献。
[0075]
其它步骤及参数与具体实施方式一至六之一相同。
[0076]
具体实施方式八:本实施方式与具体实施方式一至七之一不同的是,所述自编码器的隐藏层层数在1500~8000之间。
[0077]
其它步骤及参数与具体实施方式一至七之一相同。
[0078]
具体实施方式九:本实施方式与具体实施方式一至八之一不同的是,所述生成对抗网络的学习率初始值在10-5
~10-3
之间。
[0079]
其它步骤及参数与具体实施方式一至八之一相同。
[0080]
选择合适的学习率可以使生成器避免陷入局部最小值并更快达到损失函数的最小值。
[0081]
本发明中设置的正则化系数值、隐藏层大小、学习率初始值以及收敛阈值如表1所示:
[0082]
表1
[0083][0084]
实验结果及分析
[0085]
利用r语言cidr包模拟了5个细胞类每类40个细胞共计200个细胞的单细胞rna测序数据,数据规模为200细胞*20200基因。对这些数据分别设置随机丢失参数v=8,对应数据缺失比例为41.20%共产生1组缺失数据。缺失数据中只含数据缺失导致的零,没有基因不表达导致的真零。使用本发明方法对模拟数据进行缺失值填补,基于填补后的数据结果进行t-sne降维、可视化,再用k-means聚类。并与scimpute方法和autoimpute方法进行比较。
[0086]
将本发明、scimpute和autoimpute三种方法的聚类结果与原始数据归一化后直接聚类的结果进行对比,分别计算处理过的数据与原始数据的聚类评价指标。统计数据聚类结果,根据聚类评价指标绘图进行分析。
[0087]
如图2(a)至图2(d)所示,当随机丢失参数为8时,分别为对数据采用直接聚类以及scimpute、autoimpute、本发明方法三种方法进行填补后再聚类的结果图。
[0088]
根据聚类结果,可以计算出这三种方法在模拟数据集上聚类的评价指标,汇总后如表2所示。
[0089]
表2三种方法对应模拟数据聚类评价指标
[0090][0091]
其中:
[0092]
1、rand是兰德指数
[0093]
兰德指数计算样本预测值与真实值之间的相似度,取值范围是[0,1],值越大意味着聚类结果与真实情况越吻合。
[0094][0095]
2、ari是调整兰德指数
[0096]
对于随机结果,兰德指数并不能保证分数接近0,因此具有更高区分度的调整兰德指数被提出,取值范围是[-1,1],值越大表示聚类结果和真实情况越吻合。
[0097][0098]
3、fm是fm指数
[0099]
如果记在c中属于同一类的样本对中,在c
*
也是同一类的样本对的比例为p1,在c
*
中属于同一类的样本对中,在c也是同一类的样本对的比例为p2,那么fm就是p1和p2的几何平均,取值范围是[0,1]。
[0100][0101]
4、jaccard是jaccard系数
[0102]
jaccard系数刻画了所有属于同一类的样本对(要么在c中属于同一类,要么在c
*
中属于同一类),同时在c和c
*
中属于同一类的样本量的比值,取值范围是[0,1]。
[0103][0104]
为了更加清晰的进行分析,给出了三种方法在模拟数据集上结果好坏的排序,如
表3所示,其中1表示最优,3表示最差。
[0105]
表3三种方法对应模拟数据聚类情况排序
[0106][0107]
分析上述实验结果,可以发现,本发明提出的方法在模拟数据集的聚类实验中有着很好的表现。autoimpute的表现不太理想,scimpute则略差于本发明方法。总的来说,本发明方法在模拟数据集的表现是最好的。
[0108]
本发明的上述算例仅为详细地说明本发明的计算模型和计算流程,而并非是对本发明的实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动,这里无法对所有的实施方式予以穷举,凡是属于本发明的技术方案所引伸出的显而易见的变化或变动仍处于本发明的保护范围之列。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1