一种基于两路多分支CNN的单步逆合成方法及系统
一种基于两路多分支cnn的单步逆合成方法及系统
技术领域
1.本发明涉及利用分子序列信息以及分子指纹信息进行单步逆合成的分析领域,尤其涉及一种基于两路多分支cnn的单步逆合成方法及系统,属于卷积神经网络机器学习模型在单步逆合成中的应用。
背景技术:
2.逆合成分析是化学家广泛采用的一种技术,由corey等人于20世纪60年代提出,用于设计目标分子的合成路线,即通过递归的方式将目标分子不断的转化成可用的“前体”分子,直到确定商业上可用的起始分子。自20世纪60年代开始,化学家们已经意识到计算机辅助在化学逆合成方面的前景,同时也设计出了各种的方法,用于合成具有高价值的化合物。与化学逆合成类似,生物逆合成是一种潜在地简化生物合成路径设计的过程,它于2010年被首次提出。作为一种概念性的路径设计策略,生物逆合成与逆合成具有共同的优势,可以引导研究人员逐步获得更简单的可用中间体。
3.无论化学逆合成还是生物逆合成,都需要对反应途径中间体的反应物进行预测,即单步逆合成,用于根据目标分子预测目标分子的反应物。根据是否使用规则,单步逆合成被分为基于规则的方法与无规则的方法。基于规则的方法将目标分子与大量的反应规则相匹配,而无规则的方法将逆合成预测作为一个序列到序列的问题。
4.现有的单步逆合成预测主要利用分子的smiles序列来进行单步逆合成,基于规则的方法将目标分子与大量的反应规则相匹配,或者直接对规则以及反应物联合建模;无规则的方法则直接使用seq2seq模型预测反应物smiles序列。但是,smiles序列的一个劣势在于,smiles表征假定了分子中原子之间的顺序,这不能有效地反映分子中原子之间的复杂关系,仅使用smiles序列将会导致预测性能不佳,因此需要结合其他特征以提供更多分子信息进行预测。
技术实现要素:
5.本发明提出了一种基于两路多分支cnn的单步逆合成方法及系统,基于两路多分支cnn的单步逆合成方法用于预测能产生目标分子的反应规则,该方法不需要复杂的领域知识,直接预测反应规则并同时获得预测概率值,解决了现有技术中的方法存在的预测效果不佳的技术问题。
6.为了解决上述技术问题,本发明第一方面提供了一种基于两路多分支cnn的单步逆合成方法,包括:
7.s1:获取既定的反应数据集r,既定的反应数据集包含不同的反应,每条反应包含底物分子和产物分子,根据既定的反应数据集中反应的产物分子构造分子集合s,根据既定的反应数据集中的反应构造反应规则集t;
8.s2:根据构造的分子集合、反应规则集以及分子和反应规则之间的对应关系,构造输入数据集d={(s,t)},其中,s表示分子,t表示反应规则,s∈s,t∈t,二元组(s,t)表示反
应规则t所对应的反应中存在能产生s的反应;
9.s3:搭建两路多分支cnn,构建单步逆合成预测模型;
10.s4:利用构造的输入数据集d作为训练集训练步骤s3中构建的单步逆合成预测模型,得到训练好的预测模型a;
11.s5:将待预测的目标分子输入训练好的预测模型a,预测出反应规则集合t中每个反应规则产生该目标分子的概率,根据概率值,选择其中概率最大的前k条规则作为结果输出,k为设定的参数。
12.在一种实施方式中,步骤s1中,步骤s1中既定的反应数据集r为从公共资源收集的已知反应整理后得到的集合,r中的每一个反应包含有如下组成部分:反应id,代表一个或多个反应物的smiies序列,代表一个产物分子的smiles序列;其中,对有多个产物的原始反应,分解为多条单产物的反应,并赋予相同的id。
13.在一种实施方式中,步骤s4中搭建的两路多分支cnn包括五层,分别为输入层、两路多分支卷积层、特征拼接层、全连接层和输出层;
14.其中,输入层包含一个输入节点,用于输入分子的smiles序列;
15.两路多分支卷积层由两路结构类似的网络组成,用于得到输入分子的两种卷积特征,其中每一路均包括多条分支,每条分支依次由卷积、批量归一化、sigmoid激活和最大池化操作构成;对于每一路的输入,不同分支采用不同大小卷积核卷积,并进行批量归一化、sigmoid激活和最大池化后得到不同的卷积向量,多个卷积向量经过拼接操作得到该路输入所对应的卷积特征;
16.拼接层用于对得到的两种卷积特征进行拼接得到输入分子的融合表示特征;
17.全连接层通过softmax函数计算反应规则集t中各反应规则产生输入分子的概率,概率值取值范围为[0,1];
[0018]
输出层包含|t|个节点,分别对应反应规则集t中的每个反应规则,|t|表示集合t的大小。
[0019]
在一种实施方式中,两路多分支卷积层中,一路的输入v1是基于输入分子的smiles序列生成的半径值为2的扩展连通性指纹,另一路的输入v2是基于输入分子的smiles序列和字母表生成的单热编码矩阵,其中,字母表由预定的由所有分子smiles序列包含的符号组成。
[0020]
在一种实施方式中,两路多分支卷积层采用的卷积核均为一维卷积核,对其中一路,设第一个分支的卷积核大小为size0,则第i个分支的卷积核的大小设为size0+(i-1)
×
step,其中step为卷积核大小增加的步长。
[0021]
在一种实施方式中,步骤s5中,待预测的目标分子输入模型之前,转换smiles序列的形式。
[0022]
基于同样的发明构思,本发明第二方面提供了一种基于两路多分支cnn的单步逆合成系统,包括:
[0023]
反应数据集获取模块,用于获取既定的反应数据集r,既定的反应数据集包含不同的反应,每条反应包含底物分子和产物分子,根据既定的反应数据集中反应的产物分子构造分子集合s,根据既定的反应数据集中的反应构造反应规则集t;
[0024]
训练集构造模块,用于根据构造的分子集合、反应规则集以及分子和反应规则之
间的对应关系,构造输入数据集d={(s,t)},其中,s表示分子,t表示反应规则,s∈s,t∈t,二元组(s,t)表示反应规则t所对应的反应中存在能产生s的反应;
[0025]
模型构建模块,用于搭建两路多分支cnn,构建单步逆合成预测模型;
[0026]
模型训练模块,用于利用构造的输入数据集d作为训练集训练步骤s3中构建的单步逆合成预测模型,得到训练好的预测模型a;
[0027]
单步逆合成预测模块,用于将待预测的目标分子输入训练好的预测模型a,预测出反应规则集合t中每个反应规则产生该目标分子的概率,根据概率值,选择其中概率最大的前k条规则作为结果输出,k为设定的参数。
[0028]
在一种实施方式中,所述系统还包括:结果可视化模块,用于利用图形方式直观显示分子、分子相关的反应、预测得到的反应规则。
[0029]
本技术实施例中的上述一个或多个技术方案,至少具有如下一种或多种技术效果:
[0030]
本发明提供的基于两路多分支cnn的单步逆合成方法,首先根据获取的既定的反应数据集r分别构造分子集合s和反应规则集t,并根据构造的分子集合、反应规则集以及分子和反应规则之间的对应关系,构造输入数据集d,然后搭建两路多分支cnn,构建单步逆合成预测模型;接着利用构造的输入数据集d训练构建的单步逆合成预测模型;最后用训练好的模型a进行单步逆合成预测。该方法不需要复杂的领域知识,可以直接预测反应规则并同时获得预测概率值。同现有方法相比,能够提供更多的潜在信息,从而可以改善预测效果。
[0031]
此外,本发明还提出一种基于两路多分支cnn的单步逆合成系统,用于快速辅助化学家进行单步逆合成。
附图说明
[0032]
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
[0033]
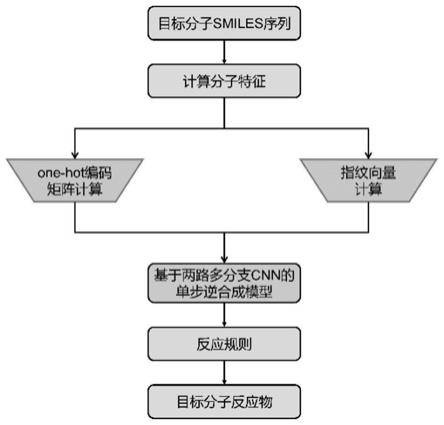
图1为本发明实施例中一种基于两路多分支cnn的单步逆合成方法流程图;
[0034]
图2为本发明实施例的使用的基于两路多分支cnn的单步逆合成模型a示意图;
[0035]
图3为本发明实施例中使用的smiles序列的one-hot编码矩阵示意图;
[0036]
图4为本发明实施例中一种基于两路多分支cnn的单步逆合成系统的模块示意图。
具体实施方式
[0037]
本发明针对现有技术的不足,提出了一种基于两路多分支cnn的单步逆合成方法。基于两路多分支cnn的单步逆合成方法用于预测能产生目标分子的反应规则,该方法不需要复杂的领域知识,直接预测反应规则并同时获得预测概率值。同现有方法相比,能够提供更多的潜在信息。本发明同时提出一种基于两路多分支cnn的单步逆合成系统,用于快速辅助化学家进行单步逆合成。
[0038]
为使本发明实施例的目的、技术方案和优点更加清楚,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例是
本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
[0039]
实施例一
[0040]
本发明实施例提供了一种基于两路多分支cnn的单步逆合成方法,包括:
[0041]
s1:获取既定的反应数据集r,既定的反应数据集包含不同的反应,每条反应包含底物分子和产物分子,根据既定的反应数据集中反应的产物分子构造分子集合s,根据既定的反应数据集中的反应构造反应规则集t;
[0042]
s2:根据构造的分子集合、反应规则集以及分子和反应规则之间的对应关系,构造输入数据集d={(s,t)},其中,s表示分子,t表示反应规则,s∈s,t∈t,二元组(s,t)表示反应规则t所对应的反应中存在能产生s的反应;
[0043]
s3:搭建两路多分支cnn,构建单步逆合成预测模型;
[0044]
s4:利用构造的输入数据集d作为训练集训练步骤s3中构建的单步逆合成预测模型,得到训练好的预测模型a;
[0045]
s5:将待预测的目标分子输入训练好的预测模型a,预测出反应规则集合t中每个反应规则产生该目标分子的概率,根据概率值,选择其中概率最大的前k条规则作为结果输出,k为设定的参数。
[0046]
具体来说,步骤s1是根据既定的反应数据集r构造分子集合与反应规则集t,步骤s2是构建输入数据集,s3是构建单步逆合成预测模型,步骤s4是模型的训练,步骤s5是使用训练好的模型a进行单步逆合成预测。
[0047]
在一种实施方式中,步骤s1中,既定的反应数据集r为从公共资源收集的已知反应整理后得到的集合,r中的每一个反应包含有如下组成部分:反应id,代表一个或多个反应物的smiies序列,代表一个产物分子的smiles序列;其中,对有多个产物的原始反应,分解为多条单产物的反应,并赋予相同的id。
[0048]
其中,当反应包含催化酶时,反应包含的信息还包括催化酶编号。
[0049]
反应规则集t可以通过调用rdchiral中的template_extractor函数来构造。该函数的输入为反应数据集r中的一条具体反应,输出为该反应所对应的反应规则。反应规则集t中的每一条反应规则被赋予一个唯一的规则标签。
[0050]
在一种实施方式中,步骤s4中搭建的两路多分支cnn包括五层,分别为输入层、两路多分支卷积层、特征拼接层、全连接层和输出层;
[0051]
其中,输入层包含一个输入节点,用于输入分子的smiles序列;
[0052]
两路多分支卷积层由两路结构类似的网络组成,用于得到输入分子的两种卷积特征,其中每一路均包括多条分支,每条分支依次由卷积、批量归一化、sigmoid激活和最大池化操作构成;对于每一路的输入,不同分支采用不同大小卷积核卷积,并进行批量归一化、sigmoid激活和最大池化后得到不同的卷积向量,多个卷积向量经过拼接操作得到该路输入所对应的卷积特征;
[0053]
拼接层用于对得到的两种卷积特征进行拼接得到输入分子的融合表示特征;
[0054]
全连接层通过softmax函数计算反应规则集t中各反应规则产生输入分子的概率,概率值取值范围为[0,1];
[0055]
输出层包含|t|个节点,分别对应反应规则集t中的每个反应规则,|t|表示集合t
的大小。
[0056]
请参见图2,为本发明实施例的使用的基于两路多分支cnn的单步逆合成模型a示意图。
[0057]
在一种实施方式中,两路多分支卷积层中,一路的输入v1是基于输入分子的smiles序列生成的半径值为2的扩展连通性指纹,另一路的输入v2是基于输入分子的smiles序列和字母表生成的单热编码矩阵,其中,字母表由预定的由所有分子smiles序列包含的符号组成。
[0058]
具体来说,扩展连通性指纹(extended connectivity fingerprints,ecfp),的长度为2048。另一路的输入v2的单热(one-hot)编码矩阵,其大小300*40。其中,300为既定的分子smiles的最大长度,如果smiles长度小于等于300,则直接在smiles后补0,直到smiles长度为300为止;如果smiles长度大于300,则将smiles字符串进行截断,把长度超过300的部分都直接截断,而40为字母表的长度。
[0059]
请参见图3,为本发明实施例中使用的smiles序列的one-hot编码矩阵示意图。
[0060]
在一种实施方式中,两路多分支卷积层采用的卷积核均为一维卷积核,对其中一路,设第一个分支的卷积核大小为size0,则第i个分支的卷积核的大小设为size0+(i-1)
×
step,其中step为卷积核大小增加的步长。
[0061]
其中,两路多分支卷积层所用卷积核均为一维卷积核,仅仅在输入通道数上有所区别,而输出通道均为40。对于输入v1的一路,该路所有的卷积核的输入通道为1,对于输入v2的一路,该路所有卷积核的输入通道为40,即字母表的长度。卷积核对其中一个路,设第一个分支的卷积核大小为size0,则第i个分支的卷积核的大小设为size0+(i-1)
×
step,其中step为卷积核大小增加的步长。
[0062]
在一种实施方式中,步骤s5中,待预测的目标分子输入模型之前,转换smiles序列的形式。
[0063]
请参见图1,本发明实施例提供的基于两路多分支cnn的单步逆合成预测的流程图。
[0064]
具体实施时,步骤s1的既定的反应数据集r是对从公共资源收集的已知反应整理后得到的集合。r中的每一个反应包含有如下组成部分:反应id,代表一个或多个反应物的smiies序列,代表一个产物分子的smiles序列,催化酶编号(如果有)。对有多个产物的原始反应,分解为多条单产物的反应,并被赋予相同的id。
[0065]
在一种情形中:既定的反应数据集r是对可公开获得的化学反应数据集uspto-50k中的所有化学反应进行整理后得到。r中每一个反应包含反应id,代表一个或多个反应物的smiies序列和代表一个产物分子的smiles序列。
[0066]
另一种情形中:既定的反应数据集r是对可公开获得的代谢反应数据集metanetx中的所有代谢反应进行整理后得到。r中的每一个反应包含有如下组成部分:反应id,代表一个或多个反应物的smiies序列,代表一个产物分子的smiles序列,催化酶编号(可以为空)。
[0067]
反应规则集t是通过调用rdchiral中的template_extractor函数构造的。该函数的输入为反应数据集r中的一条具体反应,输出为该反应所对应的反应规则。反应规则集t中的每一条反应规则被赋予一个唯一的规则标签。
[0068]
步骤s3中,两路多分支cnn网络结构主要包括五层:输入层、两路多分支卷积层、特征拼接层、全连接层和输出层。各层的处理步骤如下:
[0069]
3.1输入层包含一个输入节点,用于输入分子的smiles序列;
[0070]
3.2两路多分支卷积层由两路结构类似的网络组成,用于得到3.1中输入分子的两种卷积特征。其中每一路均包括多条分支,每条分支依次由卷积、批量归一化、sigmoid激活和最大池化操作构成。对该路的输入,不同分支采用不同大小卷积核卷积,并进行批量归一化、sigmoid激活和最大池化后的到不同的卷积向量,多个向量经过拼接操作得到该路输入所对应的卷积特征。
[0071]
3.3拼接层用于对3.2得到的两种卷积特征进行拼接得到3.1中输入分子的融合表示特征;
[0072]
3.4全连接层通过softmax函数计算反应规则集t中各反应规则产生3.1中输入分子的概率,概率值取值范围为[0,1];
[0073]
3.5输出层包含|t|个节点,分别对应反应规则集t中的每个反应规则,|t|表示集合t的大小。
[0074]
在3.2中,两路多分支卷积层中一路的输入v1是基于3.1中输入分子的smiles序列生成的半径值为2的扩展连通性指纹(extended connectivity fingerprints,ecfp),其长度为2048。另一路的输入v2是基于3.1中输入分子的smiles序列和预定的由所有分子smiles序列包含的符号组成的字母表生成的单热(one-hot)编码矩阵,大小为300*40。其中,300为既定的分子smiles的最大长度,如果smiles长度小于等于300,则直接在smiles后补0,直到smiles长度为300为止;如果smiles长度大于300,则将smiles字符串进行截断,把长度超过300的部分都直接截断,而40为字母表的长度。输入分子的smiles序列中出现的字母,单热编码矩阵中对应位置为1,否则为0。
[0075]
在3.2中所用卷积核均为一维卷集核。对于输入v1的一路,该路所有的卷积核的输入通道为1,对于输入v2的一路,该路所有卷积核的输入通道为40,即字母表的长度。对其中一个路,设第一个分支的卷积核大小为size0,则第i个分支的卷积核的大小设为size0+(i-1)
×
step,其中step为卷积核大小增加的步长。
[0076]
图2为该两路多分支cnn的一个具体实施方案的网络结构。
[0077]
一个具体的实施例中,
[0078]
3.2中的sigmoid激活函数为:
[0079][0080]
其中,e为自然常数,x表示上一层的输出,f(x)为激活函数的输出。该函数具有单增以及反函数单增等性质,平滑且易于求导,用于隐层神经元输出,取值范围为(0,1),它可以将一个实数映射到(0,1)的区间。
[0081]
在一种实施方式中,s3中的模型,针对输入为ecfp指纹的一路多分支卷积,将起始卷积核的大小size0设为32,卷积核大小增加的步长step设为32,最大卷积核的大小不超过2048(ecfp指纹向量的长度),一共构建64条分支。
[0082]
在一种实施方式中,s3中的模型,针对输入为单热编码矩阵的一路多分支卷积,将起始卷积核的大小size0设为5,卷积核大小增加的步长step设为5,最大卷积核的大小不超
过300(smiles序列的最大长度),一共构建60条分支。
[0083]
3.4中的softmax函数,具体定义为:
[0084][0085]
其中,e为自然常数,∑
jej
表示所有神经元以e为底数以该神经元为指数的幂之和,si表示第i个神经元经过softmax后的结果。
[0086]
在一种实施方式中,模型的损失函数采用交叉熵损失函数,其具体表示为:
[0087][0088]
其中,m为标签的总数,即为反应规则集t的大小,n为样本总数;y
i,c
一个二进制标识符,表示样本i的真实标签是否为c,即预测的样本i的规则是否与真实规则c相同,当样本i的真实标签与c相同时取1,否则取0;p
i,c
表示样本i的标签为c的概率,即预测的样本i的规则为c的概率。
[0089]
在一种实施方式中,采用adam优化方法进行模型的优化。
[0090]
在具体实施例中,对步骤s2构建的数据集按8:1:1的比例随机划分为训练集、验证集和测试集。训练集和验证机用于模型的训练,测试集不参与训练,用于评价训练好的模型的单步逆合成预测性能。训练轮数(epoch)设置为20,在每轮中进行多次迭代直到所有训练样本均参与一次训练,参与一次迭代的训练样本数目batch_size设置值为128。初始学习率为设置为0.001。
[0091]
实施例1:对可公开获得的化学反应数据集uspto-50k中的所有化学反应进行整理后得到既定的反应数据集r。根据r整理构建步骤三所述的数据集d,并按8:1:1的比例随机划分为训练集、验证集和测试集。其中的训练集和验证集训练模型,测试集对训练得到的单步化学逆合成预测模型的预测精度进行测试。表1展示了本发明所提出的基于两路多分支cnn的单步逆合成预测方法在单步化学逆合成的预测性能。目前该领域最好的top-1预测精度不超过52.5%,显然,基于本发明得到的模型的预测精度显著高于目前该领域的最好结果。基于本发明的模型在top-3,top-5以及top-10上的预测精度也都高于现有的模型。
[0092]
表1:基于uspto-50k构建的单步化学逆合成模型预测精度
[0093]
top-1top-3top-5top-1061.1%79.1%83.9%87.7%
[0094]
实施例2:对可公开获得的代谢反应数据集metanetx中的所有代谢反应进行整理后得到既定的反应数据集r。根据r整理构建步骤三所述的数据集d,并按8:1:1的比例随机划分为训练集、验证集和测试集。其中的训练集和验证集训练模型,测试集对训练得到的单步生物逆合成预测模型的性能进行测试。表2展示了本发明所提出的基于两路多分支cnn的单步逆合成预测方法在单步生物逆合成的预测性能。在生物逆合成中,目前还未发现单步生物逆合成的预测工作报道,本发明将化学领域中的单步逆合成方法成功用于单步生物逆合成,拓宽了生物逆合成的新思路,填补了单步生物逆合成预测的空白。
[0095]
表2:基于metanetx构建的单步生物逆合成模型预测精度
[0096][0097][0098]
本发明是单步逆合成领域里首个使用卷积神经网络来进行预测的方法,实现了一个端到端的单步逆合成框架,不需要复杂的领域参数设置。同时,本发明利用分子指纹与one-hot编码的融合特征,相比较仅采用单一信息的已有方法,本发明能够提供更多的潜在信息。
[0099]
与现有技术相比较,本发明的有益效果在于:
[0100]
1.本发明是单步逆合成领域里首个使用卷积神经网络来进行单步逆合成预测的方法,实现了一个端到端的单步逆合成框架,不需要复杂的领域参数设置。
[0101]
2.本发明利用指纹和one-hot编码的融合特征,同已有方法相比,能够提供更多的潜在信息。
[0102]
3.本发明成果既可用于化学逆合成领域,也可用于生物逆合成领域,具有比现有方法更广泛的用途。特别地,进行单步逆合成预测的思路目前在生物逆合成领域尚属空白,本发明方法可以填补该空白。
[0103]
实施例二
[0104]
基于同样的发明构思,本实施例提供了一种基于两路多分支cnn的单步逆合成系统,其特征在于,包括:
[0105]
反应数据集获取模块,用于获取既定的反应数据集r,既定的反应数据集包含不同的反应,每条反应包含底物分子和产物分子,根据既定的反应数据集中反应的产物分子构造分子集合s,根据既定的反应数据集中的反应构造反应规则集t;
[0106]
训练集构造模块,用于根据构造的分子集合、反应规则集以及分子和反应规则之间的对应关系,构造输入数据集d={(s,t)},其中,s表示分子,t表示反应规则,s∈s,t∈t,二元组(s,t)表示反应规则t所对应的反应中存在能产生s的反应;
[0107]
模型构建模块,用于搭建两路多分支cnn,构建单步逆合成预测模型;
[0108]
模型训练模块,用于利用构造的输入数据集d作为训练集训练步骤s3中构建的单步逆合成预测模型,得到训练好的预测模型a;
[0109]
单步逆合成预测模块,用于将待预测的目标分子输入训练好的预测模型a,预测出反应规则集合t中每个反应规则产生该目标分子的概率,根据概率值,选择其中概率最大的前k条规则作为结果输出,k为设定的参数。
[0110]
其中,反应数据集获取模块包括反应数据集构建单元、反应规则集生成单元以及分子集合构造单元。
[0111]
分子处理模块为单步逆合成预测模型的组成部分,包括从反应中提取相关分子的smiles序列、基于smiles序列生成指纹向量、基于smiles序列和字母表生成one-hot。
[0112]
在一种实施方式中,所述系统还包括:结果可视化模块,用于利用图形方式直观显示分子、分子相关的反应、预测得到的反应规则。
[0113]
请参见图4,是本发明实施例中一种基于两路多分支cnn的单步逆合成系统的模块示意图。
[0114]
反应数据集获取模块包括三部分,反应数据集获取单元、分子集合构建单元以及反应规则集生成单元,其中,数据集获取单元,对从公共资源收集的已知反应进行整理后得到的反应数据集合;分子集合构建单元:根据反应数据集中反应的产物分子构造分子集合,反应规则集生成单元:基于反应数据集中所有反应,生成反应规则集;训练集构造模块:生成由(分子,反应规则)组成的训练集合;模型训练模块:对双路多分支cnn进行训练优化,得到单步逆合成预测模型;单步逆合成预测模块:对输入未知的目标分子smiles序列,预测能产生该分子的前k条反应规则,k为事先设定的参数;结果可视化模块:利用图形方式直观显示分子、分子相关的反应、预测的到的反应规则等。
[0115]
总体来说,实施例一的方法在进行单步逆合成预测时,输入待预测分子的smiles序列,经过两路多分支卷积层,特征拼接层,全连接层后,输出反应规则集能产生该分子的前k条反应规则。根据输出的反应规则,结合待预测分子的smiles,最终计算得到目标分子的反应物smiles,实现单步逆合成自动化。本实施例还提出一种基于两路多分支cnn的单步逆合成系统,通过反应数据集获取,训练集构造,模型构建,模型训练,单步逆合成预测以及结果可视化等模块处理过程,实现了目标分子的单步逆合成自动化。本发明是单步逆合成领域里首个使用卷积神经网络来进行单步逆合成预测的方法,实现了一个端到端的单步逆合成框架。本发明成果既可用于化学逆合成领域,也可用于生物逆合成领域,具有比现有方法更广泛的用途。特别地,进行单步逆合成预测的思路目前在生物逆合成领域尚属空白,本发明方法可以填补该空白。
[0116]
由于本发明实施例二所介绍的系统,为实施本发明实施例一中基于两路多分支cnn的单步逆合成方法所采用的系统,故而基于本发明实施例一所介绍的方法,本领域所属技术人员能够了解该系统的具体结构,在此不再赘述。凡是本发明实施例一的方法所采用的系统都属于本发明所欲保护的范围。
[0117]
应当理解的是,上述针对较佳实施例的描述较为详细,并不能因此而认为是对本发明专利保护范围的限制,本领域的普通技术人员在本发明的启示下,在不脱离本发明权利要求所保护的范围情况下,还可以做出替换或变形,均落入本发明的保护范围之内,本发明的请求保护范围应以所附权利要求为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1