一种基因组预测方法、预测系统及应用
1.本发明属于基因组学、生物信息学、基因组预测和基因组育种技术领域,尤其涉及一种基因组预测方法、预测系统及应用。
背景技术:
2.目前,随着高通量测序技术的快速发展,研究人员可以准确、快速的对目标物种进行基因组组装和全基因组测序,从而使得遗传学和基因组学研究进入高速发展时期。高质量的基因组组装和廉价、高效的基因分型技术驱动着研究目标高精确基因型的获得,并由此推动基因组预测的发展。基因组预测是人类复杂性状、疾病风险预测和选择性育种领域中一种高效而有力的工具,它利用表型信息和基因型信息在参考群体中构建基因组预测模型,即可预测仅有基因型信息的候选群体的潜在表现。
3.最经典的基因组预测模型是基因组最佳线性无偏预测(genomic best linear unbiased prediction,gblup),它使用基因型信息构建的基因组g矩阵代替传统最佳线性无偏预测中的系谱a矩阵,避免了由孟德尔抽样造成的误差,因此取得了更好的预测效果,被广泛应用于动植物选择育种中。除了经典的gblup以外,已经有更多的基因组预测模型被开发,例如各种贝叶斯方法。这些基于贝叶斯方法的基因组预测模型已成为基因组预测中使用最广泛的模型,因其更高的预测精度。然而,由于不同物种中不同性状的复杂遗传调控机制,仍然没有一种模型在所有物种和所有性状中都是最优的,最优模型仍应根据具体的物种和性状进行选择。
4.除此以外,广泛使用的基因组预测模型利用大量的全基因组基因型和最多数千个个体进行候选群体的基因组育种值估计,这导致了基因组预测模型中存在严重的过拟合现象,影响着模型的预测性能。尽管人们早就意识到过多的基因型会影响模型的预测性能,但仍然没有很好的方法解决该问题。在各种机器学习中,过度拟合的预防已被广泛提及,但对于基因组预测模型中的过度拟合问题,几乎没有解决方案。以往的基因组预测模型通过不断优化基因型效应值的分布和效应值方差的分布来优化模型的预测性能,但很少有从预防过拟合角度出发提高基因组模型的预测性能。
5.基于上述因素,亟需解决基因组预测模型中由于高维基因型数据引入导致的过拟合问题,从而减轻模型的噪音干扰,提升基因组预测模型的预测性能,并使其更加广泛地应用于疾病风险预测、动植物育种领域。
6.通过上述分析,现有技术存在的问题及缺陷为:
7.(1)传统的基因组预测模型使用上万个基因型构建预测模型,导致了高维数据引入造成的“大p小n”问题,造成了严重的过拟合问题。尽管现有的贝叶斯套索等模型在构建模型中进行参数了优化,并添加了惩罚项,但是仍然没有解决基因组预测中的拟合问题,使得在疾病风险预测、动植物育种中的基因组预测精度较低。
8.(2)现有基因组预测模型构建过程中,需要对整个基因组上的变异进行基因分型,使得基因分型成本较高,导致基因组预测在疾病风险预测和动植物育种中的应用范围受
限,实用性差。
9.解决以上问题及缺陷的难度为:(1)忽略了基因组变异对表型影响的生物学意义,单纯从数据角度出发,对模型进行优化,没有考虑引入与性状无关的基因型信息对模型的负担;(2)过拟合现象在基因组预测领域的研究极少。
10.解决以上问题及缺陷的意义为:使得现有的基因组预测模型的预测精度大幅度提升,并通过对极少的与性状相关的基因型进行分型就可获得高精度的预测准确性,降低了基因分型的成本。
技术实现要素:
11.为克服相关技术中存在的问题,本发明公开实施例提供了一种基因组预测方法、预测系统、可读存储介质及应用。具体涉及一种提高基因组预测模型预测精度的方法。
12.所述技术方案如下:基因组预测方法,其特征在于,应用于信息数据处理终端,所述基因组预测方法包括:对在全部基因型矩阵中效应值相对较低的基因型进行逐步丢弃,仅保留具有大效应值的最优基因型矩阵构建基因组预测模型,利用基因组信息对不同物种的表型进行预测。
13.在一实施例中,所述基因组预测方法具体包括以下步骤:
14.步骤一,对目标物种性状进行表型值测定,获得表型信息,并对具有表型值的个体进行基因分型,获得基因型信息;
15.步骤二,利用基因型信息和表型信息构建基于所有基因型的基因组预测模型;
16.步骤三,依据表型信息和基因型信息的相关关系,利用最大似然估计或者基于吉布斯采样的贝叶斯方法,在构建的基因组预测模型中估计每个基因型的效应值(在构建的模型中依据不同模型对效应值及其方差分布的不同前提假设,求取效应值分布的相应参数,从而估计每个基因型的效应值,具体地,依据表型信息和基因型信息的相关关系,在构建的基因组预测模型中利用最大似然估计或者基于吉布斯采样的贝叶斯方法求取标记效应值所服从的分布的相应参数,从而估计每个基因型的效应值)。
17.步骤四,根据每个基因型效应值的大小对所有的基因型从小到大进行排序,从基因型效应值最小的基因型开始逐渐将所述基因型效应值最小的基因型从基因组预测模型中丢弃;其中,从基因型效应值最小的基因型开始,按照一定的数目(如每次丢弃200个基因型),将其从基因组预测模型中丢弃,从而利用保留的具有较大效应值的基因型构建新的基因组预测模型。
18.步骤五,利用交叉验证的方法,通过计算被掩盖表型值个体的基因组育种值和实际表型值的相关性对每次丢弃效应值较小的基因型后仅保留较大效应值基因型构建的基因组预测模型进行预测精度评估;其中,通过计算被掩盖表型值个体的基因组育种值和实际表型值的皮尔森相关系数或roc曲线下面积,对每次丢弃效应值较小的基因型后仅保留较大效应值基因型构建的基因组预测模型进行预测精度评估。
19.其中,皮尔森相关系数的计算公式为:
[0020][0021]
其中y为被掩盖的表型值,gebv为基因组育种值;roc曲线下面积的计算公式为:
[0022][0023][0024]
其中n1为存活个体数,表型值记录为1,n0为死亡个体数,表型值记录为0,r1为存活个体的排序之和。
[0025]
步骤六,所有基因型都从基因组预测模型中丢弃后,结束基因组预测模型的预测精度评估,根据基因组预测模型的最高预测精度出现的基因型数目选择相应的基因型矩阵构建基因组预测模型,即获得最高的基因组预测数据信息。
[0026]
本发明的该基因组预测模型是基于丢弃较小效应值标记后不断迭代而获得的最优基因型矩阵得到的,显著的降低了高维数据引入造成的过拟合问题。
[0027]
在一实施例中,所述步骤一基因型信息为基于单核苷酸多态性分型的基因型信息,只保留具有二等位位点的基因型,并由0,1,2所编码,分别代表纯合等位基因型aa,杂合等位基因型aa,以及次要纯合等位基因型aa,并且用于构建基因组预测模型的基因型信息不存在缺失。
[0028]
在一实施例中,所述步骤二基因组预测模型构建采用间接法使用所有的基因型数据构建,构建的基因组预测模型均为基于加性效应的线性模型;
[0029]
线性模型公式表示为y=μ+zu+e;
[0030]
其中y为个体的表型值,μ为截距,z为n行m列的基因型矩阵,其中n为构建基因组模型用到的样本数目,m为基因型数目,u为m行1列的效应值矩阵,第i行对应z矩阵中第i列基因型的效应值;e为残差;
[0031]
所有基因型与其效应值乘积的总和即为基因组育种值。
[0032]
在一实施例中,所述步骤三基因型的效应值估计在所有基因型数据构建的基因组预测模型中进行,该基因组预测模型为每个基因型生成效应值;
[0033]
所述步骤四效应值排序根据每个基因型效应值的绝对值大小由小到大进行排序。
[0034]
在一实施例中,所述步骤五交叉验证的方法包括:通过掩盖部分个体的表型信息,仅保留基因型信息,利用基因组预测模型估计被掩盖表型个体基因组育种值,并且与实际表型进行相关性比较,获得基因组预测模型的预测信息;
[0035]
所述步骤六使用的基因型矩阵通过逐次丢弃效应值较小的基因型进行不断迭代获得;
[0036]
基因组预测模型基于丢弃较小效应值标记所确定的具有最高预测准确度的最优基因型矩阵构建。
[0037]
本发明的另一目的在于提供一种基因组预测系统包括信息数据处理终端,所述信息数据处理终端包括存储器和处理器,所述存储器存储有计算机程序,所述计算机程序被所述处理器执行时,使得所述处理器执行如下步骤:
[0038]
对在全部基因型矩阵中效应值相对较低的基因型进行逐步丢弃,仅保留具有大效应值的最优基因型矩阵构建基因组预测模型,对不同物种的表型和基因型数据进行预测。
[0039]
本发明的另一目的在于提供一种所述基因组预测方法在高粱、奶牛、马、虹鳟和鲤鱼不同物种的表型和基因型数据预测上的应用。
[0040]
结合上述的所有技术方案,本发明所具备的优点及积极效果为:
[0041]
本发明通过逐渐丢弃基因组预测模型中效应值较低的基因型,仅保留具有大效应的基因型,降低了由高维基因型数据引入模型所导致的过拟合损害。相比于现在普遍利用所有基因型数据构建基因组预测模型而言,本发明显著地提高了基因组预测的准确性,并且仅对具有大效应的基因型进行分型即可获得最高的预测精度,降低了基因分型的成本,使得基因组预测在人类疾病风险预测和动植物育种中的应用范围更加广泛。
[0042]
在本发明中,“所有基因型”即为传统的利用所有基因型构建基因组预测模型,进行基因组预测的预测精度,而“最优基因型”即为本发明相比于传统的利用所有基因型构建基因组预测模型的预测提升情况,在所测试的5个物种的12个性状中,本发明均表现出显著地预测精度提升效果。
[0043]
还有,本发明显著地提升了现有基因组预测模型的预测精度,并且只需要对具有大效应的基因型进行分型即可满足需求,并且预测精度显著高于使用所有基因型构建的预测模型,使得预测精度和大规模样本分型的适用性较之以往的预测模型都有了显著的提升。
[0044]
本发明所获得的最优基因型矩阵不仅可以用于基于加性效应的各种传统的基因组预测模型,还能用于如随机森林,支持向量机等各种基于机器学习算法的预测模型。
[0045]
当理解的是,以上的一般描述和后文的细节描述仅是示例性和解释性的,并不能限制本发明的公开。
附图说明
[0046]
此处的附图被并入说明书中并构成本说明书的一部分,示出了符合本公开的实施例,并与说明书一起用于解释本公开的原理。
[0047]
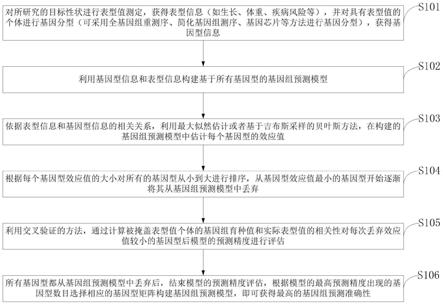
图1是本发明实施例提供的基因组预测方法流程图。
[0048]
图2是本发明实施例提供的最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在高粱数据集中的预测精度提升图;其中,图2(a)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在高粱秆长性状中的预测精度提升图;图2(b)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在高粱秆茎性状中的预测精度提升图;图2(c)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在高粱总重性状中的预测精度提升图;图2(d)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在高粱秆数性状中的预测精度提升图。
[0049]
图3是本发明实施例提供的最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在奶牛和马的数据集中的预测精度提升图;其中,图3(a)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在奶牛乳脂率性状中的预测精度提升图;图3(b)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在奶牛体细胞评分性状中的预测精度提升图;图3(c)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在奶牛产奶量性状中的预测精度提升图;图3(d)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在马毛色性状中的预测精度提升图。
[0050]
图4是本发明实施例提供的最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在虹鳟和鲤鱼的数据集中的预测精度提升图;其中,图4(a)为最优基因型矩
阵相对于使用所有基因型矩阵构建的基因组预测模型在虹鳟存活状态性状中的预测精度提升图;图4(b)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在虹鳟存活时间性状中的预测精度提升图;图4(c)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在鲤鱼体重性状中的预测精度提升图;图4(d)为最优基因型矩阵相对于使用所有基因型矩阵构建的基因组预测模型在鲤鱼体长性状中的预测精度提升图。
具体实施方式
[0051]
为使本发明的上述目的、特征和优点能够更加明显易懂,下面结合附图对本发明的具体实施方式做详细的说明。在下面的描述中阐述了很多具体细节以便于充分理解本发明。但是本发明能够以很多不同于在此描述的其它方式来实施,本领域技术人员可以在不违背本发明内涵的情况下做类似改进,因此本发明不受下面公开的具体实施的限制。
[0052]
本发明提供一种解决现有基因组预测模型中由于高维基因型数据引入造成过拟合问题的方法,即基因组预测方法,包括:对效应值较低的基因型进行逐步丢弃,仅保留具有大效应值的最优基因型矩阵用于构建基因组预测模型,可获得最高的预测精度。相比于现在常用的利用所有基因型信息构建模型并进行基因组预测,本发明显著地提高了基因组预测模型的预测性能,并且仅对最优的基因型矩阵进行分型即可获得最高的预测精度,降低了基因分型的成本。
[0053]
下面结合具体实施例对本发明的技术方案作进一步描述。
[0054]
实施例1:
[0055]
如图1所示,本发明提供一种基因组预测方法包括:
[0056]
s101,对所研究的目标性状进行表型值测定,获得表型信息(如生长、体重、疾病风险等),并对具有表型值的个体进行基因分型(可采用全基因组重测序、简化基因组测序、基因芯片等方法进行基因分型),获得基因型信息。
[0057]
s102,利用基因型信息和表型信息构建基于所有基因型的基因组预测模型。
[0058]
s103,依据表型信息和基因型信息的相关关系,利用最大似然估计或者基于吉布斯采样的贝叶斯方法,在构建的基因组预测模型中估计每个基因型的效应值。
[0059]
s104,根据每个基因型效应值的大小对所有的基因型从小到大进行排序,从基因型效应值最小的基因型开始逐渐将其从基因组预测模型中丢弃。
[0060]
s105,利用交叉验证的方法,通过计算被掩盖表型值个体的基因组育种值和实际表型值的相关性对每次丢弃效应值较小的基因型后模型的预测精度进行评估。
[0061]
s106,所有基因型都从基因组预测模型中丢弃后,结束模型的预测精度评估,根据模型的最高预测精度出现的基因型数目选择相应的基因型矩阵构建基因组预测模型,即可获得最高的基因组预测准确性。
[0062]
在本发明一优选实施例中,所述步骤s101基因型信息为基于单核苷酸多态性分型的基因型信息,只保留具有二等位位点的基因型,并由0,1,2所编码,分别代表主要纯合等位基因型aa,杂合等位基因型aa,以及次要纯合等位基因型aa,并且用于构建基因组预测模型的基因型信息不能存在缺失。
[0063]
在本发明一优选实施例中,所述步骤s102基因组预测模型构建采用间接法使用所有的基因型数据构建,构建的基因组预测模型均为基于加性效应的线性模型;
[0064]
线性模型公式表示为y=μ+zu+e;
[0065]
其中y为个体的表型值,μ为截距,z为n行m列的基因型矩阵,其中n为构建基因组模型用到的样本数目,m为基因型数目,u为m行1列的效应值矩阵第i行对应z矩阵中第i列基因型的效应值;e为残差;
[0066]
所有基因型与其效应值乘积的总和即为基因组育种值
[0067]
在本发明一优选实施例中,所述步骤s103在构建的模型中依据不同模型对效应值及其方差分布的不同前提假设,求取效应值分布的相应参数,从而估计每个基因型的效应值。
[0068]
在本发明一优选实施例中,所述步骤s103效应值估计是在所有基因型数据构建的基因组预测模型中完成的,该模型为每个基因型生成效应值。
[0069]
在本发明一优选实施例中,所述步骤s104效应值排序是根据每个基因型效应值的绝对值大小由小到大进行排序的,而不是数值大小。
[0070]
在本发明一优选实施例中,所述步骤s105基因组预测模型的预测精度是采用交叉验证的方法计算得到的,通过掩盖部分个体的表型信息,仅保留基因型信息,利用基因组预测模型估计被掩盖表型个体基因组育种值,并且与实际表型进行相关性比较,从而获得模型的预测准确性。二元性状预测准确性的度量为基因组育种值和实际表型值的roc曲线下面积,非二元性状的预测准确性度量为基因组育种值和实际表型值的皮尔森相关系数,数值越接近1表明预测准确性越高。
[0071]
其中,皮尔森相关系数的计算公式为:
[0072][0073]
其中y为被掩盖的表型值,gebv为基因组育种值;roc曲线下面积的计算公式为:
[0074][0075][0076]
其中n1为存活个体数,表型值记录为1,n0为死亡个体数,表型值记录为0,r1为存活个体的排序之和。
[0077]
在本发明一优选实施例中,基因组预测模型的最高预测精度所使用的基因型矩阵是通过逐次丢弃效应值较小的基因型不断迭代得到的。
[0078]
在本发明一优选实施例中,通过计算被掩盖表型值个体的基因组育种值和实际表型值的皮尔森相关系数或roc曲线下面积,对每次丢弃效应值较小基因型后构建模型的预测精度进行评估。
[0079]
在本发明一优选实施例中,所述步骤s106最终用于基因组预测的模型是基于丢弃较小效应值标记所确定的具有最高预测准确度的最优基因型矩阵所构建的,而非现在常用的使用所有基因型构建的基因组预测模型。
[0080]
实施例2:
[0081]
本发明提供一种基因组预测方法(提高基因组预测模型预测精度的方法),包括以下步骤:
[0082]
(1)不同物种的表型和基因型数据收集,包括高粱、奶牛、马、虹鳟和鲤鱼。
[0083]
高粱的基因型数据包括3260个基因型和1020个样本,通过限制性酶切位点相关dna测序进行基因分型,表型数据为秆长(cl)、总重(tw)、秆径(cd)和秆数(cn)。牛的数据包括42551个基因型和5024个样本,通过50ksnp芯片进行基因分型。表型数据为乳脂百分比(fp)、体细胞评分(scs)和产奶量(my)。马的数据包括50621个基因型和480个样本,通过54ksnp芯片进行基因分型,表型数据为毛色(cc)。虹鳟的数据包括1934个样本和27490个基因型,通过57ksnp芯片进行基因分型,表型数据为鲑鱼立克次氏体攻毒后的存活时间(sd)和存活状态(ss),存活状态为代表每个个体在攻毒试验后死亡或存活的二元性状,记录为0或1。鲤鱼的数据包括1259个样本和15615个基因型,通过限制性酶切位点相关dna测序进行基因分型,表型为标准长度(sl)和体重(bw)。上述数据从已经发表的文章中获得,来源分别为高粱(ishimori m,takanashi h,hamazaki k et al.dissecting the genetic architecture of biofuel-related traits in a sorghum breeding population.g3 genes|genomes|genetics 2020;10:4565-4577),奶牛(accuracy of whole-genome prediction using a genetic architecture-enhanced variance-covariance matrix.g3 genes|genomes|genetics 2015;5:615-627),马(kaml:improving genomic prediction accuracy of complex traits using machine learning determined parameters.genome biology 2020;21:146),虹鳟(yoshida gm,bangera r,carvalheiro r et al.genomic prediction accuracy for resistance against piscirickettsia salmonis in farmed rainbow trout.g3 genes|genomes|genetics 2018;8:719-726),鲤鱼(palaiokostas c,vesely t,kocour m et al.optimizing genomic prediction of host resistance to koi herpesvirus disease in carp.frontiers in genetics 2019;10:543)。
[0084]
(2)基因组预测模型构建
[0085]
在r中导入全部的基因型数据和表型数据,并构建基因组预测模型。基因组预测模型是使用r包bglr和rrblup构建的,分别对应于贝叶斯方法和岭回归最佳线性无偏预测方法所构建的基因组预测模型。
[0086]
(3)基因型效应值估计及排序
[0087]
利用构建的基因组预测模型为每个基因型生成效应值,然后根据效应值的绝对值大小进行排序。rrblup方法使用最大似然估计为每个基因型生成效应值,而各种贝叶斯方法通过吉布斯采样构建马尔可夫链蒙特卡罗链,从每个基因型参数的后验分布中抽样,从而计算每个基因型的效应值。根据效应值的大小,利用r中的order函数,按照基因型效应值由小到大的顺序对基因型进行排序。
[0088]
(4)最高预测精度的基因型矩阵确定
[0089]
根据基因型效应值的排序大小,逐渐将其从基因组预测模型中丢弃,每次丢弃数目定为200个基因型。每丢弃200个基因型后,对现有基因型矩阵构建的基因组预测模型实施五重交叉验证,从而评估其预测准确性,直至基因组预测模型中不再含有基因型数据。五重交叉验证实施的具体细节为:将全部的基因型数据和表型数据随机分为5个子集,其中一个子集的表型信息被掩盖,仅保留基因型信息,使用其他4份子集构建基因组预测模型并计算基因型的效应值,将基因型的效应值矩阵与掩盖表型子集的基因型数据进行矩阵乘法,
为每个个体生成基因组育种值,如果表型数据为连续性状,则用基因组育种值和表型的皮尔森相关系数代表准确性,如果是二元性状,则采用roc曲线下面积作为准确性。为了防止实施五重交叉验证时抽样造成的误差,所有的预测精度评估均执行50次,50次重复的均值和标准差作为最终结果。
[0090]
(5)为了验证本发明的有效性,利用(1)中的所有数据集对本发明进行了验证。图2为本发明在高粱的4个表型性状中的提升情况,相比于使用所有基因型矩阵构建的基因组预测模型,使用最优的基因型矩阵构建的模型的精确度显著提升,并且在4个性状中均适用。图3为在牛和马的数据集中的基因组预测模型的预测准确性提升情况,图4为虹鳟和鲤鱼的基因组预测模型的预测准确性提升情况。所测试的5个物种的12个性状均表现了基于丢弃小效应基因型而构建的最优基因组预测模型比使用所有基因型构建的基因组预测模型具有更高的预测精确度,表明了该方法在不同性状、不同物种中均适用。
[0091]
本领域技术人员在考虑说明书及实践这里公开的公开后,将容易想到本公开的其它实施方案。本技术旨在涵盖本公开的任何变型、用途或者适应性变化,这些变型、用途或者适应性变化遵循本公开的一般性原理并包括本公开未公开的本技术领域中的公知常识或惯用技术手段。说明书和实施例仅被视为示例性的,本公开的真正范围和精神由所附的权利要求指出。
[0092]
应当理解的是,本公开并不局限于上面已经描述并在附图中示出的精确结构,并且可以在不脱离其范围进行各种修改和改变。本公开的范围应由所附的权利要求来限制。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1