增强采样方法及计算复合物的结合自由能的方法与流程
1.本技术涉及药物研发技术领域,特别是涉及一种增强采样方法、计算目标分子-蛋白质复合物的结合自由能的方法、增强采样装置、计算目标分子-蛋白质复合物的结合自由能的装置、电子设备以及计算机可读存储介质。
背景技术:
2.药物分子与蛋白质之间的结合自由能作为一种评价药物分子活性的指标,可以能够通过多种计算方法进行预测。其中,自由能微扰(free energy perturbation,fep)是一种高精度的计算化学预测方法,已经被广泛应用于药物设计当中。但是,fep作为一种基于分子动力学模拟的方法,在模拟过程中往往面临着采样的问题。对于采样不充分的体系,得到的结合自由能并不能很好的收敛,极大的影响到预测结果的准确性。
3.为了解决这一问题,现有技术中存在各种增强采样的方法。其中,溶质回火副本交换(replica exchange with solute tempering,rest2)是一种计算化学中的增强采样方法,设计之初主要被用于蛋白折叠体系的模拟。rest2方法通过局部(hot region)升温的思想,在保证交换率的同时能够减少所需交换副本数量,实现高效可靠的增强采样。很多研究已经证明,使用rest2方法对于fep进行增强采样(以下缩写为fep/rest2),能够明显提高fep的计算精度。但是,本技术的发明人在长期研究中发现,在使用fep/rest2时面临着两个问题:1、在fep中使用rest2方法涉及到分子动力学软件底层代码的修改,实现难度较大;2、fep/rest2方法整体流程复杂,使用难度较大。
技术实现要素:
4.本技术主要的目的是提供一种增强采样方法、计算目标分子-蛋白质复合物的结合自由能的方法、增强采样装置、计算目标分子-蛋白质复合物的结合自由能的装置、电子设备以及计算机可读存储介质,以解决现有技术中在fep中使用rest2方法实现难度较大且使用难度较大的问题。
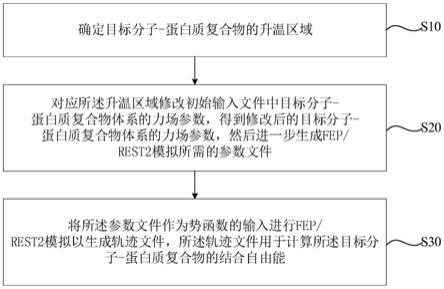
5.本技术实施例提供的一种增强采样方法,包括:
6.确定目标分子-蛋白质复合物的升温区域;
7.对应所述升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,得到修改后的目标分子-蛋白质复合物体系的力场参数,然后进一步生成fep/rest2模拟所需的参数文件;
8.将所述参数文件作为势函数的输入进行fep/rest2模拟以生成轨迹文件,所述轨迹文件用于计算所述目标分子-蛋白质复合物的结合自由能。
9.本技术实施例提供的一种计算目标分子-蛋白质复合物的结合自由能的方法,包括:
10.构建目标分子-蛋白质复合物体系的初始输入文件;
11.将所述初始输入文件输入至如上述实施例所述的增强采样方法,获得轨迹文件;
12.基于所述轨迹文件获得目标分子-蛋白质复合物的结合自由能。
13.本技术实施例提供的一种增强采样装置,包括:
14.升温区域确定模块,用于确定目标分子-蛋白质复合物的升温区域;
15.力场参数修改模块,用于对应所述升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,得到修改后的目标分子-蛋白质复合物体系的力场参数,然后进一步生成fep/rest2模拟所需的参数文件;和
16.轨迹文件生成模块,用于将所述参数文件作为势函数的输入进行fep/rest2模拟以生成轨迹文件,所述轨迹文件用于计算所述目标分子-蛋白质复合物的结合自由能。
17.本技术实施例提供的一种计算目标分子-蛋白质复合物的结合自由能的装置,包括:
18.构建模块,用于构建目标分子-蛋白质复合物体系的初始输入文件;
19.如上述实施例所述的增强采样装置,用于基于输入的所述初始输入文件,获得轨迹文件;和
20.计算模块,用于基于所述轨迹文件获得目标分子-蛋白质复合物的结合自由能。
21.本技术实施例提供的一种电子设备,包括:
22.一个或多个处理器;
23.存储器,与所述处理器耦接,用于存储一个或多个程序;
24.当所述一个或多个程序被所述一个或多个处理器执行,使得所述一个或多个处理器实现如上述实施例所述的增强采样方法或上述实施例所述的计算目标分子-蛋白质复合物的结合自由能的方法。
25.本技术实施例提供的一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现如任一实施例所述的增强采样方法或上述实施例所述的计算目标分子-蛋白质复合物的结合自由能的方法。
26.相较于现有技术,本技术的增强采样方法、增强采样方法、计算目标分子-蛋白质复合物的结合自由能的方法、电子设备以及计算机可读存储介质具有如下有益效果:
27.由于在分子动力学模拟中,目标分子-蛋白质复合物体系的能量可以通过势函数进行计算,因此本技术通过确定目标分子-蛋白质复合物的升温区域,然后对应升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,即修改势函数内的各类参数,以生成fep/rest2模拟所需的参数文件,从而以尽量少修改代码的方式快速便捷地实现rest2方法的流程,而不需要大幅修改分子动力学模拟软件的底层代码,操作简单,提高后续fep的计算效率及预测精度。
附图说明
28.本技术将结合附图对实施方式进行说明。本技术的附图仅用于描述实施例,以展示为目的。在不偏离本技术原理的条件下,本领域技术人员能够轻松地通过以下描述根据所述步骤做出其他实施例。
29.图1为本技术实施例中一种增强采样方法的流程示意图。
30.图2为本技术实施例中步骤s10中确定升温区域的具体流程示意图。
31.图3为本技术实施例中步骤s20中对应所述升温区域修改所述目标分子-蛋白质复
合物体系的力场参数的具体流程示意图。
32.图4为本技术实施例中一种计算目标分子-蛋白质复合物的结合自由能的方法的流程示意图。
33.图5为本技术实施例中利用相对自由能计算不同分子之间的自由能差异的基本原理示意图。
34.图6(a)为未使用rest2增强采样时的fep计算结果示意图。
35.图6(b)为使用本技术rest2增强采样时的fep计算结果示意图。
36.图7为本技术实施例中一种增强采样装置的结构示意图。
37.图8为本技术实施例中一种计算目标分子-蛋白质复合物的结合自由能的装置的结构示意图。
38.图9为本技术实施例中一种电子设备的结构示意图。
具体实施方式
39.下面将结合本技术实施例中的附图,对本技术实施例中的技术方案进行清楚、完整地描述。可以理解的是,此处所描述的具体实施例仅用于解释本技术,而非对本技术的限定。另外还需要说明的是,为了便于描述,附图中仅示出了与本技术相关的部分而非全部结构。基于本技术中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本技术保护的范围。
40.本技术中的术语“第一”、“第二”等是用于区别不同对象,而不是用于描述特定顺序。此外,术语“包括”和“具有”以及它们任何变形,意图在于覆盖不排他的包含。例如包含了一系列步骤或单元的过程、方法、系统、产品或设备没有限定于已列出的步骤或单元,而是可选地还包括没有列出的步骤或单元,或可选地还包括对于这些过程、方法、产品或设备固有的其它步骤或单元。
41.在本文中提及“实施例”意味着,结合实施例描述的特定特征、结构或特性可以包含在本技术的至少一个实施例中。在说明书中的各个位置出现该短语并不一定均是指相同的实施例,也不是与其它实施例互斥的独立的或备选的实施例。本领域技术人员显式地和隐式地理解的是,本文所描述的实施例可以与其它实施例相结合。
42.请参阅图1,本发明实施例提供一种增强采样方法,该方法应用在常用分子模拟软件中,以尽量少修改代码的方式快速便捷地实现rest2方法的流程。其中,常用分子模拟软件包括但不限于amber程序、gromacs程序、namd程序。
43.具体的,本技术的增强采样方法包括以下步骤:
44.s10、确定目标分子-蛋白质复合物的升温区域。
45.在本实施例中,目标分子可以为药物候选化合物。
46.自由能微扰计算(fep)是评估药物小分子和靶点结合强度的一种方法。fep通过在结合态与非结合态之间构建一系列非物理存在的中间态,从而增强采样的可靠性。在计算fep时,采用在输入文件里修改lambda的方式进行微扰。在本实施例中,根据一般的fep计算过程中的微扰区域,确定目标分子-蛋白质复合物的升温区域,从而对待检测的目标分子-蛋白质复合物实现局部升温,在保证交换率的同时能够减少所需交换副本数量,实现高效可靠的增强采样。
47.在一个具体实施例中,如图2所示,上述步骤s10包括以下步骤:
48.将fep计算过程中的微扰区域确定为目标分子-蛋白质复合物的初始升温区域。
49.本实施例将一般的fep计算过程中的微扰区域作为初始升温区域。
50.在确定初始升温区域后,针对目标分子的结构进行分析,判断与微扰区域直接相连的原子是否位于目标分子内的环上。如果位于环上,则确定微扰区域直接与环相连,如果没有位于环上,则确定微扰区域没有与环相连。如果确定与环相连,将该环添加至升温区域。如果确定并未与环相连,则不做处理。本实施例通过将额外的环增加至升温区域,可以帮助部分体系更快地收敛。
51.具体的,对目标分子的结构进行分析,以调整升温区域的步骤如下:
52.判断所述目标分子中是否有环;
53.若所述目标分子中有环,则进一步判断所述初始升温区域中的至少部分原子是否位于所述环上,或所述初始升温区域中的至少部分原子是否与所述环上的原子直接或间接相连;
54.若所述初始升温区域中的至少部分原子位于所述环上或与所述环上的原子直接相连,则将所述环所在的区域添加至所述的初始升温区域,得到第一更新升温区域;
55.若所述初始升温区域中的至少部分原子与所述环上的原子间接相连,则将所述环及与所述环间接相连部分所在的区域添加至所述的初始升温区域,得到第一更新升温区域;
56.若所述初始升温区域中的所有原子既不位于所述环上,也不与所述环上的原子直接或间接相连,则将所述初始升温区域定为第一更新升温区域;
57.若所述目标分子中无环,则将所述初始升温区域定为第一更新升温区域;
58.判断是否存在额外指定的需要升温的区域;
59.若存在,则将所述额外指定的需要升温的区域添加至所述第一更新升温区域,得升温区域;
60.若不存在,则将所述第一更新升温区域定为升温区域。
61.如此,根据目标分子的结构更新初始升温区域,从而获得第一更新升温区域,并在获得第一更新升温区域后,进一步判断是否存在额外指定的需要升温的区域,如果存在,则将其添加至第一升温区域,得升温区域,如果不存在,则不添加,将第一更新升温区域定为升温区域,从而实现了升温区域的自动选择。
62.s20、对应所述升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,得到修改后的目标分子-蛋白质复合物体系的力场参数,然后进一步生成fep/rest2模拟所需的参数文件。
63.fep/rest2指使用rest2方法对于fep进行增强采样。由于在分子动力学模拟中,目标分子-蛋白质复合物体系的能量可以通过势函数进行计算,因此在对升温区域进行rest2计算时,可以通过修改势函数内的各类参数来实现这一过程,而不需要修改分子动力学模拟软件的底层代码。
64.在确定升温区域后,进一步确定目标分子-蛋白质复合物体系需要修改的力场参数,以便对应修改。具体的,上述步骤s20,包括以下步骤:
65.将所述目标分子-蛋白质复合物体系的力场参数项划分为所述升温区域内部的参
数、所述升温区域与环境区域间的参数以及所述环境区域内部的参数;
66.至少部分修改所述升温区域内部的参数、所述升温区域与所述环境区域间的参数以及所述环境区域内部的参数,得到修改后的目标分子-蛋白质复合物体系的力场参数,然后进一步生成所述fep/rest2模拟所需的参数文件。
67.本技术在已经得到升温区域的基础上可以把目标分子-蛋白质复合物体系分为两部分,升温区域及升温区域以外的环境区域,从而可以把rest2下的目标分子-蛋白质复合物体系总能量u
mrest2
分为三部分(如下式5所示):升温区域内部的能量uc,c、升温区域与环境区域间的能量uc,e以及环境区域内部的能量ue,e,rest2基本公式为:
[0068][0069][0070]
其中,为使用rest2时目标分子-蛋白质复合物体系总能量,uc,c升温区域内部的能量,uc,e为升温区域与环境区域间的能量,ue,e为环境区域内部的能量,kb为波尔兹曼常数,t0为环境温度,一般为298k,tm为升温区域的温度,可自行选择温度。
[0071]
在分子动力学模拟中,上述各能量u可以通过势函数(如下式7所示)进行计算,基本势函数如下所示,计算过程中各参数及各变量均有分子动力学模拟软件提供计算。如此,可分别计算出升温区域内部的能量uc,c、升温区域与环境区域间的能量uc,e以及环境区域内部的能量ue,e,进而计算出目标分子-蛋白质复合物体系总能量
[0072][0073][0074]
其中,为键连参数,包括分别为键长系数、角度系数及二面角系数;∈i、∈j分别为第i个原子和第j个原子的范德华势阱深度参数,σ
ij
为范德华参数;qi、qj为第i个原子和第j个原子的静电荷参数;ri、θi、φi分别为第i个原子的键长参数、角度参数及二面角参数,r
ij
为第i个原子与第j个原子间的距离;r0、θ0、δi、ni分别为第i个原子的键长参数、角度参数、二面角参数及中间态i采样所包含的样本量。
[0075]
如图3所示,按照确定的升温区域,将参数k划分为不同的类型,包括升温区域内部的参数、升温区域与环境区域间的参数及环境区域内部的参数。同时,确定需要修改的力场参数,包括键长、键角、二面角、范德华及静电参数等。
[0076]
根据上述势函数的计算公式可得,对升温区域进行rest2计算时,可以通过修改势函数内的各类参数来实现这一过程,而不需要修改模拟软件的底层代码。
[0077]
具体的,上述步骤s22中至少部分修改所述升温区域内部的参数、所述升温区域与
所述环境区域间的参数以及所述环境区域内部的参数,包括以下步骤:
[0078]
将所述升温区域内部的参数乘以第一类参数;
[0079]
将所述升温区域与所述环境区域间的参数乘以第二类参数;
[0080]
对所述环境区域内部的参数不做修改。
[0081]
可选的,所述第一类参数包括键连系数、范德华系数及静电系数,所述第二类参数包括键连系数。
[0082]
在本实施例中,针对位于升温区域内部的参数(包括键连参数、范德华参数及静电参数),分别乘以对应的第一类参数,包括键连系数、范德华系数及静电系数等。
[0083]
具体的,如图3所示,
[0084]
针对位于升温区域与环境区域间的参数(键连参数),乘以对应第二类参数进行修改,所述第二类参数为升温区域与环境区域两者之间的键连系数。
[0085]
需要说明的是,本实施例中的键连参数为与用于连接原子的化学键相关的参数,例如键长系数、键角系数、二面角系数等。
[0086]
具体的,如图3所示,
[0087]
而对于环境区域内部的参数则不做修改。
[0088]
s30、将所述参数文件作为势函数的输入进行fep/rest2模拟以生成轨迹文件,所述轨迹文件用于计算所述目标分子-蛋白质复合物的结合自由能。
[0089]
最后,将修改过后的参数文件势函数的输入进行fep/rest2模拟。当进行fep/rest2模拟生成轨迹文件后,轨迹文件可以用于计算目标分子-蛋白质复合物的结合自由能。
[0090]
综上,本技术的增强采样方法具有如下有益效果:
[0091]
由于在分子动力学模拟中,目标分子-蛋白质复合物体系的能量可以通过势函数进行计算,因此本技术通过确定目标分子-蛋白质复合物的升温区域,然后对应升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,即修改势函数内的各类参数,以生成fep/rest2模拟所需的参数文件,从而以尽量少修改代码的方式快速便捷地实现rest2方法的流程,而不需要大幅修改分子动力学模拟软件的底层代码,操作简单,提高后续fep的计算效率及预测精度。
[0092]
请参阅图4,本发明实施例还提供完整的结合自由能计算方法。具体的,本技术的一种计算目标分子-蛋白质复合物的结合自由能的方法,包括以下步骤:
[0093]
s101、构建目标分子-蛋白质复合物体系的初始输入文件。
[0094]
其中,初始输入文件含目标分子-蛋白质复合物的三维构象的文件,具体可为pdb格式的文件。
[0095]
s102、将所述初始输入文件输入至如上述任意一个实施例所述的增强采样方法,获得轨迹文件。
[0096]
s103、基于所述轨迹文件获得目标分子-蛋白质复合物的结合自由能。
[0097]
在本实施例中,在获得轨迹文件后,解析所述轨迹文件,并将解析得到的所述目标
分子-蛋白质复合物体系在不同状态下的分子动力学轨迹。
[0098]
在一个实施例中,在所述步骤s101构建目标分子-蛋白质复合物体系的初始输入文件之前,还包括以下步骤:
[0099]
选择已知小分子-蛋白质复合物构象的小分子为参考化合物;
[0100]
确定参考化合物-蛋白质复合物的升温区域;
[0101]
对应的,所述步骤s20对应所述升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,得到修改后的目标分子-蛋白质复合物体系的力场参数,然后进一步生成fep/rest2模拟所需的参数文件,包括以下步骤:
[0102]
对应所述升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,得到修改后的目标分子-蛋白质复合物体系的力场参数;
[0103]
对应所述升温区域修改初始输入文件中参考化合物-蛋白质复合物体系的力场参数,得到修改后的参考化合物-蛋白质复合物体系的力场参数;
[0104]
基于修改后的参考化合物-蛋白质复合物体系的力场参数,和修改后的目标分子-蛋白质复合物体系的力场参数,生成fep/rest2模拟所需的参数文件。
[0105]
进一步地,所述步骤s103基于所述轨迹文件获得目标分子-蛋白质复合物的结合自由能为:基于所述轨迹文件,采用本内特接受率方法进行计算,获得所述目标分子-蛋白质复合物的结合自由能。
[0106]
因为进行fep/rest2计算时,把fep计算过程中各个态(alchemical state)的参数进行了修改,所以不能使用常见的积分方法进行分析,因此本实施例使用本内特接受率(bennett acceptance ratio,bar)方法计算目标分子-蛋白质复合物的结合自由能,具体计算方法如下公式所示。其中,可以通过重加权(reweighting)的方法,计算得到的轨迹文件在其他状态参数文件下能量的差异计算δu,通过公式最终得到两个态之间的自由能差异δg。
[0107]
具体的,上述步骤s103,进一步包括以下步骤:
[0108]
根据所述轨迹文件中两相邻的中间态i及中间态j,分别计算中间态i轨迹在中间态j对应参数下的第一能量δu
ij
,以及中间态j轨迹在中间态i对应参数下的第二能量δu
ji
;
[0109]
将所述第一能量δu
ij
和所述第二能量δu
ji
带入式1和式2,分别计算出参考化合物变换成目标分子的溶剂自由能差异δga,和参考化合物变换成目标分子的结合自由能差异δgb;
[0110][0111][0112]
基于参考化合物变换成目标分子的溶剂自由能差异δga,和参考化合物变换成目标分子的结合自由能差异δgb,按式3计算获得参考化合物变换成目标分子的相对结合自由能δδg
binding
;
[0113]
δδg
binding
=δg
b-δgaꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
式3;
[0114]
基于参考化合物变换成目标分子的相对结合自由能δδg
binding
,和已知的参考化合物-蛋白质复合物的结合自由能δg1,按照式4计算获得目标分子-蛋白质复合物的结合自由能δg2;
[0115]
δg2=δδg
binding
+δg1ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
式4;
[0116]
其中,δg为自由能差异,δu
ij
为所述第一能量,δu
ji
为所述第二能量,《*》i为中间态i下的系统平均,《*》j为中间态j下的系统平均,ni、ni为中间态i、j下的模拟轨迹的帧数,kb为波尔兹曼常数,t为模拟温度,一般为298k,δδg
binding
为相对结合自由能,δga为参考化合物变换成目标分子的溶剂自由能差异,δgb为参考化合物变换成目标分子的结合自由能差异,δg1为已知的参考化合物-蛋白质复合物的结合自由能,δg2为目标分子-蛋白质复合物的结合自由能。
[0117]
在本实施例中,利用相对自由能(relative binding free energy,rbfe)计算不同分子之间的自由能差异δδg
binding
,从而来计算目标分子-蛋白质复合物的结合自由能。
[0118]
如图5所示,图5为利用相对自由能(relative binding free energy,rbfe)计算不同分子之间的自由能差异δδg
binding
的基本原理示意图。其中,左上图表示参考化合物a与蛋白质受体分离示意图,左下图表示参考化合物a与蛋白质受体形成复合物结构a,δg1为参考化合物a与蛋白质受体形成复合物结构a的结合自由能,右上图表示目标分子b与蛋白质受体分离示意图,右下图表示目标分子b与蛋白质受体形成复合物结构b,δg2为目标分子b与蛋白质受体形成复合物结构b的结合自由能,δga为蛋白质受体将参考化合物a变换成目标分子b的溶剂结合自由能,δgb为参考化合物a变换成目标分子b的结合自由能差异。
[0119]
其中,δδg
binding
=δg
2-δg1=δg
b-δga。
[0120]
为了计算δδg
binding
,δg
2-δg1计算起来相对困难,而横向相邻的两个复合物体系差别很小,因此相对容易达到平衡,在实际计算的时候,就容易实现,因而可以计算相对简单的δg
b-δga。具体的,通过上述计算目标分子-蛋白质复合物的结合自由能的方法来计算待预测目标分子的参考化合物变换成目标分子的结合自由能差异δgb和参考化合物变换成目标分子的溶剂自由能δga,由于选择了已知小分子-蛋白质复合物构象的小分子作为参考化合物,因此参考化合物-蛋白质复合物的结合自由能δg1为确定,如此在通过δg
b-δga计算出δδg
binding
后,进而通过δg2=δδg
binding
+δg1(式4)计算出目标分子-蛋白质复合物的结合自由能δg2。
[0121]
示例性的,常用分子模拟软件以amber程序为例,选择某蛋白小分子复合物作为测试体系,选择二面角、范德华及静电参数进行修改,计算该蛋白小分子复合物的结合自由能。
[0122]
1.选择已知小分子-蛋白质复合物构象的小分子作为参考化合物,构建相对自由能的分子对。
[0123]
2.根据小分子对构建适合rbfe计算的复合物体系初始输入文件,具体为:
[0124]
a.准备参考化合物与蛋白质之间复合物结构,同时利用分子对接(docking)的方法获得上述蛋白小分子复合物。
[0125]
b.利用amber18中的antechamber工具抓取针对各小分子的gaff2力场参数,同时利用am1bcc方法计算各小分子所带电荷。
[0126]
c.在此基础上利用amber18中的tleap工具构建蛋白质和小分子体系的初始输入文件。
[0127]
同理,构建目标分子-蛋白质复合物的初始输入文件。
[0128]
3.对于目标分子-蛋白质复合物和参考化合物-蛋白质复合物的初始输入文件,按照前述图1-图3方法,分别修改二面角、范德华及静电参数,得到各中间状态计算所需的力场输入文件,具体的:
[0129]
a.分别确定目标分子-蛋白质复合物以及参考化合物-蛋白质复合物的升温区域。
[0130]
b.对应所述升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,得到修改后的目标分子-蛋白质复合物体系的力场参数。
[0131]
同理,对应所述升温区域修改初始输入文件中参考化合物-蛋白质复合物体系的力场参数,得到修改后的参考化合物-蛋白质复合物体系的力场参数;
[0132]
c.基于修改后的参考化合物-蛋白质复合物体系的力场参数,和修改后的目标分子-蛋白质复合物体系的力场参数,生成fep/rest2模拟所需的参数文件。
[0133]
4.针对各中间态的输入进行分子动力学模拟,并保存模拟过程中的轨迹文件。
[0134]
5.根据得到的轨迹文件及前述本内特接受率方法(bar)方法计算具体的相对自由能,具体过程如下:
[0135]
a.首先对于轨迹文件中两相邻的中间态i及中间态j,分别计算中间态i轨迹在中间态j对应参数下的第一能量δu
ij
,同理计算出第二能量δu
ji
;
[0136]
b.将第一能量δu
ij
及第二能量δu
ji
带入bar方法(如下式1和式2),分别计算出参考化合物变换成目标分子的溶剂自由能差异δga,和参考化合物变换成目标分子的结合自由能差异δgb;
[0137][0138][0139]
c.基于参考化合物变换成目标分子的溶剂自由能差异δga,和参考化合物变换成目标分子的结合自由能差异δgb,按如下式3计算获得参考化合物变换成目标分子的相对结合自由能δδg
binding
;
[0140]
δδg
binding
=δg
b-δgaꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
式3;
[0141]
d.基于参考化合物变换成目标分子的相对结合自由能δδg
binding
,和已知的参考化合物-蛋白质复合物的结合自由能δg1,按照如下式4计算获得目标分子-蛋白质复合物的结合自由能δg2;
[0142]
δg2=δδg
binding
+δg1ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
式4;
[0143]
其中,δg为自由能差异,δu
ij
为所述第一能量,δu
ji
为所述第二能量,《*》i为中间态i下的系统平均,《*》j为中间态j下的系统平均,ni、ni为中间态i、j下的模拟轨迹的帧数,kb为波尔兹曼常数,t为模拟温度,δδg
binding
为相对结合自由能,δga为参考化合物变换成目标分子的溶剂自由能差异,δgb为参考化合物变换成目标分子的结合自由能差异,δg1为已知的参考化合物-蛋白质复合物的结合自由能,δg2为目标分子-蛋白质复合物的结合自
由能。
[0144]
如此,在通过δg
b-δga计算出δδg
binding
后,通过δg2=δδg
binding
+δg1(式4)最终计算出目标分子-蛋白质复合物的结合自由能δg2。
[0145]
将上述方法计算得到的结果与实验值进行对比,得到的结果如图6(b)所示,其中,图6(a)为未使用rest2增强采样时的fep计算结果,图6(b)为本技术使用rest2增强采样后的计算结果。
[0146]
将图6(b)使用fep/rest2的结果与图6(a)不使用rest2的结果相比,使用fep/rest2后的平均无符号误差mue(mean unsigned error)有所降低,同时相关系数r2有所提高。由此可知,本技术计算得到的自由能与实验结果更加接近,因此,本技术的增强采样方法以及增强采样方法提高了fep预测的精度。
[0147]
需要说明的是,本技术除了采用本内特接受率方法(bar)方法计算目标分子-蛋白质复合物的结合自由能,还可以采用其他方法来计算目标分子-蛋白质复合物的结合自由能,在此不做具体限定。
[0148]
综上,本技术的计算目标分子-蛋白质复合物的结合自由能的方法具有如下有益效果:
[0149]
通过执行本技术实施例中的增强采样方法,以尽量少修改代码的方式快速便捷地实现rest2方法的流程,而不需要大幅修改分子动力学模拟软件的底层代码,并且使得fep/rest2方法整体流程自动化,提高了fep的计算效率及预测精度,从而便于更加快捷地计算出目标分子与蛋白质之间的结合自由能。
[0150]
请参阅图7,本技术实施例还提供一种增强采样装置100。增强采样装置100包括:
[0151]
升温区域确定模块110,用于确定目标分子-蛋白质复合物的升温区域;
[0152]
力场参数修改模块120,用于对应所述升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,得到修改后的目标分子-蛋白质复合物体系的力场参数,然后进一步生成fep/rest2模拟所需的参数文件;和
[0153]
轨迹文件生成模块130,用于将所述参数文件作为势函数的输入进行fep/rest2模拟以生成轨迹文件,所述轨迹文件用于计算所述目标分子-蛋白质复合物的结合自由能。
[0154]
在某一个实施例中,所述力场参数修改模块120,包括:
[0155]
参数划分模块,用于将所述目标分子-蛋白质复合物体系的力场参数项划分为所述升温区域内部的参数、所述升温区域与环境区域间的参数以及所述环境区域内部的参数;
[0156]
第一修改模块,用于至少部分修改所述升温区域内部的参数、所述升温区域与所述环境区域间的参数以及所述环境区域内部的参数,得修改后的目标分子-蛋白质复合物体系的力场参数,然后进一步生成所述fep/rest2模拟所需的参数文件。
[0157]
在某一个实施例中,所述第一修改模块,具体用于:
[0158]
将所述升温区域内部的参数乘以第一类参数;
[0159]
将所述升温区域与所述环境区域间的参数乘以第二类参数;
[0160]
对所述环境区域内部的参数不做修改。
[0161]
在某一个实施例中,所述第一类参数包括键连系数、范德华系数及静电系数,所述第二类参数包括键连系数。
[0162]
在某一个实施例中,所述升温区域确定模块110,具体用于:
[0163]
将fep计算过程中的微扰区域确定为目标分子-蛋白质复合物的初始升温区域;
[0164]
判断所述目标分子中是否有环;
[0165]
若所述目标分子中有环,则进一步判断所述初始升温区域中的至少部分原子是否位于所述环上,或所述初始升温区域中的至少部分原子是否与所述环上的原子直接或间接相连;
[0166]
若所述初始升温区域中的至少部分原子位于所述环上或与所述环上的原子直接相连,则将所述环所在的区域添加至所述的初始升温区域,得到第一更新升温区域;
[0167]
若所述初始升温区域中的至少部分原子与所述环上的原子间接相连,则将所述环及与所述环间接相连部分所在的区域添加至所述的初始升温区域,得到第一更新升温区域;
[0168]
若所述初始升温区域中的所有原子既不位于所述环上,也不与所述环上的原子直接或间接相连,则将所述初始升温区域定为第一更新升温区域;
[0169]
若所述目标分子中无环,则将所述初始升温区域定为第一更新升温区域;
[0170]
判断是否存在额外指定的需要升温的区域;
[0171]
若存在,则将所述额外指定的需要升温的区域添加至所述第一更新升温区域,得升温区域;
[0172]
若不存在,则将所述第一更新升温区域定为升温区域。
[0173]
关于增强采样装置100的具体限定可以参见上文中对于增强采样方法的限定,在此不再赘述。上述增强采样装置100中的各个模块可全部或部分通过软件、硬件及其组合来实现。上述各模块可以硬件形式内嵌于或独立于计算机设备中的处理器中,也可以以软件形式存储于计算机设备中的存储器中,以便于处理器调用执行以上各个模块对应的操作。
[0174]
请参阅图8,本技术实施例还提供一种计算目标分子-蛋白质复合物的结合自由能的装置200,该装置200包括:
[0175]
构建模块210,用于构建目标分子-蛋白质复合物体系的初始输入文件;
[0176]
如上述任一实施例所述的增强采样装置100,用于基于输入的所述初始输入文件,获得轨迹文件;和
[0177]
计算模块220,用于基于所述轨迹文件获得目标分子-蛋白质复合物的结合自由能。
[0178]
在某一个实施例中,一种计算目标分子-蛋白质复合物的结合自由能的装置200还包括:
[0179]
参考化合物选择模块,用于选择已知小分子-蛋白质复合物构象的小分子为参考化合物;
[0180]
所述升温区域确定模块110,还用于确定参考化合物-蛋白质复合物的升温区域;
[0181]
所述力场参数修改模块120,具体用于:
[0182]
对应所述升温区域修改初始输入文件中目标分子-蛋白质复合物体系的力场参数,得到修改后的目标分子-蛋白质复合物体系的力场参数;
[0183]
对应所述升温区域修改初始输入文件中参考化合物-蛋白质复合物体系的力场参数,得到修改后的参考化合物-蛋白质复合物体系的力场参数;
[0184]
基于修改后的参考化合物-蛋白质复合物体系的力场参数,和修改后的目标分子-蛋白质复合物体系的力场参数,生成fep/rest2模拟所需的参数文件。
[0185]
在某一个实施例中,所述计算模块220包括第一子计算模块,所述第一子计算模块用于:
[0186]
基于所述轨迹文件,采用本内特接受率方法进行计算,获得所述目标分子-蛋白质复合物的结合自由能;
[0187]
所述第一子计算模块,具体用于:
[0188]
根据所述轨迹文件中两相邻的中间态i及中间态j,分别计算中间态i轨迹在中间态j对应参数下的第一能量δu
ij
,以及中间态j轨迹在中间态i对应参数下的第二能量δu
ji
;
[0189]
将所述第一能量δu
ij
和所述第二能量δu
ji
带入式1和式2,分别计算出参考化合物变换成目标分子的溶剂自由能差异δga,和参考化合物变换成目标分子的结合自由能差异δgb;
[0190][0191][0192]
基于参考化合物变换成目标分子的溶剂自由能差异δga,和参考化合物变换成目标分子的结合自由能差异δgb,按式3计算获得参考化合物变换成目标分子的相对结合自由能δδg
binding
;
[0193]
δδg
binding
=δg
b-δgaꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
式3;
[0194]
基于参考化合物变换成目标分子的相对结合自由能δδg
binding
,和已知的参考化合物-蛋白质复合物的结合自由能δg1,按照式4计算获得目标分子-蛋白质复合物的结合自由能δg2;
[0195]
δg2=δδg
binding
+δg1ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
式4;
[0196]
其中,δg为自由能差异,δu
ij
为所述第一能量,δu
ji
为所述第二能量,《*》i为中间态i下的系统平均,《*》j为中间态j下的系统平均,ni、ni为中间态i、j下的模拟轨迹的帧数,kb为波尔兹曼常数,t为模拟温度,δδg
binding
为相对结合自由能,δga为参考化合物变换成目标分子的溶剂自由能差异,δgb为参考化合物变换成目标分子的结合自由能差异,δg1为已知的参考化合物-蛋白质复合物的结合自由能,δg2为目标分子-蛋白质复合物的结合自由能。
[0197]
关于计算目标分子-蛋白质复合物的结合自由能的装置200的具体限定可以参见上文中对于计算目标分子-蛋白质复合物的结合自由能的方法的限定,在此不再赘述。上述计算目标分子-蛋白质复合物的结合自由能的装置200中的各个模块可全部或部分通过软件、硬件及其组合来实现。上述各模块可以硬件形式内嵌于或独立于计算机设备中的处理器中,也可以以软件形式存储于计算机设备中的存储器中,以便于处理器调用执行以上各个模块对应的操作。
[0198]
请参阅图9,本技术实施例还提供一种电子设备,包括:
[0199]
一个或多个处理器;
[0200]
存储器,与所述处理器耦接,用于存储一个或多个程序;
[0201]
当所述一个或多个程序被所述一个或多个处理器执行,使得所述一个或多个处理器实现如上任一实施例所述的增强采样方法或上述任一实施例所述的计算目标分子-蛋白质复合物的结合自由能的方法。
[0202]
处理器用于控制该终端设备的整体操作,以完成上述的增强采样方法或上述任一实施例所述的计算目标分子-蛋白质复合物的结合自由能的方法的全部或部分步骤。存储器用于存储各种类型的数据以支持在该终端设备的操作,这些数据例如可以包括用于在该终端设备上操作的任何应用程序或方法的指令,以及应用程序相关的数据。该存储器可以由任何类型的易失性或非易失性存储设备或者它们的组合实现,例如静态随机存取存储器(static random access memory,简称sram),电可擦除可编程只读存储器(electrically erasable programmable read-only memory,简称eeprom),可擦除可编程只读存储器(erasable programmable read-only memory,简称eprom),可编程只读存储器(programmable read-only memory,简称prom),只读存储器(read-only memory,简称rom),磁存储器,快闪存储器,磁盘或光盘。
[0203]
在一示例性实施例中,终端设备可以被一个或多个应用专用集成电路(application specific integrated circuit,简称asic)、数字信号处理器(digital signal processor,简称dsp)、数字信号处理设备(digital signal processing device,简称dspd)、可编程逻辑器件(programmable logic device,简称pld)、现场可编程门阵列(field programmable gate array,简称fpga)、控制器、微控制器、微处理器或其他电子元件实现,用于执行如上述任一项实施例所述的增强采样方法或上述任一实施例所述的计算目标分子-蛋白质复合物的结合自由能的方法,并达到如上述方法一致的技术效果。
[0204]
在另一示例性实施例中,还提供一种包括计算机程序的计算机可读存储介质,该计算机程序被处理器执行时实现如上述任一项实施例所述的增强采样方法或上述任一实施例所述的计算目标分子-蛋白质复合物的结合自由能的方法的步骤。例如,该计算机可读存储介质可以为上述包括计算机程序的存储器,上述计算机程序可由终端设备的处理器执行以完成如上述任一实施例所述的增强采样方法或上述任一实施例所述的计算目标分子-蛋白质复合物的结合自由能的方法,并达到如上述方法一致的技术效果。
[0205]
以上仅为本技术的较佳实施方式,并非因此限制本技术的专利范围,凡是利用本技术说明书及附图内容所作的等效结构或等效流程变换,或直接或间接运用在其他相关的技术领域,均同理包括在本技术的专利保护范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1