细胞类型确定方法、装置、设备以及存储介质与流程
1.本技术涉及人工智能领域,尤其涉及一种细胞类型确定方法、装置、设备以及存储介质。
背景技术:
2.近年来随着分子生物学和单细胞测序技术的进步,越来越多的研究表明,细胞的基因选择性表达、组织中多细胞间的相互作用决定了单个细胞的类型,如细胞的空间协调和信号传递决定了组织微环境的动态发展过程,决定了胚胎细胞如何分化为神经细胞、肌肉细胞、免疫细胞等等。
3.现有的细胞类型确定方式往往是通过对细胞的基因表达数据(转录组测序数据)进行分析,进而从分子层面确定细胞类型。或者是基于细胞的基因表达数据和各细胞之间的距离来确定细胞类型,上述方式在确定细胞类型的准确性方面仍有较大提升空间。
4.因此,如何进一步准确确定组中细胞的细胞类型成为亟需解决的问题。
技术实现要素:
5.本技术实施例提供一种细胞类型确定方法、装置、设备以及存储介质,可提升下确定细胞类型的准确性,适用性高。
6.一方面,本技术实施例提供一种方法细胞类型确定方法,该方法包括:
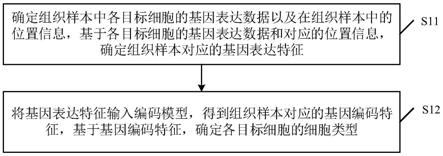
7.确定组织样本中各目标细胞的基因表达数据以及在上述组织样本中的位置信息,基于各上述目标细胞的基因表达数据和对应的位置信息,确定上述组织样本对应的目标基因表达特征;
8.将上述目标基因表达特征输入编码模型,得到上述组织样本对应的基因编码特征,基于上述基因编码特征,确定各上述目标细胞的细胞类型;
9.其中,上述编码模型是基于以下方式训练得到的:
10.将上述目标基因表达特征输入初始模型,得到上述组织样本对应的初始基因编码特征;
11.基于上述初始基因编码特征,确定各上述目标细胞的预测细胞类型、以及对应于各上述预测细胞类型的预测基因表达分布,确定对应于各上述预测细胞类型的实际基因表达分布,基于上述预测基因表达分布和上述实际基因表达分布,确定总训练损失值;
12.基于上述总训练损失值对上述初始模型进行迭代训练,直至上述总训练损失值满足训练结束条件时,停止训练并将训练结束时的初始模型确定为上述编码模型。
13.另一方面,本技术实施例提供了一种细胞类型确定装置,该装置包括:
14.数据处理模块,用于确定组织样本中各目标细胞的基因表达数据以及在上述组织样本中的位置信息,基于各上述目标细胞的基因表达数据和对应的位置信息,确定上述组织样本对应的目标基因表达特征;
15.类型确定模块,用于将上述目标基因表达特征输入编码模型,得到上述组织样本
对应的基因编码特征,基于上述基因编码特征,确定各上述目标细胞的细胞类型;
16.其中,上述编码模型是基于训练装置训练得到的,上述训练装置用于:
17.将上述目标基因表达特征输入初始模型,得到上述组织样本对应的初始基因编码特征;
18.基于上述初始基因编码特征,确定各上述目标细胞的预测细胞类型、以及对应于各上述预测细胞类型的预测基因表达分布,确定对应于各上述预测细胞类型的实际基因表达分布,基于上述预测基因表达分布和上述实际基因表达分布,确定总训练损失值;
19.基于上述总训练损失值对上述初始模型进行迭代训练,直至上述总训练损失值满足训练结束条件时,停止训练并将训练结束时的初始模型确定为上述编码模型。
20.另一方面,本技术实施例提供了一种电子设备,包括处理器和存储器,该处理器和存储器相互连接;
21.上述存储器用于存储计算机程序;
22.上述处理器被配置用于在调用上述计算机程序时,执行本技术实施例提供的细胞类型确定方法。
23.另一方面,本技术实施例提供了一种计算机可读存储介质,该计算机可读存储介质存储有计算机程序,该计算机程序被处理器执行以实现本技术实施例提供的细胞类型确定方法。
24.另一方面,本技术实施例提供了一种计算机程序产品,该计算机程序产品包括计算机程序或计算机指令,上述计算机程序或计算机指令被处理器执行时实现本技术实施例提供的细胞类型确定方法。
25.本技术实施例所确定出的组织样本对应的目标基因表达特征,可包括目标基因的基因表达数据以及位置信息,从而使得编码模型对组织样本的目标基因表达特征进行编码的过程中将各目标细胞的基因表达特征与位置信息相融合。并且由于模型训练过程中的训练损失是基于对应于预测细胞类型的预测基因表达分布和实际基因表达分布确定所确定的,因此在此基础上得到的编码模型对目标基因表达特征进行编码得到的基因编码特征具有较高的准确性。进而在基于基因编码特征确定组织样本中各目标细胞的细胞类型时可在融合细胞间位置关系的情况下,准确确定出目标细胞的细胞类型,适用性高。
附图说明
26.为了更清楚地说明本技术实施例中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本技术的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
27.图1是本技术实施例提供的细胞类型确定方法的流程示意图;
28.图2是本技术实施例提供的确定组织样本对应的特征图的场景示意图;
29.图3是本技术实施例提供的确定细胞类型的场景示意图;
30.图4是本技术实施例提供的模型训练方法的流程示意图;
31.图5a是本技术实施例通过的模型训练的一场景示意图;
32.图5b是本技术实施例提供的模型训练的另一场景示意图;
33.图6是本技术实施例提供的细胞类型确定效果的示意图;
34.图7是本技术实施例提供的细胞类型确定装置的结构示意图;
35.图8是本技术实施例提供的电子设备的结构示意图。
具体实施方式
36.下面将结合本技术实施例中的附图,对本技术实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本技术一部分实施例,而不是全部的实施例。下面通过参考附图描述的实施例是示例性的,仅用于解释本技术,而不能解释为对本技术的限制。基于本技术中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本技术保护的范围。
37.本技术领域技术人员可以理解,除非特意声明,这里使用的单数形式“一”、“一个”、“所述”和“该”也可包括复数形式。应该进一步理解的是,本技术的说明书中使用的措辞“包括”是指存在所述特征、整数、步骤、操作、元件和/或组件,但是并不排除存在或添加一个或多个其他特征、整数、步骤、操作、元件、组件和/或它们的组。应该理解,当我们称元件被“连接”或“耦接”到另一元件时,它可以直接连接或耦接到其他元件,或者也可以存在中间元件。此外,这里使用的“连接”或“耦接”可以包括无线连接或无线耦接。这里使用的措辞“和/或”包括一个或更多个相关联的列出项的全部或任一单元和全部组合。
38.本技术实施例提供的细胞类型确定方法可适用于生物医学、细胞研究、病理分析等领域,基于人工智能技术来确定细胞类型。例如,基于本技术实施例提供的细胞类型确定方法,可准确识别癌细胞,进而开发对应的靶向药物用于改善抗癌治疗。
39.其中,本技术实施例可基于人工智能领域中的机器学习技术训练得到可用于对组织样本的基因表达特征进行编码的编码模型,进而基于编码模型的编码结果确定组织样本中各目标细胞的细胞类型。
40.其中,人工智能是利用数字计算机或者数字计算机控制的机器模拟、延伸和扩展人的智能,感知环境、获取知识并使用知识获得最佳结果的理论、方法、技术及应用系统。换句话说,人工智能是计算机科学的一个综合技术,它企图了解智能的实质,并生产出一种新的能以人类智能相似的方式做出反应的智能机器。人工智能也就是研究各种智能机器的设计原理与实现方法,使机器具有感知、推理与决策的功能。
41.而机器学习是一门多领域交叉学科,涉及概率论、统计学、逼近论、凸分析、算法复杂度理论等多门学科。机器学习是人工智能的核心,是使计算机具有智能的根本途径,其应用遍及人工智能的各个领域。机器学习和深度学习通常包括人工神经网络、置信网络、强化学习、迁移学习、归纳学习、式教学习等技术,专门研究计算机怎样模拟或实现人类的学习行为,以获取新的知识或技能,如基于机器学习技术训练得到本技术实施例中的编码模型。
42.其中,本技术实施例涉及到的数据处理过程、如确定基因表达数据、确定细胞类型等过程可基于云计算技术实现。云计算指it基础设施的交付和使用模式,指通过网络以按需、易扩展的方式获得所需资源,是网格计算(grid computing)、分布式计算(distributedcomputing)、并行计算(parallel computing)、效用计算(utility computing)、网络存储(network storage technologies)、虚拟化(virtualization)、负载均衡(load balance)等传统计算机和网络技术发展融合的产物。随着互联网、实时数据流、
连接设备多样化的发展,以及搜索服务、社会网络、移动商务和开放协作等需求的推动,云计算迅速发展起来。不同于以往的并行分布式计算,云计算的产生从理念上将推动整个互联网模式、企业管理模式发生革命性的变革。
43.随着科学技术的不断发展,现有技术中主要存在以下几种方式来确定细胞类型。
44.(1)结合空间形态特征的单细胞空间转录组测序的bulk数据集聚类分析方法。该方法在提取细胞空间形态时受限于细胞轮廓的分割、形态特征的设计、图像分辨率以及细胞密集程度等因素,同时对于细胞位置的利用率较低且需要对基因表达数据进行降采样处理,从而导致确定细胞类型的准确性较低。
45.(2)基于图卷积神经网络(graph convolutional network,gcn)的空间信息嵌入的单细胞空间转录组细胞类型注释。该方法仅仅是在确定细胞类型的过程中利用细胞间的距离信息,细胞位置信息利用率较低,且由于gcn的局限性不能处理大规模的但下拨空间转录组数据。
46.(3)基于贝叶斯统计的空间转录组基因表达增强分析方法。该方法主要应用于对大体量空间转录组数据进行增强,并不适用于确定细胞类型,且基于贝叶斯统计的计算过程的计算效率较低。
47.(4)基于无监督空间信息嵌入的单细胞空间转录组细胞类型注释。该方法同样仅仅是在确定细胞类型的过程中利用细胞间的距离信息,细胞位置信息利用率较低,且细胞基因特征表达不准确,造成确定细胞类型的准确性降低。
48.综上所述,现有技术中在确定细胞类型时,存在细胞位置信息利用不充分,效率低下以及基因特征表达不准确等问题。基于此,本技术实施例提供一种细胞类型确定方法、装置、电子设备及可读存储介质,旨在解决上述中的部分或全部技术问题。
49.参见图1,图1是本技术实施例提供的细胞类型确定方法的流程示意图。如图1所示,本技术实施例提供的细胞类型确定方法可包括如下步骤:
50.步骤s11、确定组织样本中各目标细胞的基因表达数据以及在组织样本中的位置信息,基于各目标细胞的基因表达数据和对应的位置信息,确定组织样本对应的目标基因表达特征。
51.在一些可行的实施方式中,在确定组织样本中各目标细胞的基因表达数据时,可先确定组织样本的病理图像,如通过组织切片程序、共聚焦荧光成像等方式得到组织样本的病理图像。
52.其中,组织样本的病理图像可以是基于任一染色方式得到的病理图像,包括但不限于通过苏木精-伊红染色(hematoxylin-eosin staining,h&e)、ssdna染色、4',6-二脒基-2-苯基吲哚(4',6-diamidino-2-phenylindole,dapi)染色等方法得到的病理图像,染色方式具体可基于实际应用场景需求确定,在此不做限制。
53.其中,组织样本的病理图像可以是任一原始分辨率下的成像结果,如分辨率可为显微镜10倍镜、20倍镜、40倍镜等,具体也可基于实际应用场景需求确定,在此不做限制。
54.进一步地,基于病理图像可确定出组织样本中的所有细胞,从而可确定组织样本中所有细胞的转录本数量,并筛选掉组织样本中转录本数量在预设数量区间的细胞,将组织样本中转录本数量在预设数量区间内的细胞确定为目标细胞。
55.其中,转录本是由一条基因通过转录形成的一种或多种可供编码蛋白质的成熟的
mrna。即对于组织样本中的每个细胞而言,该细胞的转录本数量为一条基因通过转录行程的一种或者多种mrna。
56.其中,通过筛选掉组织样本中转录本数量未达到预设数量区间下限的细胞,可避免后续确定出的该类细胞的基因表达数据不能完全包括该类细胞的基因的情况,并且也可筛选掉发育异常的细胞。通过筛选掉组织样本中转录本数量超过预设数量区间上限的细胞,可消除细胞提前破裂或细胞重叠导致某一细胞的转录本数量增加的情况,以保证目标细胞的基因表达数据的准确性。
57.进一步地,在确定出组织样本中的目标细胞之后,可对每一目标细胞进行基因测序,得到每一目标细胞的初始基因表达数据,并可直接将该目标细胞的初始基因表达数据直接确定为该目标细胞的基因表达数据。
58.其中,每一目标细胞对的基因表达数据也可以成为该目标细胞的转录组数据。
59.在一些可行的实施方式中,在对每一目标细胞进行基因测序,得到每一目标细胞的初始基因表达数据之后,还可进一步对该目标细胞的初始基因表达数据进行数据优化,得到该目标细胞的基因表达数据。
60.具体地,在对各目标细胞的初始基因表达数据进行数据优化时,可去除各个目标细胞的初始基因表达数据中的异常基因数据,得到各目标细胞的基因表达数据。
61.需要说明的是,在组织样本的所有目标细胞中,并不是所以的目标细胞都具有异常基因,因此上述去除异常基因数据的过程可仅仅针对所有目标细胞中的部分细胞,即确定出目标细胞中包括异常基因的细胞,并将该细胞的初始基因表达数据中对应于异常基因的异常基因数据筛选掉。
62.其中,异常基因数据包括但不限于细胞中的突变基因、出现概率较低的基因或者指定的基因等,具体可基于实际应用场景需求确定,在此不做限制。
63.可选地,在得到各目标细胞的初始基因表达数据之后,可对各目标细胞的初始基因表达数据进行归一化处理得到各目标细胞的基因表达数据。通过对各目标细胞的初始基因表达数据进行归一化处理,可消除在确定初始基因表达数据时各目标细胞在组织样本中的不同位置所带来的差异性,例如rna分离不均衡、测序深度不一致等,也称之为不同空间位置的批次效应,并且可使得各目标细胞的基因表达数据处于同一数据维度之上,便于后续对各目标细胞的基因表达数据的应用。
64.可选地,在得到各目标细胞的初始基因表达数据之后,可将各目标细胞的初始基因表达数据进行数据筛选,以筛选掉各初始基因表达数据中的异常基因数据,并进一步对筛选后的初始基因表达数据进行归一化处理,得到各目标细胞的基因表达数据。
65.或者,可先对各目标细胞的初始基因表达数据进行归一化处理,再将归一化处理后的初始基因表达数据中的异常基因数据,得到各目标细胞的基因表达数据。
66.需要特别说明的是,上述对各目标细胞的基因表达数据进行数据优化的过程仅为示例,如还可以包括对各目标细胞的基因表达数据进行校对或纠正等,具体可基于实际应用场景需求确定,在此不做限制。
67.在一些可行的实施方式中,在确定组织样本中各目标细胞的基因表达数据时,还可先基于病理图像确定组织样本中的所有细胞,并对所有细胞进行基因测序得到所有细胞的初始基因表达数据。
68.进一步地,可去除组织样本中各个细胞的初始基因表达数据中的异常基因数据,得到组织样本中各细胞的基因表达数据。或者,对组织样本中各个细胞的基因表达数据进行归一化处理,得到组织样本中各细胞的基因表达数据。或者,在得到组织样本中所有细胞的初始基因表达数据之后,可基于上述异常基因数据的数据优化方式以及归一化处理方式对各细胞的初始基因表达数据进行数据优化,得到组织样本中各细胞的基因表达数据。
69.基于此,在得到组织样本中各细胞的基因表达数据之后,可再行确定各细胞的转录本数量,将转录本数量在预设数量区间内的细胞确定为目标细胞,以得到组织样本中各目标细胞的基因表达数据。
70.在一些可行的实施方式中,在确定组织样本的病理图像之后,可基于病理图像确定各目标细胞在组织样本中的位置信息,从而对于每一目标细胞而言,可基于该目标细胞在组织样本中的位置信息和该目标细胞对应的基因表达数据,确定组织样本对应的目标基因表达特征。
71.其中,组织样本对应的目标基因表达特征包括每个目标细胞在组织样本中的位置信息、以及每个目标细胞的基因表达数据。
72.其中,本技术实施例中每一目标细胞在组织样本中的位置信息可以为该目标细胞在组织样本中的二维坐标信息,也可以为该目标细胞在组织样本中的三维空间信息,在此不做限制。
73.具体地,在确定组织样本对应的目标基因表达特征时,可先确定每一目标细胞对应的基因表达特征,其中,每一目标细胞对应的基因表达特征包该目标细胞在组织样本中的位置信息以及该目标细胞的基因表达数据。
74.对于每一目标细胞,可将该目标细胞的基因表达数据和位置信息进行融合处理,得到该目标细胞的基因表达特征。例如,对于每一目标细胞,可将该目标细胞的基因表达数据和位置信息进行对齐,得到该目标细胞的基因表达特征,或者可将该目标细胞的基因表达数据和位置信息输入融合模型,通过融合模型得到该目标细胞对应的基因表达特征。
75.或者,对于每一目标细胞,可将该目标细胞的基因表达数据视为一个一维向量,该向量的长度与该目标细胞的基因数量一致,每一向量元素对应于该目标细胞的一个基因表达。进一步的,可将该目标细胞在组织样本中的位置信息与该向量进行融合,得到该目标细胞对应的基因表达特征。
76.例如,对于每一目标细胞而言,可将该目标细胞在组织样本中的二维坐标与该目标细胞的基因表达数据对应的一维向量进行融合,得到该目标细胞对应的基因表达特征。用于表示该目标细胞的空间转录组数据的三维矩阵n*h*w。其中,n表示基因数量,h和w分别表示高和宽,用于指示目标细胞在组织样本中的位置信息。其中,也可将该三维矩阵视为一个多通道的图,每一通道对应一个基因。
77.其中,对于每一目标细胞而言,该目标细胞对应的基因表达特征也可称为该细胞对应的空间转录组数据。并且本技术实施例中组织样本的目标基因表达特征可以为基于现有的空间转录组测序技术采集到的空间转录组数据,如包括但不限于基于多重抗误差矫正荧光原位杂交技术(merfish)、ouroboros荧光原位杂交技术(fluorescence in situ hybridization,fish)osmfish、slide-seq、seqfish+、seqfish、visium、空间分辨转录放大读出图(starmap)、高清空间转录组学(high definition spatial transcriptomics,
hdst)技术等得到的空间转录组数据,具体可基于实际应用场景需求确定,在此不做限制。
78.进一步地,在确定各目标细胞的基因表达特征之后,可基于各目标细胞对应的基因表达特征和对应的位置信息,确定组织样本对应的目标基因表达特征。如可将各目标细胞按照各自在组织样本中的位置,将各目标细胞对应的基因表达特征进行整合,得到组织样本对应的目标基因表达特征,从而使得组织样本对应的目标基因表达特征包括各目标细胞对应的基因表达特征以及在组织样本中的位置信息。
79.其中,可将组织样本对应的目标基因表达特征视为一个特征图,每一目标细胞对应于特征图中的一个像素,每一像素对应的特征数据为对应目标细胞所对应的基因表达特征,每一像素的一个通道对应于一个目标细胞的一个基因的基因表达数据,且每一像素的位置信息表示对应目标细胞在组织样本中的位置信息。参见图2,图2是本技术实施例提供的确定组织样本对应的特征图的场景示意图。如图2所示,对于细胞1至细胞m,可确定每一细胞对应的基因表达特征,其中每一细胞的基因表达特征包括该细胞的基因表达数据和对应的位置信息。进一步基于细胞1至细胞m在组织样本中的位置信息以及各细胞对应的基因表达特征,确定组织样本对应的特征图。
80.步骤s12、将基因表达特征输入编码模型,得到组织样本对应的基因编码特征,基于基因编码特征,确定各目标细胞的细胞类型。
81.在一些可行的实施方式中,在确定出组织样本对应的目标基因表达特征之后,可将组织样本对应的目标基因表达特征输入编码模型,得到组织样本对应的基因编码特征。
82.其中,编码模型可以为基于深度卷积网络结构或深度学习编码器构建的神经网络模型,包括但不限于基于efficientnet、resnet、densenet等神经网络结构构建的编码模型,以及基于变分自编码器(variational auto-encoder,vae)实现的编码模型,具体可基于实际应用场景需求确定,在此不做限制。
83.其中,上述编码模型的构建方式仅为示例,且编码模型中的卷积层以及对应的卷积核大小具体可基于实际应用场景需求确定,在此不做限制。
84.其中,通过编码模型中的卷积层可对目标基因表达特征进行滑窗卷积运算,以降低输出的基因编码特征中各目标细胞的基因表达维度,并且使得各目标细胞的基因表达嵌入邻近目标细胞在组织样本中的位置信息。
85.进一步地,如图3所示,图3是本技术实施例提供的确定细胞类型的场景示意图。在得到组织样本对应的目标基因表达特征之后,可将目标基因表达特征输入编码模型,得到组织样本对应的基因编码特征。进而可基于目标基因编码特征对各目标细胞进行聚类,得到组织样本中各目标细胞的细胞类型。
86.可选地,在得到组织样本对应的基因编码特征之后,可将基因编码特征输入预训练的类型预测模型,通过预训练的类型预测模型得到组织样本中各目标细胞的细胞类型。
87.其中,上述预训练的类型预测模型同样可基于神经网络构建,具体构建方式和训练方式在此不做说明。
88.在一些可行的实施方式中,在确定出组织样本中各目标细胞的细胞类型之后,可确定出各目标细胞中与该目标细胞的细胞类型相关联的目标基因。
89.其中,每一目标细胞对应的目标基因,为可代表该目标细胞的细胞类型的一个或者多个基因。如可将该目标细胞中该目标细胞的细胞类型所独有的基因确定为该目标细胞
对应的目标基因。
90.进一步地,对于每一目标细胞,可基于该目标细胞对应的目标基因对该目标细胞进行细胞类型标记,如可对该目标细胞对应的目标基因进行标记,从而可基于任一目标细胞中被标记的基因,确定对应目标细胞的细胞类型。
91.其中,本技术实施例中的编码模型是基于组织样本对应的目标基因表达特征训练得到的,具体训练方式可参见图4。图4是本技术实施例提供的模型训练方法的流程示意图。如图4所示,本技术实施例提供的模型训练方法可包括如下步骤:
92.步骤s41、将目标基因表达特征输入初始模型,得到组织样本对应的初始基因编码特征。
93.在一些可行的实施方式中,初始模型为上述编码模型进行模型训练之前的模型,其模型结构与编码模型的结构相同。也即通过对初始模型的训练过程对初始模型的训练参数进行调整,最终得到的模型即为上述编码模型。
94.具体地,在对初始模型进行训练时,可将组织样本对应的目标基因表达特征输入初始模型,得到组织样本对应的初始基因编码特征。其中,组织样本对应的目标基因表达特征的确定方式可参见图1中步骤s11所示的实现方式,在此不再赘述。
95.步骤s42、基于初始基因编码特征,确定各目标细胞的预测细胞类型、以及对应于各预测细胞类型的预测基因表达分布,确定对应于各预测细胞类型的实际基因表达分布,基于预测基因表达分布和实际基因表达分布,确定总训练损失值。
96.在一些可行的实施方式中,基于组织样本对应的初始基因编码特征,可对各目标细胞进行聚类,进而基于聚类结果确定出各目标细胞对应于初始基因编码特征的细胞类型。
97.或者可将基因编码特征输入预训练的类型预测模型,通过预训练的类型预测模型得到组织样本中各目标细胞的细胞类型。为方便描述,本技术实施例将在对初始模型训练过程中得到的各目标细胞的细胞类型称为预测细胞类型。
98.其中,在对初始模型进行训练的过程中,基于初始基因编码特征确定各目标细胞的预测细胞类型的实现方式,与图1中步骤s21所示的基于基因编码特征确定各目标细胞的细胞类型的方式相同,在此不再赘述。
99.进一步地,在得到初始基因编码特征之后,可基于初始基因编码特征确定在经过初始模型对目标基因表达特征进行处理后,每个目标对应的基因表达数据(为方便描述,以下称为训练基因表达数据)。
100.对于每一预测细胞类型,可将属于该预测细胞类型的至少一个目标细胞所对应的训练基因表达数据进行平均处理,得到对应于该预测细胞类型的平均训练基因表达数据。
101.基于对应于各预测细胞类型的平均训练基因表达数据,得到对应于各预测细胞类型的预测基因表达分布。其中,预测基因表达分布用于表示在经过初始模型对目标基因表达特征进行处理后,组织样本中所包括的每一细胞类型所对应的一个单细胞基因表达分布。并且,预测基因表达分布中各预测细胞类型的排序可基于实际应用场景需求确定,在此不做限制。
102.同时,可从预构建的单细胞基因表达数据集中确定出每一预测细胞类型对应的至少一个单细胞基因表达数据,并且对于每一预测细胞类型,可同样将属于该预测细胞类型
的至少一份单细胞基因表达数据进行评价处理,得到该预测细胞类型对应的平均基因表达数据。
103.同理,基于对应于各预测细胞类型的平均基因表达数据,得到对应于各预测细胞类型的实际基因表达分布。其中,实际基因表达分布用于表示各预测细胞类型所实际对应的一个单细胞基因表达分布。并且,预测基因表达分布中各预测细胞类型的排序与上述预测基因表达分布中各预测细胞类型的排序一致。
104.进一步地,可确定对应于各预测细胞类型的预测基因表达分布和实际基因表达分布的kl散度,将确定出的kl散度确定为模型训练过程中的总训练损失值。
105.此时,模型训练过程中的总训练损失值表征了对应于各预测细胞类型的预测基因表达分布与实际基因表达分布的差异,总训练损失值越大,说明对应于各预测细胞类型的预测基因表达分布与实际基因表达分布之间的差异越大,进而说明初始模型得到的初始基因编码在融和各目标细胞的位置信息后中各目标细胞的训练基因表达数据不能完全代表各目标细胞,需要继续对初始模型训练。反之,总训练损失值越小,说明对应于各预测细胞类型的预测基因表达分布与实际基因表达分布之间的差异越小,进而说明初始模型得到的初始基因编码在融合各目标细胞的位置信息后各目标细胞的训练基因表达数据越接近各目标细胞的基因表达数据,模型训练效果越来越好。
106.其中,预构建的单细胞基因表达数据集为已有的包括多种细胞类型下的多个细胞的基因表达数据的数据集,包括但不限于通过显微解剖技术、荧光原位杂交技术(fluorescence in situ hybridization,fish)以及原位捕获技术得到的单细胞基因表达数据,具体可基于实际应用场景需求确定,在此不做限制。
107.其中,本技术实施例中确定出目标组织细胞的预测细胞类型的粒度划分可基于实际应用场景需求确定,如可包括上皮组织细胞、结缔组织细胞、肌肉组织细胞以及神经组织细胞等,还对某一大类型的细胞进行进一步分类,如对鼠脑皮层细胞进行进一步分类,在此不做限制。
108.其中,预构建的单细胞基因表达数据集可以存储于服务器、数据库、云存储空间或者区块链中,具体可基于实际应用场景需求确定,在此不做限制。其中,数据库简而言之可视为电子化的文件柜——存储电子文件的处所,在本技术中可用于项目缓存信息。区块链是分布式数据存储、点对点传输、共识机制、加密算法等计算机技术的新型应用模式。区块链本质上是一个去中心化的数据库,是一串使用密码学方法相关联产生的数据块。在本技术中,区块链中的每一个数据块均可存储单细胞基因表达数据集。云存储是在云计算概念上延伸和发展出来的一个新的概念,是指通过集群应用、网格技术以及分布存储文件系统等功能,将网络中大量各种不同类型的存储设备(存储设备也称之为存储节点)通过应用软件或应用接口集合起来协同工作,共同存储单细胞基因表达数据集。
109.基于此,在获取预构建的单细胞基因表达数据时,可根据实际需求从服务器、数据库、云存储空间或者区块链中获取。
110.在一些可行的实施方式中,在确定总训练损失值时,还可基于对应于各预测细胞类型的预测基因表达分布和实际基因表达分布,确定第一训练损失值。
111.同时,将组织样本对应的初始基因编码特征输入解码模型,通过解码模型对初始基因编码特征进行解码,得到组织样本对应的还原基因表达特征。其中,解码模型为与初始
模型具有完全相反的神经网络结构,并且根据模型训练过程中对初始模型的模型参数的调整对自身的模型参数进行调整,以适应初始模型的模型参数的变化,从而可通过返卷积层对初始基因编码特征进行滑窗反卷积运算,将初始基因编码特征进行还原。
112.其中,在基于解码模型对初始基因编码特征进行解码的过程中,通过跳跃连接(skip connection)的方式获取此次训练过程中初始模型的各卷积层得到的卷积特征,将每一次反卷积得到的反卷积特征与对应的卷积特征进行融合,以对融合后的卷积特征进行进一步反卷积处理,以提升解码模型每一次反卷积处理过程中的特征识别率和准确性。
113.基于此,可基于组织样本对应的目标基因表达特征和还原基因表达特征,确定第二训练损失值,第二训练损失值表征了组织样本对应的目标基因表达特征和还原基因表达特征之间的差异。第二训练损失值越小,说明还原基因表达特征与目标基因表达特征相近,进而说明初始模型的编码稳定性和准确性较高。第二训练损失值越小,说明还原基因表达特征与目标基因表达特征差异性越大,进而说明初始模型的编码稳定性和准确性还有待提高,需要继续进行训练。
114.进一步地,基于第一训练损失值和第二训练损失值可确定总训练损失值,如可将第一训练损失值和第二训练损失值之和确定为总训练损失值,或者可分别确定第一训练损失值和第二训练损失值各自对应的训练权重,将第一续联损失值和第二训练损失值的权重和确定为总训练损失值,或者可将第一训练损失值和第二训练损失值进行归一化处理,并将归一化处理后的第一训练损失值和第二训练损失值之和确定为总训练损失值。
115.其中,上述基于第一训练损失值和第二训练损失值确定总训练损失值的方式仅为示例,具体可基于实际应用场景需求确定,在此不做限制。
116.其中,在基于组织样本对应的目标基因表达特征和还原基因表达特征,确定第二训练损失值时,可确定目标基因表达特征和还原基因表达特征的交叉熵损失,将得到的交叉熵损失确定为第二训练损失值。
117.或者,可确定组织样本对应的目标基因表达特征和还原基因表达特征的特征相似度,将得到的特征相似度确定为第二训练损失值。
118.或者,可基于目标基因表达特征确定各目标细胞的基因表达数据,基于还原基因表达特征确定各目标细胞的还原基因表达数据,从而确定各目标细胞的基因表达数据和还原基因表达数据的kl散度,将kl散度确定为第二训练损失值。
119.其中,上述确定第二训练损失值的方式仅为示例,具体可基于实际应用场景需求确定,在此不做限制。
120.步骤s43、基于总训练损失值对初始模型进行迭代训练,直至总训练损失值满足训练结束条件时,停止训练并将训练结束时的初始模型确定为编码模型。
121.在一些可行的实施方式中,在确定对初始模型进行训练所对应的总训练损失值后,可基于总训练损失值对初始模型进行迭代训练,直至预设训练结束条件时停止训练。
122.其中,上述训练结束条件可以为总训练损失值达到收敛,或者连续预设次数的总训练损失值小于预设阈值,或者可以为连续预设次数的相邻两次总训练损失值的差值小于预设阈值等,具体可基于实际应用场景需求确定,在此不做限制。
123.对于每次训练,在总训练损失值满足训练结束条件时刻停止训练,并将训练结束时的初始模型确定为最终的编码模型。若在对初始模型的训练过程中总训练损失值不满足
训练结束条件,则可对初始模型的模型参数进行调整,并对调整后的模型再次基于上述方式进行训练并确定总训练损失值,直至总训练损失值满足训练结束条件时停止训练。
124.如图5a所示,图5a是本技术实施例通过的模型训练的一场景示意图。在对初始模型训练过程中,可基于组织样本中各目标细胞的预测细胞类型,确定对应于各预测细胞类型的预测基因表达分布,并通过已构建的单细胞基因表达数据集确定对应于各预测细胞类型的实际基因表达分布。从而可基于对应于各预测细胞类型的预测基因表达分布和实际基因表达分布确定总训练损失值,并基于总训练损失值和组织样本对应的目标基因表达特征对初始模型进行迭代训练,直至总训练损失值满足训练结束条件时,停止训练并将停止训练时的初始模型确定为最终的编码模型。
125.如图5b所示,图5b是本技术实施例提供的模型训练的另一场景示意图。在对初始模型训练过程中,可基于组织样本中各目标细胞的预测细胞类型,确定对应于各预测细胞类型的预测基因表达分布,并通过已构建的单细胞基因表达数据集确定对应于各预测细胞类型的实际基因表达分布。从而可基于对应于各预测细胞类型的预测基因表达分布和实际基因表达分布确定第一训练损失值。同时基于此次训练过程对应于初始模型的解码器对组织样本对应的初始基因编码特征进行解码,得到组织样本对应的还原基因表达特征,从而基于组织样本的目标基因表达特征和还原基因表达特征确定第二训练损失值,进而基于第一训练损失值和第二训练损失值确定总训练损失值,并基于总训练损失值和组织样本对应的目标基因表达特征对初始模型进行迭代训练,直至总训练损失值满足训练结束条件时,停止训练并将停止训练时的初始模型确定为最终的编码模型。
126.本技术实施例提供的细胞类型确定方法以及编码模型的训练方法可基于训练装置执行,该训练装置可以是独立的物理服务器,也可以是多个物理服务器构成的服务器集群或者分布式系统,还可以是提供云计算服务的云服务器,也可以是具有数据处理能力的终端设备,在此不做限制。其中,终端设备可以是智能手机、平板电脑、笔记本电脑、台式计算机、智能音箱、智能手表、车载终端、智能电视等,但并不局限于此。
127.本技术实施例所确定出的组织样本对应的目标基因表达特征,可包括目标基因的基因表达数据以及位置信息,从而使得编码模型对组织样本的目标基因表达特征进行编码的过程中将各目标细胞的基因表达特征与位置信息相融合。并且由于模型训练过程中的训练损失是基于对应于预测细胞类型的预测基因表达分布和实际基因表达分布确定所确定的,因此在此基础上得到的编码模型对目标基因表达特征进行编码的基因编码特征具有较高的准确性。进而在基于基因编码特征确定组织样本中各目标细胞的细胞类型时可在融合细胞间位置关系的情况下,相较于传统的基于聚类算法直接确定细胞类型的方法具有更高的准确性,适用性高。
128.如图6所示,图6是本技术实施例提供的细胞类型确定效果的示意图,图中的每一个方块代表组织样本中的一个细胞,方块中的不同类型表示不同对应的细胞类型。其中,图6中左边示意图是传统的基于聚类算法直接确定细胞类型的结果示意图,由此不难发现基于聚类算法直接确定细胞类型时,可会导致大量的细胞类型出现错误。图6中右边示意图是基于本技术实施例提供的细胞类型确定方法所确定出的细胞类型,基于此方法可使得相同类型的细胞较为集中,不同类型的细胞簇之间的分节较为明显,细胞确定效果更好。
129.参见图7,图7是本技术实施例提供的细胞类型确定装置的结构示意图。本技术实
施例提供的细胞类型确定装置包括:
130.数据处理模块71,用于确定组织样本中各目标细胞的基因表达数据以及在上述组织样本中的位置信息,基于各上述目标细胞的基因表达数据和对应的位置信息,确定上述组织样本对应的目标基因表达特征;
131.类型确定模块72,用于将上述目标基因表达特征输入编码模型,得到上述组织样本对应的基因编码特征,基于上述基因编码特征,确定各上述目标细胞的细胞类型;
132.其中,上述编码模型是基于训练装置训练得到的,上述训练装置用于:
133.将上述目标基因表达特征输入初始模型,得到上述组织样本对应的初始基因编码特征;
134.基于上述初始基因编码特征,确定各上述目标细胞的预测细胞类型、以及对应于各上述预测细胞类型的预测基因表达分布,确定对应于各上述预测细胞类型的实际基因表达分布,基于上述预测基因表达分布和上述实际基因表达分布,确定总训练损失值;
135.基于上述总训练损失值对上述初始模型进行迭代训练,直至上述总训练损失值满足训练结束条件时,停止训练并将训练结束时的初始模型确定为上述编码模型。
136.在一些可行的实施方式中,上述数据处理模块71,用于:
137.确定组织样本中所有细胞的转录本数量,将转录本数量在预设数量区间的细胞确定为目标细胞;
138.确定各上述目标细胞的初始基因表达数据,并对各上述初始基因表达数据进行数据优化,得到各上述目标细胞的基因表达数据。
139.在一些可行的实施方式中,上述数据处理模块71,用于:
140.去除各上述目标细胞的初始基因表达数据中的异常基因数据;
141.将各上述初始基因表达数据进行归一化处理。
142.在一些可行的实施方式中,上述数据处理模块71,用于:
143.对于每一上述目标细胞,基于该目标细胞的基因表达数据和对应的位置信息,确定该目标细胞对应的基因表达特征;
144.基于各上述目标细胞对应的基因表达特征,确定上述组织样本对应的目标基因表达特征,上述目标基因表达特征包括各上述目标细胞的基因表达数据和对应的位置信息。
145.在一些可行的实施方式中,上述类型确定模块72,还用于:
146.对于每一上述目标细胞,确定该目标细胞中与该目标细胞的细胞类型相关联的目标基因,基于上述目标基因对上述目标细胞进行细胞类型标记。
147.在一些可行的实施方式中,上述训练装置,用于:
148.基于上述初始基因编码特征对各上述目标细胞进行聚类,得到各上述目标细胞的预测细胞类型;
149.基于上述初始基因编码特征和各上述预测细胞类型,确定对应于各上述预测细胞类型的预测基因表达分布。
150.在一些可行的实施方式中,上述训练装置,用于:
151.从预构建的单细胞基因表达数据集中确定出每一上述预测细胞类型对应的至少一份单细胞基因表达数据;
152.对于每一上述预测细胞类型,确定该预测细胞类型对应的至少一份单细胞基因表
达数据的平均基因表达数据;
153.基于各上述预测细胞类型对应的平均基因表达数据,确定对应于各上述预测细胞类型的实际基因表达分布。
154.在一些可行的实施方式中,上述训练装置,用于:
155.基于上述预测基因表达分布和上述实际基因表达分布,确定第一训练损失值;
156.对上述初始基因编码特征进行解码,得到上述组织样本对应的还原基因表达特征;
157.基于上述组织样本对应的目标基因表达特征和还原基因表达特征,确定第二训练损失值;
158.基于上述第一训练损失值和上述第二训练损失值,确定总训练损失值。
159.具体实现中,上述细胞类型确定装置可通过其内置的各个功能模块执行如上述图1中各个步骤所提供的实现方式,具体可参见上述各个步骤所提供的实现方式,在此不再赘述。
160.参见图8,图8是本技术实施例提供的电子设备的结构示意图。如图8所示,本实施例中的电子设备800可以包括:处理器801,网络接口804和存储器805,此外,上述电子设备800还可以包括:用户接口803,和至少一个通信总线802。其中,通信总线802用于实现这些组件之间的连接通信。其中,用户接口803可以包括显示屏(display)、键盘(keyboard),可选用户接口803还可以包括标准的有线接口、无线接口。网络接口804可选的可以包括标准的有线接口、无线接口(如wi-fi接口)。存储器804可以是高速ram存储器,也可以是非易失性存储器(non-volatile memory,nvm),例如至少一个磁盘存储器。存储器805可选的还可以是至少一个位于远离前述处理器801的存储装置。如图8所示,作为一种计算机可读存储介质的存储器805中可以包括操作系统、网络通信模块、用户接口模块以及设备控制应用程序。
161.在图8所示的电子设备800中,网络接口804可提供网络通讯功能;而用户接口803主要用于为用户提供输入的接口;而处理器801可以用于调用存储器805中存储的设备控制应用程序,以实现:
162.确定组织样本中各目标细胞的基因表达数据以及在上述组织样本中的位置信息,基于各上述目标细胞的基因表达数据和对应的位置信息,确定上述组织样本对应的目标基因表达特征;
163.将上述目标基因表达特征输入编码模型,得到上述组织样本对应的基因编码特征,基于上述基因编码特征,确定各上述目标细胞的细胞类型;
164.其中,上述编码模型可基于上述处理器901基于以下方式训练得到的:
165.将上述目标基因表达特征输入初始模型,得到上述组织样本对应的初始基因编码特征;
166.基于上述初始基因编码特征,确定各上述目标细胞的预测细胞类型、以及对应于各上述预测细胞类型的预测基因表达分布,确定对应于各上述预测细胞类型的实际基因表达分布,基于上述预测基因表达分布和上述实际基因表达分布,确定总训练损失值;
167.基于上述总训练损失值对上述初始模型进行迭代训练,直至上述总训练损失值满足训练结束条件时,停止训练并将训练结束时的初始模型确定为上述编码模型。
168.在一些可行的实施方式中,上述处理器801用于:
169.确定组织样本中所有细胞的转录本数量,将转录本数量在预设数量区间的细胞确定为目标细胞;
170.确定各上述目标细胞的初始基因表达数据,并对各上述初始基因表达数据进行数据优化,得到各上述目标细胞的基因表达数据。
171.在一些可行的实施方式中,上述处理器801用于:
172.去除各上述目标细胞的初始基因表达数据中的异常基因数据;
173.将各上述初始基因表达数据进行归一化处理。
174.在一些可行的实施方式中,上述处理器801用于:
175.对于每一上述目标细胞,基于该目标细胞的基因表达数据和对应的位置信息,确定该目标细胞对应的基因表达特征;
176.基于各上述目标细胞对应的基因表达特征,确定上述组织样本对应的目标基因表达特征,上述目标基因表达特征包括各上述目标细胞的基因表达数据和对应的位置信息。
177.在一些可行的实施方式中,上述处理器801还用于:
178.对于每一上述目标细胞,确定该目标细胞中与该目标细胞的细胞类型相关联的目标基因,基于上述目标基因对上述目标细胞进行细胞类型标记。
179.在一些可行的实施方式中,上述处理器801用于:
180.基于上述初始基因编码特征对各上述目标细胞进行聚类,得到各上述目标细胞的预测细胞类型;
181.基于上述初始基因编码特征和各上述预测细胞类型,确定对应于各上述预测细胞类型的预测基因表达分布。
182.在一些可行的实施方式中,上述处理器801用于:
183.从预构建的单细胞基因表达数据集中确定出每一上述预测细胞类型对应的至少一份单细胞基因表达数据;
184.对于每一上述预测细胞类型,确定该预测细胞类型对应的至少一份单细胞基因表达数据的平均基因表达数据;
185.基于各上述预测细胞类型对应的平均基因表达数据,确定对应于各上述预测细胞类型的实际基因表达分布。
186.在一些可行的实施方式中,上述处理器801用于:
187.基于上述预测基因表达分布和上述实际基因表达分布,确定第一训练损失值;
188.对上述初始基因编码特征进行解码,得到上述组织样本对应的还原基因表达特征;
189.基于上述组织样本对应的目标基因表达特征和还原基因表达特征,确定第二训练损失值;
190.基于上述第一训练损失值和上述第二训练损失值,确定总训练损失值。
191.应当理解,在一些可行的实施方式中,上述处理器801可以是中央处理单元(central processing unit,cpu),该处理器还可以是其他通用处理器、数字信号处理器(digital signal processor,dsp)、专用集成电路(application specific integrated circuit,asic)、现成可编程门阵列(field-programmable gate array,fpga)或者其他可
编程逻辑器件、分立门或者晶体管逻辑器件、分立硬件组件等。通用处理器可以是微处理器或者该处理器也可以是任何常规的处理器等。该存储器可以包括只读存储器和随机存取存储器,并向处理器提供指令和数据。存储器的一部分还可以包括非易失性随机存取存储器。例如,存储器还可以存储设备类型的信息。
192.具体实现中,上述电子设备800可通过其内置的各个功能模块执行如上述图1和/或图4中各个步骤所提供的实现方式,具体可参见上述各个步骤所提供的实现方式,在此不再赘述。
193.本技术实施例还提供一种计算机可读存储介质,该计算机可读存储介质存储有计算机程序,被处理器执行以实现图1和/或图4中各个步骤所提供的方法,具体可参见上述各个步骤所提供的实现方式,在此不再赘述。
194.上述计算机可读存储介质可以是前述任一实施例提供的细胞类型确定装置或电子设备的内部存储单元,例如电子设备的硬盘或内存。该计算机可读存储介质也可以是该电子设备的外部存储设备,例如该电子设备上配备的插接式硬盘,智能存储卡(smart media card,smc),安全数字(secure digital,sd)卡,闪存卡(flash card)等。上述计算机可读存储介质还可以包括磁碟、光盘、只读存储记忆体(read-only memory,rom)或随机存储记忆体(randomaccess memory,ram)等。进一步地,该计算机可读存储介质还可以既包括该电子设备的内部存储单元也包括外部存储设备。该计算机可读存储介质用于存储该计算机程序以及该电子设备所需的其他程序和数据。该计算机可读存储介质还可以用于暂时地存储已经输出或者将要输出的数据。
195.本技术实施例提供了一种计算机程序产品,该计算机程序产品包括计算机程序或计算机指令,上述计算机程序或计算机指令被处理器执行图1和/或图4中各个步骤所提供的方法。
196.本技术的权利要求书和说明书及附图中的术语“第一”、“第二”等是用于区别不同对象,而不是用于描述特定顺序。此外,术语“包括”和“具有”以及它们任何变形,意图在于覆盖不排他的包含。例如包含了一系列步骤或单元的过程、方法、系统、产品或电子设备没有限定于已列出的步骤或单元,而是可选地还包括没有列出的步骤或单元,或可选地还包括对于这些过程、方法、产品或电子设备固有的其它步骤或单元。在本文中提及“实施例”意味着,结合实施例描述的特定特征、结构或特性可以包含在本技术的至少一个实施例中。在说明书中的各个位置展示该短语并不一定均是指相同的实施例,也不是与其它实施例互斥的独立的或备选的实施例。本领域技术人员显式地和隐式地理解的是,本文所描述的实施例可以与其它实施例相结合。在本技术说明书和所附权利要求书中使用的术语“和/或”是指相关联列出的项中的一个或多个的任何组合以及所有可能组合,并且包括这些组合。
197.本领域普通技术人员可以意识到,结合本文中所公开的实施例描述的各示例的单元及算法步骤,能够以电子硬件、计算机软件或者二者的结合来实现,为了清楚地说明硬件和软件的可互换性,在上述说明中已经按照功能一般性地描述了各示例的组成及步骤。专业技术人员可以对每个特定的应用来使用不同方法来实现所描述的功能,但是这种实现不应认为超出本技术的范围。
198.以上所揭露的仅为本技术较佳实施例而已,不能以此来限定本技术之权利范围,因此依本技术权利要求所作的等同变化,仍属本技术所涵盖的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1