蛋白质超图的构建方法、构建装置及设备与流程
1.本技术涉及计算机技术领域,具体涉及一种蛋白质超图的构建方法、一种蛋白质超图的构建装置、介质、设备及程序产品。
背景技术:
2.蛋白质是有氨基酸折叠盘曲而成的具有3d空间结构的生物大分子,其功能也是依赖于空间结构的。其中,蛋白质表示学习是蛋白质研究领域的一个重要分支,现有的蛋白质表示学习方法基于序列和图谱,例如基于图谱的蛋白质表示学习通过某种模型或方法,将图谱中的实体和关系转化为统一的某一维度的向量表示。然而用序列以及图谱来建模蛋白质在本质上忽略了在空间上多个氨基酸之间的相互作用关系,从而不能很好地表示蛋白质的高阶关系。
技术实现要素:
3.本技术实施例提供一种蛋白质超图的构建方法、一种蛋白质超图的构建装置、介质、设备及程序产品,使得生成的蛋白质超图可以较好地表示蛋白质中的高阶信息。
4.一方面,提供一种蛋白质超图的构建方法,包括:
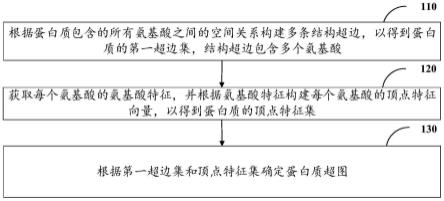
5.根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到所述蛋白质的第一超边集,所述结构超边包含多个氨基酸;
6.获取每个氨基酸的氨基酸特征,并根据所述氨基酸特征构建所述每个氨基酸的顶点特征向量,以得到所述蛋白质的顶点特征集;
7.根据所述第一超边集和所述顶点特征集确定蛋白质超图。
8.另一方面,提供一种蛋白质超图的构建装置,包括:
9.第一构建单元,用于根据蛋白质的多个氨基酸构建多条结构超边,以得到所述蛋白质的第一超边集,所述结构超边包含多个顶点氨基酸;
10.第二构建单元,用于获取每个顶点氨基酸的氨基酸特征,并根据所述氨基酸特征构建每个所述顶点氨基酸的顶点特征向量,以得到所述蛋白质的顶点特征集;
11.确定单元,用于根据所述第一超边集和所述顶点特征集确定蛋白质超图。
12.另一方面,提供一种计算机可读存储介质,计算机可读存储介质存储有计算机程序,计算机程序适于处理器进行加载,以执行如上任一实施例的方法中的步骤。
13.另一方面,提供一种计算机设备,计算机设备包括处理器和存储器,存储器中存储有计算机程序,处理器通过调用存储器中存储的计算机程序,用于执行如上任一实施例的方法中的步骤。
14.另一方面,提供一种计算机程序产品,包括计算机指令,计算机指令被处理器执行时实现如上任一实施例的方法中的步骤。
15.本技术实施例通过根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个氨基酸;获取每个氨基酸的氨基酸特
征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;根据第一超边集和顶点特征集确定蛋白质超图。本技术实施例提供的蛋白质超图的构建方法,将空间中多个氨基酸相互作用序列结构超边和空间结构超边构建了出来,并且还融入了顶点氨基酸的特征信息,使得构建的超图能够反映氨基酸之间的空间关系及氨基酸特征,在一定程度上可建模蛋白质的高阶信息。进一步地,将蛋白质建模成超图之后,可继续使用超图神经网络进行特征抽取,可以很好的得到将蛋白质的嵌入向量,用于下游任务的学习。
附图说明
16.为了更清楚地说明本技术实施例中的技术方法,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本技术的一些实施例,对于本领域技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
17.图1为本技术实施例提供的构建方法的流程示意图;
18.图2为本技术实施例提供的构建方法的示例图;
19.图3为本技术实施例提供的构建方法的另一流程示意图;
20.图4为本技术实施例提供的构建方法的另一流程示意图;
21.图5为本技术实施例提供的构建方法的另一示例图;
22.图6为本技术实施例提供的构建方法的另一流程示意图;
23.图7为本技术实施例提供的构建方法的另一示例图;
24.图8为本技术实施例提供的构建方法的另一流程示意图;
25.图9为本技术实施例提供的构建装置的结构示意图;
26.图10为本技术实施例提供的计算机设备的示意性结构图。
具体实施方式
27.下面将结合本技术实施例中的附图,对本技术实施例中的技术方法进行清楚、完整地描述,显然,所描述的实施例仅仅是本技术一部分实施例,而不是全部的实施例。基于本技术中的实施例,本领域技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本技术保护的范围。
28.首先,在对本技术实施例进行描述的过程中出现的部分名词或者术语作如下解释:
29.区块链系统:可以是由客户端、多个节点(接入网络中的任意形式的计算设备,如服务器、用户终端)通过网络通信的形式连接形成的分布式系统。节点之间形成组成的点对点(p2p,peer to peer)网络,p2p协议是一个运行在传输控制协议(tcp,transmission control protocol)协议之上的应用层协议,在分布式系统中,任何机器如服务器、终端都可以加入而成为节点,节点包括硬件层、中间层、操作系统层和应用层。
30.蛋白质(protein):是组成人体一切细胞、组织的重要成分。一般说,蛋白质约占人体全部质量的18%,最重要的还是其与生命现象有关。人体内蛋白质的种类很多,性质、功能各异,但都是由20种氨基酸(amino acid)按不同比例组合而成的,并在体内不断进行代谢与更新。蛋白质由一条或多条多肽链组成的生物大分子,每一条多肽链有二十~数百个
氨基酸残基不等;各种氨基酸残基按一定的顺序排列。产生蛋白质的细胞器是核糖体。
31.氨基酸(aminoacid):是构成蛋白质的基本单位,赋予蛋白质特定的分子结构形态,使他的分子具有生化活性。蛋白质是生物体内重要的活性分子,包括催化新陈代谢的酵素和酶。不同的氨基酸化学聚合成肽,一个蛋白质的原始片段,是蛋白质生成的前体。
32.超图(hypergraph):超图是图(graph)的一种泛化,不同与图上的边(一条边只连接2个顶点),超图的一条超边(hyperedge)可以连接多个顶点。
33.当前蛋白质表示都是基于序列或者基于简单图(graph),对应的学习蛋白质就是用深度学习里边的序列模型或者图模型。序列模型即将蛋白质中的氨基酸组合成的链作为蛋白质的表示,而图模型一般指的是将氨基酸建模成图的顶点,在一定距离内的两个氨基酸被认为有边。
34.本技术实施例提供了一种蛋白质超图的构建方法、一种蛋白质超图的构建装置、介质、设备及程序产品。本技术蛋白质超图的构建方法,将空间中多个氨基酸相互作用序列结构超边和空间结构超边构建了出来,并且还融入了顶点氨基酸的特征信息,使得构建的超图能够反映氨基酸之间的空间关系及氨基酸特征,在一定程度上可建模蛋白质的高阶信息。本技术实施例利用超图建模蛋白质的高阶信息,可应用于蛋白质表示学习领域。同时,一方面还可以应用于蛋白质分类,而这对于预测蛋白质功能非常重要,在生命科学领域具有重要意义。另一方面,可以应用于对已有的蛋白质预测好的空间结构进行打分,估计其与真正蛋白质的契合程度,模型学好之后可以用于蛋白质生成后的质量评估。
35.具体的,本技术实施例的方法可以由计算机设备执行,其中,该计算机设备可以为终端或者服务器等设备。
36.为更好地理解本技术实施例提供的技术方法,下面对本技术实施例提供的技术方法适用的应用场景做一些简单介绍,需要说明的是,以下介绍的应用场景仅用于说明本技术实施例而非限定。以构建方法由计算机设备执行为例,其中,该计算机设备可以为终端或者服务器等设备。
37.本技术实施例可结合云技术或区块链网络技术实现。如本技术实施例所公开的构建方法,其中这些数据可保存于区块链上,例如:结构超边、第一超边集、氨基酸特征、顶点特征向量、顶点特征集及蛋白质超图,均可保存于区块链上。
38.为了便于实现对结构超边、第一超边集、氨基酸特征、顶点特征向量、顶点特征集及蛋白质超图的存储和查询,可选的,该构建方法还包括:将结构超边、第一超边集、氨基酸特征、顶点特征向量、顶点特征集及蛋白质超图发送至区块链网络中,以使区块链网络的节点将结构超边、第一超边集、氨基酸特征、顶点特征向量、顶点特征集及蛋白质超图填充至新区块,且当对新区块取得共识一致时,将新区块追加至区块链的尾部。本技术实施例可以将结构超边、第一超边集、氨基酸特征、顶点特征向量、顶点特征集及蛋白质超图上链存储,实现记录的备份,当需要获取蛋白质超图时,可直接、快速地从区块链上获取相应的蛋白质超图,从而提高蛋白质超图构建方法的效率。
39.以下分别进行详细说明。需说明的是,以下实施例的描述顺序不作为对实施例优先顺序的限定。
40.本技术各实施例提供了一种蛋白质超图的构建方法,本技术实施例以构建方法由计算机设备为例来进行说明。
41.请参阅图1,图1为本技术实施例提供的蛋白质超图的构建方法的流程示意图,该方法包括:
42.步骤110:根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个氨基酸。
43.具体的,在蛋白质分子的空间结构中,所有蛋白质都是由20种不同氨基酸连接形成的多聚体,在形成蛋白质后,这些氨基酸又被称为残基。氨基酸是构成蛋白质的基本单位,它按不同的顺序和构型而组成不同的蛋白质。
44.其中,超图(hypergraph)为图(graph)的一种泛化,不同与图上的一条边只连接2个顶点,超图的一条超边(hyperedge)可以连接两个及两个以上的顶点。超边有多种构建方法,在一定程度上可以表征连接的多个顶点之间的关系。请参阅图2,图为超边的示例图,节点为n1-n8一共8个节点,e1、e2及e3为3条超边,例如,超边e1连接节点n2、n4及n8三个节点。
45.其中,氨基酸之间的空间关系包括氨基酸之间的距离关系、相邻氨基酸之间的空间关系等。
46.可选的,结构超边包括空间结构超边,根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集的步骤包括:
47.获取当前氨基酸,并计算当前氨基酸与蛋白质中的任意一个目标氨基酸之间的欧氏距离;
48.在欧氏距离在距离阈值范围内的情况下,将当前氨基酸和目标氨基酸确定为第一顶点氨基酸;
49.根据多个第一顶点氨基酸确定空间结构超边;
50.根据蛋白质的所有氨基酸确定的空间结构超边,确定第一超边集。
51.具体的,获取蛋白质分子,确定当前氨基酸,蛋白质中的其他氨基酸作为目标氨基酸。计算当前氨基酸与任一个目标氨基酸之间的欧氏距离,并与距离阈值进行比较,距离阈值为预先设定的阈值,例如4a0,6a0。
52.满足该条件的目标氨基酸可为一个或多个氨基酸:当计算得到的欧氏距离在距离阈值范围内,或者说,计算得到的欧氏距离小于或等于距离阈值时,目标氨基酸及当前氨基酸可确定为第一顶点氨基酸。同时,这些目标氨基酸及当前氨基酸确定一条空间结构超边,或者说,空间结构超边包括欧氏距离在距离阈值范围内的目标氨基酸及当前氨基酸。
53.遍历蛋白质中每一个氨基酸,每个氨基酸作为当前氨基酸,计算与任一个其他目标氨基酸的欧氏距离,得到每一个氨基酸的空间结构超边。当所有的氨基酸都确定好一条或多条空间结构超边后,根据蛋白质的所有氨基酸的空间结构超边确定第一超边集。第一超边集包括所有氨基酸的所有空间结构超边,以及每一条超边所包含的氨基酸。
54.空间结构超边可以表示为:
55.ei={vj|d(vi,vj)<d
cut_off
};
56.其中,ei表示为第i个氨基酸的超边,vj表示第j个氨基酸,d(vi,vj)表示第i个氨基酸与第j个氨基酸的欧式距离,d
cut_off
表示表示限定的距离阈值,d
cut_off
>0。
57.可选的,结构超边包括序列结构超边,根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集的步骤包括:
58.将相邻的k个氨基酸确定为第二顶点氨基酸,并进行连接以得到序列结构超边,其
中,k为整数;
59.根据蛋白质中的所有序列结构超边,和/或所有氨基酸确定的空间结构超边,确定第一超边集。
60.具体的,可将相邻的k个氨基酸连接起来作为一条序列结构超边,k个氨基酸作为第二顶点氨基酸。例如将相邻的5个氨基酸确定为第二顶点氨基酸,构成一条序列结构超边。
61.第i条序列结构超边可以表示为:ei={v
i+1
,v
i+2
,
…vi+k
},其中,v
i+1
表示为第i+1个氨基酸,即ei表示为将相邻的k个氨基酸连接在一起形成的第i条序列结构超边。
62.第一超边集可包括序列结构超边,或空间结构超边,或序列结构超边与空间结构超边共同形成第一超边集。
63.步骤120:获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;
64.可选的,顶点特征向量包括第一顶点特征向量,获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集的步骤,包括:
65.获取蛋白质中所有氨基酸的二级结构;
66.根据每个氨基酸的二级结构生成第一实数向量,以得到蛋白质的第一顶点特征向量,第一实数向量用于表征氨基酸的二级结构。
67.具体的,氨基酸具有3d空间结构信息,例如氨基酸的二级结构,二级结构可以通过已有的python工具包dssp,从存储蛋白质数据的pdb文件中获取,其中用到的方法为bio.pdb.dssp.dssp_dict_from_pdb_file()。
68.蛋白质的二级结构是氨基酸链中对应的每个氨基酸的结构,主要分为八类:
″g″
,
″h″
,
″i″
,
″
t
″
,
″e″
,
″b″
,
″s″
,
″
blank
″
。其中,h=α-helix,b=β-bridge,e=β-strand,g=3helix(3/10helix),i=5helix(pihelix),t=β-turn,s=bend,blank空值。
69.例如,根据每个氨基酸的二级结构生成7维的第一实数向量,包括生成one-hot向量,以得到蛋白质的第一顶点特征向量,第一实数向量用于表征氨基酸的二级结构。例如,常见的二级结构有hbet,则表示为one-hot形式的第一实数向量为{1,1,1,0,0,1,0,0}。
70.可选的,顶点特征包括第二顶点特征向量,获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集的步骤,还包括:
71.获取蛋白质的空间特征信息,空间特征信息包括蛋白质残基的相对可及表面积(rasa)、第一扭转角phi,及第二扭转角psi;
72.根据空间特征信息生成第二实数向量,以得到蛋白质的第二顶点特征向量。
73.具体的,蛋白质的空间特征信息包括蛋白质残基的相对可及表面积(rasa)、第一扭转角phi,及第二扭转角psi。
74.其中,可接触表面积(asa)或溶剂可接触表面积(sasa)是生物大分子可被溶剂接触的表面积。蛋白质残基的相对可及表面积(rasa)或相对溶剂可及性是衡量残基溶剂暴露的一种方法。它可以通过公式计算。
75.rasa=asa/maxasa;
76.其中,asa是溶剂可及的可接触表面积,maxasa是残留物可能的最大溶剂可及表面
积。
77.构成第一扭转角phi的四个原子是一个羰基碳、连接的α-碳、一个酰胺氮和下一个羰基碳(都标有绿色光环)。而构成第二扭转角psi的四个原子是一个酰胺氮、一个羰基碳、一个α-碳和第二个氮。
78.可以理解,四肽leu-leu-ile-tyr,可包含四个氨基酸,通过三个酰胺键或肽键连接在一起。由于酰胺键具有π键特性,构成肽键的六个原子都位于同一平面上,其中,第一平面是连接tyr和ile的肽键,第二平面连接ile和leu。与任何肽一样,骨架的构象由两个扭转角的值决定。按顺序,phi(φ)为c(i-),n(i),ca(i),c(i)扭转角,psi(ψ)为n(i),ca(i),c(i),n(i+1)扭转角。那么,第二平面用作测量两个角度的参考。则第三平面是tyr和ile(第四平面)之间的平面肽键的一部分,第四平面和第二平面之间的角度是psi。第一平面是ile和leu(第五平面)之间的平面肽键的一部分,第五平面和第二平面之间的夹角是phi。
79.计算蛋白质的空间特征信息rasa、phi及psi的数值,并根据rasa、phi及psi的数值生成第二实数向量,以得到蛋白质的3维的第二顶点特征向量。例如,第二顶点特征向量为{1.1000e+02,3.6000e+02,1.3190e+02}。
80.可选的,顶点特征包括第三顶点特征向量,获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集的步骤,还包括:
81.根据蛋白质的所有氨基酸生成第三实数向量,以得到蛋白质的第三顶点特征向量,第三实数向量用于表征蛋白质中包含的氨基酸种类。
82.具体的,组成生命体中的蛋白质的主要的氨基酸种类包括20种氨基酸:甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、甲硫氨酸(蛋氨酸)、脯氨酸、色氨酸、丝氨酸、酪氨酸、半胱氨酸、苯丙氨酸、天冬酰胺、谷氨酰胺、苏氨酸、天门冬氨酸、谷氨酸、赖氨酸、精氨酸和组氨酸。根据蛋白质中包含的所有氨基酸的种类生成第三实数向量,以得到蛋白质的第三顶点特征向量。
83.例如,使用one-hot编码方式对20种常见氨基酸进行表示,如下:
84.‘
a’:[1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0]
[0085]
‘
c’:[0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0]
[0086]
‘
d’:[0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0]
[0087]
‘
e’:[0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0]
[0088]
‘
f’:[0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0]
[0089]
‘
g’:[0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0]
[0090]
‘
h’:[0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0]
[0091]
‘
i’:[0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0]
[0092]
‘
k’:[0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0]
[0093]
‘
l’:[0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0]
[0094]
‘
m’:[0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0]
[0095]
‘
n’:[0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0]
[0096]
‘
p’:[0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0]
[0097]
‘
q’:[0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0]
[0098]
‘
r’:[0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0]
[0099]
‘
s’:[0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0]
[0100]
‘
t’:[0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0]
[0101]
‘v’
:[0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0]
[0102]
‘
w’:[0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0]
[0103]
‘
y’:[0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1]
[0104]
蛋白质的顶点特征向量可包括第一特征向量、第二特征向量、和第三特征向量连接在一起,所有特征向量一起,则每个顶点有29维向量。顶点特征矩阵x的维度则为|v|
×
29,其中v表示氨基酸。
[0105]
可选的,顶点特征包括第四顶点特征向量,方法还包括:
[0106]
根据蛋白质的每个氨基酸的结构,得到化学元素统计特性;
[0107]
根据每个氨基酸的化学元素统计特性生成第四顶点特征向量。
[0108]
具体的,根据蛋白质的每个氨基酸的结构,得到化学元素统计特性,其中化学元素统计特性可包括氨基酸包含的化学元素之间的关系,如氨基酸中苯环个数、羟基的个数等。将每个氨基酸的化学元素统计特性集合,以生成第四顶点特征向量。
[0109]
步骤130:根据第一超边集和顶点特征集确定蛋白质超图。
[0110]
具体的,当第一超边集和顶点特征集确定好后,可得到蛋白质超图,例如蛋白质超图h(v,e1)由顶点特征集v和第一超边集e1组成。蛋白质超图为一种通过多模态数据来表示蛋白质的方法,通过超边与顶点特征可以表征氨基酸之间的相互作用关系,即蛋白质中的高阶信息。
[0111]
将蛋白质建模成超图之后,可继续使用超图神经网络进行特征抽取,可以很好的得到将蛋白质的嵌入向量,用于下游任务的学习,譬如:蛋白质分类,蛋白质质量评估等等。
[0112]
如此,本技术通过根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个氨基酸;获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;根据第一超边集和顶点特征集确定蛋白质超图。本技术蛋白质超图的构建方法,将空间中多个氨基酸相互作用序列结构超边和空间结构超边构建了出来,并且还融入了顶点氨基酸的特征信息,使得构建的超图能够反映氨基酸之间的空间关系及氨基酸特征,在一定程度上可建模蛋白质的高阶信息。进一步地,将蛋白质建模成超图之后,可继续使用超图神经网络进行特征抽取,可以很好的得到将蛋白质的嵌入向量,用于下游任务的学习。
[0113]
请参阅图3,图为本技术实施例提供的蛋白质超图的另一种构建方法的流程示意图,该方法包括:
[0114]
步骤310,根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个氨基酸;
[0115]
具体实施方式同上述步骤110,此处不再展开赘述。
[0116]
步骤320,获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;
[0117]
具体实施方式同上述步骤120,此处不再展开赘述。
[0118]
步骤330,根据每个氨基酸的顶点特征向量之间的距离,构建特征超边;
[0119]
具体的,上述任一实施例得到的顶点特征向量,包括第一顶点特征向量、第二顶点
特征向量、第三顶点特征向量及第四顶点特征向量,计算各氨基酸顶点特征向量之间的距离,如欧式距离,将距离与预定的距离阈值进行比较,在距离阈值范围内的氨基酸确定为一条特征超边中的顶点,从而构建一条特征超边。
[0120]
步骤340,根据蛋白质的所有氨基酸确定的特征超边,确定第二超边集;
[0121]
步骤350,根据第一超边集、第二超边集和顶点特征集确定蛋白质超图。
[0122]
例如,将蛋白质中每个氨基酸的特征超边进行集合,得到蛋白质的第二超边集。根据第一超边集、第二超边集和顶点特征集确定蛋白质超图。例如蛋白质超图h(v,e1、e2)由顶点特征集v、第一超边集e1和第二超边集e2组成。
[0123]
如此,将蛋白质超图的超边设计扩展到顶点特征向量,可得到蛋白质更为丰富的高阶信息,提高蛋白质的表示效率。
[0124]
请参阅图4,图为本技术实施例提供的蛋白质超图的另一种构建方法的流程示意图,该方法包括:
[0125]
步骤410,根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个氨基酸;
[0126]
具体实施方式同上述步骤110,此处不再展开赘述。
[0127]
步骤420,获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;
[0128]
具体实施方式同上述步骤120,此处不再展开赘述。
[0129]
步骤430,根据每个氨基酸的顶点特征向量之间的距离,构建特征超边;
[0130]
步骤440,根据蛋白质的所有氨基酸确定的特征超边,确定第二超边集;
[0131]
步骤450,利用核函数对欧氏距离进行空间变换,以得到空间结构超边中每个第一顶点氨基酸的顶点权重,顶点权重用于表征第一顶点氨基酸与空间结构超边的连接强度;
[0132]
具体的,利用核函数将距离进行空间变换,以得到空间结构超边中每个第一顶点氨基酸的顶点权重,顶点权重q1(c,j)具体可通过如下公式得到:
[0133][0134]
其中,d(v,vc)2表示顶点v与vc的欧式距离。ej表示第j条超边,每条超边由多个顶点表示,例如序列结构超边ei={v
i+1
,v
i+2
,
…vi+k
},或空间结构超边:ei={vj|d(vi,vj)<d
cut_off
};
[0135]
表示顶点vc与其他所有顶点距离的平均值,可以表示为:
[0136][0137]
其中,v表示顶点的数量。
[0138]
步骤460,根据每个第一顶点氨基酸对应的顶点权重确定蛋白质的空间结构关联矩阵;
[0139]
当得到每个第一顶点氨基酸对应的顶点权重后,形成所有氨基酸的空间结构关联矩阵q∈r
|v|
×
|e|
,q为vxe维的矩阵。
[0140]
步骤470,根据第一超边集、第二超边集、顶点特征集及空间结构关联矩阵确定蛋
白质超图。
[0141]
例如,蛋白质超图h(v,e,q1)主要由顶点特征集v,第一超边集e1和第二超边集e2组成的超边集e,空间结构关联矩阵q1。
[0142]
请参阅图5,图为空间结构关联矩阵的表示图,q为空间结构关联矩阵,e1为空间结构关联超边1,e2为空间结构关联超边2,a、b、c、d和f为顶点氨基酸,其中,超边e1包括顶点氨基酸a、b、c、d,超边e2包括顶点氨基酸b、d和f,q(a)、q(b)、q(c)、q(d)和q(f)为顶点的顶点权重。
[0143]
如此,在蛋白质超图中增加空间结构关联矩阵,引入氨基酸的顶点权重,使得构建的超图能够反映氨基酸之间的空间关系。
[0144]
请参阅图6,图为本技术实施例提供的蛋白质超图的另一种构建方法的流程示意图,该方法包括:
[0145]
步骤610,根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个氨基酸;
[0146]
具体实施方式同上述步骤11θ,此处不再展开赘述。
[0147]
步骤620,获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;
[0148]
具体实施方式同上述步骤120,此处不再展开赘述。
[0149]
步骤630,根据每个氨基酸的顶点特征向量之间的距离,构建特征超边,并根据蛋白质的所有氨基酸确定的特征超边,确定第二超边集;
[0150]
具体实施方式同上述实施例,此处不再展开赘述。
[0151]
步骤640,利用核函数对欧氏距离进行空间变换,以得到空间结构超边中每个第一顶点氨基酸的顶点权重,并根据每个第一顶点氨基酸对应的顶点权重确定蛋白质的空间结构关联矩阵;
[0152]
具体实施方式同上述实施例,此处不再展开赘述。
[0153]
步骤650,根据多个序列结构超边确定序列结构关联矩阵;
[0154]
步骤660,将序列结构关联矩阵和空间结构关联矩阵进行拼接,以得到蛋白质的结构矩阵;
[0155]
将蛋白质中v个氨基酸的所有序列结构超边集合,得到vxv维的序列结构关联矩阵q2。将空间结构关联矩阵q1与序列结构关联矩阵q2进行拼接,得到结构矩阵q=[q1,q2]。
[0156]
步骤670,根据第一超边集、第二超边集、顶点特征集及结构矩阵确定蛋白质超图。
[0157]
蛋白质超图h(v,e,q)由顶点特征集v,第一超边集e1和第二超边集e2组成的超边集e,结构矩阵q=[q1,q2]。
[0158]
请参阅图7,图7纵坐标表示氨基酸数,横坐标表示超边数,图的左边部分框图为空间结构超边q1,右边框图为序列结构超边q2,图中q1中的白色点部分为顶点权重q1的位置表示,q2的白色线部分为序列结构关联矩阵q2的位置表示。
[0159]
请参阅图8,图为本技术实施例提供的蛋白质超图的另一种构建方法的流程示意图,该方法包括:
[0160]
步骤810,根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个氨基酸;
[0161]
具体实施方式同上述步骤110,此处不再展开赘述。
[0162]
步骤820,获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;
[0163]
具体实施方式同上述步骤120,此处不再展开赘述。
[0164]
步骤830,根据每个氨基酸的顶点特征向量之间的距离,构建特征超边,并根据蛋白质的所有氨基酸确定的特征超边,确定第二超边集;
[0165]
具体实施方式同上述实施例,此处不再展开赘述。
[0166]
步骤840,利用核函数对欧氏距离进行空间变换,以得到空间结构超边中每个第一顶点氨基酸的顶点权重,并根据每个第一顶点氨基酸对应的顶点权重确定蛋白质的空间结构关联矩阵;
[0167]
具体实施方式同上述实施例,此处不再展开赘述。
[0168]
步骤850,根据多个序列结构超边确定序列结构关联矩阵,并将序列结构关联矩阵和空间结构关联矩阵进行拼接,以得到蛋白质的结构矩阵;
[0169]
步骤860,根据第一超边集或第二超边集生成相应的边权对角矩阵,边权对角矩阵为单位矩阵;
[0170]
根据第一超边集或第二超边集生成相应的边权对角矩阵,边权对角矩阵为单位矩阵w,w与超边集的维度相同,例如w∈r
|e|
×
|e|
。
[0171]
步骤870,根据第一超边集、第二超边集、顶点特征集、结构关联矩阵及边权对角矩阵确定蛋白质超图;
[0172]
蛋白质超图h(v,e,q,w)由顶点特征集v,第一超边集e1和第二超边集e2组成的超边集e,结构矩阵q=[q1,q2],及边权对角矩阵w∈r
|e|
×
|e|
组成。
[0173]
步骤880,将蛋白质超图发送至超图神经网络,以对蛋白质进行表示学习。
[0174]
具体的,超图神经网络(hypergraph neural networks,hgnn)利用现有的超边卷积运算来处理表示学习过程中的数据相关性。通过超图神经网络,可以有效地利用超边卷积运算来进行传统的蛋白质超图学习。hgnn能够学习考虑到高阶数据结构的隐含层表示。hgnn首先将多模态的数据关系即蛋白质超图作为输入,将他们同时考虑进行超边的生成,将生成的超边组进行超图卷积得到最后的结果,根据下游任务的要求进行输出。
[0175]
需要说明的是,边权对角矩阵是根据hgnn增加的单位矩阵,蛋白质超图送入不同的网络,可根据网络的需求对蛋白质超图进行适应性调整为网络的输入格式。
[0176]
上述所有的技术方法,可以采用任意结合形成本技术的可选实施例,在此不再一一赘述。
[0177]
为便于更好的实施本技术实施例的蛋白质超图的构建方法,本技术实施例还提供一种蛋白质超图的构建装置。请参阅图9,图9为本技术实施例提供的蛋白质超图的构建装置的结构示意图。其中,该构建装置900可以包括:
[0178]
第一构建单元910,用于根据蛋白质的多个氨基酸构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个顶点氨基酸;
[0179]
第二构建单元920,用于获取每个顶点氨基酸的氨基酸特征,并根据氨基酸特征构建每个顶点氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;
[0180]
确定单元930,用于根据第一超边集和顶点特征集确定蛋白质超图。
[0181]
可选的,该构建装置900还可以包括:
[0182]
第三构建单元940,用于根据每个氨基酸的顶点特征向量之间的距离,构建特征超边;根据蛋白质的所有氨基酸确定的特征超边,确定第二超边集;确定单元930,还可以用于根据第一超边集、第二超边集和顶点特征集确定蛋白质超图。
[0183]
可选的,该构建装置900还可以包括:
[0184]
变换单元950,用于利用核函数对欧氏距离进行空间变换,以得到空间结构超边中每个第一顶点氨基酸的顶点权重,顶点权重用于表征第一顶点氨基酸与空间结构超边的连接强度;根据每个第一顶点氨基酸对应的顶点权重确定蛋白质的空间结构关联矩阵;
[0185]
相应地,确定单元930,用于根据第一超边集、第二超边集、顶点特征集及空间结构关联矩阵确定蛋白质超图。
[0186]
可选的,该构建装置900还可以包括:
[0187]
拼接单元960,用于根据多个序列结构超边确定序列结构关联矩阵;将序列结构关联矩阵和空间结构关联矩阵进行拼接,以得到蛋白质的结构矩阵;
[0188]
相应地,确定单元930,用于根据第一超边集、第二超边集、顶点特征集及结构矩阵确定蛋白质超图。
[0189]
可选的,该构建装置900还可以包括:
[0190]
生成单元970,用于根据第一超边集或第二超边集生成相应的边权对角矩阵,边权对角矩阵为单位矩阵;根据第一超边集、第二超边集、顶点特征集、结构关联矩阵及边权对角矩阵确定蛋白质超图;将蛋白质超图发送至超图神经网络,以对蛋白质进行表示学习。
[0191]
可选的,第一构建单元910,还可以用于获取当前氨基酸,并计算当前氨基酸与蛋白质中的任意一个目标氨基酸之间的欧氏距离;在欧氏距离在距离阈值范围内的情况下,将当前氨基酸和目标氨基酸确定为第一顶点氨基酸;根据多个第一顶点氨基酸确定空间结构超边;根据蛋白质的所有氨基酸确定的空间结构超边,确定第一超边集。
[0192]
可选的,第一构建单元910,还可以用于将相邻的k个氨基酸确定为第二顶点氨基酸,并进行连接以得到序列结构超边,其中,k为整数;根据蛋白质中的所有序列结构超边,和/或所有氨基酸确定的空间结构超边,确定第一超边集。
[0193]
可选的,第二构建单元920,还可以用于获取蛋白质中所有氨基酸的二级结构;根据每个氨基酸的二级结构生成第一实数向量,以得到蛋白质的第一顶点特征向量,第一实数向量用于表征氨基酸的二级结构。
[0194]
可选的,第二构建单元920,还可以用于获取蛋白质的空间特征信息,空间特征信息包括蛋白质残基的相对可及表面积(rasa)、第一扭转角phi,及第二扭转角psi;根据空间特征信息生成第二实数向量,以得到蛋白质的第二顶点特征向量。
[0195]
可选的,第二构建单元920,还可以用于根据蛋白质的所有氨基酸生成第三实数向量,以得到蛋白质的第三顶点特征向量,第三实数向量用于表征蛋白质中包含的氨基酸种类。
[0196]
可选的,第二构建单元920,还可以用于根据蛋白质的每个氨基酸的结构,得到化学元素统计特性;根据每个氨基酸的化学元素统计特性生成第四顶点特征向量。
[0197]
需要说明的是,本技术实施例中的构建装置900中各模块的功能可对应参考上述各方法实施例中任意实施例的具体实现方式,这里不再赘述。
[0198]
上述构建装置900中的各个单元可全部或部分通过软件、硬件及其组合来实现。上述各个单元可以以硬件形式内嵌于或独立于计算机设备中的处理器中,也可以以软件形式存储于计算机设备中的存储器中,以便于处理器调用执行上述各个单元对应的操作。
[0199]
构建装置900例如可以集成在具备储存器并安装有处理器而具有运算能力的终端或服务器中,或者该构建装置900为该终端或服务器。该终端可以为智能手机、平板电脑、笔记本电脑、智能电视、智能音箱、穿戴式智能设备、个人计算机(personal computer,pc)等设备,终端还可以包括客户端,该客户端可以是视频客户端、浏览器客户端或即时通信客户端等。服务器可以是独立的物理服务器,也可以是多个物理服务器构成的服务器集群或者分布式系统,还可以是提供云服务、云数据库、云计算、云函数、云存储、网络服务、云通信、中间件服务、域名服务、安全服务、内容分发网络(content delivery network,cdn)、以及大数据和人工智能平台等基础云计算服务的云服务器。
[0200]
图10为本技术实施例提供的计算机设备1000的示意性结构图,如图所示,计算机设备可以包括:通信接口1001,存储器1002,处理器1003和通信总线1004。通信接口1001,存储器1002,处理器1003通过通信总线1004实现相互间的通信。通信接口1001用于装置1000与外部设备进行数据通信。存储器1002可用于存储软件程序以及模块,处理器1003通过运行存储在存储器1002的软件程序以及模块,例如前述方法实施例中的相应操作的软件程序。
[0201]
可选的,该处理器1003可以调用存储在存储器1002的软件程序以及模块执行如下操作:
[0202]
根据蛋白质包含的所有氨基酸之间的空间关系构建多条结构超边,以得到蛋白质的第一超边集,结构超边包含多个氨基酸;
[0203]
获取每个氨基酸的氨基酸特征,并根据氨基酸特征构建每个氨基酸的顶点特征向量,以得到蛋白质的顶点特征集;
[0204]
根据第一超边集和顶点特征集确定蛋白质超图。
[0205]
可选的,计算机设备为该终端或服务器。该终端可以为智能手机、平板电脑、笔记本电脑、智能电视、智能音箱、穿戴式智能设备、个人计算机、游戏机、车载终端、智能电视等设备。该服务器可以是独立的物理服务器,也可以是多个物理服务器构成的服务器集群或者分布式系统,还可以是提供云服务、云数据库、云计算、云函数、云存储、网络服务、云通信、中间件服务、域名服务、安全服务、cdn、以及大数据和人工智能平台等基础云计算服务的云服务器。
[0206]
本技术还提供了一种计算机可读存储介质,用于存储计算机程序。该计算机可读存储介质可应用于计算机设备,并且该计算机程序使得计算机设备执行本技术实施例中的蛋白质超图的构建方法中的相应流程,为了简洁,在此不再赘述。
[0207]
本技术还提供了一种计算机程序产品,该计算机程序产品包括计算机指令,该计算机指令存储在计算机可读存储介质中。计算机设备的处理器从计算机可读存储介质读取该计算机指令,处理器执行该计算机指令,使得计算机设备执行本技术实施例中的蛋白质超图的构建方法中的相应流程,为了简洁,在此不再赘述。
[0208]
本技术还提供了一种计算机程序,该计算机程序包括计算机指令,计算机指令存储在计算机可读存储介质中。计算机设备的处理器从计算机可读存储介质读取该计算机指
令,处理器执行该计算机指令,使得计算机设备执行本技术实施例中的蛋白质超图的构建方法中的相应流程,为了简洁,在此不再赘述。
[0209]
应理解,本技术实施例的处理器可能是一种集成电路芯片,具有信号的处理能力。在实现过程中,上述方法实施例的各步骤可以通过处理器中的硬件的集成逻辑电路或者软件形式的指令完成。上述的处理器可以是通用处理器、数字信号处理器(digital signal processor,dsp)、专用集成电路(application specific integrated circuit,asic)、现成可编程门阵列(field programmable gate array,fpga)或者其他可编程逻辑器件、分立门或者晶体管逻辑器件、分立硬件组件。可以实现或者执行本技术实施例中的公开的各方法、步骤及逻辑框图。通用处理器可以是微处理器或者该处理器也可以是任何常规的处理器等。结合本技术实施例所公开的方法的步骤可以直接体现为硬件译码处理器执行完成,或者用译码处理器中的硬件及软件模块组合执行完成。软件模块可以位于随机存储器,闪存、只读存储器,可编程只读存储器或者电可擦写可编程存储器、寄存器等本领域成熟的存储介质中。该存储介质位于存储器,处理器读取存储器中的信息,结合其硬件完成上述方法的步骤。
[0210]
可以理解,本技术实施例中的存储器可以是易失性存储器或非易失性存储器,或可包括易失性和非易失性存储器两者。其中,非易失性存储器可以是只读存储器(read-only memory,rom)、可编程只读存储器(programmable rom,prom)、可擦除可编程只读存储器(erasable prom,eprom)、电可擦除可编程只读存储器(electrically eprom,eeprom)或闪存。易失性存储器可以是随机存取存储器(random access memory,ram),其用作外部高速缓存。通过示例性但不是限制性说明,许多形式的ram可用,例如静态随机存取存储器(static ram,sram)、动态随机存取存储器(dynamic ram,dram)、同步动态随机存取存储器(synchronous dram,sdram)、双倍数据速率同步动态随机存取存储器(double data rate sdram,ddr sdram)、增强型同步动态随机存取存储器(enhanced sdram,esdram)、同步连接动态随机存取存储器(synchlink dram,sldram)和直接内存总线随机存取存储器(direct rambus ram,dr ram)。应注意,本文描述的系统和方法的存储器旨在包括但不限于这些和任意其它适合类型的存储器。
[0211]
应理解,上述存储器为示例性但不是限制性说明,例如,本技术实施例中的存储器还可以是静态随机存取存储器(static ram,sram)、动态随机存取存储器(dynamic ram,dram)、同步动态随机存取存储器(synchronous dram,sdram)、双倍数据速率同步动态随机存取存储器(double data rate sdram,ddr sdram)、增强型同步动态随机存取存储器(enhanced sdram,esdram)、同步连接动态随机存取存储器(synch link dram,sldram)以及直接内存总线随机存取存储器(direct rambus ram,dr ram)等等。也就是说,本技术实施例中的存储器旨在包括但不限于这些和任意其它适合类型的存储器。
[0212]
本领域普通技术人员可以意识到,结合本文中所公开的实施例描述的各示例的单元及算法步骤,能够以电子硬件、或者计算机软件和电子硬件的结合来实现。这些功能究竟以硬件还是软件方式来执行,取决于技术方法的特定应用和设计约束条件。专业技术人员可以对每个特定的应用来使用不同方法来实现所描述的功能,但是这种实现不应认为超出本技术的范围。
[0213]
所属领域的技术人员可以清楚地了解到,为描述的方便和简洁,上述描述的系统、
装置和单元的具体工作过程,可以参考前述方法实施例中的对应过程,在此不再赘述。
[0214]
在本技术所提供的几个实施例中,应该理解到,所揭露的系统、装置和方法,可以通过其它的方式实现。例如,以上所描述的装置实施例仅仅是示意性的,例如,单元的划分,仅仅为一种逻辑功能划分,实际实现时可以有另外的划分方式,例如多个单元或组件可以结合或者可以集成到另一个系统,或一些特征可以忽略,或不执行。另一点,所显示或讨论的相互之间的耦合或直接耦合或通信连接可以是通过一些接口,装置或单元的间接耦合或通信连接,可以是电性,机械或其它的形式。
[0215]
所述作为分离部件说明的单元可以是或者也可以不是物理上分开的,作为单元显示的部件可以是或者也可以不是物理单元,即可以位于一个地方,或者也可以分布到多个网络单元上。可以根据实际的需要选择其中的部分或者全部单元来实现本实施例方法的目的。
[0216]
另外,在本技术实施例中的各功能单元可以集成在一个处理单元中,也可以是各个单元单独物理存在,也可以两个或两个以上单元集成在一个单元中。
[0217]
所述功能如果以软件功能单元的形式实现并作为独立的产品销售或使用时,可以存储在一个计算机可读取存储介质中。基于这样的理解,本技术的技术方法本质上或者说对现有技术做出贡献的部分或者该技术方法的部分可以以软件产品的形式体现出来,该计算机软件产品存储在一个存储介质中,包括若干指令用以使得一台计算机设备(可以是个人计算机,服务器)执行本技术各个实施例所述方法的全部或部分步骤。而前述的存储介质包括:u盘、移动硬盘、rom、ram、磁碟或者光盘等各种可以存储程序代码的介质。
[0218]
以上所述,仅为本技术的具体实施方式,但本技术的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本技术揭露的技术范围内,可轻易想到变化或替换,都应涵盖在本技术的保护范围之内。因此,本技术的保护范围应所述以权利要求的保护范围为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1